芝麻是世界上广泛种植的健康、特色油料作物,含油量高达55%[1],有“油料皇后”之称[2],我国已经有2 200多年的种植历史[3],至今依然是世界上重要的芝麻生产国和消费国,对全球芝麻产业的发展具有重要贡献[4]。据统计,近年来我国芝麻单产逐年增加,为提高芝麻种植效益奠定了扎实的经济基础。然而,机械化水平较低始终是困扰我国芝麻产业发展的瓶颈问题,特别是在农业劳动力不断减少情况下,这一问题更加突出。因此,培育适宜机械收获的新品种、提高机械化水平,成为当前芝麻育种的重要方向。
株高是芝麻的重要农艺性状,传统的芝麻品种多为高大型,对产量、抗倒伏能力、机械收获等有重要影响[5]。研究证明,适宜降低株高有利于提高农作物抗倒伏能力,对提高产量、改良品质有重要意义[6-8]。因此,芝麻株高相关的遗传分析、分子标记开发、QTL定位及候选基因挖掘等研究得以逐步开展。丁霞[3]研究发现,芝麻株高受1对(或2对主基因)+多基因控制或多基因控制,主茎始蒴高度和主茎果轴长度是影响株高的主要因素,并通过多态性标记筛选和遗传图谱构建,定位到26个株高相关QTL位点。朱晓凤[9]以芝麻重组自交系为试验材料,构建了一张包含344对引物的遗传图谱,利用不同软件对株高多年多点数据进行QTL定位,并对定位结果进行详细比较。Wei等[10]通过对705份芝麻材料测序,鉴定到5 407 981个SNP,关联到12个株高相关的高质量SNP标记,为挖掘株高相关基因提供了良好的研究基础。Zhang等[11]通过构建高密度SNP遗传图谱,对芝麻有限生长基因进行定位,并分析了不同突变体之间的基因差异,为深入分析芝麻顶端发育及株高的遗传控制奠定了研究基础。Wang等[12]通过构建高密度遗传图谱,在提高芝麻参考基因组的组装质量基础上,定位了41个株高相关QTL位点,为研究芝麻株型提供了更多的分子遗传学知识。Miao等[13]通过QTL定位、突变体分析、BSA测序等多种方法,发现了一个控制芝麻株高和节间长度的候选基因Sidwf1。已有研究为了解芝麻株高的分子遗传机制奠定了扎实的研究基础。然而,株高是一个复杂的数量性状,现有研究基础无法充分揭示其基因调控网络,也难以为适机采芝麻新品种的选育提供理论依据。
为了进一步挖掘株高相关基因,本研究以冀航芝1号为母本、DW607为父本构建F2群体,以株高为目标性状构建子代极端混池,利用BSA-seq方法筛选与目标性状关联的染色体区域,并基于参考基因组信息分析目标区域内的基因,通过KEGG、GO等数据库分析注释基因的功能。
1 材料和方法
1.1 供试材料
冀航芝1号由河北省农林科学院粮油作物研究所选育,于2010年通过鉴定。DW607由河南省农业科学院芝麻研究中心提供。
1.2 构建混池并测序
根据亲本及群体株高表型数据,构建2个亲本和2个极端株高混池,2个亲本各选10株取叶片,子代各选择高/矮极端株30株取叶片,采用CTAB法提取DNA,检测DNA浓度及质量后,将不同混池内单株的DNA分别等量混合,构建4个DNA混池。样品送至北京百迈克生物科技有限公司,通过Illumina HiSeq平台完成测序。亲本混池测序深度为10×,子代混池测序深度为30×。基于Wang等[14]公布的芝麻参考基因组信息,通过BWA[15]软件完成测序数据与参考基因组的比对,通过GATK[16]软件检测SNP标记,通过SnpEff[17]软件完成SNP和InDel的注释。
1.3 关联分析及候选基因注释
首先过滤以下低质量SNP/InDel:多个基因型的SNP/InDel,read支持度<4的SNP/InDel,混池之间基因型一致的SNP/InDel。最终获得高质量SNP/InDel。
通过ED(Euclidean Distance)和SNP-index两种方法进行关联分析[18],分析流程与张之昊等[19]相同。在进行ED方法分析时,本项目取原始ED的3次方作为关联值消除背景噪音。理论上,混池之间仅有目标性状相关的位点存在显著差异。因此,目标位点的ED值或ΔSNP-index较大,该位点与目标性状的关联越紧密,其他位点的ED值及ΔSNP-index趋向于0。
选择2种算法所得结果的交集作为候选区域,通过Blast[20]软件进行GO[21]、KEGG[22]等数据库注释分析。
通过Excel 2016分析数据的基本统计量。
2 结果与分析
2.1 群体表型数据呈现双向超亲分布,性状之间存在复杂的相关关系
本研究以冀航芝1号和DW607为亲本构建了F2群体,群体大小为410株。表1为群体性状基本统计量。可以看出,冀航芝1号的株高极显著大于DW607,F2群体的株高呈现双向超亲的近似正太分布,变异系数为9.90%。因此,试验群体可以用于进行BSA-seq分析。
表1 亲本及群体株高的基本统计量
Tab.1 Statistics of the parents and population plant height

冀航芝1号/cmJihangzhi 1DW607/cmF2 群体 F2 population最大值/cmMax最小值/cmMin平均值/cmAverage偏度Skew峰度Kurt变异系数/%CV127.5A87.5B164.4083.10134.98-0.810.989.90
注:A、B表示在0.01水平下,亲本之间差异达到极显著。
Note: A,B indicates the difference between the parents reached the 0.01 significance level.
2.2 测序数据量及数据质量
本研究通过Illumina HiSeq平台测序、过滤后,共得到41.71 Gb高质量数据,平均Q30达到91.11%,平均GC碱基含量36.93%,与参考基因组比对成功率平均为98.46%,对参考基因组的覆盖度平均为98.79(图1),亲本平均测序深度为10.5×,混池平均测序深度为40.5×(表2),插入片段大小呈单峰正态分布(图2)。由此可知,所得测序数据质量较好,可靠性较高。

R01.冀航芝1号;R02.DW607;R03.A池;R04.G池。图2-3同。
R01.Jihangzhi 1;R02.DW607;R03.A pool;R04.G pool.The same as Fig.2-3.
图1 样品测序数据对基因组的覆盖度
Fig.1 Coverage of the sequencing data on the reference genome
表2 测序后4个样品得到的数据量及数据质量
Tab.2 Data size and quality of the 4 sequencing datasets
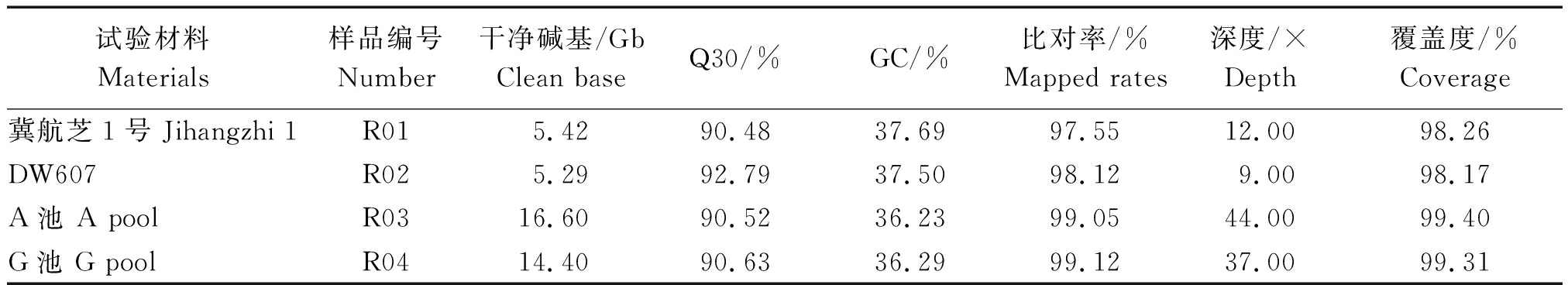
试验材料Materials样品编号Number干净碱基/GbClean base Q30/%GC/%比对率/%Mapped rates深度/×Depth 覆盖度/%Coverage冀航芝1号 Jihangzhi 1R015.4290.4837.6997.5512.0098.26DW607R025.2992.7937.5098.129.0098.17A池 A poolR0316.6090.5236.2399.0544.0099.40G池 G poolR0414.4090.6336.2999.1237.0099.31

图2 双末端测序后4个样品插入片段大小分布
Fig.2 Distribution of the insert segment size of the four paired-end sequenced samples
2.3 变异位点检测
基于高质量的测序数据,采用GATK软件进行变异位点检测,亲本之间共获得298 634个SNP和76 360个InDel,其中非同义突变的SNP共7 252个;混池之间共获得24 048个SNP和9 630个InDel,引起非同义突变的SNP共453个。样品间SNP和InDel标记的数量统计如图3所示。
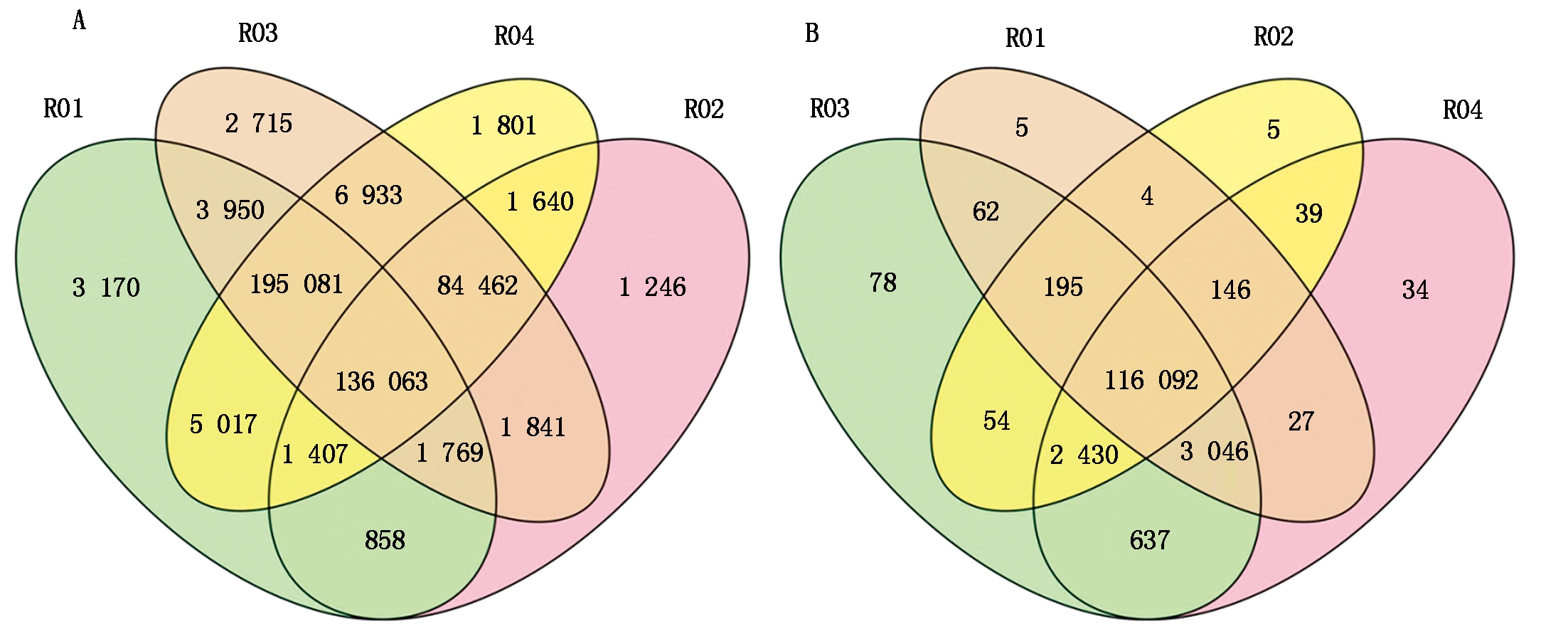
A.SNP数量统计;B.InDel数量统计。
A.The SNP statistics;B.The InDel statistics.
图3 样品间SNP及InDel标记统计数量
Fig.3 SNP and InDel marker statistics among samples
2.4 关联分析
2.4.1 基于SNP标记的关联分析 利用混池间的SNP标记,计算每个位点的ED值并进行乘方处理,取原始ED值的3次方作为关联值消除背景噪音,采用DISTANCE方法对ED值进行拟合,取所有位点拟合值的median+3s作为关联阈值,共关联到5个区域,总长度为6.53 Mb(表3)。同时,采用DISTANCE方法对ΔSNP-index进行拟合,在0.99置信度阈值下关联到3个区域,总长度为2.30 Mb(表4)。可以看出,基于ED方法的关联结果包含了基于ΔSNP-index方法的关联结果。
表3 基于SNP-ED方法关联到的染色体区域
Tab.3 Associated chromosome regions based on SNP-ED
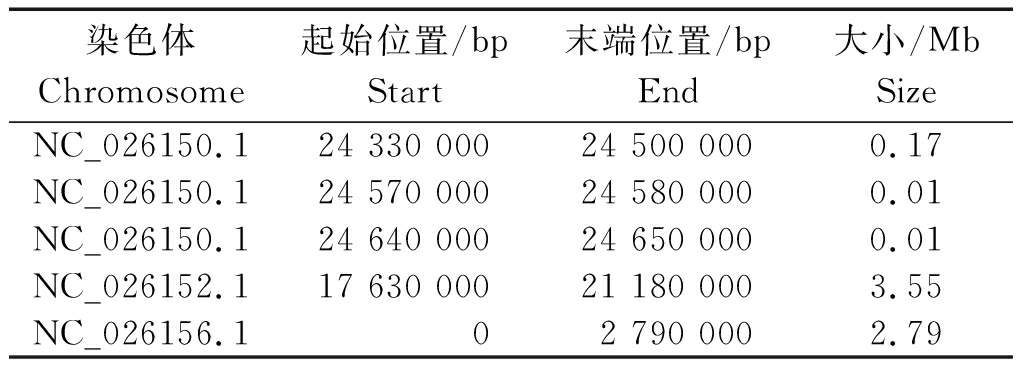
染色体Chromosome起始位置/bpStart 末端位置/bpEnd 大小/MbSize NC_026150.124 330 00024 500 0000.17NC_026150.124 570 00024 580 0000.01NC_026150.124 640 00024 650 0000.01NC_026152.117 630 00021 180 0003.55NC_026156.102 790 0002.79
表4 基于ΔSNP-index方法关联到的染色体区域
Tab.4 Associated chromosome regions based on ΔSNP-index
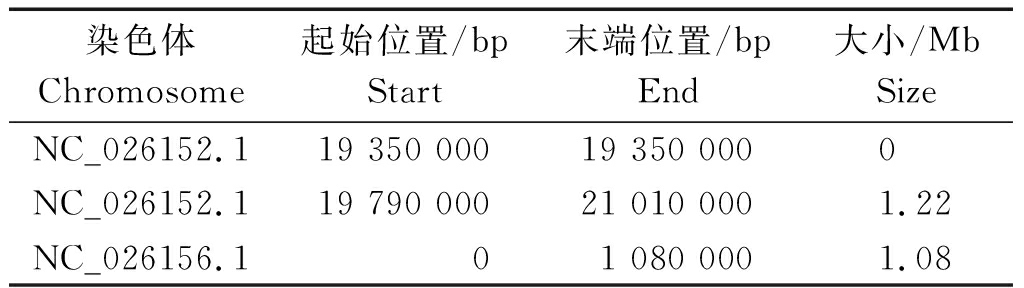
染色体Chromosome起始位置/bpStart 末端位置/bpEnd 大小/MbSize NC_026152.119 350 00019 350 0000 NC_026152.119 790 00021 010 0001.22NC_026156.101 080 0001.08
2.4.2 基于InDel标记的关联分析 利用混池间的InDel标记,以相同的方法进行关联。其中,通过ED方法共关联到5个区域,总长度为6.40 Mb(表5);通过ΔInDel-index方法关联到8个区域,总长度为2.25 Mb(表6)。可以看出,基于ED方法关联到的区域包含了基于ΔInDel-index方法关联到的区域。
表5 基于InDel-ED方法关联到的染色体区域
Tab.5 Associated chromosome regions based on InDel-ED
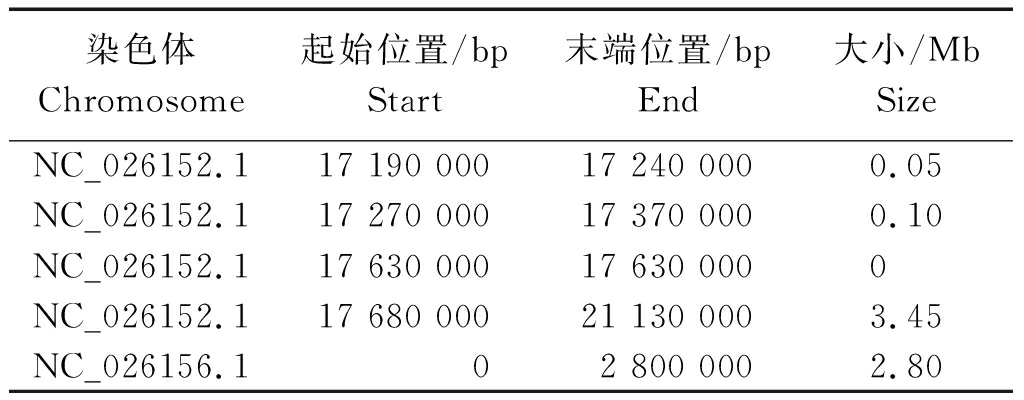
染色体Chromosome起始位置/bpStart 末端位置/bpEnd 大小/MbSize NC_026152.117 190 00017 240 0000.05NC_026152.117 270 00017 370 0000.10NC_026152.117 630 00017 630 0000NC_026152.117 680 00021 130 0003.45NC_026156.102 800 0002.80
表6 基于ΔInDel-index方法关联到的染色体区域
Tab.6 Associated chromosome regions based on ΔInDel-index
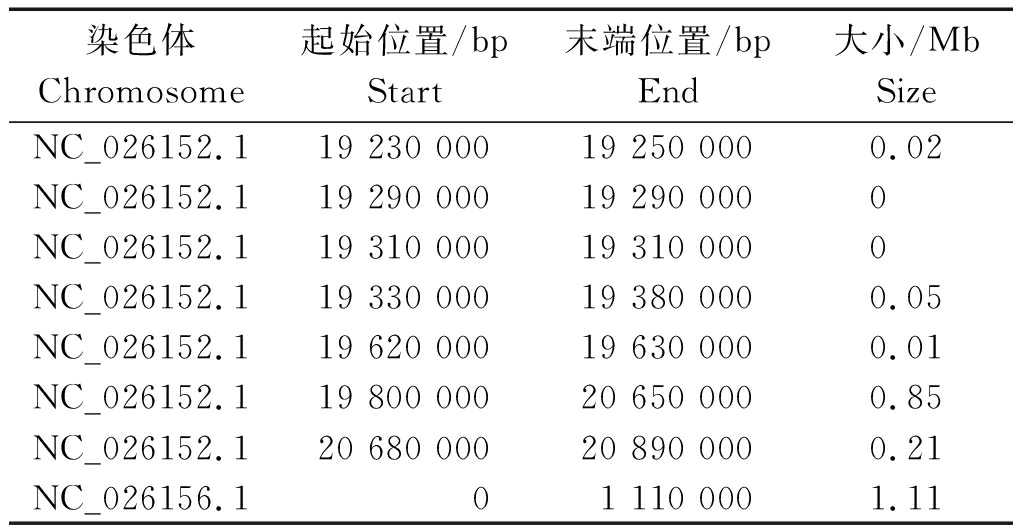
染色体Chromosome起始位置/bpStart 末端位置/bpEnd 大小/MbSize NC_026152.119 230 00019 250 0000.02NC_026152.119 290 00019 290 0000NC_026152.119 310 00019 310 0000NC_026152.119 330 00019 380 0000.05NC_026152.119 620 00019 630 0000.01NC_026152.119 800 00020 650 0000.85NC_026152.120 680 00020 890 0000.21NC_026156.101 110 0001.11
2.5 候选区域基因注释
对基于SNP和InDel关联到的区域取交集,共得到4个染色体区域,总长度为2.14 Mb(表7)。根据参考基因组信息,共注释到330个基因。KEGG代谢通路分析发现,注释基因比对到的前20个KEGG通路中,主要包括植物激素信号转导和能量代谢通路,其中有21个基因(SIN_1014691、SIN_1014702、SIN_1014697、SIN_1014704、SIN_1014699、SIN_1014700、SIN_1014709、SIN_1014693、SIN_1014707、SIN_1014695、SIN_1014705、SIN_1014692、SIN_1014703、SIN_1014694、SIN_1014701、SIN_1014696、SIN_1014512、SIN_1014698、SIN_1014689、SIN_1014688、SIN_1014708)显著富集到植物激素信号转导通路(图4)。GO富集结果显示,注释基因主要参与的前20个GO条目主要包括生长素响应、光合反应等生物过程、叶绿体、细胞壁等细胞组分以及葡萄糖跨膜转运活性等分子功能,其中富集到生长素响应过程的18个基因(SIN_1014691、SIN_1014702、SIN_1014697、SIN_1014704、SIN_1014515、SIN_1014686、SIN_1014709、SIN_1014707、SIN_1014699、SIN_1014692、SIN_1014703、SIN_1014694、SIN_1014701、SIN_1014512、SIN_1014695、SIN_1014821、SIN_1014688、SIN_1014708)可能是参与株高调控的关键基因(图5)。
表7 基于SNP和InDel 2种标记共同关联到的染色体区域
Tab.7 Common regions between SNP and InDel makers
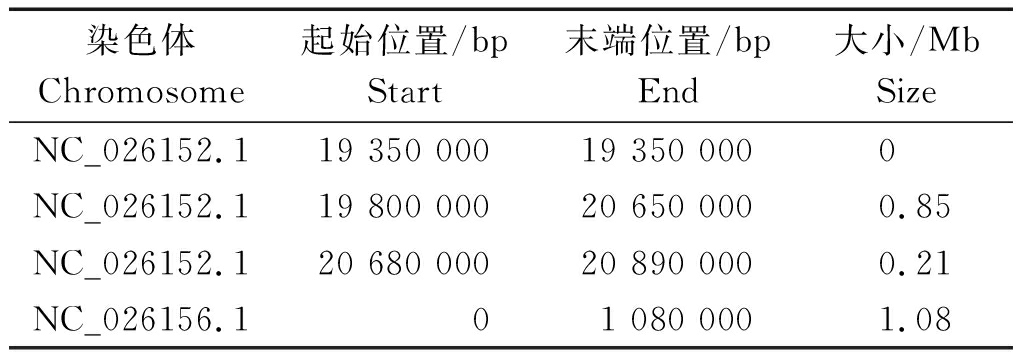
染色体Chromosome起始位置/bpStart 末端位置/bpEnd 大小/MbSize NC_026152.119 350 00019 350 0000 NC_026152.119 800 00020 650 0000.85NC_026152.120 680 00020 890 0000.21NC_026156.101 080 0001.08
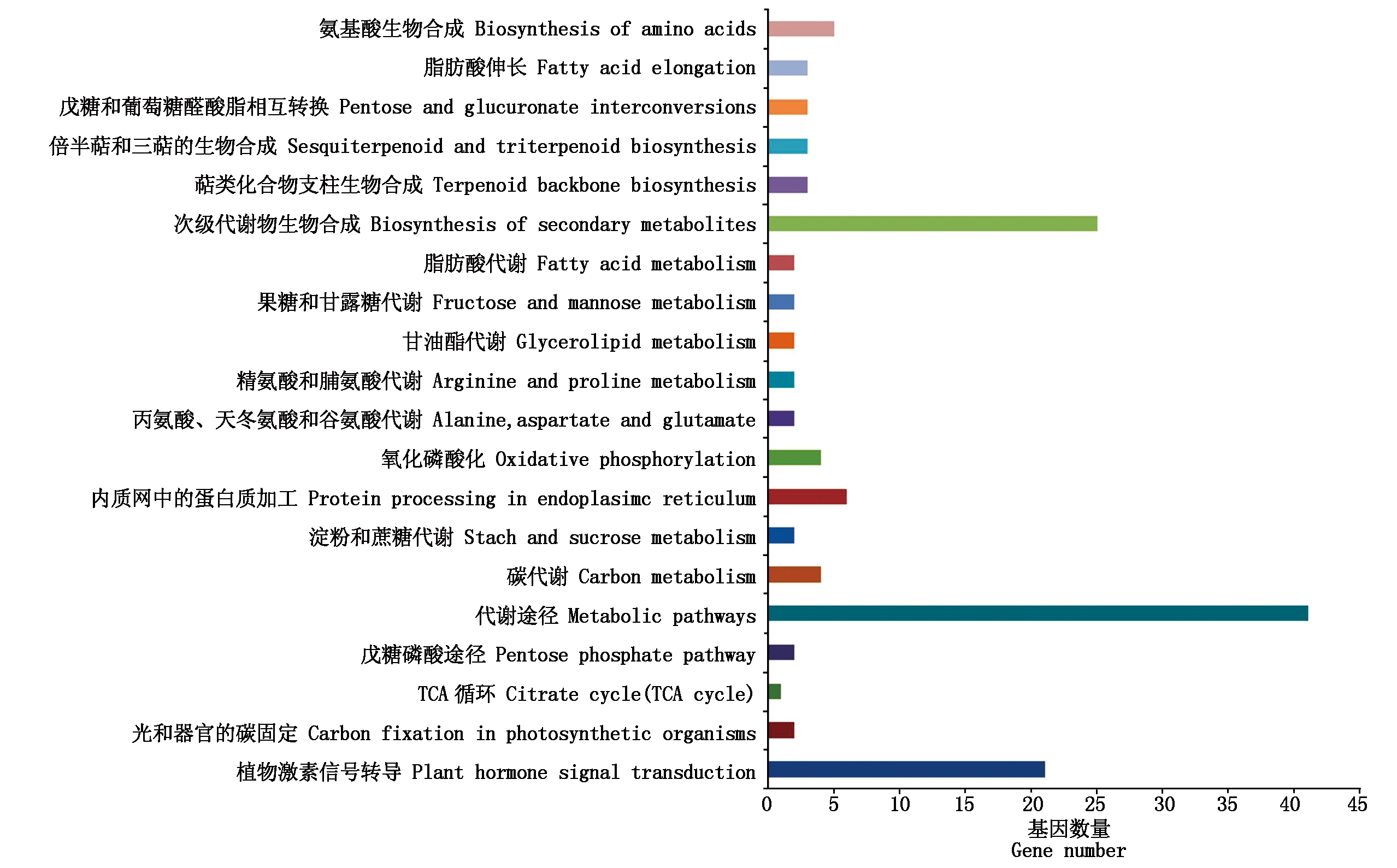
图4 注释基因KEGG通路分析
Fig.4 The mapped KEGG pathways of the annotated genes
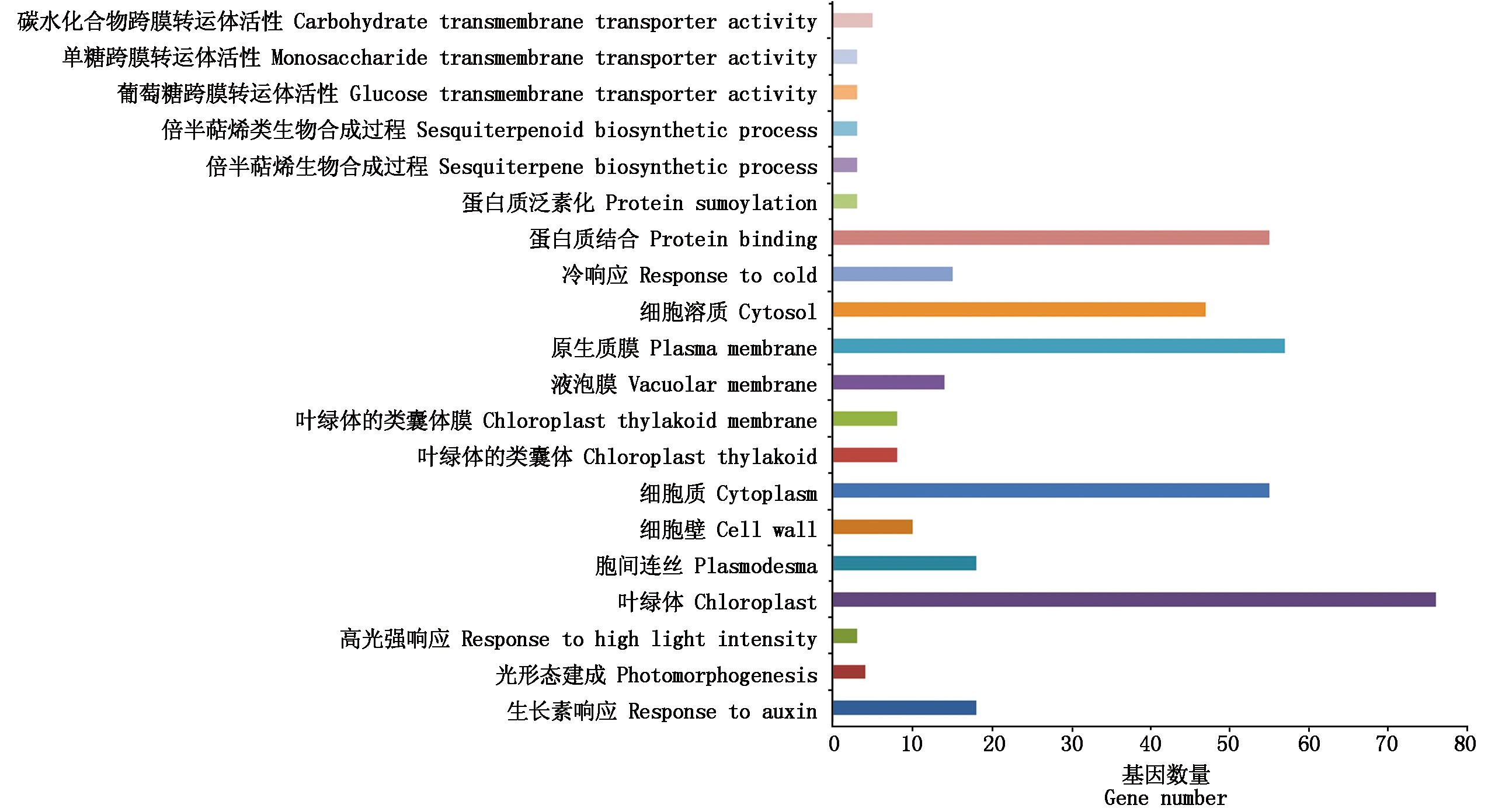
图5 注释基因的GO功能富集结果
Fig.5 GO enrichment results of the annotated genes
3 讨论
芝麻是我国四大油料作物之一,芝麻油的油酸和亚油酸总量高达85%,并且富含多种营养成分,具有很高的营养和保健价值[23]。传统的芝麻株型比较高大,不适宜现代农业的发展需求[5],特别是在全程机械化的发展趋势下,株高矮化成为新品种选育的重要内容。在水稻和小麦育种历史过程中,矮杆基因在产量提升方面的作用为作物育种带来了“绿色革命”[24]。因此,挖掘芝麻株高相关的基因信息,不仅能为了解芝麻株高的分子遗传机制奠定研究基础,还可以为培育高产品种提供理论指导。
本研究以冀航芝1号为母本、DW607为父本。其中,冀航芝1号株型高大,是当前河北省的主推品种;DW607植株较矮,且来源于不同的地理区域;所得F2群体的株高存在较大的变异范围,变异系数为9.90%,是进行株高相关基因挖掘的理想群体。丁霞等[5]以来源于我国8个省份的216份地方资源为材料,发现株高2 a的变异系数分别为11.81%,34.31%,高于本研究结果,可能是所用试验材料的来源更加广泛。BSA-seq技术是快速挖掘目标性状关键基因的有效方法,在黄瓜[25]、棉花[26]、玉米[27]、大麦[28]等作物中均有成功应用。在芝麻相关性状的研究中,Miao等[13]用芝麻株高突变体和野生型构建混池,通过BSA-seq技术得到了一个控制节间长度的基因Sidwf1,并发现赤霉素含量的变化与突变体表型吻合,该研究首次对芝麻株高和节间长度的分子遗传机制进行探讨;Liu等[29]通过BSA-seq技术对具有皱缩叶片性状的芝麻雄性不育材料进行基因定位,将目标区域缩小至219.7 kb,荧光定量实验发现了4个表达被抑制的基因。由此可知,BSA-seq技术同样适用于进行芝麻关键性状的基因挖掘工作。由于株高是复杂的数量性状,其分子遗传机制难以得到充分揭示。因此,本研究通过构建株高性状的极端混池,结合测序技术,以便快速获得芝麻株高相关的基因信息,加速其分子遗传机制研究的速度。通过SNP和InDel 2种标记进行关联,分别在NC_026152.1和NC_026156.1 2条连锁群上关联到3个和1个区域,总长度为2.14 Mb;根据基因组信息,共注释到330个基因。研究发现,植物激素在芝麻高/矮秆材料中存在显著差异,其中生长素和赤霉素含量与株高的关系最为密切[30]。本研究通过对注释的330个基因进行KEGG通路比对和GO功能富集分析,发现有21个基因参与植物激素信号转导通路、18个基因参与生长素响应过程,重叠的基因有15个,包括SIN_1014691、SIN_1014702、SIN_1014697、SIN_1014704、SIN_1014688、SIN_1014707、SIN_1014699、SIN_1014692、SIN_1014703、SIN_1014694、SIN_1014701、SIN_1014512、SIN_1014695、SIN_1014709和SIN_1014708,下一步将通过表达量分析、单倍型分析等途径,继续缩小候选基因范围、验证基因功能,深入研究芝麻株高的分子遗传机制,为适宜机械化的芝麻新品种培育奠定理论基础。
本研究通过对冀航芝1号和DW607的株高性状进行基因定位,在,NC_026152.1和NC_026156.1 2条连锁群上关联到4个区域,注释到330个基因,其中有18个基因参与生长素响应,为深入挖掘芝麻株高的分子遗传机制奠定研究基础。
[1] Wei W L,Zhang Y X,Lü H,Li D H,Wang L H,Zhang X R. Association analysis for quality traits in a diverse panel of Chinese sesame(Sesamum indicum L.)germplasm[J].Journal of Integrative Plant Biology,2013,55(8):745-758. doi:10.1111/jipb.12049.
[2] Johnson L A,Suleiman T M,Lusas E W. Sesame protein:A review and prospectus[J].Journal of the American Oil Chemists' Society,1979,56(3):463-468. doi:10.1007/BF02671542.
[3] 丁霞. 芝麻株高相关性状遗传分析和QTL定位[D].北京:中国农业科学院,2013.
Ding X. Inheritance analysis and QTL mapping on plant height related traits in sesame(Sesamum indicum L.)[D].Beijing:Chinese Academy of Agricultural Sciences,2013.
[4] 王瑞元. 中国为全球芝麻产业的发展作出了重要贡献[J].中国油脂,2019,44(12):1-2. doi:10.3969/j.issn.1003-7969.2019.12.001.
Wang R Y. China has made important contributions to the development of global sesame industry[J].China Oils and Fats,2019,44(12):1-2.
[5] 丁霞,王林海,张艳欣,黎冬华,高媛,危文亮,王蕾,张秀荣. 芝麻核心种质株高构成相关性状的遗传变异及关联定位[J].中国油料作物学报,2013,35(3):262-270. doi:10.7505/j.issn.1007-9084.2013.03.006.
Ding X,Wang L H,Zhang Y X,Li D H,Gao Y,Wei W L,Wang L,Zhang X R. Genetic variation and associated mapping for traits related to plant height constitutions in core collections of sesame(Sesamum indicum L.)[J].Chinese Journal of Oil Crop Sciences,2013,35(3):262-270.
[6] 钟明志,魏淑红,彭正松,杨在君. 小麦Rht矮秆基因研究和应用综述[J].分子植物育种,2018,16(20):6670-6677. doi:10.13271/j.mpb.016.006670.
Zhong M Z,Wei S H,Peng Z S,Yang Z J. A review of the research and application of Rht dwarf genes in wheat[J].Molecular Plant Breeding,2018,16(20):6670-6677.
[7] Liu F,Wang P D,Zhang X B,Li X F,Yan X H,Fu D H,Wu G. The genetic and molecular basis of crop height based on a rice model[J].Planta,2018,247(1):1-26. doi:10.1007/s00425-017-2798-1.
[8] Sakamoto T,Matsuoka M. Generating high-yielding varieties by genetic manipulation of plant architecture[J].Current Opinion in Biotechnology,2004,15(2):144-147. doi:10.1016/j.copbio.2004.02.003.
[9] 朱晓凤. 芝麻遗传图谱构建和株高相关性状QTL分析[D].北京:中国农业科学院,2015.doi:10.7666/d.Y2787588.
Zhu X F. Construction of genetic linkage map and QTL analysis for plant height associated traits in sesame(Sesame indicum L.)[D].Beijing:Chinese Academy of Agricultural Sciences,2015.
[10] Wei X,Liu K Y,Zhang Y X,Feng Q,Wang L H,Zhao Y,Li D H,Zhao Q,Zhu X D,Zhu X F,Li W J,Fan D L,Gao Y,Lu Y Q,Zhang X M,Tang X M,Zhou C C,Zhu C R,Liu L F,Zhong R C,Tian Q L,Wen Z R,Weng Q J,Han B,Huang X H,Zhang X R. Genetic discovery for oil production and quality in sesame[J].Nature Communications,2016,6(1):1-10. doi:10.1038/ncomms9609.
[11] Zhang H Y,Miao H M,Li C,Wei L B,Duan Y H,Ma Q,Kong J J,Xu F F,Chang S X. Ultra-dense SNP genetic map construction and identification of SiDt gene controlling the determinate growth habit in Sesamum indicum L.[J].Scientific Reports,2016,6:31556. doi:10.1038/srep31556.
[12] Wang L H,Xia Q J,Zhang Y X,Zhu X D,Zhu X F,Li D H,Ni X M,Gao Y,Xiang H T,Wei X,Yu J Y,Quan Z W,Zhang X R. Updated sesame genome assembly and fine mapping of plant height and seed coat color QTLs using a new high-density genetic map[J].BMC Genomics,2016,17:31. doi:10.1186/s12864-015-2316-4.
[13] Miao H M,Li C,Duan Y H,Wei L B,Ju M,Zhang H Y. Identification of a Sidwf1 gene controlling short internode length trait in the sesame dwarf mutant dw607[J].Theoretical and Applied Genetics,2020,133(1):73-86. doi:10.1007/s00122-019-03441-x.
[14] Wang L H,Yu S,Tong C B,Zhao Y Z,Liu Y,Song C,Zhang Y X,Zhang X D,Wang Y,Hua W,Li D H,Li D,LiF,Yu J Y,Xu C Y,Han X L,Huang S M,Tai S H,Wang J Y,Xu X,Li Y R,Liu S Y,Varshney R K,Wang J,Zhang X R. Genome sequencing of the high oil crop sesame provides insight into oil biosynthesis[J].Genome Biology,2014,15(2):39.doi:10.1186/gb-2014-15-2-r39.
[15] Li H,Durbin R. Fast and accurate long read alignment with Burrows-Wheeler transform[J].Bioinformatics,2010,26(5):1754-1760. doi:10.1093/bioinformatics/btp324.
[16] McKenna A,Hanna M,Banks E,Sivachenko A,Cibulskis K,Kernytsky A,Garimella K,Altshuler D,Gabriel S,Daly M,DePristo M A. The Genome Analysis Toolkit:A MapReduce framework for analyzing next-generation DNA sequencing data[J].Genome Research,2010,20(9):1297-1303. doi:10.1101/gr.107524.110.
[17] Cingolani P,Platts A,Wang L L,Coon M,Nguyen T,Wang L,Land S J,Lu X Y,Ruden D M. A program for annotating and predicting the effects of single nucleotide polymorphisms,SnpEff[J].Fly,2012,6(2):80-92. doi:10.4161/fly.19695.
[18] Hill J T,Demarest B L,Bisgrove B W,Gorsi B,Su Y C,Yost H J. MMAPPR:mutation mapping analysis pipeline for pooled RNA-seq[J].Genome Research,2013,23(4):687-697. doi:10.1101/gr.146936.112.
[19] 张之昊,王俊,刘章雄,邱丽娟. 基于BSA-Seq技术挖掘大豆中黄622的多小叶基因[J].作物学报,2020,46(12):1839-1849. doi:10.3724/SP.J.1006.2020.04075.
Zhang Z H,Wang J,Liu Z X,Qiu L J. Mapping of an incomplete dominant gene controlling multifoliolate leaf by BSA-seq in soybean(Glycine max L.)[J].Acta Agronomica Sinica,2020,46(12):1839-1849.
[20] Altschul S F,Madden T L,Schäffer A A,Zhang J H,Zhang Z,Miller W,Lipman D J. Gapped BLAST and PSI-BLAST:A new generation of protein database search programs[J].Nucleic Acids Research,1997,25(17):3389-3402. doi:10.1093/nar/25.17.3389.
[21] Ashburner M,Ball C A,Blake J A,Botstein D,Butler H,Cherry J M,Davis A P,Dolinski K,Dwight S S,Eppig J T,Harris M A,Hill D P,Issel-Tarver L,Kasarskis A,Lewis S,Matese J C,Richardson J E,Ringwald M,Rubin G M,Sherlock G. Gene ontology:Tool for the unification of biology[J].Nature Genetics,2000,25(1):25-29. doi:10.1038/75556.
[22] Kanehisa M,Goto S,Kawashima S,Okuno Y,Hattori M. The KEGG resource for deciphering the genome[J].Nucleic Acids Research,2004,32(Database issue):D277-D280. doi:10.1093/nar/gkh063.
[23] Anilakumar K.R,Pal A,Khanum F,Bawa A S. Nutritional,medicinal and industrial uses of sesame(Sesamum indicum L.)seeds-an overview[J].Agriculturae Conspectus Scientificus,2010. 75(4):159-168. doi:10.1083/jcb.77.3.685.
[24] Hedden P. The genes of the green revolution[J].Trends in Genetics,2003,19(1):5-9. doi:10.1016/S0168-9525(02)00009-4.
[25] Win K T,Vegas J,Zhang C Y,Song K,Lee S. QTL mapping for downy mildew resistance in cucumber via bulked segregant analysis using next-generation sequencing and conventional methods[J].Theoretical and Applied Genetics,2017,130(1):199-211. doi:10.1007/s00122-016-2806-z.
[26] Chen W,Yao J B,Chu L,Yuan Z W,Li Y,Zhang Y S. Genetic mapping of the nulliplex-branch gene(gb_nb1)in cotton using next-generation sequencing[J].Theoretical and Applied Genetics,2015,128(3):539-547. doi:10.1007/s00122-014-2452-2.
[27] Klein H,Xiao Y G,Conklin P A,Govindarajulu R,Kelly J A,Scanlon M J,Whipple C J,Bartlett M. Bulked-segregant analysis coupled to whole genome sequencing(BSA-seq)for rapid gene cloning in maize[J].G3,2018,8(11):3583-3592. doi:10.1534/g3.118.200499.
[28] Xu D D,Sun D,Diao Y L,Liu M X,Gao J,Wu B,Yuan X M,Lu P,Zhang Z W,Zhang J,Guo G G. Fast mapping of a chlorophyll b synthesis-deficiency gene in barley(Hordeum vulgare L.)via bulked-segregant analysis with reduced-representation sequencing[J].The Crop Journal,2019,7(1):58-64.doi:10.1016/j.cj.2018.07.002.
[29] Liu H Y,Zhou F,Zhou T,Yang Y X,Zhao Y Z. Fine mapping of a novel male-sterile mutant showing wrinkled-leaf in sesame by BSA-Seq technology[J]. Industrial Crops and Products,2020,156:112862.doi:10.1016/j.indcrop.2020.112862.
[30] 朱晓凤,黎冬华,王林海,张艳欣,高媛,魏鑫,张秀荣.矮秆与高秆芝麻株高建成中内源激素含量变化比较分析[J].中国油料作物学报,2015,37(1):83-89.doi:10.7505/j.issn.1007-9084.2015.01.013.
Zhu X F,Li D H,Wang L H,Zhang Y X,Gao Y,Wei X,Zhang X R. Phytohormone change during plant height development between dwarfs and high genotypes of sesame[J].Chinese Journal of Oil Crop Sciences,2015,37(1):83-89.
