养猪场中猪病频发导致的药物及抗生素滥用引发了一系列的药物残留及食品安全问题。因此,发掘抗病性状相关的分子标记,通过分子选育从遗传本质上提高猪群的抗病力,是从长远角度上解决药物残留及猪肉食品安全的有效手段之一[1-2]。我国拥有丰富的种猪资源,这些地方猪种具有许多优良的种质特性,其中最为突出的特性之一就是抗病性强。因此,研究地方猪优质抗病性状的分子调控机理,挖掘和鉴定调控猪免疫性状的候选基因对于猪抗病力的遗传改良具有重要意义。
转录组是特定组织或细胞在某种特定环境或生理条件下表达的所有RNA的总和,是连接基因组遗传信息与蛋白质组生物功能的纽带[3]。近年来,得益于高通量测序技术的快速发展和成熟,RNA-Seq技术(RNA sequencing,RNA-Seq)在畜禽产奶[4] 、肉质[5-6]、产蛋[7]等重要经济性状的分子机理研究中得到了广泛应用。利用 RNA-Seq技术,研究者也开展了一些猪抗病性状候选基因的挖掘。Li等[8]研究了莱芜猪、杂交(约克×长白)2个品种猪的肺组织在猪2型圆环病毒应答中的转录组差异,筛选到1个可能抑制肺部过度炎症反应、减轻肺部病理程度的重要候选基因。Ni等[9]比较了猪肺炎支原体感染条件下,江曲海猪和杜洛克猪肺组织之间的转录组差异,在2个猪种中分别筛选出2 250,3 526个差异表达基因,其中,共有的差异表达基因有1 669个。Nascimento等[10]比较分析了感染伊利诺伊州型支原体猪与未感染猪的全血中转录组的差异,发现55个差异表达基因。Zhang等[11]研究了猪急性腹泻综合症病毒感染肠道上皮细胞时细胞的转录组变化,挖掘到1 897个差异表达基因。Zhang等[12]研究了口蹄疫毒感染猪肾PK-15细胞时细胞的转录组变化情况,挖掘到1 212个差异表达基因。上述研究结果为从转录组层面阐明猪抗病性状的遗传机制奠定了基础。但是,这些研究均局限在某种特定疾病的研究上,并且现有研究对象也仅局限在极少数中国地方品种,大量地方猪品种优良的抗病性状的遗传机理仍需进一步深入研究。
普遍认为,中国地方猪种在抗病抗逆性状上表现普遍比外国猪种优秀,荣昌猪等中国地方猪种的抗病能力显著高于长白等外种猪,但目前依然缺乏相应的生理生化指标作为证据[13]。从猪的生命周期来看,对除种公猪种母猪以外的大多数商品猪而言,其生命周期是极其短暂的,在其免疫系统尚未发育成熟就已经完成了它的生产周期,因此,先天免疫系统对猪的抗病能力来说是至关重要的。胸腺是机体中一个极其重要的中枢淋巴器官,与B淋巴细胞、T淋巴细胞的发生、发育、成熟紧密相关[14],在机体的先天免疫中发挥着重要功能。因此,本研究拟选用荣昌猪(中国地方猪种)和长白猪(外种猪)为对象,利用RNA-seq技术比较两者胸腺组织中的差异表达基因,同时对筛选得到的差异表达基因进行GO和KEGG富集分析,并通过qRT-PCR验证RNA-seq结果的准确性,挖掘和鉴定控制荣昌猪免疫性状的候选基因。研究结果可为地方猪抗病性状的遗传机理研究提供基础数据。
1 材料和方法
1.1 试验材料
试验用出生1 d荣昌猪及长白猪由重庆市荣昌区国家级荣昌猪保种场提供;RNeasy Mini Kit购自Qiagen公司;Reverse Transcription system及GoTaq® qPCR Master Mix购自Promega公司,其他试剂均为国产分析纯。
1.2 试验方法
1.2.1 胸腺RNA提取、文库构建、测序及数据的质量控制 使用RNeasy Mini Kit提取荣昌猪和长白猪胸腺组织的总RNA,详细步骤参照试剂盒说明书。利用NanoDrop 2000测定RNA的浓度,1%琼脂糖凝胶电泳检测RNA的完整性。RNA进一步质检、cDNA文库构建、测序及测序数据的质量控制由北京诺禾致源生物信息科技有限公司完成。
1.2.2 测序数据拼接、组装 利用Bowtie建立参考基因组索引,通过Tophat 2对过滤后的测序序列进行基因组定位分析,其中mismatch设置为2,其余选用默认设置。利用HTSeq计算比对到每个基因上的reads数。
1.2.3 差异表达基因的筛选及分析 利用Deseq进行差异基因表达分析,用RPKM(每百万reads中来自某一基因每千碱基长度的reads数目)算法对转录本的表达量进行标准化,以|log2(Fold change)| > 1、P<0.05为标准筛选差异表达基因。利用DAVID软件对差异表达基因进行GO和KEGG功能富集分析,以P<0.05为显著富集条目。
1.2.4 RNA-seq结果的验证 本研究随机选取6个差异表达显著的基因,通过RT-qPCR对RNA-seq结果的准确性进行验证。首先,参考NCBI数据库中该基因的mRNA序列,以β-actin为内参,利用Primer 5软件设计RT-qPCR引物(表1)。随后,选取与RNA-seq分析用相同的组织样品,利用RNeasy Mini Kit提取总RNA,Transcription system将RNA反转录成cDNA后,利用GoTaq® qPCR Master Mix进行RT-qPCR。反转录分2步进行,第1步反应体系如下:Radom Primer 1 μL,Oligo dT 1μL,RNA 1 μg,加RNase free H2O至10 μL。反应条件为70 ℃ 5 min;第2步反应体系如下:Reaction Buffer 4 μL,MgCl2 2 μL,PCR nucleotide Mix 1 μL,RNase inhibitor 0.4 μL,RNase free H2O 1.6 μL,反转录酶1 μL。反应条件为:25 ℃ 5 min,42 ℃ 60 min,70 ℃ 15 min。实时荧光定量PCR反应体系如下:2 ×GoTaq® qPCR Master Mix 10 μL,Forward primer 0.4 μL,Reverse primer 0.4 μL,CXR 0.2 μL,cDNA 0.5 μL,H2O 8.5 μL。反应条件为:95 ℃ 10 min;95 ℃ 15 s,60 ℃ 30 s,40个循环。每个样品设置3个重复。溶解曲线采用仪器默认设置。基因的相对表达量采用2-ΔΔCT方法进行计算。RNA-seq结果用RPKM值表示,RT-qPCR结果用相对表达量表示。通过计算验证基因的RPKM值与相对表达量两者间相关系数评价RNA-seq与RT-qPCR结果的一致性。
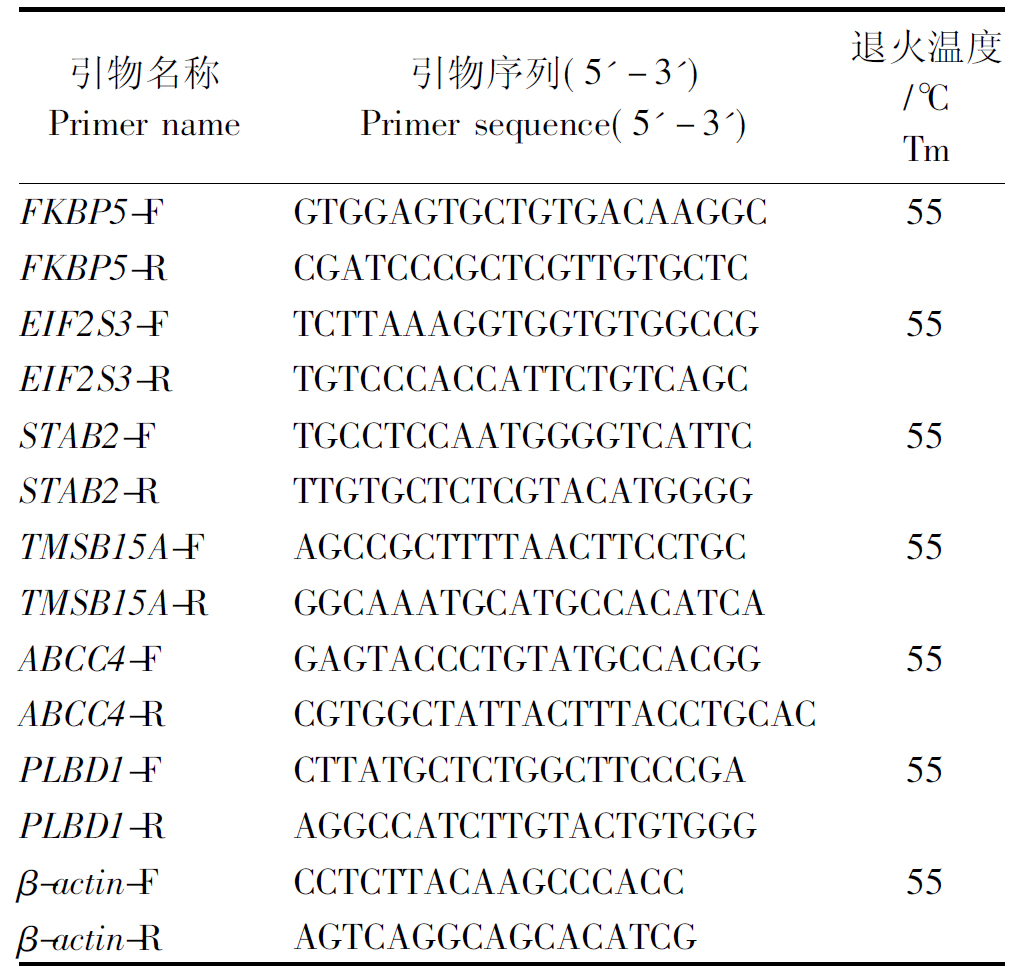
表1 RT-qPCR扩增用引物信息
Tab.1 Primer information for RT-qPCR
2 结果与分析
2.1 测序数据质量评估、拼接和组装
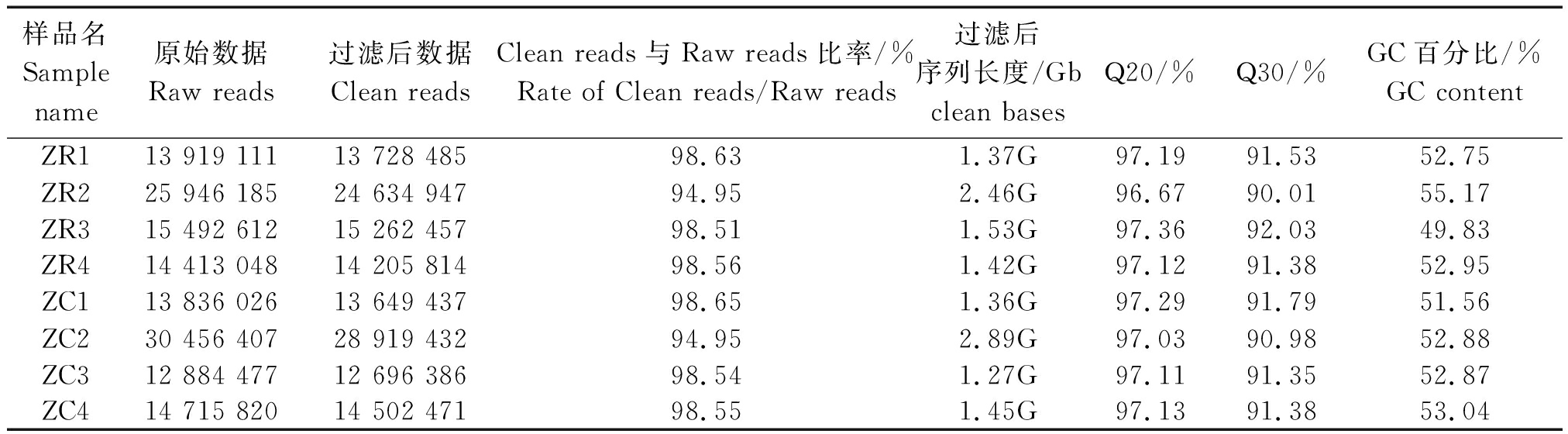
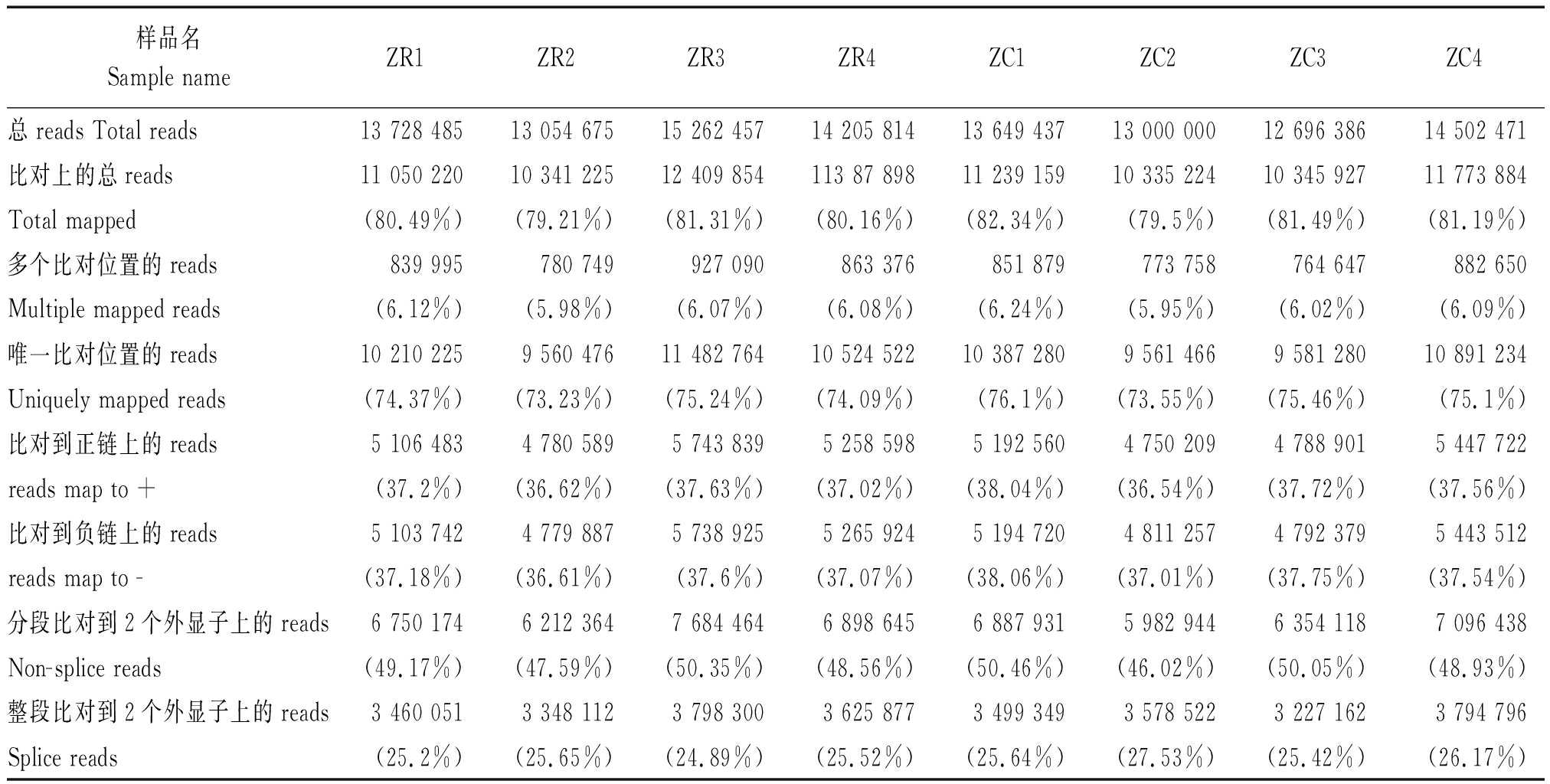
为了筛选导致初生荣昌猪、长白猪先天免疫功能差异的候选基因,本研究对荣昌猪和长白猪胸腺组织进行了RNA-seq。此次测序共计获得了24 Gb的碱基总数,序列拼接后,共获得1.42亿条reads,筛选过滤后的Clean reads 1.38亿条。样品的Clean reads/Raw reads比例最低94.95%,Q20(碱基质量大于20%)不低于96.67%,Q30不低于90.01%,GC含量均在50%左右(表2)。此外,所有样本均有80%左右的Cleans reads能够比对在猪基因组上。其中,6%左右的reads为多位点比对上(Multiple mapped),余下的74%左右均为单一位点比对上(Uniquely mapped)(表3)。Cleans reads定位在外显子(Exon)区域的百分比含量最高(均在77%以上)(图1)。
表2 测序数据质量评估
Tab.2 Quality assessment of sequencing data
样品名Samplename原始数据Raw reads过滤后数据Clean readsClean reads与Raw reads比率/%Rate of Clean reads/Raw reads过滤后序列长度/Gbclean basesQ20/%Q30/%GC百分比/%GC contentZR113 919 11113 728 48598.631.37G97.1991.5352.75ZR225 946 18524 634 94794.952.46G96.6790.0155.17ZR315 492 61215 262 45798.511.53G97.3692.0349.83ZR414 413 04814 205 81498.561.42G97.1291.3852.95ZC113 836 02613 649 43798.651.36G97.2991.7951.56ZC230 456 40728 919 43294.952.89G97.0390.9852.88ZC312 884 47712 696 38698.541.27G97.1191.3552.87ZC414 715 82014 502 47198.551.45G97.1391.3853.04
注:ZR.荣昌猪;ZC.长白猪。表3同。
Note:ZR.Rongchang pigs;ZC.Landrace.The same as Tab.3.
表3 测序Reads与参考基因组比对
Tab.3 Alignment of sequencing reads and reference genome
样品名Sample nameZR1ZR2ZR3ZR4ZC1ZC2ZC3ZC4总reads Total reads13 728 48513 054 67515 262 45714 205 81413 649 43713 000 00012 696 38614 502 471比对上的总reads11 050 22010 341 22512 409 854113 87 89811 239 15910 335 22410 345 92711 773 884Total mapped (80.49%)(79.21%)(81.31%)(80.16%)(82.34%) (79.5%) (81.49%)(81.19%)多个比对位置的reads839 995780 749927 090863 376851 879773 758764 647882 650Multiple mapped reads (6.12%)(5.98%) (6.07%)(6.08%) (6.24%) (5.95%) (6.02%) (6.09%)唯一比对位置的reads10 210 2259 560 47611 482 76410 524 52210 387 2809 561 4669 581 28010 891 234Uniquely mapped reads (74.37%)(73.23%) (75.24%) (74.09%) (76.1%) (73.55%) (75.46%) (75.1%)比对到正链上的reads5 106 4834 780 5895 743 8395 258 5985 192 5604 750 2094 788 9015 447 722reads map to +(37.2%)(36.62%)(37.63%)(37.02%)(38.04%)(36.54%)(37.72%)(37.56%)比对到负链上的reads5 103 7424 779 8875 738 9255 265 9245 194 7204 811 2574 792 3795 443 512reads map to -(37.18%)(36.61%)(37.6%)(37.07%)(38.06%)(37.01%)(37.75%)(37.54%)分段比对到2个外显子上的reads6 750 1746 212 3647 684 4646 898 6456 887 9315 982 9446 354 1187 096 438Non-splice reads(49.17%)(47.59%)(50.35%)(48.56%)(50.46%)(46.02%)(50.05%)(48.93%)整段比对到2个外显子上的reads3 460 0513 348 1123 798 3003 625 8773 499 3493 578 5223 227 1623 794 796Splice reads(25.2%)(25.65%)(24.89%)(25.52%)(25.64%)(27.53%)(25.42%)(26.17%)
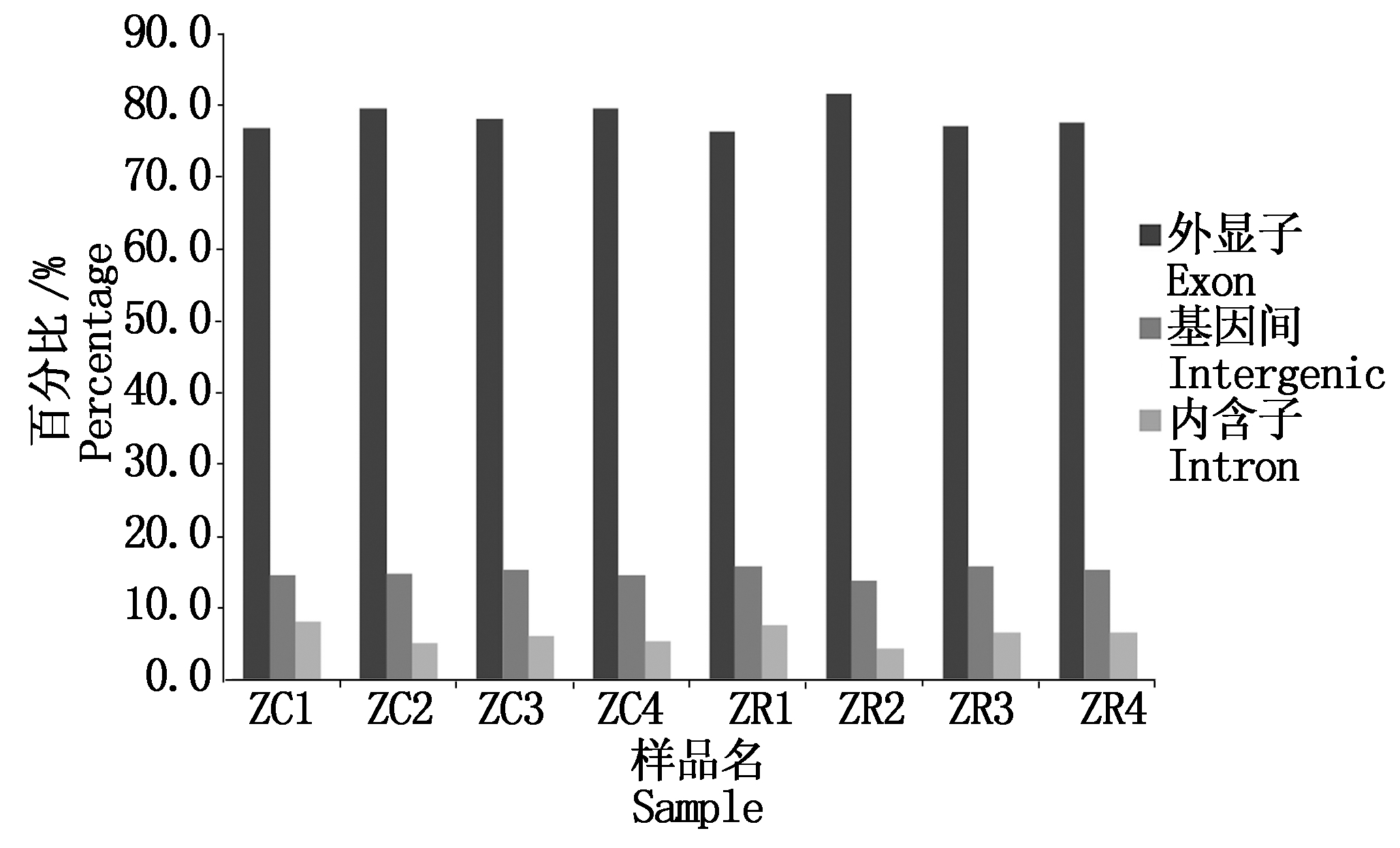
图1 Clean reads在基因组不同区域分布
Fig.1 Distribution of clean reads in
different regions of the genome
2.2 荣昌猪、长白猪胸腺RNA-seq主成分分析
为了评估此次测序数据的准确性,本研究对荣昌猪、长白猪胸腺RNA-seq数据进行了主成分(PCA)分析,结果发现,4个荣昌猪胸腺样本聚类在一起,4个长白猪胸腺样本聚类在一起(图2),表明此次RNA-seq的数据可靠性高。
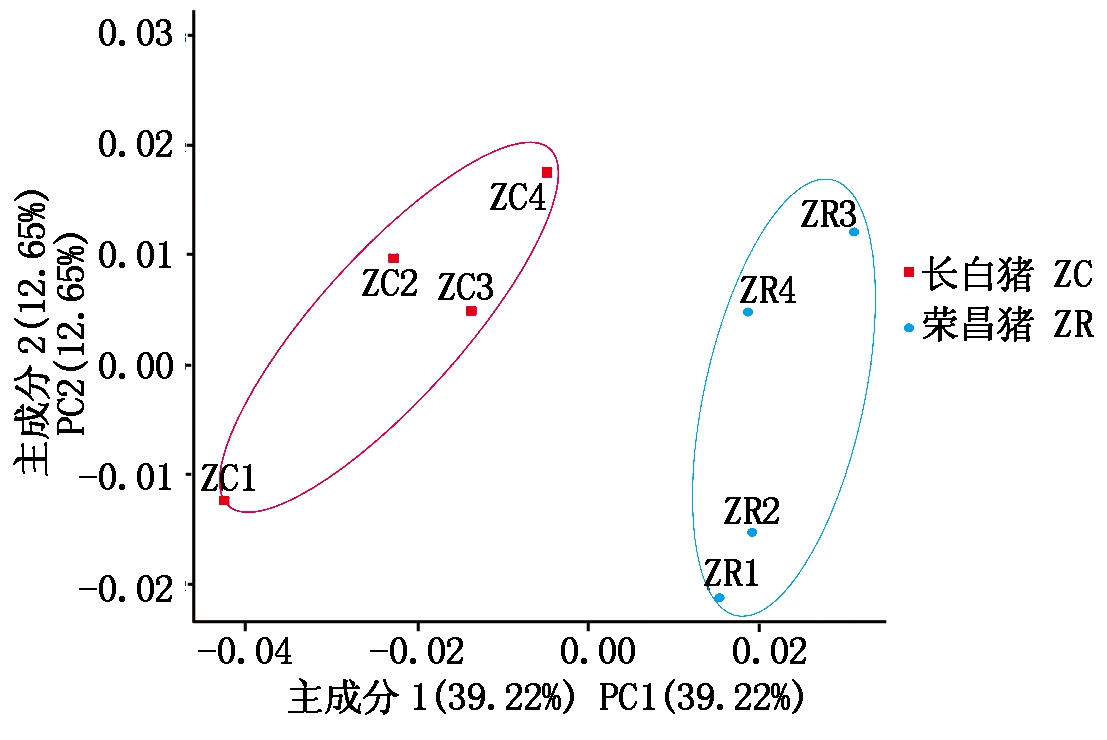
图2 RNA-seq主成分分析
Fig.2 PCA analysis of RNA-seq
2.3 荣昌猪和长白猪胸腺转录组差异表达基因
以|log2(Fold change)| > 1、P<0.05为标准筛选荣昌猪、长白猪胸腺差异表达基因,结果发现,差异倍数在2倍以上的基因共421个,其中,荣昌猪相对长白猪上调的有231个,下调的有190个(图3)。
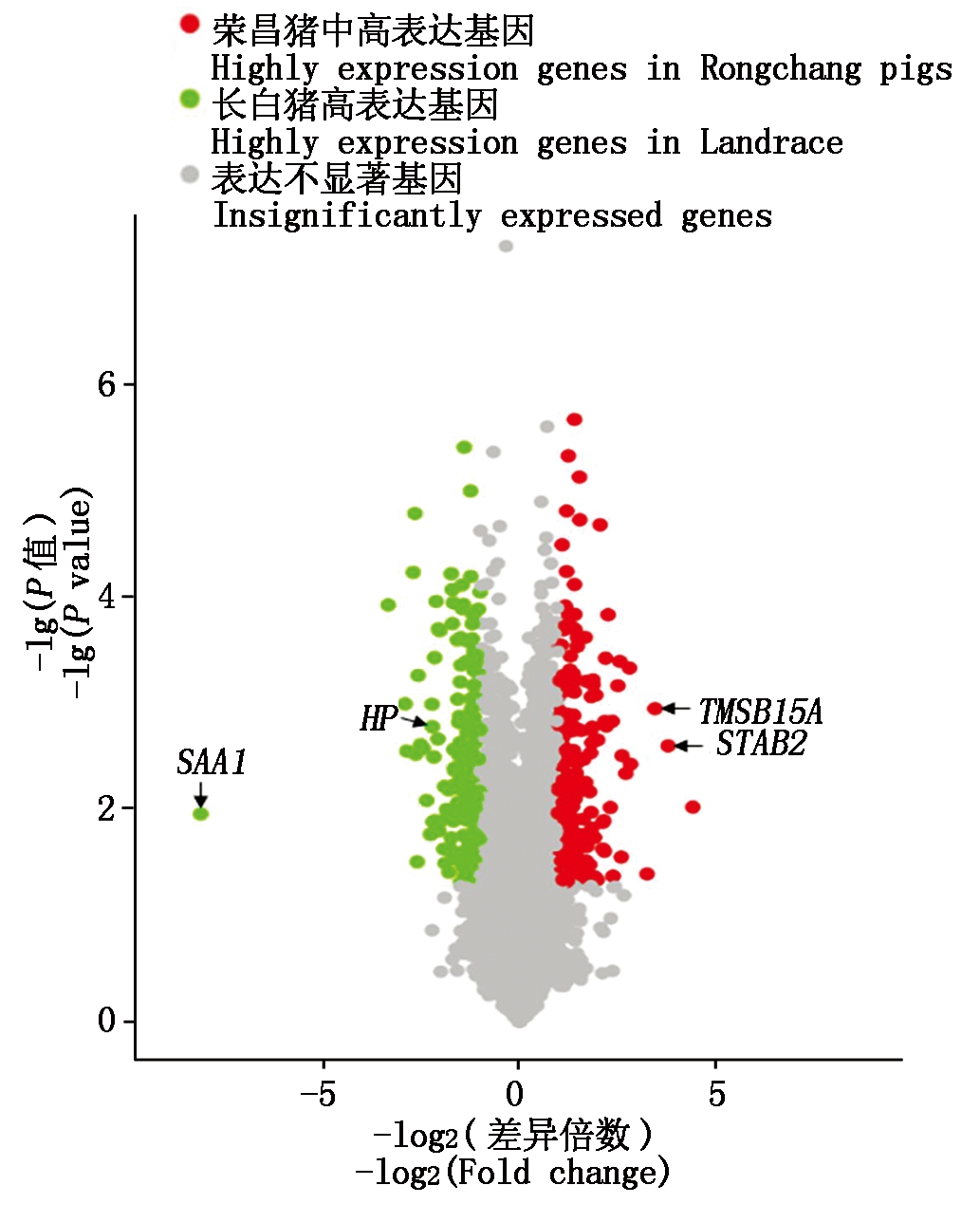
图3 荣昌猪和长白猪差异表达基因火山图
Fig.3 Volcano plot for differentially expressed genes
between Rongchang pig and Landrace
差异倍数大于5倍的基因,荣昌猪有11个,长白猪有10个;差异倍数大于10倍的基因,荣昌猪有2个,长白猪有1个;差异倍数大于20倍的基因,荣昌猪有1个,长白猪有0个;差异倍数大于100倍的基因,长白猪有1个(图4)。在荣昌猪中表达量高且与长白猪表达差异最大的基因是TMSB15A,在长白猪中表达量高且与荣昌猪表达差异最大的基因是SAA1。此外,STAB2和HP基因在荣昌猪和长白猪中表达量较高,表达倍数差异较大(图3)。
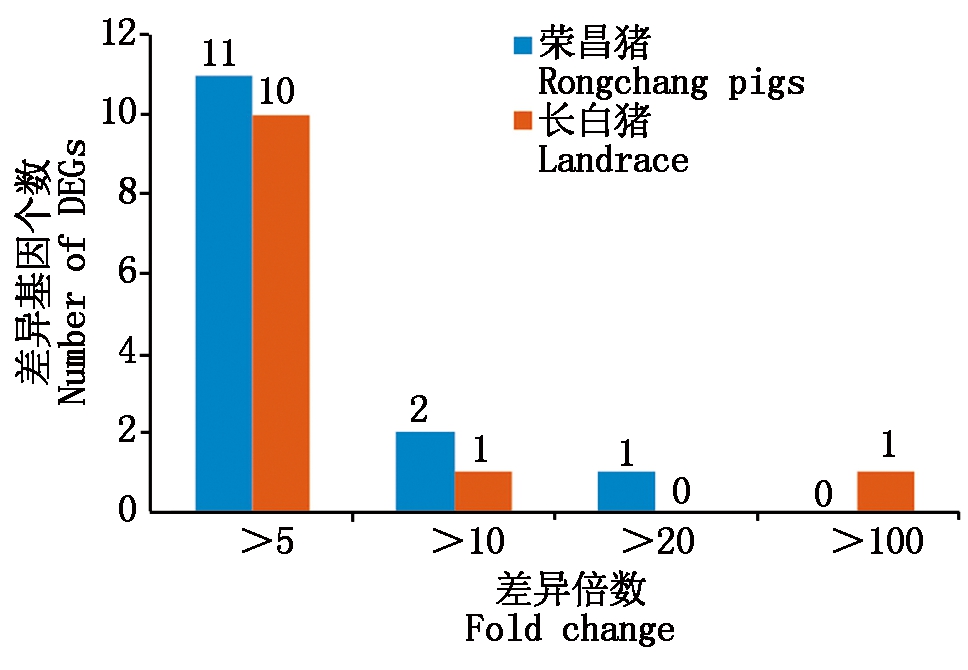
图4 荣昌猪、长白猪差异表达基因分布
Fig.4 DEGs distribution map between
Rongchang pig and Landrace
2.4 差异表达基因的GO功能富集和KEGG代谢通路分析
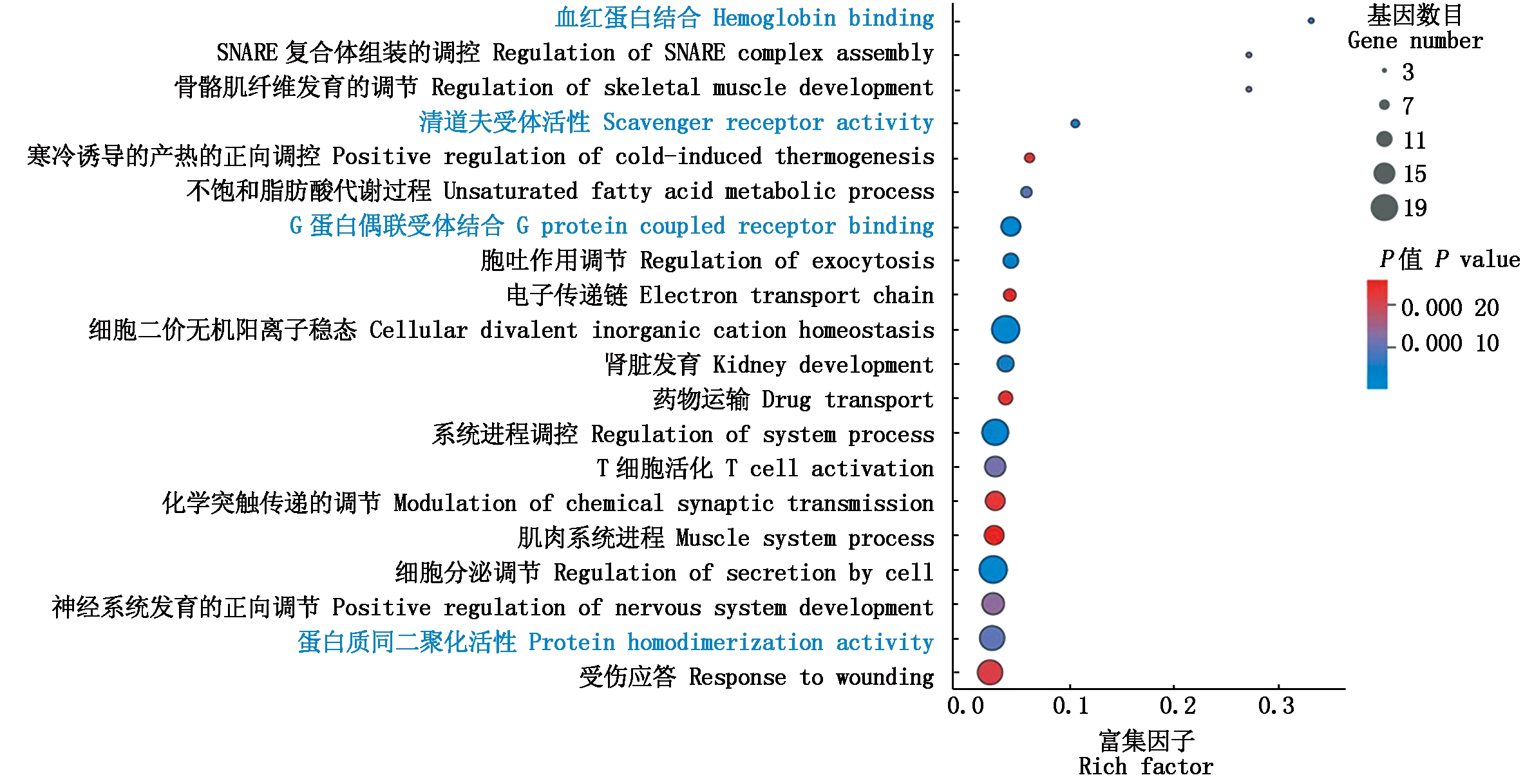
GO功能富集分析可以更好地对差异表达基因进行注释及基因功能分类,分为分子功能(Molecular functions)、生物进程(Biological processes)及细胞组分(Cellular component)3个组分。本研究差异表达基因的GO功能富集分析结果表明(图5),差异表达基因显著富集的前20条目中,分子功能4条,生物进程16条。与分子功能有关的主要是清道夫受体和G蛋白偶联受体结合过程。与生物进程有关的主要是T细胞活化、细胞分泌调节、胞吐作用调节、SNARE复合物组装调节等,暗示荣昌猪、长白猪胸腺中这些生物学进程可能导致了荣昌猪、长白猪胸腺先天免疫机能的差异。KEGG代谢通路分析有助于更好地对差异表达基因中存在的显著性富集的代谢通路进行注释。本研究KEGG代谢通路分析结果表明,差异表达基因显著富集的代谢通路有12条,主要包括细胞因子-细胞因子受体互作、cAMP信号通路、代谢(磷脂酶D、半胱氨酸和蛋氨酸)及一些疾病(非洲锥虫病、癌症)相关通路(图6),表明荣昌猪和长白猪胸腺中的这些差异表达基因主要通过调控上述代谢通路来引起荣昌猪和长白猪先天免疫差异。
蓝色字体.分子功能;黑色字体.生物进程。
Blue fonts.Molecular functions;Black fonts.Biological processes.
图5 差异表达基因GO分析
Fig.5 GO analysis of differentially expressed genes
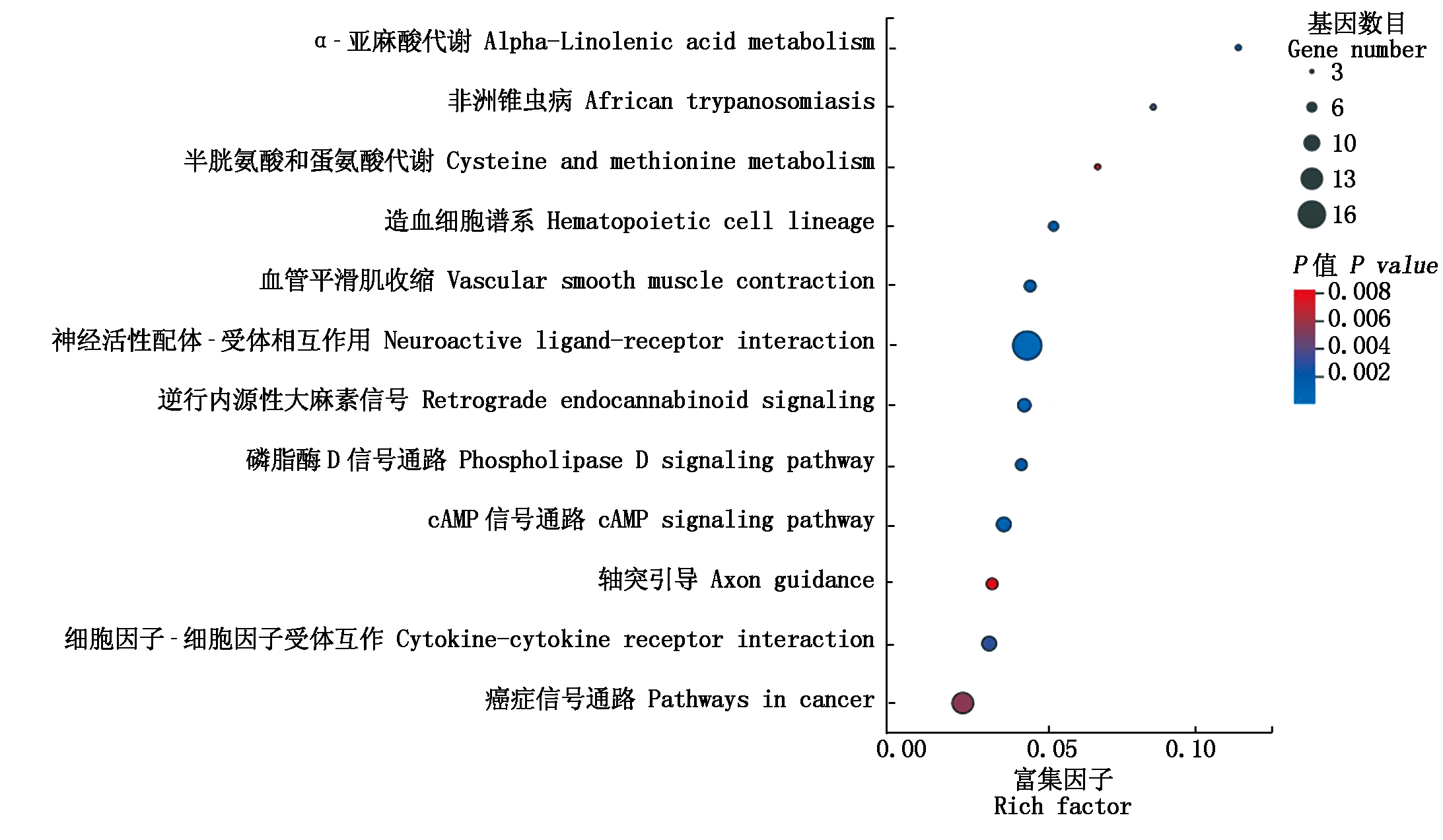
图6 差异表达基因KEGG分析结果
Fig.6 KEGG analysis of differentially expressed genes
2.5 转录组测序数据的RT-qPCR验证
在差异表达基因中随机选择6个基因进行RT-qPCR,结果发现,所选择6个基因在荣昌猪和长白猪2个品种间的表达变化模式与RNA-seq结果一致(表4),表明转录组测序结果较为准确,因此,基于此测序结果获得的差异表达基因数据也较为准确。
表4 差异表达基因RT-qPCR验证
Tab.4 RT-qPCR verification of differentially
expressed genes
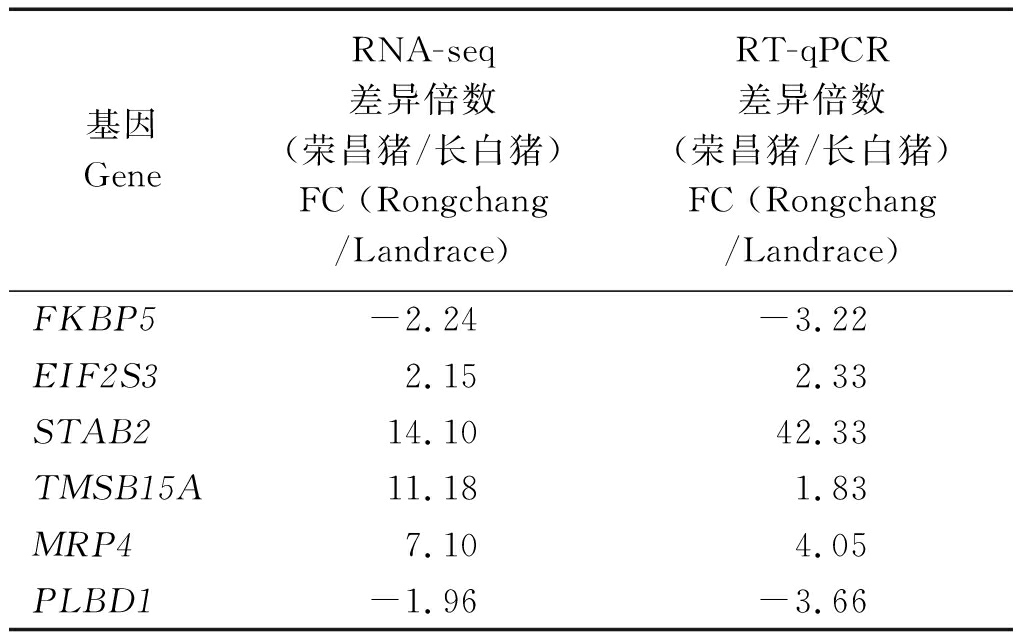
基因GeneRNA-seq差异倍数(荣昌猪/长白猪)FC (Rongchang/Landrace)RT-qPCR差异倍数(荣昌猪/长白猪)FC (Rongchang/Landrace)FKBP5-2.24-3.22EIF2S32.152.33STAB214.1042.33TMSB15A11.181.83MRP47.104.05PLBD1-1.96-3.66
3 讨论与结论
本研究利用RNA-seq技术比较分析了荣昌猪和长白猪胸腺组织中的差异表达基因,结果发现,在荣昌猪中表达量高且与长白猪表达差异最大的候选基因是TMSB15A,其他的差异高表达的免疫候选基因有STAB2。长白猪中表达量最高且表达差异最大的候选基因是SAA1,其他差异高表达候选基因有HP。研究结果可为进一步研究地方猪优质抗病性状的分子机理奠定理论基础。
急性期反应是指在感染、炎症等发生后,机体短时间内发生的非特异性应答[15],急性期反应物即参与急性期反应的物质,多为蛋白,这些蛋白通常被称为急性时相反应蛋白,是炎症早期的标志。SAA1基因编码的血清淀粉样蛋白A1,是急性时相反应蛋白之一。Smole等[16]研究发现,SAA1蛋白可与病原微生物中的脂肪酸结合蛋白FABP相互作用,激活SAA1蛋白结合受体FPR2,促进内皮细胞释放细胞因子IL-33来增强2型免疫。Cheng等[17]发现,在机体遭受革兰氏阴性感染时,SAA1蛋白可直接与细菌细胞壁上的脂多糖相结合并形成复合物,促进巨噬细胞对脂多糖的摄取,继而实现巨噬细胞对细菌的吞噬作用。中性粒细胞是炎症过程中的主要效应细胞,但它们也可以通过分泌抗炎细胞因子来控制机体过度的炎症反应。White 等[18]发现,SAA1蛋白能直接激活中性粒细胞并将其募集起来。Santo等 [19]进一步研究发现,SAA-1蛋白能够通过调节中性粒细胞的分化可塑性来调控中性粒细胞的炎症效应,即SAA1蛋白不仅可以诱导中性粒细胞的生成和活化,使其发挥免疫功能,还可以促进中性粒细胞与恒定自然杀伤细胞之间的相互作用,使中性粒细胞减少IL-10的分泌,增加IL-12的分泌,从而降低中性粒细胞引起的过度炎症反应。除了直接激活白细胞外,SAA1蛋白还可以通过toll样受体2信号来诱导趋化因子的生成。Gouwy等[20]发现,SAA诱导生成的趋化因子与完整的SAA1协同作用,可以在体外和体内对中性粒细胞进行化学吸引,继而发挥免疫功能。Zheng等[21]发现,在机体遭受金黄色葡萄球菌感染时,SAA会自发形成小囊泡或胶束来破坏金黄色葡萄球菌细胞膜上的阴离子脂质体,继而发挥抗菌活性。此外,还有研究发现,SAA1具有促进细胞吞噬功能相关基因上调表达的能力[22]。上述研究表明,SAA1可以通过多种生物学过程实现对多种病原的抑制作用。本研究发现在长白猪中表达量高且与荣昌猪表达差异最大的基因是SAA1,鉴于SAA1通常在炎症或病原感染早期出现,表明长白猪的先天免疫优势可能体现在病原感染早期的快速应答。
HP基因编码结合珠蛋白(Haptoglobin),也是一种急性时相反应蛋白。在机体感染病原体时,病原体可通过其自身的铁捕获系统,首先从血红蛋白中提取血红素并将其转运进自身的细胞质中,最终使血红素中的铁释放出来以维持自身的营养所需。HP蛋白则可以通过CD63+介导的巨噬细胞的吞噬作用[23]来清除从红细胞中漏出来的血红蛋白,从而使病原体无法摄取血红素。同时,HP蛋白还可以限制病原体对血红素中铁元素的利用[24],从而抑制病原体增殖。此外,Raju等 [25]研究发现,HP蛋白不仅可以抑制病原体增殖,还可通过抑制促炎因子的功能来维持机体的免疫耐受。本研究发现,HP基因是长白猪胸腺中显著高表达的候选基因,且HP基因与SAA1基因同属于急性时相反应蛋白,因此,进一步表明了长白猪先天免疫优势可能体现在病原感染早期的快速应答。
胸腺素是一类可以促进T淋巴细胞的生长、诱导调节性T细胞的分化及增强自然杀伤细胞的活性的生物活性分子[26-28],在机体免疫中具有重要作用。依据蛋白等电点的差异,胸腺素可以分为α、β和γ 3个家族。Zou等[29]研究发现,胸腺素β可以通过与细菌壁中的多糖结合,抑制金黄色葡萄球菌、嗜水气单胞菌、鳗弧菌、铜绿假单胞菌和肺炎克雷伯菌的生长,从而在斑马鱼的先天抗菌免疫中发挥重要作用。Shi等[30]研究证实,胸腺素β可以抑制白斑综合症病毒的复制,促进血细胞对病毒的吞噬以及提高感染后的存活率,继而在非脊椎动物的抗病毒免疫中发挥重要功能。TMSB15A(Thymosin beta 15A)蛋白属于胸腺素β家族一员,课题组前期研究发现,TMSB15A基因在猪胸腺、脾脏等免疫相关组织中特异性高表达[26],表明其在机体免疫中的确发挥着重要作用。当前关于TMSB15A基因的报道虽然主要集中在癌症[31-32]上,但是鉴于其家族成员在机体先天免疫中的功能,且其在荣昌猪中表达量高且与长白猪表达差异最大,可以推测,TMSB15A是荣昌猪先天免疫的主要候选基因,因此,荣昌猪在产生生物活性物质增强机体免疫细胞功能方面较长白猪更有优势。
STAB2蛋白是一类清道夫受体,在机体内的配体包括凋亡细胞、革兰氏阴性菌、革兰氏阳性菌和淋巴细胞[33],主要功能就是与这些配体相结合来介导机体对这些配体的吞噬作用,从而维持机体的稳态[34]。例如,STAB2蛋白识别凋亡细胞膜上的磷脂酰丝氨酸后,通过在凋亡小体吞噬过程中诱导产生抗炎介质来减轻机体炎症[35],维持机体稳态。再例如,STAB2蛋白可作为细胞黏附分子,使血液中循环的淋巴细胞与内皮细胞相互作用,将淋巴细胞募集到机体特定的组织中,从而调控机体的炎症和免疫应答[36]。本研究发现,STAB2基因在荣昌猪和长白猪中表达量较高,说明STAB2基因在长白猪、荣昌猪先天免疫中发挥着重要作用。而STAB2基因在荣昌猪和长白猪之间的表达倍数差异较大,表明荣昌猪可能在免疫应答后期的病原清除以及抗炎功能上更优于长白猪。
生物体内各种生物学功能的实现依赖于多个基因之间的协调作用,通过对差异候选基因进行GO富集和KEGG信号通路分析,可以更加深入地了解该基因的生物学功能及其与其他基因的互作关系。本研究发现,荣昌猪中高表达的基因主要富集在清道夫受体、细胞因子-细胞因子受体互作及T细胞活化、造血细胞分化等免疫相关通路,长白猪中高表达的基因主要富集在离子通道活性、钙离子转运、细胞骨架、G蛋白偶联受体通路,鲜少与机体先天免疫相关,暗示荣昌猪、长白猪胸腺中这些生物学进程和信号通路差异可能导致了新生阶段荣昌猪、长白猪胸腺免疫机能的差异。此研究结果为进一步研究地方猪的抗病力强于外种猪的分子机理奠定了理论基础。
综上,结合荣昌猪、长白猪候选基因的功能、GO富集以及KEGG信号通路综合分析发现,长白猪的先天免疫优势可能更集中体现在病原体感染早期的快速应答,这正好与长白猪的部分差异高表达基因富集在G蛋白偶联受体通路[19]结果相一致。荣昌猪的先免疫优势则更多体现在促进各种免疫细胞的增殖和功能、免疫应答后期及炎症抵抗等方面,包括T细胞、清道夫受体、细胞因子-细胞因子受体互作等。
本研究利用RNA-seq技术比较分析了荣昌猪、长白猪胸腺差异表达基因,并结合GO富集和KEGG信号通路分析筛选到荣昌猪先天免疫候选基因TMSB15A和STAB2,长白猪先天免疫候选基因SAA1和HP,研究结果为进一步研究地方猪优质抗病性状的分子机理奠定了理论基础。
[1] 戴超辉,冯海悦,吴圣龙,包文斌.猪源Toll样受体家族(TLRs)及其在抗病育种中的应用[J].浙江农业学报,2018,30(3):507-520.doi:10.3969/j.issn.1004-1524.2018.03.21.
Dai C H,Feng H Y,Wu S L,Bao W B. Advances in porcine Toll-like receptor family(TLRs)and its application in resistance breeding[J].Acta Agriculturae Zhejiangensis,2018,30(3):507-520.
[2] 王超,赵书红,朱猛进.猪抗病育种的相关问题及研究进展[J].中国畜牧杂志,2014,50(22):67-72.doi:10.3969/j.issn.0258-7033.2014.22.014.
Wang C,Zhao S H,Zhu M J. Related problems and research progress in anti-disease breeding of pig[J].Chinese Journal of Animal Science, 2014,50(22):67-72.
[3] Costa V,Angelini C,De Feis I,Ciccodicola A. Uncovering the complexity of transcriptomes with RNA-Seq[J].Journal of Biomedicine & Biotechnology,2010,2010:853916.doi:10.1155/2010/853916.
[4] 陈一丹,张昱,杨洁,张勤,姜力.基于转录组测序的奶牛产奶性状重要功能基因挖掘[J].生物技术通报,2020,36(9):244-252.doi:10.13560/j.cnki.biotech.bull.1985.2020-0255.
Chen Y D,Zhang Y,Yang J,Zhang Q,Jiang L. Exploration of key functional genes affecting milk production traits in dairy cattle based on RNA-seq[J].Biotechnology Bulletin,2020,36(9):244-252.
[5] 刘远,李文杨,吴贤锋,黄勤楼,高承芳,陈鑫珠,张晓佩.福清山羊与努比亚黑山羊背最长肌比较转录组分析[J].中国农业科学,2019,52(14):2525-2537.doi:10.3864/j.issn.0578-1752.2019.14.011.
Liu Y,Li W Y,Wu X F,Huang Q L,Gao C F,Chen X Z,Zhang X P. Transcriptome analysis of differentially gene expression associated with longissimus doris tissue in Fuqing goat and Nubian black goat[J].Scientia Agricultura Sinica,2019,52(14):2525-2537.
[6] 成志敏,张宁芳,王媛媛,乐宝玉,张万锋,宋鹏康,郭晓红,高鹏飞,蔡春波,曹果清,李步高. 基于RNA-Seq技术筛选影响猪肌纤维性状的候选基因[J].畜牧兽医学报,2019,50(5):918-929. doi:10.11843/j.issn.0366-6964.2019.05.003.
Cheng Z M,Zhang N F,Wang Y Y,Le B Y,Zhang W F,Song P K,Guo X H,Gao P F,Cai C B,Cao G Q,Li B G. Screening of candidate genes for muscle fiber characteristics in pig using RNA-seq[J].Acta Veterinaria et Zootechnica Sinica,2019,50(5):918-929.
[7] 朱志明,陈红萍,林如龙,缪中纬,辛清武,李丽,张丹青,郑嫩珠. 山麻鸭开产期和产蛋高峰期卵巢组织转录组分析[J].中国农业科学,2016,49(5):998-1007.doi:10.3864/j.issn.0578-1752.2016.05.020.
Zhu Z M,Chen H P,Lin R L,Miao Z W,Xin Q W,Li L,Zhang D Q,Zheng N Z. Transcriptome analysis of ovary tissue in early laying period and egg laying peak period of Shanma ducks[J].Scientia Agricultura Sinica,2016,49(5):998-1007.
[8] Li Y P,Liu H,Wang P F,Wang L Y,Sun Y,Liu G,Zhang P,Kang L,Jiang S J,Jiang Y L.RNA-seq analysis reveals genes underlying different disease responses to porcine Circovirus type 2 in pigs[J].PLoS One,2016,11(5):e0155502. doi:10.1371/journal.pone.0155502.
[9] Ni L G,Song C Y,Wu X S,Zhao X T,Wang X Y,Li B C,Gan Y. RNA-seq transcriptome profiling of porcine lung from two pig breeds in response to Mycoplasma hyopneumoniae infection[J].PeerJ,2019,7(2):e7900. doi:10.7717/peerj.7900.
[10] Nascimento N C D,Guimaraes A M S,Santos A P D,Chu Y F,Lucas M M,Joanne B M. RNA-seq based transcriptome of whole blood from immunocompetent pigs(Sus scrofa)experimentally infected with Mycoplasma suis strain Illinois[J]. Veterinary Research,2018,49(1):49. doi:10.1186/s13567-018-0546-6.
[11] Zhang F F,Yuan W F,Li Z Q,Zhang Y H,Ye Y,Li K,Ding Z,Chen Y Y,Cheng T,Wu Q,Tang Y X,Song D P. RNA-seq-based whole transcriptome analysis of IPEC-J2 cells during Swine acute diarrhea syndrome coronavirus infection[J]. Frontiers in Veterinary Science,2020,7:492. doi:10.3389/fvets.2020.00492.
[12] Zhang T L,Chen H T,Qi L L,Zhang J,Wu R,Zhang Y G,Sun Y F. Transcript profiling identifies early response genes against FMDV infection in PK-15 cells[J]. Viruses,2018,10(7):364. doi:10.3390/v10070364.
[13] 袁树楷,王金勇,谢和芳,李琴,白小青. 猪抗病育种候选基因研究进展[J].中国畜牧杂志,2007,43(15):50-52. doi:10.3969/j.issn.0258-7033.2007.15.018.
Yuan S K,Wang J Y,Xie H F,Li Q,Bai X Q. Advances in candidate genes of porcine disease resistance[J].Chinese Journal of Animal Science,2007,43(15):50-52.
[14]  inkora M,Butler J E. The ontogeny of the porcine immune system[J].Developmental and Comparative Immunology,2009,33(3):273-283. doi:10.1016/j.dci.2008.07.011.
inkora M,Butler J E. The ontogeny of the porcine immune system[J].Developmental and Comparative Immunology,2009,33(3):273-283. doi:10.1016/j.dci.2008.07.011.
[15] Gelain M E,Bonsembiante F. Acute phase proteins in marine mammals:State of art,perspectives and challenges[J].Frontiers in Immunology,2019,10:1220.doi:10.3389/fimmu.2019.01220.
[16] Smole U,Gour N,Phelan J,Hofer G,Köhler C,Kratzer B,Tauber P A,Xiao X,Yao N,Dvorak J,Caraballo L,Puerta L,Rosskopf S,Chakir J,Malle E,Lane A P,Pickl W F,Lajoie S,Wills-Karp M. Serum amyloid A is a soluble pattern recognition receptor that drives type 2 immunity[J].Nature Immunology,2020,21(7):756-765. doi:10.1038/s41590-020-0698-1.
[17] Cheng N,Liang Y R,Du X P,Ye R D.Serum amyloid A promotes LPS clearance and suppresses LPS-induced inflammation and tissue injury[J].EMBO Reports,2018,19(10):e45517.doi:10.15252/embr.201745517.
[18] White M R,Hsieh I H,De Luna X,Hartshorn K L. Effects of serum amyloid protein A on Influenza A virus replication and viral interactions with neutrophils[J].Journal of Leukocyte Biology,2020,10(1):155-166.doi:10.1002/JLB.4AB0220-116RR.
[19] De Santo C,Arscott R,Booth S,Karydis I,Jones M,Ashe R,Salio M,Middleton M,Cerundolo V.Invariant NKT cells modulate the suppressive activity of IL-10-secreting neutrophils differentiated with serum amyloid A[J].Nature Immunology,2010,11(11):1039-1046. doi:10.1038/ni.1942.
[20] Gouwy M,De Buck M,Salama S A,Vandooren J,Knoops S,Pörtner N,Vanbrabant L,Berghmans N,Opdenakker G,Proost P,Van Damme J,Struyf S. Matrix metalloproteinase-9-generated COOH-,but not NH2-terminal fragments of serum amyloid A1 retain potentiating activity in neutrophil migration to CXCL8,with loss of direct chemotactic and cytokine-inducing capacity[J].Frontiers in Immunology,2018,9:1081. doi:10.3389/fimmu.2018.01081.
[21] Zheng H,Li H F,Zhang J Y,Fan H L,Jia L N,Ma W Q,Ma S Q,Wang S H,You H,Yin Z N,Li X D. Serum amyloid A exhibits pH dependent antibacterial action and contributes to host defense against Staphylococcus aureus cutaneous infection[J].Journal of Biological Chemistry,2020,295(9):2570-2581. doi:10.1074/jbc.RA119.010626.
[22] Leow K Y,Goh W W B,Heng C K. Effect of serum amyloid A1 treatment on global gene expression in THP-1-derived macrophages[J].Inflammation Research,2012,61(4):391-398. doi:10.1007/s00011-011-0424-4.
[23] Nishitani K,Ishikawa M,Morita Y,Yokogawa N,Xie C,Bentley K L D M,Ito H,Kates S L,Daiss J L,Schwarz E M. IsdB antibody-mediated sepsis following S.aureus surgical site infection[J].JCI Insight,2020,5(19):e141164. doi:10.1172/jci.insight.141164.
[24] Mikkelsen J H,Runager K,Andersen C B F. The human protein haptoglobin inhibits IsdH-mediated heme-sequestering by Staphylococcus aureus[J].Journal of Biological Chemistry,2020,295(7):1781-1791. doi:10.1074/jbc.RA119.011612.
[25] Raju S M,Kumar A P,Yadav A N,Rajkumar K,MVS S,Burgula S. Haptoglobin improves acute phase response and endotoxin tolerance in response to bacterial LPS[J].Immunology Letters,2019,207:17-27. doi:10.1016/j.imlet.2019.01.002.
[26] 龙熙,蓝静,郭宗义,王金勇,赵久刚. 荣昌猪胸腺素β15A(TMSB15A)cDNA全长克隆、序列信息及组织表达分析[J].畜牧兽医学报,2017,48(9):1602-1610.doi:10.11843/j.issn.0366-6964.2017.09.005.
Long X,Lan J,Guo Z Y,Wang J Y,Zhao J G. The full-length cloning,sequence information and expression analysis of cDNA of thymosin β15A(TMSB15A)in Rongchang pig[J].Acta Veterinaria et Zootechnica Sinica,2017,48(9):1602-1610.
[27] Liao W,Lin J X,Leonard W J. IL-2 family cytokines:New insights into the complex roles of IL-2 as a broad regulator of T helper cell differentiation[J].Current Opinion in Immunology,2011,23(5):598-604. doi:10.1016/j.coi.2011.08.003.
[28] Goldstein A L,Badamchian M. Thymosins:chemistry and biological properties in health and disease[J]. Expert Opinion on Biological Therapy,2004,4(4):559-573.doi:10.1517/14712598.4.4.559.
[29] Zou S S,Wang J,Li B X,Yang G W,Sun J J,Yang H T. Thymosin participates in antimicrobial immunity in zebrafish[J].Fish & Shellfish Immunology,2019,87:371-378. doi:10.1016/j.fsi.2019.01.040.
[30] Shi X Z,Shi L J,Zhao Y R,Zhao X F,Wang J X. β-Thymosins participate in antiviral immunity of red swamp crayfish(Procambarus clarkii)[J].Developmental and Comparative Immunology,2015,51(2):213-225. doi:10.1016/j.dci.2015.04.003.
[31] Anjum S,Sahar T,Nigam A,Wajid S. Transcriptome analysis of mRNA in uterine leiomyoma using next-generation RNA sequencing[J].Anti Cancer Agents in Medicinal Chemistry,2019,19(14):1703-1718. doi:10.2174/1871520619666190409102855.
[32] Darb-Esfahani S,Kronenwett R,Minckwitz G V,Denkert C,Gehrmann M,Rody A,Budczies J,Brase J C,Mehta M K,Bojar H,Ataseven B,Karn T,Weiss E,Zahm D M,Khandan F,Dietel M,Loibl S. Thymosin beta 15A(TMSB15A)is a predictor of chemotherapy response in triple-negative breast cancer[J].British Journal of Cancer,2012,107(11):1892-1900. doi:10.1038/bjc.2012.475.
[33] Weigel P H. Discovery of the liver hyaluronan receptor for endocytosis(HARE)and its progressive emergence as the multi-ligand scavenger receptor Stabilin-2[J].Biomolecules,2019,9(9):454. doi:10.3390/biom9090454.
[34] Weigel P H. Systemic glycosaminoglycan clearance by HARE/Stabilin-2 activates intracellular signaling[J].Cells,2020,9(11):2366. doi:10.3390/cells9112366.
[35] Penberthy K K,Ravichandran K S. Apoptotic cell recognition receptors and scavenger receptors[J].Immunological Reviews,2016,269(1):44-59. doi:10.1111/imr.12376.
[36] Jung M Y,Park S Y,Kim I S. Stabilin-2 is involved in lymphocyte adhesion to the hepatic sinusoidal endothelium via the interaction with αMβ2 integrin[J].Journal of Leukocyte Biology,2007,82(5):1156-1165. doi:10.1189/jlb.0107052.
