南方根结线虫(Meloidogyne incognita)是危害黄瓜(Cucumis sativus)的重要病原,已成为影响黄瓜生产的严重障碍[1-2]。实践证明,应用抗线虫品种是防治根结线虫最经济有效和无污染的方法[3]。迄今,国内外尚无抗南方根结线虫的黄瓜品种应用于生产,对根结线虫缺乏有效的防控措施。黄瓜属(Cucumis)一些野生资源,如酸黄瓜(C. hystrix)、角瓜(C.metuliferus)等对南方根结线虫具有抗性[1-2],Chen等[4]成功实现了酸黄瓜与栽培黄瓜间的种间杂交,选育出抗南方根结线虫的黄瓜-酸黄瓜渐渗系[5]。可见,野生抗源在黄瓜根结线虫抗性遗传改良中的潜力已逐步凸现,但种间杂交障碍仍使一些优异抗源未能得到有效利用[6]。角瓜为原产非洲的黄瓜属近源野生物种,角瓜对南方根结线虫的优异抗性得到了黄瓜遗传学家的广泛关注[1,7-10],然而种间杂交障碍制约了其在黄瓜遗传改良中的利用。因此,筛选鉴定黄瓜抗南方根结线虫的关键基因,探明其根结线虫抗性形成的分子机制,可为抗线虫品种的选育提供理论依据。
MicroRNA(miRNAs)是一类长度为21~24 nt的非编码小RNA(sRNA),通过与靶基因的3′UTR区结合抑制靶基因表达或直接降解mRNA,在转录和转录后水平发挥调控作用[11]。miRNAs调控参与植物-线虫互作的基因表达,且miRNAs的靶标多为转录因子[12-14],一些转录因子与植物的线虫抗性密切相关[15]。目前,有关研究miRNAs在植物-线虫互作中的功能以孢囊线虫(CNs)居多[16-18]。Hewezi等[19]从甜菜孢囊线虫(Heterodera schachtii,BCN)侵染拟南芥(Arabidopsis thaliana)的根系内鉴定出30个miRNAs,大部分miRNAs负调控靶基因表达。进一步研究发现,miR396-GRF1/F3是BCN诱导的合胞体中基因表达与细胞重排重要的调控体系,miR396控制合胞体发育阶段的转换,而GRF1/F3通过调控激素合成和信号传导的基因表达控制合胞体的细胞数目与体积[16]。拟南芥miR827介导了BCN合胞体中靶基因泛素E3连接酶的转录后基因沉默,增强了拟南芥对BCN的感病性[20],拟南芥miR858则通过调控其靶基因MYB83的表达从而在合胞体的形成中发挥重要作用[21]。有关miRNAs在植物-根结线虫互作中的功能研究报道较少,番茄根结线虫抗性受miR319/TCP4与miR396/GRF的互作调控,根系内miR319/TCP4-miR396/GRF调控细胞增殖,茎内miR319/TCP4调控JA合成[22],而miR319通过信号传导调控TCP4进而正调控miR396表达[23],从而抑制GRFs表达与细胞增殖[24]。拟南芥受南方根结线虫侵染后,miR159通过抑制其靶基因MYB33的翻译从而在根结发育中发挥重要作用[25]。爪哇根结线虫(M. javanica)侵染拟南芥后,根结中大部分miRNAs下调表达,而miR390在线虫侵染早期根结和巨型细胞中上调表达,miR390启动子在巨型细胞和根结维管束组织中具有活性,调控根结中tasiRNAs(trans-acting short interfering RNAs)的形成,tasiRNAs来源于TAS3,TAS3通过调控生长素响应因子(ARFs)来促进根结的生长发育[14]。可见,不同miRNAs在不同作物、不同线虫胁迫中发挥的作用不同。miRNAs作为基因表达的非编码调控sRNA,通过转录和转录后水平在植物-线虫互作中发挥重要作用。野生黄瓜miRNAs遗传多样性较栽培黄瓜丰富[26],一些miRNAs表达与种、组织及发育阶段的特异性相关[27],部分miRNAs进化上高度保守[28],目前有关miRNAs在黄瓜-根结线虫互作中的研究国内外鲜见报道。
本研究以角瓜为试材分别构建角瓜与南方根结线虫亲和、非亲和互作的sRNA文库,通过Solexa 测序和生物信息学分析,筛选鉴定角瓜与南方根结线虫非亲和互作密切相关的miRNAs并预测其靶基因,利用qRT-PCR检测miRNAs的表达,为深入解析角瓜与根结线虫非亲和互作的调控机理奠定基础。该项研究对于完善角瓜与根结线虫非亲和互作的分子机理具有重要的理论价值,相关研究结果可为通过miRNAs的遗传操作来改良黄瓜的根结线虫抗性提供技术支撑,具有潜在的应用前景。
1 材料和方法
1.1 试验材料
角瓜品系CmR07和CmS12,二者分别高抗、高感南方根结线虫[9]。采用穴盘育苗,将育苗基质(沙∶泥炭土=2∶1)经120 ℃高温灭菌2 h后装入穴盘,种子用0.1% HgCl2表面消毒15~20 min后催芽播种,待幼苗长至2叶1心时,采用2龄幼虫法接种南方根结线虫1号小种,接种液浓度为1 000条 J2/株。分别于接种线虫后4,30 d采集两材料的根系样品,同时采集未接种的根系样品做对照,每个处理随机取3份样品,每份样品随机取3株根系,用液氮速冻后保存于-80 ℃冰箱中备用。
1.2 总RNA提取、sRNA文库构建与高通量测序
采用TRIzol(上海生工)提取根系总RNA,利用Qubit 2.0 RNA检测试剂盒(Life)检测RNA浓度,通过1%琼脂糖凝胶电泳检测RNA完整性以及基因组污染情况。依据Qubit 2.0 RNA检测试剂盒对总RNA精确定量后取适量根系RNA,通过RNA Mix制备、3′端Adapter连接、逆转录引物杂交、5′端Adapter连接、逆转录反应、PCR反应进行sRNA文库构建,文库PCR产物用12% PAGE胶电泳检测,并回收140~150 bp左右的PCR产物条带,文库质检合格后利用Qubit 2.0 DNA检测试剂盒(Life)对回收的DNA精确定量,然后按照1∶1的比例等量混合,采用Soleax技术在HiSeq2500平台进行高通量测序。
1.3 测序数据统计和sRNA分类注释
对测序的原始数据通过FastQC进行质量评估,去除3′端接头,删除两端质量值低于20的碱基,过滤掉连续4个碱基平均质量值低于20,以及处理后长度低于17的reads,得到高质量序列(Clean reads)。利用Bowtie将Clean reads与Rfam数据库、物种mRNA进行比对,设置比对条件为:Gapopen为0、Evalue小于0.01、Mismatch小于等于1,过滤掉比对上的rRNA、sRNA、snRNA、snoRNA以及物种mRNA 的降解片段,然后将Clean reads转化为Fasta格式,得到的reads称Total reads,去除重复后的reads称Unique reads,最后对Raw reads、Clean reads和Unique reads进行注释统计。
1.4 miRNAs鉴定、差异表达筛选及qRT-PCR验证
由于野生角瓜没有已知miRNAs序列数据,可从miRBase数据库获取近缘物种(黄瓜、甜瓜)miRNAs序列,使用BlastN软件对所有样本Clean reads与成熟miRNAs序列进行比对,与数据库序列完全相同的reads即可视为已知miRNAs。采用TPM算法对各样本中miRNAs表达量进行归一化处理,使用edgeR包进行miRNAs差异表达分析,以|log2FoldChange|≥1、FDR≤0.05作为筛选条件,筛选得到差异显著的miRNAs。采用加尾法合成cDNA第一链,合成参照Mir-X miRNAs First-Strand Synthesis Kit说明书(TaKaRa)。根据筛选出的miRNAs设计荧光定量引物,miRNAs上游引物即成熟的miRNAs 序列本身,下游是通用引物(表1)。根据TaKaRa公司的SYBR® Premix ExTaqTMⅡ(Tli RNaseH PLUS)试剂盒说明书配置20 μL反应体系,包括2 μL的cDNA模板,各0.8 μL的上、下游引物,10 μL的2×Premix ExTaqⅡ(Tli RNaseH PLUS)和6.4 μL的ddH2O,每个反应包括3个重复。PCR反应程序为:预变性94 ℃ 30 s;变性94 ℃ 5 s,退火60 ℃ 15 s,延伸72 ℃ 45 s,40个循环。然后进行扩增曲线、熔解曲线分析,检测引物特异性。以黄瓜U6作内参,目的基因的相对表达量计算采用2-ΔΔCT 法。
表1 miRNAs荧光定量PCR引物序列
Tab.1 Primer sequences for miRNAs qPCR

1.5 miRNAs靶基因预测与数据分析
利用PsRNATarget软件预测miRNAs的靶基因,根据miRNAs与相关物种靶mRNA的基因注释,了解差异表达的miRNAs及其靶基因的功能。试验数据采用Excel 2010整理,使用SPSS 19.0进行方差分析。
2 结果与分析
2.1 sRNA的分类注释与长度统计
为了解析miRNAs 在角瓜应对根结线虫胁迫中的作用,利用Solexa测序技术对CmR07和CmS12在接种和未接种根结线虫处理下的18个根系样品(包含3个生物学重复)进行了sRNA测序分析。测序后的原始测序数据去除低质量的测序片段、接头序列、mRNA降解片段以及重复序列后,18份样品获得的unique reads数量在808 262~3 926 308条,所有样品中绝大多数是未知序列,占比高达80%~90%,注释序列中大部分为rRNA,占14%~30%,其次为tRNA,占2%~6%,snRNA和snoRNA占比较少,大约占0.6%~1.5%,miRNAs仅占所有sRNA的0.3%~0.5%左右。随后对各样品中sRNA的长度进行统计,结果显示,所有样品的sRNA长度在24 nt存在一个峰值,即24 nt的sRNA丰度最高(图1)。
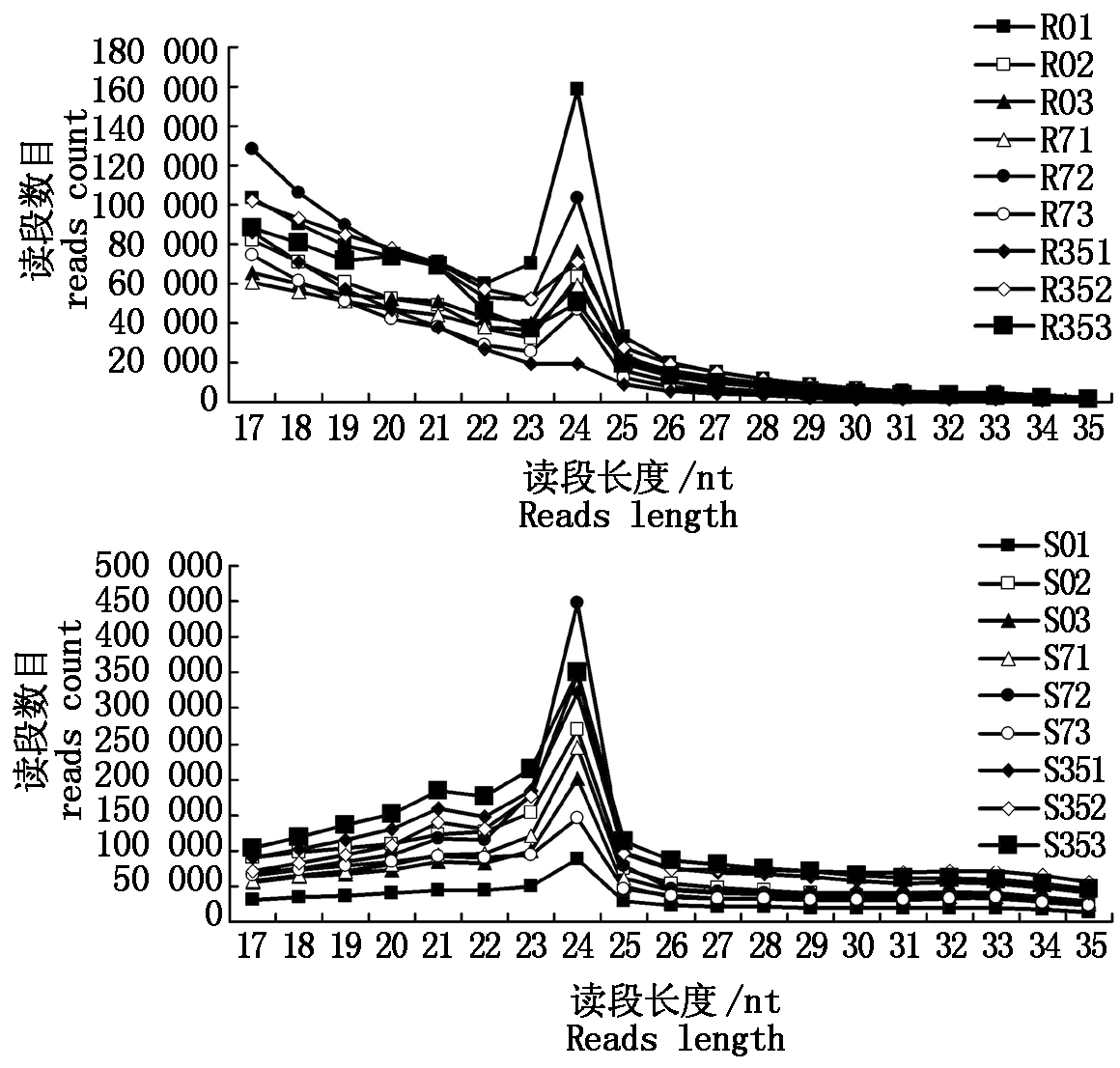
图1 18个根系样品中sRNA(17~35 nt)的长度分布
Fig.1 Sequence length distribution of
17-35 nt small RNAs in 18 root samples
2.2 已知miRNAs的鉴定
为了鉴定各样品中的miRNAs,首先需要去除样品所有sRNA中和miRNAs不相干的reads。首先将sRNA比对到GenBank和Rfam 11.0的数据库中,以获得rRNA、tRNA、snRNA和snoRNA的注释信息,然后进行miRNAs鉴定。将属于同一家族的miRNAs的reads 数进行合并,得到了75个已知miRNAs家族,包含565个miRNAs。每个家族所包含的成员数差别较大,数量在1~51不等,大部分家族(38个家族)仅包含1个成员,有11个家族的成员数超过10个,成员数由多到少依次分别为miR156、miR166、miR159、miR396、miR172、miR171、miR399、miR167、miR477、miR160、miR390,其中miR156包含51个成员,包含的家族成员数目最多。此外,抗病品系CmR07各样本中的miRNAs数量明显少于感病品系CmS12,但同一材料不同样本间的miRNAs的数量差别不大。
2.3 差异表达miRNAs的筛选与表达分析
为了筛选差异表达的miRNAs,首先对miRNAs的表达量进行归一化处理,获得每个miRNAs对应的TPM值,并将各生物学重复之间以平均值进行合并,然后以|log2FoldChange|≥1、FDR≤0.05作为筛选条件,比较分析样本组内miRNAs的表达量变化,共获得2组样本内212个差异表达的miRNAs。为了明确miRNAs的表达模式,对上述筛选获得的差异表达miRNAs进一步严格筛选条件,以|log2FoldChange|≥2、FDR≤0.01作为筛选标准共获得26条miRNAs。根据sRNA测序结果(接种线虫后30 d),将miRNAs响应根结线虫胁迫的表达模式分为4种类型。Type Ⅰ:在CmR07中上调表达而在CmS12中下调表达的miRNAs,包括6条miRNAs:aly-miR165a-3p、crt-miR166b、osa-miR166g-3p、ptc-miR166p、vvi-miR396a、aly-miR172b-5p。Type Ⅱ:在CmR07中下调表达而在CmS12中上调表达的miRNAs,包括5条miRNAs:bna-miR2111c、ath-miR159a、peu-miR2916、cpa-miR319、gma-miR9736。Type Ⅲ:在CmR07和CmS12中均上调表达的miRNAs,包括10条miRNAs:smo-miR156b、atr-miR396d、cme-miR164b、pde-miR396、ptc-miR396f、stu-miR390-3p、ptc-miR396g-5p、smo-miR396、cme-miR399d、aly-miR827-3p。Type Ⅳ:在CmR07和CmS12中均下调表达的miRNAs,包括5条miRNAs:ppt-miR1222e、osa-miR166k-3p、cme-miR858、hbr-miR6173、gma-miR5376。
为了检测sRNA测序结果的可靠性,同时为了获得角瓜与根结线虫非亲和互作密切相关的miRNAs,对上述获得的差异表达miRNAs进一步筛选,从中筛选出不同表达类型、表达量较高的10条miRNAs进行qRT-PCR验证。结果显示,10条差异表达的miRNAs扩增曲线完整,熔解曲线单一。除aly-miR172b-5p(图2-A)、cpa-miR319(图2-B)的部分测序结果与qRT-PCR 结果不相吻合外,其余miRNAs的qRT-PCR 结果与sRNA测序结果的趋势基本一致。在非亲和互作中(CmR07),除aly-miR172b-5p外,其他miRNAs在接种根结线虫后4 d均下调表达,下调幅度最大的分别为smo-miR396(图2-C)和smo-miR156b(图2-D),其余miRNAs的下调幅度较小;接种根结线虫后30 d,除ath-miR159a(图2-E)和cme-miR858(图2-F)外,其他miRNAs均上调表达,其中smo-miR156b(图2-D)上调幅度最大,其次为smo-miR396(图2-C)。在亲和性互作中(CmS12)中,cme-miR164b(图2-G)和aly-miR827-3p(图2-H)在接种根结线虫后4 d均上调表达,但仅有aly-miR827-3p的表达存在显著差异,其余miRNAs均下调表达,其中cpa-miR319(图2-B)下调幅度最大,其次为aly-miR165a-3p(图2-I);接种根结线虫后30 d,除aly-miR165a-3p(图2-I)和cme-miR858(图2-F)外,其他miRNAs均上调表达,但与抗病材料CmR07相比,感病材料CmS12中miRNAs的上调表达幅度较小,其中cpa-miR319(图2-B)和stu-miR390-3p(图2-J)的上调幅度最大。
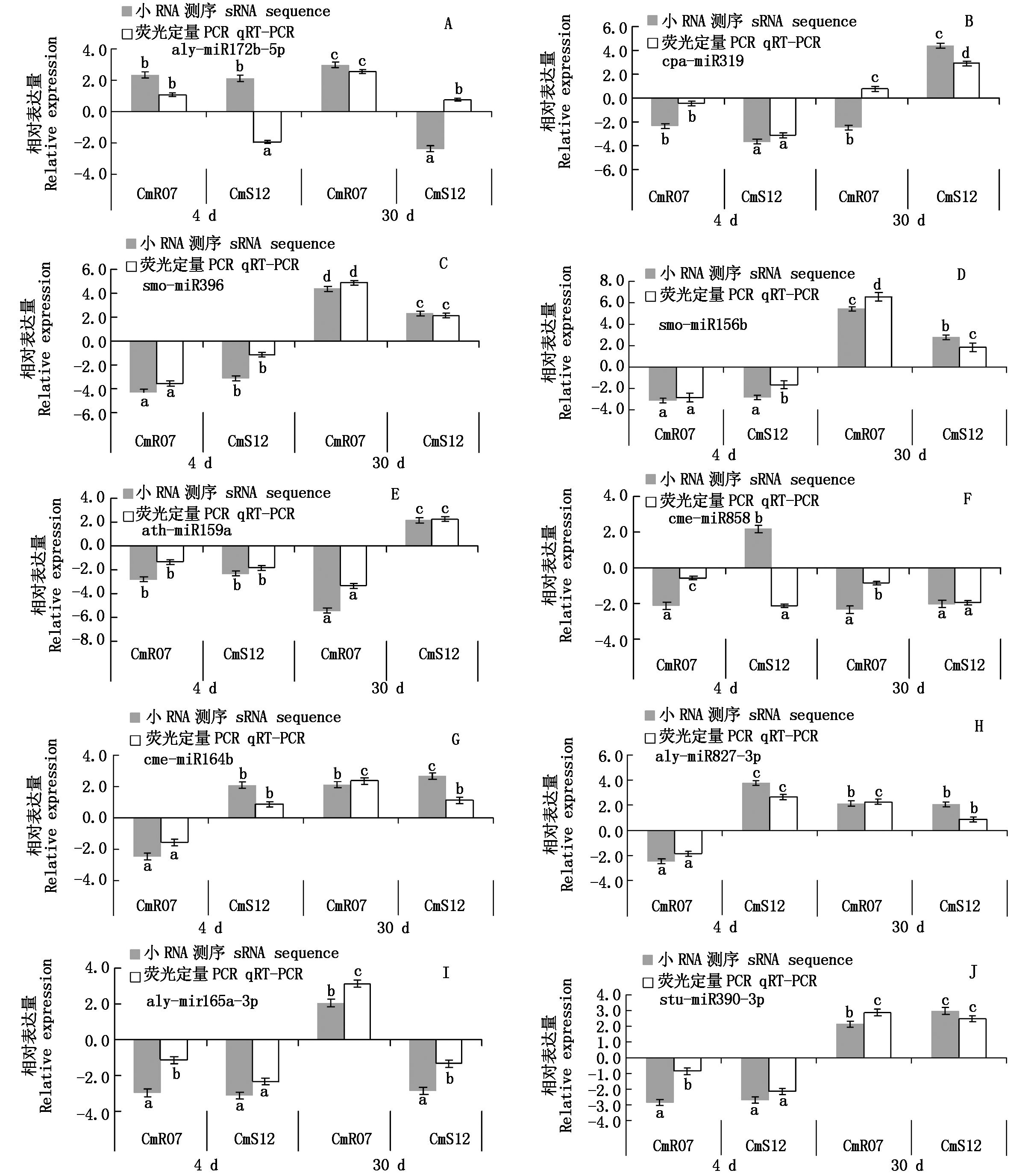
不同小写字母表示差异显著。
Different lowercase indicate significant different.
图2 差异表达miRNAs的qRT-PCR分析
Fig.2 qRT-PCR analysis of miRNAs with differential expression in sRNA sequencing
2.4 miRNAs的靶基因预测与功能注释
利用psRNATarget Finder对筛选获得的具有显著差异表达的10条miRNA进行靶基因预测,并进行功能注释。从表2可以看出,一个miRNAs可调控多个靶基因,其中smo-miR156b、aly-miR172b-5p和smo-miR396的预测靶基因较多,而且同一miRNA的靶基因同源性较高,如smo-miR156b的靶基因主要为编码Squamosa启动子结合蛋白类的基因,而smo-miR396的大部分靶基因为生长调控因子GRF。miRNAs的靶基因功能主要涉及转录激活,如编码亮氨酸拉链蛋白ATHB等的基因,有的靶基因与植物生长发育和调控相关,如转录因子GRF,有的靶基因编码信号传导和细胞代谢相关蛋白,如LRR受体类蛋白激酶AT1G51890等。
表2 差异表达miRNAs及其靶基因
Tab.2 miRNAs with differential expression
and their target genes

miRNAs名称miRNAs name靶基因Target gene基因注释Gene annotationsmo-miR156bCsa_3G117960Squamosa启动子结合蛋白7Csa_1G051590Squamosa启动子结合蛋白13ACsa_3G809420Squamosa启动子结合蛋白9Csa_6G094760Squamosa启动子结合蛋白16Csa_1G015680Squamosa启动子结合蛋白6Csa_1G074980Squamosa启动子结合类蛋白3Csa_4G631590Squamosa启动子结合类蛋白1Csa_5G149870短链脱氢酶TIC32Csa_4G075190阳离子氨基酸转运蛋白8Csa_6G3570301,4-α-葡聚糖分支酶1ath-miR159aCsa_7G043580转录因子MYB104Csa_1G077140CREB调控转录激活子Csa_4G083490BES1/BZR1同源蛋白4Csa_6G105150MYB相关蛋白MYB4Csa_5G182630端粒重复结合蛋白3Csa_2G264010预测酰胺酶C869.01Csa_7G201900阿拉伯糖转移酶ARAD2cme-miR164bCsa_3G523580含有NAC结构域的21/22类蛋白Csa_1G009870含有NAC结构域的100类蛋白aly-miR165a-3pCsa_1G538230亮氨酸拉链蛋白ATHB-14Csa_6G141360亮氨酸拉链蛋白REVOLUTACsa_3G895630亮氨酸拉链蛋白ATHB-8Csa_7G452940亮氨酸拉链蛋白ATHB-15Csa_6G150540组蛋白去乙酰化酶HDT1Csa_3G776860脱落酸非敏性蛋白aly-miR172b-5pCsa_3G073920葡聚糖内酯-1,3-β-葡萄糖苷酶13Csa_2G382620含U盒结构域蛋白12Csa_6G502750五肽重复序列蛋白AT5G66520Csa_1G181360ABC转运体I家族成员20Csa_2G061550含蛋白质胡椒的RNA结合KH结构域Csa_7G374660GPN环GTP酶Csa_6G423430MEI2类蛋白5Csa_4G293860水溶性无机焦磷酸酶4cpa-miR319Csa_6G524000转录因子TCP2Csa_1G045510果胶裂解酶Csa_3G799120锌指蛋白8Csa_4G088720转录因子TCP4Csa_2G006850葡聚糖内-1,3-β-葡萄糖苷酶Csa_2G258850含蛋白质MRL1的五肽重复序列stu-miR390-3pCsa_3G120440含蛋白质AT5G18950的五肽重复序列Csa_6G518210生长素响应因子3Csa_6G507130重金属相关异戊烯化植物蛋白26smo-miR396Csa_6G496380生长调控因子GRF1Csa_6G014490腺嘌呤/鸟嘌呤渗透酶AZG1Csa_3G585360线粒体输入受体亚基TOM20Csa_4G337310含环指和跨膜结构域蛋白2Csa_5G148650含BTB/POZ结构域蛋白AT3G05675Csa_2G000170生长调控因子GRF5Csa_2G432230生长调控因子GRF4Csa_3G751470生长调控因子GRF3Csa_3G124990生长调控因子GRF9Csa_5G623770F盒蛋白AT3G54460Csa_3G149990碱性7S球蛋白类Csa_7G452180LRR受体类蛋白激酶AT1G51890aly-miR827-3pCsa_5G156110含SPX结构域膜蛋白AT4G22990Csa_2G415520反式白藜芦醇二-O-甲基转移酶Csa_6G041190PTI-1类酪氨酸蛋白激酶1cme-miR858Csa_3G182040转录因子LAF1Csa_6G018600丝氨酸/苏氨酸蛋白激酶AT1G54610Csa_3G076520转录因子MYB104Csa_7G375780MYB相关蛋白MYB4
3 讨论
根结线虫是农业生产上的重要病原。根结线虫侵入作物根系后,在维管束内诱导巨型细胞(GCs)进而形成根结(Galls),Galls是由GCs的扩张、邻近维管束组织的增殖以及皮层细胞的肥大形成的[29]。本研究对根结线虫侵染后的整株根系进行了取样,因此,通过Solexa检测到的sRNA可能来源于Galls或GCs或这些细胞类型中的任意一种,获得24 nt的sRNA在根结线虫侵染的根系中占比最多,是测序文库中最丰富的序列,这一结果符合Galls中含有高丰度的24 nt的sRNA这一分子特征[14],这与从孢囊线虫[16]、根结线虫[14]取食位点中获得的sRNA测序结果相一致,说明sRNA和角瓜与根结线虫互作密切相关。陆地棉(Gossypium hirsutum)感染黄萎病菌(Verticillium dahlia)后,同样发现了24 nt的sRNA的富集[30]。24 nt的sRNA可能在GCs从其维管束前体分化的早期阶段通过染色质重塑介导了基因表达的表观遗传调控,进而协调参与GCs和Gall分化的发育进程[14]。
植物miRNAs是在转录和转录后水平对基因表达具有调控作用的sRNA,在应对多种胁迫中发挥重要作用[11]。前人研究表明,miRNAs参与植物-线虫互作的基因表达调控,如在线虫与拟南芥[14]、大豆[18]、番茄[31]、棉花[32]等作物互作中,miRNAs对线虫取食位点的形成发挥调控作用。角瓜对南方根结线虫具有高度抗性[1,7-10],是黄瓜属中为数不多的抗线虫野生资源。因此,探索miRNAs在角瓜对根结线虫抗性中的作用,有助于全面解析角瓜与根结线虫非亲和互作的分子机制。本研究利用角瓜抗线虫品系CmR07和感病品系CmS12,构建了2个品系在2个不同接种时间点的根系sRNA文库,并采用Solexa技术进行了sRNA测序,生物信息学分析在18个sRNA文库中获得的unique reads数量在808 262~3 926 308条,但由于目前尚无角瓜基因组测序信息[9-10],因此,所有样品中绝大多数是未知序列,注释序列中miRNAs仅占sRNA的0.3%~0.5%,最终获得了75个已知的miRNAs家族,包含565个已知miRNAs。这些大数据包含未接种对照和3次生物学重复,为分析鉴定与根结线虫互作相关的miRNAs奠定了基础。
为了鉴定根结线虫侵染过程中的差异表达miRNAs,明确miRNAs的表达模式,本研究首先通过测序分析比较接种与未接种对照miRNAs的表达水平,鉴定出212个响应根结线虫胁迫的差异表达miRNAs。在此基础上,严格筛选条件,选择了10条测序miRNAs通过qRT-PCR进行了表达验证,绝大部分miRNAs的qRT-PCR结果与Solexa测序结果一致,说明本次测序数据可靠。进一步分析发现,在根结线虫侵染前期(4 d),无论亲和(CmS12)还是非亲和互作(CmR07),大部分miRNAs呈现下调表达,说明miRNAs在Galls发育早期普遍下调表达,有利于根结线虫的寄生,这与孢囊线虫侵染拟南芥4 d后合胞体细胞中miRNAs的表达相似[19]。在番茄(Solanum lycopersicum)与南方根结线虫互作中,番茄大部分miRNAs在抗病材料中下调表达,而在感病材料中上调表达,如miR169[33]。相似地,在大豆(Glycine max)与CNs互作中,miR169在感病反应中上调表达而在抗性反应中下调表达[34]。与此不同,本研究中根结线虫侵染后期(30 d),10条miRNAs在抗感品系中表现出了4种不同的表达模式,如aly-miR165a-3p、aly-miR172b-5p在抗性品系CmR07(非亲和互作)中上调表达而在感病品系CmS12(亲和性互作)中下调表达,ath-miR159a、cpa-miR319在CmR07中下调表达而在CmS12中上调表达,smo-miR156b、cme-miR164b、stu-miR390-3p、smo-miR396、aly-miR827-3p在抗感品系中均上调表达,cme-miR858在抗感品系中均下调表达,说明miRNAs在不同植物与线虫互作中的表达模式不同[35]。
miRNAs主要通过抑制mRNAs翻译或降解mRNAs介导靶基因的表达与调控,而研究miRNAs功能需首先进行miRNAs靶基因的预测与鉴定。Kohli等[36]通过高通量测序获得了181个鹰嘴豆(Cicer arietinum)miRNAs,并利用psRNA Target预测到358个miRNAs靶标基因。本研究利用ps RNA Target Finder预测到61个miRNAs靶基因,靶基因功能涉及转录激活、植物生长发育和调控以及编码信号传导和细胞代谢相关蛋白等,为开展角瓜与南方根结线虫非亲和互作相关miRNAs及其靶基因的功能鉴定奠定了基础。
[1] Weng Y Q. Genetic diversity among Cucumis metuliferus populations revealed by cucumber microsatellites[J].HortScience,2010,45(2):214-219.doi:10.21273/hortsci.45.2.214.
[2] Ye D Y,Qian C T,Kurowski C. Identification of a novel source of resistance to the root-knot nematode Meloidogyne Incognita in Cucumis[J].Russian Journal of Nematology,2012,20(1):45-51.
[3] López-Gómez M,Flor-Peregrín E,Talavera M,Verdejo-Lucas S. Suitability of zucchini and cucumber genotypes to populations of Meloidogyne arenaria,M. incognita,and M. javanica[J].Journal of Nematology,2015,47(1):79-85.
[4] Chen J F,Adelberg J. Interspecific hybridization in Cucumis-progress,problems,and perspectives[J].HortScience,2000,35(1):11-15. doi:10.21273/hortsci.35.1.11.
[5] 叶德友,钱春桃,陈劲枫. 抗南方根结线虫黄瓜-酸黄瓜渐渗系的筛选及鉴定[J].园艺学报,2011,38(12):2281-2288.doi:10.16420/j.issn.0513-353x.2011.12.012.
Ye D Y,Qian C T,Chen J F. Screening and identification of cucumber-sour cucumber introgression lines resistant to the root-knot nematode Meloidogyne incognita[J].Acta Horticulturae Sinica,2011,38(12):2281-2288.
[6] de Souza Galatti F,Franco A J,Ito L A,de Oliveira Charlo H,Gaion L A,Braz L T. Rootstocks resistant to Meloidogyne incognita and compatibility of grafting in net melon[J].Revista Ceres,2013,60(3):432-436.doi:10.1590/s0034-737x2013000300018.
[7] 马金慧,茆振川,李惠霞,谢丙炎. 刺角瓜对南方根结线虫的抗性及特征分析[J].园艺学报,2014,41(1):73-79. doi:10.16420/j.issn.0513-353x.2014.01.011.
Ma J H,Mao Z C,Li H X,Xie B Y. Resistance identification of Cucumis metuliferus to Meloidogyne incognita and characteristic analysis[J].Acta Horticulturae Sinica,2014,41(1):73-79.
[8] 魏偲,史倩倩,马玉琴,马金慧,茆振川,凌键,杨宇红,谢丙炎. 不同温度下刺角瓜过氧化物酶基因的表达及其对抗南方根结线虫作用的影响[J].园艺学报,2016,43(8):1537-1544.doi:10.16420/j.issn.0513-353x.2016-0233.
Wei C,Shi Q Q,Ma Y Q,Ma J H,Mao Z C,Ling J,Yang Y H,Xie B Y. Effects of peroxidase gene to the resistance of Cucumis metuliferus against Meloidogyne incognita in different temperature[J].Acta Horticulturae Sinica,2016,43(8):1537-1544.
[9] Ye D Y,Qi Y H,Cao S F,Wei B Q,Zhang H S. Histopathology combined with transcriptome analyses reveals the mechanism of resistance to Meloidogyne incognita in Cucumis metuliferus[J].Journal of Plant Physiology,2017,212:115-124. doi:10.1016/j.jplph.2017.02.002.
[10] Ling J,Mao Z C,Zhai M J,Zeng F,Yang Y H,Xie B Y. Transcriptome profiling of Cucumis metuliferus infected by Meloidogyne incognita provides new insights into putative defense regulatory network in Cucurbitaceae[J].Scientific Reports,2017,7:3544. doi:10.1038/s41598-017-03563-6.
[11] Li R J,Rashotte A M,Singh N K,Weaver D B,Lawrence K S,Locy R D. Integrated signaling networks in plant responses to sedentary endoparasitic Nematodes:A perspective[J].Plant Cell Reports,2015,34(1):5-22. doi:10.1007/s00299-014-1676-6.
[12] Hewezi T,Baum T J. Gene silencing in nematode feeding sites[J].Advances in Botanical Research,2015,73:221-239. doi:10.1016/bs.abr.2014.12.007.
[13] Shukla N,Kaur P,Kumar A. Molecular aspects of plant-nematode interactions[J].Indian Journal of Plant Physiology,2016,21(4):477-488.doi:10.1007/s40502-016-0263-y.
[14] Cabrera J,Barcala M,García A,Rio-Machín A,Medina C,Jaubert-Possamai S,Favery B,Maizel A,Ruiz-Ferrer V,Fenoll C,Escobar C. Differentially expressed small RNAs in Arabidopsis galls formed by Meloidogyne javanica:A functional role for miR390 and its TAS3-derived tasiRNAs[J].The New Phytologist,2016,209(4):1625-1640.doi:10.1111/nph.13735.
[15] Ali M,Anjam M,Nawaz M,Lam H M,Chung G. Signal transduction in plant nematode interactions[J].International Journal of Molecular Sciences,2018,19(6):1648. doi:10.3390/ijms19061648.
[16] Hewezi T,Maier T R,Nettleton D,Baum T J. The Arabidopsis MicroRNA396-GRF1/GRF3 regulatory module Acts as a developmental regulator in the reprogramming of root cells during cyst nematode infection[J].Plant Physiology,2012,159(1):321-335.doi:10.1104/pp.112.193649.
[17] Xu M Y,Li Y H,Zhang Q X,Xu T,Qiu L J,Fan Y L,Wang L. Novel miRNA and phasiRNA biogenesis networks in soybean roots from two sister lines that are resistant and susceptible to SCN race 4[J].PLoS One,2014,9(10):e110051. doi:10.1371/journal.pone.0110051.
[18] Tian B,Wang S C,Todd T C,Johnson C D,Tang G L,Trick H N. Genome-wide identification of soybean microRNA responsive to soybean cyst Nematodes infection by deep sequencing[J].BMC Genomics,2017,18(1):572. doi:10.1186/s12864-017-3963-4.
[19] Hewezi T,Howe P,Maier T R,Baum T J. Arabidopsis small RNAs and their targets during cyst nematode parasitism[J].Molecular Plant-Microbe Interactions ,2008,21(12):1622-1634. doi:10.1094/mpmi-21-12-1622.
[20] Hewezi T,Piya S,Qi M S,Balasubramaniam M,Rice J H,Baum T J.Arabidopsis miR827 mediates post-transcriptional gene silencing of its ubiquitin E3 ligase target gene in the syncytium of the cyst nematode Heterodera schachtii to enhance susceptibility[J].The Plant Journal,2016,88(2):179-192. doi:10.1111/tpj.13238.
[21] Piya S,Kihm C,Rice J H,Baum T J,Hewezi T. Cooperative regulatory functions of miR858 and MYB83 during cyst nematode parasitism[J].Plant Physiology,2017,174(3):1897-1912. doi:10.1104/pp.17.00273.
[22] Zhao W C,Li Z L,Fan J W,Hu C L,Yang R,Qi X,Chen H,Zhao F K,Wang S H. Identification of jasmonic acid-associated microRNAs and characterization of the regulatory roles of the miR319/TCP4 module under root-knot nematode stress in tomato[J].Journal of Experimental Botany,2015,66(15):4653-4667.doi:10.1093/jxb/erv238.
[23] Curaba J,Singh M B,Bhalla P L. miRNAs in the crosstalk between phytohormone signalling pathways[J].Journal of Experimental Botany,2014,65(6):1425-1438.doi:10.1093/jxb/eru002.
[24] Schommer C,Debernardi J M,Bresso E G,Rodriguez R E,Palatnik J F. Repression of cell proliferation by miR319-regulated TCP4[J].Molecular Plant,2014,7(10):1533-1544. doi:10.1093/mp/ssu084.
[25] Medina C,da Rocha M,Magliano M,Ratpopoulo A,Revel B,Marteu N,Magnone V,Lebrigand K,Cabrera J,Barcala M,Silva A C,Millar A,Escobar C,Abad P,Favery B,Jaubert-Possamai S. Characterization of microRNAs from Arabidopsis galls highlights a role for miR159 in the plant response to the root-knot nematode Meloidogyne incognita[J].The New Phytologist,2017,216(3):882-896. doi:10.1111/nph.14717.
[26] Ling J,Jiang W J,Zhang Y,Yu H J,Mao Z C,Gu X F,Huang S W,Xie B Y. Genome-wide analysis of WRKY gene family in Cucumis sativus[J].BMC Genomics,2011,12:471. doi:10.1186/1471-2164-12-471.
[27] Mao W H,Li Z Y,Xia X J,Li Y D,Yu J Q. A combined approach of high-throughput sequencing and degradome analysis reveals tissue specific expression of MicroRNAs and their targets in cucumber[J].PLoS One,2012,7(3):e33040. doi:10.1371/journal.pone.0033040.
[28] Hu J H,Sun L L,Zhu Z X,Zheng Y,Xiong W,Ding Y. Characterization of conserved microRNAs from five different cucurbit species using computational and experimental analysis[J].Biochimie,2014,102:137-144. doi:10.1016/j.biochi.2014.03.002.
[29] Escobar C,Barcala M,Cabrera J,Fenoll C. Overview of root-knot Nematodes and giant cells[J].Advances in Botanical Research,2015,73:1-32.doi:10.1016/bs.abr.2015.01.001.
[30] Yang X Y,Wang L C,Yuan D J,Lindsey K,Zhang X L. Small RNA and degradome sequencing reveal complex miRNA regulation during cotton somatic embryogenesis[J].Journal of Experimental Botany,2013,64(6):1521-1536.doi:10.1093/jxb/ert013.
[31] Koter M D,![]() wi
wi cicka M,Matuszkiewicz M,Pacak A,Derebecka N,Filipecki M. The miRNAome dynamics during developmental and metabolic reprogramming of tomato root infected with potato cyst nematode[J].Plant Science,2018,268:18-29.doi:10.1016/j.plantsci.2017.12.003.
cicka M,Matuszkiewicz M,Pacak A,Derebecka N,Filipecki M. The miRNAome dynamics during developmental and metabolic reprogramming of tomato root infected with potato cyst nematode[J].Plant Science,2018,268:18-29.doi:10.1016/j.plantsci.2017.12.003.
[32] Pan X P,Nichols R L,Li C,Zhang B H. MicroRNA-target gene responses to root knot nematode(Meloidogyne incognita)infection in cotton(Gossypium hirsutum L.)[J].Genomics,2019,111(3):383-390.doi:10.1016/j.ygeno.2018.02.013.
[33] Kaur P,Shukla N,Joshi G,VijayaKumar C,Jagannath A,Agarwal M,Goel S,Kumar A. Genome-wide identification and characterization of miRNAome from tomato(Solanum lycopersicum)roots and root-knot nematode(Meloidogyne incognita)during susceptible interaction[J].PLoS One,2017,12(4):e0175178.doi:10.1371/journal.pone.0175178.
[34] Li X Y,Wang X,Zhang S P,Liu D W,Duan Y X,Dong W. Identification of soybean microRNAs involved in soybean cyst nematode infection by deep sequencing[J].PLoS One,2012,7(6):e39650.doi:10.1371/journal.pone.0039650.
[35] Siddique S,Grundler F M. Parasitic Nematodes manipulate plant development to establish feeding sites[J].Current Opinion in Microbiology,2018,46:102-108. doi:10.1016/j.mib.2018.09.004.
[36] Kohli D,Joshi G,Deokar A A,Bhardwaj A R,Agarwal M,Katiyar-Agarwal S,Srinivasan R,Jain P K. Identification and characterization of wilt and salt stress-responsive microRNAs in chickpea through high-throughput sequencing[J].PLoS One,2014,9(10):e108851.doi:10.1371/journal.pone.0108851.
