中国是栗属植物的起源中心,主要分布有板栗、茅栗、锥栗和日本栗4个种,是世界上栗属植物分布最广泛的区域[1]。燕山地区是中国板栗的主要产区,包括北京市怀柔区、密云区和河北省的遵化、迁安、迁西、兴隆、青龙和宽城等市县。得益于得天独厚的地理条件和气候优势,燕山板栗以口味甘甜、肉质细糯、品质优良以及适宜糖炒等优良特性享誉国内外市场。
燕山地域独特的生境条件和长久的栽培历史使得燕山板栗拥有丰富独特的表型性状,为板栗遗传特点研究和种质改良提供了材料和基因来源[2]。如何从数量繁多的种质资源中快速准确地挖掘出育种研究所需要的优异基因是板栗遗传育种工作所需解决的现实问题。此外,板栗是树体高大的多年生木本植物,占地面积大,管理费用高,而当前资源保存通常以活体种植保存为主,随着资源数量不断增加,给资源保存单位带来一定困难,耗费人力、金钱和时间。据此,提出了构建燕山板栗核心种质的策略,一方面为燕山板栗资源开发和种质创新奠定基础,提高资源的有效利用率,另一方面通过利用科学的方法探索最优化的燕山板栗样本保存策略,提高资源的保存效率。
目前,植物上主要基于农艺、形态等表型性状数据或分子标记信息来构建核心种质[3-4]。分子标记如RAPD[5-6]、AFLP[7-8]和SSR[9-10]具有高效、快速、稳定的优点,常应用于栗属植物遗传多样性评价与核心种质构建当中[11]。程丽莉[12]利用AFLP标记对燕山区136份实生板栗进行遗传多样性分析,初选37份材料为核心种质;马玉敏[13]以120份中国板栗野生株系为材料,探讨了采用荧光AFLP标记构建野生板栗核心种质的方法。有关燕山板栗遗传多样性分析和核心种质构建研究有待进一步完善,前期研究选用样本数量有限且取样范围相对狭窄,没有涵盖燕山板栗几个主要产区,遗传信息缺乏丰富性。本研究利用SSR标记,以来源燕山板栗主产区10个市(县)的161份资源为材料,分别利用SM系数、Dice系数和Jaccard系数进行UPGMA聚类,比较位点优先取样法和随机取样法的不同,确定构建燕山板栗核心种质的适宜方法,并筛选出燕山板栗核心种质,以期为板栗种质资源的管理、保存和开发利用提供科学依据,从而有效提高整个行业资源利用率和育种效率。
1 材料和方法
1.1 试验材料
供试植株定植于河北省农林科学院昌黎果树研究所板栗种质资源圃(119°15′E,39°72′N,属温带半湿润大陆性气候区,年平均气温11 ℃,年平均降水量638 mm,无霜期186 d)。所有资源均是在燕山地区分布范围内选取的实生良种、地方品种或古树资源,2004年统一嫁接入圃保存。供试材料为来自青龙(25份)、抚宁(4份)、平泉(5份)、遵化(17份)、迁安(4份)、昌黎(14份)、迁西(30份)、宽城(24份)、兴隆和(26份)北京(12份)10个市(县)的161份板栗种质。2018年5-6月,每个植株选取新鲜健康嫩叶5片,置入装有硅胶的塑封袋带回实验室,在室温下保存备用。
1.2 SSR分析
本研究从220对引物中筛选出扩增产物多态性较好、条带清晰的21对引物用于全部样品分析,引物具体信息、基因组DNA提取方法和SSR扩增程序参见本课题组前期研究内容[14]。
1.3 核心种质的构建
利用NTSYS Version 2.10软件,分别采用SM遗传相似系数(GSSM)、Dice相似系数(GSD)和Jaccard相似系数(GSJ)基于UPGMA法对扩增结果进行聚类分析。SM系数、Dice系数和Jaccard系数是一种相似性配对系数,即相似匹配数与总匹配数的比值[15]。
GSSM=(a+d)/(b+c)[16],GSD=2a/(2a+b+c)[17],GSJ=a/(a+b+c)[18]
其中,a表示品种i、j共有条带,b表示品种i特有条带,c表示品种j特有条带,d表示品种i、j共同缺失条带。
本研究以随机取样法为对照,应用位点优先取样策略采取多次聚类的方法构建核心种质[19]。位点优先取样策略即根据聚类树状图,在最低分类水平的2份材料中选取具有较多稀有等位基因数(等位基因频率小于5%)或稀有等位基因频率值更小的材料进入下一轮聚类,如果这2个值相等则随机选择一份材料,若组内只有一个材料则直接进入下一轮聚类。随机取样法即在最低分类水平的2份材料中随机选取一个进入下一轮聚类。进行聚类抽样时保证每个市(县)都有种质被抽取,多次聚类直到某地再次聚类时无种质被抽取。
1.4 核心种质有效性分析检验
对初始种质、保留种质和核心种质有效等位基因数(Ne)、Nei′s遗传多样性指数(H)、Shannon′s信息指数(I)进行t检验,评价核心种质的代表性。以上数据采用POPGENE Version 1.32和SPSS 20.0软件进行分析。
1.5 核心种质的确认
采用主坐标分析法和表型数据分析对构建的核心种质进行确认。基于SSR数据信息,利用NTSYS Version 2.10软件中的EIGEN模块对核心种质的代表性进行主坐标分析,判定核心种质是否在主坐标图中均匀分布。
使用数显游标卡尺和电子天平对板栗坚果纵径、坚果横径、厚度、单粒质量4个数量性状进行测定,并根据公式计算果形指数(坚果纵径/坚果横径)。参照Hu等[20]评价方法,用均值差异百分率(MD%)、方差差异百分率(VD%)、极差符合率(CR)和变异系数变化率(VR)来评价核心种质的代表性。若均值差异百分率小于20%,同时极差符合率大于80%,可认为核心种质能代表原种质资源的遗传多样性。
其中,n为数量性状总数,St为核心种质与原种质t测验得到的均值差异显著的性状数,SF为核心种质与原种质F测验得到的方差差异显著的性状数,RC为核心种质的极差,RI为原种质的极差,CVC为核心种质的变异系数,
2 结果与分析
2.1 SSR分子标记遗传多样性
采用SSR标记对161份板栗种质资源进行遗传多样性分析(表1),21对SSR引物检测到71个等位基因,每对引物检测到的等位基因数变化为2~6,平均3.38个,各引物的Nei′s多样性指数为0.135 5~0.427 2,平均为0.301 3,Shannon′s信息指数为0.235 4~0.615 3,平均为0.463 4,表明燕山板栗种质资源具有较丰富的遗传多样性。
表1 161份燕山板栗种质遗传多样性参数
Tab.1 The genetic diversity parameters of Castanea
mollissima germplasm resources in Yanshan Region
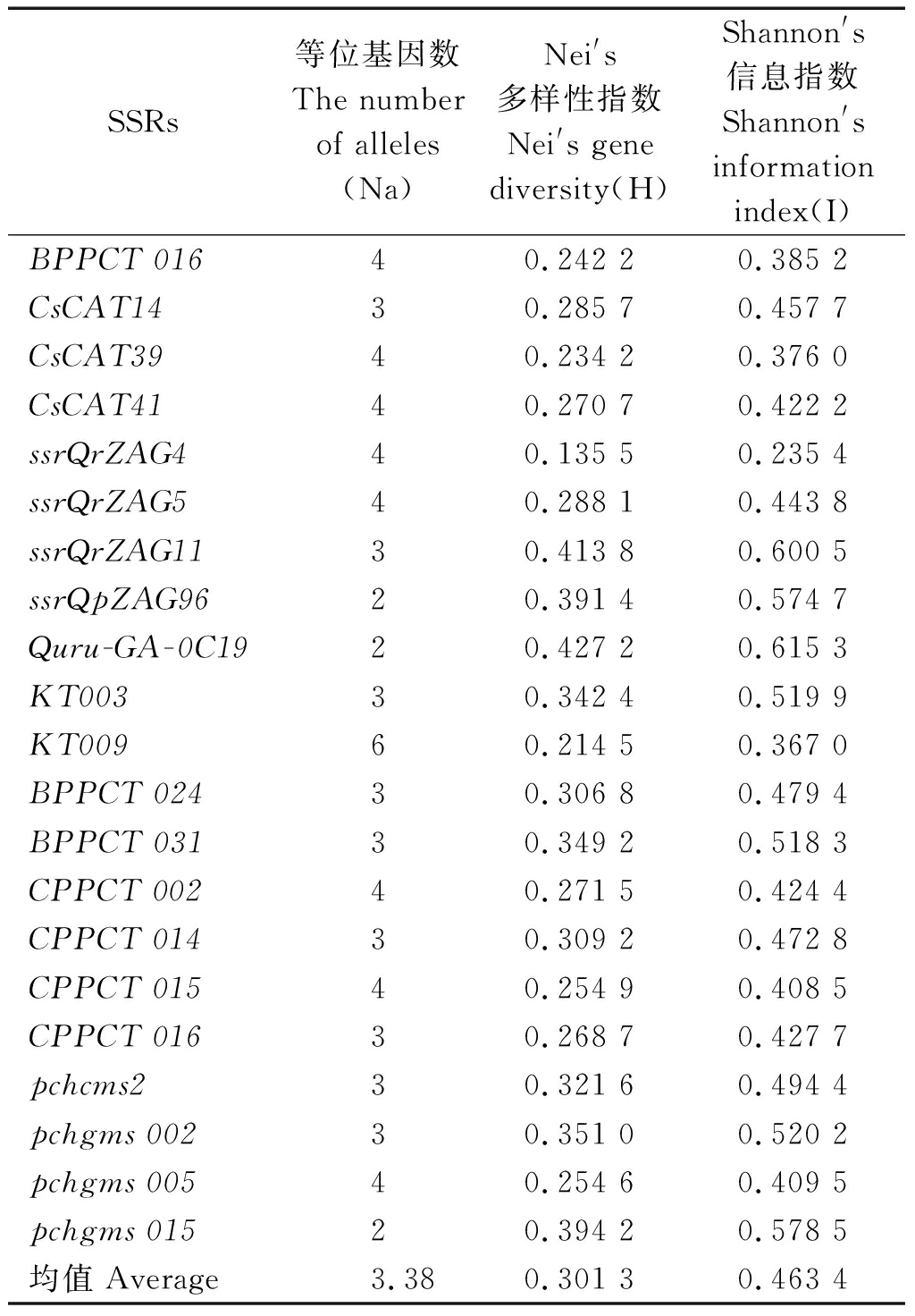
SSRs等位基因数The number of alleles(Na)Nei's多样性指数Nei's gene diversity(H)Shannon's信息指数Shannon's information index(I)BPPCT 01640.242 20.385 2CsCAT1430.285 70.457 7CsCAT3940.234 20.376 0CsCAT4140.270 70.422 2ssrQrZAG440.135 50.235 4ssrQrZAG540.288 10.443 8ssrQrZAG1130.413 80.600 5ssrQpZAG9620.391 40.574 7Quru-GA-0C1920.427 20.615 3KT00330.342 40.519 9KT00960.214 50.367 0BPPCT 02430.306 80.479 4BPPCT 03130.349 20.518 3CPPCT 00240.271 50.424 4CPPCT 01430.309 20.472 8CPPCT 01540.254 90.408 5CPPCT 01630.268 70.427 7pchcms230.321 60.494 4pchgms 00230.351 00.520 2pchgms 00540.254 60.409 5pchgms 01520.394 20.578 5均值Average3.380.301 30.463 4
2.2 核心种质构建
由表2可见,无论采用SM相似系数或Dice相似系数或Jaccard相似系数进行UPGMA聚类,均为位点优先取样法得到的种质有效等位基因数(Ne)、Nei′s多样性指数(H)和Shannon′s信息指数(I)高于随机取样法,说明从遗传多样性角度考虑构建燕山板栗核心种质时位点优先取样法要优于随机取样法。此外,利用SM系数多次聚类取得的种质Ne、H和I均高于原种质,利用Dice系数或Jaccard系数得到的种质遗传多样性指数低于原种质。
表2 板栗各种质遗传多样性比较
Tab.2 Genetic diversity analysis of different collections of China chestnut
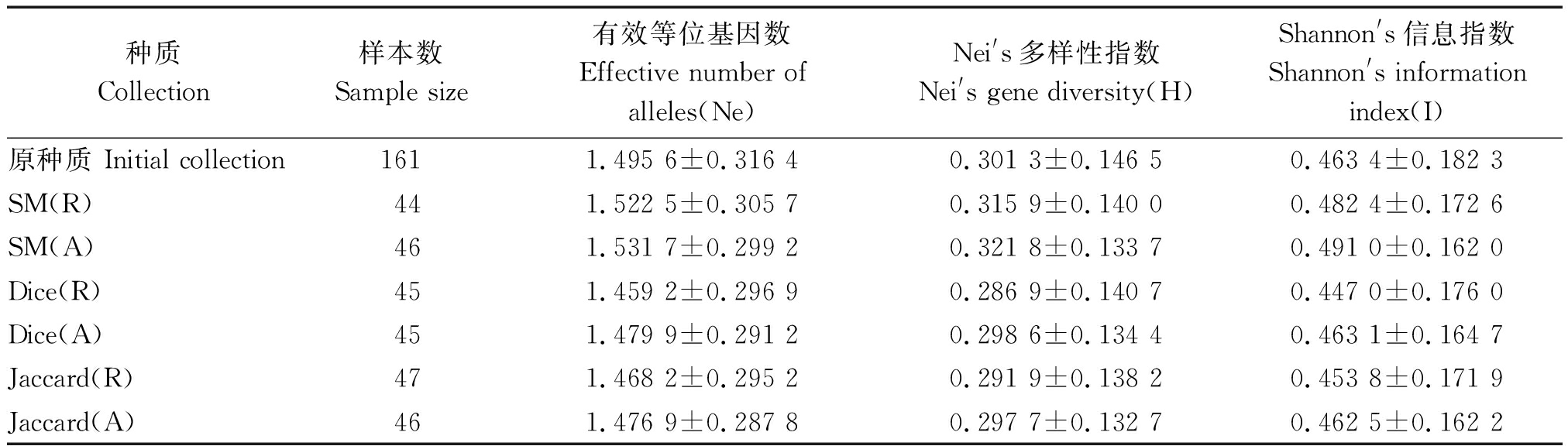
种质Collection样本数Sample size有效等位基因数Effective number of alleles(Ne)Nei's多样性指数Nei's gene diversity(H)Shannon's信息指数Shannon's information index(I)原种质 Initial collection1611.495 6±0.316 40.301 3±0.146 50.463 4±0.182 3SM(R)441.522 5±0.305 70.315 9±0.140 00.482 4±0.172 6SM(A)461.531 7±0.299 20.321 8±0.133 70.491 0±0.162 0Dice(R)451.459 2±0.296 90.286 9±0.140 70.447 0±0.176 0Dice(A)451.479 9±0.291 20.298 6±0.134 40.463 1±0.164 7Jaccard(R)471.468 2±0.295 20.291 9±0.138 20.453 8±0.171 9 Jaccard(A)461.476 9±0.287 80.297 7±0.132 70.462 5±0.162 2
注:A.位点优先取样法;R.随机取样法。
Note:A.Allele preferred sampling method;R.Random sampling method.
表3 核心种质和保留种质、原种质遗传多样性对比
Tab.3 Genetic diversity comparison and t-test between core collection,reserved collection and initial collection
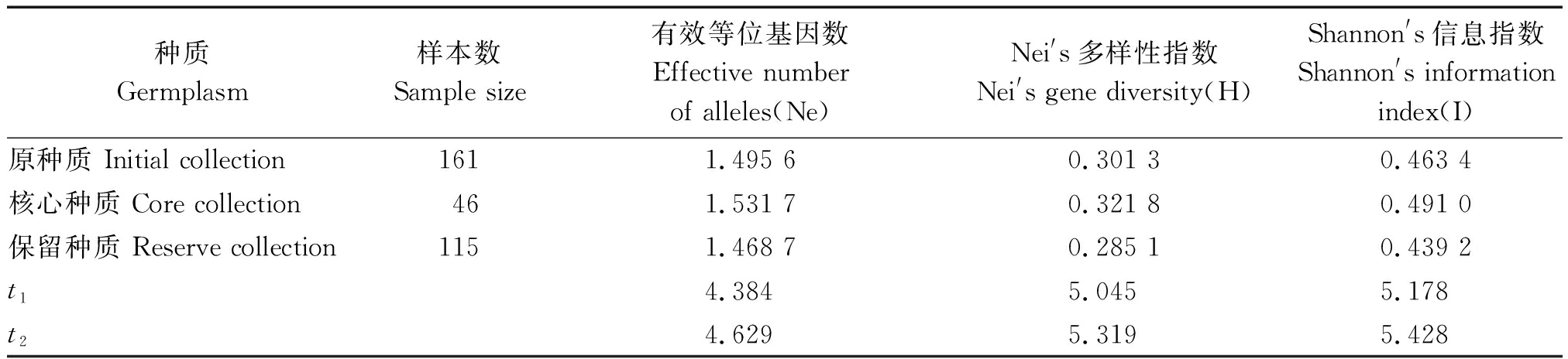
种质Germplasm样本数Sample size有效等位基因数Effective number of alleles(Ne)Nei's多样性指数Nei's gene diversity(H)Shannon's信息指数Shannon's information index(I)原种质 Initial collection1611.495 60.301 30.463 4核心种质 Core collection461.531 70.321 80.491 0保留种质 Reserve collection1151.468 70.285 10.439 2t14.384 5.0455.178t24.6295.3195.428
注:t1.核心种质与原种质各遗传参数t检验值;t2.核心种质与保留种质各遗传参数t检验值。
Note:t1 .The t value of genetic diversity parameters between core collection and initial collection;t2.The t value of genetic diversity parameters between core collection and reserve collection.
利用SM遗传相似系数和位点优先取样法进行多次聚类抽样,得到的种质SM(A)遗传多样性指数(Ne、H、I)最高,所以由46份资源构成的核心样本群SM(A)为核心种质资源,有效等位基因数(Ne)、Nei′s多样性指数(H)和Shannon′s信息指数(I)分别为1.531 7,0.321 8,0.491 0。
2.3 核心种质有效性分析检验
保留种质指从原种质中除去核心种质后剩余的部分,它可以作为核心种质的后备资源。对核心种质、原种质和保留种质的遗传多样性进行评价,以此判定核心种质代表性。核心种质和保留种质各遗传多样性指标及保留率见表3。结果显示,核心种质保留了原种质28.57%的样品,Ne、H和I分别为1.531 7,0.321 8和0.491 0,均高于原种质遗传多样性参数值,由此可见,核心种质在遗传多样性上能较好地代表原种质,可以作为燕山板栗核心种质资源。保留种质保留了原种质71.43%的样品,Ne、H和I分别为1.468 7,0.285 1和0.4392。再对核心种质和原种质、核心种质和保留种质的Ne、H和I值分别作t检验,核心种质Ne、H和I在概率0.05水平上显著大于原种质和保留种质,说明在进行遗传研究时优先选择核心种质会具有更丰富的遗传多样性。
2.4 核心种质主坐标分析
采用主坐标分析法对核心种质进行确认(图1),发现核心种质在原种质全部资源的主坐标图分布均匀,表明筛选出的核心种质具有较好的代表性。
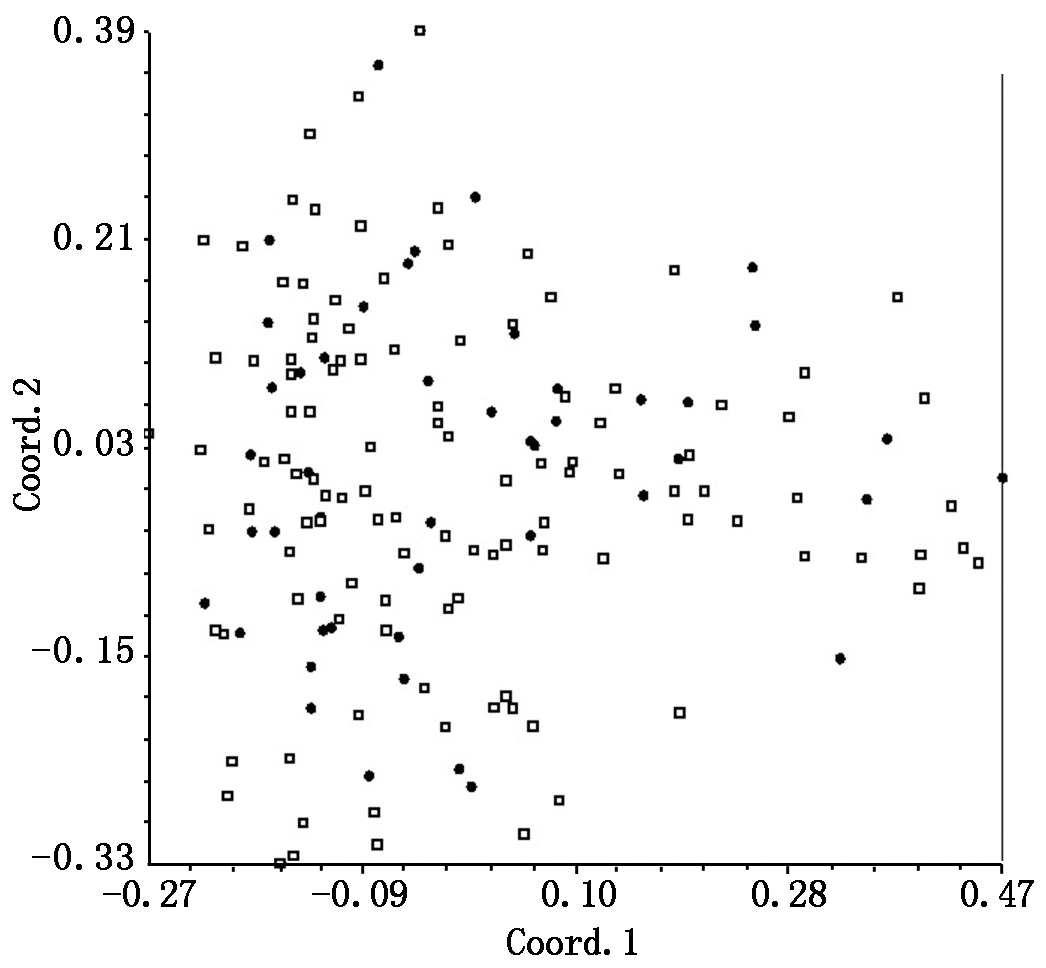
□.原有种质;●.核心种质;横纵坐标分别表示贡献率
较高的1、2主坐标。
□.Initial collection;●.Core colletion;Horizontal and vertical
coordinates mean the first two principal
coordinates with higher contribution rate.
图1 核心种质与原种质的主坐标分布图
Fig.1 Principal coordinates plots of core collection
and initial collection
2.5 核心种质表型性状确认
核心种质与原种质5个表型特征数据见表4。根据公式计算出均值差异百分率(MD)、方差差异百分率(VD)、极差符合率(CR)和变异系数变化率(VR)分别为20.00%,0,86.75%和97.92% 。符合胡晋等[21]提出的核心种质均值同原种质群体存在显著差异的性状小于20%、核心种质与原种质的极差符合率大于80%作为燕山板栗核心种质代表原群体遗传变异的最低标准。
表4 核心种质与原种质5个坚果表型特征基本参数
Tab.4 Basic parameter value of 5 phenotypic traits of core collection and initial collection
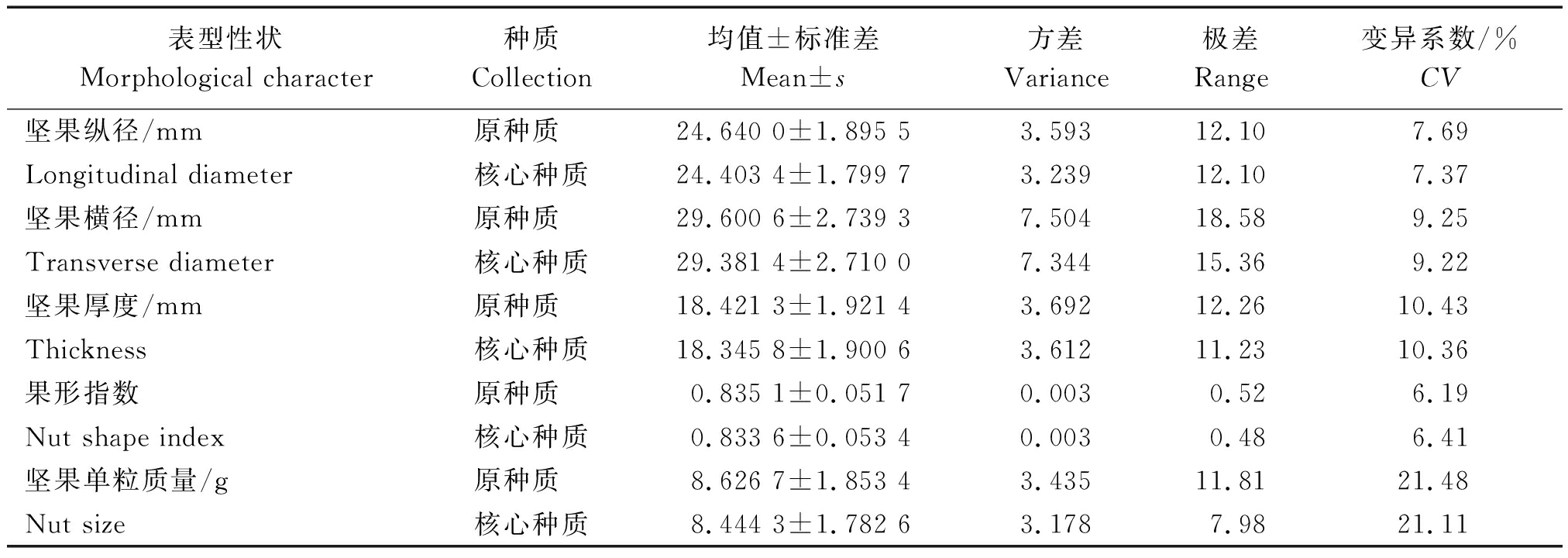
表型性状Morphological character种质Collection均值±标准差Mean±s方差Variance极差Range变异系数/%CV坚果纵径/mm原种质 24.640 0±1.895 53.59312.107.69Longitudinal diameter核心种质24.403 4±1.799 73.23912.107.37坚果横径/mm 原种质 29.600 6±2.739 37.50418.589.25Transverse diameter核心种质29.381 4±2.710 07.34415.369.22坚果厚度/mm 原种质 18.421 3±1.921 43.69212.2610.43Thickness核心种质18.345 8±1.900 63.61211.2310.36果形指数原种质 0.835 1±0.051 70.0030.526.19Nut shape index核心种质0.833 6±0.053 40.0030.486.41坚果单粒质量/g原种质 8.626 7±1.853 43.43511.8121.48Nut size核心种质8.444 3±1.782 63.1787.9821.11
3 讨论
3.1 核心种质构建方法
核心种质的构建是近几年种质资源研究的热门问题。目前,主要基于表型特征数据或分子信息数据来构建植物核心种质[22-24]。表型性状是多种因素综合作用的结果,容易受到外界环境影响。板栗是高大的多年生植物,要获得可靠的表型特征数据需要较长时间。DNA分子标记具有高效、快速、稳定的特点,不易受到外界环境的影响,是评价植物遗传多样性和构建核心种质的有效手段。因此,本研究选择利用SSR分子标记技术构建燕山板栗核心种质资源,SSR标记是共显性遗传,能直接反映出DNA序列水平上的变化,并提供丰富的等位基因信息,较其他标记具有更大优势[25]。
聚类分析近几年被广泛应用于品种分子鉴别、种质间亲缘关系评价及遗传多样性研究方面。遗传相似系数的选择对聚类分析至关重要,基于不同遗传相似系数进行聚类分析,结果也会存在较大差异。本研究采用UPGMA法进行聚类抽样,具有最大遗传相似系数的两份材料往往最先聚在一起,去除相近材料后可得到遗传特性差异较大的种质。本研究通过比较利用不同遗传相似系数(SM系数、Dice系数和Jaccard系数)聚类取样获得样本的遗传多样性指标,发现利用SM系数聚类得到的种质有效等位基因数(Ne)、Nei′s多样性指数(H)和Shannon′s信息指数(I)显著高于利用Dice系数和Jaccard系数聚类取得的样本,说明基于SSR分子标记构建燕山板栗核心种质时使用SM系数要优于Dice系数和Jaccard系数。刘娟等[26]研究表明,利用ISSR分子标记构建新疆野杏核心种质时采用SM遗传系数要优于Dice遗传系数和Jaccard遗传系数;张春雨等[19]构建新疆野苹果核心种质时提出了显性分子标记如AFLP和RAPD多使用单匹配相似系数SM,而共显性分子标记如SSR和RFLP采用Dice和Jaccard系数更理想。本研究与前人研究结果不太一致,分析原因一方面可能是不同研究选择的分子标记不同,与之相匹配的遗传相似系数也会有所差异;另一方面,基于遗传相似系数的聚类分析虽在一定程度上能反映材料间亲缘关系的远近,但是不同植物具有自身独特的遗传特性,对遗传相似系数的应用不能固定化和格式化,应根据物种遗传结构和实际工作而定。
取样策略即取样方法是核心种质构建的又一重要环节。研究表明,通常不推荐采用在整个群体内完全随机的方法进行取样,因为一个物种的遗传多样性是以某种特定组织或结构分布,而不是完全随机地分布在整个群体中[27-28]。李自超等[29]发现构建水稻核心种质时聚类法要优于随机法。Quero-Garcia等[30]在芋头核心种质构建研究中,比较了完全随机法取样、组内完全随机法取样和组内先聚类后取样的方法,结果表明,组内先聚类后取样是一种比前两者更加有效的方法。参照前人的研究经验,本研究即采用先聚类后取样的方法。结果表明,无论利用何种遗传相似系数(SM系数、Dice系数和Jaccard系数)聚类,通过位点优先法取得的样本均比随机取样法得到的样本具有较大的有效等位基因数(Ne)、Nei′s多样性指数(H)和Shannon′s信息指数(I),说明构建燕山板栗核心种质时位点优先法优于随机取样法,与前人研究结果一致[19,31]。原因可能是生物的基因多样性是决定其表型特征的遗传因子,所有位点的等位基因频率和等位基因数目构成了生物基因多样性的基础。因此,可以认为等位基因数目和频率承载着生物的遗传多样性,尤其是稀有等位基因可能具有某种有利特征来帮助该物种适应不同的环境变化,防止群体衰落,在维持生物多样性方面具有重要意义[32-34]。因此建议,构建核心种质时优先选取稀有基因(等位基因频率小于5%),同时尽可能地保留原种质等位基因的多样性,这样既可以减少等位基因的丢失,又尽可能保留原种质群体的基因多样性。
3.2 核心种质取样比例
样本量的规模是判定核心种质是否合理有效的重要因子。核心种质能以最小的资源份数最大限度地代表原种质的遗传多样性,因此,确定合理恰当的取样比例十分关键。李自超等[35]提出应根据具体植物的遗传结构和数量规模来确定相应的取样比例。作物群体一般原种质资源数量较多,选取较小规模的核心样品也能充分保留原种质的遗传特性,如稻类等农作物核心样品一般占总收集品的10%左右[36]。目前国内外构建的林木资源核心种质取样规模一般在10%~40%[37-39]。取样比例应根据具体树种遗传特性和群体资源数量等实际工作而定,对遗传多样性丰富、原种质资源数量庞大的群体可适当减小取样比例。本研究选取了46份燕山板栗核心种质,保留了原种质28.57%的样品,核心种质来源地范围涵盖原种质所有来源地,经t检验核心种质能较充分代表原种质的遗传多样性。此外,本研究从主坐标分析和表型特征方面对构建的核心种质进行确认,发现核心种质均匀分布在整个主坐标图,且表型特征符合胡晋等提出的核心种质均值同原种质群体存在显著差异的性状小于等于20%、核心种质与原种质的极差符合率不低于80%作为燕山板栗核心种质代表原群体遗传变异的最低标准[21],所以通过位点优先取样策略结合SM遗传系数进行多次聚类确定的28.57%的取样比例是合理有效的。
建议燕山地区优先对这部分核心种质进行繁殖、交换、评价和利用,以促进各主产区板栗基因交流,提高资源利用率和育种效率。而对于资源保存单位来说,这部分核心种质的确定可以提高整个燕山板栗资源库的管理、保存和利用水平。尽管理论上本研究构建的核心种质在遗传多样性方面能最大程度地代表整个燕山板栗资源,但其他种质仍具有重要的保存意义。因为本研究仅是借助21对SSR引物对其遗传多样性进行了初级挖掘,今后随着资源评价内容的拓展、评价水平的提高,资源遗传多样性将得到进一步挖掘,从中仍可能筛选出极具利用潜力的基因资源,其他植物对非核心种质的留存亦可参考如此。
与前人研究相比[5,12],本研究选用的161份燕山板栗资源取材于燕山主产区10个市(县),是迄今为止进行燕山板栗核心种质群构建选用材料(样本)数量最多、覆盖面最广的研究案例。尽管如此,本研究也存在原种质取样不均的问题,其中抚宁、平泉、迁安地区的资源数量不足10份,可能遗漏部分具有潜在利用价值的种质资源,因此,本研究选用样本在代表燕山板栗资源的全部遗传信息方面仍有欠缺,在今后的工作中需不断补充新搜集的优良品种进入原种质,完善、丰富能够代表原始群体遗传多样性的样本数据。
[1] 张宇和,柳鎏,梁维坚. 中国果树志-板栗 榛子卷[M].北京:中国林业出版社,2005.
Zhang Y H,Liu L,Liang W J. Fruit trees of China·Chestnut[M].Beijing:China Forestry Press,2005.
[2] 杨振立,王广鹏,张树航,郭燕,张馨方,李颖. 燕山板栗15个花序相关表型性状遗传多样性研究[J].华北农学报,2020,35(S1):93-101. doi:10.7668/hbnxb.20191320.
Yang Z L,Wang G P,Zhang S H,Guo Y,Zhang X F,Li Y. Genetic diversity analysis of 15 phenotypic traits related to catkin in Yanshan chestnut[J].Acta Agriculturae Boreali-Sinica,2020,35(S1):93-101.
[3] 王建成,胡晋,黄歆贤,徐盛春. 植物核心种质构建数据和代表性评价参数的研究进展[J].种子,2008,27(8):52-55. doi:10.3969/j.issn.1001-4705.2008.08.014.
Wang J C,Hu J,Huang X X,Xu S C. Progress in constructing data and evaluating parameters of representativeness for plant core collection[J].Seed,2008,27(8):52-55.
[4] 刘艳阳,梅鸿献,杜振伟,武轲,郑永战,崔向华,郑磊. 基于表型和SSR分子标记构建芝麻核心种质[J].中国农业科学,2017,50(13):2433-2441. doi:10.3864/j.issn.0578-1752.2017.13.003.
Liu Y Y,Mei H X,Du Z W,Wu K,Zheng Y Z,Cui X H,Zheng L. Construction of core collection of sesame based on phenotype and molecular markers[J].Scientia Agricultura Sinica,2017,50(13):2433-2441.
[5] 王同坤,董超华,齐永顺,张京政,柏素花,宋文静,张开慧. 燕山板栗种质资源遗传多样性的RAPD分析[J].果树学报,2006,23(4):547-552. doi:10.3969/j.issn.1009-9980.2006.04.014.
Wang T K,Dong C H,Qi Y S,Zhang J Z,Bai S H,Song W J,Zhang K H. Analysis of genetic diversity of Yanshan chestnut by RAPD[J].Journal of Fruit Science,2006,23(4):547-552.
[6] 高捍东,黄宝龙. 板栗主要栽培品种的分子鉴别[J].林业科学,2001,37(1):64-71. doi:10.3321/j.issn:1001-7488.2001.01.010.
Gao H D,Huang B L. Genetic analysis and identification of main cultivated varities of chestnut(Castanea mollissima)by rapd molecular markers[J].Scientia Silvae Sinicae,2001,37(1):64-71.
[7] Abdelhamid S. Genetic variation and characterization of Swiss chestnut cultivars(Castanea sativa Mill.)using RAPD and AFLP markers[J].The Africa Journal of Plant Science and Biotechnology,2012,6(1):21-25.
[8] 周连第,兰彦平,韩振海. 板栗品种资源分子水平遗传多样性研究[J].华北农学报,2006,21(3):81-85. doi:10.3321/j.issn:1000-7091.2006.03.019.
Zhou L D,Lan Y P,Han Z H. Study on heritance diversity of Chinese chestnut(Castanea mollissima)variety resources at molecular level[J].Acta Agriculturae Boreali-Sinica,2006,21(3):81-85.
[9] Jiang X B,Tang D,Gong B C. Genetic diversity and association analysis of Chinese chestnut(Castanea mollissima Blume)cultivars based on SSR markers[J].Brazilian Journal of Botany,2017,40(1):235-246.doi:10.1007/s40415-016-0321-8.
[10] Gailing O,Nelson C D. Genetic variation patterns of American chestnut populations at EST-SSRs[J].Botany,2017,95(8):799-807.doi:10.1139/cjb-2016-0323.
[11] 韩继成,刘庆香,王广鹏,孔德军,张新忠.栗属植物遗传多样性研究进展[J].华北农学报,2006,21(S2):129-132.doi:10.3321/j.issn:1000-7091.2006.Z2.032.
Han J C,Liu Q X,Wang G P,Kong D J,Zhang X Z. The progress of the genetic diversity studies on Castanea (Tourn.)L.[J].Acta Agriculturae Boreali-Sinica,2006,21(S2):129-132.
[12] 程丽莉.燕山板栗实生居群遗传多样性研究与核心种质初选[D].北京:北京林业大学,2005.
Cheng L L. Studies on genetic diversity and core collection selection of Castanea mollissima Bl.populations[D].Beijing:Beijing Forestry University,2005.
[13] 马玉敏.中国野生板栗(Castanea mollissim Blume)群体遗传结构和核心种质构建方法[D].泰安:山东农业大学,2009.
Ma Y M. Population genetic structure and method of constructing core collection for Castanea mollissim Blume[D].Taian:Shandong Agricultural University,2009.
[14] 张馨方,张树航,李颖,郭燕,王广鹏.基于SSR标记的燕山板栗种质资源遗传多样性分析[J].中国农业大学学报,2020,25(4):61-71.doi:10.11841/j.issn.1007-4333.2020.04.07
Zhang X F,Zhang S H,Li Y,Guo Y,Wang G P. Genetic diversity analysis of chestnut germplasm in Yanshan region based on SSR markers[J].Journal of China Agricultural University,2020,25(4):61-71.
[15] 黄平,崔娇鹏,郑勇奇,张川红.基于月季微卫星标记的7个遗传相似系数比较[J].林业科学,2013,49(1):68-76.doi:10.11707/j.1001-7488.20130111.
Huang P,Cui J P,Zheng Y Q,Zhang C H. Comparison of 7 genetic similarity coefficients based on microsatellite markers in rose variety[J].Scientia Silvae Sinicae,2013,49(1):68-76.
[16] Sneath P H A,Sokal R R. Numerical Taxonomy. The principles and practice of numerical classification[J].Taxon,1963,12(5):190-199.
[17] Dice L R. Measures of the amount of ecologic association between species[J].Ecology,1945,26(3):297-302.doi:10.2307/1932409.
[18] Duarte J M,dos Santos J B,Melo L C. Comparison of similarity coefficients based on RAPD markers in the common bean[J].Genetics and Molecular Biology,1999,22(3):427-432.doi:10.1590/S1415-47571999000300024.
[19] 张春雨,陈学森,张艳敏,苑兆和,刘遵春,王延龄,林群.采用分子标记构建新疆野苹果核心种质的方法[J].中国农业科学,2009,42(2):597-604.doi:10.3864/j.issn.0578-1752.2009.02.026.
Zhang C Y,Chen X S,Zhang Y M,Yuan Z H,Liu Z C,Wang Y L,Lin Q. A method for constructing core collection of Malus sieversii using molecular markers[J].Scientia Agricultura Sinica,2009,42(2):597-604.
[20] Hu J,Zhu J,Xu H M. Methods of constructing core collections by stepwise clustering with three sampling strategies based on the genotypic values of crops[J].Theoretical and Applied Genetics,2000,101(1/2):264-268.doi:10.1007/s001220051478.
[21] 胡晋,徐海明,朱军.基因型值多次聚类法构建作物种质资源核心库[J].生物数学学报,2000,15(1):103-109.doi:10.3969/j.issn.1001-9626.2000.01.016.
Hu J,Xu H M,Zhu J. Constructing core collection of crop germplasm by multiple clusters based on genotypic values[J].Journal of Biomathematics,2000,15(1):103-109.
[22] Huang C Q,Long T,Bai C J,Wang W Q,Tang J,Liu G D. Establishment of a core collection of Cynodon based on morphological data[J].Tropical Grasslands-Forrajes Tropicales,2020,8(3):203-213.doi:10.17138/tgft(8)203-213.
[23] Girma G,Bhattacharjee R,Lopez-Montes A,Gueye B,Ofodile S,Franco J,Abberton M. Re-defining the yam(Dioscorea spp.)core collection using morphological traits[J].Plant Genetic Resources,2018,16(3):193-200.doi:10.1017/s1479262117000144.
[24] Espósito,Andrea M,Gatti,ILeana,Cointry,Enrique. Construction and validation of core collections in Pisum sp.using different methodologies[J].Legume Research,2019,42(6):743-749.doi:10.18805/lr-451.
[25] 张君玉. 利用SSR和SRAP分子标记构建葡萄核心种质[D].洛阳:河南科技大学,2012.doi:10.7666/d.D01125248.
Zhang J Y. Construction of core collections for grape germplasm using SSR and SRAP molecular makers[D].Luoyang:Henan University of Science and Technology,2012.
[26] 刘娟,廖康,赵世荣,曹倩,孙琪,刘欢.利用ISSR分子标记构建新疆野杏核心种质资源[J].中国农业科学,2015,48(10):2017-2028.doi:10.3864/j.issn.0578-1752.2015.10.014.
Liu J,Liao K,Zhao S R,Cao Q,Sun Q,Liu H. The core collection construction of Xinjiang wild apricot based on ISSR molecular markers[J].Scientia Agricultura Sinica,2015,48(10):2017-2028.
[27] 王建成,胡晋,黄歆贤,徐盛春.植物遗传资源核心种质新概念与应用进展[J].种子,2008,27(5):47-50.doi:10.3969/j.issn.1001-4705.2008.05.014.
Wang J C,Hu J,Huang X X,Xu S C. New concept and application on core collection of plant germplasm resources[J].Seed,2008,27(5):47-50.
[28] Balakrishnan R,Nair N V,Sreenivasan T V. A method for establishing a core collection of Saccharum officinarum L. germplasm based on quantitative-morphological data[J].Genetic Resources and Crop Evolution,2000,47(1):1-9. doi:10.1023/A:1008780526154.
[29] 李自超,张洪亮,曾亚文,杨忠义,申时全,孙传清,王象坤.云南地方稻种资源核心种质取样方案研究[J].中国农业科学,2000,33(5):1-7.
Li Z C,Zhang H L,Zeng Y W,Yang Z Y,Shen S Q,Sun C Q,Wang X K. Study on sampling schemes of core collection of local varieties of rice in Yunnan,China[J].Scientia Agricultura Sinica,2000,33(5):1-7.
[30] Quero-Garcia J,Noyer J L,Perrier X,Marchand J L,Lebot V. A germplasm stratification of taro(Colocasia esculenta)based on agro-morphological descriptors,validation by AFLP markers[J].Euphytica,2004,137(3):387-395. doi:10.1023/B:EUPH.0000040521.00303.ac.
[31] 刘娟,廖康,曼苏尔·那斯尔,曹倩,江振斌,贾杨.利用ISSR分子标记构建南疆杏种质资源核心种质[J].果树学报,2015,32(3):374-384.doi:10.13925/j.cnki.gsxb.20140447.
Liu J,Liao K,Mansur N,Cao Q,Jiang Z B,Jia Y. Core-germplasm construction of apricot collections in South of Xinjiang by ISSR molecular markers[J].Journal of Fruit Science,2015,32(3):374-384.
[32] Richter T S,Soltis P S,Soltis D E. Genetic variation within and among populations of the narrow endemic,Delphinium viridescens(Ranunculaceae)[J].American Journal of Botany,1994,81(8):1070-1076.doi:10.1002/j.1537-2197.1994.tb15596.x.
[33] Bengtsson B O,Weibull P,Ghatnekar L. The loss of alleles by sampling:A study of the common outbreeding grass Festuca ovina over three geographic scales[J].Hereditas,1995,122(3):221-238.doi:10.1111/j.1601-5223.1995.00221.x.
[34] Caujap-Castells J,Pedrola-Monfort J. Designing ex-situ conservation strategies through the assessment of neutral genetic markers:Application to the endangered Androcymbium gramineum[J].Conservation Genetics,2004,5(2):131-144. doi:10.1023/B:COGE.0000029997.59502.88.
[35] 李自超,张洪亮,曹永生,裘宗恩,魏兴华,汤圣祥,余萍,王象坤.中国地方稻种资源初级核心种质取样策略研究[J].作物学报,2003,29(1):20-24.doi:10.3321/j.issn:0496-3490.2003.01.004.
Li Z C,Zhang H L,Cao Y S,Qiu Z E,Wei X H,Tang S X,Yu P,Wang X K. Studies on the sampling strategy for primary core collection of Chinese ingenious rice[J].Acta Agronomica Sinica,2003,29(1):20-24.
[36] 黎毛毛,黄永兰,余丽琴,王记林,芦明,熊玉珍,束爱萍,范志洁,万建林.利用SSR标记构建江西稻种资源核心种质库的研究[J].植物遗传资源学报,2012,13(6):952-957.doi:10.3969/j.issn.1672-1810.2012.06.005.
Li M M,Huang Y L,Yu L Q,Wang J L,Lu M,Xiong Y Z,Shu A P,Fan Z J,Wan J L. Development of a core collection for Jiangxi traditional rice germplasm by SSR markers[J].Journal of Plant Genetic Resources,2012,13(6):952-957.
[37] 吴涛,陈少瑜,肖良俊,宁德鲁,潘莉,贺娜,朱云凤.基于SSR标记的云南省核桃种质资源遗传多样性及核心种质构建[J].植物遗传资源学报,2020,21(3):767-774.doi:10.13430/j.cnki.jpgr.20190813001.
Wu T,Chen S Y,Xiao L J,Ning D L,Pan L,He N,Zhu Y F. Genetic diversity analysis and core collection construction of walnut germplasm in Yunnan Province[J].Journal of Plant Genetic Resources,2020,21(3):767-774.
[38] 毛秀红,朱士利,李善文,华辉,田书勇,仲伟国,董玉峰,安新民.基于荧光SSR标记的毛白杨核心种质构建[J].北京林业大学学报,2020,42(7):40-47.doi:10.12171/j.1000-1522.20190413.
Mao X H,Zhu S L,Li S W,Hua H,Tian S Y,Zhong W G,Dong Y F,An X M. Core germplasm construction of Populus tomentosa based on the fluorescent SSR markers[J].Journal of Beijing Forestry University,2020,42(7):40-47.
[39] 杨汉波,张蕊,王帮顺,徐肇友,周志春.基于SSR标记的木荷核心种质构建[J].林业科学,2017,53(6):37-46.doi:10.11707/j.1001-7488.20170605.
Yang H B,Zhang R,Wang B S,Xu Z Y,Zhou Z C. Construction of core collection of Schima superba based on SSR molecular markers[J].Scientia Silvae Sinicae,2017,53(6):37-46.
