解淀粉芽孢杆菌属于革兰氏阳性杆菌的一种,具有好氧、产芽孢、嗜温的特点[1]。此细菌分布广泛,代谢产物丰富,如大环内酯类、抗菌蛋白、脂肽类等物质,应用前景十分广阔[2-3]。由于解淀粉芽孢杆菌的巨大工业价值,对于菌株基因操作的各种方法要求,研究人员也提出了更高的要求。在基因敲除过程中,由于抗生素抗性标记的存在,可能会存在一些负面影响。相关研究表明,反向筛选标记技术能有效降低重组菌株对抗性筛选标记的携带率,是目前研究特异基因型生物的有效手段。该技术利用自杀基因的特性,通过2次同源重组,大幅度提高重组菌株的筛选率。目前,在枯草芽孢杆菌中已报道的反向筛选标记有upp、mazF、galK基因。由于mazF基因编码毒性蛋白,对细胞有损伤;galK基因与半乳糖类似物不能共存,否则会杀死细胞等因素,大多选用upp基因作为反向筛选标记,利用upp基因编码尿嘧啶磷酸转移酶(UPRTase),它能将5-氟尿嘧啶(5-FU)通过生物代谢作用转化为5-F-dUMP。5-F-dUMP能起到抑制细胞生长的作用。2002年,Fabret等[4]用upp作为反向筛选标记,转化后使upp表达盒和多余的2个正向重复从染色体上脱落。这也是第一个报道upp在芽孢杆菌中的使用的研究。吴果果等[5]利用upp反向筛选标记在链霉菌中构建了完整的遗传操作系统。
γ-聚谷氨酸(Poly-γ-glutamic acid,γ-PGA)是一种由重复谷氨酸单体构成的聚氨基酸。由于具有极佳的水溶性、生物亲和性、吸附性等广泛应用于医药行业、化妆品行业、污水吸附处理等诸多方面[6-10]。γ-PGA大多数由芽孢杆菌产生。目前,在研究和生产中使用的芽孢杆菌大多都是谷氨酸依赖型菌株,即需要在培养基中添加高浓度谷氨酸作为合成γ-PGA的前体,这是推高γ-PGA生产成本的重要因素之一。而另外一类菌株即谷氨酸非依赖性菌株,例如枯草芽孢杆菌TAM-4、C1等[11-12],利用这类菌株发酵合成γ-PGA不需要在培养基中添加谷氨酸,菌体自身以糖类为原料以从头合成途径(de novo pathway)产生内源谷氨酸前体,可有效降低生产成本。目前提高γ-PGA产量主要有3个思路:筛选新的γ-PGA合成菌株、优化发酵工艺和遗传改造已有的γ-PGA合成菌。但筛选新的γ-PGA合成菌株、优化发酵工艺存在周期较长,稳定性方面的问题,因此,通过代谢工程手段提高谷氨酸非依赖性菌株的发酵产率,将会最大程度挖掘出此类芽孢杆菌生物合成系统的潜力,开辟一条将生物质原料(糖类)转化为生物聚合物(γ-PGA)的最有效途径。Wang等[13]利用基因组重组技术由枯草芽孢杆菌GXA-28出发获得了突变株F3-178,其突变菌株中γ-PGA相关合成基因的过度表达使得产量提高了1.9倍((34.3±1.2)g/L)。张伟[14]构建了解淀粉芽孢杆菌LL3ΔUR(ΔrocR)和解淀粉芽孢杆菌LL3 ΔUBG(ΔrocG ΔgudB)2株γ-PGA合成突变菌株,与野生菌株的4.08 g/L相比,γ-PGA产量分别提高至5.68,5.83 g/L。冯俊[15]在解淀粉芽孢杆菌LL3基础上双敲除itu和srf基因簇的菌株,γ-PGA产量从3.3 g/L提高到4.5 g/L。
前期研究发现在培养基中添加少量草酸能够触发解淀粉芽孢杆菌合成高浓度γ-PGA[16]。通过查询KEGG代谢途径数据库发现,在枯草芽孢杆菌中,草酸的代谢是由草酸脱羧酶(Oxalate decarboxylase,OXDC)转化为甲酸,甲酸被甲酸脱氢酶进一步氧化为二氧化碳。解淀粉芽孢杆菌C10是新疆石河子大学生命科学学院发酵实验室筛选出来的谷氨酸非依赖性菌株,目前,对于oxdC基因在γ-PGA合成中的作用鲜有报道。本研究拟将pKSV7即温度敏感型质粒与upp反向筛选标记进行结合,在谷氨酸非依赖性菌株解淀粉芽孢杆菌C10中构建一个基因无痕替换方法,为解淀粉芽孢杆菌C10的代谢工程改造提供工具。在此基础上敲除oxdC基因,验证草酸在不能被分解的情况下,对γ-聚谷氨酸合成的影响。
1 材料和方法
1.1 试验材料
1.1.1 主要试剂 Taq DNA聚合酶、细菌基因组提取试剂盒、质粒提取试剂盒、DNA 片段回收试剂盒均购于北京天根生化科技有限公司,ClonExpressⅡOne Step Cioning Kit试剂盒购于南京诺唯赞生物科技有限公司,T载体及限制性核酸内切酶均购于宝日医生物技术(北京)有限公司,Bradford蛋白浓度测定试剂盒购于北京索莱宝科技有限公司。
1.1.2 菌株及质粒 解淀粉芽孢杆菌C10、枯草芽孢杆菌168、温度敏感型质粒pKSV7。
1.1.3 引物 本研究引物合成于上海生工生物工程,具体引物见表1。
表1 本研究所用的引物
Tab.1 Primers used in this study

1.1.4 培养基 生长培养基/发酵培养基:A组分为葡萄糖80 g/L;FeCl3·6H2O 0.04 g/L;MnSO4·H2O 0.104 g/L;CaCl2 0.11 g/L。B组分为蛋白胨10 g/L;NH4Cl 18 g/L;K2HPO4 0.5 g/L;MgSO4·7H2O 0.5 g/L;草酸钠14.98 g/L。A组分和B组分单独灭菌后在超净工作台内混合。
复苏培养基:酵母提取物5 g/L,胰蛋白胨10 g/L,NaCl 10 g/L,山梨醇0.5 mol/L,甘露醇0.38 mol/L,定容至1 L,1×105 Pa灭菌30 min。
1.2 试验方法
1.2.1 解淀粉芽孢杆菌对5-氟尿嘧啶(5-FU)耐受浓度的确定 首先制备添加5-FU的LB琼脂平板,终浓度分别为0.10,0.15,0.20,0.25,0.30,0.35,0.40 mmol/L。为了排除DMSO对细菌生长的影响,同时配制对应浓度的DMSO平板。活化解淀粉芽孢杆菌C10菌株,挑单菌落于LB液体培养基中,180 r/min,32 ℃过夜培养,将其稀释10-7~10-9倍,吸取100 μL的稀释物涂布于5-FU及DMSO平板上。在温度为32 ℃的条件下,过夜培养,对每个平板上的菌落数进行观察。计算存活率:5-FU平板上的菌落数与DMSO平板上的菌落数之比。试验结果取3个生物学重复的平均值。
1.2.2 解淀粉芽孢杆菌C10upp基因敲除菌的构建
以解淀粉芽孢杆菌C10基因组DNA为模板,利用引物对UppUP-F、UppUP-R及UppDN-F、UppDN-R扩增upp基因上、下游同源臂片段以overlapping PCR得到融合片段UP-DN。与pKSV7的KpnⅠ和EcoRⅠ的酶切位点连接,转化至E. coli JM110,质粒提取送测序验证,用BamH Ⅰ甲基转移酶进行甲基化处理,电转化至解淀粉芽孢杆菌C10感受态。电转条件为将OD600为4.2时制备的解淀粉芽孢杆菌C10感受态细胞在电压2 200 V的条件下电击时间5 ms,涂布氯霉素平板,48 h后长出转化子进行筛选,筛选过程见参考文献[14]。筛选出的菌落用引物UppOUT-F/UppOUT-R筛选出突变菌株即C10Δupp(图1)。
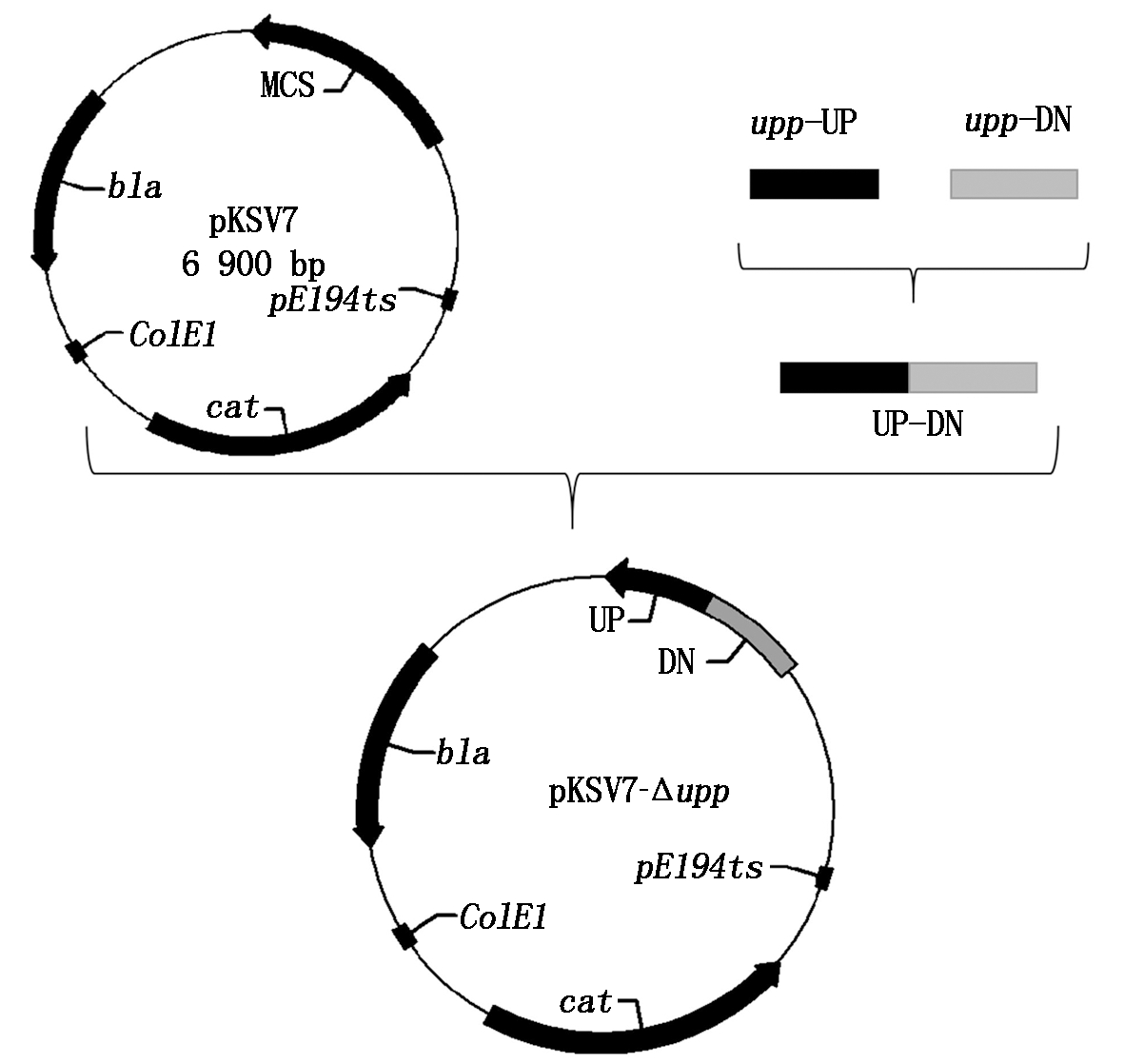
图1 载体pKSV7-Δupp的构建流程
Fig.1 Construction flow chart of vector pKSV7-Δupp
1.2.3 敲除菌株C10ΔuppΔoxdC的构建 利用无缝克隆的方法以解淀粉芽孢杆菌C10基因组DNA为模板,以oxdC UP-F/oxdC UP-R和oxdC DN-F/oxdC DN-R为引物对草酸脱羧酶oxdC基因上、下游同源臂扩增,与Sal Ⅰ酶切线性化的pKSU(pKSV7-Δupp)质粒连接转化至E.coli JM110。对长出的单菌落进行PCR及酶切验证,经测序后得到敲除载体pKSU-ΔoxdC。将敲除载体电转化转入解淀粉芽孢杆菌C10Δupp中,筛选得到敲除菌株C10ΔuppΔoxdC(图2)。
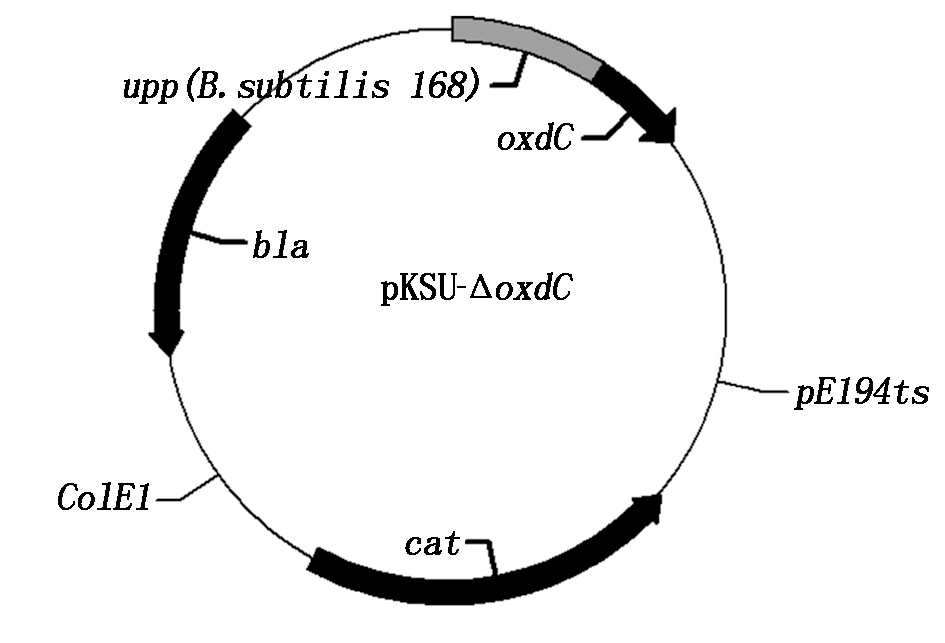
图2 敲除载体pKSU-ΔoxdC的构建
Fig.2 Knock out carrier pKSU-ΔoxdC build
1.2.4 oxdC基因敲除菌株C10ΔuppΔoxdC的发酵培养 挑取LB固体培养基平板上的C10ΔuppΔoxdC单菌落接种于30 mL LB培养基中,32 ℃,200 r/min过夜培养至OD600至4.5。按照10%的接种量将菌液接种于装有30 mL发酵培养基的250 mL锥形瓶中,32 ℃,200 r/min培养36 h。
1.2.5 敲除菌株C10ΔuppΔoxdC草酸脱羧酶活力测定
1.2.5.1 粗酶液的提取 发酵液6 700 r/min离心15 min收集菌体,除上清,将沉淀悬浮于含有0.5 mol/L NaCl的50 mmol/L磷酸钠缓冲液(pH值7.6)中,缓冲液与培养液体积的比为1∶10,于冰浴条件下使用超声波破碎仪进行细胞破碎,破碎条件:破碎1 s,暂停2 s,破碎总时间为8 min,功率为50%。随后将细胞破碎液于4 ℃、8 200 r/min离心30 min,上清液即为粗酶液。对照为C10Δupp菌株。
1.2.5.2 蛋白浓度测定 采用考马斯亮蓝法蛋白浓度测定试剂盒测定。草酸脱羧酶活力检测参考白雪等[17]的方法。反应液用0.22 μm微孔过滤膜过滤酶蛋白后,采用液相色谱法(HPLC)检测反应生成的甲酸。HPLC条件:流动相为0.005 mol/L H2SO4,流速为0.5 mL/min,进样量20 μL,色谱柱为BENSON 8100-0 BP-800Ca柱,柱温60 ℃,紫外检测波长210 nm。甲酸标准品的浓度梯度为0,0.004,0.006,0.008,0.010,0.012,0.014,0.016,0.018,0.020,0.022 mmol/L,根据甲酸浓度与HPLC 210 nm处吸收峰面积的线性关系绘制甲酸标准曲线。酶活单位定义为:1 min催化转化草酸产生1 μmo1甲酸的酶量。酶的比活力即为单位质量(mg)酶所具有的酶活力单位数,草酸脱羧酶比活力即为酶活力与蛋白浓度之比。
1.2.6 γ-PGA的测定 发酵结束将菌液在12 000 r/min的条件下离心20 min,去除菌体沉淀,在上清中加入上清体积2倍的无水乙醇,4 ℃冰箱过夜保存,次日于3 500 r/min离心10 min,去上清,将沉淀物于60 ℃烘干至恒质量后称质量。试验结果取3个生物学重复的平均值。
2 结果与分析
2.1 解淀粉芽孢杆菌 C10对5-氟尿嘧啶(5-FU)耐受浓度的确定
如图3所示,解淀粉芽孢杆菌C10对5-FU具有敏感性。C10细胞的存活率,随着5-FU浓度的增加,逐步降低,直至存活率为0。这时0.25 mmol/L的5-FU就可作为后续筛选时的工作浓度。
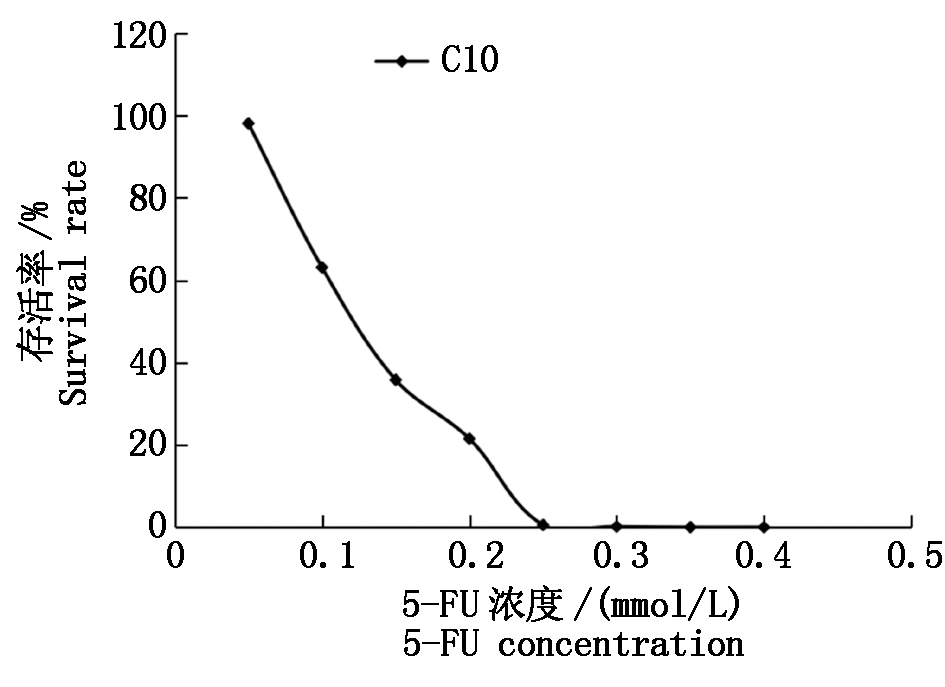
试验结果是3个生物学重复的平均值,标准偏差小于10%。
The experimental result is the average of three
biological replicates with a standard deviation of less than 10%.
图3 解淀粉芽孢杆菌C10耐受5-FU试验
Fig.3 5-FU tolerance test of Bacillus amyloliquefaciens C10
2.2 upp基因的敲除
敲除载体pKSV7-Δupp经酶切得到约7 000,1 100 bp的条带,经过测序确认载体构建成功(图4)。解淀粉芽孢杆菌C10中的转化子通过2次同源重组后筛选得到解淀粉芽孢杆菌C10Δupp菌株,具体原理如图5。分析图6可知,野生型的C10可以获得片段约1 800 bp,因upp阅读框的缺少,突变株只获得约1 200 bp的片段。对该片段的进一步DNA测序证明,成功敲除了upp基因。命名为解淀粉芽孢杆菌C10Δupp,作为随后敲除的初始菌株。
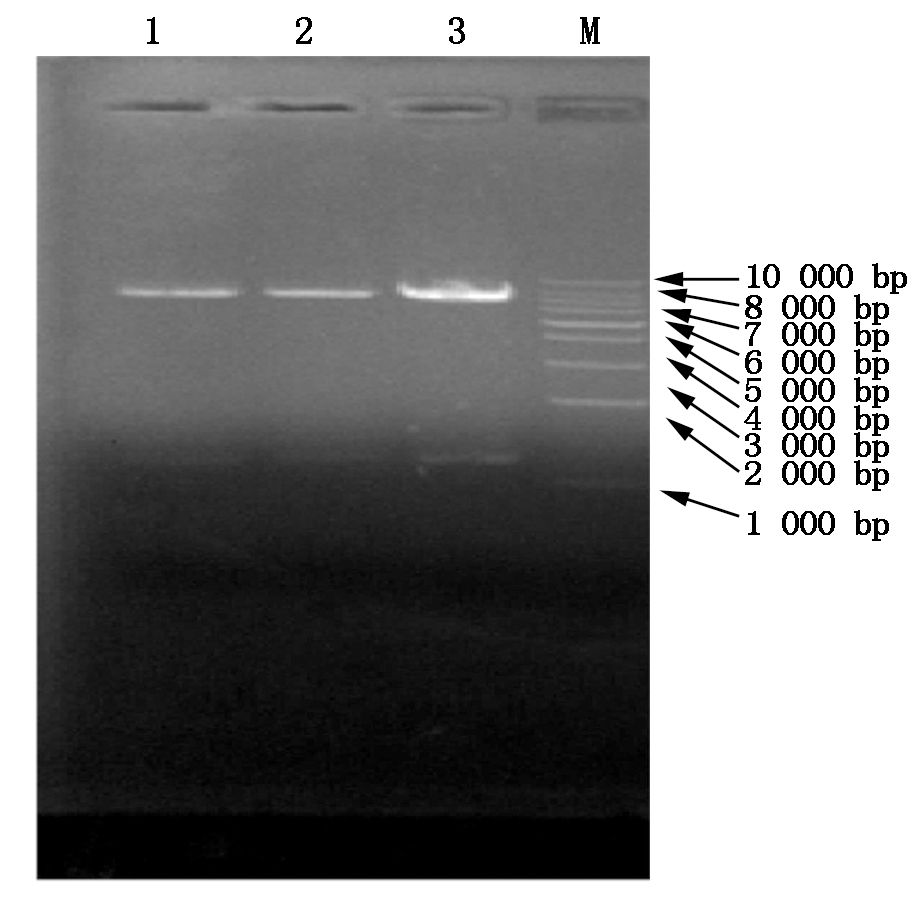
1-3.pKSV7-Δupp质粒。
1-3.pKSV7-Δupp plasmid.
图4 pKSV7-Δupp载体酶切
Fig.4 pKSV7-Δupp vector digestion
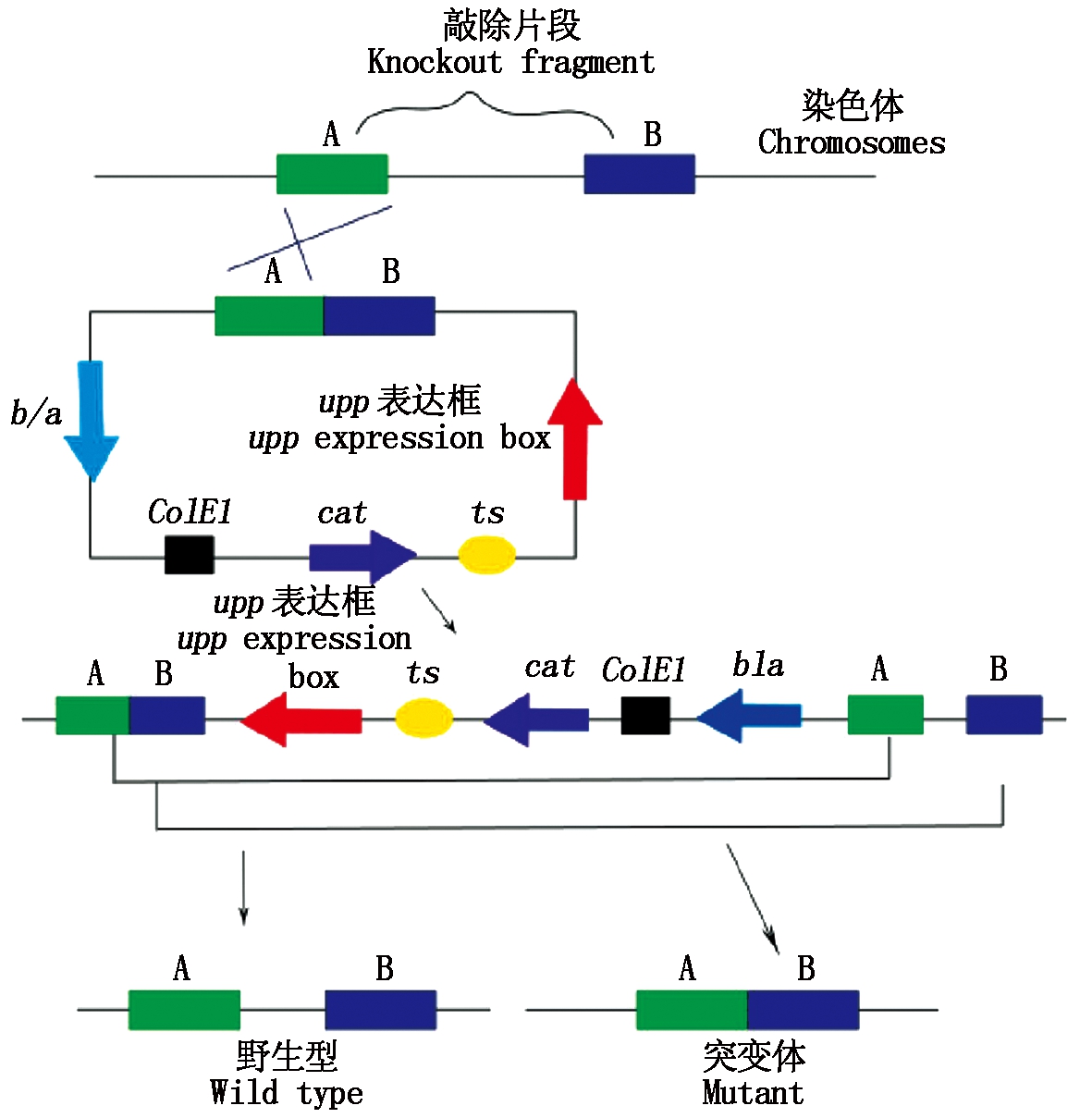
图5 细菌中基于upp基因的无痕基因敲除方法原理图
Fig.5 Schematic diagram of the method of non-trace gene
knockout based on the upp gene in bacteria
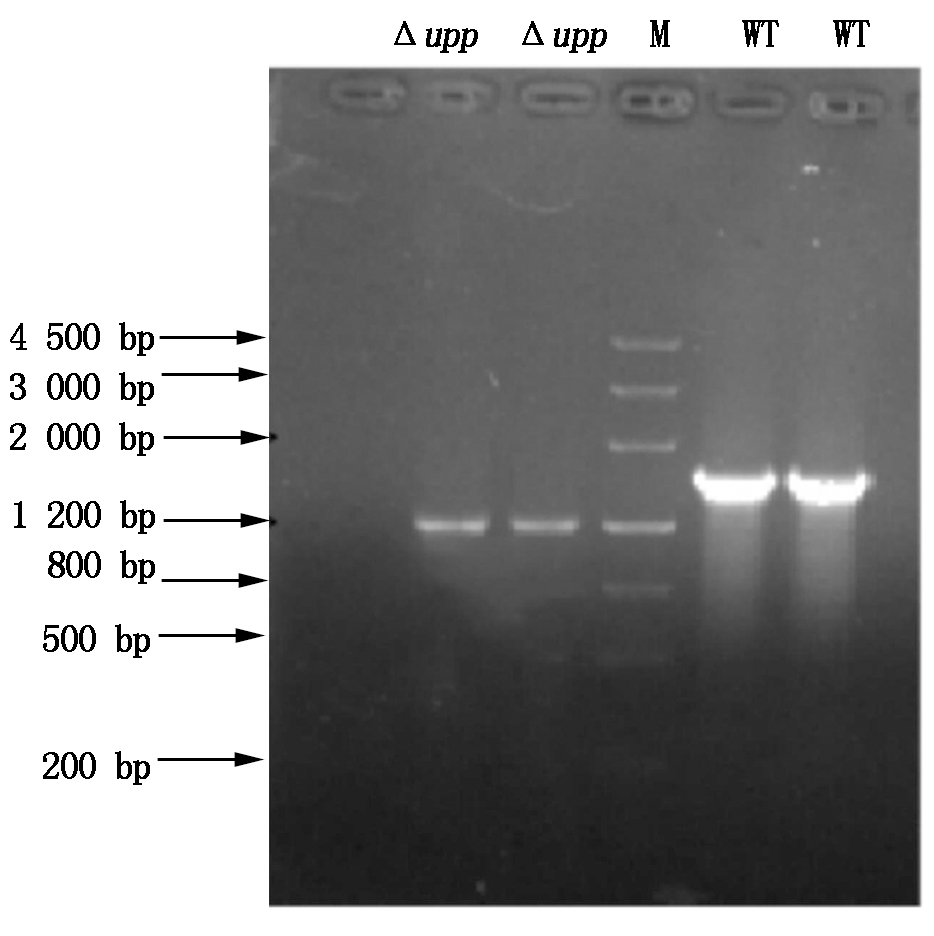
WT.模板为野生型染色体DNA;Δupp.模板为突变株染色体DNA。
WT uses wild-type chromosomal DNA as a template;
Δupp uses mutant chromosomal DNA as a template.
图6 upp基因敲除PCR验证的电泳图
Fig.6 upp gene knockout PCR verification electrophoresis
2.3 oxdC基因的敲除
使用敲除菌株C10ΔuppΔoxdC的染色体DNA作为模板,通过PCR扩增可获得约1 100 bp的片段,而对照组C10Δupp可获得约2 300 bp的片段,如图7所示。由此,可以得出,C10Δupp基因组中的oxdC基因已经成功敲除,得到敲除菌株C10ΔuppΔoxdC。
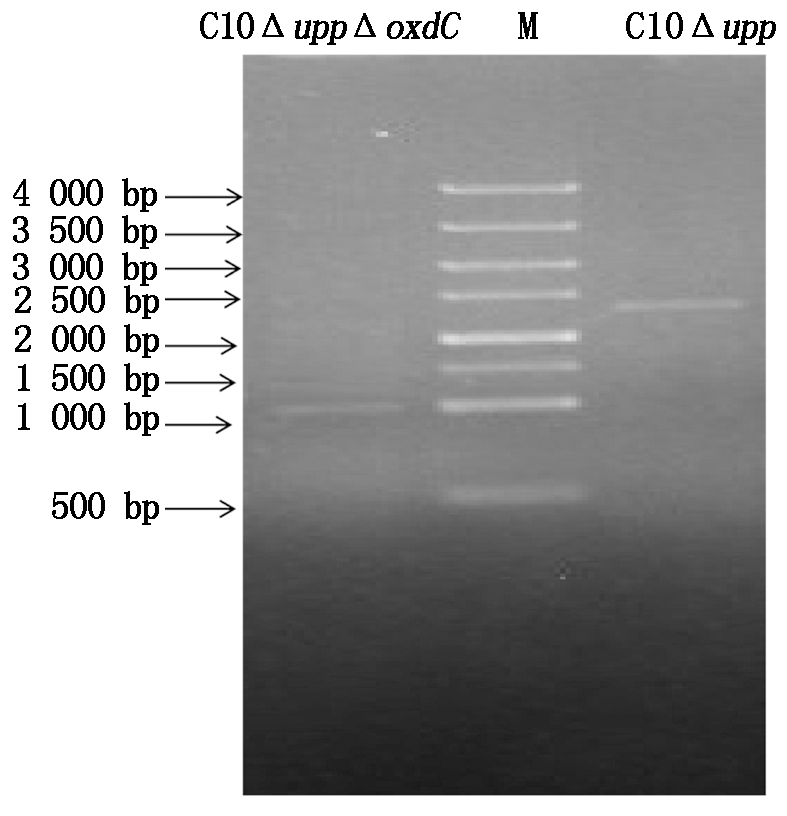
M.DNA Marker Ⅲ。
图7 oxdC基因敲除菌株PCR检测电泳
Fig.7 PCR electrophoresis of oxdC due
to the knockout strain
2.4 突变菌株C10ΔuppΔoxdC的发酵验证
根据蛋白标准品BSA的不同浓度梯度与595 nm处吸光度的关系,绘制标准曲线,回归方程为y=1 885x+0.011 4,R2=0.996 7。敲除菌株C10ΔuppΔoxdC及对照菌株发酵后破碎细胞提取的蛋白质含量分别为0.003 45,2.293 4 mg/mL。根据甲酸浓度与HPLC 210nm处吸收峰面积的线性关系绘制的标准曲线,回归方程为y=3 046.1x+4.127 9,R2=0.990 4。HPLC对草酸脱羧酶催化反应液中甲酸的检测结果见图8,通过HPLC可以有效检测产物甲酸(16.57 min)。根据回归方程求出对照C10Δupp的草酸脱羧酶活力为1.378 U,比活力为0.600 8 U/mg,而C10ΔuppΔoxdC的草酸脱羧酶无酶活力,可以证实oxdC基因已经成功敲除。
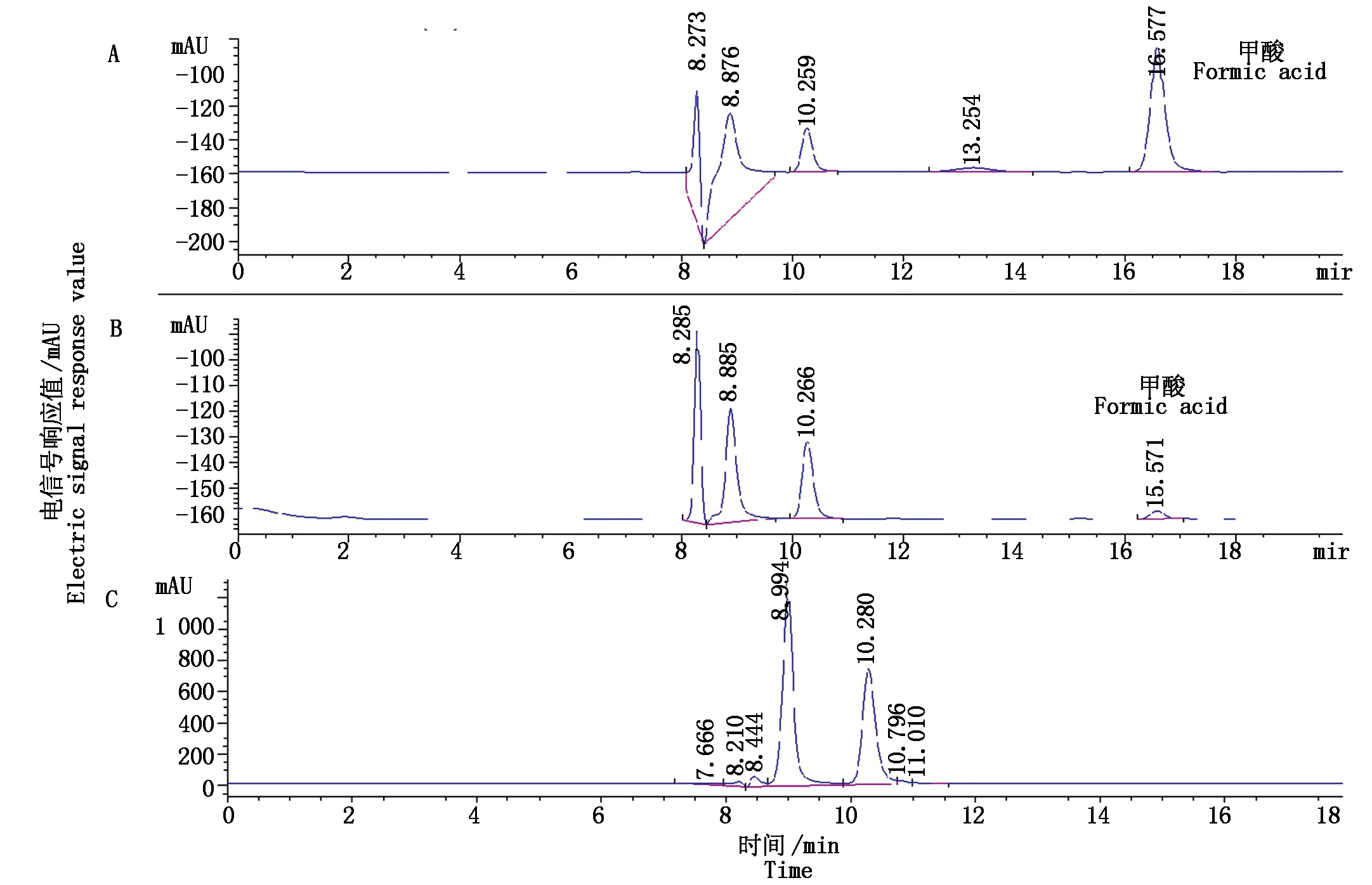
A.甲酸标准品;B. C10Δupp样品;C. C10ΔuppΔoxdC,未检测到甲酸。
A. Formic acid standard;B. C10Δupp sample;C. C10ΔuppΔoxdC, no formic acid was detected.
图8 HPLC对甲酸的检测
Fig.8 HPLC detection of formic acid
C10ΔuppΔoxdC和C10Δupp在发酵36 h后OD600值分别为13.96,15.56,C10Δupp较C10ΔuppΔoxdC有轻微的优势,说明oxdC基因的缺失对菌株生长有一定影响,结果如图9所示。发酵试验结果(图10)显示,初始菌株C10Δupp的γ-PGA产量为22.22 g/L,突变菌株的γ-PGA产量为12.33 g/L,产量与初始菌株相比有所下降,推测草酸的积累会对菌体生长造成负担。
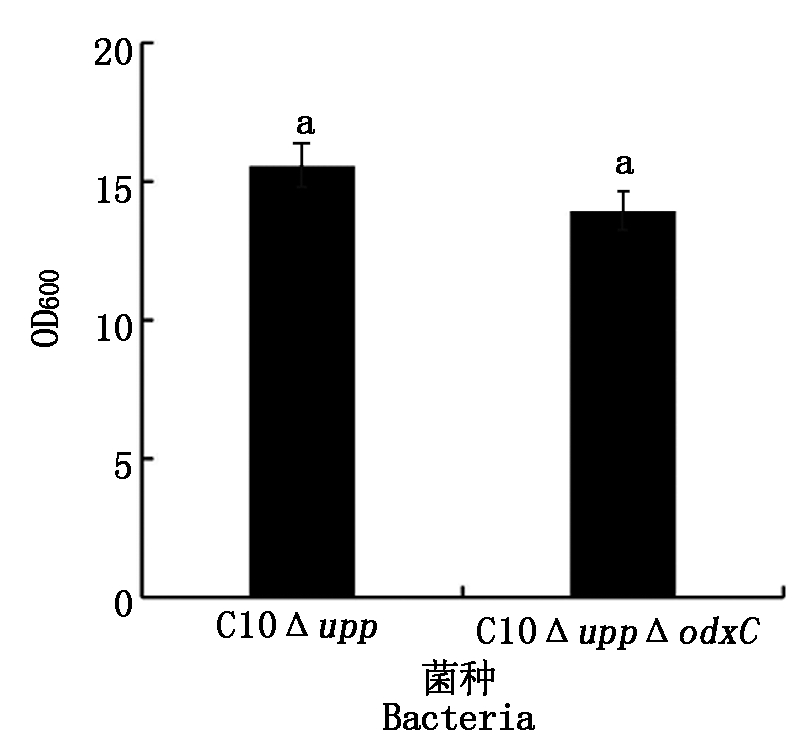
不同小写字母表示差异达到显著水平(P<0.05)。图10同。
Different lowercase letters indicate the difference was
significant(P<0.05).The same as Fig.10.
图9 C10Δupp和C10ΔuppΔoxdC的OD600比较
Fig.9 An OD600 comparison of
C10Δupp and C10ΔuppΔoxdC
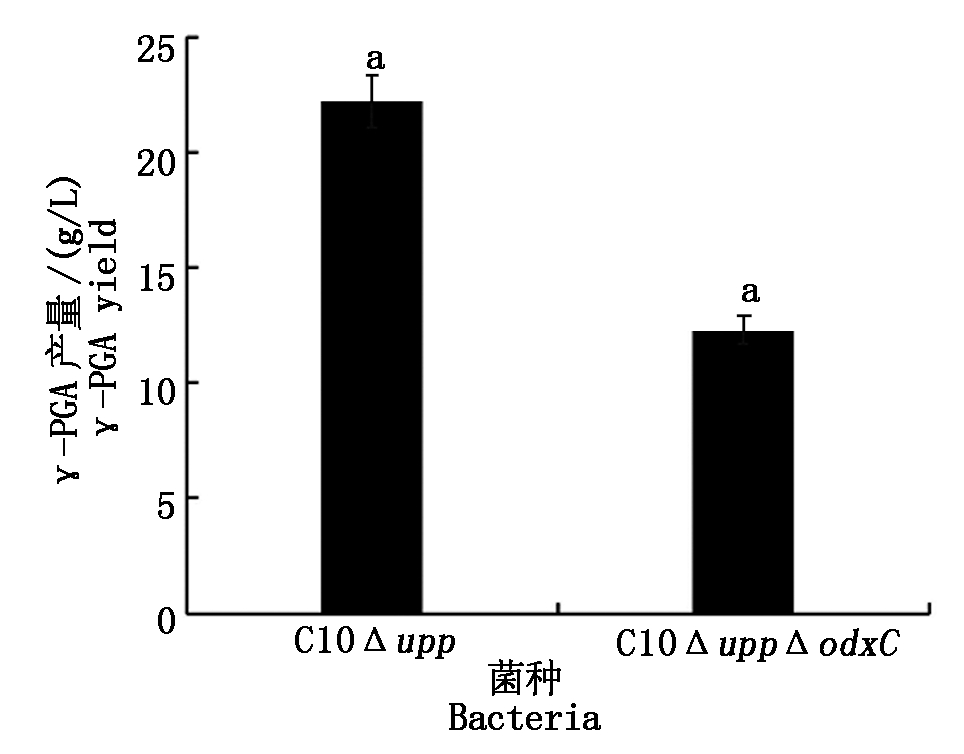
图10 C10Δupp和C10ΔuppΔoxdC的γ-PGA产量对比
Fig.10 Panel a is a comparison of γ-PGA production of
C10Δupp and C10ΔuppΔoxdC
3 讨论
芽孢杆菌的染色体无痕修饰改造的技术方法在目前看来已经属于一种成熟的技术,在菌种的合成生物学和遗传改造方面也取得非常优秀的成绩。Wang等[18]在假单胞菌Pf-5菌株中构建了upp基因缺失菌株,在此基础上成功敲除了17 kb的片段,upp作为反向筛选标记已被广泛使用[19]。
解淀粉芽孢杆菌易于分离和培养,具有丰富的代谢产物,非常适合生物的大规模生产和应用。γ-PGA因具备优异特性广泛应用于医药、食品、化妆品等行业[20-22]。近年来利用基因工程技术改造γ-PGA合成菌来提高γ-PGA产量的方法已非常成熟,并取得了不错的成就。Yeh等[23]在枯草芽孢杆菌DB430菌株中的pgsBCA基因簇前插入表达序列-SECS,突变菌株γ-PGA产量为28 g/L。Mitsui等[24]将cwlO基因敲除后,与对照相比,菌株的γ-PGA产量提高了2倍。Liu等[25]在解淀粉芽孢杆菌C06菌株中构建了epsA基因缺失菌株,γ-PGA产量从3.2 g/L提高到6.8 g/L。但是关于oxdC基因对于γ-PGA合成的影响还鲜有报道。
本研究利用upp反向筛选标记,首先构建了upp基因缺失菌株。温敏型质粒pKSV7用于获得单交换菌株,upp反向筛选标记用于筛选双交换菌株,在获得突变菌株C10Δupp的基础上,对oxdC基因进行敲除,抑制草酸转化为甲酸和二氧化碳,然而,结果显示,草酸的积累反而不利于菌体生长和聚谷氨酸的合成。推测可能的原因是草酸在分解过程中产生还原力(NADH),抑制了菌体的TCA循环,未能促进碳流进入谷氨酸合成途径。然而,具体分子机理尚不清楚,后续工作可继续探究。该方法为其他解淀粉芽孢杆菌菌株的代谢工程改造提供了技术方法的借鉴,因此值得进一步推广应用。
[1] 严婉荣,赵廷昌,肖彤斌,肖敏,赵志祥,陈绵才. 生防细菌在植物病害防治中的应用[J].基因组学与应用生物学,2013,32(4):533-539. doi:10.3969/gab.032.000533.
Yan W R,Zhao T C,Xiao T B,Xiao M,Zhao Z X,Chen M C. Applications of Biocontrol bacteria in plant disease control[J].Genomics and Applied Biology,2013,32(4):533-539.
[2] 杨敏馨. 解淀粉芽孢杆菌在动物生产中的应用研究进展[J].饲料广角,2018(6):49-51.
Yang M X. Research progress of application of Bacilus amyloliquefaciens in animal production[J].Feed China,2018(6):49-51.
[3] 杜丽华,张维瑞,袁王俊. 解淀粉芽孢杆菌次生代谢产物的研究进展[J].科技资讯,2018,16(6):128,130. doi:10.16661/j.cnki.1672-3791.2018.06.128.
Du L H,Zhang W R,Yuan W J. Research progress on secondary metabolites of Bacillus amyloliquefaciens[J].Science & Technology Information,2018,16(6):128,130.
[4] Fabret C,Ehrlich S D,Noirot P. A new mutation delivery system for genome-scale approaches in Bacillus subtilis[J].Molecular Microbiology,2002,46(1):25-36. doi:10.1046/j.1365-2958.2002.03140.x.
[5] 吴果果,宋淑婷,岳荣,张晶,关莹,王玥,刘宝爱,吕学敏,魏建军,张会图. 反向筛选标记基因upp在杀真菌链霉菌遗传改造中的应用[J].中国生物工程杂志,2019,39(11):78-86. doi:10.13523/j.cb.20191109.
Wu G G,Song S T,Yue R,Zhang J,Guan Y,Wang Y,Liu B A,Lü X M,Wei J J,Zhang H T. Application of counterseletable gene upp in genetic manipulation of Streptomyces fungicidicus[J].China Biotechnology,2019,39(11):78-86.
[6] Zhiting Luo,Yuan Guo,Jidong Liu,Hua Qiu,Mouming Zhao,Wei Zou,Shubo Li. Microbial synthesis of poly-γ-glutamic acid:current progress,challenges,and future perspectives[J].Biotechnol Biofuels,2016,9:134.doi:10.1186/s13068-016-0537-7.
[7] Ogata F,Nagai N,Kawasaki N. Adsorption capability of cationic dyes(methylene blue and crystal violet)onto poly-γ-glutamic acid[J].Chemical and Pharmaceutical Bulletin,2017,65(3):268-275. doi:10.1248/cpb.c16-00827.
[8] Hsueh Y H,Huang K Y,Kunene S,Lee T Y. Poly-γ-glutamic acid synthesis,gene regulation,phylogenetic relationships,and role in fermentation[J].International Journal of Molecular Sciences,2017,18(12):2644. doi:10.3390/ijms18122644.
[9] Sakamoto S,Kawase Y. Adsorption capacities of poly-γ-glutamic acid and its sodium salt for cesium removal from radioactive wastewaters[J].Journal of Environmental Radioactivity,2016,165:151-158. doi:10.1016/j.jenvrad.2016.10.004.
[10] 李文婧,赵祥颖,田延军,张家祥,韩延雷,刘建军. γ-聚谷氨酸产生菌的发酵培养基优化[J].食品与发酵工业,2010,36(3):108-111,116. doi:10.13995/j.cnki.11-1802/ts.2010.03.009.
Li W J,Zhao X Y,Tian Y J,Zhang J X,Han Y L,Liu J J. Optimization of γ-PGA fermentation medium of Bacillus amyloliquefaciens by response surface methodology[J].Food and Fermentation Industries,2010,36(3):108-111,116.
[11] Ito Y,Tanaka T,Ohmachi T,Asada Y. Glutamic acid independent production of poly(γ-glutamic acid)by Bacillus subtilis TAM-4[J].Bioscience,Biotechnology,and Biochemistry,1996,60(8):1239-1242. doi:10.1271/bbb.60.1239.
[12] Shih I L,Wu P J,Shieh C J. Microbial production of a poly(γ-glutamic acid)derivative by Bacillus subtilis[J].Process Biochemistry,2005,40(8):2827-2832. doi:10.1016/j.procbio.2004.12.009.
[13] Wang Z,Chen G G,Wu H,Wang J,Liu Y L,Guo Y,Liang Z Q. Improvement of Bacillus subtilis for poly-γ-glutamic acid production by genome shuffling[J].Microbial Biotechnology,2016,9(6):824-833. doi:10.1111/1751-7915.12405.
[14] 张伟. 解淀粉芽孢杆菌LL3中无痕基因敲除方法的建立及其应用研究[D].天津:南开大学,2015.doi:10.7666/d.D795780.
Zhang W. The establishment and application of the method of seamless gene knockout in Bacillus amyloliquefaciens LL3[D]. Tianjin:Nankai University,2015.
[15] 冯俊. 代谢工程改造解淀粉芽胞杆菌提高γ-PGA产量[D].天津:南开大学,2016.doi:10.7666/d.Y3159102.
Feng J. Metabolic engineering of Bacillus amyloliquefaciens for γ-PGA overproduction[D].Tianjin:Nankai University,2016.
[16] 张慧莉. 微生物高效生产γ-聚谷氨酸和β-聚苹果酸的研究[D].杭州:浙江大学,2012.
Zhang H L. Studies on efficient bioproductions of poly(γ-glutamic acid)and poly(β-L-malic acid)[D].Hangzhou:Zhejiang University,2012.
[17] 白雪,张慧莉,黄冲,遆秀秀,冯斌. 解淀粉芽孢杆菌草酸脱羧酶基因的克隆、原核表达与活力测定[J].江苏农业科学,2019,47(12):66-70. doi:10.15889/j.issn.1002-1302.2019.12.013.
Bai X,Zhang H L,Huang C,Ti X X,Feng B. Cloning,prokaryotic expression and activity determination of Bacillus amyloliquefaciens oxalate decarboxylase gene[J].Jiangsu Agricultural Sciences,2019,47(12):66-70.
[18] Wang X L,Dai S Y,Wang Q J,Xu H N,Shi H Q,Kang Y B,Zha D M. Efficient markerless gene deletions in Pseudomonas protegens Pf-5 using a upp-based counterselective system[J].Biotechnology Letters,2020,42(2):277-285. doi:10.1007/s10529-019-02772-5.
[19] Ara K,Ozaki K,Nakamura K,Yamane K,Sekiguchi J,Ogasawara N. Bacillus minimum genome factory:Effective utilization of microbial genome information[J].Biotechnology and Applied Biochemistry,2007,46(3):169-178.doi:10.1042/ba20060111.
[20] Sakai K ,Sonoda C,Murase K. Bitterness relieving agent. JP Patent WO0021390,2000.
[21] Ogunleye A,Bhat A,Irorere V U,Hill D,Williams C,Radecka I. Poly-γ-glutamic acid:Production,properties and applications[J].Microbiology ,2015,161(Pt 1):1-17. doi:10.1099/mic.0.081448-0.
[22] Sung M H,Park C,Kim C J,Poo H,Soda K,Ashiuchi M. Natural and edible biopolymer poly-γ-glutamic acid:Synthesis,production,and applications[J].The Chemical Record,2005,5(6):352-366. doi:10.1002/tcr.20061.
[23] Yeh C M,Wang J P,Lo S C,Chan W C,Lin M Y. Chromosomal integration of a synthetic expression control sequence achieves poly-γ-glutamate production in a Bacillus subtilis strain[J].Biotechnology Progress,2010,26(4):1001-1007. doi:10.1002/btpr.417.
[24] Mitsui N,Murasawa H,Sekiguchi J. Disruption of the cell wall lytic enzyme CwlO affects the amount and molecular size of poly-γ-glutamic acid produced by Bacillus subtilis(natto)[J].The Journal of General and Applied Microbiology,2011,57(1):35-43. doi:10.2323/jgam.57.35.
[25] Liu J,Ma X,Wang Y,Liu F,Qiao J Q,Li X Z,Gao X W,Zhou T. Depressed biofilm production in Bacillus amyloliquefaciens C06 causes γ-polyglutamic acid(γ-PGA)overproduction[J].Current Microbiology,2011,62(1):235-241. doi:10.1007/s00284-010-9696-0.
