大麻(Cannabis sativa L.)是大麻科一年生草本植物[1]。大麻的种子为火麻仁,是一种传统中药材,药理作用包括润肠、通便、抗疲劳、护肝等。大麻在我国具有悠久的种植和利用历史,同时也是起源地之一[2]。大麻素为大麻植株产生的特有化学物质,主要在雌蕊及萼片的腺毛中含量较高[3]。大麻二酚(CBD)和四氢大麻酚(THC)是2种主要的大麻素物质,THC具有成瘾性,可导致人体焦虑、致幻及免疫力下降,是被严格管控的物质[4-5]。大麻二酚(CBD)具有止痛消炎、抗癫痫、抗焦虑及抗失眠等生理活性功能,被广泛用于药品、保健品等领域[5]。
CBD在大麻植株体内的前体物质为大麻二酚酸(CBDA),在大麻植株的生物合成路径已较为清楚[3]。它主要包括甲基赤藓糖醇-4-磷酸、脂肪酸和大麻素(Cannabinoid)3个代谢途径。MEP代谢途径的IPP异构酶以及香叶基二磷酸(GPP)合成酶催化顺式异戊烯己基二磷酸(IPP)形成;脂肪酸代谢途径的橄榄酸环化酶和橄榄醇合成酶能将丙二酰辅酶A经过一系列的酶促反应形成2,4-二羟基-6-戊基苯甲酸。然后在大麻素途径中,在大麻醇酸合成酶的作用下,底物2,4-二羟基-6-戊基苯甲酸和香叶基二磷酸进一步合成了大麻醇酸(CBGA)。最后,在大麻二酸合成酶催化下CBGA合成大麻二酚酸(CBDA)。CBDA进一步在光、热等物理作用下发生脱羧基反应生成CBD。
大麻二酚酸合成酶(CBDAS)是大麻植株体内合成大麻二酚酸的限速酶,它主要由大麻二酚酸合成酶基因CsCBDAS编码[6-7]。CBDAS基因是第一个在生物界中报道的大麻二酚酸合成酶基因,它从墨西哥纤用大麻中分离,酶活试验证明它具有利用CBGA催化合成CBDA的功能[8-9]。该基因除了在大麻CBD合成具有一定功能报道外,其他生物学功能未见相关研究报道。FAD结构域和BBE结构域为CBDAS蛋白的典型结构域,FAD结构域为氧化还原反应所必需,BBE结构域与酶的催化活性有关[9]。这些研究证明,CBDAS在大麻素合成中有一定功能,但是大麻CBDAS基因家族的全基因组分析未见相关研究报道。本研究旨在对大麻CBDAS基因进行全基因组分析,分析其理化性质、进化关系、染色体分布、基因结构和表达模式,以期为后期大麻CBDAS基因功能研究奠定基础。
1 材料和方法
1.1 大麻CBDAS基因的全基因组鉴定、理化特性分析及基因染色体定位分析
从NCBI网站库(https://www.ncbi.nlm.nih.gov/)下载大麻的全基因组、蛋白组及注释文件(GCA_900626175.2)。利用TBtools软件[10],分别以已报道的CBDAS基因FAD domain和BBE domain的氨基酸序列为输入序列进行本地Blast序列比对(E-vaule<1E5),筛选出大麻中包含FAD domain和BBE domain的CBDAS候选基因。将初筛获得的候选基因序列输入在线数据库SMART(http://smart.embl-heidelberg.de/)分析其结构域特征,同时具有FAD domain和BBE domain结构域的序列即为CBDAS家族基因。利用ExPASy(http://web.expasy.org/protparam/)在线预测大麻CBDAS蛋白的理化性质。使用TBtools绘制CBDAS家族基因所对应的染色体物理位置图。
1.2 大麻CBDAS蛋白系统进化分析、基因结构预测及启动子区域响应元件分析
大麻CBDAS蛋白系统进化树的构建采用邻接法(Neigh-Joining,NJ)(MEGA 7.0),Bootstrap值设置为1 000。根据基因组序列及注释文件使用TBtools软件分析基因结构,包括编码序列及非翻译区。MEME(http://meme-suite.org/tools/meme)网站预测CBDAS蛋白保守基序(Motif),设定预测值为10。利用TBtools软件提取5个CBDAS基因上游2 kb序列,PlantCare(http://www.plantcare.co.uk/)预测其顺式作用元件,并使用TBtools软件对其可视化构图。DNAMAN软件进行CBDAS蛋白序列的比对分析。
1.3 大麻CBDAS基因共线性预测
从NCBI网站下载大豆、水稻和大麻的基因组、蛋白组及注释文件,利用软件TBtools计算并绘制大麻CBDAS基因3种作物间的共线性关系并进行共线性关系进行可视化分析。
1.4 大麻CBDAS基因组织特异性、光照及Cd胁迫处理下基因表达量分析
通过大麻品种雌性植株的雌蕊、茎、叶、根等组织的表达量检测来反映大麻CBDAS基因的组织特异性。大麻高大麻二酚(CBD)品种C8和低CBD含量品种庆大麻1号第1对真叶展开后,挑选若干植株移植室外培养,开花后,挑选3株长势均匀的、且已开花的C8雌株,分别取其雌蕊、茎、叶、根等组织,迅速放入液氮,用于提取RNA。同时取庆大麻1号雌蕊和叶片进行RNA的提取。每个组织设3个生物学重复。
大麻品种C8发芽后,挑选若干长势基本一致的植株置于室外培养。待长至10对叶时,分别进行光照处理和黑暗处理。光照处理为将植株培养于光强度700 μmol/(m·s),温度为24 ℃,湿度为60%的光照培养箱;黑暗处理除无光照之外,其他处理与光照处理一致。分别在处理4,8 h后取C8的叶片组织进行RNA提取,每个处理设3个生物学重复。根据中国农业科学院麻类研究所一年生麻类作物遗传育种团队Cd胁迫下的RNA-seq数据找到所有大麻CBDAS家族基因表达量值(FPKM)用于表达量分析[11]。cDNA合成用Prime Script RT reagent Kit(Perfect Real Time)试剂盒(TaKaRa),采用2-ΔΔCT 值计算CBDAS家族基因的相对表达量,所用引物如表1所示。
表1 CsCBDAS基因家族实时荧光定量引物
Tab.1 qRT-PCR for CsCBDAS
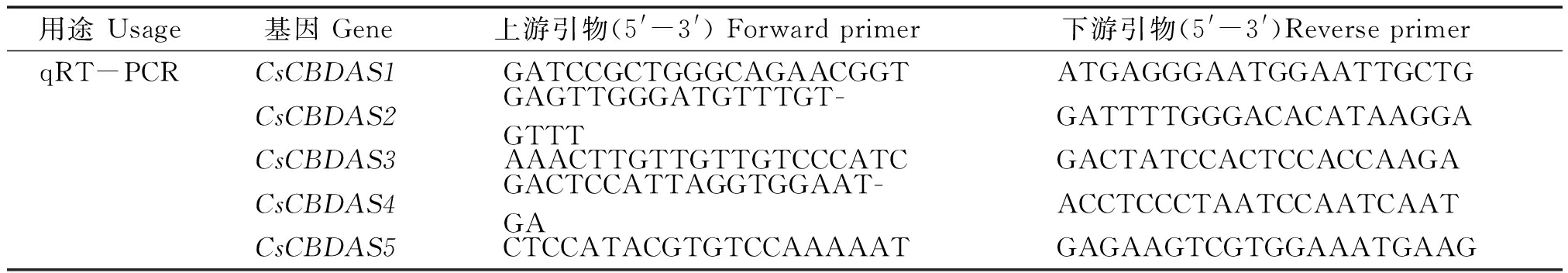
用途 Usage基因 Gene上游引物(5'-3') Forward primer下游引物(5'-3')Reverse primerqRT-PCRCsCBDAS1GATCCGCTGGGCAGAACGGTATGAGGGAATGGAATTGCTGCsCBDAS2GAGTTGGGATGTTTGT-GTTTGATTTTGGGACACATAAGGACsCBDAS3AAACTTGTTGTTGTCCCATCGACTATCCACTCCACCAAGACsCBDAS4GACTCCATTAGGTGGAAT-GAACCTCCCTAATCCAATCAATCsCBDAS5CTCCATACGTGTCCAAAAATGAGAAGTCGTGGAAATGAAG
2 结果与分析
2.1 CBDAS基因家族成员的鉴定与理化性质分析
利用CsCBDAS蛋白序列中FAD及BBE结构域保守序列分别与大麻蛋白序列进行比对,从大麻基因组中鉴定出5个成员,依次命名CsCBDAS1~CsCBDAS5。如表2所示,大麻CBDAS基因编码区序列1 626~1 638 bp,其氨基酸介于541~545 aa,蛋白相对分子质量大小为61.496 66~62.414 59 ku,等电点介于6.90~9.90;脂肪指数介于82.61~90.02,其中CsCBDAS3的脂肪指数最低,CsCBDAS4的脂肪指数最高。
表2 大麻中CBDAS基因家族成员基本信息
Tab.2 Basic information of the CBDAS family in cannabis
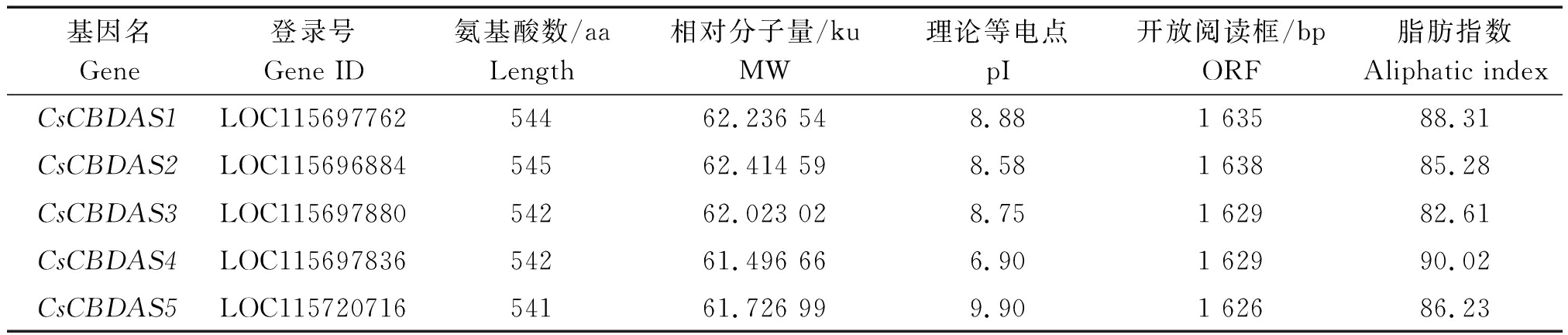
基因名Gene登录号Gene ID氨基酸数/aaLength相对分子量/kuMW理论等电点pI开放阅读框/bpORF脂肪指数Aliphatic indexCsCBDAS1LOC11569776254462.236 548.881 63588.31CsCBDAS2LOC11569688454562.414 598.581 63885.28CsCBDAS3LOC11569788054262.023 028.751 62982.61CsCBDAS4LOC11569783654261.496 666.901 62990.02CsCBDAS5LOC11572071654161.726 999.901 62686.23
2.2 大麻CBDAS基因结构、蛋白保守序列、系统进化分析及启动子顺式作用元件分析
大麻CBDAS基因仅在第2,7染色体分布,分别含有1,4个基因成员,CsCBDAS5位于第2号染色体,其余4个CBDAS基因家族成员位于第7染色体(图1)。系统发育表明,5个CsCBDAS基因分为3个亚家族。大麻CBDAS基因家族不含内含子,仅含有1个外显子(图2)。利用MEME在线预测CsCBDAS家族motif,大麻CBDAS家族成员都含有7种保守基序:motif 1-4、motif 6、motif 9、motif 10,表明这些motif基序在进化过程种具有较高的保守性(图2)。
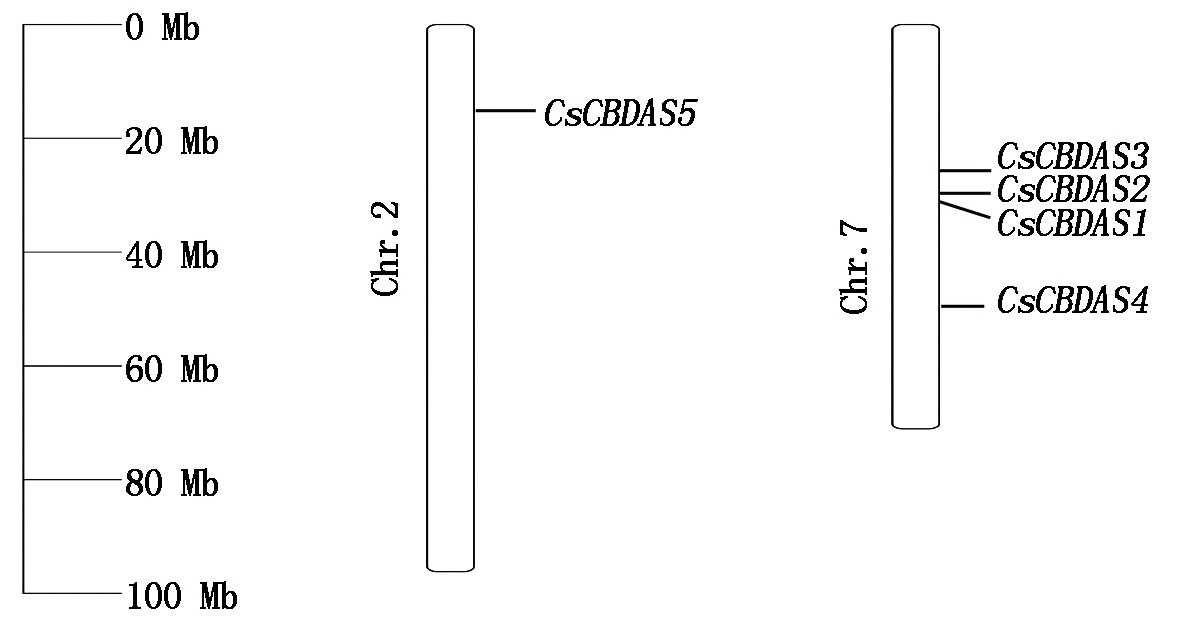
图1 CsCBDAS基因家族成员在大麻染色体上的定位
Fig.1 Chromosomal locations of CsCBDAS genes
in cannabis
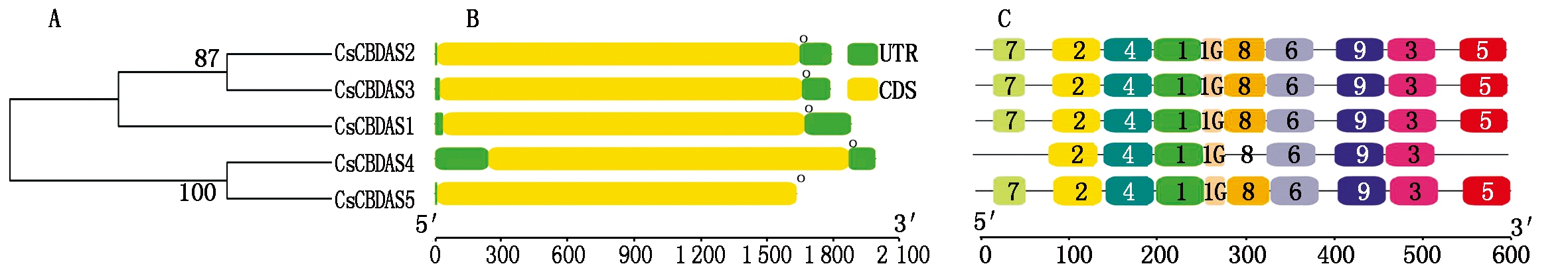
图2 CsCBDAS基因家族系统发育分析(A)、基因结构(B)及蛋白保守基序(C)
Fig.2 Phylogenetic analyses of CsCBDAS(A),gene structure of CsCBDAS(B)and conserved motif of CsCBDAS proteins(C)
CBDAS基因首次在大麻植株中发现,目前在其他作物未有相应的研究报道。为了研究大麻CBDAS基因系统进化关系,首先在NCBI数据库中查找CBDAS基因在物种中的分布情况。如表3所示,生物界共报道CBDAS基因家族成员299个,植物、动物、真菌都存在CBDAS基因家族成员。植物包含了289个CBDAS基因家族成员,且以双子叶植物分布居多。进一步挑选了大麻、水稻、二倍体棉花、海蜗牛(动物界)、尖端赛多孢菌(微生物界)等5个物种进行了系统进化树的构建(图3)。如图3所示,CBDAS基因包含了GroupⅠ、GroupⅡ和GroupⅢ。其中大麻CBDAS基因家族成员主要属于Group Ⅰ,与二倍体棉花亲缘关系较近;GroupⅡ只包含单子叶植物水稻1个成员;Group Ⅲ 包含海蜗牛(动物界)和尖端赛多孢菌(真菌界)的CBDAS基因成员各1个。且除CsCBDAS4外,大麻CBDAS基因家族4个成员都聚类在一个分枝上(图3)。
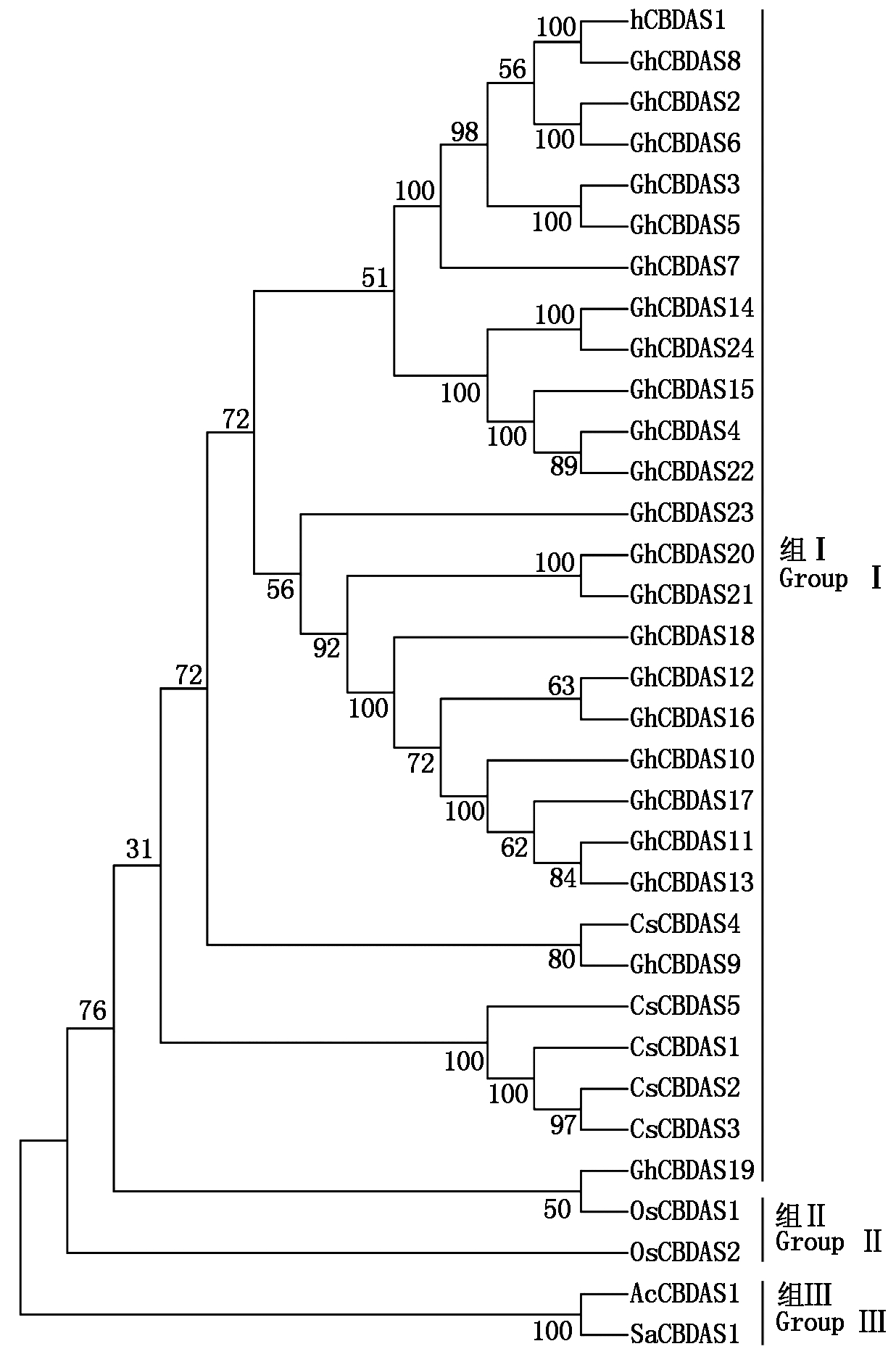
图3 大麻(Cs)、水稻(Os)、二倍体棉花(Gh)、海蜗牛
(Ac)、尖端赛多孢菌(Sa)CBDAS基因家族系统发育分析
Fig.3 Phylogenetic analysis of CBDAS gene family in Cannabis
sativa (Cs),Oryza sativa (Os),Gossypium hirsutum (Gh),
Aplysia californica (Ac),Scedosporium apiospermum (Sa)
利用在线软件PlantCare分析5个大麻CBDAS成员启动子区域顺式作用元件,如图4所示,CBDAS基因启动子包含光响应、伤诱导响应、激素响应元件、MYB和MYC位点结合等4类顺式作用元件。其中光响应元件、MYB和MYC结合元件尤为丰富。
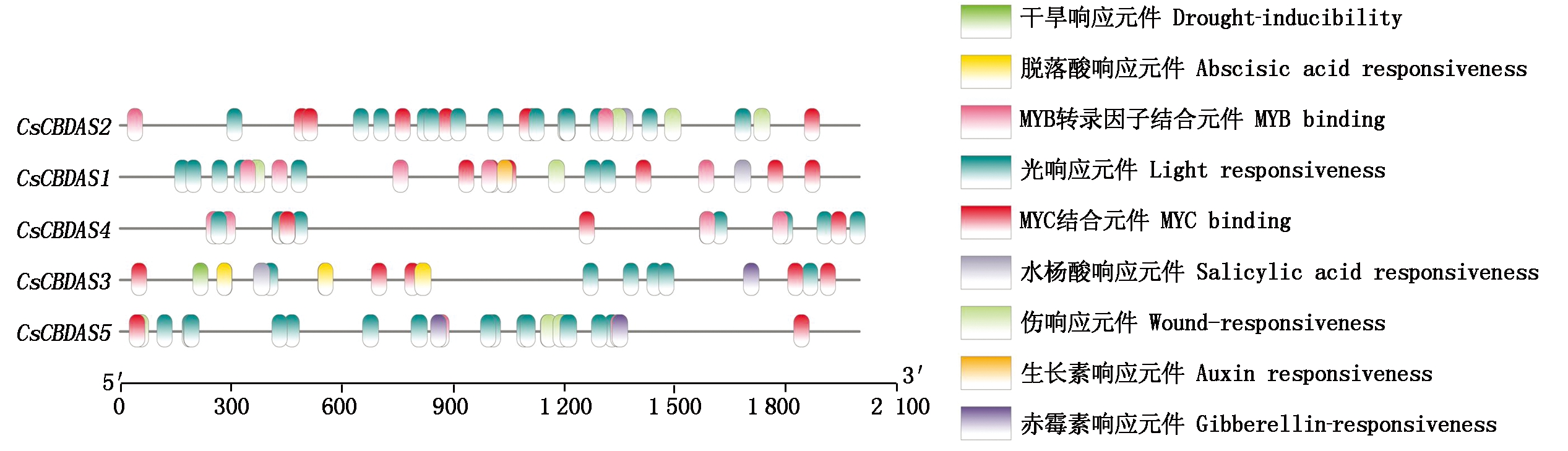
图4 CsCBDAS基因启动子顺式作用元件分析
Fig.4 Analysi of cis-elements in CsCBDAS promoters
2.3 大麻CBDAS基因的共线性分析
为了研究大麻CBDAS基因的进化机制,构建了大麻与大豆、水稻的CBDAS家族共线性关系图,这2种作物分别属于双子叶和单子叶植物。如图5所示,大麻CsCBDAS2只在双子叶大豆鉴定出直系同源基因,而在单子叶水稻中未鉴定出。暗示着除CsCBDAS2外,大麻CBDAS基因家族其他成员可能是大麻与其他单、双子叶功能差异的相关基因。
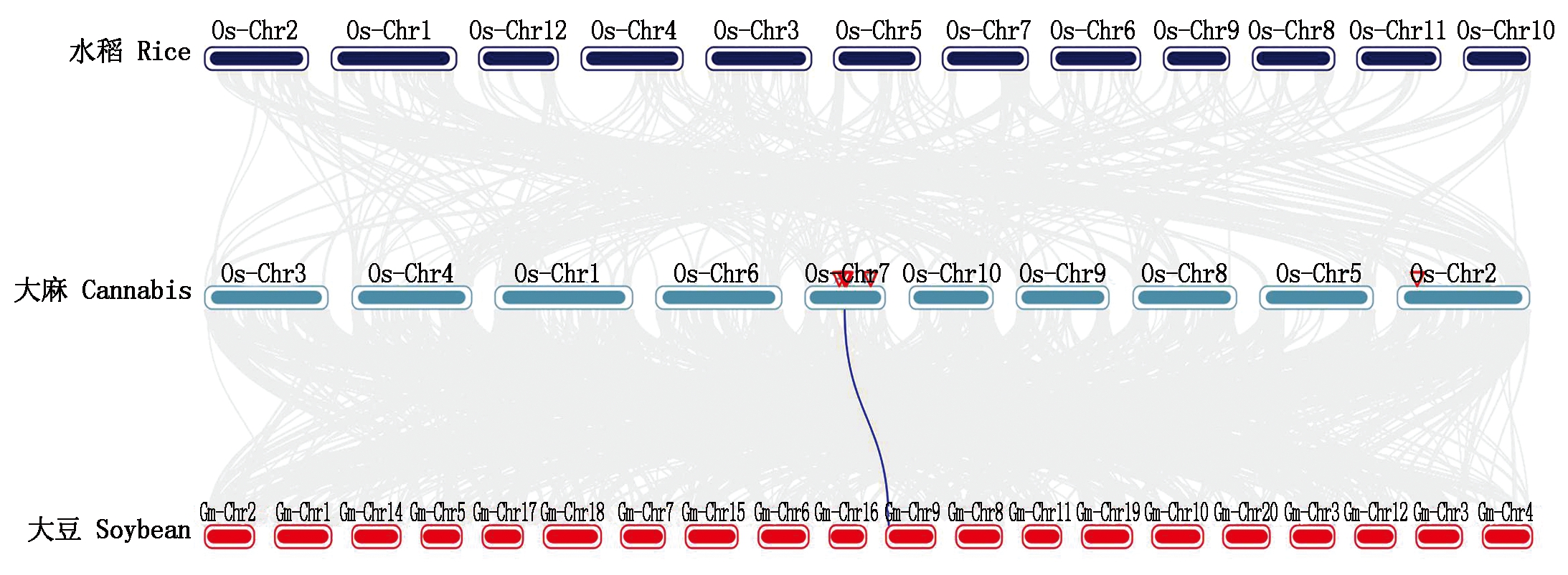
灰色线条代表大麻与水稻、大豆共线性区域;蓝色线条突出显示了共线性CsCBDAS2基因对。
Gray lines in the background indicate the collinear blocks between Cannabis;Rice and soybean,
while the blue lines highlight the syntenic CsCBDAS2 gene pairs.
图5 大麻与水稻、大豆的CBDAS基因共线性分析
Fig.5 Synteny analysis of CBDAS genes between cannabis,rice and soybean
2.4 CsCBDAS基因组织特异性表达分析
为了探讨大麻CsCBDAS基因在大麻不同组织中的表达特性,利用qRT-PCR对雌蕊盛开时期植株的不同组织(雌蕊、根、茎、叶)进行表达量分析,并利用TBtools软件进行了热图绘制。如图6所示,CsCBDAS1和CsCBDAS2在雌蕊中表达量最高,CsCBDAS4和CsCBDAS5在根中表达量最高,而CsCBDAS3在大麻叶中表达量最高。这些结果暗示着这些基因可能在不同组织中发挥着不同功能。
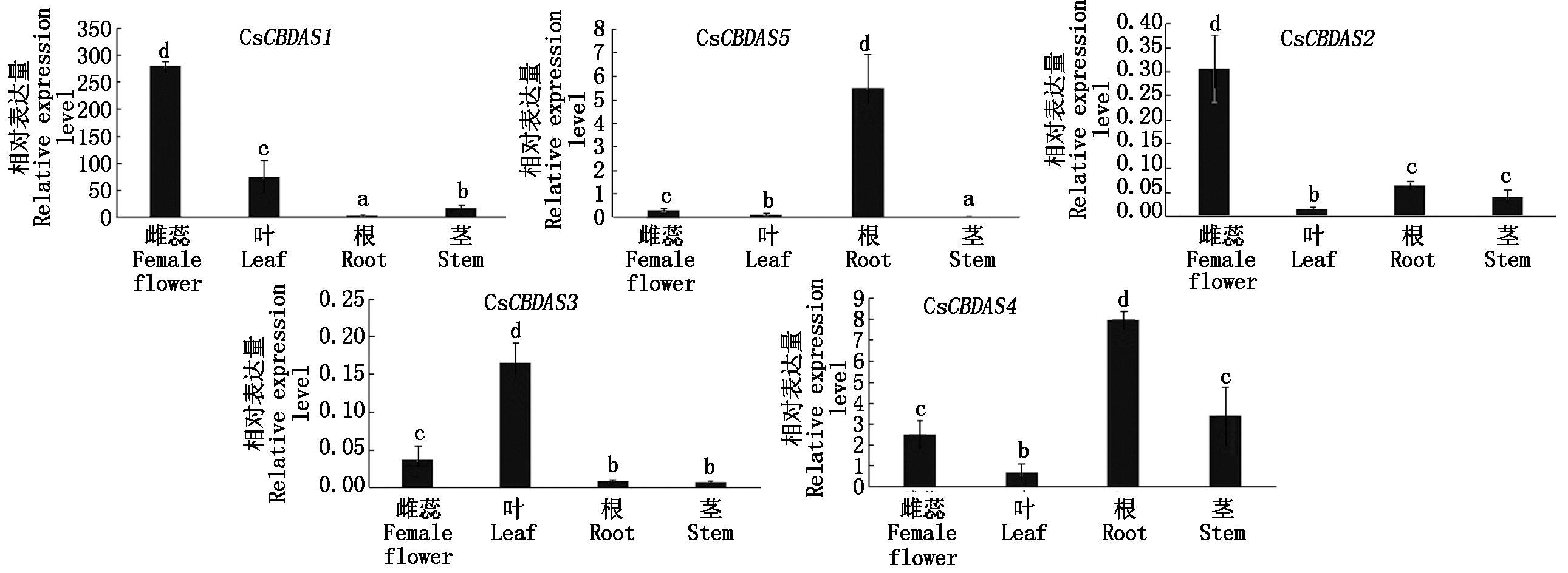
不同组织间差异显著用不同小写字母表示(P<0.05)。
Significant differences among different tissues were indicatied by different small letters(P<0.05)
图6 CsCBDAS基因家族成员在大麻不同组织中的表达模式分析
Fig.6 Transcript profiles of CsCBDAS genes in different tissues
2.5 光照及镉胁迫处理下CsCBDAS基因家族成员表达分析
前人研究结果表明,不同光照处理直接影响大麻植株CBD含量。同时由图4结果可知,大麻CsCBDAS基因启动子富含光响应元件,其表达水平可能受到光照影响。为了验证这一猜想,进一步对光照和黑暗处理下CsCBDAS表达量进行了分析。如图7所示,CsCBDAS2和CsCBDAS5在光照处理下其表达量显著高于黑暗处理,尤其CsCBDAS5,在光照处理下其表达约为黑暗处理的5倍;CsCBDAS1在光照条件下的表达量显著降低,CsCBDAS3和CsCBDAS4在光照处理和黑暗处理间其表达量无显著变化。
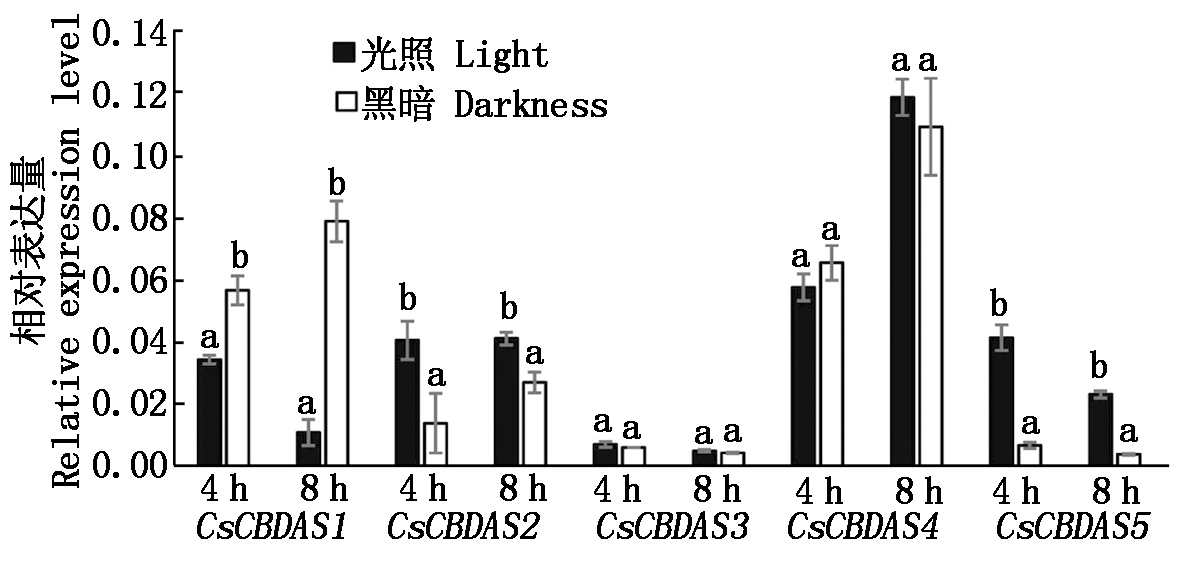
同一时间点光照处理和黑暗处理间显著差异
用不同小写字母表示(P<0.05)。
Significant differences among light and darkness treatment at the
same point were indicated by different small letters(P<0.05).
图7 光照和黑暗处理下大麻CBDAS基因家族表达量分析
Fig.7 Transcript analysis of CBDAS family genes under
light and dark treatment in cannabis
植物次生代谢产物被广泛报道参与植物Cd胁迫反应[12-13]。CBD为大麻特有的次生代谢产物,为了探究其合成相关基因在大麻应对镉胁迫反应中的功能,对其在镉胁迫下的基因表达量进行了分析。如图8所示,在镉处理中,CsCBDAS1和CsCBDAS2在Cd处理下其表达量显著低于对照处理,CsCBDAS4和CsCBDAS5受到镉处理的诱导表达,而CsCBDAS3表达量无显著变化。
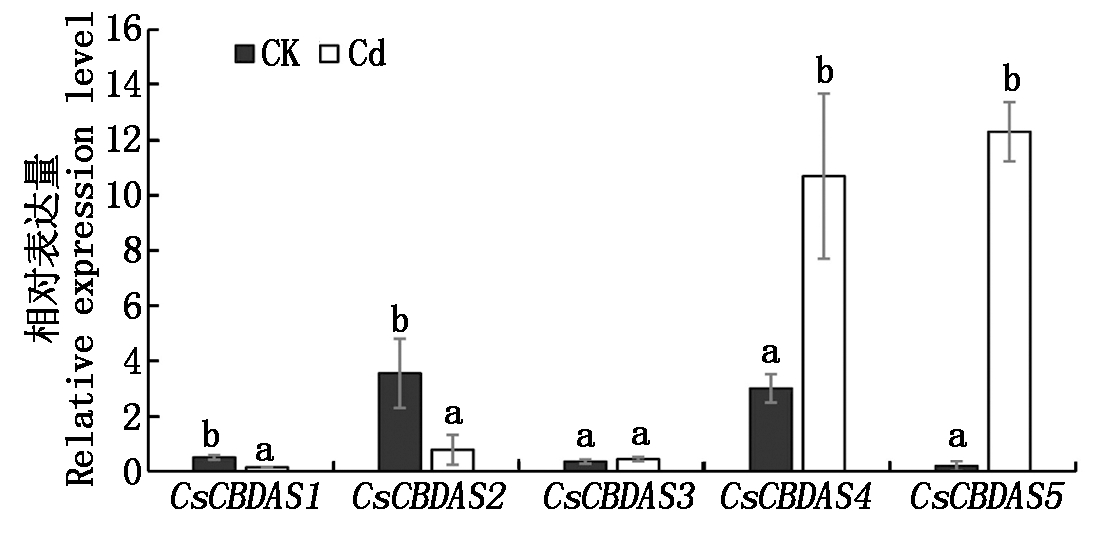
不同处理间差异显著用不同小写字母表示(P<0.05)。
Significant differences among different treatment were indicated
by different small letters(P<0.05).
图8 Cd胁迫处理下大麻CBDAS基因家族表达量分析
Fig.8 Expression analysis of CBDAS family
genes under Cd treatment in cannabis
为了进一步研究大麻CBDAS家族基因在大麻CBD合成中的功能,对不同CBD含量材料的叶和雌蕊中的表达量进行了分析。如图9所示,大麻品种C8在雌蕊和叶片中其CBD的含量显著高于庆大麻1号。C8品种雌蕊和叶片中CsCBDAS1表达量显著高于庆大麻1号品种,CsCBDAS3在庆大麻1号品系雌蕊组织中表达量高于C8品种,其在两品种叶片组织中的表达量无显著差异,而CsCBDAS2、CsCBDAS4和CsCBDAS5基因在C8和庆大麻1号两品种雌蕊和叶片中表达量无显著差异。
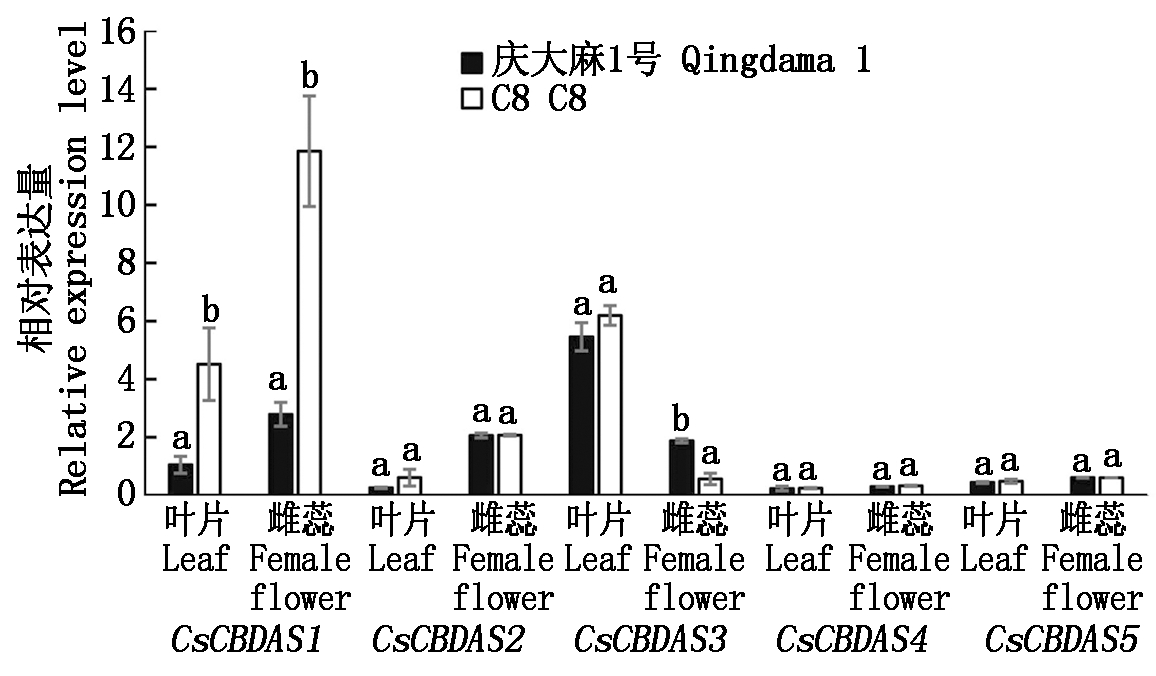
不同品种在同一组织中差异显著用不同小写字母表示(P<0.05)。
Significant difference among the same tissue of different
varieties were indicated by different small letters(P<0.05).
图9 大麻CBDAS家族基因在庆大麻1号和C8
品种雌蕊和叶片中基因表达量分析
Fig.9 Expression analysis of CBDAS family genes
in female and leaf of Qingdama 1 and C8
3 讨论
大麻二酚酸(CBDA)是大麻特有的酚类物质,它是大麻二酚(CBD)的前体物质。CBDAS合酶在大麻CBDA合成中起着重要的作用。随着高通量技术的发展,大麻基因组测序质量得到了显著的提高,基因家族分析成为研究基因功能有效的生物信息学手段。目前,COL、MYB、bHLH、GRAS等基因家族成员在大麻中被鉴定出来,并对其成员进行了系统的研究和分析[14-16],但CBDAS基因家族在大麻中未见相关研究报道。
本研究利用生物信息学方法,在大麻中鉴定出5个CBDAS基因家族成员,这些成员只分布在第2,第7号染色体。基因结构分析表明,5个CBDAS基因家族成员不含内含子,属于一类无内含子基因家族,暗示大麻CBDAS基因可能具有较快的进化速度[17]。构建了与单子叶植物水稻、双子叶植物二倍体棉花、动物海蜗牛及真菌类尖端赛多孢菌中CBDAS基因系统进化树后发现,CBDAS在来自生物界不同物种中分为3个家族,大麻CBDAS基因家族成员与双子叶二倍体棉花亲缘关系较近,与水稻次之,和动物海蜗牛及真菌类尖端赛多孢菌亲缘关系最远,这也符合大麻与这几个物种之间的进化关系[16]。此外,大麻4个CBDAS家族成员较好分在同一亚家族,暗示大麻CBDAS基因的独特性。进一步研究发现,大麻CBDAS基因与双子叶大豆有1对直系同源基因,而在单子叶水稻中未鉴定其直系同源基因,表明这些基因存在于单双子叶植物分化后。
顺式调控元件的预测可初步判断该基因家族成员的基本功能[18]。如图4所示,CsCBDAS1、CsCBDAS2及CsCBDAS5启动子区域富含光响应元件,同时研究发现,CsCBDAS2和CsCBDAS5基因受到光照处理诱导表达,表明这些光响应元件可能作为正调控元件诱导CsCBDAS1和CsCBDAS2基因的表达,同时也作为负调控元件抑制CBDAS1的表达,暗示这些基因可能通过不同的光响应方式来参与大麻的生长发育。前人研究表明,光照能够显著提高叶片和雌蕊中CBD含量[19]。综合本试验研究结果推测,光照处理可能通过提高CsCBDAS2和CsCBDAS5的表达量来增加大麻植株CBD含量。
前人研究报道表明,植物主要通过根部来响应重金属胁迫[20]。本研究中,CsCBDAS4和CsCBDAS5在根中表达量最高,且这2个基因受到重金属Cd胁迫的诱导表达,上调倍数高达5倍以上,说明这2个基因可能参与大麻镉胁迫响应。前人研究报道表明,在重金属污染土壤种植大麻能显著提高其CBD含量,且CsCBDAS1基因受到重金属诱导表达[21]。与Husain等[21]研究结果不同,本试验中,CsCBDAS1未受重金属镉胁迫的诱导,可能是由于本研究中重金属胁迫为单一镉胁迫,而Husain等[21]研究中为多种重金属胁迫,也可能是镉胁迫主要调控CsCBDAS1转录后水平而非转录水平。
前人研究报道表明,大麻CsCBDAS1基因在雌蕊表达量最高,且其表达量在高CBD含量品种中显著高于毒品大麻品种[3]。与前人研究结果类似[3],大麻CsCBDAS1基因在雌蕊中表达量最高,且其表达量在高CBD含量品系叶和雌蕊中高于低CBD含量品种。在未来大麻高CBD含量品种选育中,可以借助转基因手段通过提高CsCBDAS1基因的表达量来提高大麻品种中CBD的含量。
本研究中利用生物信息学分析和时空表达分析筛选出的这些差异基因可为大麻CBDAS基因家族成员生物学功能研究奠定基础,但后续研究中仍需要进一步通过转基因技术对这些候选基因进行生物学功能验证。
[1] Hurgobin B,Tamiru-Oli M,Welling M T,Doblin M S,Bacic A,Whelan J,Lewsey M G.Recent advances in Cannabis sativa genomics research[J].New Phytologist,2021,230(1):73-89.doi:10.1111/nph.17140.
[2] Crocq M A.History of Cannabis and the endocannabinoid system?[J].Dialogues in Clinical Neuroscience,2020,22(3):223-228.doi:10.31887/DCNS.2020.22.3/mcrocq.
[3] van Bakel H,Stout J M,Cote A G,Tallon C M,Sharpe A G,Hughes T R,Page J E.The draft genome and transcriptome of Cannabis sativa[J].Genome Biology,2011,12(10):R102.doi:10.1186/gb-2011-12-10-r102.
[4] Grof C P L.Cannabis,from plant to pill[J].British Journal of Clinical Pharmacology,2018,84(11):2463-2467.doi:10.1111/bcp.13618.
[5] Salami S A,Martinelli F,Giovino A,Bachari A,Arad N,Mantri N.It is our turn to get Cannabis high:put cannabinoids in food and health baskets[J].Molecules,2020,25(18):4036.doi:10.3390/molecules25184036.
[6] Taura F,Sirikantaramas S,Shoyama Y,Yoshikai K,Shoyama Y,Morimoto S.Cannabidiolic-acid synthase,the chemotype-determining enzyme in the fiber-type Cannabis sativa[J].FEBS Letters,2007,581(16):2929-2934.doi:10.1016/j.febslet.2007.05.043.
[7] Laverty K U,Stout J M,Sullivan M J,Shah H,Gill N,Holbrook L,Deikus G,Sebra R,Hughes T R,Page J E,van Bakel H.A physical and genetic map of Cannabis sativa identifies extensive rearrangements at the THC/CBD acid synthase loci[J].Genome Research,2019,29(1):146-156.doi:10.1101/gr.242594.118.
[8] Taura F,Morimoto S,Shoyama Y.Purification and characterization of cannabidiolic-acid synthase from Cannabis sativa L.[J].The Journal of Biological Chemistry,1996,271(29):17411-17416.doi:10.1074/jbc.271.29.17411.
[9] Zirpel B,Kayser O,Stehle F.Elucidation of structure-function relationship of THCA and CBDA synthase from Cannabis sativa L.[J].Journal of Biotechnology,2018,284:17-26.doi:10.1016/j.jbiotec.2018.07.031.
[10] Chen C J,Chen H,Zhang Y,Thomas H R,Frank M H,He Y H,Xia R.TBtools:an integrative toolkit developed for interactive analyses of big biological data[J].Molecular Plant,2020,13(8):1194-1202.doi:10.1016/j.molp.2020.06.009.
[11] Huang Y M,Li D F,Zhao L N,Chen A G,Li J J,Tang H J,Pan G,Chang L,Deng Y,Huang S Q.Comparative transcriptome combined with physiological analyses revealed key factors for differential cadmium tolerance in two contrasting hemp(Cannabis sativa L.)cultivars[J].Industrial Crops and Products,2019,140:111638.doi:10.1016/j.indcrop.2019.111638.
[12] Zhu H H,Ai H L,Hu Z R,Du D Y,Sun J,Chen K,Chen L.Comparative transcriptome combined with metabolome analyses revealed key factors involved in nitric oxide(NO)-regulated cadmium stress adaptation in tall fescue[J].BMC Genomics,2020,21(1):601.doi:10.1186/s12864-020-07017-8.
[13] Mishra B,Chand S,Singh Sangwan N.ROS management is mediated by ascorbate-glutathione-α-tocopherol triad in co-ordination with secondary metabolic pathway under cadmium stress in Withania somnifera[J].Plant Physiology and Biochemistry,2019,139:620-629.doi:10.1016/j.plaphy.2019.03.040.
[14] Pan G,Li Z,Yin M,Huang S Q,Tao J,Chen A G,Li J J,Tang H J,Chang L,Deng Y,Li D F,Zhao L N.Genome-wide identification,expression,and sequence analysis of CONSTANS-like gene family in Cannabis reveals a potential role in plant flowering time regulation[J].BMC Plant Biology,2021,21(1):142.doi:10.1186/s12870-021-02913-x.
[15] Bassolino L,Buti M,Fulvio F,Pennesi A,Mandolino G,Milc J,Francia E,Paris R.In silico identification of MYB and bHLH families reveals candidate transcription factors for secondary metabolic pathways in Cannabis sativa L.[J].Plants,2020,9(11):1540-1069.doi:10.3390/plants9111540.
[16] 尹明,杨大为,唐慧娟,潘根,李德芳,赵立宁,黄思齐.大麻GRAS转录因子家族的全基因组鉴定及镉胁迫下表达分析[J].作物学报,2021,47(6):1054-1069.doi:10.3724/SP.J.1006.2021.04078.
Yin M,Yang D W,Tang H J,Pan G,Li D F,Zhao L N,Huang S Q.Genome-wide identification of GRAS transcription factor and expression analysis in response to cadmium stresses in hemp(Cannabis sativa L.)[J].Acta Agronomica Sinica,2021,47(6):1054-1069.
[17] Liu H,Lyu H M,Zhu K K,van de Peer Y,Max Cheng Z M.The emergence and evolution of intron-poor and intronless genes in intron-rich plant gene families[J].The Plant Journal,2021,105(4):1072-1082.doi:10.1111/tpj.15088.
[18] Zhou S M,Zhang H L,Li R S,Hong Q,Li Y,Xia Q F,Zhang W.Function identification of the nucleotides in key cis-element of DYSFUNCTIONAL TAPETUM1 (DYT1)promoter[J].Frontiers in Plant Science,2017,8:153.doi:10.3389/fpls.2017.00153.
[19] Wei X Y,Zhao X L,Long S H,Xiao Q M,Gou Y,Qiu C S,Qiu H J,Wang Y F.Wavelengths of LED light affect the growth and cannabidiol content in Cannabis sativa L.[J].Industrial Crops and Products,2021,165:113433.doi:10.1016/j.indcrop.2021.113433.
[20] Lin Y F,Aarts M G M.The molecular mechanism of zinc and cadmium stress response in plants[J].Cellular and Molecular Life Sciences,2012,69(19):3187-3206.doi:10.1007/s00018-012-1089-z.
[21] Husain R,Weeden H,Bogush D,Deguchi M,Soliman M,Potlakayala S,Katam R,Goldman S,Rudrabhatla S.Enhanced tolerance of industrial hemp(Cannabis sativa L.)plants on abandoned mine land soil leads to overexpression of cannabinoids[J].PLoS One,2019,14(8):e0221570.doi:10.1371/journal.pone.0221570.
