生长素在植物生长发育以及对环境适应方面具有重要作用,如参与调控植物器官发育、对生物逆境胁迫和非生物逆境胁迫的响应等[1-3]。生长素主要通过一系列蛋白组成的生长素信号通路发挥功能,该信号转导通路上游主要包含3个基因家族成员,即TIR1/AFB生长素受体家族蛋白、AUX/IAA家族蛋白和生长素响应因子ARF家族蛋白[4]。当植物内源生长素含量升高时,生长素分子结合并激活生长素受体TIR1/AFB,被激活的TIR1/AFB可与AUX/IAA蛋白结合并通过泛素-26S蛋白酶体途径将其降解,随着AUX/IAA蛋白的降解,该蛋白对ARF转录因子的抑制作用被解除,进而引发对下游生长素响应基因的转录调控,激活生长素信号通路[5]。
ARF家族基因作为生长素信号通路的组分之一,在植物生长发育过程的许多方面均具有调控作用。在拟南芥中,共有23个ARF基因被鉴定出来,不同的ARF基因所发挥的功能也具有多样性。如ARF1和ARF2可调节拟南芥叶片衰老和花的凋谢,且在功能上存在冗余[6]。ARF3通过与细胞分裂素信号的互作调控拟南芥花原基的形成[7]。ARF8可影响拟南芥中茉莉酸的含量并在调节下胚轴伸长、顶端优势和侧根形成方面发挥功能[8]。在番茄中,相比于野生型番茄,ARF4基因缺失突变体对盐胁迫和渗透胁迫的耐受性增强[9]。在杨树中,过表达ARF1可以增加植株对木霉菌侵染的响应[10]。
茎瘤芥(Brassica juncea var.tumida)为十字花科芸薹属植物,是重庆地区的重要经济作物之一,其膨大茎是制作中国名特产品“涪陵榨菜”的主要原料。榨菜产业可贡献很大的经济效益,仅重庆涪陵区2020年的榨菜产业总产值为120亿元,为大量茎瘤芥种植农户带来收益。为实现重庆地区的优势榨菜产业可持续发展,重庆蔬菜产业发展“十三五”规划中把强化科技创新,注重研发高产优质的榨菜加工专用品种作为工作重点。在茎瘤芥栽培过程中,瘤茎产量低、盐胁迫和根肿菌胁迫等问题仍对榨菜产业发展带来巨大挑战。基于生长素在调控植物器官发育、植物抵御生物和非生物逆境胁迫中的多方面作用,本研究以生长素信号通路中的重要组分ARF家族基因为研究对象,对这些基因在茎瘤芥生长发育过程和逆境条件下的表达模式进行检测,挖掘出一些在瘤茎中特异表达的以及对盐胁迫和根肿菌胁迫有响应的ARF基因成员,为进一步研究它们的功能奠定基础,同时为研发高产优质茎瘤芥品种提供潜在的基因资源。
1 材料和方法
1.1 植物材料及培养
试验所用茎瘤芥材料为重庆地区栽培品种永安小叶。将种子播种在含蛭石∶营养土(3∶1)的花盆中,在植物培养室中进行培养,培养温度为22 ℃,光周期为16 h光照/8 h黑暗。对生长至生育期的植物各组织(根、茎、叶、花、种荚)进行取样,液氮速冻后放置于-80 ℃冰箱,用于后续表达检测试验。盐胁迫处理方法为:选取培养室中培养2周龄的茎瘤芥幼苗,用200 mmol/L的氯化钠灌根处理0,6,12,24,48 h后对幼苗根系进行取样,其中以灌根处理的0 h的幼苗根系作为对照;根肿菌胁迫处理方法为:选取2周龄的茎瘤芥幼苗,用5 mL根肿菌休眠孢子提取液(OD600=0.07)灌根处理0,12,24,36,72 h后对幼苗根系取样,其中以灌根处理的0 h的幼苗根系作为对照,根肿菌休眠孢子的提取参照杜艳等[11]的试验方法。每个处理的样品各取3株材料混样,取样进行3次生物学重复,用于后续基因表达分析。
1.2 生物信息学分析
在芸薹属基因组网站(http://brassicadb.cn/#/)中的茎瘤芥基因组数据库(Brassica juncea V1.5)中下载茎瘤芥ARF家族基因,基因ID参照Li等[12]发表的文章。利用DNAMAN和MEGA 7软件将茎瘤芥ARF家族蛋白与拟南芥ARF蛋白序列进行同源比对,依据比对结果对茎瘤芥ARF家族基因重新命名,以便直观显示与拟南芥ARF家族各成员高度相似的茎瘤芥基因。通过茎瘤芥基因组网站提供的信息统计茎瘤芥ARF的基因组长度、蛋白编码序列(CDS)长度、氨基酸长度及外显子数目。采用在线软件ExPASy(https://web.expasy.org/compute_pi/)预测蛋白等电点及分子量。采用TBtools软件进行基因串联重复分析以及计算ARF片段复制基因对的Ka/Ks值[13]。选择茎瘤芥ARF家族各基因“ATG”上游2 kb序列作为启动子,采用在线分析软件PLACE(https://www.dna.affrc.go.jp/PLACE/?action=newplace)进行启动子顺式作用元件分析。
1.3 茎瘤芥总RNA提取及基因的qPCR检测
采用SimGEN公司(杭州,中国)的植物总RNA提取试剂盒,参照试验方法说明对茎瘤芥材料的总RNA进行提取,使用全式金公司(北京,中国)的TransScript® One-Step gDNA Removal and cDNA Synthesis SuperMix试剂盒将提取出的总RNA反转录为cDNA,反转录加入模板RNA的用量为500 ng。将反转录得到的cDNA稀释10倍用于qPCR检测。采用罗氏(Roche)Light Cycler 480 Ⅱ荧光定量PCR仪进行qPCR分析,使用全式金公司TransStart® Tip Green qPCR SuperMix试剂盒配制10 μL反应体系:2×TransStart® Tip Green qPCR SuperMix,5 μL;cDNA 0.5 μL;正向引物(10 μmol/mL)0.5 μL;反向引物(10 μmol/mL)0.5 μL;ddH2O 3.5 μL。反应程序为:94 ℃ 30 s;(94 ℃ 5 s,60 ℃ 15 s,72 ℃ 15 s,收集荧光信号),40个循环;运行溶解曲线程序。qPCR反应所用,内参基因为茎瘤芥Bju18S rRNA(BjuA046942)[14],所用引物序列见表1。
表1 BjARF基因qPCR引物序列
Tab.1 Primer sequences of qPCR for BjARF genes

基因IDGene ID正向引物(5′-3′) Forward primer (5′-3′)反向引物(5′-3′)Reverse primer (5′-3′)BjuO001242CCTCTTGTAACCCTACCTCGTGCATTTGTTGCTCCAAACCTTGABjuA005337TTCCGAGTTACTGTTGCTTGCGCTTCGAGATACCTATTGACGCTBjuA004135GCATCTGATACAAGCACCCACACATCTTCGGCTACTAACTCCTGBjuB022969AGCACAATGCCTACTTCGGTTCCCCTACTATGAATTGGCTTATCBjuA036279CAGGTTGTTATCAGGATCAGCTGCCATGTGACCCTGAGGGAAGBjuA036277GCTGGAGATGATCCATGGAATGCGTTCCAAGAAGAAGAAGGCBjuA043023CCTAAGAGCCACGAGCCAAGTCGAATCCGAATAACCTACAGCAABjuO002114TTCTTGCCATCCACGCTAACAGGACTCCCTGTGATTTTCABjuB014200AGCAAACCCCAACCCAAGAGGTGCTCCAGCCTGTAGTCAABjuA032544GGAAGTTGGAGCCAGCACTATGAGCACGGAAGAGTCGGBjuA022872AAGGTACGGTGGGATGAGACCCGGAGTTGAAGGAGCTAGATTAGABjuB039668CCCGAGGCTATTCAAGACGGTGTCCGAAGCAGTCAAGGTBjuA012362GGAAGCCATGAGCAACAAGTCCAACAACCGTGCCAGAABjuB020448CCGTGAAGAATAACTACTCCATTGTCCATTTTGATTTTGCCCACBjuA015563GGACAACCCAAACGGCATCTGTGGACGAGTCGCTCTACGBjuB029506ATGCGGTTTAGGATGCTGTTCCTTGACGGATCTCCAATGAGBjuA034753AACGAGTGGAAGTTCAGGCATACAGAGTCACCAGCGACAAGCBjuB043964ATCCAACTCTACCACCACAACTAAGTACAAACGTATCCTTCTGCTCCBjuA029862CAAGTGTTCGTCGGTACATGGCACCCTCGGTTGCCTATCTBjuO010408ACAGCACAGCCGCCTACTCAAGCCCACAAACAAACTCCATCCTBjuA026141TAATCCAAGAGCGTGTCCAGCCAAACATCATCCCAAACCTCATACBjuB005807AGGATCAGTCAGGGACGAGAAGAGGGAGACAAACCAGAGGGBjuB043412GACTTTCCCACTACCATCATTCGGCATCAGGAGTTACTGCTTCAGGBjuA030797TCGCTGGTGATTCAGTTTTGTGAGTCGGTGTTTGTCTGTTTGCBjuA030401GAGCCTGGATGTAATGATAAGCCTGAGGTCAGTGAGGGAAAGATGBjuB029338CCCAGCATTCCCTTTAAGTCGCCAGATTCCACCACCGTTCABjuA041527TCGGGTTTCTTCCCTTCGTTGCGGATTCGGTTTGTTAABjuB043428AAGAGGAAGCAAGCACCCATTACGTGGCACTGAGAAACCCBjuA047156TTAGATGGCGGGGAAAACATGGCTCCTCCTCCAAACAABjuO008939AGCAGTTTCCCTCGTCCTGTCGGTGGGTTTTGTTTGTCCBjuO009800CTGGGTGGAGTGTATTTGTTAGTGAATTGTCTGTGGACGAGTGGCBjuA031864GTGGTGATCCTCTGAGACAGCCATTTGCTGCTGTTGGTGABjuB026602GGGAAGCCTTGTGCTGTATTTTGAGGAGGAAGCCCGTAAATBjuA008875GTTTGAAACTGAGGATTCAGGGAAACGCCACTTAGAACCAGACCATBjuA033621AATGGGGAATCACGAGTTGGCAATAGCATTCAGTGCAGTAGCABjuO007821TCACCCTCAAGCGGTCCTACCGTGGTCTCATCCCTCAACTBjuA018466ACCCTCAAGCGGTCCTAACGCCTCAACGGGAAACCAAACABjuA016899GATACAAGCACTCATGGAGGCTCGATAAATGTGGCGAAAACGBjuA038747CAAGGATCTCCATGGAGTAGAATGCCGCATCACCAGAGACAAGACBjuB034138GCTTGTGCTGGTCCTCTCTCCTGCAGAAGATTTGAGGACTGAGBjuO003639CAAGTTTCCGATGTTCTCCCCTGCCTGGCTCCCTGTATTCCBjuO011636TGACGGCTGAGTCGGTTATAGCAACGCCTTGACGCAAAABjuA003598ACAATGTTCCCGTGGGTTTACGATGGAGGAGGATGAGGTCGBjuB040611GTATGCCACCCGTGAACTCCTGAAATCGACATGATCGTAAGCBjuA042400CTACTTCCCTCAAGGACACGCCAAGACACGGCATAGAACCATABjuO012772GATGGAGATAAGATGCTTCTTGGACCTATCTTGGCACGGGTTTTBjuB008810TGGAGATAAGATGCTTCTTGGAGTGTTAGTGTTGGGTCATCCTCAG
表1(续)

基因IDGene ID正向引物(5′-3′) Forward primer (5′-3′)反向引物(5′-3′)Reverse primer (5′-3′)BjuB036675CGAGCCTTTCTCACATTCAGCGCACCTTCCATGCGAACCABjuA026944GCAGGGAGCCAGGCATTATCTTTTCTCAGCCCAGGAAGTGBjuB029911ATGAACTTTGCCAGTCCGCGCTCCTGCACGTTTATCACCTTBjuA027703TGGGACTTTCGCCACATCTCGTGAACGGACTTCCTCATGBjuB016738GCAAAACTTGAAGAGGGTGAATCTTTGATGGAGGGTAAGTGGCBjuA031544GTTATGGAAGTTATGTGCTGGACCCAAGTTGTCATTTGCTGATGCTACBj18S rRNATCTGACGCAAGCACAACTATCTTCTCAGCTTTAGCCATT
1.4 数据统计分析
采用2-ΔΔCT方法对qPCR结果数据进行分析,计算各基因相对于内参基因的表达量。使用Heml软件对ARF基因在茎瘤芥不同组织中表达的qPCR检测结果进行热图分析,利用SigmaPlot 10.0(Systat Software,Inc.,Chicago,IL,United States)软件对盐胁迫和根肿菌侵染胁迫的茎瘤芥根中ARF基因表达检测结果进行分析并作图,使用SPSS 16.0软件进行基因表达水平比较,选择One-way ANOVA的Duncan方法进行基因表达差异显著性检验,P<0.05时认为不同样本间存在显著差异。
2 结果与分析
2.1 茎瘤芥ARF基因基本信息分析
通过蛋白序列比对分析了茎瘤芥ARF蛋白与拟南芥ARF蛋白的序列一致性,并根据序列一致性的高低分别对茎瘤芥ARF基因重新命名(表 2)。结果表明,茎瘤芥中与拟南芥ARF5序列相似度较高的基因数目最多,为6个,分别命名为BjARF5A~BjARF5F,用同样的方法也对其他基因进行了重新命名。基因长度分析结果表明,茎瘤芥ARF基因长度在1 685(BjARF17B)~4 814 bp(BjARF7C),CDS长度在1 167(BjARF13C)~3 594 bp(BjARF7C),蛋白氨基酸序列长度在388(BjARF13C)~1 197 aa(BjARF7C)。对蛋白等电点的分析结果表明,茎瘤芥ARF蛋白等电点大小在5.19(BjARF17A)~8.55(BjARF17B),其中大部分等电点小于7,呈弱酸性。蛋白分子量分析结果表明,茎瘤芥ARF蛋白分子量在44.44(BjARF13C)~132.64 ku(BjARF7C)。
表2 茎瘤芥ARF基因信息及蛋白理化性质
Tab.2 Gene information and protein physicochemical properties of ARF genes of tuber mustard
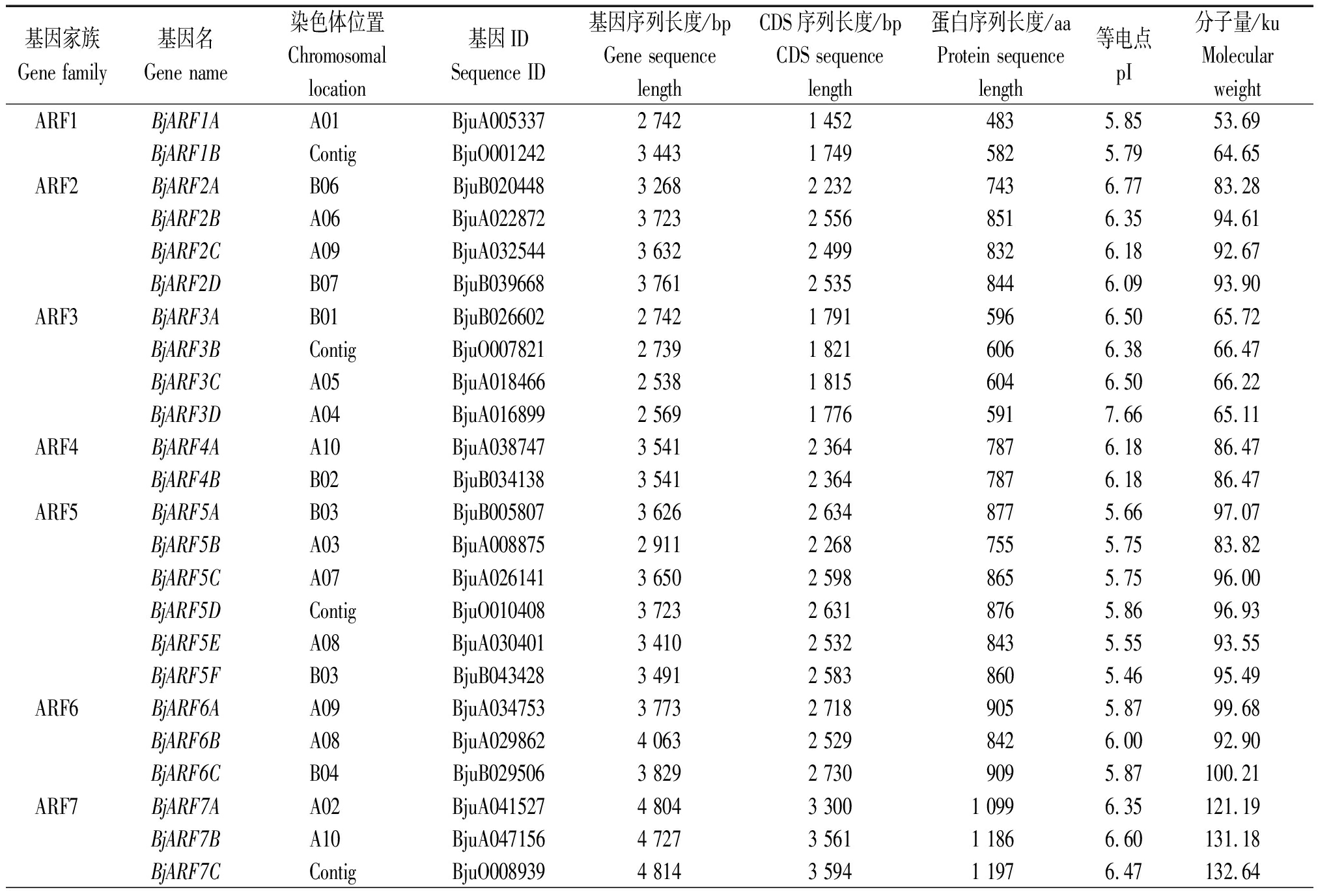
基因家族Gene family基因名Gene name染色体位置Chromosomallocation基因IDSequence ID基因序列长度/bpGene sequencelength CDS序列长度/bpCDS sequencelength 蛋白序列长度/aaProtein sequencelength 等电点pI分子量/kuMolecularweight ARF1BjARF1AA01BjuA0053372 7421 4524835.8553.69BjARF1BContigBjuO0012423 4431 7495825.7964.65ARF2BjARF2AB06BjuB0204483 2682 2327436.7783.28BjARF2BA06BjuA0228723 7232 5568516.3594.61BjARF2CA09BjuA0325443 6322 4998326.1892.67BjARF2DB07BjuB0396683 7612 5358446.0993.90ARF3BjARF3AB01BjuB0266022 7421 7915966.5065.72BjARF3BContigBjuO0078212 7391 8216066.3866.47BjARF3CA05BjuA0184662 5381 8156046.5066.22BjARF3DA04BjuA0168992 5691 7765917.6665.11ARF4BjARF4AA10BjuA0387473 5412 3647876.1886.47BjARF4BB02BjuB0341383 5412 3647876.1886.47ARF5BjARF5AB03BjuB0058073 6262 6348775.6697.07BjARF5BA03BjuA0088752 9112 2687555.7583.82BjARF5CA07BjuA0261413 6502 5988655.7596.00BjARF5DContigBjuO0104083 7232 6318765.8696.93BjARF5EA08BjuA0304013 4102 5328435.5593.55BjARF5FB03BjuB0434283 4912 5838605.4695.49ARF6BjARF6AA09BjuA0347533 7732 7189055.8799.68BjARF6BA08BjuA0298624 0632 5298426.0092.90BjARF6CB04BjuB0295063 8292 7309095.87100.21ARF7BjARF7AA02BjuA0415274 8043 3001 0996.35121.19BjARF7BA10BjuA0471564 7273 5611 1866.60131.18BjARF7CContigBjuO0089394 8143 5941 1976.47132.64
表2(续)
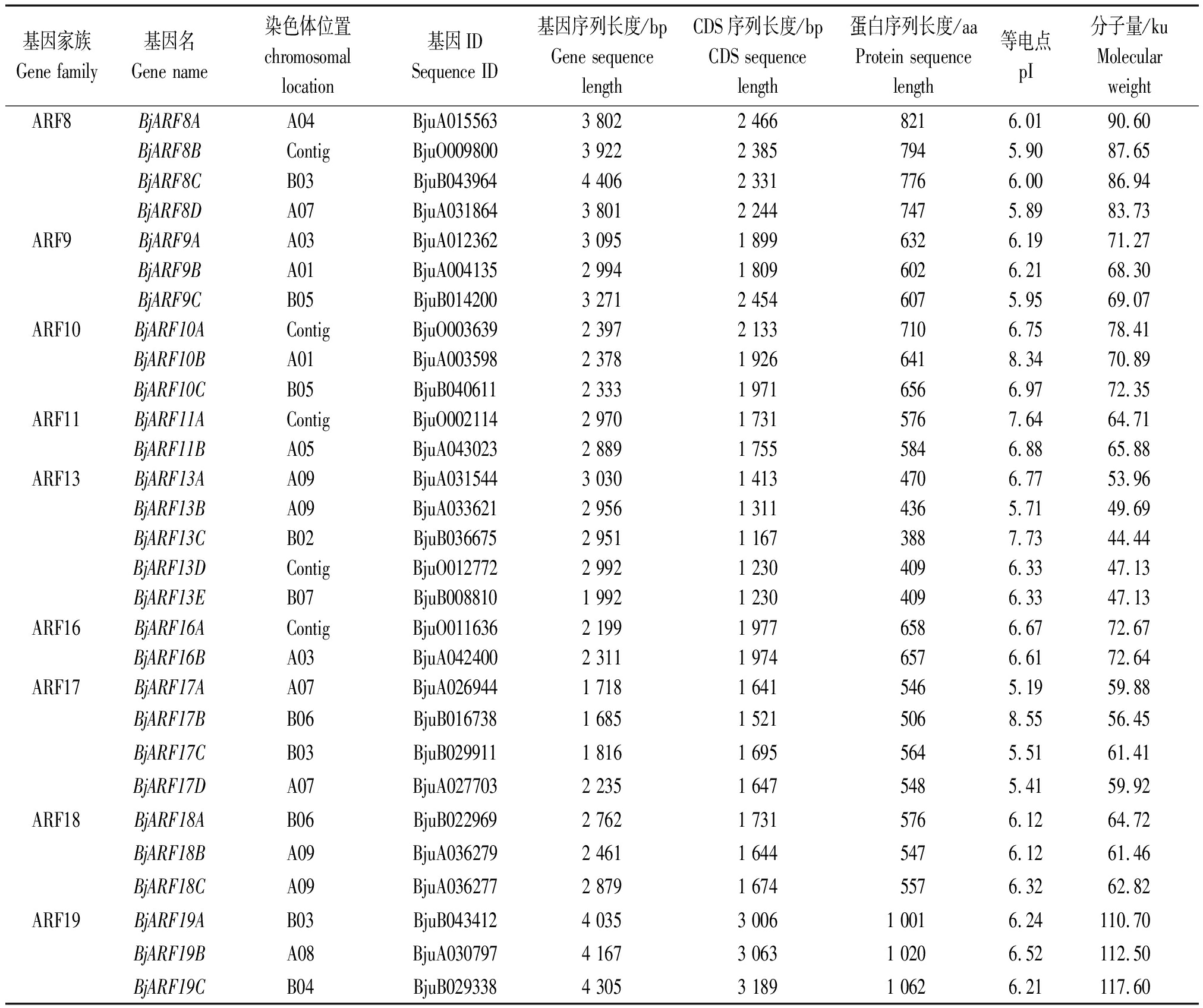
基因家族Gene family基因名Gene name染色体位置chromosomallocation基因IDSequence ID基因序列长度/bpGene sequencelength CDS序列长度/bpCDS sequencelength 蛋白序列长度/aaProtein sequencelength 等电点pI分子量/kuMolecularweight ARF8BjARF8AA04BjuA0155633 8022 4668216.0190.60BjARF8BContigBjuO0098003 9222 3857945.9087.65BjARF8CB03BjuB0439644 4062 3317766.0086.94BjARF8DA07BjuA0318643 8012 2447475.8983.73ARF9BjARF9AA03BjuA0123623 0951 8996326.1971.27BjARF9BA01BjuA0041352 9941 8096026.2168.30BjARF9CB05BjuB0142003 2712 4546075.9569.07ARF10BjARF10AContigBjuO0036392 3972 1337106.7578.41BjARF10BA01BjuA0035982 3781 9266418.3470.89BjARF10CB05BjuB0406112 3331 9716566.9772.35ARF11BjARF11AContigBjuO0021142 9701 7315767.6464.71BjARF11BA05BjuA0430232 8891 7555846.8865.88ARF13BjARF13AA09BjuA0315443 0301 4134706.7753.96BjARF13BA09BjuA0336212 9561 3114365.7149.69BjARF13CB02BjuB0366752 9511 1673887.7344.44BjARF13DContigBjuO0127722 9921 2304096.3347.13BjARF13EB07BjuB0088101 9921 2304096.3347.13ARF16BjARF16AContigBjuO0116362 1991 9776586.6772.67BjARF16BA03BjuA0424002 3111 9746576.6172.64ARF17BjARF17AA07BjuA0269441 7181 6415465.1959.88BjARF17BB06BjuB0167381 6851 5215068.5556.45BjARF17CB03BjuB0299111 8161 6955645.5161.41BjARF17DA07BjuA0277032 2351 6475485.4159.92ARF18BjARF18AB06BjuB0229692 7621 7315766.1264.72BjARF18BA09BjuA0362792 4611 6445476.1261.46BjARF18CA09BjuA0362772 8791 6745576.3262.82ARF19BjARF19AB03BjuB0434124 0353 0061 0016.24110.70BjARF19BA08BjuA0307974 1673 0631 0206.52112.50BjARF19CB04BjuB0293384 3053 1891 0626.21117.60
通过采用TBtools软件在茎瘤芥全基因组进行搜索并未发现串联重复的ARF基因对,但存在多个片段复制的ARF基因对,同时对这些基因对的Ka/Ks值进行了计算,结果表明,除BjARF4A∶BjARF4B基因对的Ka/Ks值没有得出外,其他16个基因对的Ka/Ks值均小于1,说明茎瘤芥ARF基因在进化过程中受到纯化选择压力的影响(表3)。
表3 茎瘤芥ARF片段复制基因对Ka/Ks分析
Tab.3 Analysis of Ka/Ks in ARF fragment replication
gene pairs in tuber mustard
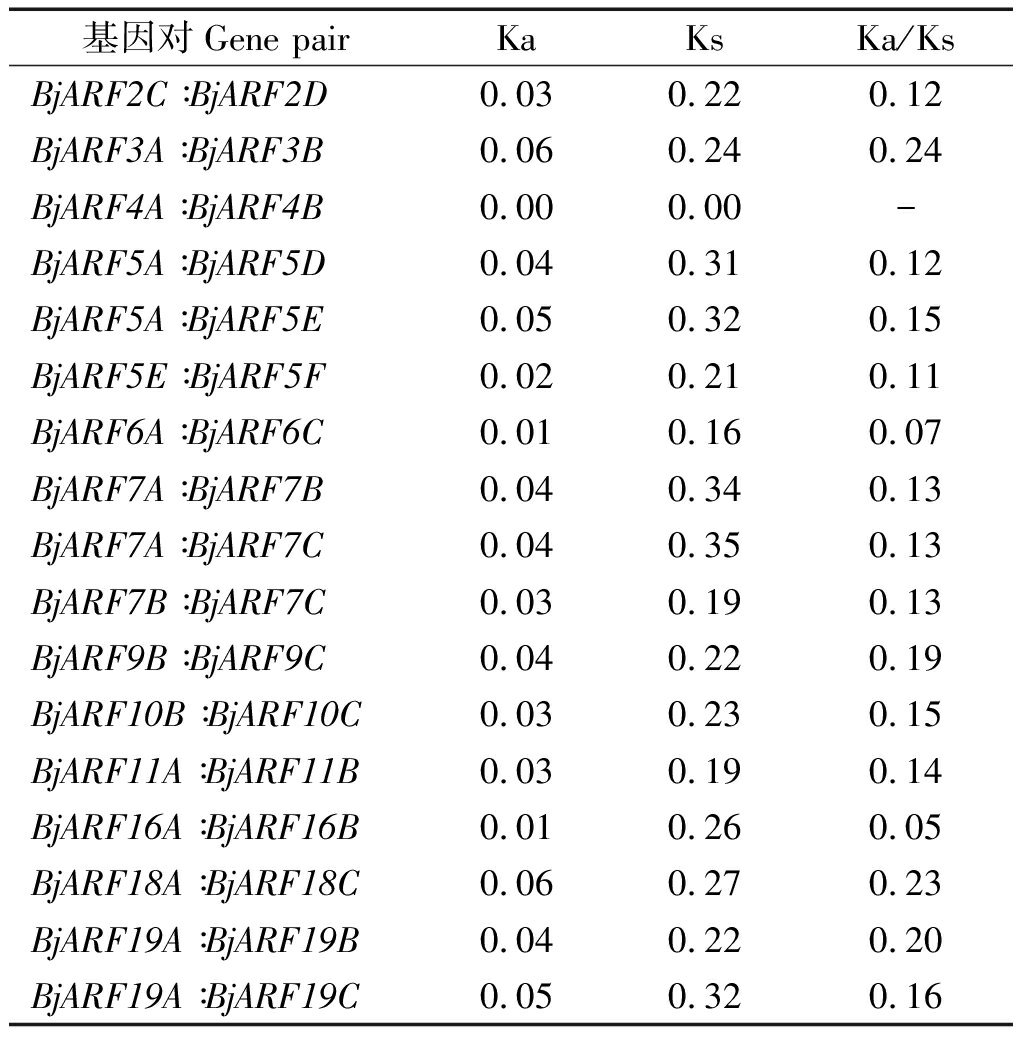
基因对Gene pairKaKsKa/KsBjARF2C∶BjARF2D0.030.220.12BjARF3A∶BjARF3B0.060.240.24BjARF4A∶BjARF4B0.000.00-BjARF5A∶BjARF5D0.040.310.12BjARF5A∶BjARF5E0.050.320.15BjARF5E∶BjARF5F0.020.210.11BjARF6A∶BjARF6C0.010.160.07BjARF7A∶BjARF7B0.040.340.13BjARF7A∶BjARF7C0.040.350.13BjARF7B∶BjARF7C0.030.190.13BjARF9B∶BjARF9C0.040.220.19BjARF10B∶BjARF10C0.030.230.15BjARF11A∶BjARF11B0.030.190.14BjARF16A∶BjARF16B0.010.260.05BjARF18A∶BjARF18C0.060.270.23BjARF19A∶BjARF19B0.040.220.20BjARF19A∶BjARF19C0.050.320.16
2.2 茎瘤芥ARF基因启动子顺式作用元件分析
为预测茎瘤芥ARF基因在响应盐胁迫和病原菌胁迫方面可能发挥的功能,对ARF各基因启动子所含有的相关顺式作用元件进行了分析。结果表明(图1),主要包含2种响应生长素相关的顺式作用元件(ARFAT和CATATGGMSAUR),一种响应生长素和水杨酸的顺式作用元件(ASF1MOTIFCAMV),2种响应病原菌相关的顺式作用元件(GCCCORE和MYB1LEPR),一种响应病原菌侵染和盐胁迫相关的顺式作用元件(GT1GMSCAM4)。其中,35个ARF基因启动子含有ARFAT元件,27个启动子含有CATATGGMSAUR元件,这2个元件是受生长素诱导表达的基因通常含有的,说明大部分茎瘤芥ARF基因均与生长素信号通路密切相关。所有ARF基因均含有2个以上GT1GMSCAM4元件,其中多个基因启动子上该元件数目超过10个,这些基因可能在茎瘤芥响应盐胁迫过程中具有功能。响应病原菌侵染的顺式作用元件GCCCORE和MYB1LEPR仅在少数ARF基因启动子上存在,且只含有一个,而响应效应子和病原菌侵染的顺式作用元件ELRECOREPCRP1则在25个启动子上被预测到,由于茎瘤芥主要病害根肿菌的效应子在其侵染茎瘤芥过程中具有重要作用,因此,启动子含有多个该元件的ARF基因,如BjARF9C(5个)、BjARF3D(3个)、BjARF13B(3个)可能在茎瘤芥抵御根肿菌过程中发挥功能。
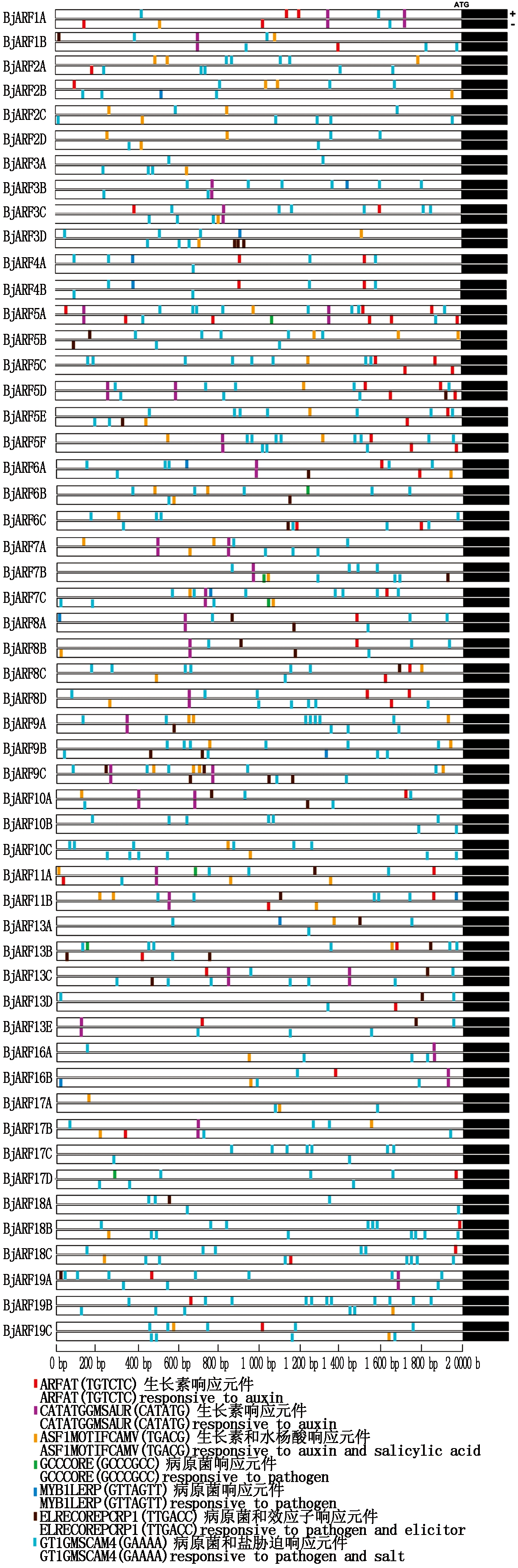
图1 茎瘤芥ARF启动子顺式作用元件分析
Fig.1 Analysis of cis-elements in the promoter of
ARF genes of tuber mustard
2.3 茎瘤芥ARF基因在不同器官中的表达分析
为探究不同ARF基因在茎瘤芥各个组织中可能发挥的功能,采用qPCR的方法对茎瘤芥根、茎、膨大茎、叶、花和种荚中的基因表达水平进行了检测,并利用2-ΔΔCT法计算了各个ARF基因相对于内参基因Bju18S rRNA的表达量。结果表明,相比于其他基因,BjARF1A、BjARF3D、BjARF4B和BjARF18B在检测的6个组织中相对表达量均较高(图2),推测这4个基因在茎瘤芥的各个组织中可能类似看家基因一样具有广泛的调控作用。BjARF1B、BjARF2A、BjARF2B、BjARF2C、BjARF2D、BjARF3A、BjARF4A、BjARF5E、BjARF5F、BjARF13A、和BjARF17D则至少在1个组织中具有较高的表达水平,说明这些基因可能对1个或几个组织生长发育的调控作用均有贡献。其他所检测的基因在各组织中的表达水平差异不大。通过组织表达水平检测还鉴定到一些在某一特定器官中具有高表达的基因,如相比于其他组织,BjARF1A和BjARF4B在叶中表达水平高,BjARF7C在根中表达水平高,BjARF2B和BjARF2C在种荚中表达水平最高;而BjARF6A虽然在各组织中的表达水平均较低,但其在膨大茎中的表达水平要高于其他5个组织,类似的BjARF7A在花组织中有特异的表达。这些在单一组织中具有特异表达模式的ARF基因值得进一步对其在调控该组织生长发育中的功能进行研究。

图2 茎瘤芥ARF基因在不同器官中的表达模式检测
Fig.2 The investigation of expression patterns of ARF genes in different organs of tuber mustard
2.4 茎瘤芥ARF基因在盐胁迫条件下的表达分析
为鉴定出可能参与茎瘤芥响应盐胁迫过程中发挥功能的ARF基因,对200 mmol/L的NaCl处理0~48 h的茎瘤芥根中的ARF基因表达水平进行了检测。结果表明,相比于对照,BjARF1B和BjARF2B在盐处理3 h后受到强烈的诱导表达,分别是未处理对照的40,303倍(图3),推测这2个基因在茎瘤芥响应盐胁迫的最初期可能发挥较大作用。此外,BjARF13E在盐胁迫处理6 h后表达水平上调14倍,BjARF9A在盐胁迫处理6 h后表达水平上调6倍,这2个基因也很可能在盐胁迫响应过程中发挥功能。同时,还检测到多个基因在盐胁迫处理48 h内持续下调表达,如BjARF2D、BjARF3A、BjARF6B、BjARF6C、BjARF10B和BjARF13A,其中BjARF2D和BjARF6B在盐处理3~48 h持续显著下调,而另外4个基因则在盐处理3 h后即显著下调,后期则在某一时间点有所恢复(图3)。这些鉴定到的受盐胁迫处理诱导表达或抑制表达的ARF基因可能参与茎瘤芥根系对盐胁迫的响应及可塑性发育,值得作为研究重点进一步研究它们在茎瘤芥抗盐方面的功能。

图3 茎瘤芥ARF基因在盐胁迫处理条件下表达模式检测
Fig.3 The investigation of expression patterns of ARF genes in tuber mustard under salt stress treatment
2.5 茎瘤芥ARF基因在根肿菌胁迫条件下的表达分析
为鉴定出可能参与茎瘤芥响应根肿菌侵染过程中发挥功能的ARF基因,对根肿菌休眠孢子处理0~72 h的茎瘤芥根中的ARF基因表达水平进行了检测。结果表明,相比于对照,BjARF3A和BjARF3D的表达水平分别在根肿菌侵染12,24 h后显著上调,如BjARF3D在72 h的表达水平为对照的4.6倍,在24 h的表达水平则达到对照的82倍(图4),这2个强烈受根肿菌侵染诱导表达的基因很可能在这一过程中发挥重要作用。BjARF13B在根肿菌处理6~72 h的表达量连续下调,且趋势逐渐加强,在处理72 h后,表达量已下降至0 h对照的2%,说明茎瘤芥很可能通过抑制该基因的表达完成对根肿菌侵染的响应,进一步验证该基因在茎瘤芥抵抗根肿病中的作用有重要意义。BjARF17C和BjARF18A在根肿菌处理12~72 h显著下调表达,说明这2个基因可能在这几个时间段发挥重要功能。这些在根肿菌侵染过程中表达显著变化的基因,无论受到诱导还是抑制作用,均可能在此过程中发挥作用,其具体生物学功能有待进一步验证。

图4 茎瘤芥ARF基因在根肿菌胁迫处理条件下表达模式检测
Fig.4 The investigation of expression patterns of ARF genes in tuber mustard under
Plasmodiophora brassicae stress treatment
3 讨论
在进化上,片段重复、串联重复和基因组重复是基因扩张的主要方式[15]。茎瘤芥是白菜(Brassica rapa)和黑芥(B.nigra)杂交所得的异源四倍体进化而来,基因组加倍是茎瘤芥ARF基因数目增加的主要动力[16]。同时,在茎瘤芥中存在多个ARF片段复制基因对,且受到纯化选择压力的影响,这也是ARF基因进化的动力之一。但是,在茎瘤芥中不存在串联重复产生的ARF基因对,与之相似,在大麦(Hordeum vulgare)和谷子(Setaria italica)中也未发现串联重复的ARF基因对[17-18]。启动子上的顺式作用元件对于基因的表达具有重要作用,经启动子预测发现,BjARF3D启动子含有3个响应病原菌和效应子的ELRECOREPCRP1元件,基因表达检测结果也发现该基因在根肿菌侵染24 h后其表达水平上调82倍,充分利用生物信息学分析与表达检测分析可为挖掘有价值的基因进行后续功能研究提供重要依据。已有研究表明,在大豆GmARF启动子上同样发现6个与生长素密切相关的CATATGGMSAUR作用元件,且该基因在下胚轴的表达受到生长素2,4-D的显著诱导[19],由该顺式作用元件在不同物种ARF基因启动子中被发现这一情况可推测其对生长素信号通路基因发挥功能具有重要意义。
基因的组织表达模式通常和它们所发挥的功能密切相关,在茎瘤芥中鉴定到一些在某一特定组织中高表达的基因,如BjARF1A和BjARF4B在叶中的表达水平高于其他组织,说明这2个基因与茎瘤芥叶的发育及功能相关。已有研究报道,在棉花(Gossypium raimondii)和桐油树(Physic nut)的叶组织中也检测到ARF4的高表达[20-21],这些证据进一步说明ARF4在调控叶片功能方面在多个物种中相对保守。生长素在调控植物根的可塑性发育方面发挥作用,其中ARF7和ARF19在植物侧根发育和根的向地性方面具有较大贡献,拟南芥arf7 arf19双重缺失突变体表现为侧根发育受抑制的表型[22]。在茎瘤芥中,BjARF7C在根中的相对高表达水平预示着该基因在茎瘤芥根系发生发育过程中可能也具有类似的调控作用。此外,茎瘤芥是一种茎食作物,其膨大茎的生长状况与其产量密切相关,通过基因表达模式检测发现,BjARF6A在膨大茎中的相对表达水平较高,可对该基因进一步深入研究以明确其调控瘤茎发育的具体功能,可为培育优质茎瘤芥品种提供潜在的基因资源。
盐胁迫是主要的非生物逆境胁迫之一,也是茎瘤芥栽培过程中面临的主要逆境,在茎瘤芥中挖掘一些影响植物耐盐相关的ARF基因可为缓解茎瘤芥因盐胁迫造成减产的现状提供帮助。在甘薯(Ipomoea batatas)中,ARF5基因在盐胁迫处理2~48 h之间受到诱导表达,同时在拟南芥中过表达该基因可增加植株对盐胁迫的耐受性[23]。在本研究中,4个茎瘤芥ARF基因受盐胁迫的诱导表达,预测它们也很有可能在茎瘤芥抗盐过程中发挥正调控作用。而在番茄(Solanum lycopersicum)中,抑制ARF4的表达则可增强植株对盐胁迫的耐受性[9],茎瘤芥中有7个ARF基因的表达水平受到盐胁迫的抑制作用,说明它们调控植物响应盐胁迫的方式可能与番茄ARF5类似。同为生长素信号通路的ARF基因,在盐胁迫条件下有的被诱导,而有的被抑制,可能是由于它们的结构不同而导致发挥功能的方式上存在差异,产生这种情况的可能是由于ARF作为转录因子,一些具有转录激活活性,一些具有转录抑制活性有关[24]。
生长素信号通路基因通常参与到植物对病原菌的防御反应中,如木薯对炭疽病的响应和拟南芥对丁香假单胞菌的抗性[25-26]。在单子叶植物慈竹(Bambusa emeiensis)中也发现,受害虫胁迫后,多个ARF基因上调表达[27]。在根肿菌处理条件下,在茎瘤芥中鉴定出2个显著上调表达的ARF基因(BjARF3A和BjARF3D),目前,已知ARF3在调控雌蕊发育中发挥功能且该功能在十字花科不同物种中保守[28],但其在其他方面的功能还未见报道,深入对它们在茎瘤芥响应根肿菌方面的功能研究可增加对该基因功能的认识。目前,在芥菜类植物中鉴定到的抗病相关的ARF基因仍然较少,在芥菜(Brassica juncea)中过表达来源于白芥(Sinapis alba)的ARF10可增强芥菜对脱落酸的敏感性和对甘蓝链格孢菌(Alternaria brassicicola)的抗性[29]。在茎瘤芥中采用过表达或基因沉默的方法可进一步验证这些受根肿菌侵染诱导或抑制表达的ARF基因在茎瘤芥抗根肿病方面的相关功能。
[1] Mironova V,Teale W,Shahriari M,Dawson J,Palme K.The systems biology of auxin in developing embryos[J].Trends in Plant Science,2017,22(3):225-235.doi:10.1016/j.tplants.2016.11.010.
[2] Ribba T,Garrido-Vargas F,O′Brien J A.Auxin-mediated responses under salt stress:From developmental regulation to biotechnological applications[J].Journal of Experimental Botany,2020,71(13):3843-3853.doi:10.1093/jxb/eraa241.
[3] Djami-Tchatchou A T,Harrison G A,Harper C P,Wang R H,Prigge M J,Estelle M,Kunkel B N.Dual role of auxin in regulating plant defense and bacterial virulence gene expression during Pseudomonas syringae PtoDC3000 pathogenesis[J].Molecular Plant-Microbe Interactions,2020,33(8):1059-1071.doi:10.1094/MPMI-02-20-0047-R.
[4] Lavy M,Estelle M.Mechanisms of auxin signaling[J].Development,2016,143(18):3226-3229.doi:10.1242/dev.131870.
[5] Gallei M,Luschnig C,Friml J.Auxin signalling in growth:Schrödinger's cat out of the bag[J].Current Opinion in Plant Biology,2020,53:43-49.doi:10.1016/j.pbi.2019.10.003.
[6] Ellis C M,Nagpal P,Young J C,Hagen G,Guilfoyle T J,Reed J W.AUXIN RESPONSE FACTOR1 and AUXIN RESPONSE FACTOR2 regulate senescence and floral organ abscission in Arabidopsis thaliana[J].Development,2005,132(20):4563-4574.doi:10.1242/dev.02012.
[7] Zhang K,Wang R Z,Zi H L,Li Y P,Cao X W,Li D M,Guo L,Tong J H,Pan Y Y,Jiao Y L,Liu R Y,Xiao L T,Liu X G.AUXIN RESPONSE FACTOR3 regulates floral meristem determinacy by repressing cytokinin biosynthesis and signaling[J].The Plant Cell,2018,30(2): 324-346.doi:10.1105/tpc.17.00705.
[8] Mao Z L,He S B,Xu F,Wei X X,Jiang L,Liu Y,Wang W X,Li T,Xu P B,Du S S,Li L,Lian H L,Guo T T,Yang H Q.Photoexcited CRY1 and phyB interact directly with ARF6 and ARF8 to regulate their DNA-binding activity and auxin-induced hypocotyl elongation in Arabidopsis[J].New Phytologist,2020,225(2):848-865.doi:10.1111/nph.16194.
[9] Bouzroud S,Gasparini K,Hu G J,Barbosa M A M,Rosa B L,Fahr M,Bendaou N,Bouzayen M,Zsögön A,Smouni A,Zouine M.Down regulation and loss of auxin response factor 4 function using CRISPR/Cas9 alters plant growth,stomatal function and improves tomato tolerance to salinity and osmotic stress[J].Genes, 2020,11(3):272.doi:10.3390/genes11030272.
[10] Wang Y F,Hou X Y,Deng J J,Yao Z H,Lyu M M,Zhang R S.AUXIN RESPONSE FACTOR 1 acts as a positive regulator in the response of poplar to Trichoderma asperellum inoculation in overexpressing plants[J].Plants, 2020,9(2):272.doi:10.3390/plants9020272.
[11] 杜艳,刘邮洲,刘永锋,李建斌,乔俊卿,许凌宇.甘蓝根肿病菌休眠孢子的生物学特性及侵染寄主的显微观察[J].西南农业学报,2016,29(6):1323-1327.doi:10.16213/j.cnki.scjas.2016.06.016.
Du Y,Liu Y Z,Liu Y F,Li J B,Qiao J Q,Xu L Y.Biological characteristics of Plasmodiophora brassicae resting spore and microscopic observation during pathogen infection[J].Southwest China Journal of Agricultural Sciences,2016,29(6):1323-1327.
[12] Li W B,Chen F B,Wang Y P,Zheng H Y,Yi Q Q,Ren Y,Gao J.Genome-wide identification and functional analysis of ARF transcription factors in Brassica juncea var.tumida[J].PLoS One,2020,15(4):e0232039.doi:10.1371/journal.pone.0232039.
[13] Chen C J,Chen H,Zhang Y,Thomas H R,Frank M H,He Y H,Xia R.TBtools:an integrative toolkit developed for interactive analyses of big biological data[J].Molecular Plant,2020,13(8):1194-1202.doi:10.1016/j.molp.2020.06.009.
[14] 孙全,蔡应繁,何晓红,曾鑫,江怀仲,王微娜.茎瘤芥根和茎组织转录组比较分析[J].四川大学学报(自然科学版),2013,50(5): 1090-1096.doi:10.3969/j.issn.0490-6756.2013.05.034.
Sun Q,Cai Y F,He X H,Zeng X,Jiang H Z,Wang W N.The root and stem's transcriptome datas analysis of Tumourous stem mustard(Brassica juncea var.tumida Tsen et Lee)[J].Journal of Sichuan University (Natural Science Edition),2013,50(5): 1090-1096.
[15] De Grassi A,Lanave C,Saccone C.Genome duplication and gene-family evolution:The case of three OXPHOS gene families[J].Gene,2008,421(1/2):1-6.doi:10.1016/j.gene.2008.05.011.
[16] Yang J H,Liu D Y,Wang X W,Ji C M,Cheng F,Liu B N,Hu Z Y,Chen S,Pental D,Ju Y H,Yao P,Li X M,Xie K,Zhang J H,Wang J L,Liu F,Ma W W,Shopan J,Zheng H K,Mackenzie S A,Zhang M F.The genome sequence of allopolyploid Brassica juncea and analysis of differential homoeolog gene expression influencing selection[J].Nature Genetics,2018,50(11): 1616.doi:10.1038/s41588-018-0227-4.
[17] 郭宝健,李赢,袁泽宸,吕超,张新忠,许如根.大麦ARF基因家族的全基因组分析[J].麦类作物学报,2016,36(11):1426-1432.doi:10.7606/j.issn.1009-1041.2016.11.03.
Guo B J,Li Y,Yuan Z C,Lü C,Zhang X Z,Xu R G.Genome-wide analysis of auxin response factor(ARF)family in barley[J].Journal of Triticeae Crops,2016,36(11):1426-1432.
[18] 赵艳,瓮巧云,马海莲,宋晋辉,袁进成,王凌云,董志平,刘颖慧.谷子ARF基因家族的鉴定与生物信息学分析[J].植物遗传资源学报,2016,17(3):547-554.doi:10.13430/j.cnki.jpgr.2016.03.022.
Zhao Y,Weng Q Y,Ma H L,Song J H,Yuan J C,Wang L Y,Dong Z P,Liu Y H.Genome-wide identification and bioinformatics analysis of ARF gene family in foxtail millet Setaria italica[J].Journal of Plant Genetic Resources,2016,17(3):547-554.
[19] 李思楠,苏安玉,于以成,张超,马彦龙,王玲爽,金杨媚,李文滨,武小霞.大豆再生相关基因GmARF的生物信息学及表达分析[J].基因组学与应用生物学,2015,34(10):2187-2193.doi:10.13417/j.gab.034.002187.
Li S N,Su A Y,Yu Y C,Zhang C,Ma Y L,Wang L S,Jin Y M,Li W B,Wu X X.Bioinformatics and expression analysis of regeneration related gene GmARF in soybean[J].Genomics and Applied Biology,2015,34(10):2187-2193.
[20] Sun R R,Wang K B,Guo T L,Jones D C,Cobb J,Zhang B H,Wang Q L.Genome-wide identification of auxin response factor(ARF)genes and its tissue-specific prominent expression in Gossypium raimondii[J].Functional & Integrative Genomics,2015,15(4): 481-493.doi:10.1007/s10142-015-0437-0.
[21] Tang Y H,Bao X X,Liu K,Wang J,Zhang J,Feng Y W,Wang Y Y,Lin L X,Feng J C,Li C W.Genome-wide identification and expression profiling of the auxin response factor(ARF)gene family in physic nut[J].PLoS One,2018,13(8): e0201024.doi:10.1371/journal.pone.0201024.
[22] Okushima Y,Overvoorde P J,Arima K,Alonso J M,Chan A,Chang C,Ecker J R,Hughes B,Lui A,Nguyen D,Onodera C,Quach H,Smith A,Yu G X,Theologis A.Functional genomic analysis of the AUXIN RESPONSE FACTOR gene family members in Arabidopsis thaliana:Unique and overlapping functions of ARF7 and ARF19[J].The Plant Cell,2005,17(2): 444-463.doi:10.1105/tpc.104.028316.
[23] Kang C,He S Z,Zhai H,Li R J,Zhao N,Liu Q C.A sweetpotato auxin response factor gene(IbARF5)is involved in carotenoid biosynthesis and salt and drought tolerance in transgenic Arabidopsis[J].Frontiers in Plant Science,2018,9: 1307.doi:10.3389/fpls.2018.01307.
[24] Roosjen M,Paque S,Weijers D.Auxin response factors:Output control in auxin biology[J].Journal of Experimental Botany,2018,69(2):179-188.doi:10.1093/jxb/erx237.
[25] Navarro L,Dunoyer P,Jay F,Arnold B,Dharmasiri N,Estelle M,Voinnet O,Jones J D G.A plant miRNA contributes to antibacterial resistance by repressing auxin signaling[J].Science,2006,312(5772): 436-439.doi:10.1126/science.1126088.
[26] Pinweha N,Asvarak T,Viboonjun U,Narangajavana J.Involvement of miR160/miR393 and their targets in cassava responses to anthracnose disease[J].Journal of Plant Physiology,2015,174: 26-35.doi:10.1016/j.jplph.2014.09.006.
[27] 李沅秋,罗朝兵,廖鸿,罗芮,杨瑶君.慈竹ARF基因的鉴定及虫害胁迫下的表达[J].东北林业大学学报,2019,47(2):56-63.doi:10.3969/j.issn.1000-5382.2019.02.013.
Li Y Q,Luo C B,Liao H,Luo R,Yang Y J.Identification and gene expression under herbivore stress of auxin response factors in Bambusa emeiensis[J].Journal of Northeast Forestry University,2019,47(2):56-63.
[28] Simonini S,Stephenson P,Φstergaard L.A molecular framework controlling style morphology in Brassicaceae[J].Development, 2018,145(5):dev158105.doi:10.1242/dev.158105.
[29] Mukherjee A,Mazumder M,Jana J,Srivastava A K,Mondal B,De A,Ghosh S,Saha U,Bose R,Chatterjee S,Dey N,Basu D.Enhancement of ABA sensitivity through conditional expression of the ARF10 gene in Brassica juncea reveals fertile plants with tolerance against Alternaria brassicicola[J].Molecular Plant Microbe Interactions,2019,32(10):1429-1447.doi:10.1094/MPMI-05-19-0132-R.