延边牛属寒温带山区的役肉兼用品种,适应性较强。在延边等地温度极低的冬季,延边牛同样可正常生产、生存,且体现出遗传稳定性与较高生产性能。在-26 ℃时才出现明显的不安[1],但依然能保持正常食欲和反刍,因此,延边牛既是我国东北部宝贵的抗寒品种又是研究大型哺乳动物抗寒不可多得的优秀模型。
恒温动物面临的一个重要挑战是在冷应激期间对产热的需求增加。当保温机制(例如蜷缩行为、血管收缩、立毛)不足以抵抗外界温度过低时,则机体需要通过震颤和非震颤产热机制来维持恒定的体温。然而持续震颤产热会影响到动物的运动、睡眠反而会出现适得其反的效果,出生于寒冷环境的动物肌肉没有充分发育,无法有效地通过震颤产热[2],所以处于寒冷环境中的哺乳动物震颤产热逐渐被非震颤产热所替代。众所周知,棕色脂肪是非震颤产热的主要部位,通过其产热可以维持机体核心体温、促进在冬眠中动物的苏醒。棕色脂肪产热受中枢神经所控制,其机理是中枢神经系统释放去肾上腺素以激活β-肾上腺受体和细胞内的网络信号,从而增加线粒体底物的供应和呼吸能力,使机体内的甘油三酯分解为游离的脂肪酸并作为线粒体氧化的燃料,同时诱导解偶联蛋白-1的活性。在棕色脂肪线粒体中,丰富的UCP1活性加速了高氧化能力,这是发热过程的核心[3]。解偶联蛋白是位于线粒体内膜上的蛋白质家族的成员,这些蛋白质与能量消耗、生热、游离脂肪酸的调节和活性氧的减少有关[4]。UCP属于一类线粒体内膜(IMM)蛋白,在其作用下,IMM两侧可无跨膜质子浓度差,下调由质子浓度差介导的氧化磷酸化(OXPHOS)速度,对三磷酸腺苷(ATP)的生成造成干扰,UCP释放效能的本质在于解除局部正常呼吸链内应有的电子传递和磷酸化双方的偶联性,导致OXPHOS过程呈空转状态[5]。根据其功能分为:UCP1、UCP2、UCP3、UCP4、UCP5[6]。解偶联蛋白-1(Uncoupling protein-1,UCP1以前称为Thermogenin)是线粒体内膜的转运离子载体,由于UCP1在非颤动产热(NST)中的作用而被称之为热生成素。大多数研究认为,UCP1基因主要在脂肪组织中表达,细胞内线粒体通过UCP1蛋白的激活从而使能量代谢增加,该蛋白将质子原动力作为热量散发。作为一种解偶联蛋白,UCP1在长链脂肪酸(FA)的存在下通过线粒体内膜转运H+,这使棕色脂肪线粒体产生热量[7]。延边牛在抗寒适应性上有着较好表现,现今还未明确UCP1参与延边牛体温调节与否。
20世纪70年代David nichollis和Daninel Ricquier 首先发现了UCP1基因[8],且成功克隆并定位于4号染色体上,基因大小为13 kb,有6个外显子,并发现其在人体内主要表达于棕色脂肪组织内。但现今已有学者证实,部分其他组织内同样表达UCP1,诸如胰腺、白色脂肪组织、胸腺、纵向平滑肌与骨骼肌[9];目前,经过测序发现各物种的基因长度、外显子数目、编码区长度、表达区域都有一定的差异,小鼠的UCP1基因位于8号染色体中且长度为8 109 bp、外显子数目有6个、CDS区长度为924 bp。山羊的UCP1位于17号染色体中且基因长度为6 899 bp、外显子数目有6个、CDS区长度为918 bp。在进一步探究UCP1基因下,其基因序列不断在其他哺乳动物内被检出。研究家畜领域,通过分析牛UCP1基因的多态性发现,(C+G)%的含量水平正向相关于动物适应环境温度,可见UCP1和牛抗寒性相关[10]。因此,本研究以延边牛皮下脂肪组织为研究对象,克隆测序得到其UCP1的CDS区序列,同时针对此序列特征开展生物信息学研究,了解UCP1基因在延边牛不同组织中的表达变化,为延边牛UCP1基因功能分析提供了新的数据,可有效指导延边牛UCP1功能的深入研究。
1 材料和方法
1.1 样品采集
于2020年11月,在延边德海牧场选取3头为20月龄的雌性延边牛,借助无菌剪刀逐一提取其心、腿部肌肉、肝、皮下组织、脾、肾与肺作为组织样品放入冷冻管中,至冷冻管内,同时马上移至液氮内,放于实验室,存储于冰箱(-80 ℃)内。
1.2 主要试剂
核酸裂解液(Life)、GoldView I 型核酸染剂(Solarbio)、pMD18-T(TaKaRa)、FastKing一步法除基因组cDNA第一链合成预混试剂(TIANGEN)、荧光定量试剂盒(TIANGEN)、琼脂糖凝胶DNA回收试剂盒、T4 DNA Ligase、普通质粒提取试剂盒、氨苄青霉素、琼脂糖、引物(均购自与上海生工)。
1.3 仪器与设备
干式恒温器(GL-1800)购自海门其林贝尔仪器制造有限公司,小型台式冷冻型离心机(5417R)购自Eppendorf公司,超微量分光光度计(NanoDrop 2000)购自Thermo公司,电泳仪(Model 300 V)购自LABNET公司,qRT-PCR仪(Mx3005P)购自Agilent公司,梯度PCR仪(TC-5000)购自TECHNE公司,多功能分子成像系统(型号Azure 600)购自Azure Biosystems公司。
1.4 引物设计合成
使用Primer Premier 5.0软件,根据GenBank中已公布的牛UCP1基因(登录号:KR711477.1)序列设计普通PCR(扩增UCP1基因的CDS区)及实时荧光定量PCR引物,将管家基因GAPDH(登录号NM_001034034.2)当作内参。表1所示为引物详情,上海生工负责合成引物。
表1 引物序列
Tab.1 Primer sequences
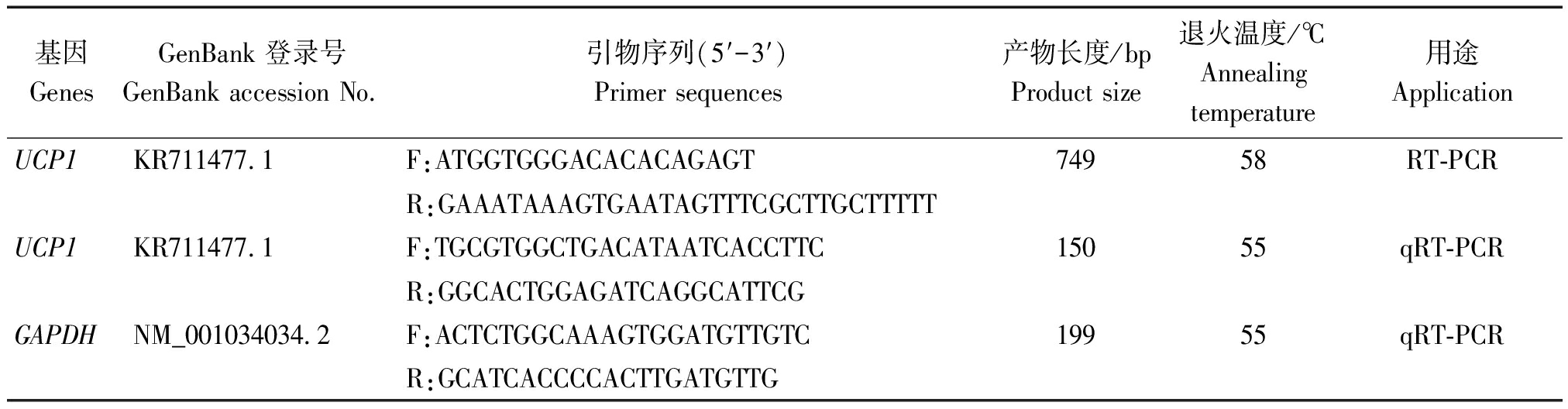
基因GenesGenBank 登录号GenBank accession No.引物序列(5′-3′)Primer sequences产物长度/bpProduct size退火温度/℃Annealing temperature用途ApplicationUCP1KR711477.1F:ATGGTGGGACACACAGAGT74958RT-PCRR:GAAATAAAGTGAATAGTTTCGCTTGCTTTTTUCP1 KR711477.1F:TGCGTGGCTGACATAATCACCTTC15055qRT-PCRR:GGCACTGGAGATCAGGCATTCGGAPDHNM_001034034.2F:ACTCTGGCAAAGTGGATGTTGTC19955qRT-PCRR:GCATCACCCCACTTGATGTTG
1.5 延边牛组织总RNA的提取及cDNA的合成
借助TRIzol Reagent试剂盒,同时遵循此试剂盒说明,完成小块脂肪组织标本总RNA的提取,由超微量分光光度计辅助,对此RNA质量(OD260/280)完成测定,对与要求相符的总RNA实施逆转录,得到cDNA,存放于-20 ℃环境中,待用。逆转录采用天根FastKing一步法试剂盒,反转录体系为50 μL:Total RNA 5 μL,5×FastKing-RT SuperMIX 10 μL,RNase-free water 35 μL。
1.6 延边牛UCP1基因CDS区克隆
将cDNA当作模板,通过上述引物(表1),并借助PCR法,对延边牛UCP1基因CDS区实施扩增。设定0.02 mL的PCR反应总体系:cDNA模板(1 μg/μL)、正向引物(10 μmol/L)、负向引物(10 μmol/L)、2×Taq PCR MasterMixⅡ、RNase-free water 依次为2.0,0.5,0.5,10.0,7.0 μL。以下为扩增步骤:94 ℃预变性3 min;94 ℃变性0.5 min,58 ℃退火0.5 min,72 ℃延伸2 min,循环35次;72 ℃ 5 min;4 ℃ 1 min。待反应结束,对0.01 mL反应产物,开展琼脂糖凝胶电泳检测。经由琼脂糖凝胶DNA回收试剂盒,完成与预期大小相符的PCR产物的回收。将回收所得目标DNA片段连接pMD18-T载体,制备0.01 mL的连接反应体系:目标DNA片段4 μL(0.1 μmol/L),pMD18-T载体1 μL(0.01 μmol/L),10×T4 DNA Ligase Buffer 4 μL,以及1 μL 的T4 DNA连接酶于4 ℃下进行一整夜连接,转化为感受态细胞,通过含有氨苄抗性的LB培养基实施涂布,经由单菌落开展扩繁,再提取质粒,同时PCR鉴定,生工生物(上海)负责对鉴定出的阳性质粒进行测序。
1.7 延边牛UCP1基因生物信息学分析
采用Blast对印度牛(Bos indicus,登录号:XM_019977126.1)、普通牛(Bos taurus,登录号:NM_001166528.1)、绵羊(Ovis aries,登录号:NM_001280694.1)、古猿(Odocoileus virginianus texanus,登录号:XM_020891215.1)、美洲草原野牛(Bison bison,登录号:XM_010830300.1)、南美羊驼(Vicugna pacos,登录号:XM_031692232.1)、白犀(Ceratotherium simum,登录号:XM_004426473.1)、东北虎(Panthera tigris altaica,登录号:XM_007091783.2)进行同源性对比分析,针对延边牛UCP1对应序列,借助表2所示在线软件实施生物信息学分析。
表2 生物信息学软件
Tab.2 Bioinformatics software

软件名称Software brand网址Uniform resource locator功能FunctionMEGA 7.0构建进化树ProtScalehttps://web.expasy.org/protscale/蛋白的理化性质NetOGlyc 4.0http://www.cbs.dtu.dk/services/NetOGlyc/糖基化位点NetPhos 3.1http://www.cbs.dtu.dk/services/NetPhos/蛋白磷酸化位点NetOGlyc 4.0http://www.cbs.dtu.dk/services/NetOGlyc/ O端糖基化位点Scratch Protein Predictorhttp://scratch.proteomics.ics.uci.edu/蛋白二硫键分析SignalP 4.1 Serverhttp://www.cbs.dtu.dk/services/ Signal P-4.1/蛋白信号肽TargetP 1.1 Server PSORThttps://www.genscript.com/psort.html蛋白亚细胞定位SOPMAhttps://npsa-prabi.ibcp.fr/cgi-bin/npsaautomat.pl?Page=npsasopma.html蛋白质二级结构PHYRE2 http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index蛋白质三级结构
1.8 延边牛各组织相对表达量分析
将GAPDH当作内参基因,借助qRT-PCR对延边牛每组织样品内UCP1基因相对表达水平展开测定,反应体系为20 μL:2×SuperReal Premixplus 10 μL,50×Rox Reference Dye 0.3 μL,正向、负向引物皆为0.6 μL(10 μmol/L),另有1 μL 的cDNA模板(1 μg/μL)以及7.5 μL 的RNase-free ddH2O。各待检样品皆安排3个重复,以下为反应步骤:95 ℃ 15 min;95 ℃ 10 s,55 ℃ 0.5 min,72 ℃ 32 s,循环35次;95 ℃ 15 s,60 ℃ 1 min,95 ℃ 15 s。借助2-ΔΔCT法,对所得数据开展分析,将脾当作对比组织。利用Graphpad软件对UCP1基因在延边牛不同组织中的表达水平进行单因素方差分析(ANOVA),差异具显著水平的标准为P<0.05。
2 结果与分析
2.1 PCR扩增结果
借助超微量分光光度计,对RNA质量(OD260/280之比)展开测定,发现此项指标处于1.8~2.0,RNA结构具良好完整性,未见DNA与蛋白污染,能够参与后续研究。对PCR扩增产物实施琼脂糖凝胶(1%)电泳分析,显示出1条约750 bp的DNA片段(图1),在长度上基本相符于预期DNA片段(长度为749 bp)。
2.2 UCP1基因序列的生物信息学分析
2.2.1 序列分析结果 通过NCBI中Blast对延边牛UCP1基因CDS区核苷酸序列与印度牛、普通牛、绵羊、古猿、美洲草原野牛、南美羊驼、白犀、东北虎的基因核苷酸序列进行同源性比对发现,延边牛UCP1基因与印度牛的同源性为99.47%、普通牛97.40%、绵羊96.72%、古猿95.56%、美洲草原野牛97.40%、南美羊驼90.11%、白犀90.12%、东北虎88.29%。基于各物种的UCP1基因编码区核苷酸(Nt)序列,借助软件MEGA 7.0完成系统发育树的制作,结果显示,在亲缘关系上,延边牛和印度牛亲缘关系最近,和南美羊驼最远(图2)。
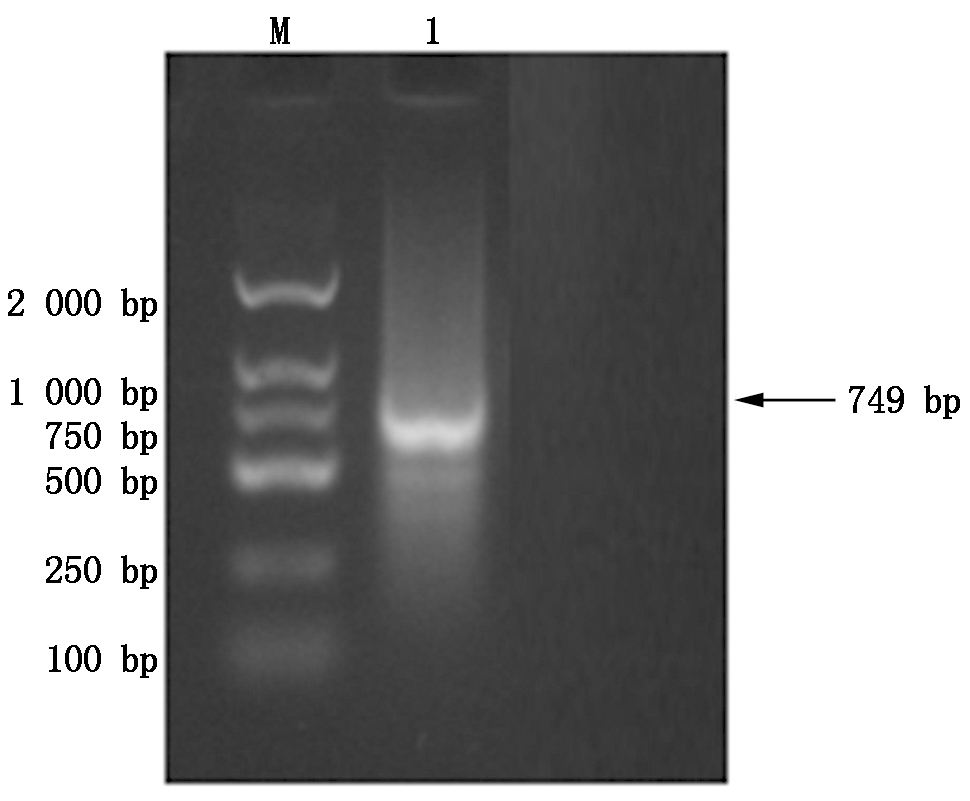
M.DL2000 DNA Marker;1.延边牛UCP1 CDS区序列。
M.DL2000 DNA Marker;1.UCP1 CDS sequence of Yanbian cattle.
图1 延边牛UCP1基因CDS区扩增结果
Fig.1 Amplification results of CDS region of
UCP1 gene in Yanbian cattle
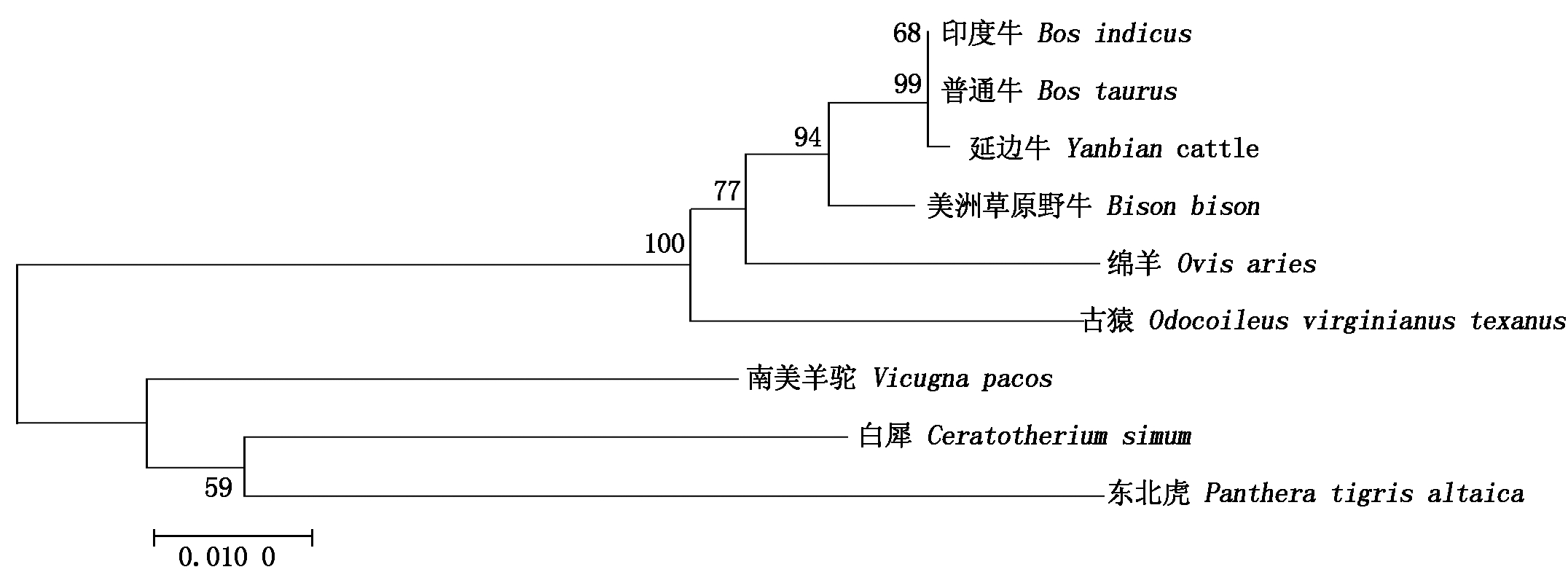
图2 UCP1基因系统进化树
Fig.2 Phylogenetic tree of UCP1 gene
2.2.2 UCP1蛋白质的理化性质 借助ProtParam在线工具分析延边牛UCP1的理化属性,发现此蛋白质的分子质量为27.065 61 ku,理论等电点是8.92,蛋白分子式是C1206H1938N320O350S17,总原子数为3 831。UCP1基因共编码249个氨基酸,氨基酸组成如表3所示,其中Val(V)最多,且不含Pyl(O),氨基酸构成为携负电残基(Asp+Glu)、正电残基(Arg+Lys)的个数各为19,25个,不稳定指数与消光系数依次是48.24,13 450,可见,此蛋白缺乏稳定性,脂溶系数是91.53,网状红细胞(RBC)于体外的半衰期是30 h,总平均亲水性(GRAVY)等于0.212,可见此蛋白具备一定亲水性。
表3 UCP1蛋白的氨基酸组成
Tab.3 Composition of amino acid of UCP1
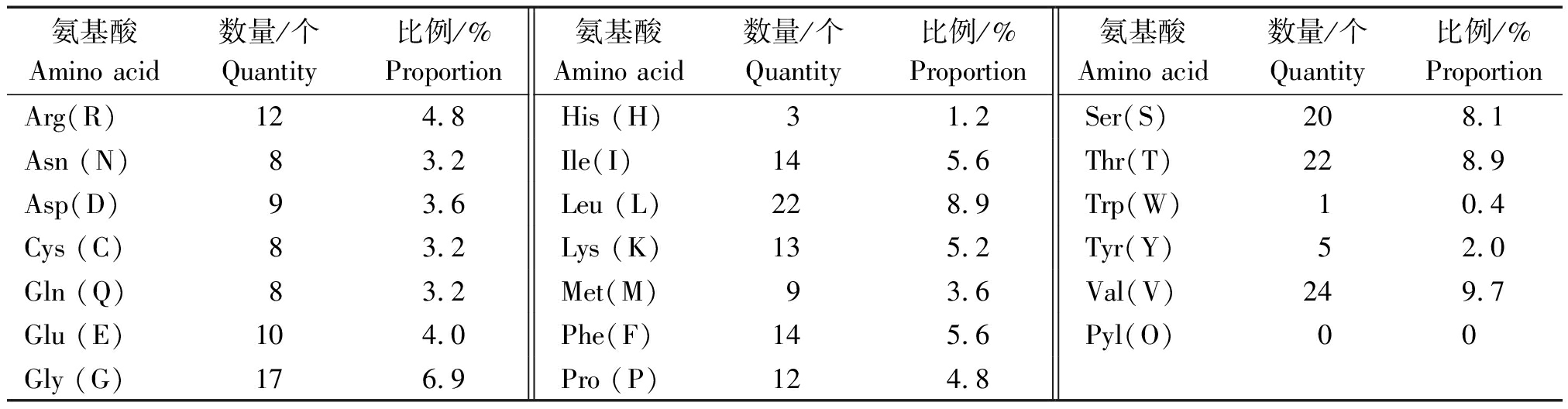
氨基酸Amino acid数量/个Quantity比例/%Proportion氨基酸Amino acid数量/个Quantity比例/%Proportion氨基酸Amino acid数量/个Quantity比例/%ProportionArg(R)124.8His (H)31.2Ser(S)208.1Asn (N)83.2Ile(I)145.6Thr(T)228.9Asp(D)93.6Leu (L)228.9Trp(W)10.4Cys (C)83.2Lys (K)135.2Tyr(Y)52.0Gln (Q)83.2Met(M)93.6Val(V)249.7Glu (E)104.0Phe(F)145.6Pyl(O)00Gly (G)176.9Pro (P)124.8
借助在线软件ProtScale,对延边牛UCP1的疏水性展开预测,获得图3所示结果,在UCP1所含氨基酸中,占比较高的为亲水性残基,总体显示具亲水性,此结果相符于软件Protparam预测结果。
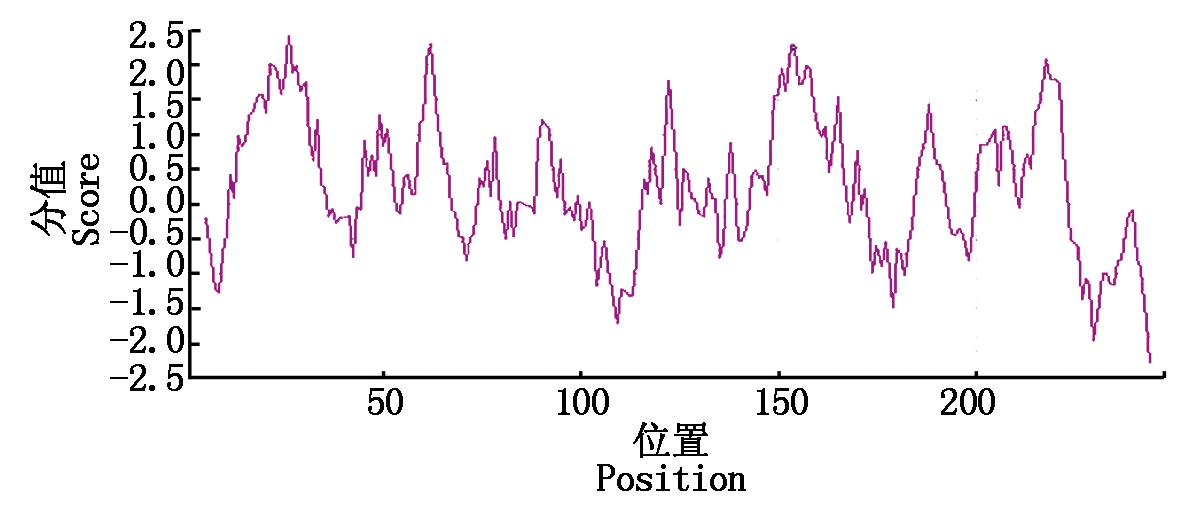
图3 UCP1的疏水性分析
Fig.3 Hydrophobicity analysis of UCP1
2.2.3 延边牛UCP1蛋白的磷酸化位点与糖基化位点 在蛋白质的功能与活性中,蛋白质翻译后修饰发挥着十分关键的调节作用,其中最为广泛的翻译后修饰为蛋白质的糖基化与磷酸化。借助Net Phos 3.1分析UCP1潜在的磷酸化位点,发现UCP1所含磷酸位点总计21个,其中丝氨酸(S)、苏氨酸(T)与酪氨酸(Y)依次占11,9,1个(图4)。分别借助NetNGlyc 1.0与NetOGlyc 4.0,对UCP1潜在的N-糖基化、O-糖基化位点展开分析,发现O-糖基化位点有4个,所处位置为第5,7,104,241位氨基酸;N-糖基化潜在位点有3个,所处位置为第113,121位氨基酸。
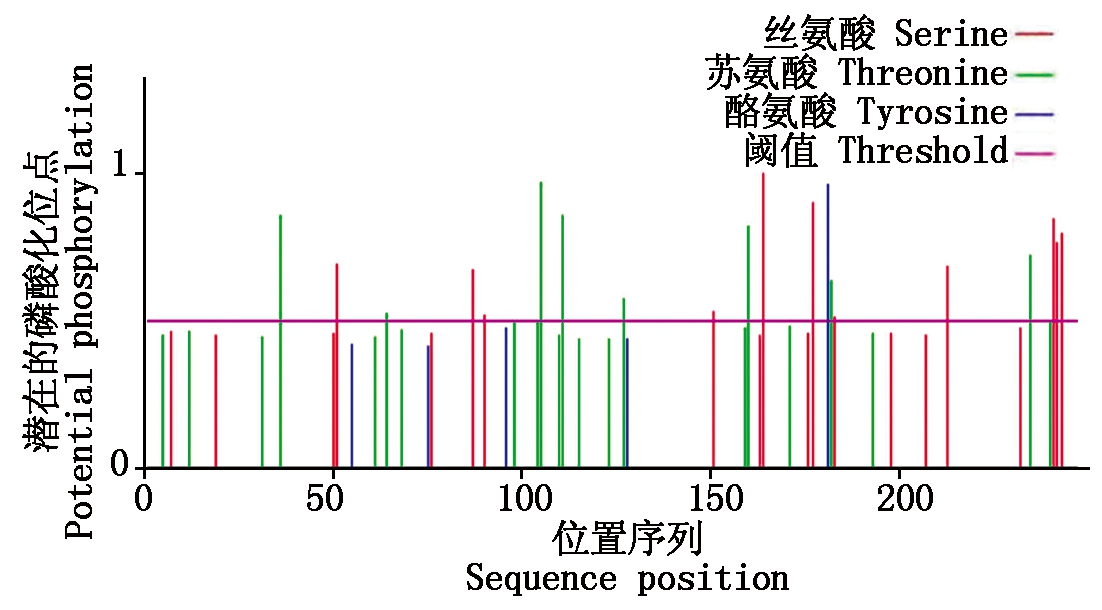
图4 UCP1蛋白潜在的磷酸化位点分析
Fig.4 Potential phosphorylation sites of UCP1 protein
2.2.4 延边牛UCP1蛋白跨膜区预测及信号肽、亚细胞定位预测 借助在线软件TMHMM 2.0Server,对跨膜结构域展开分析,发现此基因的编码产物无跨膜螺旋(TMHs)结构,跨膜螺旋氨基酸残基数量的预测值19.008 57,蛋白质前60个氨基酸的跨膜螺旋数预测值为10.980 09,位于膜细胞质侧的总概率22.995%(图5)。
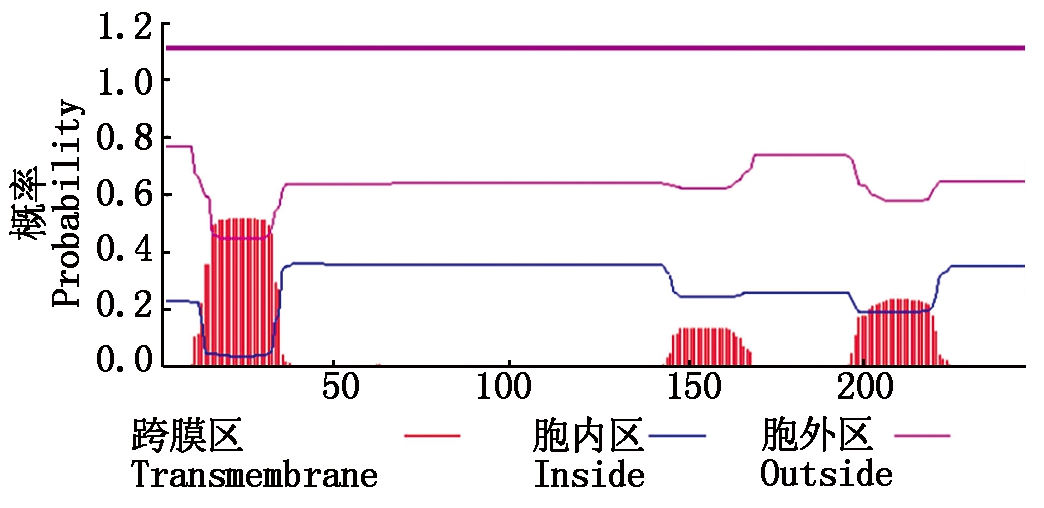
图5 延边牛UCP1蛋白跨膜结构预测
Fig.5 Transmembrane domain prediction of
Yanbian cattle UCP1 protein
利用SignalP 4.1 Server在线软件进行分析,区分信号肽和非信号肽临界值为D=0.450,UCP1预测D=0.158。参考信号肽假说,判断UCP1编码蛋白无信号肽分布,是一类非分泌蛋白(图6)。
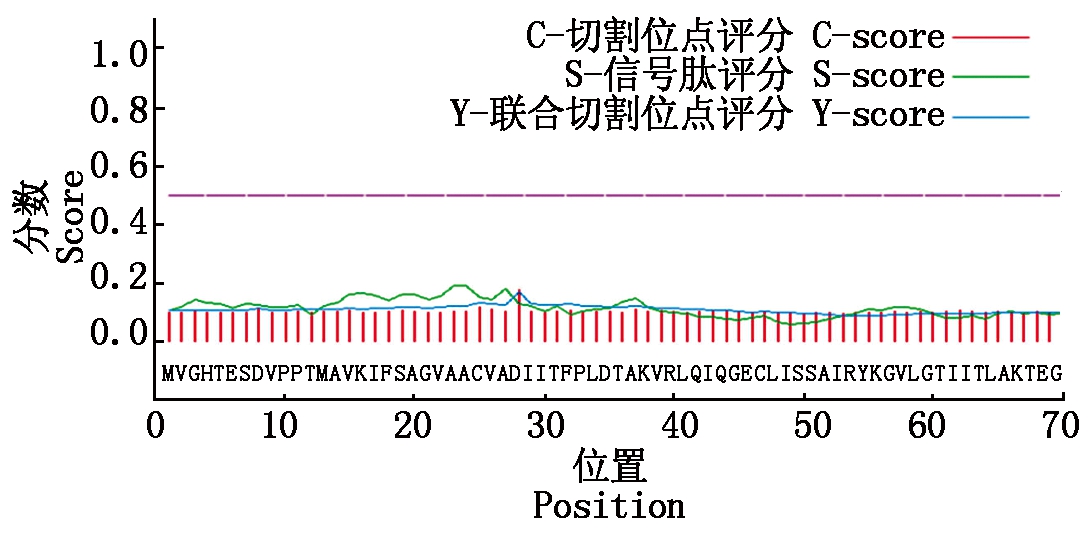
图6 延边牛UCP1蛋白信号肽预测
Fig.6 The prediction of signal peptide of
Yanbian cattle UCP1 protein
借助TargetP 1.1 Server与PSORT,对UCP1的亚细胞定位进行分析的结果显示,线粒体靶向肽(mTP)占比为0.029%,分泌通路信号位点(SP)占比为0.171%,同时不存在信号肽,故判断其合成后未出现转运;UCP1的分布以细胞核为主(表4)。
表4 UCP1蛋白亚细胞定位
Tab.4 Subcellular localization of UCP1 protein
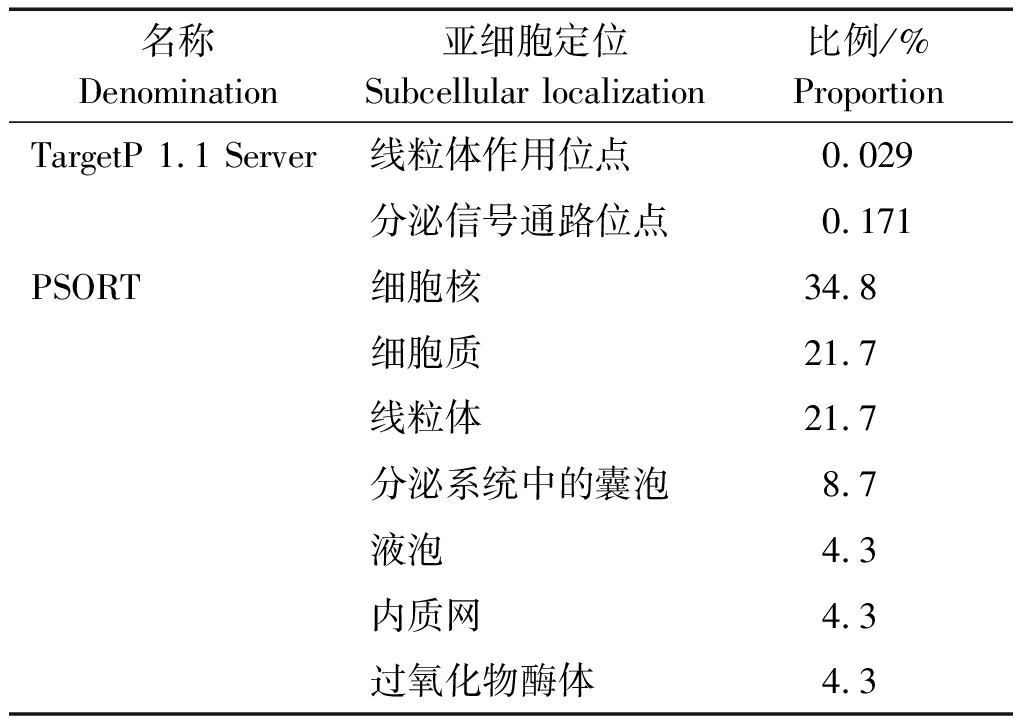
名称Denomination亚细胞定位Subcellular localization比例/%ProportionTargetP 1.1 Server线粒体作用位点0.029分泌信号通路位点0.171PSORT细胞核34.8细胞质21.7线粒体21.7分泌系统中的囊泡8.7液泡4.3内质网4.3过氧化物酶体4.3
2.2.5 UCP1蛋白高级结构预测 借助SOPMA软件,对UCP1的二级结构展开预测,该序列主要是由α-螺旋、延伸链、无规卷曲和β-转角组成的混合型蛋白(图7)。α-螺旋的氨基酸含量为134个,占比为54.03%;无规卷曲的氨基酸含量为68个,占比为27.42%;延伸链的氨基酸含量为32个,占比为12.90%;β-转角的氨基酸含量为14个,占比为5.65%。利用ExPASy中 Swiss-model平台构建延边牛UCP1蛋白可能的三级结构模型(图8),全局模型质量估计值为0.53,与模板相似性为65.45%。
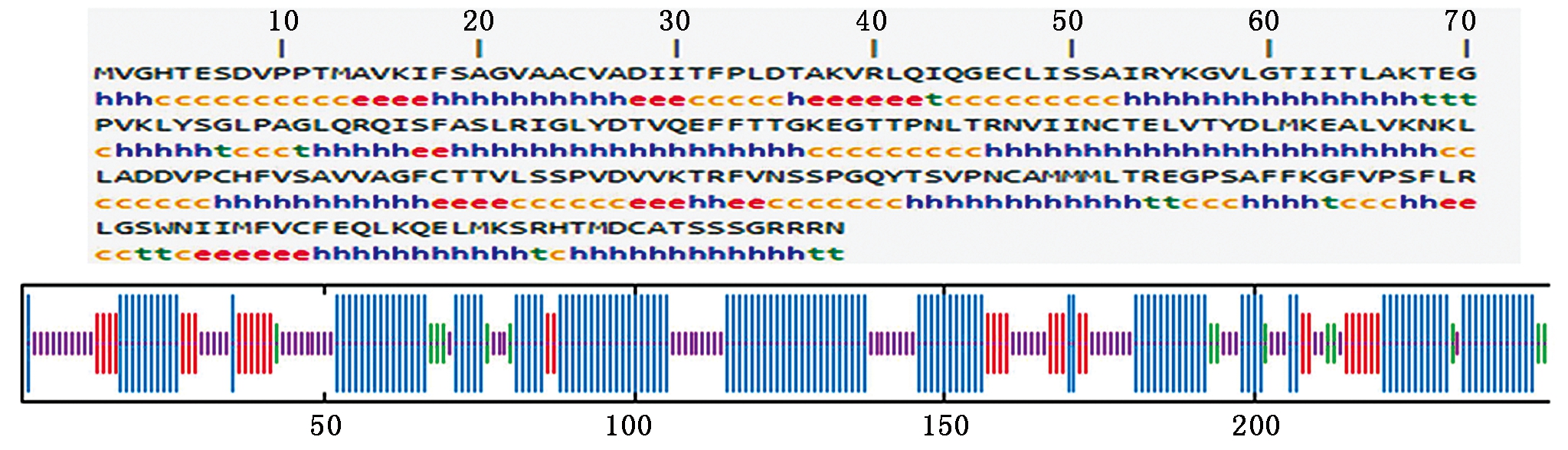
h.α-螺旋;e.延伸链;c.无规则卷曲;t.β-转角。
h.Alpha helix;e.Extended strand;c.Random coil;t.Beta turn.
图7 预测UCP1蛋白的二级结构
Fig.7 Secondary structure prediction of UCP1 protein
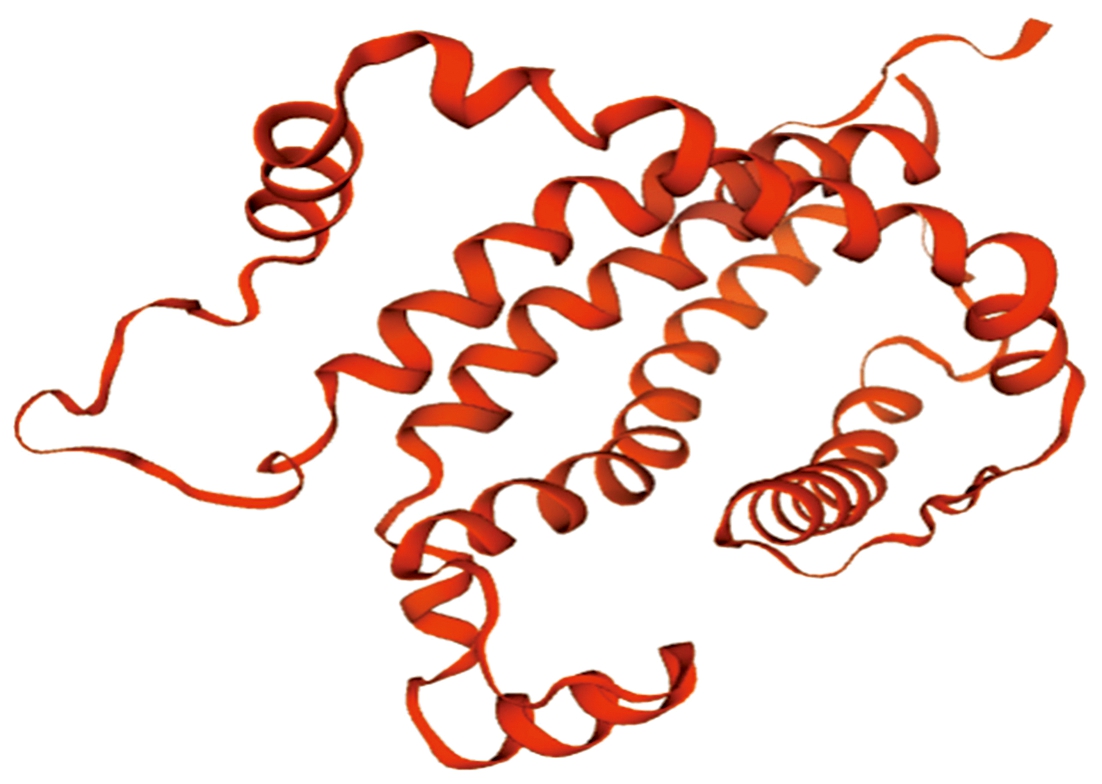
图8 UCP1的三级结构模型
Fig.8 3D structure model of UCP1
2.3 UCP1基因在延边牛不同组织间的差异表达
如图9所示,对8个组织部位进行UCP1基因表达水平的检测。结果发现,UCP1基因在延边牛的小肠和脂肪组织中表达量最高。在脾、肾脏中也有较高的相对表达量且差异显著(P<0.05),腿部肌肉和心脏表达量相对较低,且无显著差异(P>0.05)。
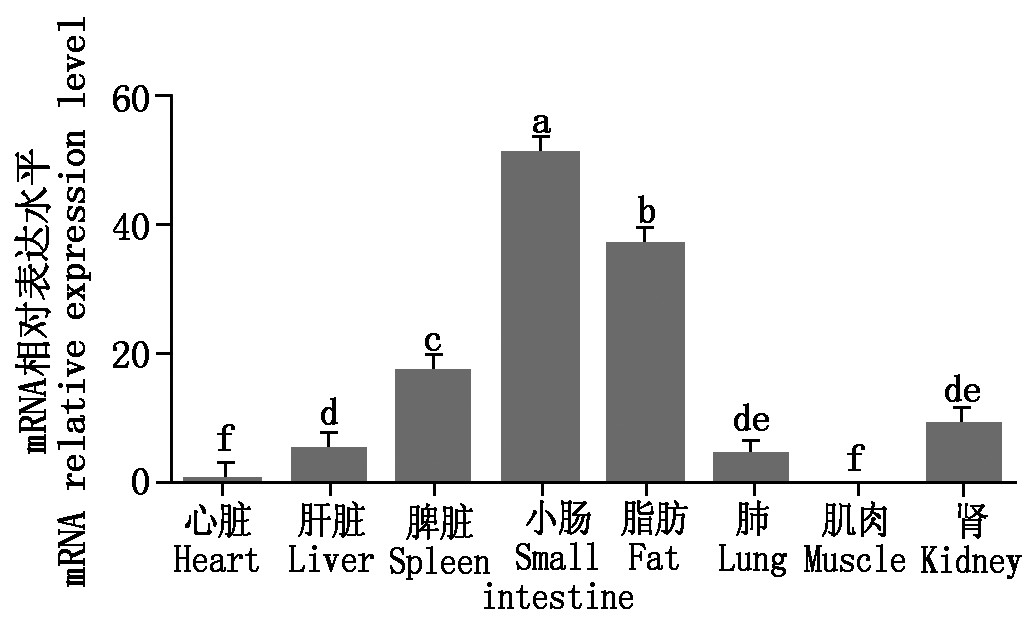
图9 UCP1基因在延边牛各组织中
的mRNA相对表达量
Fig.9 UCP1 mRNA relative expression of
different tissues in Yanbian cattle
3 讨论与结论
解偶联蛋白1在棕色脂肪和白色脂肪中[11],它是属于线粒体阴离子转运蛋白家族的解偶联蛋白亚家族中研究最多的载体[12]。UCP1是20世纪70年代中期在仓鼠、大鼠和豚鼠棕色脂肪的线粒体中发现的[13]。UCP1被多项研究视为唯一的“真正的”解偶联蛋白[14-15],因为如果在寒冷的环境下在哺乳动物中上调其质子梯度,则会耗散线粒体内膜上的质子梯度,从而产生大量热量。经研究发现,UCP1活性在褐色脂肪细胞中得到很好的调节,在寒冷环境暴露或过量喂养时,UCP1活性被诱导,从而促进了适应性生热作用[16]。Riley等[17]证明了去甲肾上腺素诱导的产热依赖于UCP1的存在,当下丘脑中传递冷感时,儿茶酚胺会从交感神经系统释放出来,从而激活褐色脂肪细胞的肾上腺素受体,进而刺激依赖cAMP的蛋白激酶PKA,从而导致三酰甘油脂肪酶的活化,从而增加脂解作用。经由脂解效应释放的游离脂肪酸(FFA)激活UCP1。激活后,UCP1使ATP合成的电化学质子梯度短路,从而刺激电子传输并增强呼吸作用。可利用的底物燃烧产生热量并通过血液循环散发到身体的其他部位[18-19]。
本研究以延边牛皮下脂肪组织为材料成功克隆出UCP1基因,并对其生物信息学进行分析。结果显示,延边牛UCP1蛋白理化性质不稳定,且亲水性残基多于疏水性残基,所以该蛋白是亲水性蛋白。参考信号肽假说,判断UCP1编码蛋白无信号肽分布,是一类非分泌蛋白。UCP1蛋白二级结构主要由α螺旋、无规卷曲构成,此相符于其转录模式。蛋白质翻译后修饰(PTM)过程很复杂,其中羟基化、磷酸化、泛素化、甲基化与乙酰化等较为常见[20]。蛋白质PTM是指,在mRNA翻译后形成蛋白质环节的一类化学修饰。具体可理解为,在蛋白质合成环节,受到修饰酶的作用,部分生化功能性基团(碳水化合物、磷酸基、脂类、乙酰基与糖类等)和蛋白质骨架(或侧链)共价结合。蛋白质PTM可使蛋白质的生理活性、成熟度、物理化学性质发生变化,导致蛋白质类型增多,功能更完善、结构更复杂、作用更专一、调控更精准,对于细胞活动发挥非常关键的作用。经预测延边牛UCP1蛋白PTM,发现潜在磷酸化位点有 21个,O端糖基化位点有4个,这些修饰能够经由将新基团连接上特定氨基酸侧链途径,使蛋白质的功能与结构发生变化。
大部分研究显示,UCP1在棕色脂肪(BAT)的线粒体内特异表达,为成熟BAT特异性的产热基因[21]。本试验通过Real-time PCR定量分析延边牛不同组织中UCP1基因相对表达量,结果显示,UCP1基因表达却在小肠较多。孙国权等[22]研究发现,UCP1基因在西门塔尔牛各组织内皆存在表达,但就表达水平而言,背膘相较其他组织皆偏高,同时发现,UCP1与产热同维持体温相关。赵丹等[23]的研究结果显示,UCP1基因于牦牛肾内表达水平相较其他组织显著偏高。说明UCP1基因不仅在不同物种中表达规律有所差异,而且在不同品种间表达规律也有差异。这可能由于UCP1基因的表达受能量、动物年龄、暴露温度、胰岛素等因素的影响[24-26]。因现今仅有少量研究涉及延边牛UCP1基因分析,未找到其确切的功能与调控机制,需开展更为深入的探究。
此项研究以延边牛脂肪组织为材料,进行UCP1完整CDS区的克隆,其呈749 bp大小,编码氨基酸量为249个,为亲水性蛋白,所含磷酸化、O-糖基化、N-糖基化潜在位点的量依次为21,4,3个,无信号肽。预测UCP1蛋白高级结构,发现其属于混合型蛋白,构成以α-螺旋、β-转角、延伸链与无规卷曲为主。RT-PCR结果表明,延边牛小肠组织内的UCP1基因表达水平最高,其次为脂肪和脾组织。此研究发现可为延边牛UCP1蛋白性质与更为深入分析此基因功能给予指导。
[1] 朴政玉,罗晓彤,金鑫,闫研,严昌国.延边黄牛肾周棕色脂肪的鉴定与UCP1的表达[J].东北农业科学,2019,44(5):58-60.doi:10.16423/j.cnki.1003-8701.2019.05.013.
Piao Z Y,Luo X T,Jin X,Yan Y,Yan C G.Identification of perirenal brown adipose tissue and seasonal variation of UCP1 expressions in Yanbian yellow cattle[J].Northeast Agricultural Sciences,2019,44(5):58-60.
[2] Oelkrug R, Polymeropoulos E T,Jastroch M.Brown adipose tissue:physiological function and evolutionary significance[J].Comp Physiol B,2015,185(6):587-606.doi:10.1007/s00360-015-0907-7.
[3] Brondani L A, Assmann T S, de Souza B M, Bouças A P, Canani L H, Crispim D. Meta-analysis reveals the association of common variants in the uncoupling protein (UCP) 1-3 genes with body mass index variability[J].PLoS One, 2014, 9(5):e96411. doi:10.1371/journal.pone.0096411.
[4] Rousset S, Alves-Guerra M C, Mozo J, Miroux B, Cassard-Doulcier A M, Bouillaud F, Ricquier D. The biology of mitochondrial uncoupling proteins[J].Diabetes, 2004, 53(S1):S130-S135.doi:10.2337/diabetes.53.2007.s130.
[5] 李海莲.UCP3基因的研究进展[J].上海畜牧兽医通讯,2011(3):15-16.doi:10.3969/j.issn.1000-7725.2011.03.006.
Li H L.Research progress of UCP3 gene[J].Shanghai Journal of Animal Husbandry and Veterinary Medicine,2011(3) :15-16.
[6] Bertholet A M, Kirichok Y. The mechanism FA-dependent H+ transport by UCP1[J].Handbook of Experimental Pharmacology, 2019, 251:143-159. doi:10.1007/164_2018_138.
[7] Cassard A M, Bouillaud F, Mattei M G, Hentz E, Raimbault S, Thomas M, Ricquier D. Human uncoupling protein gene:Structure, comparison with rat gene, and assignment to the long arm of chromosome 4[J].Journal of Cellular Biochemistry, 1990, 43(3):255-264. doi:10.1002/jcb.240430306.
[8] Gura T. Uncoupling proteins provide new clue to obesity′s causes[J].Science, 1998, 280(5368):1396-1370. doi:10.1126/science.280.5368.1369.
[9] Lee Y, Willers C, Kunji E R S, Crichton P G. Uncoupling protein 1 binds one nucleotide per monomer and is stabilized by tightly bound cardiolipin[J].Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(22):6973-6978.10.1073/pnas.1503833112.
[10] 罗刚,王杰,赖添赋,甘明川,赖松家.UCP1基因多态性与牛耐寒性的关联分析[J].四川农业大学学报,2018,36(6):808-814.doi:10.16036/j.issn.1000-2650.2018.06.015.
Luo G, Wang J, Lai T F, Gan M C,Lai S J. Association analysis of UCP1 gene polymorphism with cole resistance of cattles[J].Journal of Sichuan Agricultural University, 2018, 36(6):808-814.
[11] Stuart J A, Harper J A, Brindle K M, Jekabsons M B, Brand M D. A mitochondrial uncoupling artifact can be caused by expression of uncoupling protein 1 in yeast[J].Biochemical Journal, 2001, 356(3):779-789. doi:10.1042/0264-6021:3560779.
[12] Hodges M R, Tattersall G J, Harris M B, McEvoy S D, Richerson D N, Deneris E S, Johnson R L, Chen Z F, Richerson G B. Defects in breathing and thermoregulation in mice with near-complete absence of central serotonin neurons[J].The Journal of Neuroscience, 2008, 28(10):2495-2505. doi:10.1523/JNEUROSCI.4729-07.2008.
[13] Rodriguez-Cuenca S, Cochem H M, Logan A, Abakumova I, Prime T A, Rose C, Vidal-Puig A, Smith A C, Rubinsztein D C, Fearnley I M, Jones B A, Pope S, Heales S J R, Lam B Y H, Neogi S G, McFarlane I, James A M, Smith R A J, Murphy M P. Consequences of long-term oral administration of the mitochondria-targeted antioxidant MitoQ to wild-type mice[J].Free Radical Biology and Medicine, 2010, 48(1):161-172. doi:10.1016/j.freeradbiomed.2009.10.039.
[14] Nicholls D G.The physiological regulation of uncoupling proteins[J].Biochimica et Biophysica Acta,2006,1757(5-6):459-466.doi:10.1016/j.bbabio.2006.02.005.
[15] Enerbäck S, Jacobsson A, Simpson E M, Guerra C, Yamashita H, Harper M E, Kozak L P. Mice lacking mitochondrial uncoupling protein are cold-sensitive but not obese[J].Nature, 1997, 387(6628):90-94. doi:10.1038/387090a0.
[16] Finkel T. From sulfenylation to sulfhydration:What a thiolate needs to tolerate[J].Science Signaling, 2012, 5(215):pe10. doi:10.1126/scisignal.2002943.
[17] Riley C L, Boutz D, Bean C.UCP1 is essential for mitochondrial structural integrity and function in brown adipose tissue[J].Am J Physiol Endocrinol Metab,2003,285(6):1230-1236.doi:10.1152/ajpendo.00197.2003.
[18] Li Y G, Fromme T, Schweizer S, Schöttl T, Klingenspor M. Taking control over intracellular fatty acid levels is essential for the analysis of thermogenic function in cultured primary brown and brite/beige adipocytes[J].EMBO Reports, 2014, 15(10):1069-1076. doi:10.15252/embr.201438775.
[19] Stier A,Bize P,Habold C,Bouillaud F,Massemin S,Criscuolo F.Mitochondrial uncoupling prevents cold-induced oxidative stress:a case study using UCP1 knockout mice[J].J Exp Biol,2014,217(4):624-630.doi:10.1242/jeb.092700.
[20] 王欣悦, 张莉. 蛋白质组学及蛋白质翻译后修饰在畜牧领域中的应用研究进展[J].中国畜牧兽医, 2019, 46(4):1063-1073. doi:10.16431/j.cnki.1671-7236.2019.04.013.
Wang X Y, Zhang L. Research progress on application of proteomics and post-translational modification of proteins in animal husbandry[J].China Animal Husbandry & Veterinary Medicine, 2019, 46(4):1063-1073.
[21] Guo Y Y, Li B Y, Peng W Q, Guo L, Tang Q Q. Taurine-mediated browning of white adipose tissue is involved in its anti-obesity effect in mice[J].Journal of Biological Chemistry, 2019, 294(41):15014-15024. doi:10.1074/jbc.RA119.009936.
[22] 孙国权, 高树新, 吴慧光, 李俊雅, 丽春, 王景山, 王玉泉, 王国富. 解偶联蛋白1、2和3基因在中国西门塔尔牛组织器官中的表达水平及其与胴体品质关系分析[J].华北农学报, 2014, 29(4):116-120.doi:10.7668/hbnxb.2014.04.019.
Sun G Q, Gao S X, Wu H G, Li J Y, Li C, Wang J S, Wang Y Q, Wang G F. Analyze the ucp1, ucp2, ucp3 genes expression level in tissues/organs and the relationship with the carcass traits in Chinese Simmental cattle[J].Acta Agriculturae Boreali-Sinica, 2014, 29(4):116-120.
[23] 赵丹,熊燕,华永琳,岳永起,郭玉,熊显荣,字向东,殷实,李键.牦牛UCP1基因生物学特性及组织表达分析[J].西南民族大学学报(自然科学版),2021,47(2):139-148.doi:10.11920/xnmdzk.2021.02.005.
Zhao D, Xiong Y, Hua Y L,Yue Y Q,Guo Y,Xiong X R,Zi X D,Yin S,Li J.Biological characteristics and tissue expression analysis of UCP1 gene in yak(Bos grunniens)[J].Journal of Southwest University for Nationalities,2021,47(2):139-148.
[24] 付荣霞, 孙长颢, 杨树成. 不同膳食构成对解偶联蛋白-1、2、3基因表达的影响研究[J].食品科学, 2006, 27(12):710-714. doi:10.3321/j.issn:1002-6630.2006.12.185.
Fu R X, Sun C H, Yang S C. Study on the effects of different diets on the expression of gene of uncoup Ling proteins 1, 2 and 3[J].Food Science, 2006, 27(12):710-714.
[25] 刘畅. 线粒体解偶联蛋白在小鼠皮肤组织中的鉴定与功能研究[D].南京:南京大学, 2017.
Liu C. Identification and functional study of mitochondrial uncoupling protein in mice skin tissue[D].Nanjing:Nanjing University, 2017.
[26] 刘玉洁, 杨曦, 马慧娟. 棕色脂肪组织与糖代谢的关系[J].国际内分泌代谢杂志, 2016, 36(3):198-201. doi:10.3760/cma.j.issn.1673-4157.2016.03.14.
Liu Y J, Yang X, Ma H J. Relationship between brown adipose tissue and glucose metabolism[J].International Journal of Endocrinology and Metabolism, 2016, 36(3):198-201.