2010年春季以来,在我国华东地区,如上海、江苏、浙江等地陆续爆发一种导致鸭、鹅产蛋量急剧下降的新发疾病,鸭、鹅感染后一般表现出高热、食欲下降甚至废绝、运动障碍、产蛋减少甚至停止,死亡率可达5%~10%[1-5]。鸭、鹅死亡后剖检病理变化多集中于卵巢,表现为卵巢出血、萎缩、破裂,患病动物后期出现典型的神经症状,偏瘫、倒地震颤,最终衰竭死亡。该病爆发后从华东地区向中西部地区快速扩散,几乎席卷了大部分水禽养殖密集地区,给我国鸭、鹅养殖业造成了巨大损失[6-11]。通过实验室检测,目前已经证明引起该病的病原为坦布苏病毒(Tembusu virus,TMUV)[12]。
坦布苏病毒属于黄病毒科(Flaviviridae),黄病毒属(Flavivirus genus),是一种不分节段的单股正链RNA 病毒,含有单一的开放读码框,编码3种结构蛋白(C、prM和E)和7种非结构蛋白(NS1、NS2A、NS2B、NS3、NS4A、NS4B和NS5)[13]。非结构蛋白NS5大约100 ku,是这10种编码蛋白中分子量最大的,整个NS5蛋白一般认为可以划分成2个功能域,N端为甲基转移酶结构域,C端为RNA依赖的RNA聚合酶(RNA-dependent RNA polymerase,RdRp)结构域。黄病毒属病毒如乙型脑炎病毒、西尼罗河病毒、登革病毒、寨卡病毒等都编码有RNA依赖的RNA聚合酶功能基团,该功能基团与病毒编码的其他蛋白酶(如NS3蛋白的解旋酶)以及一些宿主因子共同构成病毒的复制酶系统,在宿主细胞内调控病毒基因组的复制功能。其中,RdRp蛋白在整个复制酶系统中处于核心地位[14]。研究证明,黄病毒属病毒RdRp功能基团起始合成病毒基因组RNA的分子机制主要有2种:一种是引物依赖的RNA合成(Primer-dependent RNA synthesis),另一种是引物非依赖的RNA合成,即从头合成(de-novo RNA synthesis)[15]。因此,明晰坦布苏病毒非结构蛋白NS5重要功能基团-RdRp蛋白的结构与功能,对于深入研究坦布苏病毒的复制机制和调控机理等具有十分重要的意义。本研究通过开展DTMUV RdRp分子重组真核质粒的构建和表达,同时对其蛋白理化性质、结构及功能进行生物信息学分析与预测,旨在阐述DTMUV的复制机制以及DTMUV蛋白与宿主蛋白的相互作用提供基础数据,也为研制治疗DTMUV的药物提供靶向位点信息和理论依据。
1 材料和方法
1.1 病毒、细胞和载体
坦布苏病毒分离株(JS804)、BHK-21细胞株、真核表达载体pCMV-N-Flag(购自碧云天公司)均由禽病与生物兽药研究室保存。E.coli DH5α感受态细胞购自宝生物工程(大连)有限公司。
1.2 主要试剂
体液病毒核酸提取试剂盒、质粒小量提取试剂盒、PCR产物纯化试剂盒均购自Axygen生物科技有限公司;Ex Taq DNA聚合酶、各类限制性内切酶、T4 DNA连接酶、pMD19-T载体均购自宝生物工程(大连)有限公司;蛋白质相对分子质量标准购自Thermo Fisher公司;细胞培养用DMEM细胞培养基、胎牛血清均购自Gibco公司;脂质体转染试剂lipofectamine 3000购自Invitrogen公司;辣根过氧化物酶标记的山羊抗小鼠IgG购于北京中杉金桥公司;异硫氰酸荧光素(FITC)标记的山羊抗小鼠IgG购于碧云天公司。
1.3 引物设计与合成
根据GenBank中登录的坦布苏病毒JS804株(GenBank No.JF895923)NS5基因序列,设计1对PCR 扩增引物,其中上游引物为RdRp-F:5′-CAAGAATTCCCTTACAAGACTTGGAACTA-3′,划线部分为插入的EcoR Ⅰ酶切位点。下游引物为RdRp-R:5′-CTCCTCGAGTTACAAGACACCTTCACTCC-3′,划线部分为插入的Xho Ⅰ酶切位点。设计好的引物送南京金斯瑞生物有限公司合成,预期PCR扩增片段大小约为1 800 bp。
1.4 TMUV RdRp基因的扩增
按照Axygen公司体液病毒核酸提取试剂盒说明书进行操作,首先提取鸭坦布苏病毒JS804株的总RNA,进一步根据反转录试剂盒说明书要求,生成cDNA。PCR以生成的cDNA为模板,公司合成的RdRp-F和RdRp-R为引物进行扩增。PCR扩增体系(25 μL)如下:其中双蒸水15 μL,10×Ex PCR Buffer 2.5 μL,25 mmol/L MgCl2 1.5 μL,2.5 mmol/L dNTP 2 μL,RdRp-F和RdRp-R(20 pmol/mL)各1 μL,cDNA模板1.5 μL,Ex Taq(5 U/μL)0.5 μL。PCR反应程序如下:95 ℃预变性5 min;95 ℃变性30 s,56 ℃退火30 s,72 ℃延伸2 min,33次循环;最后72 ℃再延伸10 min。PCR反应结束后,经1%琼脂糖凝胶电泳鉴定,如有疑似目的条带,按照Axygen公司琼脂糖DNA凝胶回收试剂盒操作说明,进行PCR产物的回收与纯化。回收的片段根据TaKaRa公司pMD19-T载体说明书进行连接,连接产物转化入DH5α感受态细胞中,涂布于LB固体培养基上,置于37 ℃恒温培养箱中,倒置过夜培养,第2天挑取单菌落于LB液体培养基中,摇床振荡培养5 h,用PCR引物对菌液进行PCR鉴定。取鉴定正确的菌液1 mL送至上海生工生物科技有限公司作测序分析,鉴定正确后,按照Axygen公司质粒小量提取试剂盒操作说明,提取重组克隆质粒pMD19-T-RdRp。
1.5 重组质粒pCMV-Flag-RdRp的构建
利用TaKaRa公司限制性内切酶EcoR Ⅰ、Xho Ⅰ对重组克隆质粒pMD19-T-RdRp和真核表达载体pCMV-N-Flag进行双酶切处理,1%琼脂糖凝胶电泳鉴定后切胶回收,将目的基因片段、pCMV-N-Flag真核表达载体片段按照适宜比例与T4 DNA连接酶16 ℃连接过夜。第2天将过夜的连接产物转化入DH5α感受态细胞中,并涂布于含有卡那霉素抗性的琼脂平板,倒置后置于37 ℃恒温培养箱中过夜培养,第2天先挑取单菌落进行菌液PCR鉴定,将PCR鉴定正确的目标菌液再次扩大培养并提取真核质粒,EcoR Ⅰ、Xho Ⅰ双酶切鉴定,琼脂糖凝胶电泳分析,酶切鉴定有目的条带的克隆产物送至上海生工生物科技有限公司测序,将测序结果正确的重组质粒命名为pCMV-Flag-RdRp。
1.6 BHK-21细胞的转染
根据Axygen公司质粒小量提取试剂盒要求提取真核表达质粒pCMV-Flag-RdRp,核酸定量分析仪测定其浓度。10%胎牛血清传代培养BHK-21细胞,待其处于对数生长期时,胰酶消化细胞,取一定数目的细胞接种于24孔细胞培养板中,加入含有10%胎牛血清的DMEM营养液继续培养,待细胞铺至细胞板约80%~90%时进行转染。按照Invitrogen公司lipofectamine 3000转染试剂操作要求,将真核表达质粒pCMV-Flag-RdRp与脂质体按照适宜比例混合,制备DNA-脂质体混合物。混合物置于室温孵育20 min,然后将DNA-脂质体混合物均匀加入BHK-21细胞中,继续培养5 h,移除含有混合物的营养液,加入含10%胎牛血清的DMEM营养液继续培养。
1.7 间接免疫荧光鉴定
真核质粒pCMV-Flag-RdRp转染BHK-21细胞24 h后,移除培养液,用PBS洗涤BHK-21细胞3次,加入300 μL 4%多聚甲醛固定BHK-21细胞8 min;移除多聚甲醛溶液,PBS洗涤细胞3次,每次5 min,继续加入0.5% Triton X-100作用10 min,对细胞进行透化处理,PBS洗涤细胞3次,每次5 min;每孔加入1% BSA,室温封闭30 min;PBS洗涤细胞3次,每次5 min,然后加入水禽疫病防控团队先期制备的坦布苏病毒阳性小鼠血清(1∶1 000),室温孵育1 h,PBS洗涤3次,每次5 min,最后加入FITC标记的山羊抗小鼠IgG(1∶400),于室温环境孵育30 min,加入PBS洗涤细胞3次,每次5 min,倒置荧光显微镜下观察,照相记录。
1.8 Western Blot鉴定
pCMV-N-Flag以及pCMV-Flag-RdRp分别转染BHK-21细胞,转染48 h后分别加入400 μL RIPA裂解液裂解细胞,离心后取蛋白上清,长时间放置可分装保存。于蛋白定量检测仪上分别测定2种蛋白浓度后,经Western Blot检测RdRp蛋白的表达情况。首先对2种蛋白样品进行SDS-PAGE电泳,通过电转仪转到PVDF膜上,加入1% BSA,于37 ℃封闭1 h。然后以本实验室制备保存的抗坦布苏病毒阳性小鼠血清(稀释比例1∶1 000)为一抗,辣根过氧化物酶标记的山羊抗小鼠IgG(稀释比例1∶10 000)为二抗进行反应,ECL显影观察结果并拍照记录。
1.9 生物信息学分析
针对非结构蛋白NS5重要功能基团RdRp,采用一系列生物信息学软件对其展开分析。首先采用在线分析软件Prot-Param Tool(http://web.expasy.org/protparam)分析RdRp蛋白氨基酸组成、等电点等基础理化性质。采用ProtScale(http://www.ncbi.nlm.nih.gov/StructureE-Cdd/wrpsb.cgi#userconsent#)分析RdRp蛋白的疏水性。采用TMpred(http://www.ch.embnet.org/software/TMPRED_form.html)针对RdRp蛋白的跨膜区展开预测。采用SignalIP 4.0 Server(http://www.cbs.dtu.dk/services/SignalIP-4.0)就RdRp蛋白的信号肽展开预测。磷酸化位点、N-糖基化位点和O-糖基化位点分别使用NetPhos 3.1、NetNGlyc 1.0、YinOYang 1.2在线软件进行预测;采用Prabi服务器的SOPMA程序(http://npsa-prabi.Ibcp.fr/)预测RdRp蛋白的二级结构;采用SWISS-MODEL软件(http://npsa-prabi.Ibcp.fr/cgi-bin/npsa_automat.pl)预测RdRp蛋白的三级结构。
2 结果与分析
2.1 鸭坦布苏病毒RdRp基因的PCR扩增
以鸭坦布苏病毒cDNA为模板,加以一对针对RdRp基因的特异性引物,通过PCR方法扩增RdRp基因全长,结果PCR扩增的片段大小约为1 800 bp(图1),与预期片段大小相符。
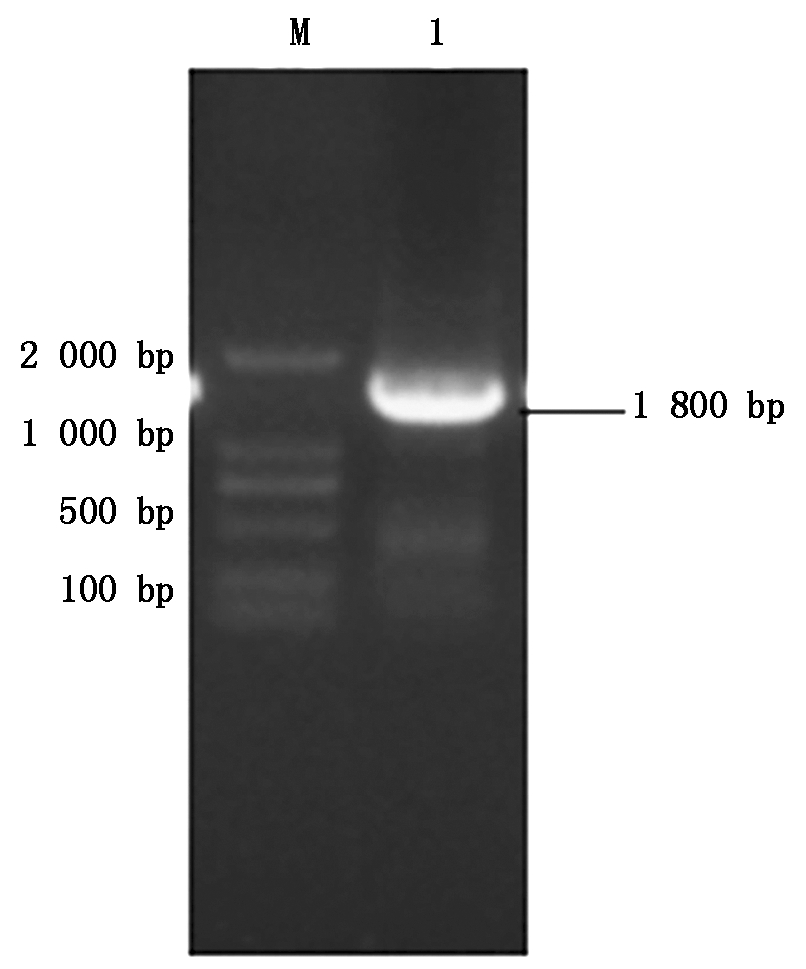
M.DL2000 分子量Marker;1.RdRp扩增片段。
M.DL2000 Marker;1.RdRp fragment.
图1 鸭坦布苏病毒RdRp基因PCR扩增
Fig.1 Amplification of DTMUV RdRp gene
2.2 重组质粒pCMV-Flag-RdRp的PCR和双酶切鉴定
菌液PCR结果显示,挑取的单克隆可以扩增得到与RdRp大小一致的DNA片段(图2)。同时利用限制性内切酶EcoR Ⅰ、Xho Ⅰ对重组质粒pCMV-Flag-RdRp进行双酶切鉴定,通过凝胶电泳分析,结果出现2条特异性的目的条带(图2),一条大小约为4 300 bp,为pCMV-N-Flag载体条带,另一条大小1 800 bp,与RdRp大小一致。测序结果经Blast比对显示,通过PCR方式扩增得来的RdRp基因序列与GenBank上发布的RdRp基因序列同源性达99%,氨基酸同源性100%,说明PCR过程中产生的基因突变对RdRp蛋白编码没有影响。以上结果都表明,真核质粒pCMV-Flag-RdRp构建成功。
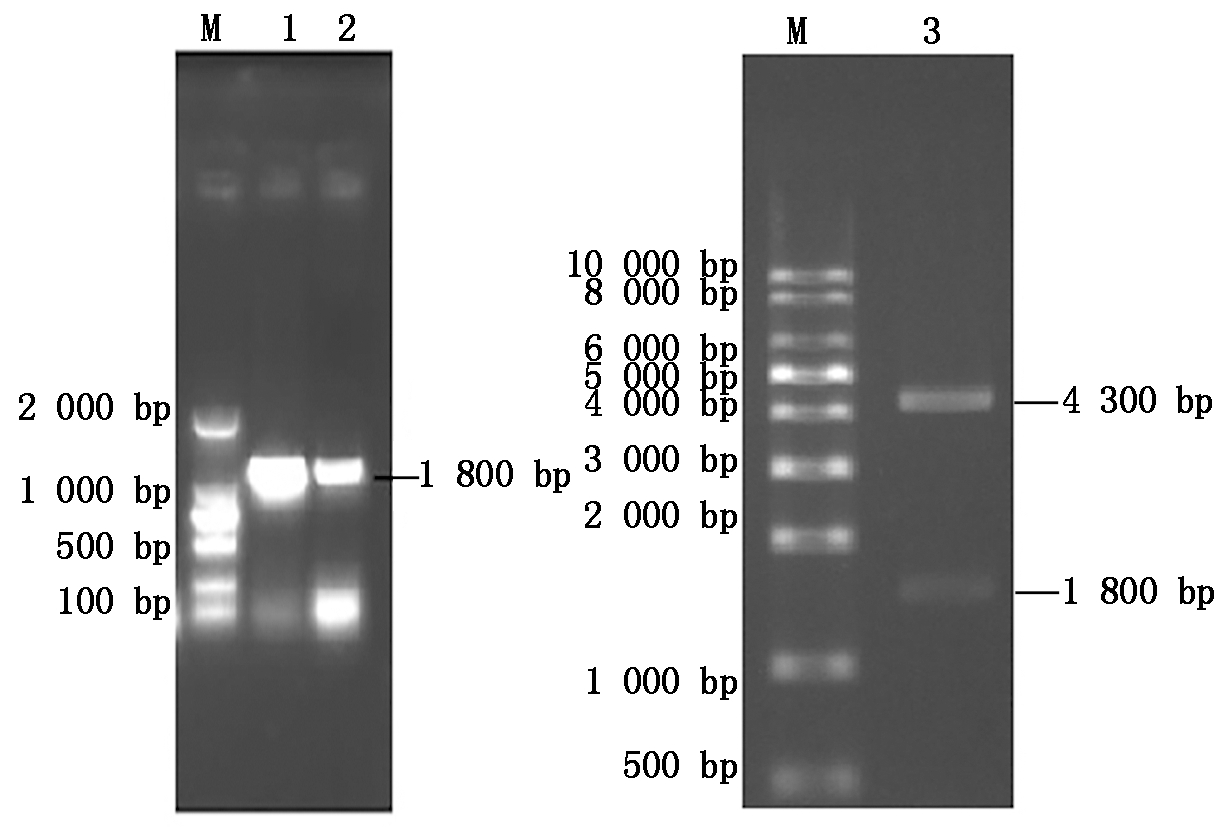
M.DNA分子质量标准;1-2.DTMUV RdRp基因的PCR产物;
3.pCMV-Flag-RdRp双酶切产物。
M.DL2000 Marker;1-2.PCR products of DTMUV RdRp;3.Double
enzyme digestion analysis of recombinant plasmid pCMV-Flag-RdRp.
图2 重组质粒pCMV-Flag-RdRp鉴定
Fig.2 Identification of recombinant plasmid
pCMV-Flag-RdRp
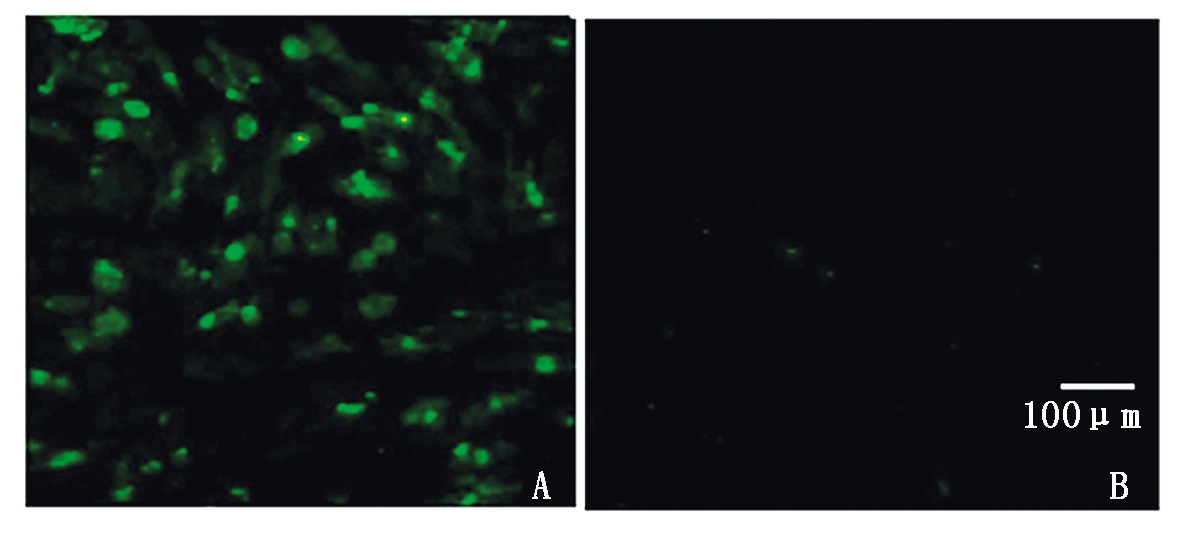
A. pCMV-Flag-RdRp重组真核质粒;B.正常BHK-21空白对照。
A.Recombinant plasmid pCMV-Flag-RdRp; B.Normal BHK-21 cell control.
图3 重组质粒pCMV-Flag-RdRp在BHK-21细胞中的表达
Fig.3 The expression of pCMV-Flag-RdRp
plasmid in BHK-21 cells
2.3 重组质粒pCMV-Flag-RdRp转染检测
用脂质体转染试剂lipofectamine 3000介导pCMV-Flag-RdRp转染至BHK-21细胞,继续培养24 h后,间接免疫荧光检测RdRp表达情况。结果发现,重组质粒pCMV-Flag-RdRp转染细胞能够观测到绿色荧光,且荧光多数位于细胞质中,正常未转染细胞无绿色荧光(图3),以上结果表明,重组质粒pCMV-Flag-RdRp能于BHK-21细胞中转染并表达,其转染效率未受外源插入蛋白的影响。
2.4 DTMUV RdRp在BHK-21细胞中的表达
经Western Blot检测,重组DTMUV RdRp蛋白与坦布苏病毒阳性小鼠血清能发生特异性结合,条带分子量大小位于约70 ku处,表明重组DTMUV RdRp蛋白具有免疫反应活性,结果见图4。
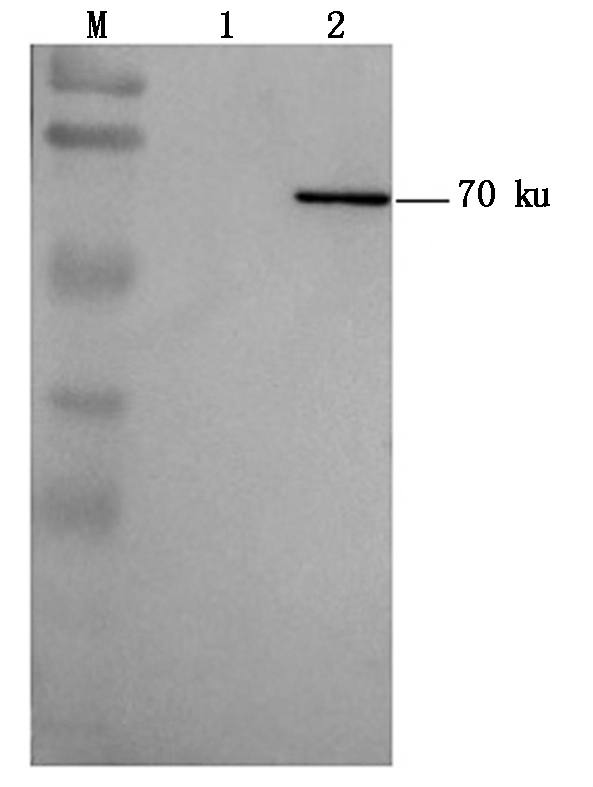
M.蛋白质分子质量标准;1.pCMV-N-Flag载体;
2.pCMV-Flag-RdRp目的蛋白。
M. Protein standard ladder;1. pCMV-N-Flag vector;
2. pCMV-Flag-RdRp target protein.
图4 Western Blot鉴定pCMV-Flag-RdRp的表达
Fig.4 Determination of fusion protein
expression using Western Blot
2.5 DTMUV RdRp的生物信息学分析
首先使用ProtParam软件对DTMUV RdRp的基础理化性质进行分析。结果表明,DTMUV RdRp基因全长1 818 bp,共编码606个氨基酸,总分子式为C3091H4810N870O894S41,理论蛋白质分子质量为69.777 9 ku,正电荷残基(精氨酸+赖氨酸)83个,负电荷残基(天冬氨酸+谷氨酸)81个,等电点为7.94。同时发现,RdRp蛋白质亲水性平均系数为-0.536,为亲水性蛋白质。进一步分析发现其消光系数为160 475,脂肪指数为70.15,不稳定指数为42.15(>40),表明该蛋白可能为不稳定性蛋白质。半衰期预测显示,该蛋白质在大肠杆菌中的半衰期大于10 h,在酵母中的半衰期大于20 h,在哺乳动物网织红细胞中的半衰期为30 h。
采用软件ProtScale对RdRp蛋白的疏水性进行分析,结果如图5所示,其中负值表示亲水,正值表示疏水,结果显示,该蛋白第529位氨基酸的亲疏水性分值最低,为-2.911,323位氨基酸分值最高为1.689(图5)。
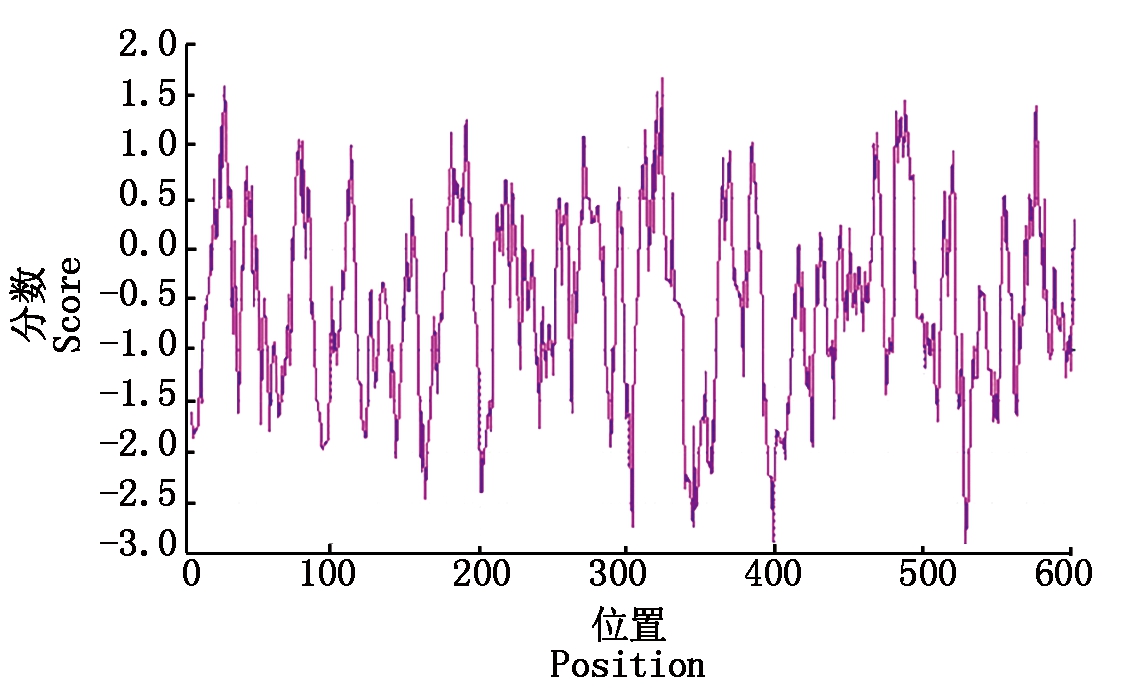
图5 DTMUV RdRp蛋白的疏水性分析
Fig.5 Hydrophobicity analysis of DTMUV RdRp protein
采用软件Tmpred对RdRp蛋白的二级结构进行分析,结果显示该蛋白存在3个潜在跨膜区,位于第173-192位,306-324位,481-497位氨基酸(图6)。
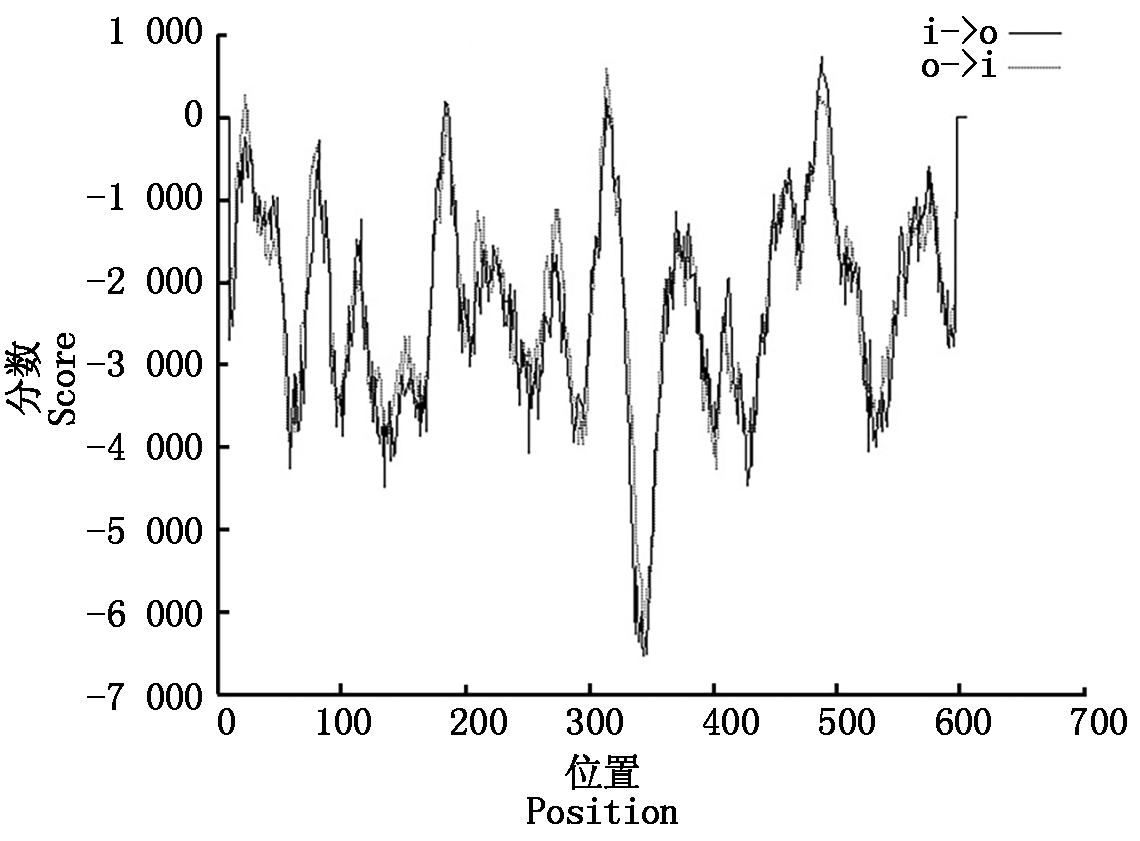
图6 DTMUV RdRp蛋白的潜在跨膜区分析
Fig.6 The potential transmembrane domain
analysis of DTMUV RdRp protein
磷酸化位点的预测结果表明,该蛋白含有84个潜在的磷酸化位点。N-糖基化位点预测结果显示,该蛋白序列没有潜在的N-糖基化位点,O-糖基化位点预测结果显示,该蛋白序列有9个潜在的O-糖基化位点,分别位于17,310,367,487,488,492,534,537,564位。SignalP 5.0 Server对其信号肽的预测分析表明,DTMUV RdRp蛋白不存在信号肽区域。DTMUV RdRp蛋白的二级结构主要由α-螺旋和无规则卷曲组成(图7),其中α-螺旋占46.37%,无规则卷曲占35.31%,延伸链占12.54%。运用SWISS-MODEL软件预测了DTMUV RdRp蛋白可能存在的三级结构(图8)。
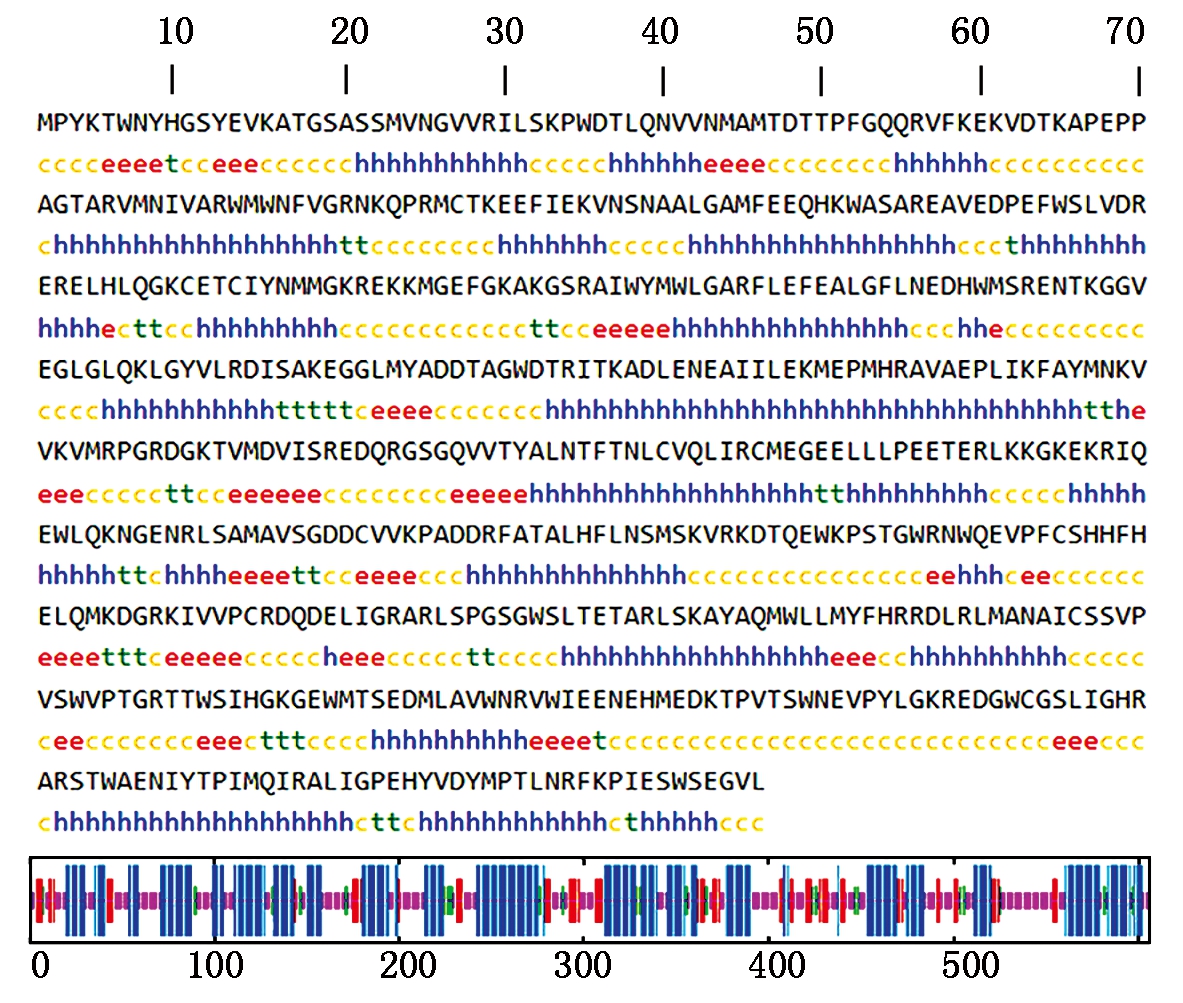
c和短线.无规则卷曲;
h和长竖线.α-螺旋;e和中长竖线.延伸链。
c and short line.Random coil;h and long line.α-helix;
e and mid-long line.Extending strand.
图7 DTMUV RdRp蛋白二级结构预测
Fig.7 The prediction of secondary
structure of DTMUV RdRp protein
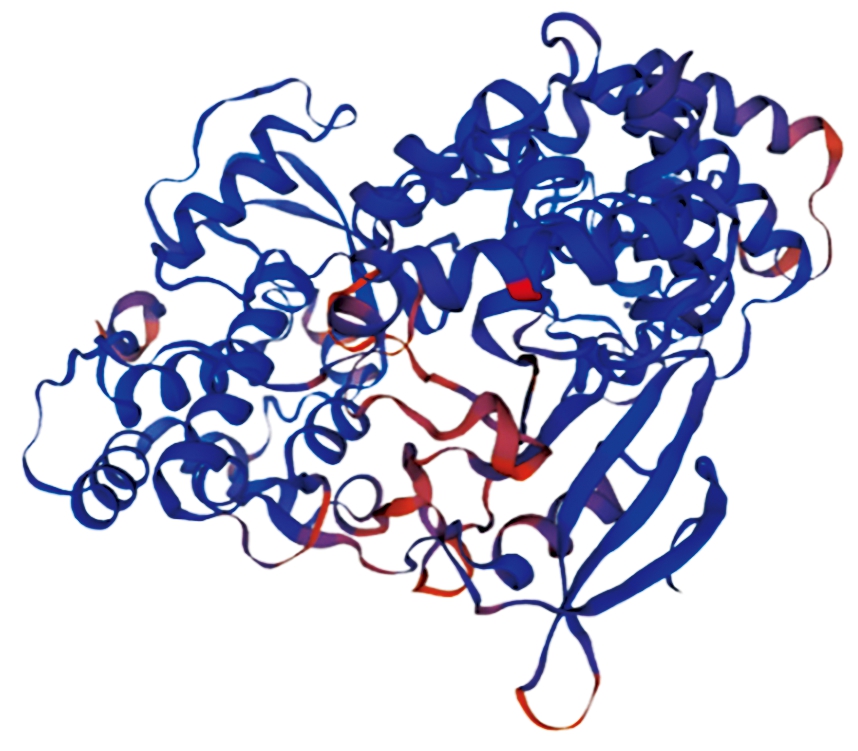
图8 DTMUV RdRp蛋白三级结构预测
Fig.8 The prediction of tertiary
structure of DTMUV RdRp protein
3 讨论和结论
本研究利用RT-PCR方法扩增出DTMUV RdRp基因,亚克隆至pCMV-N-Flag真核表达载体中,并转染至BHK-21细胞,通过间接免疫荧光观察和Western Blot鉴定该蛋白的表达。同时利用生物信息学软件分别对RdRp蛋白的序列进行分析,对结构和功能进行预测。本研究结果显示,DTMUV RdRp能够在真核细胞中稳定高效表达,对DTMUV RdRp的生物信息学预测结果同时显示,该目的蛋白含有84个潜在的磷酸化位点和9个潜在的O-糖基化位点。糖基化修饰具有重要的调节蛋白质功能作用,蛋白经过糖基化修饰后,对后续参与免疫反应有重要的实际意义[16]。而磷酸化的作用则主要体现在调节、控制蛋白质活力与功能等,同时拥有控制细胞增殖、分化等生命活动能力[17]。非结构蛋白NS5重要功能基团RdRp蛋白是否通过糖基化或磷酸化、或两者兼有参与坦布苏病毒在体内复制过程中病毒多聚蛋白的生成,还需要后续更多试验研究加以验证。
黄病毒属病毒RdRp属于非结构蛋白NS5功能基团,该功能基团与病毒编码的其他蛋白酶(如NS3蛋白的解旋酶)以及一些宿主因子共同构成病毒的复制酶系统,在宿主细胞内调控病毒基因组的复制。前期研究表明,重组黄病毒RdRp蛋白可以通过依赖引物和直接合成2种方式行使病毒RNA的合成功能,同时发现,仅仅只有3′端并不具有模板活性,还需要与5′端同时存在才能合成RNA[18]。近年来,随着对虫媒病毒的研究深入,人们发现NS5重要功能基团RdRp蛋白除了参与病毒在宿主体内的复制,还能够与宿主Ⅰ型干扰素信号转导通路中的信号分子发生相互作用,进而抑制宿主的天然免疫反应,为病毒自身的复制创造有利条件。Best等[19]在研究蜱传脑炎病毒时发现,当病毒在树突状细胞中复制时,非结构蛋白NS5能够与Ⅰ型干扰素受体相互作用,进而抑制了Janus激酶的磷酸化,最终阻断Ⅰ型干扰素信号转导通路,随着研究的继续深入,发现能够阻断I型干扰素信号通路的结构域就存在于RdRp中,为2段不连续的氨基酸序列。由于RdRp蛋白拥有调控病毒复制的特殊功能,通过抑制RdRp活性,可以抑制病毒在宿主体内的复制,因此RdRp成为黄病毒属病毒抑制剂研究的天然靶点。现在研究较多的抑制RdRp活性的核苷(酸)类抑制剂,可以作为竞争性的原料类似物终止新生病毒RNA子链的延伸,从而抑制病毒在体内的复制,在前期研究中已被证明具有较好疗效。埃默里大学的Kolykhalov课题组研究发现,UTP核苷酸的竞争抑制剂能够有效抑制寨卡病毒RdRp活性,从而阻断病毒复制[20]。此外,一种丙型肝炎病毒NS5B聚合酶的核苷酸抑制剂-索非布韦,已经通过FDA批准美国上市,该抑制剂能够有效抑制寨卡病毒在人类细胞中的复制[21]。综上,深入研究黄病毒属病毒NS5-RdRp蛋白不仅对于深入研究鸭坦布苏病毒的致病机制、明晰该疫病全面的防控措施具有重要的意义,而且对其他黄病毒属病毒病的综合防控具有重要的参考价值。
[1] Li N,Zhao J,Yang Y D,Zeng Y Q,Liu S D. Innate immune responses to duck Tembusu virus infection[J].Veterinary Research,2020,51(1):87. doi:10.1186/s13567-020-00814-9.
[2] Zhao D M,Han K K,Huang X M,Zhang L J,Wang H L,Liu N,Tian Y J,Liu Q T,Yang J,Liu Y Z,Li Y. Screening and identification of B-cell epitopes within envelope protein of Tembusu virus[J].Virology Journal,2018,15(1):142. doi:10.1186/s12985-018-1052-1.
[3] Wang X L,Yang Z Y,Wang X Q,Duan H J,Liu L X,Cheng H M,Yang C H,Hou L D,Pan J,Zhao J C,Liu Y H,Lin J. Development of a hemagglutination inhibition assay for Duck tembusu virus[J].Avian Diseases,2019,63(2):298-301. doi:10.1637/11954-082018-Reg.1.
[4] Zhang W,Chen S,Mahalingam S,Wang M S,Cheng A C. An updated review of avian-origin Tembusu virus:A newly emerging avian Flavivirus[J].The Journal of General Virology,2017,98(10):2413-2420. doi:10.1099/jgv.0.000908.
[5] Zhang L J,Zhao D M,Han K K,Huang X M,Liu Y Z,Liu Q T,Yang J,Li S,Li Y. Tembusu virus enters BHK-21 cells through a cholesterol-dependent and clathrin-mediated endocytosis pathway[J].Microbial Pathogenesis,2020,147:104242. doi:10.1016/j.micpath.2020.104242.
[6] Sun X J,Liu E X,Iqbal A,Wang T Z,Wang X D,Haseeb A,Ahmed N,Yang P,Chen Q S. The dynamic distribution of Duck tembusu virus in the spleen of infected shelducks[J].BMC Veterinary Research,2019,15(1):112. doi:10.1186/s12917-019-1860-6.
[7] Li L L,Zhang Y,Dong J W,Zhang J Q,Zhang C H,Qin J R,Sun M H,Xu Z H. Development of chimeric virus-like particles containing the E glycoprotein of Duck tembusu virus[J].Veterinary Microbiology,2019,238:108425. doi:10.1016/j.vetmic.2019.108425.
[8] Lü J,Yang L X,Qu S H,Meng R Z,Li Q,Liu H C,Wang X Y,Zhang D B. Detection of neutralizing antibodies to Tembusu virus:Implications for infection and immunity[J].Frontiers in Veterinary Science,2019,6:442. doi:10.3389/fvets.2019.00442.
[9] Zhao D M,Yang J,Han K K,Liu Q T,Wang H L,Liu Y Z,Huang X M,Zhang L J,Li Y. The unfolded protein response induced by Tembusu virus infection[J].BMC Veterinary Research,2019,15(1):34. doi:10.1186/s12917-019-1781-4.
[10] Chen L,Yu B,Hua J G,Ni Z,Ye W C,Yun T,Zhang C. Optimized expression of Duck tembusu virus E gene delivered by a vectored Duck enteritis virus in vitro[J].Molecular Biotechnology,2019,61(10):783-790. doi:10.1007/s12033-019-00206-1.
[11] Wang Y W,Zhang S,Tang Y,Diao Y X. Screening of Duck tembusu virus NS3 interacting host proteins and identification of its specific interplay domains[J].Viruses,2019,11(8):740. doi:10.3390/v11080740.
[12] Peng S H,Su C L,Chang M C,Hu H C,Yang S L,Shu P Y. Genome analysis of a novel Tembusu virus in Taiwan [J].Viruses,2020,12(5):567. doi:10.3390/v12050567.
[13] Han K K,Zhao D M,Liu Y Z,Liu Q T,Huang X M,Yang J,Zhang L J,Li Y. The ubiquitin-proteasome system is necessary for the replication of Duck tembusu virus[J].Microbial Pathogenesis,2019,132:362-368. doi:10.1016/j.micpath.2019.04.044.
[14] 刘强强,王明连,李艳. 登革病毒非结构蛋白NS5研究进展[J].中国病毒病杂志,2016,6(4):317-321. doi:10.16505/j.2095-0136.2016.04.015.
Liu Q Q,Wang M L,Li Y. Research progress on Dengue virus nonstructural protein 5 [J].Chin J Viral Dis,2016,6(4):317-321.
[15] Selisko B,Dutartre H,de Guillemot J C,Debarnot C,Benarroch D,Khromykh A,de Desprès P,Egloff M P,Canard B. Comparative mechanistic studies of de novo RNA synthesis by Flavivirus RNA-dependent RNA polymerases[J].Virology,2006,351(1):145-158. doi:10.1016/j.virol.2006.03.026.
[16] 王晓龙,王秀然,卢天成. 蛋白质糖基化修饰的研究进展[J].基因组学与应用生物学,2017,36(10):4380-4384. doi:10.13417/j.gab.036.004380.
Wang X L,Wang X R,Lu T C. The research progress of protein glycosylation modification[J].Genomics and Applied Biology,2017,36(10):4380-4384.
[17] Korkuc′ P,Walther D. Spatial proximity statistics suggest a regulatory role of protein phosphorylation on compound binding[J].Proteins,2016,84(5):565-579. doi:10.1002/prot.25001.
[18] 周希珍,赵慧,高岚,秦成峰. 虫媒黄病毒NS5蛋白的生物学功能研究进展[J].军事医学,2012,36(1):70-72.doi:10.3969/j.issn.1674-9960.2012.01.018.
Zhou X Z,Zhao H,Gao L,Qin C F. Advance in research on biological functions of nonstructural protein 5 of arthropod-borne Flavivirus[J].Military Medical Sciences,2012,36(1):70-72.
[19] Best S M,Morris K L,Shannon J G,Robertson S J,Mitzel D N,Park G S,Boer E,Wolfinbarger J B,Bloom M E. Inhibition of interferon-stimulated JAK-STAT signaling by a tick-borne flavivirus and identification of NS5 as an interferon antagonist[J].Journal of Virology,2005,79(20):12828-12839.doi:10.1128/jvi.79.20.12828-12839.2005.
[20] Lu G F,Bluemling G R,Collop P,Hager M,Kuiper D,Gurale B P,Painter G R,Rosa A D L,Kolykhalov A A. Analysis of ribonucleotide 5′-triphosphate analogs as potential inhibitors of Zika virus RNA-dependent RNA polymerase by using nonradioactive polymerase assays [J].Antimicrob Agents Chemother,2017,61(3):e01967. doi:10.1128/AAC.01967-16.
[21] Reznik S E,Ashby C R. Sofosbuvir:an antiviral drug with potential efficacy against Zika infection[J].International Journal of Infectious Diseases,2017,55:29-30. doi:10.1016/j.ijid.2016.12.011.