NAC的命名源于矮牵牛基因NAM,拟南芥基因ATAF1、ATAF2及CUC2的首字母缩略词,它是植物所特有的特异性转录因子[1-2]。NAC家族N末端高度保守,含有相似的DNA结合结构域,但其C末端高度多样化,不包含任何已知的蛋白结构域[3-4]。利用植物基因组全测序,鉴定出多个作物中含有NAC转录因子,例如拟南芥(Arabidopsis thaliana)中有117个NAC基因,大豆(Glycine max)中有152个,水稻(Oryza sativa)中有151个,烟草(Nicotiana tabacum)中有152个,芸豆(Phaseolus vulgaris)中有106个等[5-6]。同时,已有大量研究发现NAC转录因子在植物的生长发育过程中、抗逆反应过程中、应对各种胁迫过程中发挥着举足轻重的作用[7-9]。如玉米ZmNAC33基因可以诱导包括DRE、MYC和MYB等基因的表达,从而改善植物的耐旱性[10];大豆GmNAC15基因可以应对多种非生物胁迫,并且具有耐盐性[11];PwNAC2通过多种信号通路增强了植物对干旱的耐受性[12];番茄SlNAC1通过提高过氧化物歧化酶、过氧化氢酶和保持较高的光化学效率等机制来提高植株对寒冷的耐受性[13];GmNAC109参与了生长素信号通路,有助于调控毛状根的形成[14];BrNAC041参与ABA对GA在白菜叶片衰老过程中的拮抗作用[15];在低温、甘露醇和NaCl处理后,CaNAC035的基因沉默会抑制辣椒幼苗的生长[16]等。目前,越来越多的NAC转录因子被鉴定,但是对芸豆NAC转录因子的功能研究较少,对盐碱响应的分析更是鲜有报道。
植物的非生物胁迫包括干旱胁迫、低温胁迫、盐碱胁迫、高温胁迫及重金属胁迫等,在不同的理化因素逆境条件下植物会相应地产生抗性[17]。盐碱是影响植物生长的主要环境因子之一,会对植物造成大量的影响[18],如植株不能正常生长、黄化、萎蔫[19-20]、导致植株严重减产等[21-22]。芸豆是豆科植物,可食用,学名为普通菜豆。芸豆含有丰富的营养成分,如蛋白质、脂肪、碳水化合物、膳食纤维等,除供人们食用之外,在药用方面也存在超高的价值,如提高免疫力[23]。近年来,芸豆渐渐进入人们的视野,我国的种植面积也不断扩大,然而芸豆为盐碱敏感作物,土壤盐碱严重制约了芸豆的生长发育[24]。
本研究利用高通量测序技术,建立100 mmol/L NaHCO3和100 mmol/L NaCl处理芸豆叶片组织24,48 h的转录组,以水处理为对照,共获得8个盐碱响应NAC转录因子,利用染色体定位、进化发育关系、基因及蛋白结构、启动子所含元件和组织表达模式等多方面进行综合分析,为进一步研究芸豆NAC转录因子的抗逆功能和调控机制提供参考。
1 材料和方法
1.1 构建芸豆叶片组织盐碱胁迫转录组
本研究使用芸豆品种为耐盐碱品种HYD,用10% NaClO溶液浸泡10 min,用清水和无菌水各清洗3遍进行消毒,将消毒后的种子放置在发芽盒中,并在恒温培养箱中25 ℃培养3 d催芽,选取发芽整齐一致的种子移栽到1/4霍格兰营养液中,培养条件为光14 h/暗10 h (光强400 μmol/(m2·s))。培养5 d后换1/2霍格兰营养液,当芸豆长至第2对新生复叶时更换霍格兰全营养液并加入100 mmol/L NaHCO3和100 mmol/L NaCl溶液处理,分别在处理24,48 h对芸豆幼苗叶片的同一部位取样,以0 h为对照,每个处理3次重复。取样后放入液氮中冷冻并放置-80 ℃冰箱备用,后将所取样品送至百迈客公司进行高通量测序。
1.2 总RNA的提取和cDNA的合成
用所保存的0,24,48 h芸豆叶片样品,根据TRIzol法提取芸豆叶片RNA,用1%琼脂糖凝胶电泳检测RNA的完整性。之后使用东洋纺的反转录试剂盒,以总RNA为模板合成cDNA。
1.3 芸豆NAC基因系统进化发育分析
利用Phytozome数据库(https://phytozome.jgi.doe.gov)下载芸豆NAC基因的蛋白质序列,Blast与芸豆NAC基因同源的拟南芥及水稻NAC蛋白。利用Mega 6.0软件进行序列比对,并构建进化树。
1.4 芸豆NAC序列分析
在NCBI数据库 (https://www.ncbi.nlm.nih.gov)下载芸豆NAC基因的CDS序列和蛋白序列,利用在线软件MEME (http://meme-suite.org)预测保守序列,通过GSDS (http://gsds.cbi.pku.edu.cn)绘制基因结构图,利用在线软件SOSUI (http://harrier.nagahama-i-bio.ac.jp/sosui/sosui_submit.html)进行蛋白跨膜结构预测,通过在线网站NetPhos 2.0 (http://www.cbs.dtu.dk/services/NetPhos-2.0/)预测磷酸化位点数目及种类,利用PSORT (http://psort1.hgc.jp/form.html)进行蛋白亚细胞定位预测,利用在线生物信息学工具ExPASy (https://web.expasy.org/protparam)对基因理化性质进行分析。
1.5 芸豆NAC蛋白二级结构预测
利用在线网站PSIPRED (http://bioinf.cs.ucl.ac.uk/psipred/#)预测NAC蛋白二级结构。
1.6 芸豆NAC基因的染色体定位分析
在NCBI数据库下载芸豆染色体信息、基因信息及所在染色体位置,利用MapChart软件制作染色体定位图。
1.7 芸豆NAC基因的启动子分析
在Phytozome数据库下载芸豆NAC基因转录起始位点上游1 500 bp的启动子序列,利用在线数据库plantCARE (http://bioinformatics.psb.ugent.be/webtools/plantcare/html)预测芸豆NAC基因启动子所包含逆境相关顺式作用元件,使用IBS 1.0软件绘制芸豆NAC启动子分析图。
1.8 芸豆NAC基因表达特性分析
将保存的芸豆cDNA稀释10倍做模板进行qRT-PCR分析,利用Primer 5软件设计引物,以ACT11为内参[25] (表1),使用2-ΔΔCt法计算相对表达量,利用Excel软件分析并绘制柱形图,每个样品进行3次生物学重复。同时在Phytozome数据库中获取芸豆在不同组织中的表达数据,利用TBtools软件绘制热图。
表1 荧光定量PCR扩增引物序列
Tab.1 Primer sequence amplified by
fluorescence quantitative PCR

基因名称Gene name引物序列 (5′-3′)Primer sequence (5′-3′)Phvul.001G192000F:CCACAGGGTCTCCAAATCAR:ACTCGTTCATCTTCCAATCAGTPhvul.002G283600F:GGAATTGTGATCCGCAAGAGR:CATGCAAGACCCAGTCAGTTPhvul.004G077400F:TGAGATTGGATGACTGGGTTCTR:TGTGGAGGAGGGACAAGAGTPhvul.005G084500F:GCAGGGAACACACCCAATACR:GCACCCAGGTTCTCAATGTTPhvul.006G188900F:GCCACCACAAAGTAGGAGTCAR:ACCCAATCATCAAGCCTCAAPhvul.008G159200F:CCAGGATACCAAGGCAATCR:TGTCGTCCAAGCCATCATAPhvul.009G156300F:CATCCCAACATAATCCCAGACR:CGTTCCCACTTTCTCGTCAAPhvul.011G024700F:CTGCCCTTGTTGCAGAATGTR:GCTGCAGATCAGGAAGGTTGACT11F:TGCATACGTTGGTGATGAGGR:AGCCTTGGGGTTAAGAGGAG
2 结果与分析
2.1 芸豆盐碱胁迫响应NAC基因的鉴定
芸豆中含有106个NAC基因,根据转录组数据分析,共获得8个盐碱响应NAC转录因子,这8个基因能够不同程度响应盐碱胁迫,分子量为23 676.36~44 354.33 ku,蛋白编码203~394个氨基酸,等电点4.74~8.86 (表2),可见这8个基因理化性质差异不大。此外,有3个基因具有1~8个跨膜结构域。
表2 8个盐碱胁迫响应NAC基因信息和理化性质分析
Tab.2 NAC gene information and physicochemical properties analysis of 8 genes in response to saline-alkali stress
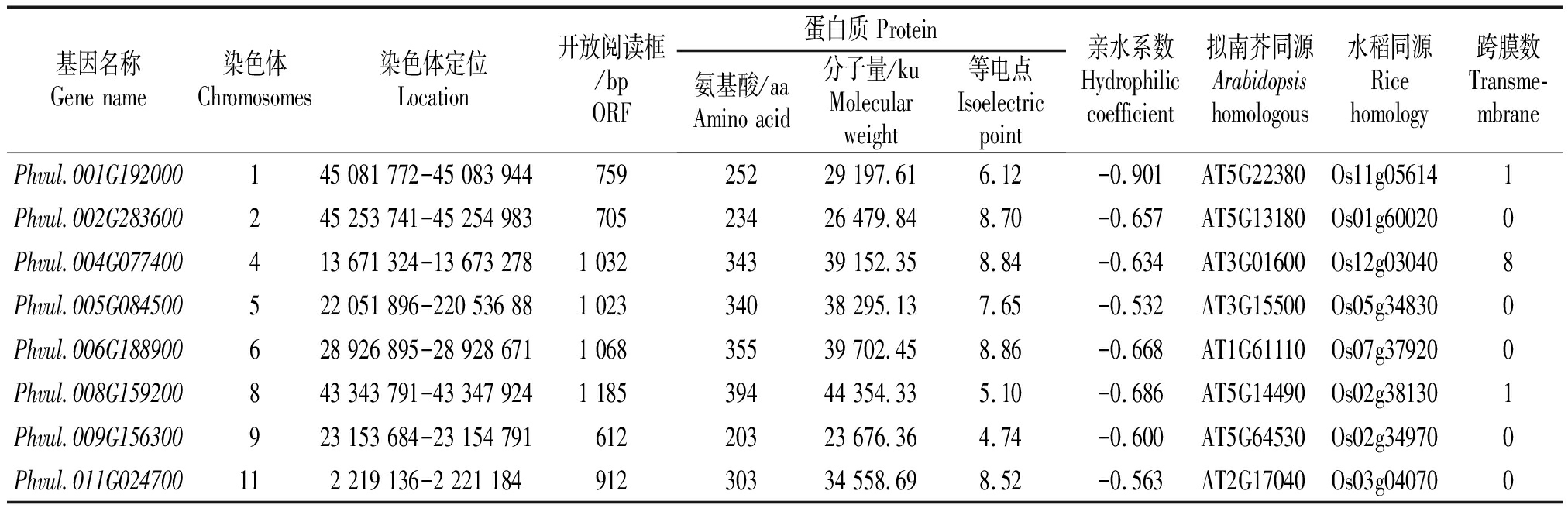
基因名称Gene name染色体Chromosomes染色体定位Location开放阅读框/bpORF蛋白质 Protein氨基酸/aaAmino acid分子量/kuMolecularweight等电点Isoelectricpoint亲水系数Hydrophiliccoefficient拟南芥同源Arabidopsishomologous水稻同源Rice homology跨膜数Transme-mbrane Phvul.001G192000145 081 772-45 083 94475925229 197.616.12-0.901AT5G22380Os11g056141Phvul.002G283600245 253 741-45 254 98370523426 479.848.70-0.657AT5G13180Os01g600200Phvul.004G077400413 671 324-13 673 2781 03234339 152.358.84-0.634AT3G01600Os12g030408Phvul.005G084500522 051 896-220 536 881 02334038 295.137.65-0.532AT3G15500Os05g348300Phvul.006G188900628 926 895-28 928 6711 06835539 702.458.86-0.668AT1G61110Os07g379200Phvul.008G159200843 343 791-43 347 9241 18539444 354.335.10-0.686AT5G14490Os02g381301Phvul.009G156300923 153 684-23 154 79161220323 676.364.74-0.600AT5G64530Os02g349700Phvul.011G024700112 219 136-2 221 18491230334 558.698.52-0.563AT2G17040Os03g040700
2.2 芸豆盐碱胁迫响应NAC基因系统进化发育分析
为了进一步了解芸豆耐盐碱相关的NAC基因与其他植物来源的NAC基因系统进化关系,通过NCBI获取了8个芸豆及其同源拟南芥和同源水稻NAC序列,利用Mega 6.0软件,构建系统进化发育树。进化树分析结果显示(图1),8个芸豆NAC被分为4个亚家族(a~d),其中在a族中,芸豆盐碱胁迫NAC基因最多,c族中含有2个芸豆NAC基因,而b族和d族中都仅含有1个芸豆NAC基因,与其他基因亲缘关系较远。
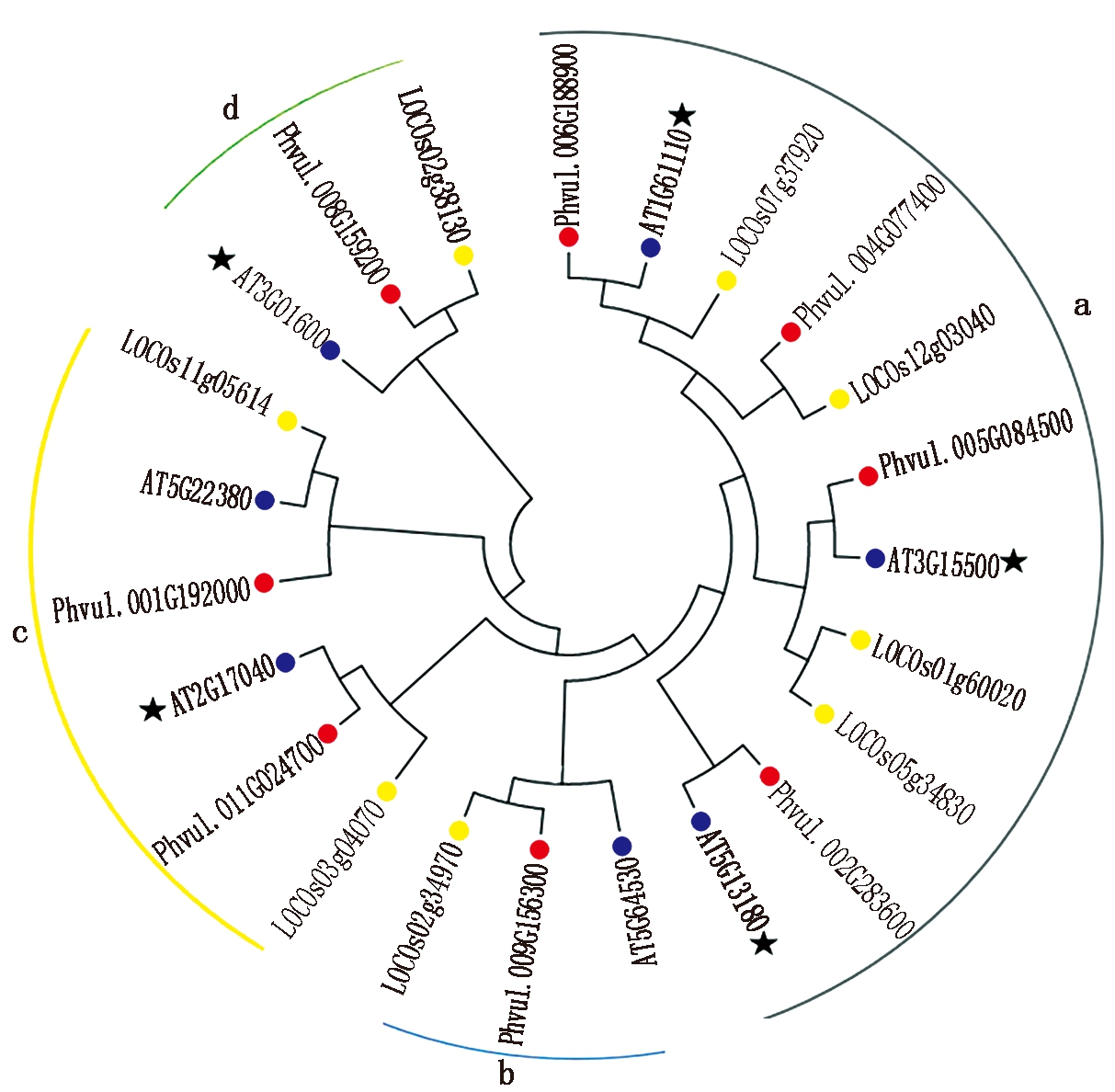
星号标识代表该基因已有相关报道。
The asterisk indicates that the gene has been reported.
图1 芸豆拟南芥、水稻NAC蛋白系统进化树
Fig.1 Phylogenetic relationship of NAC protein from
common bean, Arabidopsis and rice
2.3 盐碱胁迫下芸豆NAC基因结构和保守域分析
为了揭示芸豆中NAC家族基因序列的多样性,利用MEME在线预测工具,预测响应盐碱胁迫的NAC蛋白中10个不同的保守元件 (图2),大部分都在N端,8个芸豆NAC基因的N段均含有Motif 3,这些元件组成了芸豆NAC转录因子特有的保守的DNA结合域。所有NAC基因的N端都含有4~6个不等的Motif,仅AT5G64530中不含有Motif 6,这表明该元件并不是在所有的基因中都发挥功能。所有亚族中都能看到一些特异性Motif,如Motif 7,8,10仅在d亚家族中所特有,且在b和d亚家族中不含有Motif 1。基因结构分析显示,多数亚族的基因显示了相似的外显子-内含子结构,表明这些基因具有相似的特征。
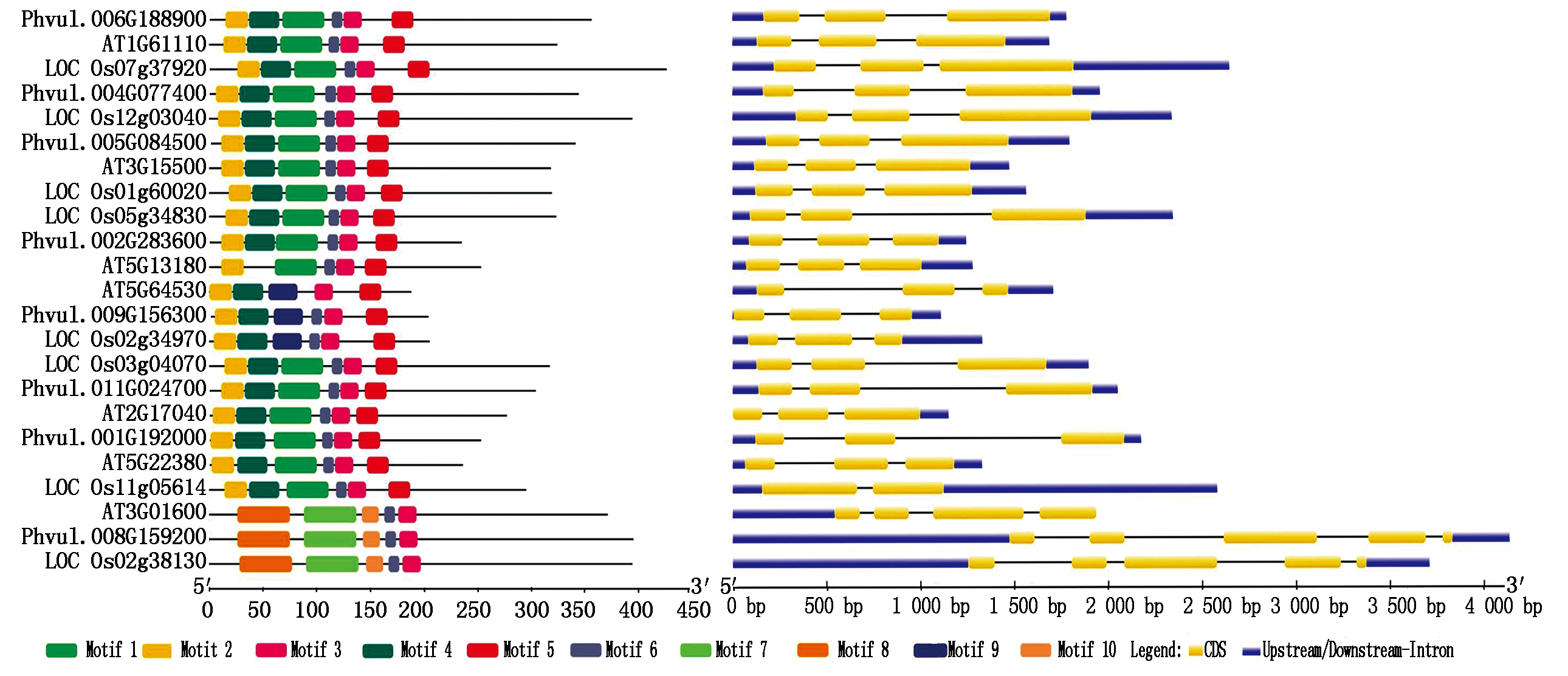
图2 8个芸豆NAC转录因子保守基序及基因结构分析
Fig.2 Conserved motif and gene structure analysis of eight common bean NAC transcription factors
2.4 芸豆盐碱胁迫响应NAC基因染色体定位分析
对芸豆8个盐碱胁迫响应NAC基因在染色体上的分布统计发现,8个基因分别分布在8个不同的染色体上,除染色体3、7、10外,其他染色体上均有盐碱响应基因分布(图3)。
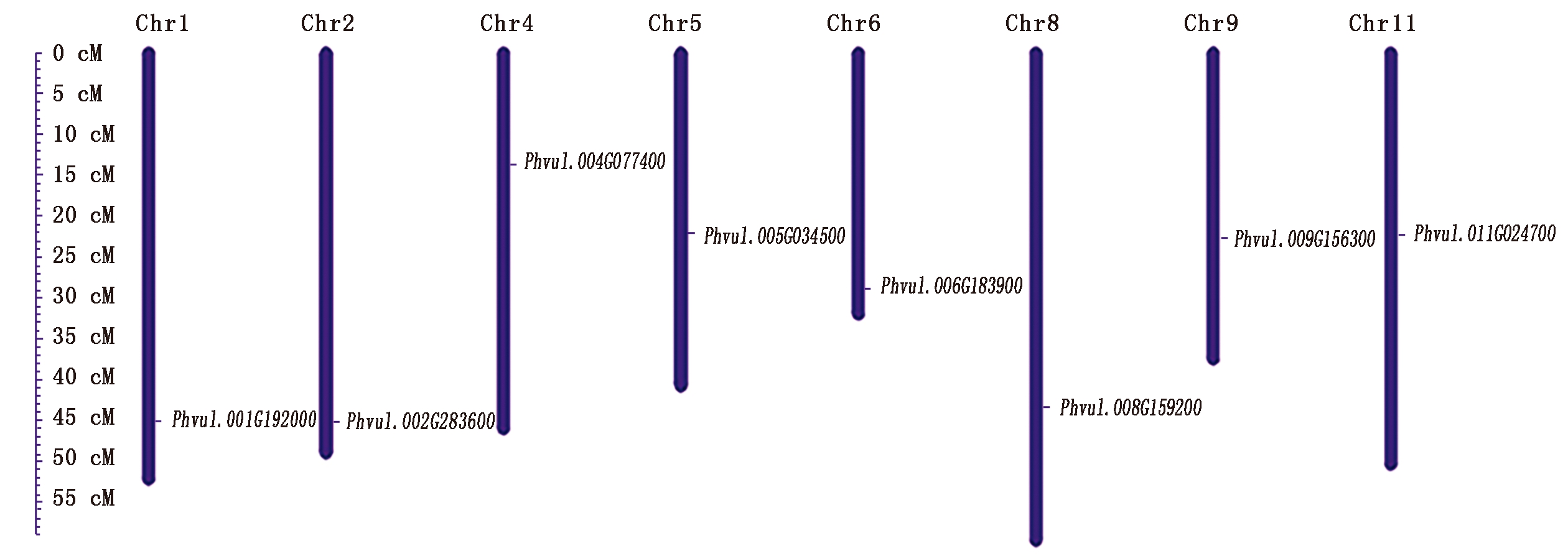
图3 8个芸豆NAC基因染色体定位分析
Fig.3 Chromosomal localization of eight common bean NAC transcription factors
2.5 芸豆盐碱胁迫响应NAC蛋白磷酸化位点和亚细胞定位点分析
8个NAC蛋白的磷酸化位点总数在15~33,不同蛋白间磷酸化位点数目相差较大,从3种磷酸化位点数目来看,丝氨酸数目最多,分别在10~22,其次为苏氨酸,酪氨酸数目最少。通过Softberry分析发现8个芸豆盐碱响应NAC蛋白都定位在细胞核上(表3)。
表3 芸豆盐碱响应NAC蛋白磷酸化位点及亚细胞定位
Tab.3 Phosphorylation site and subcellular localization of NAC protein in response to salt-alkali of common bean
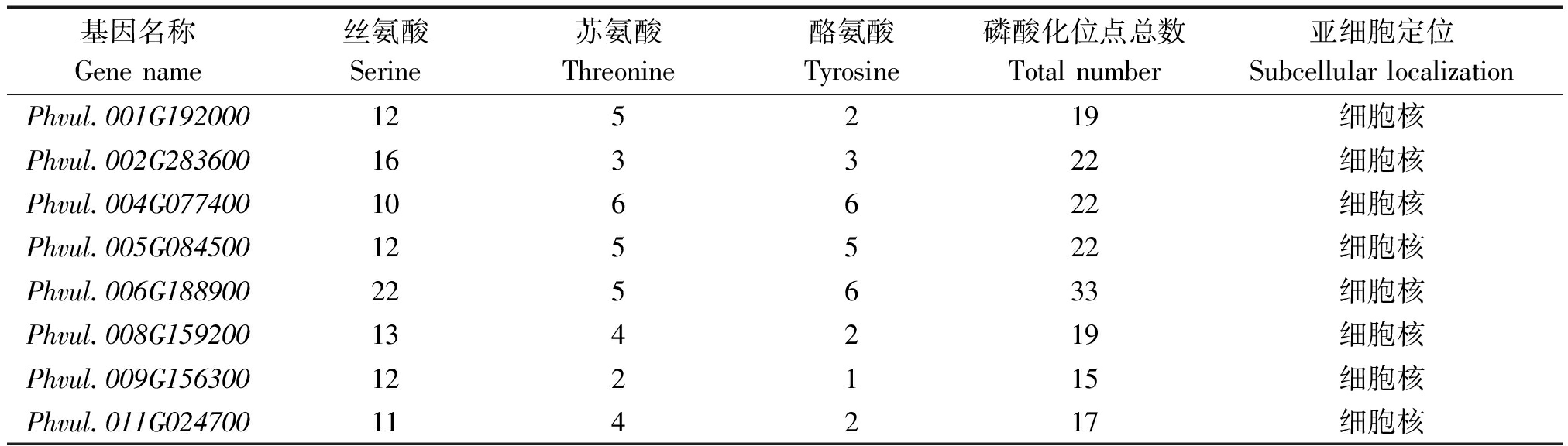
基因名称Gene name丝氨酸Serine苏氨酸Threonine酪氨酸Tyrosine磷酸化位点总数Total number亚细胞定位Subcellular localizationPhvul.001G192000125219细胞核Phvul.002G283600163322细胞核Phvul.004G077400106622细胞核Phvul.005G084500125522细胞核Phvul.006G188900225633细胞核Phvul.008G159200134219细胞核Phvul.009G156300122115细胞核Phvul.011G024700114217细胞核
2.6 芸豆盐碱胁迫响应NAC蛋白二级结构预测
通过在线软件PSIPRED预测8个芸豆NAC蛋白二级结构,可以发现,所有NAC蛋白的无规则卷曲结构比例最高(表4),α-螺旋的结构比例在7.9%~20.6%,而β-折叠的结构比例在7.6%~15.9%。
表4 芸豆NAC蛋白二级结构预测
Tab.4 Prediction of secondary structure of
common bean NAC protein %
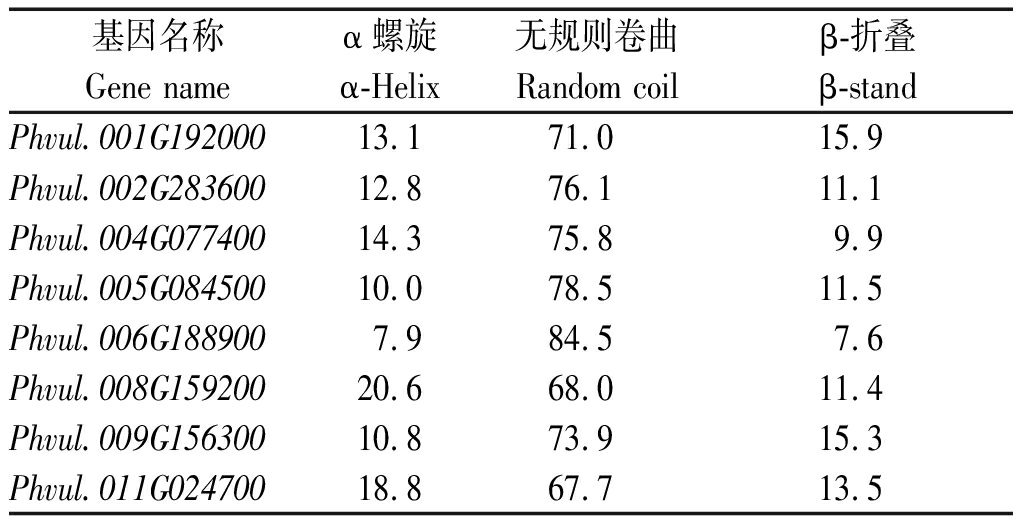
基因名称Gene nameα螺旋α-Helix无规则卷曲Random coilβ-折叠β-standPhvul.001G19200013.171.015.9Phvul.002G28360012.876.111.1Phvul.004G07740014.375.8 9.9Phvul.005G08450010.078.511.5Phvul.006G188900 7.984.5 7.6Phvul.008G15920020.668.011.4Phvul.009G15630010.873.915.3Phvul.011G02470018.867.713.5
2.7 盐碱胁迫响应芸豆NAC基因的组织部位表达模式
利用芸豆各NAC的编码序列,在Phytozome数据库中进行表达数据的检索,共得到了8个芸豆NAC基因的9个不同表达部位的表达谱。其中Phvul.005G084500基因在花蕾、花和未成熟的豆荚中均有较高的表达量,而Phvul.004G077400基因在幼叶中特异性表达,Phvul.009G156300基因成熟豆荚中有较高表达,Phvul.011G024700在各个部位均有所表达,其余4个芸豆NAC基因在芸豆部分组织中表达较低或不表达(图4)。
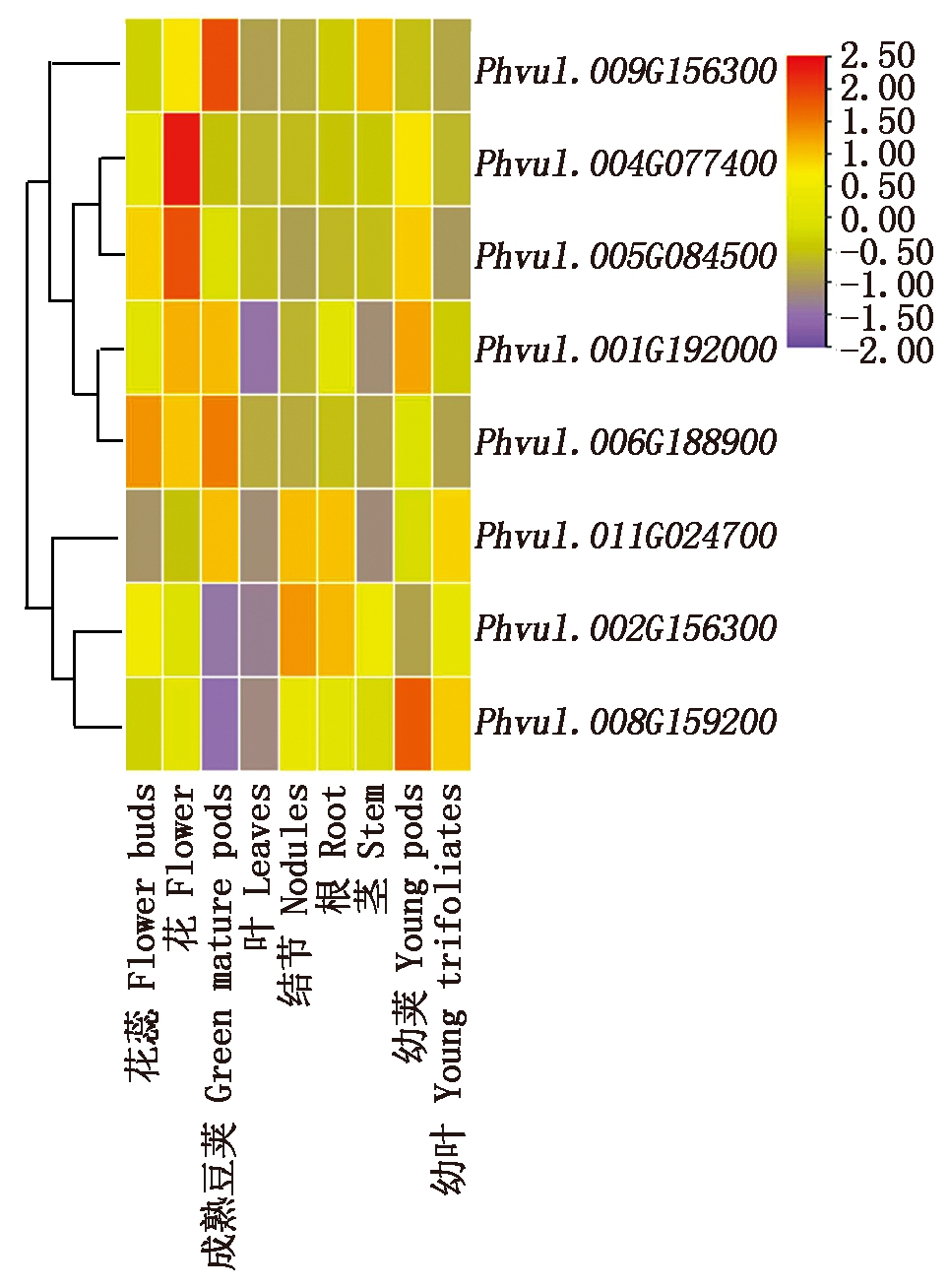
图4 8个盐碱胁迫响应的NAC基因组织部位表达分析
Fig.4 Tissue expression analysis of 8
saline-alkali response NAC
2.8 芸豆盐碱胁迫响应NAC基因启动子分析
为了更深入地了解8个芸豆NAC在盐胁迫下的功能,利用PlantCARE数据库分析芸豆NAC的转录起始位点上游1 500 bp启动子区域。结果表明,7个芸豆NAC基因含有光响应元件(ABRE),6个芸豆NAC基因含有水分胁迫响应元件(MYB)和部分参与光响应的保守DNA模块(Box 4),5个芸豆NAC基因含有光响应元件(G-box)、逆境响应元件(TC-rich)和厌氧反应元件(ARE),4个芸豆NAC基因含有ABA响应元件(MYC)和茉莉酸响应元件(CGTCA-motif),2个芸豆NAC基因含有光响应模块(AE-box),仅有一个芸豆NAC基因含有WRKY响应元件(W box)。其中有一些元件在同一个基因中重复出现3次及以上,如ABRE元件在Phvul.008G159200启动子中出现了3次,在Phvul.006G188900启动子中出现了7次,在Phvul.005G084500启动子中出现10次,Box 4元件在Phvul.008G159200启动子中出现了5次,在Phvul.002G283600和Phvul.011G024700启动子中出现了4次,ARE元件在Phvul.002G283600启动子中出现过3次,G-box元件在Phvul.004G077400中出现3次,在Phvul.005G084500中出现5次,CGTCA-motif元件在Phvul.005G084500启动子中出现3次,而MYC和MYB则出现4次,并且8个芸豆NAC启动子中都含有3~7种不同元件(图5)。

图5 8个盐碱胁迫响应NAC基因启动子序列分析
Fig.5 Sequence analysis of 8 saline-alkali response NAC gene promoters
2.9 芸豆NAC基因在不同时间点盐碱处理下的表达分析
为了更深入地了解芸豆NAC基因在不同时间点下盐碱处理的表达,对从转录组中筛选出的8个盐碱胁迫响应的NAC基因做了qRT-PCR分析,结果表明,在NaHCO3处理下4个NAC基因上调表达,2个NAC基因下调表达(图6);在NaCl处理下,1个NAC基因表现出了上调表达,4个NAC基因下调表达(图7)。同时发现,有3个基因同时响应盐胁迫与碱胁迫(图8),其中Phvul.001G192000和Phvul.008G159200在盐碱胁迫下均下调表达,Phvul.009G156300无论在碱胁迫或是盐胁迫都上调表达。
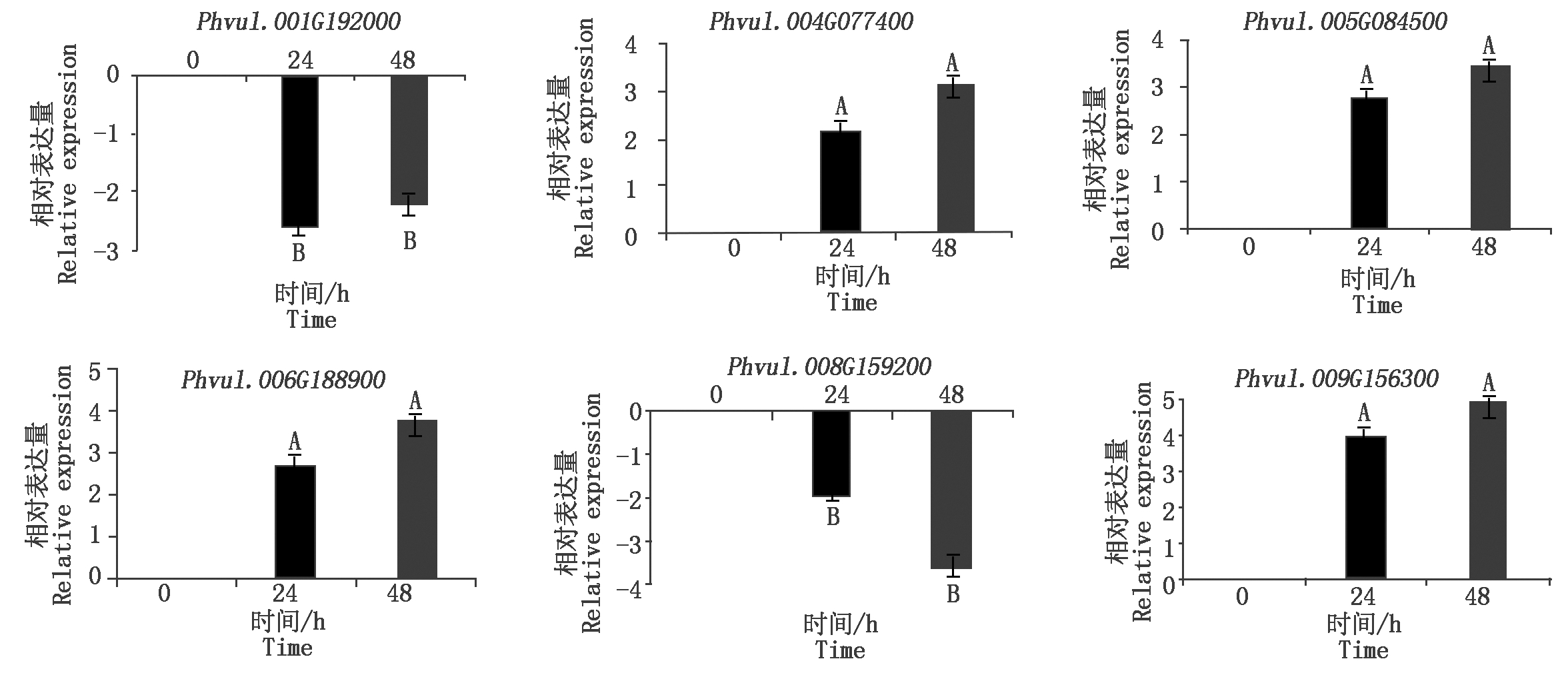
3个生物学重复,以0 h为对照,不同大写字母表示差异极显著(P<0.01)。图7同。
Three biological replicates, with 0 h as the control, different capital letters indicate extremely significant differences (P<0.01).The same as Fig.7.
图6 NaHCO3胁迫下芸豆NAC基因的表达
Fig.6 Expression profile of NAC gene in common bean under NaHCO3 stress
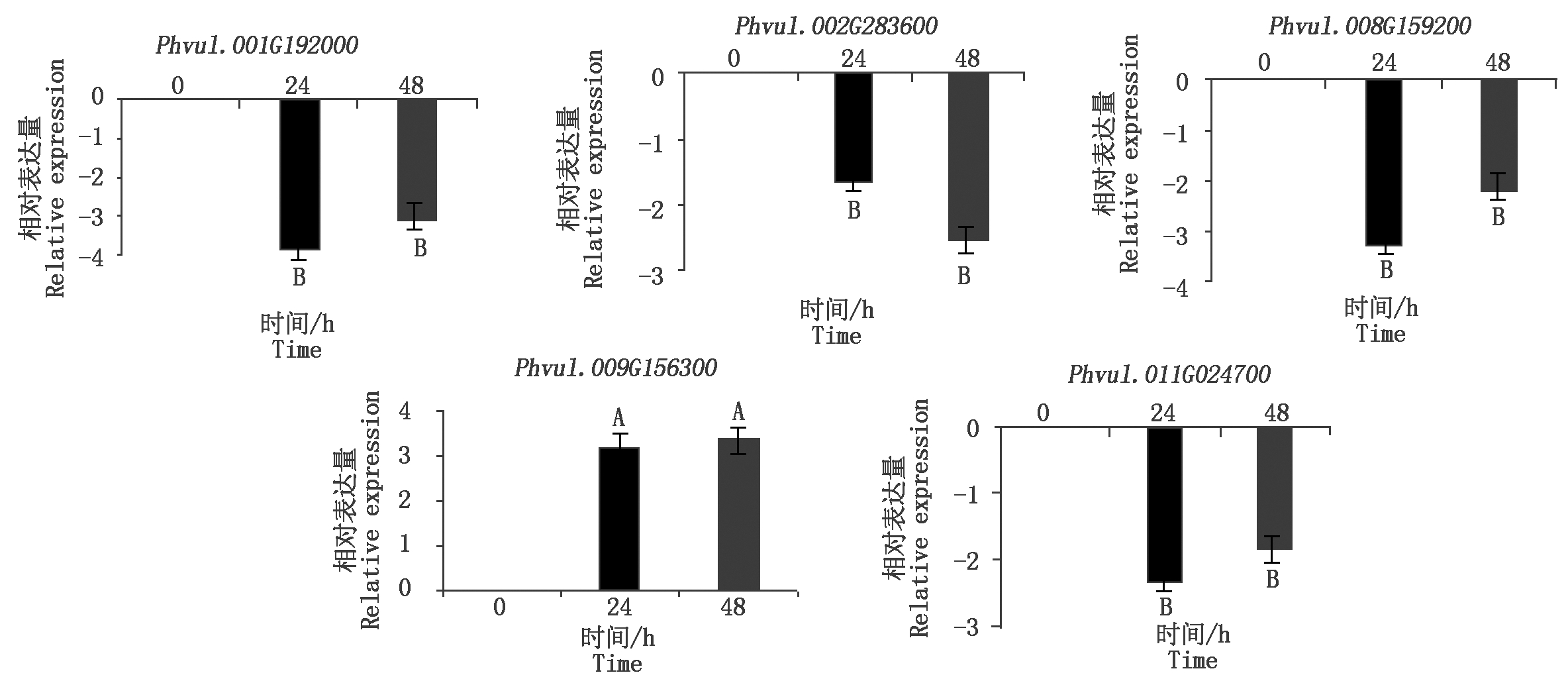
图7 NaCl胁迫下芸豆NAC基因的表达
Fig.7 Expression profile of NAC gene in common bean under NaCl stress

图8 8个芸豆NAC盐碱响应基因重叠韦恩图
Fig.8 Overlapped Venn plots of NAC salt-alkali
response genes in eight common bean
3 讨论与结论
NAC基因广泛的参与植物生长发育和逆境胁迫响应,并且调控着植物生长发育的整个生命进程[26-27],如调控细胞分裂[28]、调节植物的细胞生长[29]、诱导植株抗生素的合成[30]以及植物次生壁的形成等[31-32]。NAC转录因子被证明可以提高拟南芥的抗逆性之后,大量的NAC基因在作物中发挥的功能被陆续的报道出来,例如过表达胁迫诱导的SNAC1基因水稻在四叶期表现出更好的抗旱性和耐盐性[33];小麦中的TaNAC29基因参与盐胁迫的响应,并能增强植株抗氧化酶的活性,减少盐胁迫对植株的损伤[34];SNAC2/OsNAC6转基因水稻在低温下细胞膜更稳定[35]。然而并不是所有NAC转录因子都可以提高作物的抗逆性,如甜瓜的CmNAC14,发现其过表达的转基因拟南芥增加了对盐胁迫的敏感性等[36]。现在,越来越多的NAC转录因子功能被鉴定出来,但在芸豆中,NAC转录因子研究却很有限,本研究通过芸豆叶片组织的转录组数据,鉴定了芸豆8个响应盐碱NAC转录因子,表明这些基因在盐碱反应中可能发挥重要的作用。
本研究通过进化发育分析、基因结构分析及保守域分析可以发现,8个芸豆NAC基因被分为4个亚家族,包含已知的逆境相关NAC基因,其中AT3G15500基因被验证和大豆的抗旱性相关[37],Wu等[38]也提及Phvul.005G084500与干旱有关,而本研究中发现Phvul.005G084500与抗碱性相关,由此猜测a亚族中的基因可能与干旱胁迫及碱胁迫相关,可以发现有的基因不止在一种逆境下有所表达,一个基因可能同时响应多种非生物胁迫,本研究分析也得到3个基因同时响应盐胁迫和碱胁迫。
转录因子通过与DNA上的特异性元件结合,会驱动下游基因的转录。像GmDGAT1A基因通过启动子在转录水平调控油脂代谢相关基因的表达,从而影响大豆种子中油脂的合成与分解[39],冯珊珊等[40]发现了与CBF冷反应通路相关的CRT/DRE元件,证明了TaEXPB12-A/B/D可能在CBF冷反应通路中承担着一定的角色。本研究中一些逆境相关元件在启动子中高频出现,如ABRE、Box 4和MYB等,这表明NAC转录因子在参与芸豆盐碱胁迫信号调控网络中,可能作为相关基因的上游调控因子,结合启动子中相应结合位点从而激活或抑制基因的表达。
磷酸化位点是一种特殊的分子区域,它们对细胞中蛋白质的调节特别重要,它可以激活或停用蛋白质,在细胞信号转导过程中起着重要的作用。有大部分的研究表明,转录因子可以通过蛋白磷酸化来提高植物的抗逆能力,像转录因子的MAPK可以改变细胞定位、蛋白质稳定性、反式激活或抑制活性、DNA结合活性和核小体结构的重塑。MAPK介导的WRKY8的磷酸化激活其DNA结合和转移活性,并且WRKY8诱导下游防御基因[41]。本研究中8个NAC蛋白均有磷酸化位点,但磷酸化位点数目及类型有所不同,说明这8个芸豆NAC蛋白的磷酸化位点都有可能被特定的蛋白激酶磷酸化从而影响植物的抗逆能力。通过对NAC蛋白的二级结构预测,可以发现无规则卷曲结构占据着主导地位,无规则卷曲有明确而稳定的结构,受侧链相互作用的影响很大,它们经常构成酶活性部位和其他蛋白质特异的功能部位。
本研究对各组织表达分析发现,芸豆的NAC基因在芸豆植株各部位均有不同程度的表达。通过qRT-PCR验证分析筛选出的8个芸豆NAC基因在盐碱胁迫下出现了不同程度的上调或下调表达,表明这8个基因在盐碱胁迫中都具有一定的功能。
本研究从基因组范围内对芸豆NAC转录因子进行系统分析,从中鉴定得到了8个芸豆NAC基因,表明这些基因可能在芸豆盐碱胁迫应答中发挥重要作用。利用生物学信息的方法对这些NAC基因的进化关系,启动子分析和组织表达模式以及qRT-PCR等多方面进行了系统分析,发现芸豆NAC出现不同程度的表达,表明其对盐碱有一定的响应。本研究可以提供关于逆境诱导候选基因的有用信息,这些潜在基因发现对于提高作物抗逆性研究及芸豆后续NAC转录因子的研究具有重要意义。
[1] Aida M, Ishida T, Fukaki H, Fujisawa H, Tasaka M. Genes involved in organ separation in Arabidopsis:An analysis of the cup-shaped cotyledon mutant[J].The Plant Cell, 1997, 9(6):841-857. doi:10.1105/tpc.9.6.841.
[2] Souer E, van Houwelingen A, Kloos D, Mol J, Koes R. The no apical meristem gene of Petunia is required for pattern formation in embryos and flowers and is expressed at meristem and primordia boundaries[J].Cell, 1996, 85(2):159-170. doi:10.1016/S0092-8674(00)81093-4.
[3] Liu C, Wang B M, Li Z X, Peng Z H, Zhang J R. TsNAC1 is a key transcription factor in abiotic stress resistance and growth[J].Plant Physiology, 2018, 176(1):742-756. doi:10.1104/pp.17.01089.
[4] Jin C, Li K Q, Xu X Y, Zhang H P, Chen H X, Chen Y H, Hao J, Wang Y, Huang X S, Zhang S L. A novel NAC transcription factor, PbeNAC1, of Pyrus betulifolia confers cold and drought tolerance via interacting with PbeDREBs and activating the expression of stress-responsive genes[J].Frontiers in Plant Science, 2017, 8:1049. doi:10.3389/fpls.2017.01049.
[5] Shim J S, Oh N, Chung P J, Kim Y S, Choi Y D, Kim J K. Overexpression of OsNAC14 improves drought tolerance in rice[J].Frontiers in Plant Science, 2018, 9:310. doi:10.3389/fpls.2018.00310.
[6] Hussain R M, Ali M, Feng X, Li X. The essence of NAC gene family to the cultivation of drought-resistant soybean (Glycine max L. Merr.) cultivars[J].BMC Plant Biology, 2017, 17(1):55. doi:10.1186/s12870-017-1001-y.
[7] So H A, Lee J H. NAC transcription factors from soybean (Glycine max L.) differentially regulated by abiotic stress[J].Journal of Plant Biology, 2019, 62(2):147-160. doi:10.1007/s12374-018-0285-2.
[8] Meng X Q, Li G, Yu J, Cai J, Dong T T, Sun J, Xu T, Li Z Y, Pan S Y, Ma D F, Zhu M K. Isolation, expression analysis, and function evaluation of 12 novel stress-responsive genes of NAC transcription factors in sweetpotato[J].Crop Science, 2018, 58(3):1328-1341. doi:10.2135/cropsci2017.12.0738.
[9] Hoang X L T, Nhi D N H, Thu N B A, Thao N P, Tran L P. Transcription factors and their roles in signal transduction in plants under abiotic stresses[J].Current Genomics, 2017, 18(6):483-497. doi:10.2174/1389202918666170227150057.
[10] Liu W P, Zhao B G, Chao Q, Wang B C, Zhang Q, Zhang C X, Li S F, Jin F X, Yang D G, Li X H. Function analysis of ZmNAC33, a positive regulator in drought stress response in Arabidopsis[J].Plant Physiology and Biochemistry, 2019, 145:174-183. doi:10.1016/j.plaphy.2019.10.038.
[11] Li M, Hu Z, Jiang Q Y, Sun X J, Guo Y, Qi J C, Zhang H. GmNAC15 overexpression in hairy roots enhances salt tolerance in soybean[J].Journal of Integrative Agriculture, 2018, 17(3):530-538. doi:10.1016/S2095-3119(17)61721-0.
[12] Zhang H H, Cui X Y, Guo Y X, Luo C B, Zhang L Y. Picea wilsonii transcription factor NAC2 enhanced plant tolerance to abiotic stress and participated in RFCP1-regulated flowering time[J].Plant Molecular Biology, 2018, 98(6):471-493. doi:10.1007/s11103-018-0792-z.
[13] Ma N N, Zuo Y Q, Liang X Q, Yin B, Wang G D, Meng Q W. The multiple stress-responsive transcription factor SlNAC1 improves the chilling tolerance of tomato[J].Physiologia Plantarum, 2013, 149(4):474-486. doi:10.1111/ppl.12049.
[14] Yang X F, Kim M Y, Ha J, Lee S H. Overexpression of the soybean NAC gene GmNAC109 increases lateral root formation and abiotic stress tolerance in transgenic Arabidopsis plants[J].Frontiers in Plant Science, 2019, 10:1036. doi:10.3389/fpls.2019.01036.
[15] Fan Z Q, Tan X L, Shan W, Kuang J F, Lu W J, Lin H T, Su X G, Lakshmanan P, Zhao M L, Chen J Y. Involvement of BrNAC041 in ABA-GA antagonism in the leaf senescence of Chinese flowering cabbage[J].Postharvest Biology and Technology, 2020, 168:111254. doi:10.1016/j.postharvbio.2020.111254.
[16] Zhang H F, Ma F, Wang X K, Liu S Y, Saeed U H, Hou X M, Zhang Y M, Luo D, Meng Y C, Zhang W, Abid K, Chen R G. Molecular and functional characterization of CaNAC035, an NAC transcription factor from pepper (Capsicum annuum L.)[J].Frontiers in Plant Science, 2020, 11:14. doi:10.3389/fpls.2020.00014.
[17] 徐红云. 拟南芥trihelix转录因子AST1调控植物抗旱、耐盐的机制研究[D].哈尔滨:东北林业大学, 2017.
Xu H Y. The mechanism of Arabidopsis trihelix transcription factor AST1 in drought or salt stress response regulation[D].Harbin:Northeast Forestry University, 2017.
[18] 王臻昱,才华, 柏锡,纪巍,李勇,魏正巍,朱延明.野生大豆GsGST19基因的克隆及其转基因苜蓿的耐盐碱性分析[J].作物学报, 2012, 38(6):971-979. doi:10.3724/SP.J.1006.2012.00971.
Wang Z Y, Cai H, Bai X, Ji W, Li Y, Wei Z W, Zhu Y M. Isolation of GsGST19 from Glycine soja and analysis of saline-alkaline tolerance for transgenic Medicago sativa[J].Acta Agronomica Sinica, 2012, 38(6):971-979.
[19] 郭运娜. MdNAC29基因在苹果干旱和盐胁迫中的作用和机制[D].沈阳:沈阳农业大学, 2018.
Guo Y N. Effect and mechanism of MdNAC29 in apple to salt and drought stress[D].Shenyang:Shenyang Agricultural University, 2018.
[20] Cui M H, Yoo K S, Hyoung S, Nguyen H T K, Kim Y Y, Kim H J, Ok S H, Yoo S D, Shin J S. An Arabidopsis R2R3-MYB transcription factor, AtMYB20, negatively regulates type 2C serine/threonine protein phosphatases to enhance salt tolerance[J].FEBS Letters, 2013, 587(12):1773-1778. doi:10.1016/j.febslet.2013.04.028.
[21] Li H, Gao Y, Xu H, Dai Y, Deng D Q, Chen J M. ZmWRKY33, a WRKY maize transcription factor conferring enhanced salt stress tolerances in Arabidopsis[J].Plant Growth Regulation, 2013, 70(3):207-216. doi:10.1007/s10725-013-9792-9.
[22] 李琳, 于崧, 蒋永超, 张婷婷, 邹春雷, 金珊珊, 郭建华, 梁海芸, 段君君, 于立河. 芸豆苗期耐盐碱性鉴定及品种筛选研究[J].植物生理学报, 2016, 52(1):62-72. doi:10.13592/j.cnki.ppj.2015.0620.
Li L, Yu S, Jiang Y C, Zhang T T, Zou C L, Jin S S, Guo J H, Liang H Y, Duan J J, Yu L H. Identification and screening of different kidney bean cultivars for saline-alkaline tolerance during seedling stage[J].Plant Physiology Journal, 2016, 52(1):62-72.
[23] 张晓艳, 王坤, 王述民. 普通菜豆种质资源遗传多样性研究进展[J].植物遗传资源学报, 2007, 8(3):359-365. doi:10.13430/j.cnki.jpgr.2007.03.026.
Zhang X Y, Wang K, Wang S M. Advances in genetic diversity research on germplasm resources of common bean (Phaseolus vulgaris L.)[J].Journal of Plant Genetic Resources, 2007, 8(3):359-365.
[24] Talaat N B. Effective microorganisms enhance the scavenging capacity of the ascorbate-glutathione cycle in common bean(Phaseolus vulgaris L.)plants grown in salty soils[J].Plant Physiology and Biochemistry, 2014, 80:136-143. doi:10.1016/j.plaphy.2014.03.035.
[25] 高国丽. 普通菜豆PvGF14a/g基因在盐胁迫下的功能分析[D].长春:吉林大学, 2018.
Gao G L. Functional analysis of common bean gene PvGF14a/g under salt stress[D].Changchun:Jilin University, 2018.
[26] Zhang L N, Zhang L C, Xia C, Zhao G Y, Jia J Z, Kong X Y. The novel wheat transcription factor TaNAC47 enhances multiple abiotic stress tolerances in transgenic plants[J].Frontiers in Plant Science, 2015, 6:1174. doi:10.3389/fpls.2015.01174.
[27] 张慧珍, 白雪芹, 曾幼玲. 植物NAC转录因子的生物学功能[J].植物生理学报, 2019, 55(7):915-924. doi:10.13592/j.cnki.ppj.2019.0107.
Zhang H Z, Bai X Q, Zeng Y L. Biological functions of plant NAC transcription factors[J].Plant Physiology Journal, 2019, 55(7):915-924.
[28] Kim S Y, Kim S G, Kim Y S, Seo P J, Bae M, Yoon H K, Park C M. Exploring membrane-associated NAC transcription factors in Arabidopsis:Implications for membrane biology in genome regulation[J].Nucleic Acids Research, 2007, 35(1):203-213. doi:10.1093/nar/gkl1068.
[29] Kato H, Motomura T, Komeda Y, Saito T, Kato A. Overexpression of the NAC transcription factor family gene ANAC036 results in a dwarf phenotype in Arabidopsis thaliana[J].Journal of Plant Physiology, 2010, 167(7):571-577. doi:10.1016/j.jplph.2009.11.004.
[30] Saga H, Ogawa T, Kai K, Suzuki H, Ogata Y, Sakurai N, Shibata D, Ohta D. Identification and characterization of ANAC042, a transcription factor family gene involved in the regulation of camalexin biosynthesis in Arabidopsis[J].Molecular Plant Microbe Interactions, 2012, 25(5):684-696. doi:10.1094/MPMI-09-11-0244.
[31] Mitsuda N, Iwase A, Yamamoto H, Yoshida M, Seki M, Shinozaki K, Ohme-Takagi M. NAC transcription factors, NST1 and NST3, are key regulators of the formation of secondary walls in woody tissues of Arabidopsis[J].The Plant Cell, 2007, 19(1):270-280. doi:10.1105/tpc.106.047043.
[32] Zhao Q, Gallego-Giraldo L, Wang H Z, Zeng Y N, Ding S Y, Chen F, Dixon R A. An NAC transcription factor orchestrates multiple features of cell wall development in Medicago truncatula[J].The Plant Journal, 2010, 63(1):100-114. doi:10.1111/j.1365-313X.2010.04223.x.
[33] Hu H H, Dai M Q, Yao J L, Xiao B Z, Li X H, Zhang Q F, Xiong L Z. Overexpressing a NAM, ATAF, and CUC (NAC) transcription factor enhances drought resistance and salt tolerance in rice[J].PNAS,2006,103(35):12987-12992.doi:10.1073/pnas.0604882103.
[34] Xu Z Y, Gongbuzhaxi, Wang C Y, Xue F, Zhang H, Ji W Q. Wheat NAC transcription factor TaNAC29 is involved in response to salt stress[J].Plant Physiology and Biochemistry, 2015, 96:356-363. doi:10.1016/j.plaphy.2015.08.013.
[35] Hu H H, You J, Fang Y J, Zhu X Y, Qi Z Y, Xiong L Z. Characterization of transcription factor gene SNAC2 conferring cold and salt tolerance in rice[J].Plant Molecular Biology, 2008, 67(1/2):169-181. doi:10.1007/s11103-008-9309-5.
[36] Wei S W, Gao L W, Zhang Y D, Zhang F R, Yang X, Huang D F. Genome-wide investigation of the NAC transcription factor family in melon (Cucumis melo L.) and their expression analysis under salt stress[J].Plant Cell Reports, 2016, 35(9):1827-1839. doi:10.1007/s00299-016-1997-8.
[37] Chai C L, Wang Y Q, Joshi T, Valliyodan B, Prince S, Michel L, Xu D, Nguyen H T. Soybean transcription factor ORFeome associated with drought resistance:A valuable resource to accelerate research on abiotic stress resistance[J].BMC Genomics, 2015, 16:596. doi:10.1186/s12864-015-1743-6.
[38] Wu J, Wang L F, Wang S M. Comprehensive analysis and discovery of drought-related NAC transcription factors in common bean[J].BMC Plant Biology, 2016, 16(1):193. doi:10.1186/s12870-016-0882-5.
[39] 晁毛妮, 胡喜贵, 张晋玉, 王润豪, 温青玉, 孙新凯, 黄中文. 大豆二酰甘油酰基转移酶基因GmDGAT1A启动子的克隆与功能分析[J].华北农学报, 2020, 35(4):27-34.doi:10.7668/hbnxb.20190821.
Chao M N, Hu X G, Zhang J Y, Wang R H, Wen Q Y, Sun X K, Huang Z W. Cloning and functional analysis of promoter of diacylglycerol acyltransferase gene GmDGAT1A in soybean[J].Acta Agriculturae Boreali-Sinica, 2020, 35(4):27-34.
[40] 冯珊珊, 徐永清, 赵梓颐, 李凤兰, 胡宝忠. 寒地冬小麦膨胀素基因TaEXPB12同源基因的克隆及功能分析[J].华北农学报, 2020, 35(6):74-80.doi:10.7668/hbnxb.20191210.
Feng S S, Xu Y Q, Zhao Z Y, Li F L, Hu B Z. Cloning and function verification of TaEXPB12 homologous genes in frigid region winter wheat[J].Acta Agriculturae Boreali-Sinica, 2020, 35(6):74-80.
[41] Ishihama N, Yamada R, Yoshioka M, Katou S, Yoshioka H. Phosphorylation of the Nicotiana benthamiana WRKY8 transcription factor by MAPK functions in the defense response[J].The Plant Cell, 2011, 23(3):1153-1170. doi:10.1105/tpc.110.081794.