由于长期以来采用密植和高水肥的栽培方式,小麦白粉病在世界范围内日趋严重。该病害由布氏白粉菌 (Blumeria graminis f.sp.tritici,Bgt)引起,常年流行于我国各小麦主产区,严重影响小麦的产量和品质。发掘抗病基因、选育抗病品种是缓解小麦白粉病最经济有效的措施[1]。
目前,国内外已从小麦基因组中鉴定并正式命名了68个抗白粉病位点 (Pm1~Pm68)[2],并且有12个位点上的Pm基因已被克隆。其中,9个基因(Pm1[3]、Pm2[4]、Pm3[5]、Pm5[6]、Pm8[7]、Pm17[8]、Pm21[9]、Pm41[10]和Pm60 [11])均为核苷酸结合位点(Nucleotide binding site,NBS)基因。NBS基因广泛存在于植物中,是数量最多的一类抗病基因,其编码蛋白通过结合三磷酸腺苷或三磷酸鸟苷来参与抗病信号传导,进而引发植株对病原菌的抗病反应[12]。水稻[13]、玉米[14]、大豆[15]等作物中的NBS序列已被分离。
2015年,Bouktila等[16]利用普通小麦品种中国春的第一代测序数据,首次分离了436条小麦NBS序列,并初步分析了家族保守域结构。随后,乔麟轶等[17]利用中国春第二代测序数据,从全基因组范围内分离了2 406条小麦NBS序列,分析了序列邻近的SSR位点,并在2AL染色体上筛选出3个可能与抗白粉病基因Pm4a连锁的NBS-SSR标记。上述研究成果为小麦基因组中NBS家族的分离鉴定奠定了基础,但尚未从转录组水平研究NBS对白粉菌的响应情况。
本研究利用小麦受白粉菌胁迫后的转录组数据[18]分析TaNBS家族的表达水平变化,并选择差异表达显著基因进行qRT-PCR和病毒诱导基因沉默(Virus-induced gene silencing,VIGS)验证,以期发掘抗白粉病相关TaNBS基因,为小麦抗病育种提供分子依据。
1 材料和方法
1.1 试验材料和菌株
小麦种质CH7124由作物遗传与分子改良山西省重点实验室提供。该种质由山西省农业科学院畅志坚研究员选育,对白粉菌生理小种E09表现为免疫[19]。白粉菌种E09由中国农业科学院段霞瑜研究员惠赠,由本实验室繁殖、保存。
1.2 试验方法
1.2.1 转录组数据分析 以注册号PRJNA243835检索SRA数据库(http://www.ncbi.nlm. nih. gov/sra/),下载小麦接种白粉菌后0,24,48,72 h的转录组数据。利用Tophat 2.0软件将reads比对到小麦参考基因组上(中国春TGACv1版本,http://tgac-browser.tgac.ac.uk/);之后用cufflinks组装转录本,并用edger获得转录本的FPKM(Expression fragment per kilobase of exon model per million mapped reads)值。
利用小麦NBS家族序列[17]从上述转录本中检索并筛选受白粉菌胁迫后表达量差异显著的TaNBS,筛选标准为log2|FPKMt/FPKMt0|≥1。所得结果用MeV软件输出。
1.2.2 白粉菌接种与鉴定 将CH7124播于育苗盘内,种子萌发后移入人工培养箱,设置22 ℃光照16 h/16 ℃黑暗8 h。待植株生长至三叶期,用扫拂法充分接种白粉菌种E09的分生孢子。并在接菌的0,24,48,72 h剪取植株叶片,于-80 ℃保存。
1.2.3 qRT-PCR 用RNA提取试剂盒 (DP420,天根生化,北京 )提取样品的总RNA,利用反转录试剂盒 (RR036A,宝生物,北京 )反转录cDNA。在罗氏Light Cycler® 96-PCR仪上进行qRT-PCR,所用引物序列列于表1。使用的酶为SYBR Premix Ex Taq Ⅱ(宝生物,北京 ),每个反应重复3次,所得结果采用Fold-change法进行分析。
1.2.4 大麦条纹花叶病毒 (BSMV)接种液制备 涉及的载体均购自北京信捷创辉生物科技有限公司。使用表1中的引物扩增目的基因片段,经测序验证后用无缝克隆试剂盒 (B632219,生工生物,上海 )将扩增产物连接到载体BSMV-γ上,将其与BSMV-α、BSMV-β线性化处理后用体外转录试剂盒(AM1340,Thermo Fisher,美国)进行体外转录。之后将上述BSMV-α、BSMV-β+BSMV-γ-目的片段、BSMV-γ的转录物各取10 μL混合,加入90 μL DEPC水和120 μL GKP Buffer,制成240 μL接种液。
表1 使用的qRT-PCR引物
Tab.1 Primers for qRT-PCR used

引物名称Primer name前引物序列(5′-3′) Forward primer sequence( 5′-3′) 后引物序列(5′-3′) Reverse primer sequence( 5′-3′) 长度/bpSize 退火温度/℃AnnealingtemperatureTa1bsLoc018729GAATCTTTGGCTCAGAGAATTCCTGGCTTTTGTTCAAGTCAGAACCAAT18660Ta1dsLoc003044GGTTTGGTGGATTGGGAAAGACCTGTAAGAAACTTGGGAGATCACAT14860Ta2asLoc021716CTGACGGTGTTATCCAGTCTTCAGTATATTATCACCATCGTCCAT13158Ta2dlLoc029209GGCTGATGTTAAAGTTAATGGTCTCTTTAGTGCTTTGGCAATCTCT13758Ta3asLoc007663CGTGATGTTCAGTACATCAATGATGGGCAAAGTCATCAATGCAATC16058Ta3dlLoc008638GACAAGAAGAAGTATTCATGCATTGGCAAGGAATTCATTATTGCTCT12356Ta4dsLoc004039CTGCTCGAAAGTTTGCAATTATGGTTGTTAGACCTAATGTCTGTTGTGAAACT14960Ta6alLoc012531CAAGGAGATCAAGTCTCTCACTAATGATGCTTTGCATGAAGTCATCGAT16660Ta7dlLoc000139CAGCTAGCAGCAGATGGTGTGTCAGCATCAGCCAAGAAGTT10560Ta7dlLoc004854GTACGATGTGGAAGACTGCATGCCTTGAGCTGCTCGATCT14258TaGAPDHTTAGACTTGCGAAGCCAGCAAAATGCCCTTGAGGTTTCCC20458
1.2.5 小麦VIGS体系建立 当CH7124幼苗第2片叶展开时,滴加10 μL接种液并摩擦接种,之后保持湿润和黑暗24 h,移入正常生长环境,待第3片叶产生病毒斑表型,表明接种成功。当VIGS植株第4片叶展开时,剪取有病毒斑的叶片,一部分置于含有苯并咪唑的MS培养基上接种白粉菌E09,7 d后采用0~4级标准调查反应型[20],每个目的基因调查3株VIGS植株;另一部分样本提取总RNA,按照1.2.3描述的方法鉴定目的基因的表达水平。
1.2.6 基因序列分析 利用基因的表达序列比对小麦参考基因组(中国春IWGSCv1.0版本,http://wheat-urgi.versailles.inra.fr/)得到基因组序列,进而获得基因结构特征。将基因起始密码子前2 000 bp基因组序列递交PlantCARE数据库(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/ )进行启动子元件分析。
2 结果与分析
2.1 转录组数据组装结果
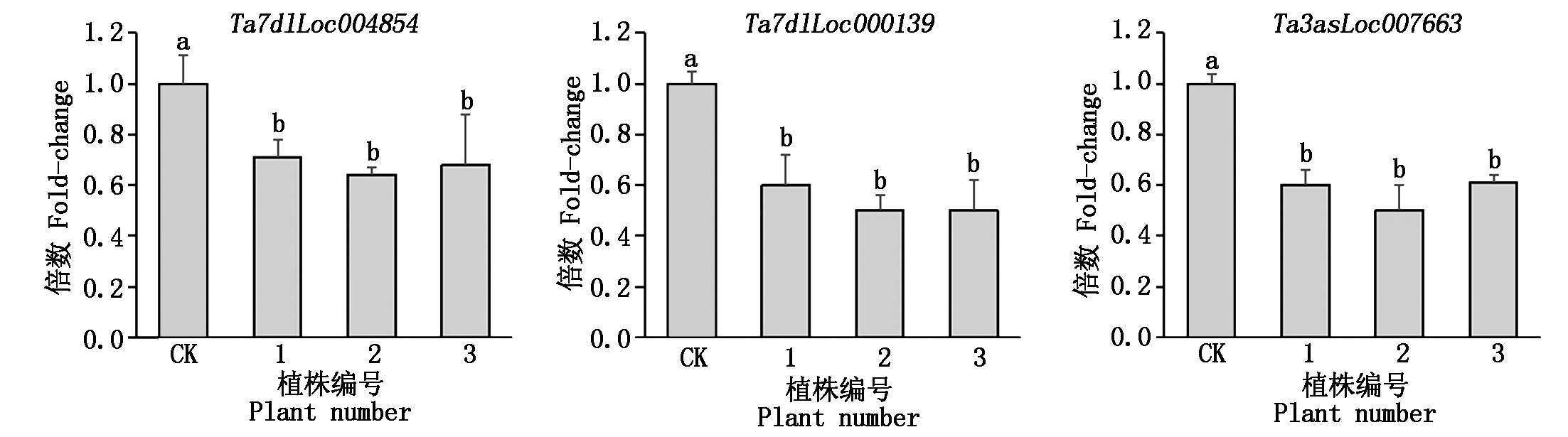
通过组装转录组数据,获得小麦在接种白粉菌4个时间点后的基因表达数据;根据FPKM值,将基因分为不表达 (FPKM=0 )、低表达 (FPKM≤2)、中表达(2 图1 小麦接种白粉菌后的转录组数据组装结果 利用2 406条小麦NBS家族序列检索组装的转录组数据,共获得1 283条分布于21条染色体上的、具有表达数据的TaNBS(FPKM>0 ),占比53.3%;其中,在小麦4A和7D染色体上分布的TaNBS最多,各有101条,而4B和4D染色体分布较少,分别为16,15条 (图2)。 图2 具有表达数据的TaNBS在染色体上的分布 基于差异显著性分析,进一步筛选出395条接种Bgt后表达水平变化显著的TaNBS基因,这些基因可大致分为5类,第1类在接种后24 h内表达水平降低,之后持续升高;第2类在接种48 h内表达水平升高,之后下降;第3类在3个时间点表现出升-降-升的变化趋势;第4类在接种48 h内表达水平降低,之后上升;第5类在接种后持续上升(图3)。 图3 小麦接种白粉菌E09后表达水平 从上述第5类基因中选择10个表达水平变化差异最为显著的TaNBS进行qRT-PCR验证,有3个基因在接种白粉菌24,48,72 h均保持上调,与转录组数据一致。其中,位于7D染色体的Ta7dlLoc004854在接种24,72 h显著上调(P<0.05),48 h显著上调(P<0.05);同样位于7D染色体的Ta7dlLoc000139在接种24,48,72 h均保持显著上调(P<0.05),但上调倍数呈下降趋势;而位于3A染色体的Ta3asLoc007663在接种24 h显著上调(P<0.05),48,72 h显著上调(P<0.05)(图4)。 其余基因的表达水平变化与转录组不一致。Ta4dsLoc004039在接种24 h 无显著变化,在48 h呈极显著(P<0.01)上调,随后在72 h显著下调(P<0.05);Ta1dsLoc003044仅在24 h显著上调(P<0.05);Ta2asLoc021716仅在72 h显著上调(P<0.05);Ta3dlLoc008638、Ta1bsLoc018729、Ta2dlLoc029209 和Ta6alLoc012531的表达水平无显著变化(图4)。 使用倍数法分析表达数据,设0 h为对照;不同小写字母代表显著差异,P<0.05。图5同。 图4 10个TaNBS在小麦抗白粉病种质CH7124中受白粉菌侵染后的表达水平 利用BSMV侵染CH7124植株,分别诱导Ta7dlLoc004854、Ta7dlLoc000139和Ta3asLoc007663的表达水平下调,获得VIGS植株;qRT-PCR结果显示,VIGS植株中Ta7dlLoc004854、Ta7dlLoc000139和Ta3asLoc007663的表达水平与对照植株相比显著下调(图5)。剪取VIGS植株第4片叶进行离体接种白粉菌E09,结果如表2所示,VIGS植株BSMV-Ta7dlLoc004854和BSMV-Ta3asLoc007663对E09表现为免疫(IT=0),与未侵染BSMV的CK植株表型一致;而VIGS植株BSMV-Ta7dlLoc000139的叶片上出现零星孢子,其中,2株表现为高抗(IT=1),1株表现为中抗(IT=2)。推测Ta7dlLoc000139表达水平的降低引起了植株抗性减弱(图5)。 表2 VIGS植株离体接种白粉菌E09的反应型 植株Plants反应型 Infection type对照 CK第1株 Plant 1第2株 Plant 2第3株 Plant 3BSMV-Ta7dlLoc0048540000BSMV-Ta7dlLoc0001390112BSMV-Ta3asLoc0076630000 序列分析表明,Ta7dlLoc000139全长4 042 bp,包含3个外显子和2个内含子,无可变剪切;在其起始密码子前2 000 bp区域内包含大量CGCG-Box、GATA-Box和CAAT-Box等调控元件,显示该基因的表达可能受到调控(图6)。 CK.未侵染BMSV的植株;1~3.侵染BMSV的植株。 图5 目的基因沉默后VIGS植株中对白粉菌E09的感染型 图6 Ta7dlLoc000139的基因结构和启动子区调控元件预测 鉴定抗病基因用于品种选育可有效缓解小麦白粉病病害。一些抗白粉病基因如Pm2、Pm8和Pm21,已成功应用于小麦生产。然而,由于小麦白粉菌种致病型多且变异快,大多数抗病品种在推广应用5 a左右会逐渐丧失抗性[21]。因此,广泛、持续地发掘小麦抗白粉病基因尤为重要。NBS基因在植物抵御病原菌入侵过程中发挥重要作用。目前,小麦全基因组中的NBS序列已被分离[16-17],但有关NBS家族基因受白粉菌侵染后转录水平变化的研究尚未见报道。本研究从小麦转录组数据中分离了1 283条具有表达水平的NBS序列,约有46.7%的NBS基因未表达,可能是由于其表达具有时期或组织特异性,或者受其他病原菌诱导,或者一些基因已经是失去功能的假基因,显示了多倍体物种中基因家族功能的复杂性。 由于已克隆的9个NBS类抗白粉病基因在受白粉菌侵染后均持续上调[3-11],本研究从转录组数据中选择受侵染24,48,72 h后表达水平均持续上调并且差异最大的10个TaNBS进行qRT-PCR验证,最终获得3个受白粉菌诱导的TaNBS。其中,Ta3asLoc007663位于小麦3A染色体,与抗白粉病基因Pm44[22]均位于短臂;而Ta7dlLoc004854和Ta7dlLoc000139所在的7D染色体尚无正式命名的Pm基因,目前只有TaRPP13-3被报道[23]。TaRPP13-3属于NBS家族,同样对白粉菌种E09表现为抗病,但是该基因位于7D短臂18 Mb的基因组位置,而Ta7dlLoc004854和Ta7dlLoc000139分别位于长臂717,632 Mb位置,不是同一基因。因此,Ta7dlLoc004854和Ta7dlLoc000139可能是2个新的白粉菌响应基因位点。 本研究利用VIGS技术下调Ta7dlLoc000139后,植株抗病性减弱,表明Ta7dlLoc000139与抗病性相关,可能是一个抗白粉病基因。该基因启动子区包含大量的调控元件,推测其表达水平会受到转录因子或者上游基因的调控。后续研究将利用遗传转化等技术对Ta7dlLoc000139进行功能验证,如最终证实其为抗白粉病基因,将为小麦抗病分子育种提供新的抗源。 本研究从小麦转录组数据中分离了395个差异表达TaNBS基因,利用qRT-PCR证实3个TaNBS在小麦抗病材料CH7124中受Bgt胁迫后持续上调,其中,Ta7dlLoc000139经VIGS下调后,植株抗性从免疫(IT=0)变为高抗(IT=1)或中抗(IT=2),推测Ta7dlLoc000139是1个新的抗白粉病基因。 [1] Line R F,Chen X M.Success in breeding for and managing durable resistance to wheat rusts[J].Plant Disease,1995,79(12):1254-1255.doi:10.1094/pd-79-1251. [2] He H G,Liu R K,Ma P T,Du H N,Zhang H H,Wu Q H,Yang L J,Gong S J,Liu T L,Huo N X,Gu Y Q,Zhu S Y.Characterization of Pm68,a new powdery mildew resistance gene on chromosome 2BS of Greek durum wheat TRI 1796[J].Theoretical and Applied Genetics,2021,134(1):53-62.doi:10.1007/s00122-020-03681-2. [3] Hewitt T,Müller M C,Moln [4] Sínchez-Martín J,Steuernagel B,Ghosh S,Herren G,Hurni S,Adamski N,Vr [5] Brunner S,Hurni S,Streckeisen P,Mayr G,Albrecht M,Yahiaoui N,Keller B.Intragenic allele pyramiding combines different specificities of wheat Pm3 resistance alleles[J].The Plant Journal,2010,64(3):433-445.doi:10.1111/j.1365-313X.2010.04342.x. [6] Xie J Z,Guo G H,Wang Y,Hu T Z,Wang L L,Li J T,et al.A rare single nucleotide variant in Pm5e confers powdery mildew resistance in common wheat[J].The New Phytologist,2020,228(3):1011-1026.doi:10.1111/nph.16762. [7] Hurni S,Brunner S,Buchmann G,Herren G,Jordan T,Krukowski P,Wicker T,Yahiaoui N,Mago R,Keller B.Rye Pm8 and wheat Pm3 are orthologous genes and show evolutionary conservation of resistance function against powdery mildew[J].The Plant Journal,2013,76(6):957-969.doi:10.1111/tpj.12345. [8] Singh S P,Hurni S,Ruinelli M,Brunner S,Sanchez-Martin J,Krukowski P,Peditto D,Buchmann G,Zbinden H,Keller B.Evolutionary divergence of the rye Pm17 and Pm8 resistance genes reveals ancient diversity[J].Plant Molecular Biology,2018,98(3):249-260.doi:10.1007/s11103-018-0780-3. [9] He H G,Zhu S Y,Zhao R H,Jiang Z N,Ji Y Y,Ji J,Qiu D,Li H J,Bie T D.Pm21,encoding a typical CC-NBS-LRR protein,confers broad-spectrum resistance to wheat powdery mildew disease[J].Molecular Plant,2018,11(6):879-882.doi:10.1016/j.molp.2018.03.004. [10] Li M M,Dong L L,Li B B,Wang Z Z,Xie J Z,Qiu D,et al.A CNL protein in wild emmer wheat confers powdery mildew resistance[J].The New Phytologist,2020,228(3):1027-1037.doi:10.1111/nph.16761. [11] Zou S H,Wang H,Li Y W,Kong Z S,Tang D Z.The NB-LRR gene Pm60 confers powdery mildew resistance in wheat[J].The New Phytologist,2018,218(1):298-309.doi:10.1111/nph.14964. [12] Bourne H R,Sanders D A,McCormick F.The GTPase superfamily:Conserved structure and molecular mechanism[J].Nature,1991,349(6305):117-127.doi:10.1038/349117a0. [13] Monosi B,Wisser R J,Pennill L,Hulbert S H.Full-genome analysis of resistance gene homologues in rice[J].Theoretical and Applied Genetics,2004,109(7):1434-1447.doi:10.1007/s00122-004-1758-x. [14] 汪结明,江海洋,赵阳,项艳,朱苏文,程备久.玉米自交系B73全基因组NBS类型抗病基因分析[J].作物学报,2009,35(3):566-570.doi:10.3724/SP.J.1006.2009.00566. Wang J M,Jiang H Y,Zhao Y,Xiang Y,Zhu S W,Cheng B J.Genome-wide analysis of NBS-encoding disease resistance genes in maize inbred line B73[J].Acta Agronomica Sinica,2009,35(3):566-570. [15] Wang B J,Wang Y J,Wang Q,Luo G Z,Zhang Z G,He C Y,He S J,Zhang J S,Gai J Y,Chen S Y.Characterization of an NBS-LRR resistance gene homologue from soybean[J].Journal of Plant Physiology,2004,161(7):815-822.doi:10.1016/j.jplph.2004.01.007. [16] Bouktila D,Khalfallah Y,Habachi-Houimli Y,Mezghani-Khemakhem M,Makni M,Makni H.Full-genome identification and characterization of NBS-encoding disease resistance genes in wheat[J].Molecular Genetics and Genomics,2015,290(1):257-271.doi:10.1007/s00438-014-0909-2. [17] 乔麟轶,常建忠,郭慧娟,高建刚,郑军,畅志坚.小麦全基因组NBS类R基因分析及2AL染色体NBS-SSR特异标记开发[J].作物学报,2016,42(6):795-802.doi:10.3724/SP.J.1006.2016.00795. Qiao L Y,Chang J Z,Guo H J,Gao J G,Zheng J,Chang Z J.Genome-wide analysis of TaNBS resistance genes and development of chromosome 2AL-specific NBS-SSR markers in wheat[J].Acta Agronomica Sinica,2016,42(6):795-802. [18] Zhang H,Yang Y Z,Wang C Y,Liu M,Li H,Fu Y,Wang Y J,Nie Y B,Liu X L,Ji W Q.Large-scale transcriptome comparison reveals distinct gene activations in wheat responding to stripe rust and powdery mildew[J].BMC Genomics,2014,15(1):898.doi:10.1186/1471-2164-15-898. [19] 李欣,张晓军,张丛卓,詹海仙,杨足君,畅志坚.兼抗白粉、条锈病小偃麦渗入系CH7124抗性遗传及细胞学鉴定[J].植物遗传资源学报,2012,13(4):577-582,588.doi:10.3969/j.issn.1672-1810.2012.04.011. Li X,Zhang X J,Zhang C Z,Zhan H X,Yang Z J,Chang Z J.Cytological characterization and resistance inheritance in alien introgression CH7124 from Thinopyrum intermedium[J].Journal of Plant Genetic Resources,2012,13(4):577-582,588. [20] 盛宝钦.用反应型记载小麦苗期白粉病[J].植物保护,1988,14(1):49. Sheng B Q.Using infection type records the wheat powdery mildew at seedling stage[J].Plant Protection,1988,14(1):49. [21] Huang X Q,Hsam S L K,Zeller F J.Identification of powdery mildew resistance genes in common wheat(Triticum aestivum L.em Thell.).IX.Cultivars,land races and breeding lines grown in China[J].Plant Breeding,1997,116(3):233-238.doi:10.1111/j.1439-0523.1997.tb00988.x. [22] Alam M A,Xue F,Wang C Y,Ji W Q.Powdery mildew resistance genes in wheat:identification and genetic analysis[J].Journal of Molecular Biology Research,2011,1(1):1-20.doi:10.5539/jmbr.v1n1p20. [23] Liu X Y,Zhang C Q,Zhang L L,Huang J G,Dang C,Xie C J,Wang Z Y.TaRPP13-3,a CC-NBS-LRR-like gene located on chr 7D,promotes disease resistance to wheat powdery mildew in Brock[J].Journal of Phytopathology,2020,168(11/12):688-699.doi:10.1111/jph.12949.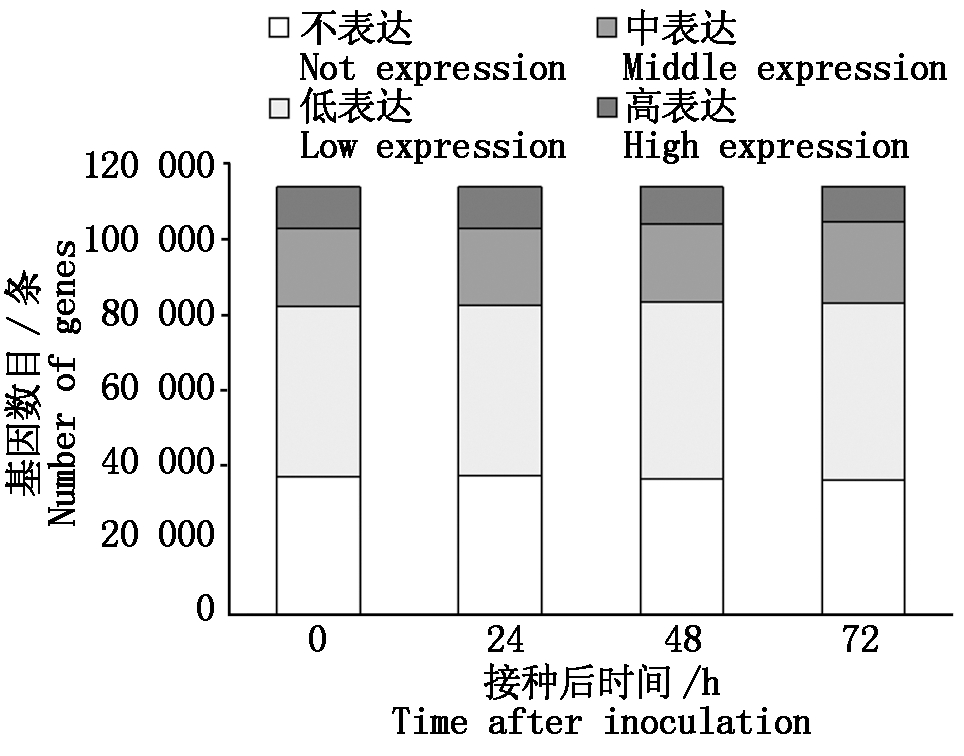
Fig.1 The assembly of transcriptome data for
wheat after inoculation with Bgt2.2 NBS序列的分离
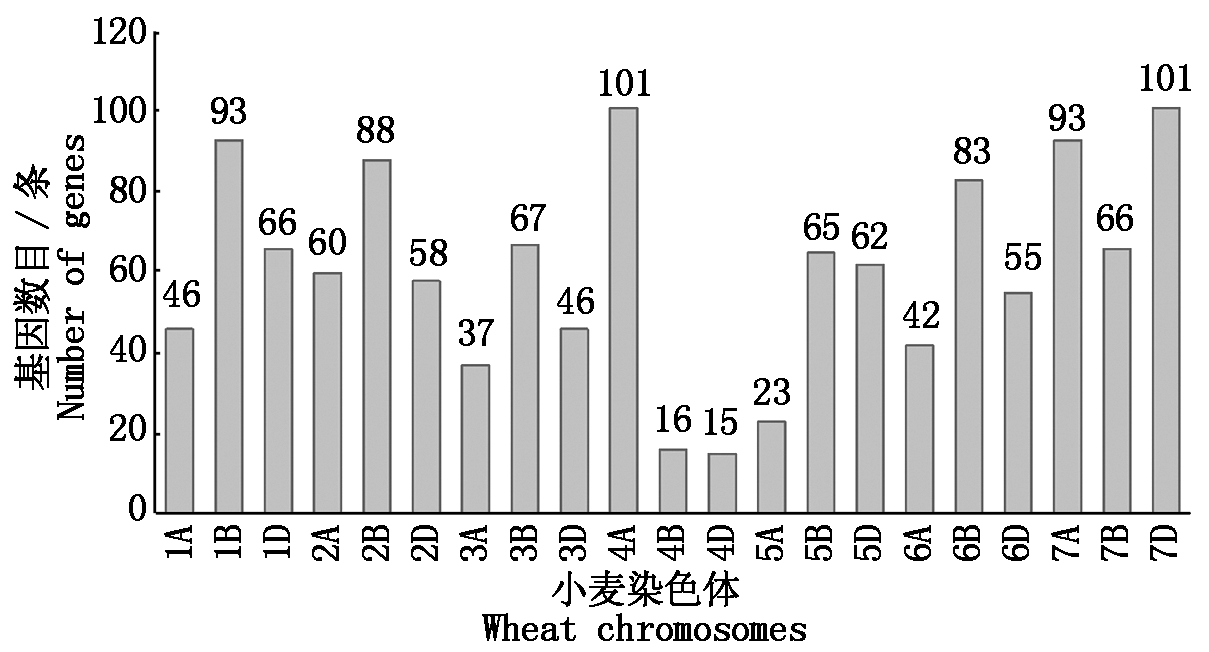
Fig.2 Distribution of TaNBSs with expression
data on wheat chromosomes2.3 TaNBS对白粉菌的响应
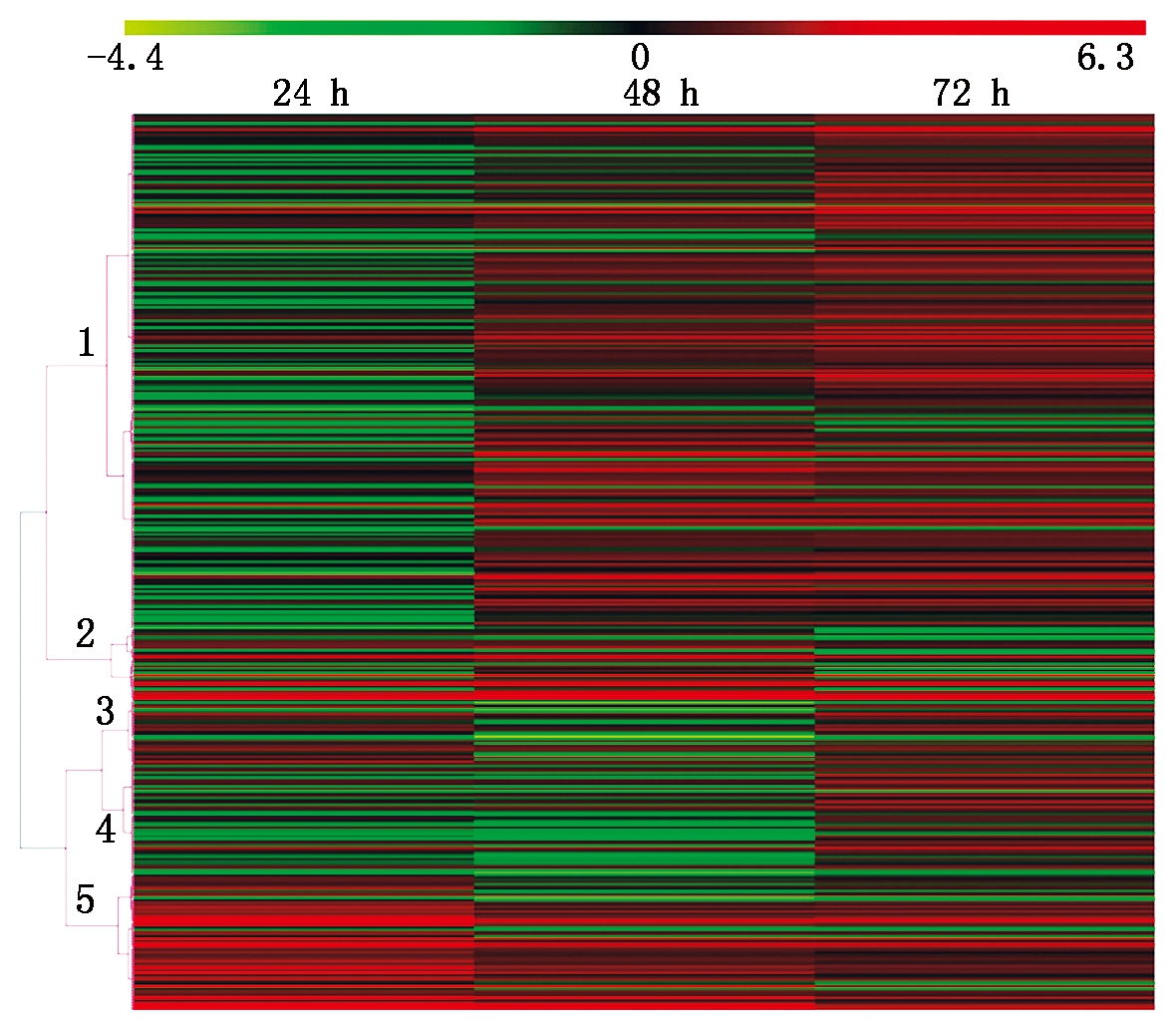
变化差异显著的TaNBS
Fig.3 TaNBSs with significant changes in
expression levels after inoculation with Bgt2.4 TaNBS的qRT-PCR验证

The expression data are processed by the Fold-change method setting 0 h as control;
different lowercase letters represent significant differences,P<0.05.The same as Fig.5.
Fig.4 Expression levels of 10 chosen TaNBSs in wheat powdery mildew resistant germplasm CH7124 after inoculation with Bgt2.5 TaNBS的VIGS验证
Tab.2 The infection type of VIGS plants inoculated with Bgt race E09 in vitro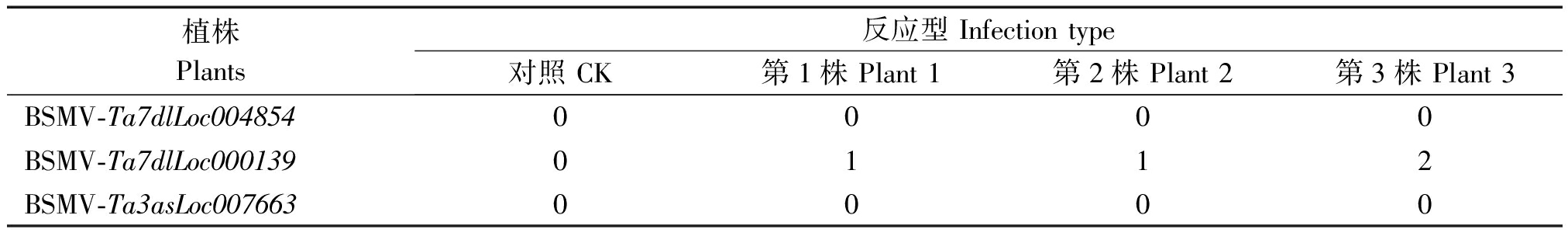
2.6 Ta7dlLoc000139序列特征
CK and number 1 to 3 refer to plants not inoculated and inoculated with BMSV,respectively.
Fig.5 Infective type of the plants against Bgt race E09 after silencing the target gene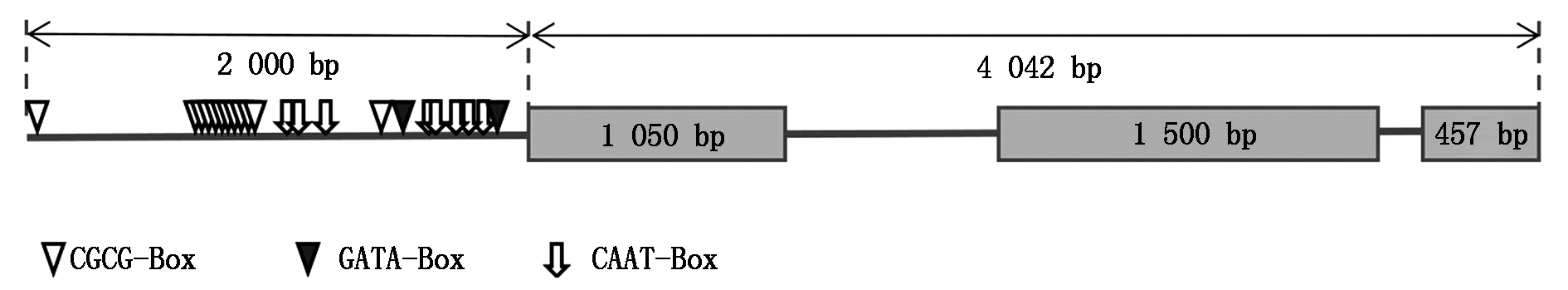
Fig.6 Gene structure and prediction of regulatory elements in promoter region of Ta7dlLoc0001393 结论与讨论
 r I,Mascher M,Holušov
r I,Mascher M,Holušov K,
K, imkov
imkov H,Kunz L,Zhang J P,Li J B,Bhatt D,Sharma R,Schudel S,Yu G T,Steuernagel B,Periyannan S,Wulff B,Ayliffe M,McIntosh R,Keller B,Lagudah E,Zhang P.A highly differentiated region of wheat chromosome 7AL encodes a Pm1 a immune receptor that recognizes its corresponding AvrPm1 a effector from Blumeria graminis[J].The New Phytologist,2021,229(5):2812-2826.doi:10.1111/nph.17075.
H,Kunz L,Zhang J P,Li J B,Bhatt D,Sharma R,Schudel S,Yu G T,Steuernagel B,Periyannan S,Wulff B,Ayliffe M,McIntosh R,Keller B,Lagudah E,Zhang P.A highly differentiated region of wheat chromosome 7AL encodes a Pm1 a immune receptor that recognizes its corresponding AvrPm1 a effector from Blumeria graminis[J].The New Phytologist,2021,229(5):2812-2826.doi:10.1111/nph.17075. na J,Kubal
na J,Kubal kov
kov M,Krattinger S G,Wicker T,Doležel J,Keller B,Wulff B B H.Rapid gene isolation in barley and wheat by mutant chromosome sequencing[J].Genome Biology,2016,17(1): 221.doi:10.1186/s13059-016-1082-1.
M,Krattinger S G,Wicker T,Doležel J,Keller B,Wulff B B H.Rapid gene isolation in barley and wheat by mutant chromosome sequencing[J].Genome Biology,2016,17(1): 221.doi:10.1186/s13059-016-1082-1.