作物种类繁多,生长环境复杂多样,包括盐碱、干旱、高温或低温等。全世界约有7%的土地面积受盐碱化影响,我国土壤盐碱化现象日趋严重,盐碱地面积约9.91×107 hm2,农业生产中不合理的施肥灌溉措施加剧了土壤次生盐渍化进程,高盐使农作物产量大幅降低,甚至造成绝收,土壤盐渍化成为制约农业发展的巨大环境压力,每年造成数十亿的农业经济损失[1]。花生具有耐瘠、固氮、培肥地力和中度耐盐碱等特性,可作为盐碱土区种植业结构调整中较为适宜的替代作物,但较高盐度条件仍然严重影响花生的生长和产量。因此,研究盐胁迫对不同生育时期花生根际土壤细菌群落和产量的影响,对盐碱地区发展花生生产具有重要的指导意义。
根际微生物是紧密附着于根际土壤颗粒中的微生物,其与植物根系相互作用、相互促进,参与并担负着根际重要的生理过程,维系着根际的生态平衡[2],是土壤与植物间物质交换活跃界面的重要成员[3-4],具有多种生物功能[5],在植物生长以及对各种生物和非生物胁迫的适应中起着至关重要的作用[6-8]。关于花生对耐盐胁迫适应性的研究大多集中在品种耐盐性鉴选[9-12]、光合产物积累与光合效能[13-14]、保护酶活性变化[15]、养分吸收积累以及种子际细菌群落变化等方面[16-17],同时,植物生长促进菌的接种,可为植物根系提供抵抗生物和非生物胁迫的缓冲区,产生各种植物生长促进素和生物防治剂,诱导系统抗性并增强养分矿化和有效性[18-19],研究了黄河三角洲不同含盐量盐碱地上花生旺盛生长期0~40 cm根层和旱盐同时胁迫下花生根际土壤细菌群落组成及其多样性[20-21]。目前,关于盐胁迫对不同生育期花生根际微生物群落组成的影响研究较少,针对盐碱土壤花生根际微生物群落结构、优势种群及花生生长阶段之间的关系等研究尚不系统[20-22]。本研究拟通过盐胁迫条件对花生不同生育期根际微生物群落结构变化和产量的影响,旨在发现和选择有益的耐盐微生物,明确非生物胁迫环境对微生物群落及功能的调节作用,发掘新的功能类群,发展盐碱地花生生产。
1 材料和方法
1.1 试验材料
供试土壤采用耕地表层土(0~30 cm),基础肥力:土壤有机质含量14.08 g/kg,全磷(P2O5)0.84 g/kg,全钾(K2O)11.26 g/kg,全氮1.70 g/kg,水解氮(N)102.1 mg/kg,速效磷(P2O5)14.7 mg/kg,速效钾(K2O)113.2 mg/kg,土壤pH值7.1,土壤交换性Na+ 0.82 cmol/kg,Cl- 28.5 mg/kg,土壤含水量9.82%。品种为花育25号(Huayu 25)。
1.2 试验设计
采用盆栽试验,选择底部直径为36 cm、高为26 cm的塑料盆,每盆装土量为18 kg。先将供试土壤风干、过1 cm筛后备用。选取饱满均匀的种子,每盆播6粒,播深均为3 cm,留4株长势一致的幼苗。
设置3个盐胁迫梯度:0.0(Y0),1.5(Y1),3.0 g/kg(Y2),盐胁迫梯度采用分析纯NaCl重量法控制。随机区组排列,6次重复。分别于开花下针期(播后70 d)、收获期(播后125 d)以处理和重复为单位采集根际土壤样本。采用“抖土法”多点混合方法,即以每处理重复为单元,“抖土法”收集紧密附着于根系的土壤,每3个重复根际土壤样品混合为1个生物学样本重复,每处理分别获取2个生物学重复,密封于灭菌后的离心管中,于-80 ℃超低温冰箱中保存备用。于收获期各处理收获实测计产,室内考察花生出米率、百果质量、百仁质量,计算荚果产量。
开花下针期各处理土壤样本分别表示为CK、Y1、Y2,收获期各处理土壤样本分别表示为HCK、HY1、HY2。相关测试由北京诺赛基因公司完成。
1.3 土壤DNA提取
土壤总DNA提取采用OMEGA soil DNA kit试剂盒进行,DNA的纯度和浓度检测采用1.5%琼脂糖凝胶电泳和Nanodrop2000 分光光度计进行。
1.4 16S rRNA文库构建及高通量测序
首先对16S rRNA基因V3-V4区进行扩增,引物340F:CCTACGGGNBGCASCAG以及805R:GACTACNV
GGGTATCTAATCC。扩增程序:95 ℃预变性3 min;30个循环包括(95 ℃ 30 s,50 ℃ 30 s,72 ℃ 60 s);72 ℃ 7 min。使用1.5%浓度的琼脂糖凝胶对PCR 产物进行电泳检测,使用0.5×TBE 浓度1.5%的琼脂糖凝胶电泳纯化PCR 产物,回收目标条带。产物纯化试剂盒为QIAGEN公司MinElute胶回收试剂盒。
将纯化的扩增子等摩尔合并,并在Illumina HiSeq2500/NovaSeq 250PE平台(Illumina,美国圣地亚哥)上进行配对末端测序。使用SOAPdenovo对原始数据进行拼接、过滤,将相似性值≥97%的序列定义为同一个OTU进行相关、聚类等分析。
1.5 生物信息学分析
基于有效数据进行OTUs(Operational taxonomic units)聚类和物种分类分析,得到每个样品OTUs 和分类谱系的基本分析结果,再进行其丰度和多样性指数分析,并在门和纲水平上进行群落结构统计分析。最后进行物种组成聚类分析、PCoA和PLS-DA统计比较分析,挖掘样品间的物种组成差异,并结合环境因素进行关联分析。
2 结果与分析
2.1 样本根际细菌群落测序质量分析
表1显示,通过对原始数据拼接、过滤,共获得有效序列(Clean data)893 527条,多数样本序列长度在400~500 bp,平均长度在400~500 bp(图1-A)。各处理花生根际样本共有2 507个OTUs,存在于CK、Y1和HY2中的OTUs数分别为59,36,29个,其余3个处理样本HCK、Y2和HY1中分别为21,16,17个,表明各处理样本微生物组成基本相似(图1-B)。
表1 各处理根际样本测序信息统计
Tab.1 The sequencing information statistics of peanut rhizosphere bacterial communities in different treatments
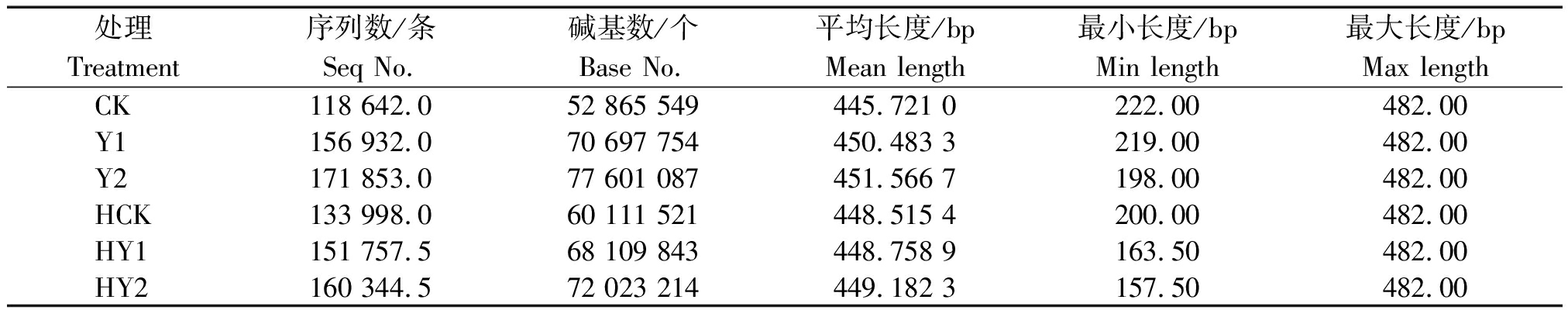
处理Treatment序列数/条Seq No.碱基数/个Base No.平均长度/bpMean length最小长度/bpMin length最大长度/bpMax lengthCK118 642.052 865 549445.721 0222.00482.00Y1156 932.070 697 754450.483 3219.00482.00Y2171 853.077 601 087451.566 7198.00482.00HCK133 998.060 111 521448.515 4200.00482.00HY1151 757.568 109 843448.758 9163.50482.00HY2160 344.572 023 214449.182 3157.50482.00
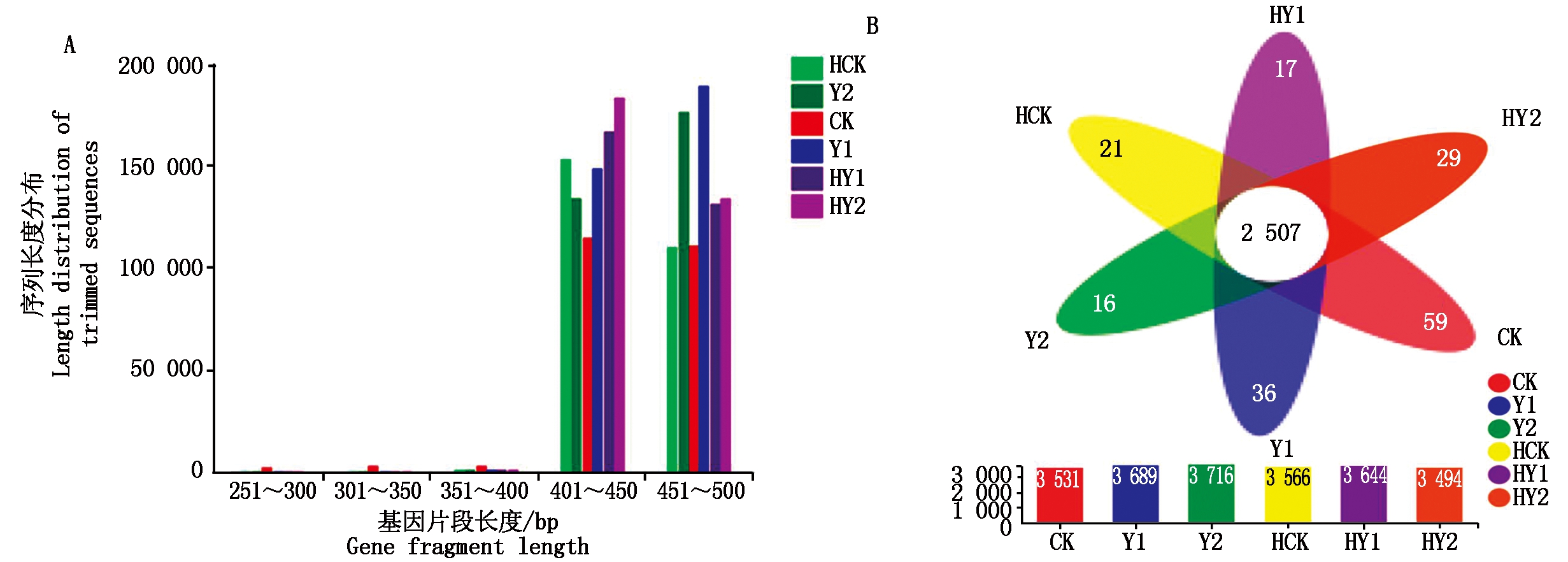
图1 花生根际土壤细菌群落测序分析
Fig.1 The sequence analysis of microbial communities in the peanut rhizosphere
2.1.1 多样性指数 表2所示,花生根际细菌种类丰富度和多样性受盐胁迫强度和生育时期的影响。各样本细菌群落中OTUs个数丰富度的Chao1、Ace和Sobs指数分别为3 308.915 1~3 722.820 3,3 316.230 4~3 684.215 7,2 797.50~3 371.00。反映群落多样性的Shannon和Simpson指数分别为5.606 5~6.979 7,0.002 170~0.027 914。各样本群落覆盖度Coverage指数均大于0.989 4。Y2样本的Chao1、Ace丰富度指数和Sobs指数及HY2的Simpson多样性指数均较高,HY2处理的丰富度指数和多样性指数均表现最低。收获期土壤根际细菌种类丰富度和多样性受盐胁迫强度的影响较大,较高强度的盐胁迫浓度使收获期根际土壤细菌种类丰富度和多样性均降低。
表2 各处理样本Alpha多样性指数
Tab.2 Alpha diversity index of rhizosphere soil samples in each treatment
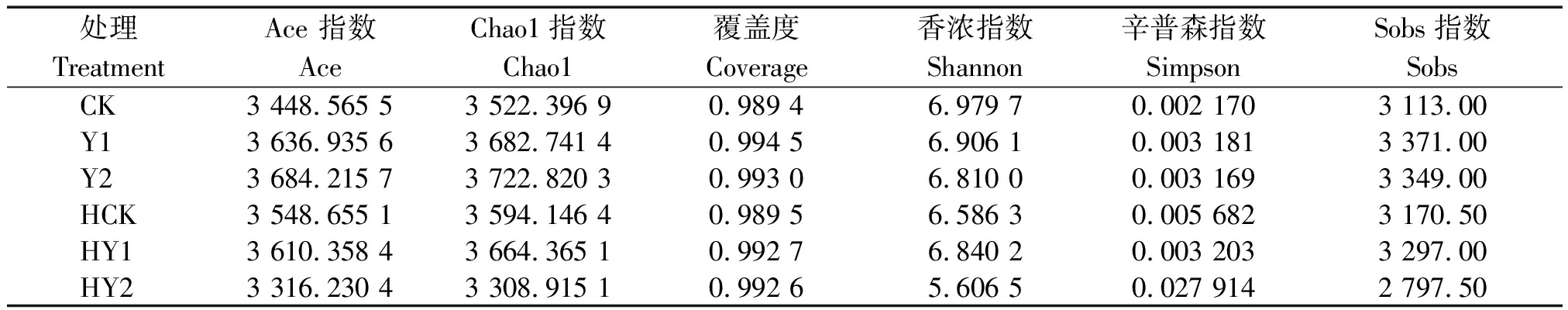
处理TreatmentAce 指数AceChao1指数Chao1覆盖度Coverage香浓指数Shannon辛普森指数SimpsonSobs指数SobsCK3 448.565 53 522.396 90.989 46.979 70.002 1703 113.00Y13 636.935 63 682.741 40.994 56.906 10.003 1813 371.00Y23 684.215 73 722.820 30.993 06.810 00.003 1693 349.00HCK3 548.655 13 594.146 40.989 56.586 30.005 6823 170.50HY13 610.358 43 664.365 10.992 76.840 20.003 2033 297.00HY23 316.230 43 308.915 10.992 65.606 50.027 9142 797.50
2.1.2 Rank Abundance曲线 各处理下各物种按相对丰度均从高到低依次排列,当OTU数量小于1 000时对应的物种相对丰度较高,曲线下降迅速且不平缓,表明其均匀度较低。当OTU数量大于2 000时,曲线较为平缓,表明其在土壤样本分布较为均匀(图2)。各处理样本间的曲线宽度和平滑程度基本一致,不同样本间物种均匀度无显著差异。
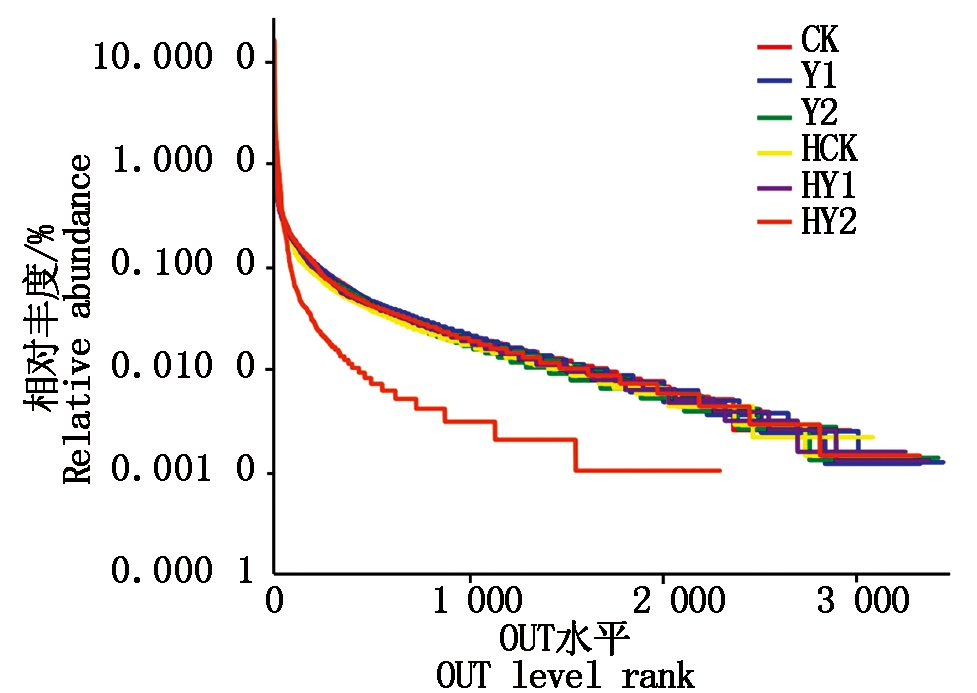
图2 Rank Abundance曲线
Fig.2 Rank Abundance curve
2.1.3 稀释性曲线 稀释曲线可以反映各样本在不同测序数量时的微生物多样性,并间接反映样本中物种的丰富程度。各样本在测序量达到3 500时,曲线均趋于平缓,测序深度足够,其结果可覆盖所测样本内的所有物种,可对微生物群落结构进行深入分析(图3)。花生不同生长期各处理OTU数量基本一致,均无显著差异。
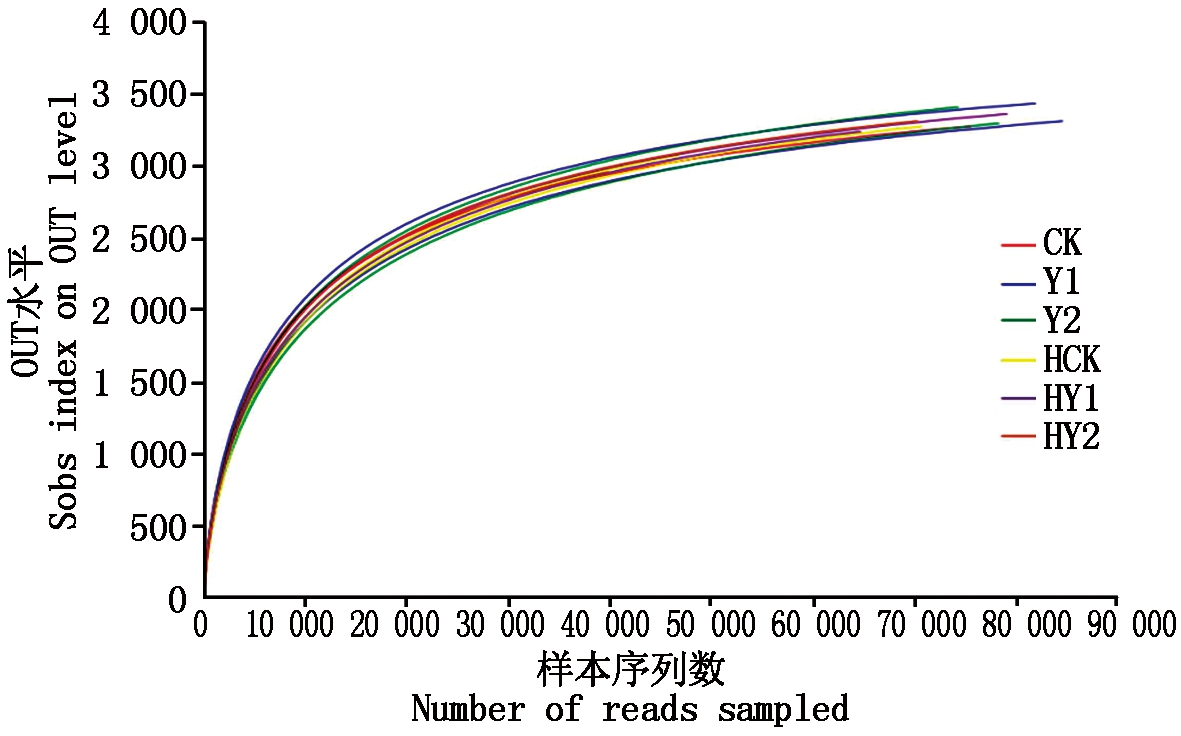
图3 稀释曲线
Fig.3 Rarefaction curve
2.2 物种组成及样本间差异比较
2.2.1 门水平全样本菌落结构分析 所有处理样本的29个门水平群落物种组成中,其Top11个门(其他合并<0.025)相对丰度之和达94.87%以上。各处理样本的优势菌门均为变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、绿弯菌门(Chloroflexi)、酸杆菌门(Acidobacteria)、疣微菌门(Verrucomicrobia)、拟杆菌门(Bacteroidetes)、Patescibacteria 7种,其相对丰度均在5.73%以上,其中以变形菌门平均相对丰度最高为31.50%,放线菌门相对丰度为15.48%,绿弯菌门和酸杆菌门相对丰度仅分别为9.96%,8.28%(图4)。
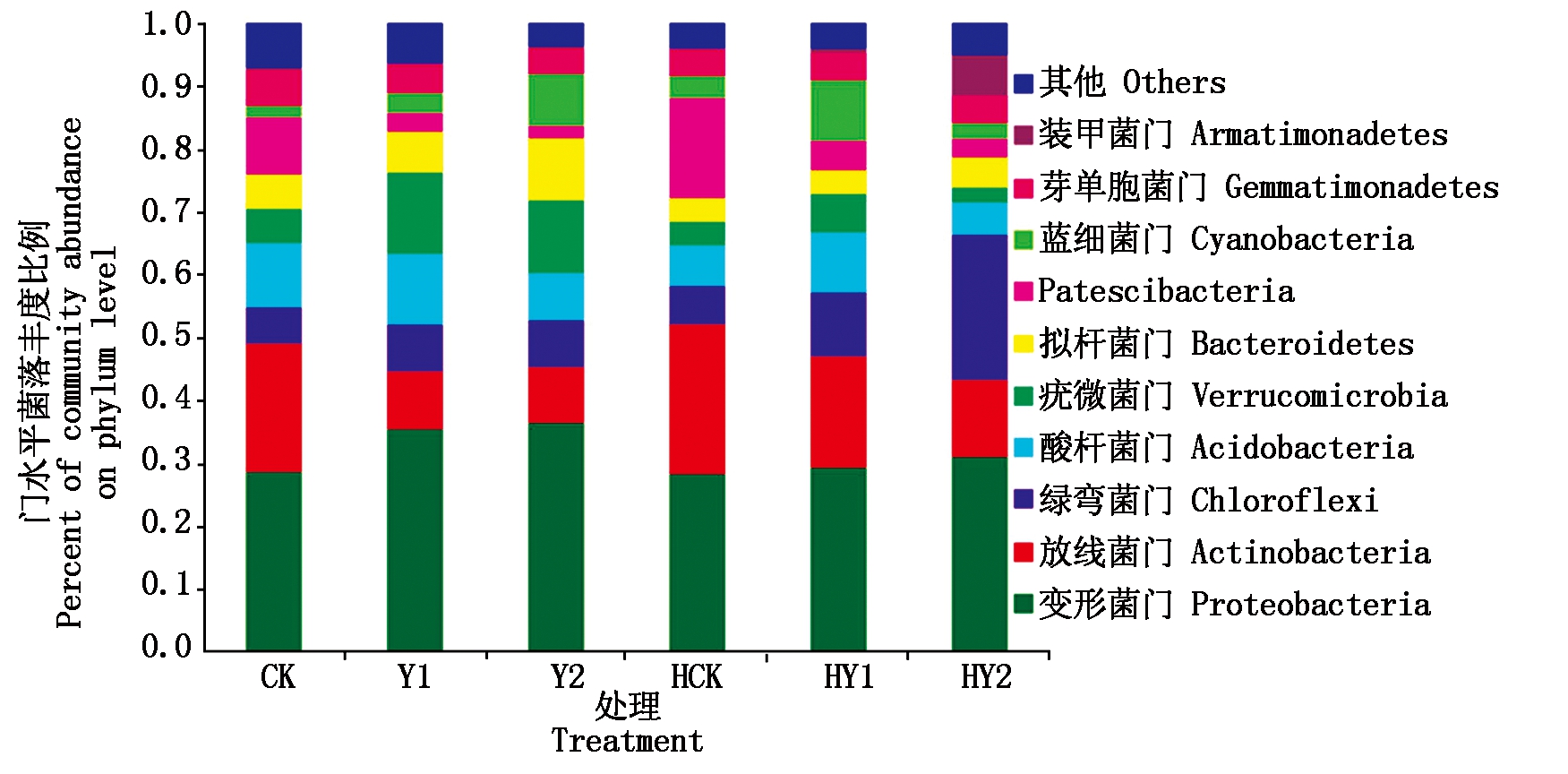
图4 门水平下各样本菌落结构分析
Fig.4 The relative abundance of the microbial communit at the phylum level
盐胁迫明显提高变形菌门、疣微菌门、拟杆菌门和蓝细菌门的相对丰度,尤以开花下针期明显,其中,变形菌门、疣微菌门和拟杆菌门3个优势菌门Y2处理较其CK分别升高27.00%,114.78%,74.57%,非优势菌门蓝细菌门是其CK的4.6倍。盐胁迫明显降低优势菌门放线菌门、酸杆菌门和Patescibacteria的相对丰度,较其相应对照降幅分别为55.58%,26.69%,78.78%。花生根际微生物群落组成受盐胁迫强度和生育时期的影响较大。
2.2.2 纲水平全样本菌落结构分析 所有处理样本的72个纲水平群落物种组成中,其top15(其他合并<0.025)的相对丰度之和平均达86.36%,优势菌纲(相对丰度在5%以上)为放线菌纲(Actinobacteria)、α-变形菌纲(Alphaproteobacteria)、δ-变形菌纲(Deltaproteobacteria)、γ-变形菌纲(Gammaproteobacteria)、芽单胞菌纲(Gemmatimonadetes)、拟杆菌纲(Bacteroidia)、疣微菌纲(Verrucomicrobiae)等7个菌纲。相对丰度在10%以上的优势菌纲仅有放线菌纲13.15%、α-变形菌纲12.68%和γ-变形菌纲14.65%(图5)。
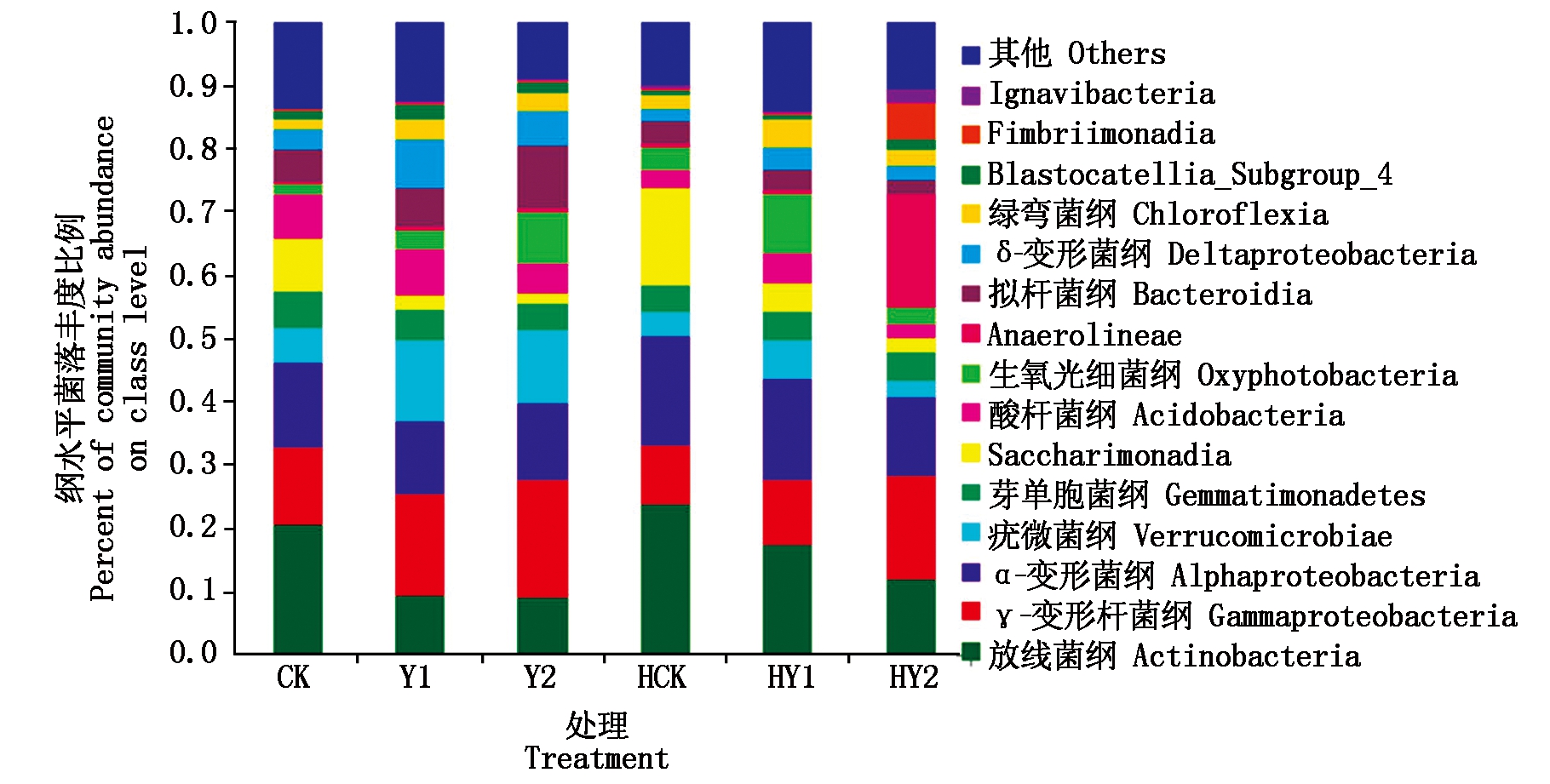
图5 纲水平下细菌群落结构分析
Fig.5 Analysis of the microbial community abundance at the class level
盐胁迫显著降低两生育时期放线菌纲和Saccharimonadia的相对丰度,而明显升高γ-变形菌纲的相对丰度,并均随盐胁迫强度的升高而升高。疣微菌纲和拟杆菌纲丰度于开花下针期丰度显著升高但至成熟期显著降低。
2.3 根际微生物群落组成比较分析
2.3.1 Beta多样性分析 基于Unweighted unifrac距离的PCoA(Principal co-ordinates analysis)分析表明,各处理样本分布于除第Ⅰ象限外的第Ⅱ、Ⅲ、Ⅳ象限,表明各处理样本间细菌群落组成存在明显差异,除HY2一重复的样本点距离较大外,其余处理同一样本的两重复间差异均较小,平行间重复性较好。横坐标(PC1)和纵坐标(PC2)分别可解释样品中所有差异的37.3%,24.8%。盐胁迫下开花下针期样品集中分布于第Ⅲ象限,物种组成相似度高;CK与收获期所有处理样本集中分布于第Ⅱ象限,其物种组成相近(图6-A)。样本层级聚类(Hierarchical clustering)结果表明,所有样本划分为4个显著不同的类群,盐胁迫下开花下针期和成熟期样本各聚为一类,两生育时期对CK各聚为一类。表明盐胁迫下花生根际细菌菌群受生长发育时期影响较大,与无盐胁迫根际细菌群落组成有明显不同(图6-B)。盐胁迫处理使根际土壤微生物菌群多样性的差异增大,受花生生育阶段和盐胁迫强度的影响较大。
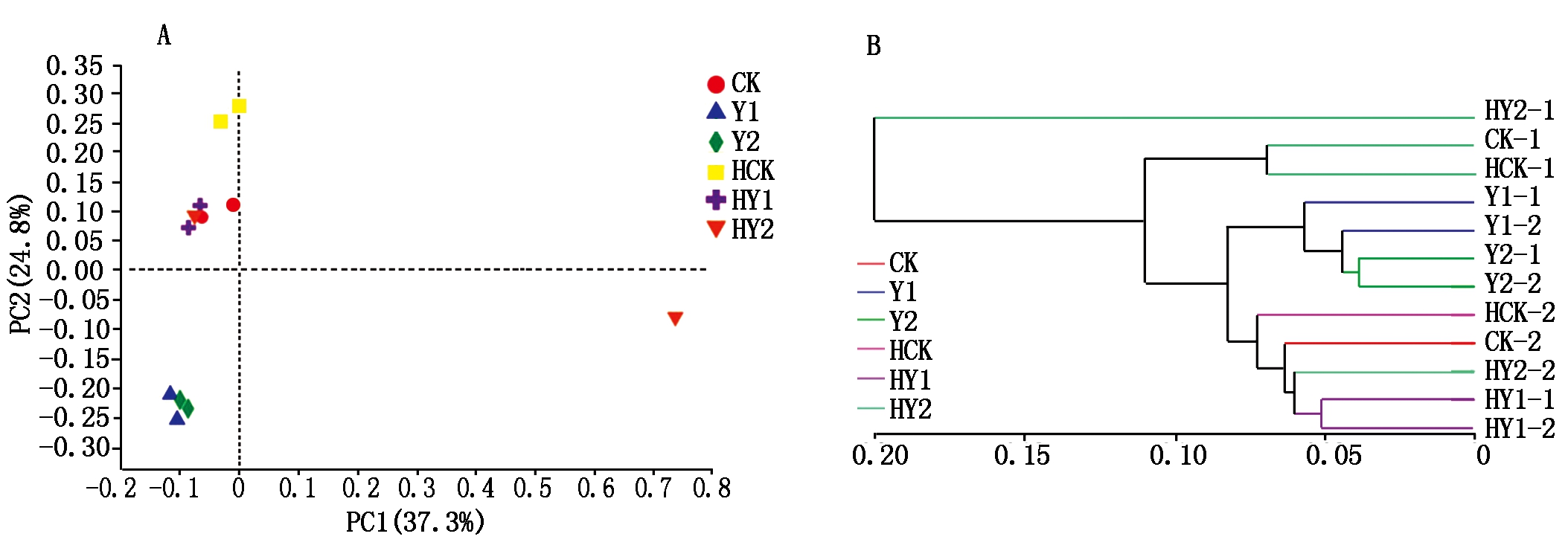
图6 Beta多样性分析
Fig.6 Beta diversity analysis
2.3.2 PLS-DA(Partial least squares discriminant analysis)分析 通过PLS-DA分析,图7-A、B可以看出,各处理样本点离散分布,各样本菌群组成差异较大,且在目水平和属水平上组成差异的影响因素相同,即各样本菌群组成差异受花生生育时期和盐胁迫程度的影响,盐胁迫下受生育时期影响较大。
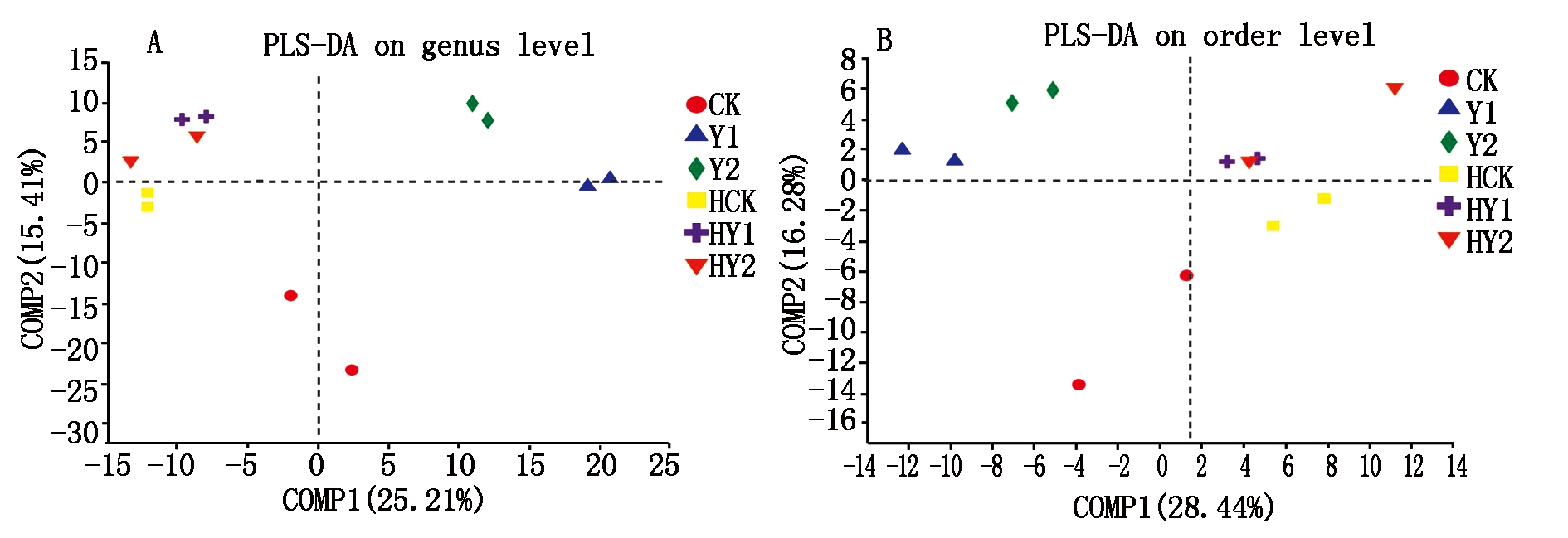
图7 PLS-DA分析
Fig.7 Partial least squares discriminant analysis
2.4 细菌群落代谢功能特征
通过PICRUSt2预测了花生根际细菌群落的代谢功能特征(COG)。各处理根际细菌富集的功能主要包括:氨基酸转运与代谢、能量生产和转化、信号转导机制、细胞壁/膜/包膜生物发生、转录和碳水化合物的运输和代谢等。盐胁迫使各功能基因丰度提高,且与生育时期和盐胁迫强度有关,开花下针期以Y1处理各功能基因丰度较高,而至收获期各功能基因丰度则随盐胁迫强度的升高而升高,尤以信号传递(翻译、转录、复制重组和修复)、细胞周期变化(细胞周期控制、细胞分裂、染色体分区、细胞壁/膜/包膜生物发生)和物质能量代谢运输(能源生产与转化、碳
水化合物的运输和代谢、氨基酸转运和代谢和无机离子的运输与代谢等)等功能丰度升幅较大。除未知功能基因外,各处理功能基因总数为71.07×106~105.23×106,Y2处理最大,Y1处理次之(99.68×106个),CK和HCK处理最小,分别为71.07×106和71.12×106个。CK预测功能基因总数比HY2处理降低32.47%,较Y2降低28.71%(图8-A)。
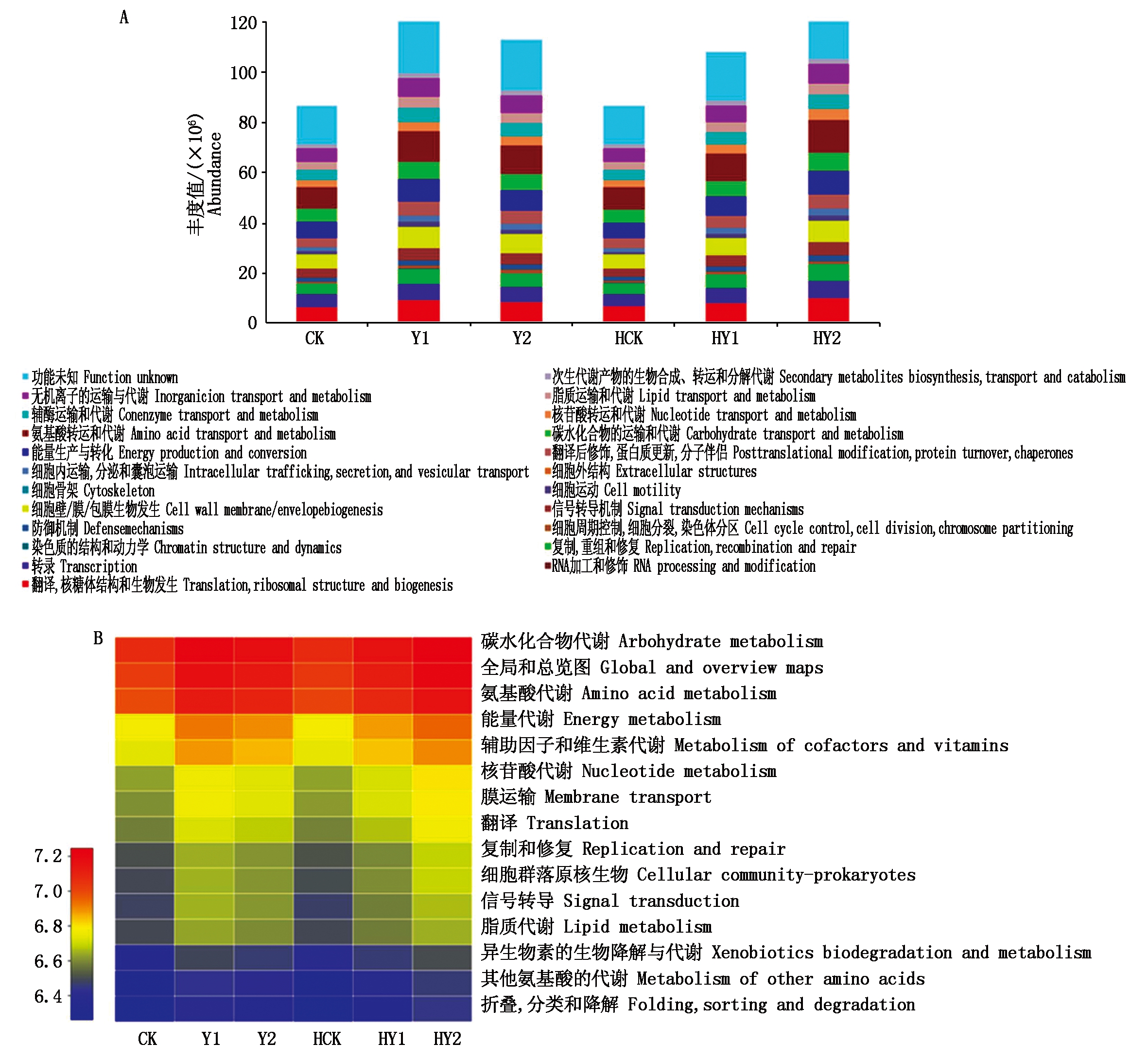
图8 样本中微生物群落功能组成预测
Fig.8 The microbial functional features of Orthologous Groups(COG)and KEGG level 2 analysis
使用热图分析比较了KEGG level 2级种群代谢功能基因(Top15)表明(图8-B),物质能量代谢(碳水化合物代谢、氨基酸代谢、核苷酸代谢、辅助因子和维生素代谢、脂质代谢)、翻译、复制和修复、信号转导及膜运输等为主要代谢功能。其中,所有样本中菌群涉及碳水化合物代谢、氨基酸代谢、能量代谢、辅助因子和维生素的代谢等功能基因丰度增加,而涉及信号转导、脂质代谢、复制和修复、异种生物的生物降解和代谢、其他氨基酸的代谢、折叠,分类和降解等功能的丰度降低。盐胁迫处理使各功能基因丰度明显升高,尤以Y1和HY2处理丰富升高明显,CK和HCK各种功能丰度降低明显。盐胁迫和花生生育时期在细菌种群功能方面起重要作用,有利于优势细菌功能群丰度提高。
2.5 产量性状
表2看出,盐胁迫处理显著影响花生出米率、百果质量、百仁质量和产量,且随盐胁迫程度的提高作用明显。盐胁迫处理的百仁质量较CK分别降低32.74%,37.63%,盐胁迫处理的产量仅为CK处理的50%左右。可见,盐胁迫严重抑制花生籽仁发育和产量形成。
表2 盐胁迫对花生产量及构成因素的影响
Tab.2 Effects on peanut yield and its components under salt stress
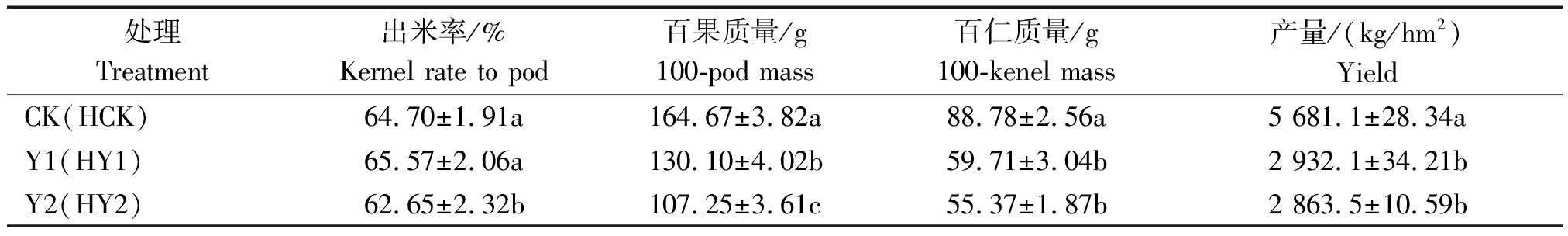
处理Treatment出米率/%Kernel rate to pod百果质量/g100-pod mass百仁质量/g100-kenel mass产量/(kg/hm2)YieldCK(HCK)64.70±1.91a164.67±3.82a88.78±2.56a5 681.1±28.34aY1(HY1)65.57±2.06a130.10±4.02b59.71±3.04b2 932.1±34.21bY2(HY2)62.65±2.32b107.25±3.61c55.37±1.87b2 863.5±10.59b
注:同一组不同小写字母表示处理间差异显著(P<0.05)。
Note:Different letters in the same group indicated significant differences among treatments at 0.05 level.
3 讨论与结论
植物根际和根内微生物群落具有明显的根际效应,根系分泌物影响附近土壤微环境中微生物活性和菌群结构,并对微生物群落结构和功能多样性施加选择性压力[23]。此外,土壤特性、植物种类、生长阶段和其他胁迫因素也会影响微生物群落组成。黄河三角洲滨海盐碱土高含盐量土壤中根际土壤微生物种类、优势种群数量和群落功能多样性更为丰富。盐碱地花生//棉花间作影响二者根层微生物群落的组成,花生//棉花间作使得花生根层土壤变形菌门和放线菌门丰度值降低,花生根际微生物菌群类型与土壤含盐量和根系分布深度有关[21,24-26]。本研究结果表明,盐胁迫和正常条件下,花生根际微生物群落组成无显著差异,但其多样性和丰富度差异受盐胁迫强度和生育阶段的影响。较高强度的盐胁迫使花生开花下针期根际微生物种类丰富度和多样性升高,可能由于花生生长阶段不同,土壤微环境和物理化学性质有差异,在根系分泌物因长势和盐胁迫逆境调节微生物群落组成的同时,也因各种微生物对盐胁迫逆境的耐受性不同而凋亡或存活,随后可能会改善植物的生长和耐盐性。
根系对微生物的“选择招募”过程与微生物自身的移动性、趋化性、多糖降解性等因素有关[20,27-29]。根际环境中纤维弧菌属(Cellvibrio)和假单胞菌(Pseudomonas)的相对丰度远远高于非根际土壤,而二者在降解纤维素和好氧反硝化细菌的过程中发挥着重要作用[30-31]。 芽单胞菌门对盐胁迫环境具有很好的适应能力[32],蓝细菌可在盐碱土中生存,通过转化积累土壤中的碳和氮以改善土壤微环境[33-35]。本试验条件下,盐胁迫导致花生根际微生物群落转移和特定微生物种类富集,变形菌门、拟杆菌门、疣微菌门和蓝细菌等优势菌门的相对丰度明显提高,尤以开花下针期明显。较高的蓝细菌和芽孢杆菌及旺盛的生长阶段,可能有助于花生在盐胁迫下维持各种生理功能,从而增强花生耐盐能力。
土壤微生物群落对于植物在盐胁迫下维持基本功能可能很重要。研究发现盐胁迫条件下,花生根际细菌群落涉及物质和能量代谢、膜运输、翻译、复制和修复以及信号转导等优势细菌功能基因丰度增加,可以推测这些途径可能有助于提高花生的耐盐性。因此,基于植物-微生物互作关系,充分利用和发挥根际微生物资源,调控管理根际微生物群落并保持有益微生物和有害微生物之间的平衡,开发生物肥料和生物防控剂,对改善土壤肥力、提高作物产量、维持农业生态环境健康等具有重要意义。
[1] 山仑,邓西平,康绍忠.我国半干旱地区农业用水现状及发展方向[J].水利学报,2002,33(9):27-31. doi :10.3321/j.issn:0559-9350.2002.09.005.
Shan L,Deng X P,Kang S Z. Current situation and perspective of agricultural water used in semiarid area of China[J].Journal of Hydraulic Engineering,2002,33(9):27-31.
[2] 陈禹竹,唐琦勇,顾美英,朱静,刘晓静,崔卫东,张志东.盐爪爪根部微生物分布特征及盐浓度对碳源代谢分析的影响[J].微生物学通报,2019,46(11):2900-2908.doi: 10.13344/j.microbiol.china.190020.
Chen Y Z,Tang Q Y,Gu M Y,Zhu J,Liu X J,Cui W D,Zhang Z D.Microbial distribution characteristics around the roots of Kalidium foliatum and the effect of salt concentrations on microbial metabolism analysis[J].Microbiology China,2019,46(11):2900-2908.
[3] 徐文静,靳晓东,杨秋生.植物根际微生物的影响因素研究进展[J].河南农业科学,2014,43(5):6-12. doi:10.15933/j.cnki.1004-3268.2014.05.009.
Xu W J,Jin X D,Yang Q S.Research progress on factors influencing plant rhizosphere microorganism[J]. Journal of Henan Agricultural Sciences,2014,43(5):6-12.
[4] Barea J M,Pozo M J,Azcón R,Azcón-Aguilar C. Microbial co-operation in the rhizosphere[J].Journal of Experimental Botany,2005,56(417):1761-1778. doi:10.1093/jxb/eri197.
[5] 陈智裕,马静,赖华燕,马祥庆,吴鹏飞.植物根系对根际微环境扰动机制研究进展[J].生态学杂志,2017,36(2):524-529.doi:10.13292/j.1000-4890.201702.021.
Chen Z Y,Ma J,Lai H Y,Ma X Q,Wu P F.Research advances in the mechanisms of plant root systems disturbance in rhizosphere micro-environment[J].Chinese Journal of Ecology,2017,36(2):524-529.
[6] Bai Y,Müller D B,Srinivas G,Garrido-Oter R,Potthoff E,Rott M,Dombrowski N,Münch P C,Spaepen S,Remus-Emsermann M,Hüttel B,McHardy A C,Vorholt J A,Schulze-Lefert P. Functional overlap of the Arabidopsis leaf and root microbiota[J].Nature,2015,528(7582):364-369. doi:10.1038/nature16192.
[7] Correa-Galeote D,Bedmar E J,Fern ndez-Gonz
ndez-Gonz lez A J,Fern
lez A J,Fern ndez-Gonz
ndez-Gonz lez A J,Fern
lez A J,Fern ndez-López M,Arone G J,Arone G J. Bacterial communities in the rhizosphere of amilaceous maize(Zea mays L.)as assessed by pyrosequencing[J].Frontiers in Plant Science,2016,7:1016. doi:10.3389/fpls.2016.01016.
ndez-López M,Arone G J,Arone G J. Bacterial communities in the rhizosphere of amilaceous maize(Zea mays L.)as assessed by pyrosequencing[J].Frontiers in Plant Science,2016,7:1016. doi:10.3389/fpls.2016.01016.
[8] Singh A,Sarma B K,Upadhyay R S,Singh H B.Compatible rhizosphere microbes mediated alleviation of biotic stress in chickpea through enhanced antioxidant and phenylpropanoid activities[J].Microbiological Research,2013,168(1):33-40. doi:10.1016/j.micres.2012.07.001.
[9] 张智猛,慈敦伟,丁红,宋文武,符方平,康涛,戴良香.花生品种耐盐性指标筛选与综合评价[J].应用生态学报,2013,24(12):3487-3494.doi:10.13287/j.1001-9332.2013.0584.
Zhang Z M,Ci D W,Ding H,Song W W,Fu F P,Kang T,Dai L X.Indices selection and comprehensive evaluation of salinity tolerance for peanut varieties[J].Chinese Journal of Applied Ecology,2013,24(12):3487-3494.
[10] 李瀚,杨吉顺,张冠初,慈敦伟,丁红,秦斐斐,张智猛,石书兵.花生品种萌发期耐盐性比较鉴定[J].花生学报,2015,44(4):48-52,57.doi: 10.14001/j.issn.1002-4093.2015.04.009.
Li H,Yang J S,Zhang G C,Ci D W,Ding H,Qin F F,Zhang Z M,Shi S B.Identification of salt tolerance in germination period of peanut varieties[J].Journal of Peanut Science,2015,44(4):48-52,57.
[11] 慈敦伟,戴良香,宋文武,张智猛.花生萌发至苗期耐盐胁迫的基因型差异[J].植物生态学报,2013,37(11):1018-1027.doi:10.3724/SP.J.1258.2013.00105.
Ci D W,Dai L X,Song W W,Zhang Z M.Genotypic differences in salt tolerance from germination to seedling stage in peanut[J].Chinese Journal of Plant Ecology,2013,37(11):1018-1027.
[12] 温赛群.苗期花生品种耐盐性鉴定及生理生化评价[D].保定:河北农业大学,2019.
Wen S Q. Identification of salt tolerance of peanut varieties at seedling stage and physiological-biochemical evaluation[D].Baoding:Hebei Agricultural University,2019.
[13] 田家明,张智猛,戴良香,张冠初,慈敦伟,丁红,杨吉顺,史晓龙,石书兵.外源钙对盐碱土与非盐碱土花生生长发育与光合特性的影响[J].华北农学报,2018,33(6):130-136.doi: 10.7668/hbnxb.2018.06.018.
Tian J M,Zhang Z M,Dai L X,Zhang G C,Ci D W,Ding H,Yang J S,Shi X L,Shi S B.Effects of exogenous calcium on the development and photosynthetic characteristics of peanut in saline-alkali soil and normal soil[J].Acta Agriculturae Boreali-Sinica, 2018,33(6):130-136.
[14] 张冠初,史晓龙,慈敦伟,丁红,杨吉顺,田家明,张智猛,戴良香.干旱和盐胁迫对花生干物质积累及光合特性的影响[J].核农学报,2019,33(5):999-1005.doi: 10.11869/j.issn.100-8551.2019.05.0999.
Zhang G C,Shi X L,Ci D W,Ding H,Yang J S,Tian J M,Zhang Z M,Dai L X.Effect of drought and salt stress on accumulation of plant dry weight and photosynthetic characteristics[J].Journal of Nuclear Agricultural Sciences,2019,33(5):999-1005.
[15] 张冠初,张智猛,慈敦伟,丁红,杨吉顺,史晓龙,田家明,戴良香.干旱和盐胁迫对花生渗透调节和抗氧化酶活性的影响[J].华北农学报,2018,33(3):176-181.doi: 10.7668/hbnxb.2018.03.026.
Zhang G C,Zhang Z M,Ci D W,Ding H,Yang J S,Shi X L,Tian J M,Dai L X. Effects of drought and salt stress on osmotic regulator and antioxidase activities[J].Acta Agriculturae Boreali-Sinica,2018,33(3):176-181.
[16] 史晓龙,张智猛,戴良香,张冠初,慈敦伟,丁红,田家明.外源施钙对盐胁迫下花生营养元素吸收与分配的影响[J].应用生态学报,2018,29(10):3302-3310.doi: 10.13287/j.1001-9332.201810.026.
Shi X L,Zhang Z M,Dai L X,Zhang G C,Ci D W,Ding H,Tian J M. Effects of calcium fertilizer application on absorption and distribution of nutrients in peanut under salt stress[J].Chinese Journal of Applied Ecology,2018,29(10):3302-3310.
[17] Dai L X,Zhang G C,Yu Z P,Ding H,Xu Y,Zhang Z M.Effect of drought stress and developmental stages on microbial community structure and diversity in peanut rhizosphere soil[J].International Journal of Molecular Sciences,2019,20(9):2265.doi:10.3390/ijms20092265.
[18] Evelin H,Kapoor R,Giri B. Arbuscular mycorrhizal fungi in alleviation of salt stress:A review[J].Ann Bot,2009,104(7):1263-1280. doi:10.1093/aob/mcp251.
[19] Geng L L,Shao G X,Raymond B,Wang M L,Sun X X,Shu C L,Zhang J. Subterranean infestation by Holotrichia parallela larvae is associated with changes in the peanut(Arachis hypogaea L.)rhizosphere microbiome[J].Microbiological Research,2018,211:13-20.doi:10.1016/j.micres.2018.02.008.
[20] 戴良香,康涛,慈敦伟,丁红,徐扬,张智猛,张岱,李文金.黄河三角洲盐碱地花生根层土壤菌群结构多样性[J].生态学报,2019,39(19):7169-7178. doi: 10.5846/stxb201807051469.
Dai L X,Kang T,Ci D W,Ding H,Xu Y,Zhang Z M,Zhang D,Li W J.Comparison of the microbial community in the rhizosphere of peanuts between saline-alkali and non-saline soil at different soil depths and intercropping cultivation in the Yellow River Delta[J].Acta Ecologica Sinica,2019,39(19):7169-7178.
[21] 徐扬,张冠初,丁红,慈敦伟,秦斐斐,张智猛,戴良香. 干旱与盐胁迫对花生根际土壤细菌群落结构和花生产量的影响[J].应用生态学报,2020,31(4):1305-1313.doi: 10.13287/j.1001-9332.202004.036.
Xu Y,Zhang G C,Ding H,Ci D W,Qin F F,Zhang Z M,Dai L X.Effects of salt and drought stresses on rhizosphere soil bacterial community structure and peanut yield[J].Chinese Journal of Applied Ecology,2020,31(4):1305-1313.
[22] 杜滢鑫,谢宝明,蔡洪生,唐璐,郭长虹.大庆盐碱地九种植物根际土壤微生物群落结构及功能多样性[J].生态学报,2016,36(3):740-747.doi:10.5846/stxb201404020621.
Du Y X,Xie B M,Cai H S,Tang L,Guo C H.Structural and functional diversity of rhizosphere microbial community of nine plant species in the Daqing saline-alkali soil region[J].Acta Ecologica Sinica,2016,36(3):740-746.
[23] Fu C,Liu X X,Yang W W,Zhao C M,Liu J.Enhanced salt tolerance in tomato plants constitutively expressing heat-shock protein in the endoplasmic reticulum[J].Genetics and Molecular Research,2016,15(2):15028301. doi:10.4238/gmr.15028301.
[24] Wu L K,Wang J Y,Wu H M,Chen J,Xiao Z G,Qin X J,Zhang Z Y,Lin W X. Comparative metagenomic analysis of rhizosphere microbial community composition and functional potentials under Rehmannia glutinosa consecutive monoculture[J].International Journal of Molecular Sciences,2018,19(8):2394.doi:10.3390/ijms19082394.
[25] Guan P Y,Wang J,Li H,Xie C,Zhang S Z,Wu C G,Yang G D,Yan K,Huang J G,Zheng C C.SENSITIVE to SALT1,an endoplasmic reticulum-localized chaperone,positively regulates salt resistance[J].Plant Physiology,2018,178(3):1390-1405.doi: 10.1104/pp.18.00840.
[26] Peiffer J A,Spor A,Koren O,Jin Z,Tringe S G,Dangl J L,Buckler E S,Ley R E.Diversity and heritability of the maize rhizosphere microbiome under field conditions[J].Proceedings of the National Academy of Sciences of the United States of America,2013,110(16):6548-6553. doi:10.1073/pans.1302837110.
[27] 丁新景,敬如岩,黄雅丽,陈博杰,马风云.黄河三角洲刺槐根际与非根际细菌结构及多样性[J].土壤学报,2017,54(5):1293-1302.doi: 10.11766/trxb201703230510.
Ding X J,Jing R Y,Huang Y L,Chen B J,Ma F Y.Bacterial structure and diversity of rhizosphere and bulk soil of Robinia pseudoacacia forests in Yellow River Delta[J].Acta Pedologica Sinica,2017,54(5):1293-1302.
[28] Ofek-Lalzar M,Sela N,Goldman-Voronov M,Green S J,Hadar Y,Minz D. Niche and host-associated functional signatures of the root surface microbiome[J].Nature Communications,2014,5:4950.doi:10.1038/ncomms5950.
[29] Lebeis S L.The potential for give and take in plant-microbiome relationships[J].Frontiers in Plant Science,2014,5:287.doi:10.3389/fpls.2014.00287.
[30] Vandenkoornhuyse P,Quaiser A,Duhamel M,Van A L,Dufresne A. The importance of the microbiome of the plant holobiont[J].New Phytologist,2015,206(4):1196-1206. doi: 10.1111/nph.13312.
[31] 王芬,段洪利,刘亚飞,王天弋.人工湿地处理含盐富营养化水的植物根际与非根际菌群分析[J].环境工程学报,2020,14(7):1844-1851.doi:10.12030/j.cjee.201909029.
Wang F,Duan H L,Liu Y F,Wang T Y. Analysis of bacterial community at the rhizosphere and non-rhizosphere of plants in constructed wetland treating brackish eutrophic water[J].Chinese Journal of Environmental Engineering,2020,14(7):1844-1851.
[32] Fozo E M,Quivey R G. Shifts in the membrane fatty acid profile of Streptococcus mutans enhance survival in acidic environments[J].Applied and Environmental Microbiology,2004,70(2):929-936.doi:10.1128/aem.70.2.929-936.2004.
[33] DeBruyn J M,Nixon L T,Fawaz M N,Johnson A M,Radosevich M. Global biogeography and quantitative seasonal dynamics of gemmatimonadetes in soil[J].Appl Environ Microbiol,2011,77(17):6295-6300.doi:10.1128/aem.05005-11.
[34] Doolotkeldieva T,Konurbaeva M,Bobusheva S. Microbial communities in pesticide-contaminated soils in Kyrgyzstan and bioremediation possibilities[J].Environmental Science and Pollution Research,2018,25(32):31848-31862.doi:10.1007/s11356-017-0048-5.
[35] Jeske O,Surup F,Ketteniβ M,Rast P,Förster B,Jogler M,Wink J,Jogler C. Developing techniques for the utilization of planctomycetes as producers of bioactive molecules[J].Frontiers in Microbiology,2016,7:1242.doi:10.3389/fmicb.2016.01242.