植物对开花时间的调控是对环境的自我适应,也是谷类作物产量的主要决定因素,此过程有许多基因家族一起参与,通过错综复杂的分子途径相结合来完成。CCT结构域是在拟南芥中最早被报道的,由拟南芥中CONSTANS(CO)蛋白、CO-LIKE蛋白和TIMING OF CABl(TOCl)蛋白C末端的43个同源保守氨基酸序列组成[1-2]。根据CCT中结构域的数量和类别,可划分为3个亚家族,分别是CMF、COL和PRR亚家族,其中,COL亚家族含有B-box锌指结构域和一个CCT结构域,PRR亚家族含有PRR结构域和一个CCT结构域,而CMF亚家族只有一个CCT结构域[3]。Cockram等[4]对禾本科植物不同物种CCT家族的基因序列进行比对和系统发育分析,结果表明,CMF亚家族是在单子叶植物/双子叶植物进化分支后,从COL基因的共同祖先进化演变来的,推测COL基因的持续进化导致B-box结构域消失。在相关的研究中,许多CCT基因都直接或者间接地调节植物开花时间,参与生物钟调控,是光信号途径中的重要一员[1,5-6]。有关长日照植物拟南芥和短日照植物水稻中CCT家族基因的研究报道较多,其中CO、TOCl、COL1、COL2、COL9等成员参与花期调控的遗传机制已基本清楚[7-8]。水稻中的Ghd7和玉米中的ZmCCT等,仅仅具有单个CCT结构域并且没有任何其他结构域,在调节水稻和玉米的开花时间和适应不同环境中发挥着关键的作用[9-10]。
河南科技大学农学院谷子分子育种团队前期克隆了一个谷子CCT家族基因(SiCCT),分析了该基因在不同光周期条件下的昼夜表达模式和不同光温组合条件下不同生长期的表达特点[11],本研究进一步分析SiCCT基因在不同光温组合条件下的昼夜表达模式,并检测该基因在160份谷子资源的单核苷酸多态性位点(SNP),开展SNP位点与10个农艺性状间的关联分析,获得与农艺性状显著相关的SNP位点,以进一步了解SiCCT基因的光温响应特点和可能的功能效应,为揭示该基因参与谷子光温互作调控作用机理奠定基础。
1 材料和方法
1.1 试验材料和地点
用于SiCCT基因表达分析的黄毛谷品种来自河北省,用于SiCCT基因SNP位点检测及关联分析的160份谷子资源来自河南、青海、甘肃、山东、山西、陕西、黑龙江、西藏、宁夏、吉林、辽宁、新疆、内蒙古、河北等国内各地区及印度、美国、日本、德国等国家,由中国农业科学院作物科学研究所、河北农林科学院谷子研究所提供。分别于2015-2016年在吉林、河南和海南3个省种植,3个地点坐标分别是吉林省吉林市农业科学院试验田(42°31′N,125°40′E)、河南省洛阳市河南科技大学试验农场(34°35′N,112°24′E)、海南省乐东县九所镇(18°45′N,109°10′E)。每个材料种植1行,行长2 m,田间管理按当地方法,调查性状包括株高、穗长、抽穗期、穗粗、穗码数、叶片数、穗质量、穗粒质量、千粒质量、码粒数。
1.2 试验方法
1.2.1 谷子的光温处理及实时荧光定量分析 本研究设置了长日照高温(15 h 光照/9 h 黑暗,27 ℃)、短日照高温(9 h 光照/15 h 黑暗,27 ℃)、长日照低温(15 h 光照/9 h 黑暗,22 ℃)、短日照低温(9 h光照/15 h黑暗,22 ℃)共4种不同的光温组合处理,除光照和温度外,其余环境保持一致。挑选大小一致、籽粒饱满的黄毛谷种子于10 cm×10 cm小方盆中种植80盆,每盆8~10粒种子,于25 ℃、12 h光照/12 h黑暗的温室萌发长至二叶期定苗,每盆保留4株长势一致的小苗,待三叶期分别转移至4个不同光温处理的人工气候箱培养,每个处理20盆。小苗长至六叶期时,从6:00(光照开始)开始取样,每隔3 h 取一次样,连续24 h取样,采集谷苗顶端1~2片嫩叶于液氮保存。
RNA提取采用康为世纪有限公司的OmniPlant RNA Kit(DNase Ⅰ),反转录使用TaKaRa公司的PrimeScript Ⅱ 1st Strand cDNA Synthesis Kit。SiCCT基因的实时荧光定量分析以反转录得到的cDNA作为模板,用引物SiCCT-F-RT/SiCCT-R-RT扩增SiCCT基因的特异保守片段,同时以引物SiActin-F和SiActin-R扩增作内参,引物信息见表1。每个样品做3个重复,方法参照TB GreenTM Premix Ex TaqTM Ⅱ(Tli RNaseH Plus)试剂(TaKaRa)的说明书。
表1 荧光定量PCR引物信息
Tab.1 Primer information for fluorescence quantitative PCR
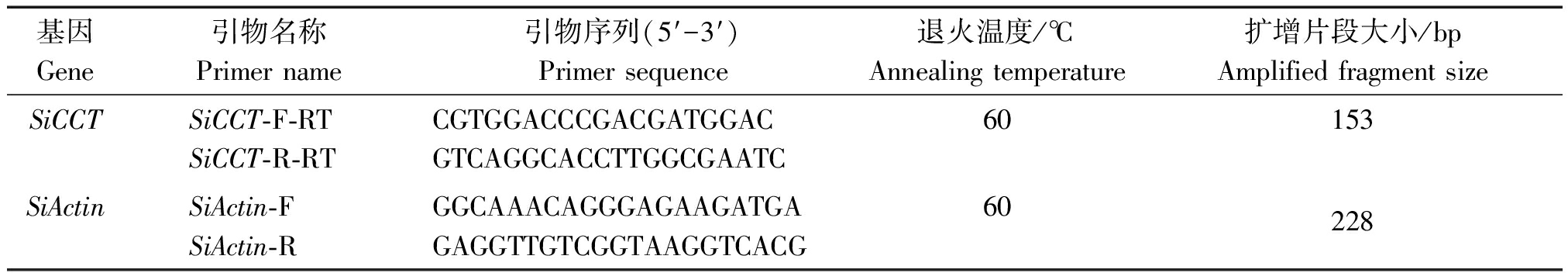
基因Gene引物名称Primer name引物序列(5′-3′)Primer sequence退火温度/℃Annealing temperature扩增片段大小/bpAmplified fragment sizeSiCCTSiCCT-F-RTSiCCT-R-RTCGTGGACCCGACGATGGACGTCAGGCACCTTGGCGAATC60153SiActinSiActin-FSiActin-RGGCAAACAGGGAGAAGATGAGAGGTTGTCGGTAAGGTCACG60228
1.2.2 SiCCT基因SNP位点检测及群体结构分析、单倍型分析 根据SiCCT基因在谷子基因组位置信息,从160份谷子重测序数据中提取SNP位点,采用Haploview 4.2软件,对160份谷子中SiCCT基因中的SNP位点进行连锁不平衡分析、单倍型分析,绘制单体型块图(LD Plot),分析两两位点的相关性,具体方法参见软件说明书。选取均匀分布于谷子9条染色体上的3 000个SNP,用Structure 1.8软件对160份谷子材料进行群体结构分析。
1.2.3 基于候选基因的关联分析 通过TASSEL 5.0软件对160份谷子中SiCCT基因的SNP数据进行分析,选取最小等位基因频率大于0.05的多态性位点,按要求过滤掉没有多态性的位点,然后计算160份谷子品种间的亲缘关系(K);将群体结构(Q矩阵)和亲缘关系(K矩阵)作为协变量,结合2 a三地相关的表型数据,采用TASSEL软件中的混合线性模型(Mixed linear model,MLM)方法进行分析,筛选出候选基因与主要农艺性状显著关联的SNP位点,并估计关联位点对主要农艺性状的效应及贡献率。
2 结果与分析
2.1 不同光温组合条件下SiCCT 基因的昼夜表达规律
由图1可知,昼夜24 h内SiCCT基因表达具有明显的节律性,无论高温还是低温,从6:00到21:00均表现相似的表达模式,即6:00到15:00光照期长日照条件基因表达量极显著高于短日照(P<0.01),18:00长、短日照条件基因表达水平接近,21:00长日照条件基因表达量显著或极显著高于短日照(P<0.05,P<0.01)。而在24:00到3:00黑暗期高温低温间基因表达模式存在差异,高温条件只在3:00短日照基因表达量极显著高于长日照(P<0.01),低温条件24:00,3:00短日照基因表达量均极显著高于长日照(P<0.01),说明温度对SiCCT基因在黑暗期的表达有明显影响。
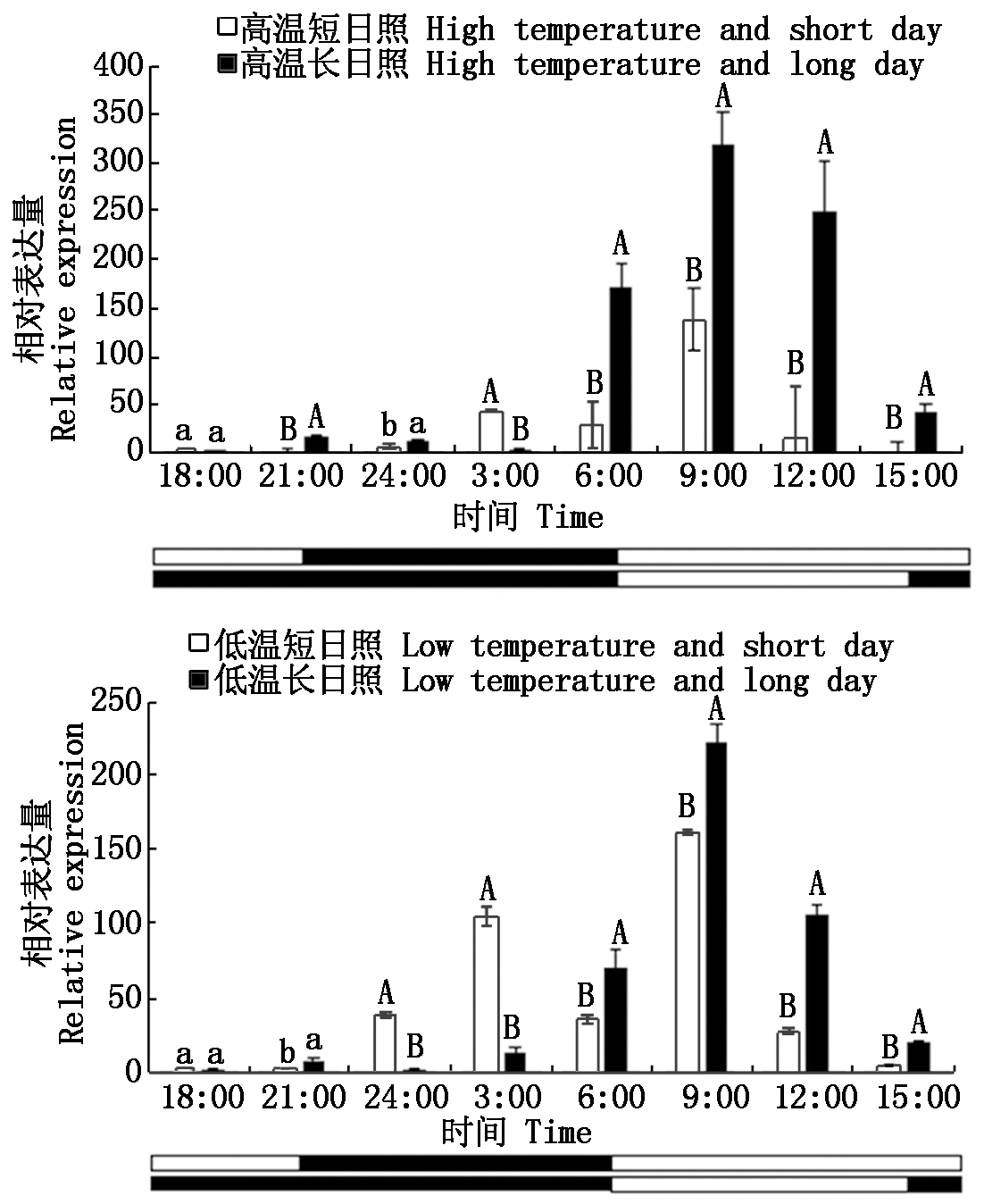
黑条表示黑暗时段,白条表示光照时段;柱上不同大小写字母分别表示在0.01和0.05水平差异显著。图2同。
Black bars represent dark periods,and white bars represent light periods. Values followed by different uppercase letters and lowercase letters above the bar represent significant difference at the 0.01 and 0.05 probability level,respectively.The same as Fig.2.
图1 高温和低温不同光周期条件下SiCCT的昼夜表达分析
Fig.1 The circadian expression of SiCCT at high and low
temperatures under different photoperiod conditions
由图2可知,在短日照条件下,除了18:00,21:00 2个时间点高温低温SiCCT基因表达无显著差异(P>0.05),在其余时间点均表现为低温环境基因表达水平显著或极显著高于高温环境的特点(P<0.05,P<0.01)。无论高温还是低温,SiCCT基因均有2个表达峰,分别在9:00和3:00,为见光3 h之后和黑暗结束前3 h,说明短日照条件下SiCCT基因的表达水平受到了温度的影响,但表达峰出现的时间点不受影响。在长日照条件下,温度的作用与短日照相反,除了18:00高温与低温间SiCCT基因表达无显著差异(P>0.05),3:00低温条件基因表达量极显著高于高温(P<0.01),其余时间点均表现为高温环境基因表达水平显著或极显著高于低温环境的特点(P<0.05,P<0.01),不管是高温还是低温,SiCCT基因在9:00均有1个表达峰。说明温度对SiCCT基因的调控作用受光周期条件的制约。
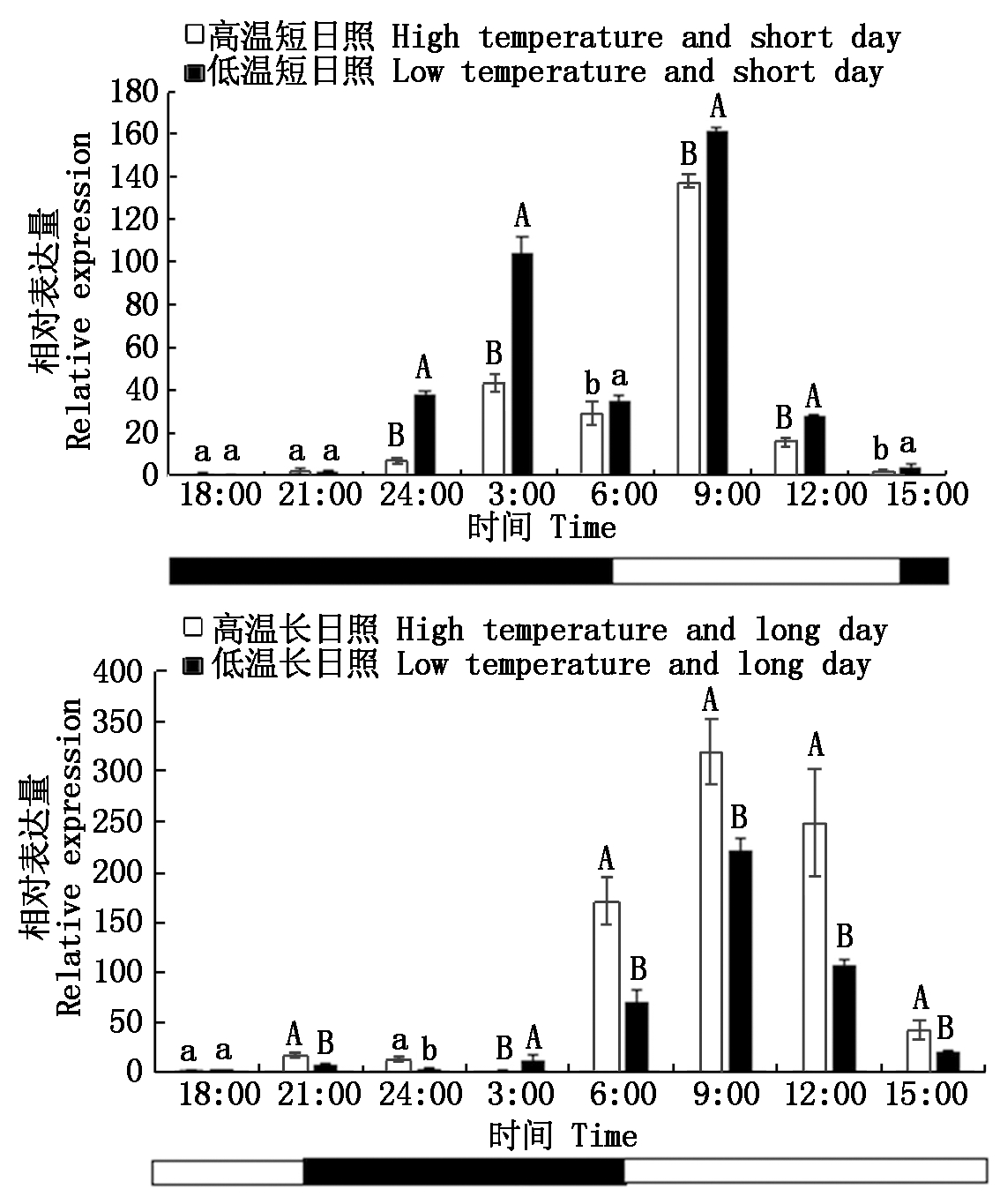
图2 短日照和长日照不同温度条件下
SiCCT的昼夜表达分析
Fig.2 The circadian expression analysis
of SiCCT gene at different temperature
under short-day and long-day conditions
2.2 SiCCT基因的核苷酸多态性分布
SiCCT基因在160份谷子材料中共检测出117个SNP位点,这些位点大部分存在于基因3′非翻区,内含子次之,基因5′非翻译区和编码区最少,这说明SiCCT基因序列的内含子区域和3′非翻译区域核苷酸变化的频率比较高(表2)。
表2 160份谷子材料SiCCT基因的多态性位点
Tab.2 Polymorphic sites of SiCCT gene in 160 millet materials

参数Parameter5′非翻译区5′UTR编码区Coding region内含子区Intron region3′非翻译区3′UTR总数Total序列长度/bp Sequence length3358612 2421 6075 045SNP数目/个 SNP number114867117
2.3 SiCCT基因内SNP的连锁不平衡和单倍型分析
本研究对160份谷子品种的SiCCT基因的SNP位点进行连锁不平衡结构分析,结果显示,该基因所有SNP的连锁情况,各个方块的颜色由浅至深表示连锁程度由低到高,深红色表示完全连锁(图3),发现在整个基因序列上分散分布着一些较强的LD结构。结果表明,SNP-53与SNP-50、SNP-45、SNP-46、SNP-55、SNP-48,SNP-78与SNP-79、SNP-80、SNP-83、SNP-74,SNP-36与SNP-42、SNP-47、SNP-37,SNP-43与SNP-41、SNP-39、SNP-40,SNP-114与SNP-115、SNP-111、SNP-113,SNP-93与SNP-91、SNP-95,SNP-100与SNP-104,SNP-72与SNP-77,SNP-94与SNP-97,SNP-107与SNP-106,SNP-103与SNP-109,SNP-102与SNP-101之间均存在较强的连锁不平衡结构(r2>0.8)。在染色体的某一些区域,有一些位点紧密相关,共同遗传,这样的区域一般称为“单体型块”,而某些区域位点相关性较弱,重组频繁形成重组区块。通过分析得到3个单体型块,在第1个区块具有2个标记,分别是SNP-100、SNP-104,有2个单倍型;第2个区块具有2个标记,分别是SNP-105、SNP-108,有3个单倍型;在第3个区块具有4个标记,分别是SNP-111、SNP-113、SNP-114、SNP-115,显示3个单倍型。

每一个方格的颜色代表2个SNP之间的LD参数r2(P<0.01),深红色方块表示r2=1,白色方块表示r2=0;黑色线框为单体型块区域。
The color of each square represents the LD parameter r2 between two SNPs(P<0.01),dark red squares indicate r2=1,and white squares indicate r2=0;Black wireframes are haploid block regions.
图3 基于SiCCT基因SNP连锁不平衡分析的单体型块图
Fig.3 The patterns of linkage disequilibrium
among the SNPs of SiCCT gene
2.4 谷子材料的群体结构分析
采用Structure 1.8软件对160份谷子品种进行群体结构分析,生成LnP(D)和ΔK随着K值变化的曲线图。最佳K值在2个曲线的拐点处,通过分析比较发现,当K=3时,得到一个稳定的a值,此时ΔK也达到了峰值,因此最佳K值为3,这表明160个品种被划分为3个主要亚群(图4)。图4中3种颜色分别代表了3个不同的亚群,纵坐标表明每个品种所属亚群的概率(即Q值)。再采用TASSEL 5.0软件计算供试品种间的亲缘关系(K),相关亲缘关系信息和群体结构信息应用于候选基因的关联分析中。
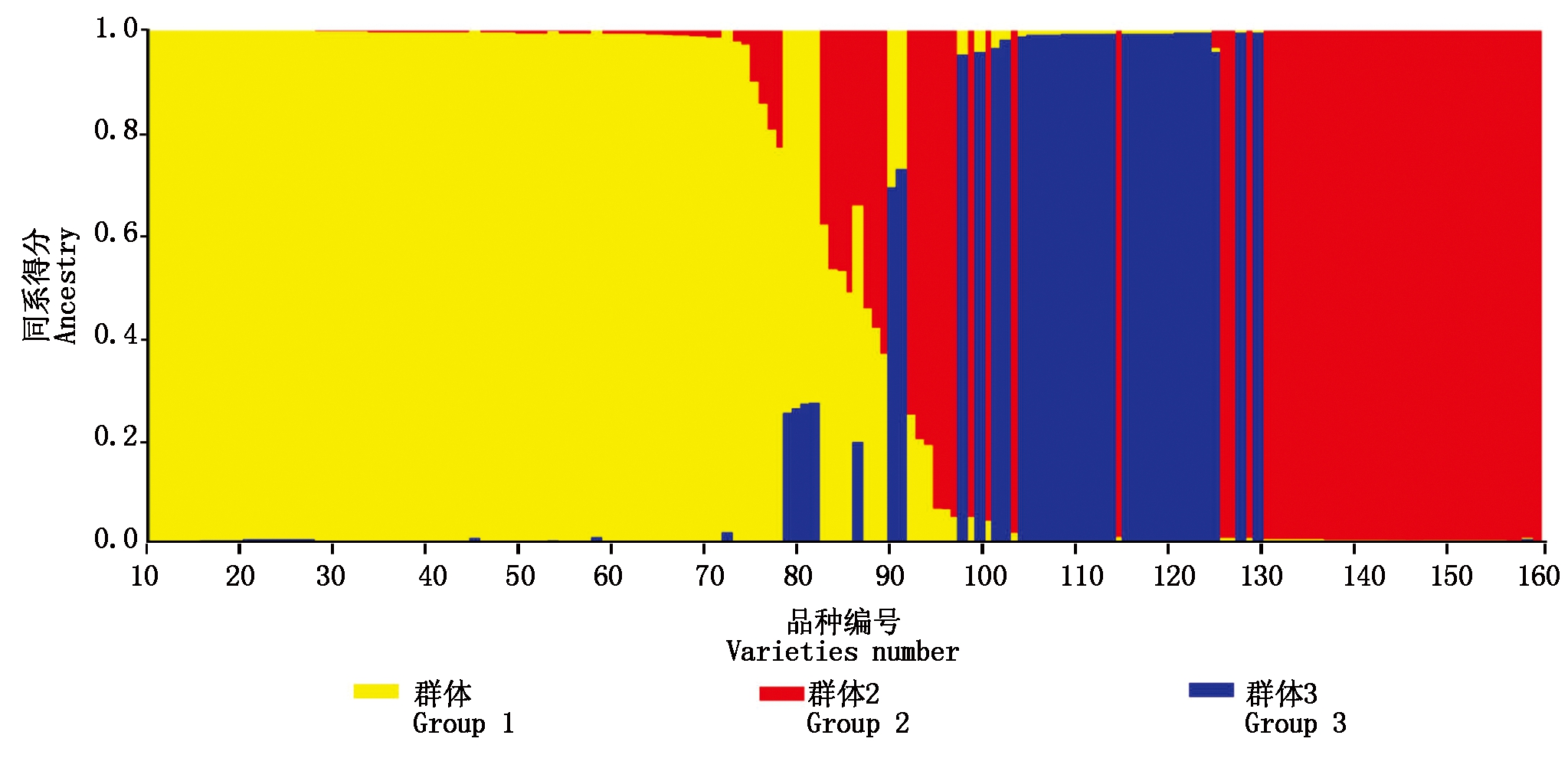
图4 160份谷子材料亚群聚类图
Fig.4 Subgroup clustering of 160 millet materials
2.5 SiCCT基因与主要农艺性状的关联分析
利用TASSEL 5.0软件,将SiCCT基因117个SNP数据与160份谷子材料2 a三地的10个主要农艺性状进行关联分析。结果表明,一共11个SNP位点具有统计学显著性效应(P<0.05),与8个主要农艺性状显著关联,其中在海南乐东短日照环境检测到多数位点与穗粗关联,如SNP-49、SNP-51、SNP-52、SNP-54,而在河南洛阳、吉林吉林长日照环境检测到多数位点与抽穗期、叶片数、株高、穗长、穗码数关联,SNP-49、SNP-52、SNP-70 3个位点例外,其中SNP-52仅在海南乐东环境检测到,SNP-49在河南洛阳也检测到与穗粗关联,SNP-70在吉林吉林检测到与穗粒质量关联(表 3)。11个SNP位点多数能在河南洛阳、吉林吉林或者河南洛阳、海南乐东2个环境中同时检测到,还有一个位点SNP-51在3个环境均能检测到,在海南乐东检测到与穗粗关联,在河南洛阳检测到与穗码数关联,在吉林吉林检测到与叶片数关联。
表3 谷子主要农艺性状与SiCCT基因SNP的关联分析
Tab.3 Association analysis between main agronomic traits and SNPs of SiCCT in foxtail millet
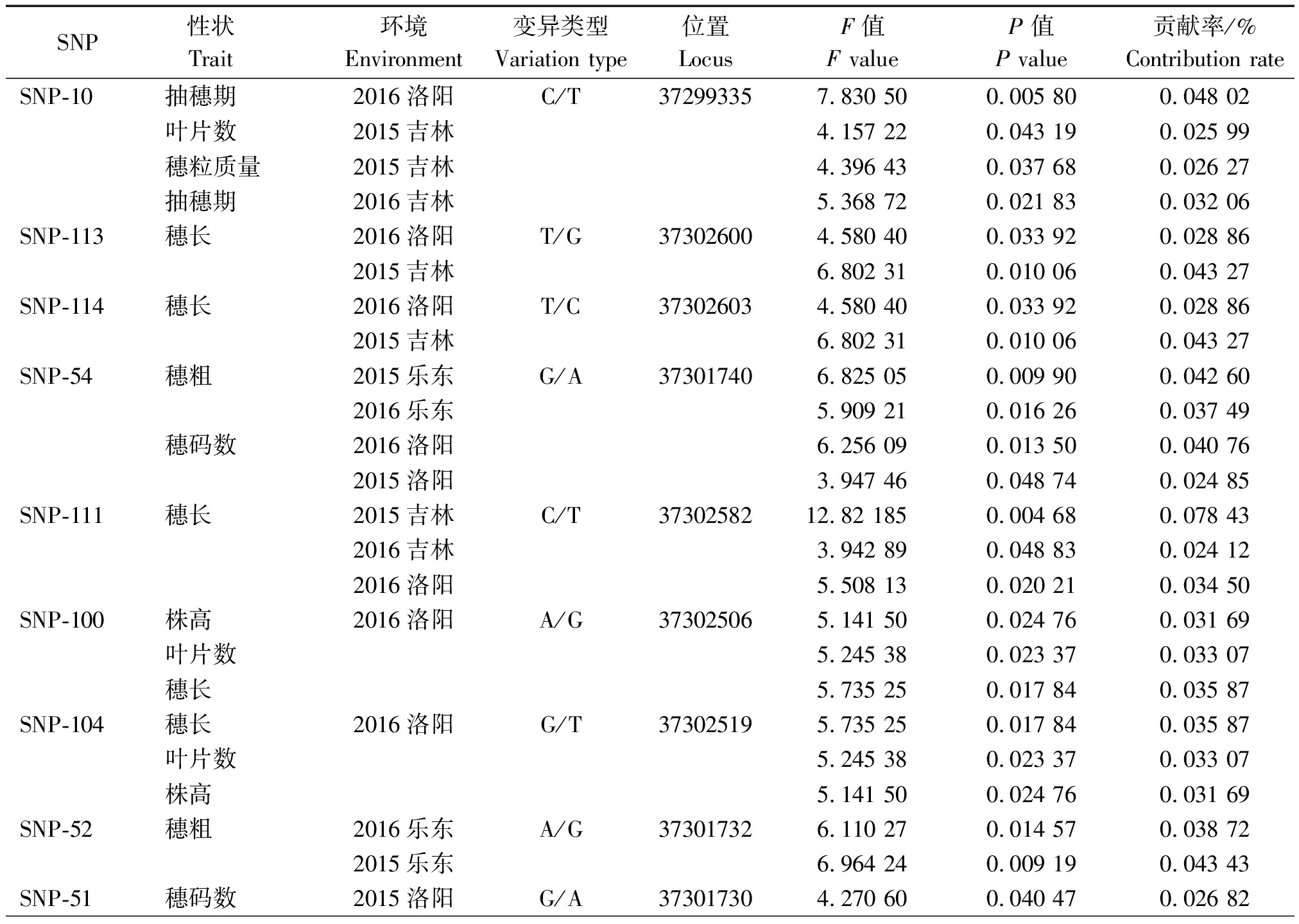
SNP性状 Trait 环境Environment变异类型Variation type位置LocusF值F valueP 值P value贡献率/%Contribution rateSNP-10抽穗期2016洛阳C/T372993357.830 500.005 800.048 02叶片数2015吉林4.157 220.043 190.025 99穗粒质量2015吉林4.396 430.037 680.026 27抽穗期2016吉林5.368 720.021 830.032 06SNP-113穗长2016洛阳T/G373026004.580 400.033 920.028 862015吉林6.802 310.010 060.043 27SNP-114穗长2016洛阳T/C373026034.580 400.033 920.028 862015吉林6.802 310.010 060.043 27SNP-54穗粗2015乐东G/A373017406.825 050.009 900.042 602016乐东5.909 210.016 260.037 49穗码数2016洛阳6.256 090.013 500.040 762015洛阳3.947 460.048 740.024 85SNP-111穗长2015吉林C/T3730258212.82 1850.004 680.078 432016吉林3.942 890.048 830.024 122016洛阳5.508 130.020 210.034 50SNP-100株高2016洛阳A/G373025065.141 500.024 760.031 69叶片数5.245 380.023 370.033 07穗长5.735 250.017 840.035 87SNP-104穗长2016洛阳G/T373025195.735 250.017 840.035 87叶片数5.245 380.023 370.033 07株高5.141 500.024 760.031 69SNP-52穗粗2016乐东A/G373017326.110 270.014 570.038 722015乐东6.964 240.009 190.043 43SNP-51穗码数2015洛阳G/A373017304.270 600.040 470.026 82
表3(续)
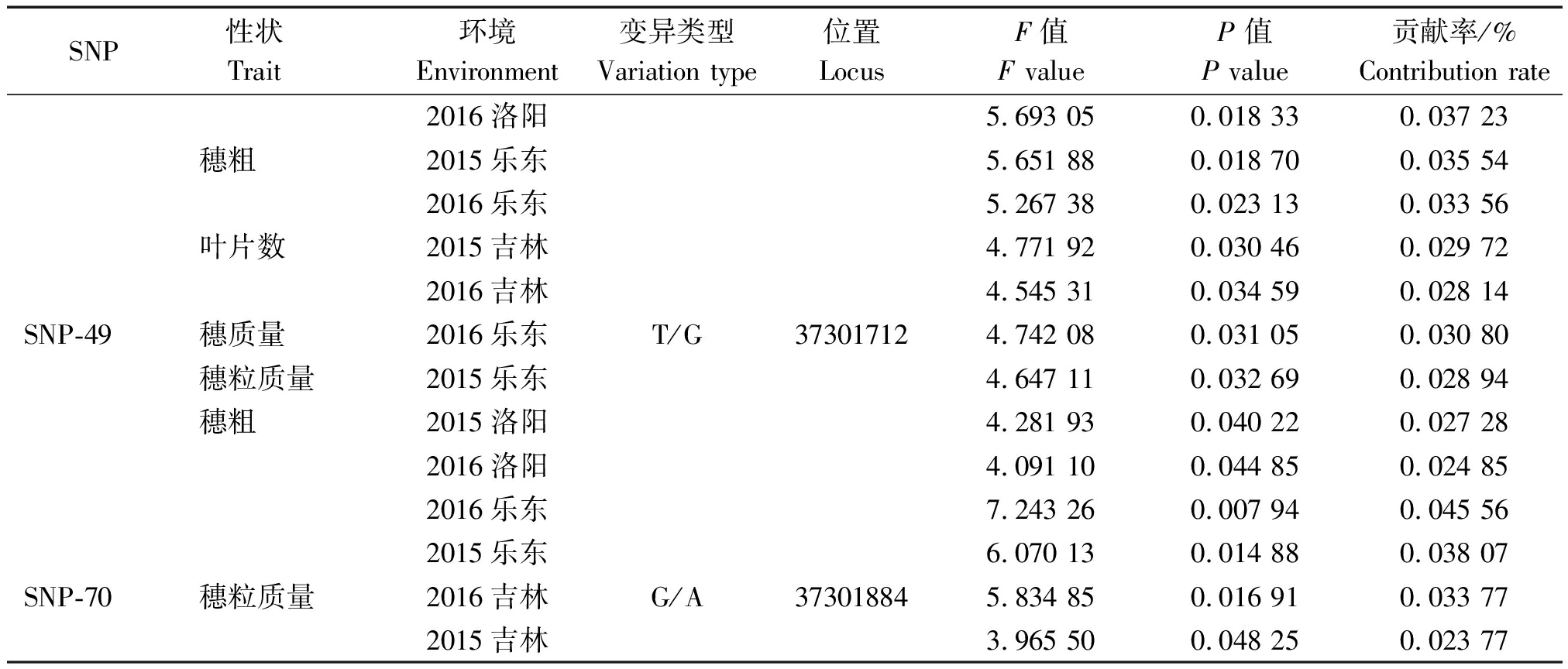
SNP性状 Trait 环境Environment变异类型Variation type位置LocusF值F valueP 值P value贡献率/%Contribution rate2016洛阳5.693 050.018 330.037 23穗粗2015乐东5.651 880.018 700.035 542016乐东5.267 380.023 130.033 56叶片数2015吉林4.771 920.030 460.029 722016吉林4.545 310.034 590.028 14SNP-49穗质量2016乐东T/G373017124.742 080.031 050.030 80穗粒质量2015乐东4.647 110.032 690.028 94穗粗2015洛阳4.281 930.040 220.027 282016洛阳4.091 100.044 850.024 852016乐东7.243 260.007 940.045 562015乐东6.070 130.014 880.038 07SNP-70穗粒质量2016吉林G/A373018845.834 850.016 910.033 772015吉林3.965 500.048 250.023 77
3 结论与讨论
光周期和温度是植物开花的2个关键调控因素,植物的成花转变决定于植物对光周期和温度变化的精确测量[6]。前期的研究表明,SiCCT基因在表达模式上具有24 h的节律性变化,在不同光周期条件下具有不同的昼夜表达模式,不同叶期的相对表达量也有所不同[11]。为了研究温度和光周期互作对谷子SiCCT基因昼夜表达的影响,本试验设置4个不同光温组合处理来分析SiCCT基因在黄毛谷叶片中的24 h昼夜表达情况。结果发现,SiCCT基因在黄毛谷叶片中的表达受光周期和温度共同调节。宋远丽等[12]研究中发现,在长日照条件下,低温(23 ℃)处理促进了水稻开花抑制子Ghd7的表达,表明低温条件和长日照条件对Ghd7的表达具有协同作用。本研究中,黄毛谷叶片SiCCT 基因在4种不同光温组合条件下都呈现24 h昼夜表达规律,不管高温还是低温,长日照条件下SiCCT基因的昼夜整体表达水平高于短日照;短日照条件下,低温处理SiCCT基因的昼夜整体表达水平高于高温处理,然而在长日照条件下,低温处理SiCCT基因的昼夜整体表达水平低于高温处理,这与水稻中获得的结果相反。前期的研究发现,高温与长日照组合对谷子生殖生长抑制作用强于低温长日照组合[11],说明长日照条件下温度的提高使谷子抽穗期延迟,这与SiCCT基因在长日照高温条件下表达量增加是一致的,因此,谷子与水稻基因表达模式的差异可能反映了2种作物光温互作模式的不同。
随着测序技术的进步,近年来全基因组关联分析研究进展很快。通过对不同品种的重测序,结合表型进行连锁不平衡分析可直接快速地找到控制重要性状的位点[13]。利用全基因组关联分析法已经检测到谷子很多农艺性状的关联位点[14-19]。本研究利用前期对160份谷子资源重测序数据和表型性状调查数据,开展基于候选基因的关联分析,结果发现,SiCCT基因共检测到11个SNP位点与8个主要农艺性状显著关联,且多数性状在河南洛阳、吉林吉林长日照环境下检测到,只有穗粗、穗粒质量、穗质量在海南乐东短日照环境检测到,说明SiCCT基因的功能受光周期影响。除影响抽穗期外,光周期途径的许多基因还对其他农艺性状产生影响,具有多重效应。蔡云婷等[20]通过克隆得到了玉米的TOC1基因,其与拟南芥光周期调控基因TOC1高度同源,且位于细胞核中。小麦中光敏色素互作因子TaPIF4基因不仅受光周期调控,而且还可能参与植物ABA、干旱、低温胁迫的信号通路[21]。水稻中Ghd7基因除影响开花期外,还影响穗粒数[11]。在本研究关联到的位点中,SNP-10不仅与抽穗期相关,还与叶片数和穗粒质量显著关联;SNP-100和SNP-104位点在河南洛阳分别同时都关联到株高、叶片数和穗长3个性状;SNP-51位点连续2 a在海南乐东与穗粗相关联,连续2 a在河南洛阳与穗码数关联,还连续2 a在吉林吉林与叶片数关联。以上研究结果说明,SiCCT基因具有一因多效。
综上,SiCCT基因受光周期调控,温度对SiCCT基因的调节作用受光周期条件的制约,具体表现为温度不能改变SiCCT基因于光照期长日照表达水平下高于短日照的基本表达模式,温度在不同光周期条件下的作用效果相反,短日照条件下低温表达水平高于高温,长日照条件下高温表达水平高于低温。SiCCT基因对主要农艺性状的作用受光周期环境的影响,该基因具有多效性。
[1] Li Y P,Xu M L. CCT family genes in cereal crops:A current overview[J].The Crop Journal,2017,5(6):449-458.doi:10.1016/j.cj.2017.07.001.
[2] Shim J S,Kubota A,Imaizumi T. Circadian clock and photoperiodic flowering in Arabidopsis:CONSTANS is a hub for signal integration[J].Plant Physiology,2017,173(1):5-15. doi:10.1104/pp.16.01327.
[3] Cockram J,Jones H,Leigh F J,O′Sullivan D,Powell W,Laurie D A,Greenland A J. Control of flowering time in temperate cereals:Genes,domestication,and sustainable productivity[J].Journal of Experimental Botany,2007,58(6):1231-1244. doi:10.1093/jxb/erm042.
[4] Cockram J,Thiel T,Steuernagel B,Stein N,Taudien S,Bailey P C,O′Sullivan D M. Genome dynamics explain the evolution of flowering time CCT domain gene families in the Poaceae[J].PLoS One,2012,7(9):e45307. doi:10.1371/journal.pone.0045307.
[5] Salomé P A,McClung C R. PSEUDO-RESPONSE REGULATOR 7 and 9 are partially redundant genes essential for the temperature responsiveness of the Arabidopsis circadian clock[J].The Plant Cell,2005,17(3):791-803. doi:10.1105/tpc.104.029504.
[6] Yoo S K,Chung K S,Kim J,Lee J H,Hong S M,Yoo S J,Yoo S Y,Lee J S,Ahn J H. CONSTANS activates SUPPRESSOR OF OVEREXPRESSION OF CONSTANS 1 through FLOWERING LOCUS T to promote flowering in Arabidopsis[J].Plant Physiology,2005,139(2):770-778. doi:10.1104/pp.105.066928.
[7] Cheng X F,Wang Z Y. Overexpression of COL9,a CONSTANS-LIKE gene,delays flowering by reducing expression of CO and FT in Arabidopsis thaliana[J].The Plant Journal,2005,43(5):758-768. doi:10.1111/j.1365-313X.2005.02491.x.
[8] Ledger S,Strayer C,Ashton F,Kay S A,Putterill J. Analysis of the function of two circadian-regulated CONSTANS-LIKE genes[J].The Plant Journal,2001,26(1):15-22. doi:10.1046/j.1365-313x.2001.01003.x.
[9] Jin M L,Liu X G,Jia W,Liu H J,Li W Q,Peng Y,Du Y F,Wang Y B,Yin Y J,Zhang X H,Liu Q,Deng M,Li N,Cui X Y,Hao D Y,Yan J B. ZmCOL3,a CCT gene represses flowering in maize by interfering with the circadian clock and activating expression of ZmCCT[J].Journal of Integrative Plant Biology,2018,60(6):465-480. doi:10.1111/jipb.12632.
[10] Xue W Y,Xing Y Z,Weng X Y,Zhao Y,Tang W J,Wang L,Zhou H J,Yu S B,Xu C G,Li X H,Zhang Q F. Natural variation in Ghd7 is an important regulator of heading date and yield potential in rice[J].Nature Genetics,2008,40(6):761-767. doi:10.1038/ng.143.
[11] 贾小平,袁玺垒,李剑峰,王永芳,张小梅,张博,全建章,董志平. 不同光温条件谷子光温互作模式研究及SiCCT基因表达分析[J].作物学报,2020,46(7):1052-1062. doi:10.3724/SP.J.1006.2020.94144.
Jia X P,Yuan X L,Li J F,Wang Y F,Zhang X M,Zhang B,Quan J Z,Dong Z P. Photo-thermal interaction model under different photoperiod-temperature conditions and expression analysis of SiCCT gene in foxtail millet(Setaria italica L.)[J].Acta Agronomica Sinica,2020,46(7):1052-1062.
[12] 宋远丽,高志超,栾维江. 温度和光周期对水稻抽穗期调控的交互作用[J].中国科学(生命科学),2012,42(4):316-325. doi:10.1007/s11427-012-4300-4.
Song Y L,Gao Z C,Luan W J. Interaction of temperature and photoperiod on regulation of heading date of rice [J].Science in China (Series C),2012,42(4):316-325.
[13] 金亮,包劲松. 植物性状-标记关联分析研究进展[J].分子植物育种,2009,7(6):1048-1063. doi:10.3969/mpb.007.001048.
Jin L,Bao J S. Progress on the trait-marker association analysis in plants[J].Molecular Plant Breeding,2009,7(6):1048-1063.
[14] Jia G Q,Huang X H,Zhi H,Zhao Y,Zhao Q,Li W J,et al. A haplotype map of genomic variations and genome-wide association studies of agronomic traits in foxtail millet(Setaria italica)[J].Nature Genetics,2013,45(8):957-961. doi:10.1038/ng.2673.
[15] Jaiswal V,Gupta S,Gahlaut V,Muthamilarasan M,Bandyopadhyay T,Ramchiary N,Prasad M. Genome-wide association study of major agronomic traits in foxtail millet(Setaria italica L.)using ddRAD sequencing[J].Scientific Reports,2019,9(1):5020. doi:10.1038/s41598-019-41602-6.
[16] Jaiswal V, Bandyopadhyay T, Gahlaut V, Gupta S, Dhaka A, Ramchiary N, Prasad M. Genome-wide association study(GWAS)delineates genomic loci for ten nutritional elements in foxtail millet(Setaria italica L.)[J].Journal of Cereal Science,2019,85:48-55.doi:10.1016/j.jcs.2018.11.006.
[17] 贾小平,张博,董志平,全建章,王永芳,张小梅,袁玺垒,李剑峰,戴凌峰.海南短日照条件下谷子穗部性状的全基因组关联分析[J].河南农业科学,2018,47(9):33-40. doi:10.15933/j.cnki.1004-3268.2018.09.006.
Jia X P,Zhang B,Dong Z P,Quan J Z,Wang Y F,Zhang X M,Yuan X L,Li J F,Dai L F. Genome-wide association analysis of panicle traits of foxtail millet under Hainan short-day condition[J]. Journal of Henan Agricultural Sciences,2018,47(9):33-40.
[18] 袁玺垒,戴凌峰,张小梅,郁飞燕,贾小平. 谷子Hd1-like基因的克隆及其与农艺性状的关联分析[J].河南农业科学,2018,47(6):24-30. doi:10.15933/j.cnki.1004-3268.2018.06.005.
Yuan X L,Dai L F,Zhang X M,Yu F Y,Jia X P. Putative Hd1-like gene cloning and its preliminary association with agronomic traits in foxtail millet(Setaria italica)[J].Journal of Henan Agricultural Sciences,2018,47(6):24-30.
[19] 贾小平,张博,全建章,李剑峰,王永芳,袁玺垒. 不同光周期条件下谷子株高的全基因组关联分析[J].华北农学报,2019,34(4):16-23. doi:10.7668/hbnxb.201750856.
Jia X P,Zhang B,Quan J Z,Li J F,Wang Y F,Yuan X L. Genome-wide association analysis of plant height in foxtail millet under different photoperiod conditions[J].Acta Agriculturae Boreali-Sinica,2019,34(4):16-23.
[20] 蔡云婷,贾力,拓昊苑. 玉米ZmTOC1a、ZmTOC1b基因的克隆、表达及亚细胞定位分析[J].华北农学报,2019,34(4):24-31.doi:10.7668/hbnxb.201751601.
Cai Y T,Jia L,Tuo H Y. Cloning,expression and subcellular localization of ZmTOC1a and ZmTOC1b genes in maize[J].Acta Agriculturae Boreali-Sinica,2019,34(4):24-31.
[21] 李璐,王翔,王状元,曹云,胡格,王留壹,尹钧. 小麦光敏色素互作因子TaPIF4基因的克隆与表达分析[J].华北农学报,2019,34(1):74-82. doi:10.7668/hbnxb.201750739.
Li L,Wang X,Wang Z Y,Cao Y,Hu G,Wang L Y,Yin J. Cloning and expression analysis of phytochrome interacting factor TaPIF4 in wheat[J].Acta Agriculturae Boreali-Sinica,2019,34(1):74-82.