水稻(Oryza sativa L.)是世界上最重要的粮食作物之一,抽穗期(Heading date,HD)(开花时间)是影响水稻产量的一个重要的农艺性状,也是决定水稻季节和地区适应性的重要因素[1]。水稻抽穗期受遗传、光照周期、温度和养分有效性(如氮水平)等内源和外源因素的影响,其中遗传是影响水稻抽穗期的决定因素[2-3]。
国内外遗传研究显示,自分子标记辅助选择(Maker-assisted selection,MAS)应用于水稻抽穗期QTL(Quantitative trait locus)定位以来,已报道超过100个QTLs(包括主效和微效)参与水稻开花时间的调控[4]。过去20 a来已经克隆了诸多水稻开花调控基因,受长日照植物拟南芥成熟开花调控途径的启发,通过分析不同水稻遗传背景下抽穗期基因的表达,提出了调控水稻光周期开花途径的2条基本通路:以Hd1为核心的OsGI-Hd1-Hd3a通路和以Ehd1为核心的Ehd1-Hd3a/RFT1通路[5-6]。水稻促开花基因Hd1含有一个锌指结构域,与拟南芥的CO基因在进化上具有保守性。Ehd1-Hd3a/RFT1通路是水稻在进化上独有的[7-8]。Hd1在光周期敏感性开花作用中起到了权衡的作用,在长日照条件下,Hd1招募Ghd7和DTH8形成不同的二聚体复合体,DTH8-Hd1通过结合Hd3a启动子上的CORE2元件抑制Hd3a的表达[9-10],协同抑制Ehd1-Hd3a/RFT1通路以延迟抽穗。但在短日照条件下,Ghd7的表达量很低,因此,出现Hd1与Ghd7-Hd1、DTH8-Hd1二聚体复合物竞争的现象,降低了Ehd1-Hd3a/RFT1通路被二聚体复合物抑制的作用[11-15]。具有泛素蛋白转移酶活性的HAF1参与Hd1的降解,进而调控Hd1的积累[16]。因此,实现对Hd1的精确调控是培育水稻抽穗期多样性的重要育种目标。此外,在水稻中还存在一些独立于Hd1和Ehd1调控路径的特异开花调控因子,如se14、OsDof12。se14编码含有Jumonji C(JmjC)结构域的组蛋白脱甲基酶,在长日照条件下,se14通过修饰水稻成花素基因RFT1启动子区域H3K4me3去甲基化而抑制水稻抽穗[17]。Dof(DNA-binding one finger)转录因子OsDof12编码一种与DNA结合的单指蛋白,在水稻中的表达没有组织特异性,但有时间特异性,孕育穗时期的表达量最高。受光感影响强烈,在长日照条件下OsDof12通过上调Hd3a的表达促进抽穗,在短日条件下没有明显的作用效果[18-19]。
水稻群体中DTH8、Hd1、se14、OsDof12等抽穗期等位基因的自然变异可能会导致出现不同的水稻抽穗期表型性状。利用5个供体来源不同的SSSLs(Single segment substitution lines),除代换片段外的其余部分与受体亲本HJX74完全相同,因此,它们都是HJX74的近等基因系。本试验采用正向遗传学的方法,结合HJX74和SSSLs在田间的水稻抽穗期表型性状,通过比对CDS序列、氨基酸序列,比较其氨基酸理化性质、表达的差异,进一步分析SSSLs代换片段上抽穗期等位基因对SSSLs抽穗期的影响。
1 材料和方法
1.1 试验材料种植
本试验所用到的SSSLs有:WY18、WY24、WY26、WY60、WY61。SSSLs的具体信息见表1。试验材料种植地点为山东省农业科学院济南饮马泉试验基地(北纬35° 26′,东经116°54′),该地区平均日照时长大于14 h,属于长日照地区。单株间距15 cm,行距25 cm,常规管理。2018年试验材料于5月4日播种,6月4日插秧,7月2日起进行抽穗期调查,每隔3 d进行1次,9月30日结束。2019年试验材料于5月7日播种,6月7日插秧,7月5日起进行抽穗期调查,每隔3 d进行1次,9月30日结束。水稻抽穗期的差异显著性利用t检验来比较,以概率α=0.01为阈值,当SSSLs与HJX74抽穗天数之间的t检验的P ≤ 0.01时,证明该SSSLs的抽穗期变化显著。
表1 本试验涉及的SSSLs信息
Tab.1 The information of SSSLs in this experiment
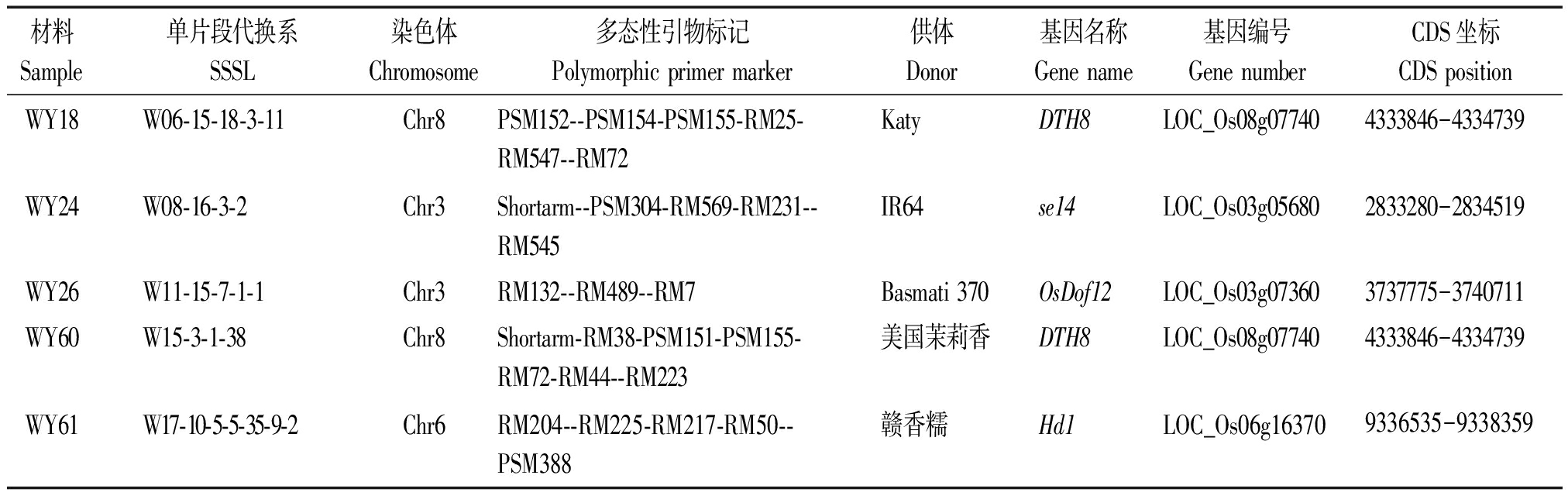
材料Sample单片段代换系SSSL染色体Chromosome多态性引物标记Polymorphic primer marker供体 Donor 基因名称Gene name基因编号Gene numberCDS坐标CDS positionWY18W06-15-18-3-11Chr8PSM152--PSM154-PSM155-RM25-RM547--RM72KatyDTH8LOC_Os08g077404333846-4334739WY24W08-16-3-2Chr3Shortarm--PSM304-RM569-RM231--RM545IR64se14LOC_Os03g056802833280-2834519WY26W11-15-7-1-1Chr3RM132--RM489--RM7Basmati 370OsDof12LOC_Os03g073603737775-3740711WY60W15-3-1-38Chr8Shortarm-RM38-PSM151-PSM155-RM72-RM44--RM223美国茉莉香DTH8LOC_Os08g077404333846-4334739WY61W17-10-5-5-35-9-2Chr6RM204--RM225-RM217-RM50--PSM388赣香糯Hd1LOC_Os06g163709336535-9338359
注:引物标记间的单线(-)表示代换片段;双线(--)表示可能发生重组的区段。
Note:The single lines(-)between primer makers means substituted segments;While the double lines(--)means a interval of recombination possibly.
1.2 主要试剂
RNAiso(TaKaRa),PrimeScriptTM Ⅱ 1st Strand cDNA Synthesis Kit(TaKaRa),Golden Star T6 SuperPCR Mix(1.1×)(TSINGKE),SYBR-Green Mix(TaKaRa),PCR产物纯化回收试剂盒(Axygen®),pMD®18-T Vector Cloning Kit(TaKaRa),DH5α大肠杆菌感受态(Vazyme)。本研究所有引物均由生工(上海)股份有限公司合成。
1.3 引物设计
本试验所用引物均使用Primer Primier 5.0软件设计。根据NCBI(https://www.ncbi.nlm.nih.gov/)已经录入的水稻核苷酸序列,分别在5′UTR和3′UTR设计基因克隆所需的引物,在3′端保守区设计荧光定量所需引物。本试验所用引物见表2。
表2 本试验所用的引物
Tab.2 Primers in this experiment

引物名称Primer name引物序列(5′-3′)Primer sequence (5′-3′)用途 Application DTH8-FATGAAGAGTAGGAAGAGCTA克隆正向引物DTH8-RCTTTAGTCTAAACCATTGCA克隆反向引物se14-FGGCTTGGCTCAGGGGGCT克隆正向引物se14-RTGGGGACATTAGTGTTGT克隆反向引物OsDof12-FGCTTGTGCGAATGAAAAGATT克隆正向引物OsDof12-RCGCTGTGTGAGTTGATGC克隆反向引物Hd1-FGCCATGCGAGGTAGAGGAA克隆正向引物Hd1-RTATATATCTGCTATCCGGAAATTACA克隆反向引物M13-FCGCCAGGGTTTTCCCAGTCACGAC克隆正向引物M13-RAGCGGATAACAATTTCACACAGGA克隆反向引物ubq-FACCCTGGCTGACTACAACATCqRT-PCR正向引物ubq-RAGTTGACAGCCCTAGGGTGqRT-PCR反向引物DTH8-qPCR-FCAGGAGTGCGTGTCGGAGTTqRT-PCR正向引物DTH8-qPCR-RGGTCGTCGCCGTTGATGGTqRT-PCR反向引物OsDof12-qPCR-FATCAAGCTGTTCGGGAAGACqRT-PCR正向引物OsDof12-qPCR-RCGATACGCTCTTGCTCTTGCqRT-PCR反向引物
1.4 基因克隆与序列分析
提取材料的总RNA,通过反转得到cDNA,以此为模板,使用高保真酶对目的基因的编码区进行扩增,扩增程序为:预变性94 ℃ 5 min;循环(变性94 ℃ 45 s,退火58 ℃ 30 s;延伸72 ℃ 45 s)30次;延伸72 ℃ 10 min。PCR产物纯化后与pMD® 18-T Vector Cloning Kit试剂盒的T载体连接,连接体系详见说明书。使用热激法将重组质粒转化DH5α大肠杆菌感受态,涂板,37 ℃过夜培养,挑取单克隆,10 mL LB培养基,37 ℃ 200 r/min过夜培养。经菌液PCR检测,挑取阳性克隆送至山东省农业科学院生物测序中心进行测序。使用ClustalX软件进行CDS序列以及氨基酸序列比对,输出的比对结果使用Genedoc软件分析[20],使用ProtParam(https://web.expasy.org/protscale/,https://web.expasy.org/protparam/)在线分析氨基酸序列理化性质。
1.5 用qRT-PCR验证代换片段上抽穗期等位基因的表达
于2019年播种后45 d(6月21日)起,采集HJX74、WY18、WY26、WY60的第1片展开叶,每隔10 d(标记为45,55,65,75,85,95,105,115,125)采集1次。采集时需使用无RNA酶的EP管液氮速冻,未及时使用的叶片速冻后可放于-80 ℃冰箱保存。用RNAiso法提取叶片总RNA,使用PrimeScriptTM Ⅱ 1st Strand cDNA Synthesis Kit试剂盒合成cDNA的第1链,合成体系详见说明书。qRT-PCR扩增反应体系:cDNA模板 2 μL,SYBR-Green Mix 12.5 μL,正向引物 1 μL,反向引物 1 μL,ddH2O 8.5 μL。qRT-PCR扩增程序:预变性95 ℃ 30 s;95 ℃变性10 s,58 ℃退火15 s,延伸72 ℃ 30 s;40个循环,利用2-ΔΔCt的方法计算基因的相对表达量[21]。
2 结果与分析
2.1 单片段代换系抽穗期表型性状分析
HJX74与SSSLs的抽穗期数据见表3。结果表明,5个SSSLs的抽穗期表型与HJX74有显著差异,WY18、WY60、WY61表现为晚抽穗,WY24、WY26表现为早抽穗。
表3 HJX74与SSSLs的抽穗期
Tab.3 Heading date investigation of HJX74 and SSSLs
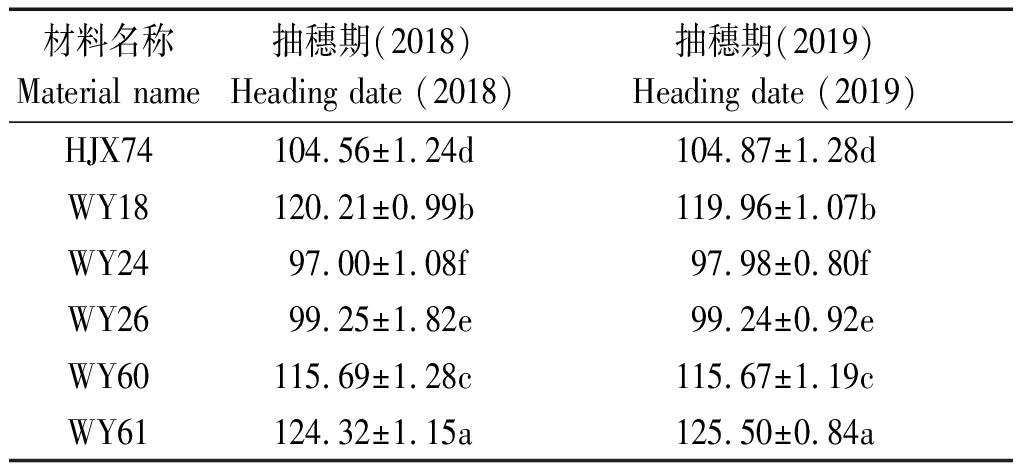
材料名称Material name抽穗期(2018)Heading date (2018)抽穗期(2019)Heading date (2019)HJX74104.56±1.24d104.87±1.28dWY18120.21±0.99b119.96±1.07bWY2497.00±1.08f97.98±0.80fWY2699.25±1.82e99.24±0.92eWY60115.69±1.28c115.67±1.19cWY61124.32±1.15a125.50±0.84a
注:表中数据为平均值±标准差,不同小写字母表示HJX74与SSSLs抽穗期差异达到显著水平,采用t检验分析数据显著性(P<0.05)。
Note:The data in the table are average±s,different lowercase letters represent significant difference heading date between HJX74 and SSSLs,the significance of the data is tested by t-test(P<0.05).
2.2 代换片段上的抽穗期等位基因的变异
WY18 DTH8 CDS序列和HJX74均为894 bp,有4个SNP(Single nucleotide polymorphism)的差异,氨基酸序列全长均为297,有1个氨基酸的替换。WY24 se14 CDS序列全长988 bp,较HJX74相比缺少74 bp。WY26 OsDof12 CDS序列和HJX74均为1 323 bp,有7个SNP的替换,氨基酸序列全长均为440,有3个氨基酸的差异。WY60 DTH8 CDS序列和HJX74均为894 bp,有6个SNP的差异,氨基酸序列为297,有2个氨基酸的替换。WY61 Hd1 CDS序列全长971 bp,较HJX74相比缺少217 bp。HJX74与SSSLs代换片段上抽穗期等位基因的琼脂糖电泳结果见图1,基因结构绘图见图2。

图1 HJX74与SSSLs代换片段上抽穗期等位基因琼脂糖凝胶电泳
Fig.1 The agarose gel electrophoresis of heading date alleles in HJX74 and SSSLs substitution fragments
2.3 单片段代换系代换片段上抽穗期等位基因氨基酸理化性质分析
HJX74&WY18 DTH8、HJX74&WY26 OsDof12和HJX74&WY60 DTH8 3组编码的氨基酸序列理化性质没有明显的差异。HJX74&WY24 se14、HJX74&WY61 Hd1 2组的CDS比对序列缺失差异显著,故不再进行氨基酸理化性质分析。
2.4 抽穗期等位基因的qRT-PCR分析
对HJX74和WY18 DTH8、WY26 OsDof12、WY60 DTH8进行RNA表达水平的检测。提取了HJX74和WY18、WY26、WY60在45,55,65,75,85,95,105,115,125 d叶片的总RNA,通过qRT-PCR技术定量分析代换片段上抽穗期等位基因的表达。结果发现,WY18 DTH8的表达整体高于HJX74。WY60 DTH8的表达曲线与HJX74基本相似,符合两者的孕育穗时期。HJX74与WY18、WY26、WY60代换片段上抽穗期等位基因的表达曲线见图3。
3 结论与讨论
我国约60%以上的人口都以稻米为主食,据国家统计局公报(http://www.stats.gov.cn/tjsj/zxfb/202012/t20201210_1808377.html),2020年全国水稻种植面积3 007.6万hm2,单产达到7 044 kg/hm2,总产达21 186.0万t,连续第10年稳定在2亿t以上水平。随着耕地面积持续减少,提高水稻单产已成为维持我国粮食安全最现实的途径,选择适宜抽穗期的水稻品种是特定种植区水稻高产的关键,因此,精确调控水稻抽穗期对培育优良性状的水稻品种具有重要的意义[3]。
水稻是一种兼性短日照植物,在水稻的光周期敏感阶段,在外源影响因素(营养条件、光周期、温度)一致的情况下,内源影响因素基因型遗传分化是
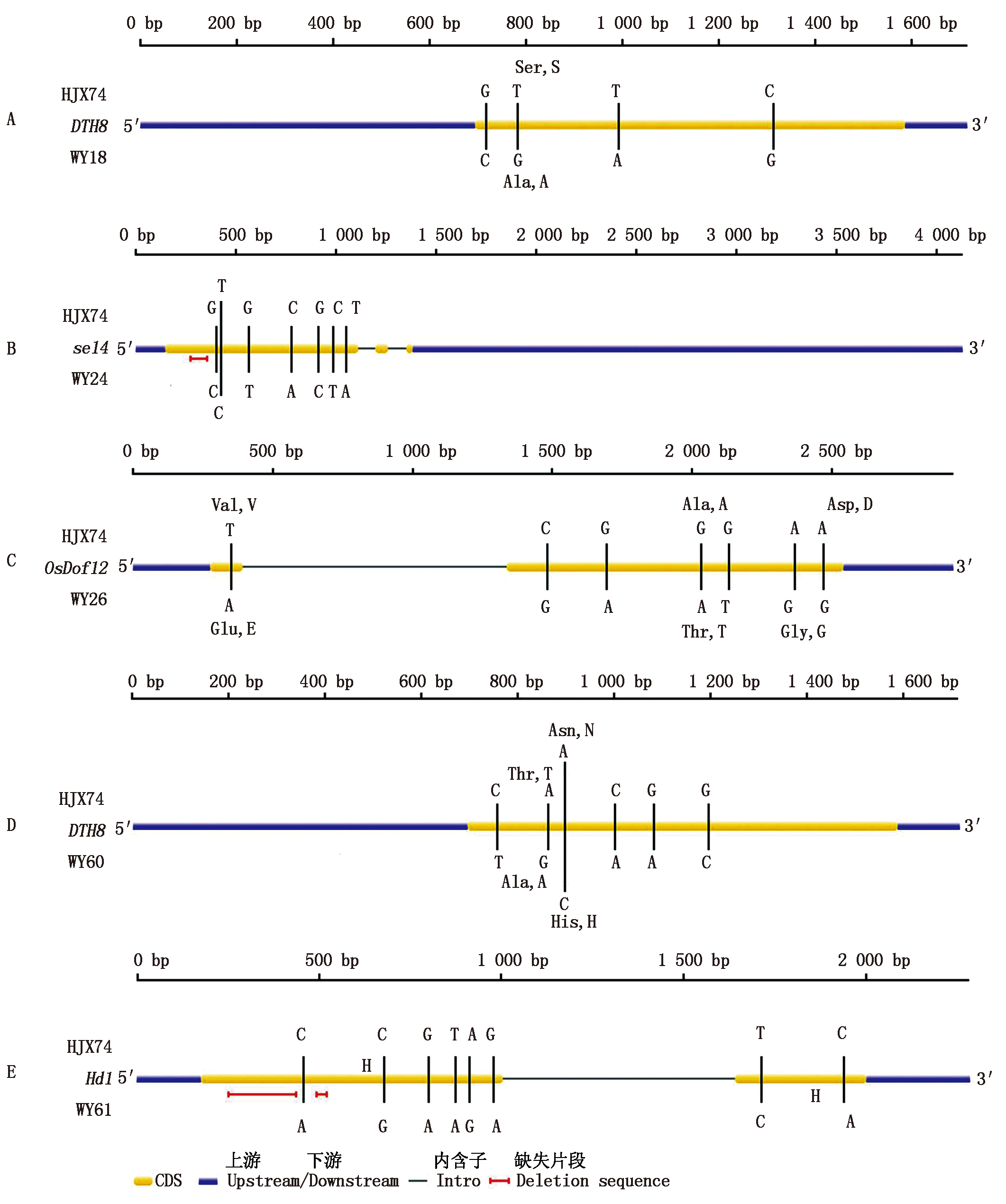
黑色垂直线表示SNP位点;红色水平线表示相应位点在HJX74或SSSLs中存在缺失变异;红色字表示相应位点SNP导致的氨基酸替换。
The vertical black bars represent SNP variations; The horizontal red bars indicate deletion variations of HJX74 or SSSLs
depending on which side; Words in red indicate the amino acid polymorphic site derived from the SNP nearby.
图2 HJX74与SSSLs代换片段上抽穗期等位基因的多态性分析
Fig.2 Polymorphism analysis of the heading date alleles between HJX74 and SSSLs substitution fragments
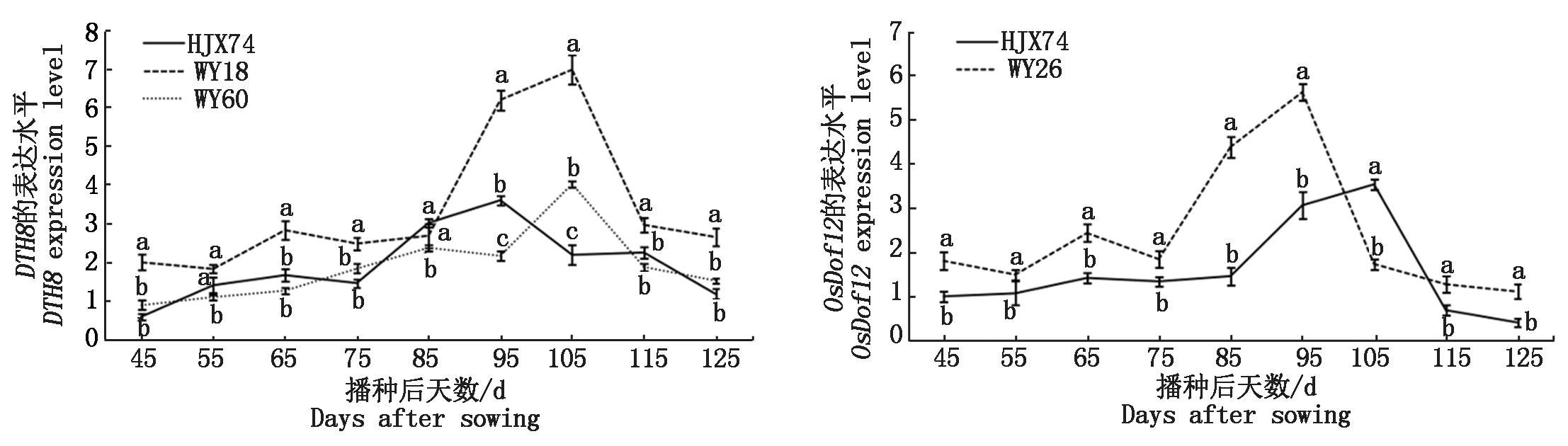
不同小写字母表示HJX74与SSSLs抽穗期差异达到显著水平,采用t检验分析数据显著性(P<0.05)。
Different lowercase letters represent significant difference heading date between HJX74 and SSSLs, the significance of the data is tested by t-test (P<0.05).
图3 HJX74和SSSLs代换片段上抽穗期等位基因的表达分析
Fig.3 Expression analysis of heading date alleles between HJX74 and SSSLs substitution fragments
决定水稻光周期响应差异的一个基本因素,会导致水稻抽穗期发生广泛的变异。为了研究5个代换片段来源不同的SSSLs和受体亲本HJX74抽穗期差异的原因,利用正向遗传学的研究方法,结合2018-2019年田间种植试验的抽穗期表型性状,对代换片段上的抽穗期等位基因的变异进行研究,表明WY18 DTH8、WY26 OsDof12的变异可能并不是引起抽穗期改变的主要原因,可能是该基因的上游调节区域对该基因的表达进行了调控,进而改变其抽穗期。WY24、WY61抽穗期的变化可能是由其代换片段上等位基因的突变所致,WY60 DTH8的变异可能并不是其抽穗期改变的原因。
对受体亲本HJX74与SSSLs代换片段上已克隆的水稻抽穗期等位基因进行遗传分化的研究,通过发现等位基因的差异,分析不同等位基因型对抽穗期表型性状的影响,对于不同等位基因在水稻育种的有效利用具有重要意义。另外,对于WY18、WY26和WY60,下一步试验可以通过配置合适的杂交组合,进一步挖掘代换片段上可能存在的新的抽穗期基因,以希望能够对水稻抽穗期主基因或QTLs的作用机制方面得到更多的启示。
[1] Gao H,Jin M N,Zheng X M,Chen J,Yuan D Y,Xin Y Y,Wang M Q,Huang D Y,Zhang Z,Zhou K N,Sheng P K,Ma J,Ma W W,Deng H F,Jiang L,Liu S J,Wang H Y,Wu C Y,Yuan L P,Wan J M. Days to heading 7,a major quantitative locus determining photoperiod sensitivity and regional adaptation in rice[J].PNAS,2014,111(46):16337-16342. doi:10.1073/pnas.1418204111.
[2] Hori K,Matsubara K,Yano M. Genetic control of flowering time in rice:integration of mendelian genetics and genomics[J].Theoretical and Applied Genetics,2016,129(12):2241-2252. doi:10.1007/s00122-016-2773-4.
[3] Zhou S R,Zhu S S,Cui S,Hou H G,Wu H Q,Hao B Y,Cai L,Xu Z,Liu L L,Jiang L,Wang H Y,Wan J M. Transcriptional and post-transcriptional regulation of heading date in rice[J].New Phytologist,2021,230(3):943-956. doi:10.1111/nph.17158.
[4] 徐仅婷,李广贤,柳絮,蔚亦沛,姚方印,刘开启. 水稻抽穗期 QTL 鉴定及重叠群作图[J].山东农业科学,2014,46(2):1-7. doi:10.3969/j.issn.1001-4942.2014.02.001.
Xu J T,Li G X,Liu X,Wei Y P,Yao F Y,Liu K Q. QTL identification of rice heading date and contig mapping[J].Shandong Agricultural Sciences,2014,46(2):1-7.
[5] Sun C H,Chen D,Fang J,Wang P R,Deng X J,Chu C C. Understanding the genetic and epigenetic architecture in complex network of rice flowering pathways[J].Protein & Cell,2014,5(12):889-898. doi:10.1007/s13238-014-0068-6.
[6] Shim J S,Jang G. Environmental signal-dependent regulation of flowering time in rice[J].International Journal of Molecular Sciences,2020,21(17):6155. doi:10.3390/ijms21176155.
[7] Galbiati F,Chiozzotto R,Locatelli F,Spada A,Genga A,Fornara F. Hd3a,RFT1 and Ehd1 integrate photoperiodic and drought stress signals to delay the floral transition in rice[J].Plant,Cell & Environment,2016,39(9):1982-1993. doi:10.1111/pce.12760.
[8] Cho L H,Yoon J,Pasriga R,An G. Homodimerization of Ehd1 is required to induce flowering in rice[J].Plant Physiology,2016,170(4):2159-2171. doi:10.1104/pp.15.01723.
[9] Du A P,Tian W,Wei M H,Yan W,He H,Zhou D,Huang X,Li S G,Ouyang X H. The DTH8-Hd1 module mediates day-length-dependent regulation of rice flowering[J].Molecular Plant,2017,10(7):948-961. doi:10.1016/j.molp.2017.05.006.
[10] Zhu S S,Wang J C,Cai M H,Zhang H,Wu F Q,Xu Y,Li C N,Cheng Z J,Zhang X,Guo X P,Sheng P K,Wu M M,Wang J L,Lei C L,Wang J,Zhao Z C,Wu C Y,Wang H Y,Wan J M. The OsHAPL1-DTH8-Hd1 complex functions as the transcription regulator to repress heading date in rice[J].Journal of Experimental Botany,2017,68(3):553-568. doi:10.1093/jxb/erw468.
[11] Zong W B,Ren D,Huang M H,Sun K L,Feng J L,Zhao J,Xiao D D,Xie W H,Liu S Q,Zhang H,Qiu R,Tang W J,Yang R Q,Chen H Y,Xie X R,Chen L T,Liu Y G,Guo J X. Strong photoperiod sensitivity is controlled by cooperation and competition among Hd1,Ghd7 and DTH8 in rice heading[J].New Phytologist,2021,229(3):1635-1649. doi:10.1111/nph.16946.
[12] Adachi S,Yoshikawa K,Yamanouchi U,Tanabata T,Sun J,Ookawa T,Yamamoto T,Sage R F,Hirasawa T,Yonemaru J. Fine mapping of carbon assimilation rate 8,a quantitative trait locus for flag leaf nitrogen content,stomatal conductance and photosynthesis in rice[J].Frontiers in Plant Science,2017,8:60. doi:10.3389/fpls.2017.00060.
[13] Zhang C Y,Liu J,Zhao T,Gomez A,Li C,Yu C S,Li H Y,Lin J Z,Yang Y Z,Liu B,Lin C T. A drought-inducible transcription factor delays reproductive timing in rice[J].Plant Physiology,2016,171(1):334-343. doi:10.1104/pp.16.01691.
[14] Goretti D,Martignago D,Landini M,Brambilla V,Gómez-Ariza J,Gnesutta N,Galbiati F,Collani S,Takagi H,Terauchi R,Mantovani R,Fornara F. Transcriptional and post-transcriptional mechanisms limit heading date 1(Hd1)function to adapt rice to high latitudes[J].PLoS Genetics,2017,13(1):e1006530. doi:10.1371/journal.pgen.1006530.
[15] Nemoto Y,Nonoue Y,Yano M,Izawa T. Hd1,a CONSTANS ortholog in rice,functions as an Ehd1 repressor through interaction with monocot-specific CCT-domain protein Ghd7[J].The Plant Journal,2016,86(3):221-233. doi:10.1111/tpj.13168.
[16] Zhu C M,Peng Q,Fu D B,Zhuang D X,Yu Y M,Duan M,Xie W B,Cai Y H,Ouyang Y D,Lian X M,Wu C Y. The E3 ubiquitin ligase HAF1 modulates circadian accumulation of EARLY FLOWERING3 to control heading date in rice under long-day conditions[J].The Plant Cell,2018,30(10):2352-2367. doi:10.1105/tpc.18.00653.
[17] Yokoo T,Saito H,Yoshitake Y,Xu Q,Asami T,Tsukiyama T,Teraishi M,Okumoto Y,Tanisaka T. Se14,encoding a JmjC domain-containing protein,plays key roles in long-day suppression of rice flowering through the demethylation of H3K4me3 of RFT1[J].PLoS One,2014,9(4):e96064. doi:10.1371/journal.pone.0096064.
[18] Li D J,Yang C H,Li X B,Ji G B,Zhu L H. Sense and antisense OsDof12 transcripts in rice[J].BMC Molecular Biology,2008,9:80. doi:10.1186/1471-2199-9-80.
[19] Li D J,Yang C H,Li X B,Gan Q,Zhao X F,Zhu L H. Functional characterization of rice OsDof12[J].Planta,2009,229(6):1159-1169. doi:10.1007/s00425-009-0893-7.
[20] Larkin M A,Blackshields G,Brown N P,Chenna R,McGettigan P A,McWilliam H,Valentin F,Wallace I M,Wilm A,Lopez R,Thompson J D,Gibson T J,Higgins D G. Clustal W and Clustal X version 2.0[J].Bioinformatics,2007,23(21):2947-2948. doi:10.1093/bioinformatics/btm404.
[21] Livak K J,Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J].Methods,2001,25(4):402-408. doi:10.1006/meth.2001.1262.