肉牛是畜牧业中重要的经济动物,骨骼肌的生长发育不仅会影响肉牛的产肉量,还会对牛肉品质有一定的影响,进而改变食用口感。因此,调节肌肉发育过程、增加肌肉量是肉牛遗传育种工作的主要研究内容。当前,越来越多的研究集中在动物骨骼肌生长发育的机理上,在这一过程中,microRNAs(miRNAs)的研究为热点。
miRNAs是一类进化上保守的内源性非编码小RNA分子,长度约为18~22个碱基,广泛存在于动物、植物以及病毒等生物的基因组中,通过在转录后水平调控基因的表达,成为许多生物过程的重要调节因子。研究证明,家畜骨骼肌发育的几乎所有方面都涉及miRNAs的调控,包括细胞迁移、增殖、分化和凋亡等[1]。已有研究表明,许多miRNAs以组织特异性或阶段特异性的方式表达,而最具特征的肌肉特异性miRNAs是miR-1、miR-206和miR-133家族,它们在心肌和骨骼肌中特异性表达,在骨骼肌卫星细胞的增殖和分化中发挥关键作用[2]。Safa等[3]研究报道,miR-1可通过靶向PAX7和HDAC4的3′UTR区,抑制PAX7和HDAC4基因的表达,促进牛骨骼肌卫星细胞的肌原性分化。过表达miR-1/miR-206,可以使TNF-α表达下调,降低其对成肌细胞分化的抑制程度[4]。Mok等[5]研究表明,miR-133靶向调控Hedgehog通路中Gli3基因,抑制miR-133表达会破坏细胞增殖、细胞外基质沉积和上皮化,进而损害肌节的形成和生长,调控胚胎肌肉发生。miR-133通过抑制山羊骨骼肌卫星细胞HDAC4和SRF基因的翻译,参与调控山羊骨骼肌细胞增殖和分化过程[6],通过调控TGFBR1的表达,参与鸭成肌细胞的增殖分化[7]。miR-133a通过靶向BIRC5基因,抑制骨骼肌细胞凋亡,影响肌肉质量[8]。此外,有的研究报道,miR-133a可通过降解生长激素基因的转录抑制子-胰岛素样生长因子 1(IGF1),进而造成肌肉肥大等异常现象[9]。
目前,研究虽然证实了bta-miR-133a参与调控骨骼肌的生长发育,但并未见bta-miR-133a调控靶基因参与成肌细胞增殖与分化过程的直接相关报道。本实验室(动物胚胎工程中心)选用布莱凯特黑牛和鲁西黄牛背最长肌构建转录组高通量测序的sRNA数据库[10-11],布莱凯特黑牛是运用体细胞克隆技术、超数排卵技术、胚胎分割及胚胎冷冻技术以及杂交育种与分子标记辅助育种的方法培育而成的优良肉牛新种质[12-13];鲁西黄牛是我国优秀的地方良种,素有“五花三层肉”和“山东膘牛”的美称。本试验基于布莱凯特黑牛和鲁西黄牛的数据库,利用生物信息学方法预测bta-miR-133a的靶基因,分析其保守性,并对靶基因进行GO富集和KEGG通路富集分析,筛选并确定目标miRNA(bta-miR-133a);以C2C12细胞作为细胞模型进行功能验证,转染bta-miR-133a 模拟剂、模拟剂阴性对照、bta-miR-133a抑制剂和抑制剂阴性对照,2%马血清诱导分化,检测靶基因和增殖分化标志性基因的表达量变化,探究bta-miR-133a通过调控靶基因参与肉牛骨骼肌发育过程的分子机制,旨在为揭示bta-miR-133a调节肉牛骨骼肌生长发育的机制提供科学依据,为肉牛肉质性状的分子选育提供参考。
1 材料和方法
1.1 主要材料与试剂
选取的12月龄布莱凯特黑牛和鲁西黄牛各3头,采集背最长肌组织置于液氮保存。小鼠C2C12细胞系和bta-miR-133a转染载体,购自普健生物(武汉)科技有限公司;miRNA反转录试剂盒,购自TaKaRa Bio USA;Lipofectamine 3000试剂,购自赛默飞世尔科技;CCK-8检测试剂盒(CK04),购自东仁化学科技(上海)有限公司。
1.2 小RNA文库的构建测序与质量控制
用TRIzol试剂(Invitrogen)提取总RNA,使用RIN≥7.0且A260/280的比值在0.8~2.0的高质量RNA样品,构建序列文库。采用Illumina Genome Analyzer对文库进行测序。原始测序数据由Fastp软件处理,删除包含适配器,ploy-N和低质量读取数据,获得干净原始读取。所有下游分析均基于高质量的数据。通过比对分析软件Bowtie 1.1.2(设置允许一个错配)将sRNA测序的干净读数定位到参考基因组(Bos_taurus.NCBI.795.1)进行比对。
1.3 bta-miR-133a的生物信息学分析
为了探索 bta-miR-133a 的生物学功能,从miRBase(http://www.mirbase.org/)检索牛(Bos taurus,Bta)、人(Homo sapiens,Hsa)、黑猩猩(Pan troglodytes,Ptr)、大鼠(Rattus norvegicus,Rno)、小鼠(Mus musculus,Mmu)、马(Equus caballus,Eca)的miR-133a成熟序列,分析其保守性。使用 TargetScan 7.2(http://www.targetscan.org/vert_72/)软件对bta-miR-133a进行靶基因预测,以物种“cow”为背景,选择目的miR-133a,Total context++score数值大小,代表miRNA与该靶基因结合的概率;根据Aggregate PCT,代表靶基因的保守定位可能性,获得预测靶基因。使用DAVID 6.8(https://david.ncifcrf.gov/)和KOBAS 3.0(http://kobas.cbi.pku.edu.cn/kobas3/)在线软件对预测所有bta-miR-133a靶基因进行GO分析和KEGG 通路富集分析。使用DAVID 6.8上传靶基因信息,选择背景基因物种“Bos taurus”,使用“Functional Annotation Tool”选择“KEGG_PATHWAY” 进行通路富集分析。使用KOBAS 3.0中的“Gene-list Enrichment”,上传基因信息,选择“GO”数据库进行功能富集分析。
1.4 双荧光素酶报告试验
使用胶回收纯化试剂盒(Shanghai Generay Biotech Co.,Ltd.)对PCR产物进行回收,将胶回收产物连接在pSI-Check2载体上,连接产物经感受态细胞DH5α转化后,37 ℃ 250 r/min摇菌14 h,用菌液进行PCR鉴定,将阳性克隆菌液进行测序。构建靶基因PAX7的双荧光素酶突变载体。HEK-293T细胞在96孔板中避光接种培养,同时将DMEM、0.8 mg/mL转染试剂(普健生物科技有限公司)、0.16 μg PAX7质粒和0.5 μg bta-miR-133a或NC mimic共转染到细胞中,转染48 h后收集细胞检测。使用双荧光素酶检测试剂盒(Promega Dual-Luciferase System)检测双荧光素酶活性。
1.5 成肌细胞转染及诱导分化
正常培养的C2C12细胞,吸弃原培养液,PBS洗后加入胰酶消化2 min后终止消化,剩余细胞悬液用培养基重悬,4 000个/孔(96孔板)进行铺板(200 μL),3个复孔,5% CO2,37 ℃过夜培养。当细胞密度达到90%时,用Lipofectamine 3000试剂转染浓度为20 μmol/L的空白组(Blank)、模拟剂阴性对照组(NC mimic)、bta-miR-133a模拟剂(bta-miR-133a mimic)、抑制剂阴性对照组(NC inhibitor)和bta-miR-133a抑制剂组(bta-miR-133a inhibitor),转染6 h后,换GM培养基,12 h后用 2%马血清诱导分化,分别再培养24(1 d),48(2 d),120(5 d),144 h(6 d)观察细胞生长状态,48 h后提取细胞总RNA及总蛋白。
1.6 Cell Counting Kit-8(CCK-8)细胞增殖率检测
转染后24,48,72 h分别从培养箱中取出1个96孔板,向每孔中加入20 μL CCK8 溶液,培养4 h后,检测450 nm处的OD值。
1.7 实时荧光定量PCR(qRT-PCR)
随机选取4个miRNAs(bta-miR-133a、bta-miR-196b、bta-miR-200b、bta-miR-11977)进行qRT-PCR检测验证。检测成肌细胞增殖分化标志基因PCNA、CCND1、Myh1、Myod1和靶基因PAX7的表达量。使用TRIzol法提取空白组(Blank)、模拟剂阴性对照组(NC mimic)、bta-miR-133a模拟剂(bta-miR-133a mimic)、抑制剂阴性对照组(NC inhibitor)和bta-miR-133a抑制剂组(bta-miR-133a inhibitor)5组转染48 h后细胞总RNA。将提取的细胞总RNA逆转录为cDNA,以cDNA为模板进行荧光定量PCR。荧光定量PCR反应体系为20 μL:RNA-free Water 7.8 μL,上游引物0.6 μL(10 μmol/L),下游引物0.6 μL(10 μmol/L),SYBRGreen mix 10 μL,cDNA 1 μL(80 ng/μL)。PCR反应程序:94 ℃预变性10 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸40 s,40个循环[14-15]。以GAPDH作为内参基因,对PAX7、PCNA、CCND1、Myh1、Myod1基因进行标准化;以U6作为内参,对bta-miR-196b、bta-miR-200b、bta-miR-11977、Novel_445、Novel_130、Novel_334进行标准化,采用2-ΔΔCT值法计算。实时荧光定量PCR引物序列见表1。
表1 实时荧光定量PCR引物
Tab.1 qRT-PCR primers

基因/miRNA名称Gene/miRNA name引物序列(5′-3′)Sequence of primers产物长度/bpProducts size退火温度/℃Melting temperaturePAX7F: CCTCGCAGTCGTACTGTCC10259R: AATCAACAGCAGTCTGGCCGPCNAF:AGAAAGTGCTGGAGGC12752R:TCGGAGCGAAGGGTTACCND1F:CTGGTCCTGGTGAACAAA14854R:TGGCACAGAGGGCAACMyh1F:AGGTTGCATCTCCAAGGCAG24760R:GTCTTGGCCGTCACCTTCCMyod1F:GCGGAACTGCTACGACC28460R:CCTGGTAAATCGGGTTGGGAPDHF: GATGCTGGTGCTGAGTATGT10460R: GCAGAAGGTGCAGAGATGATbta-miR-196bF: AGGTAGTTTCATGTTGTTGGGA60R: GTATCAACGCAGAGTACTTTbta-miR-200bF: CTAATACTGCCTGGTAATGATGA58R: GTATCAACGCAGAGTACTTTbta-miR-11977F: TGTCTCAGTTCAGCAGGAAGAA59R: GTATCAACGCAGAGTACTTTNovel_445F: ATAGGGTTCTATTGTTGGTGGA59R: GTATCAACGCAGAGTACTTTNovel_130F: TTGCTCGATCTGACGAAGAGA60R: GTATCAACGCAGAGTACTTTNovel_334F: AGTGATGTCTTATGGTTGTGTGA58R: GTATCAACGCAGAGTACTTTU6F: GGAACGATACAGAGAAGATTAGC60R: TGGAACGCTTCACGAATTTGCG
1.8 数据分析
采用 SPSS 20. 0 统计分析软件中的One-way ANOVA进行方差分析和显著性检验。
2 结果与分析
2.1 背最长肌组织中 miRNA的筛选
使用Illumina基因组分析仪系统测序后,构建sRNA文库。获得原始数据,去除低质量reads(质量值低于20、碱基数超过1或含有N的reads)、不带有3′接头的reads、含有5′接头的reads、插入片段缺失的reads和长度小于18 nt的reads以及含有polyA的reads(一条reads中大于70%的碱基都是A)后得到高质量片段(clean reads)。通过比对分析软件Bowtie(设置允许一个错配)将sRNA测序的clean reads定位到参考基因组上,共获得26 061 624个clean reads。根据比对上参考序列的reads,与miRBase数据库的牛物种的参考序列进行比较,匹配到已知miRNA上的Total和Unique clean reads的总数量分别为29 849 254,37 946。根据差异miRNA筛选标准:|log2(Fold_Change)|≥1且 Q≤0.05,获得差异表达的miRNA 71个,其中上调39个,下调32个,其结果见图1-A。根据与肉牛骨骼肌发育相关的功能数据分析,筛选出表达量(Transcripts per million,TPM)前10位的差异miRNA分别为bta-miR-1、bta-miR-133a、bta-miR-206、bta-miR-26a、bta-miR-27b、bta-miR-143、bta-miR-378、bta-miR-10b、bta-let-7g、bta-miR-499,其结果见图1-B。
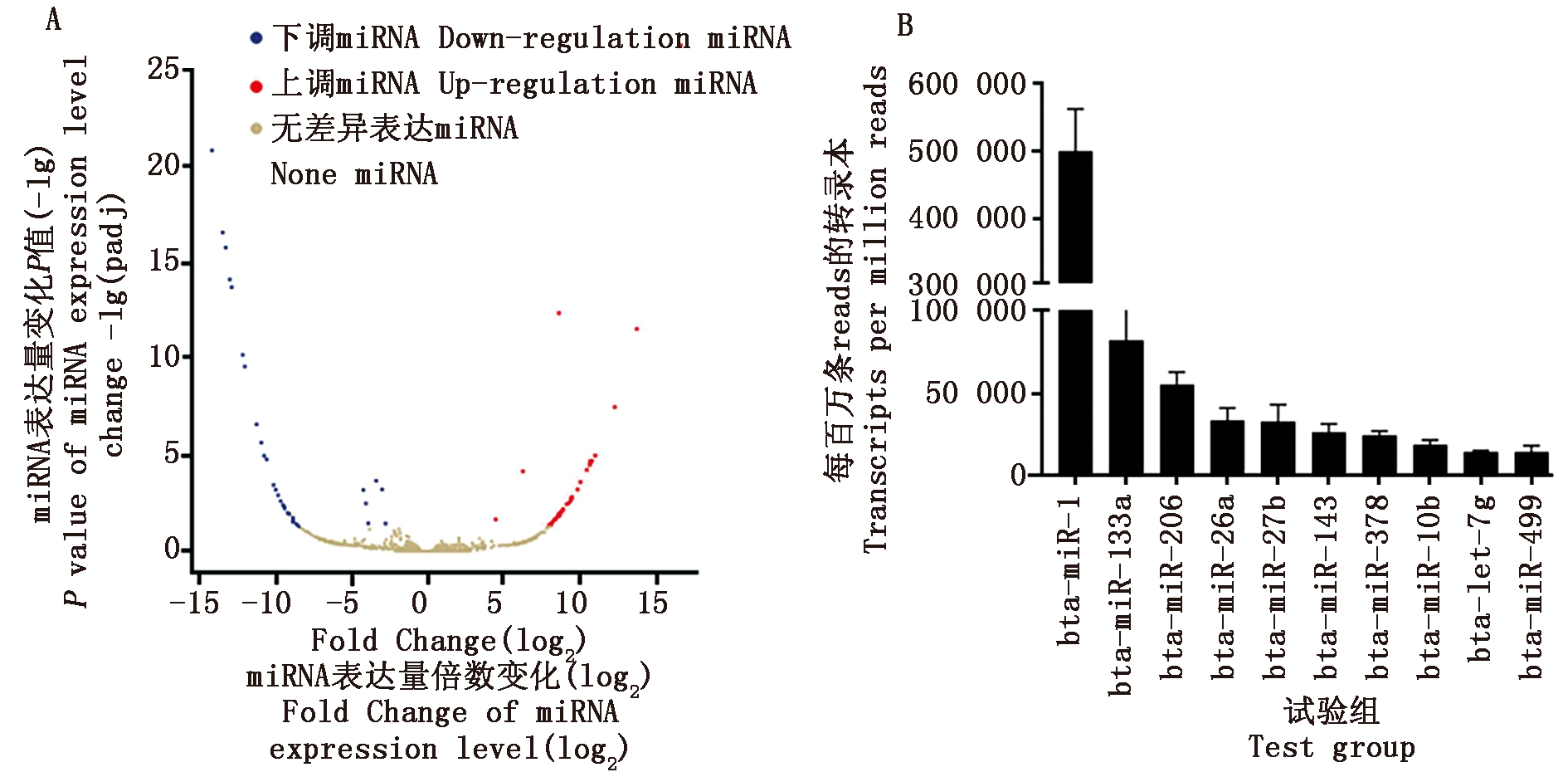
A.miRNA差异分析结果的火山图:上调或者下调指的是以鲁西黄牛为对照组,
布莱凯特黑牛背最长肌中miRNA的变化情况;B.TPM表达量前10位的miRNA。
A.Volcano plot of miRNA difference analysis results:Up-regulation or down-regulation refers to the changes
of miRNAs in the longissimus dorsi of Black cattle with Luxi cattle as the control group;B.The top ten TPM expression level of miRNAs.
图1 转录组文库中差异表达miRNA
Fig.1 Differentially expressed miRNAs in the transcriptome library
2.2 miRNA 数据验证
为了验证sRNA测序鉴定的miRNA表达谱,随机选取sRNA文库中已知表达量的6个miRNA(bta-miR-196b、bta-miR-200b、bta-miR-11977、Novel_445、Novel_130、Novel_334),通过qRT-PCR验证测序结果的准确性,其结果见图2。通过比较qRT-PCR与高通量测序的结果发现(表2),这6个miRNA在RNA-Seq和qRT-PCR分析中表现出一致的结果,表明RNA-Seq的测序结果是真实可靠的。
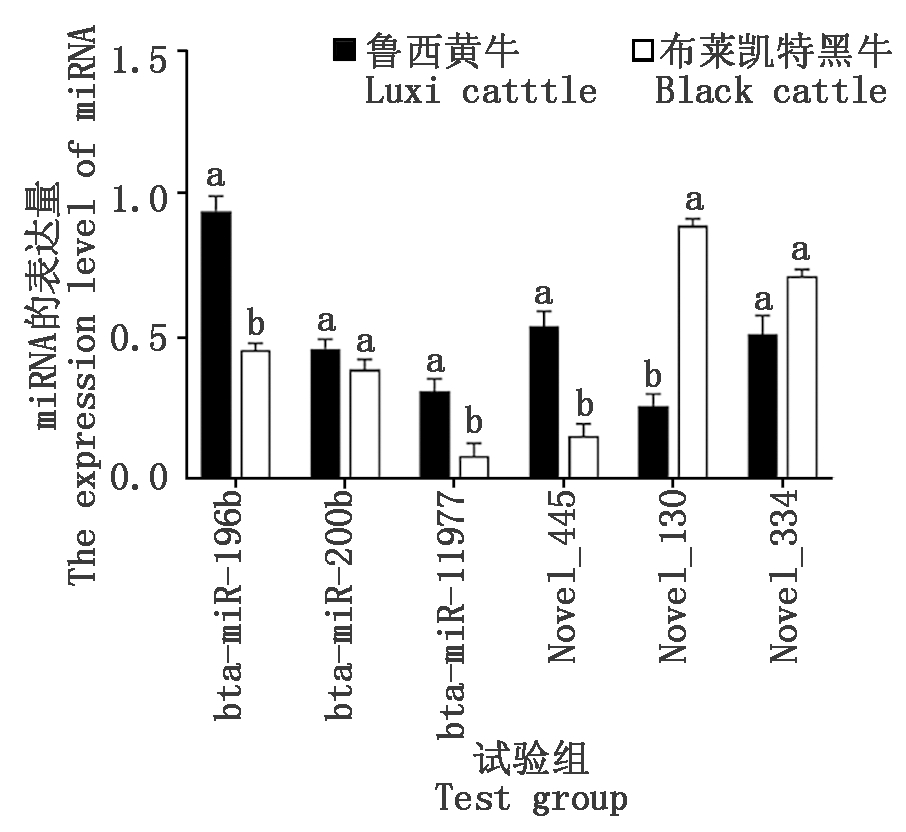
不同小写字母表示差异显著(P<0.05)。图4,6,7同。
Different lowercase letters indicate significant differences
(P<0.05).The same as Fig.4,6,7.
图2 miRNA的qRT-PCR验证分析
Fig.2 qRT-PCR verification analysis of miRNA
表2 miRNA测序表达量数据
Tab.2 miRNA sequencing expression data
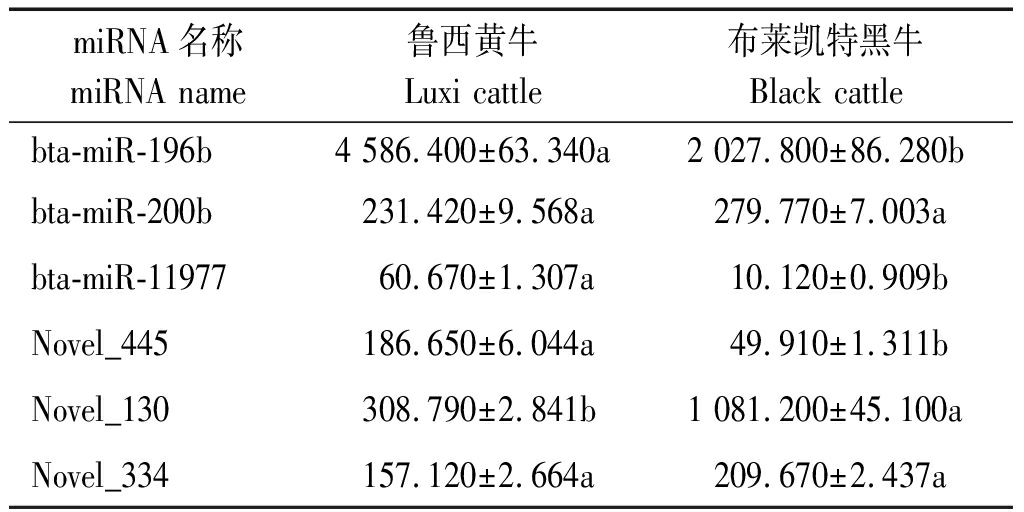
miRNA名称miRNA name鲁西黄牛Luxi cattle布莱凯特黑牛Black cattlebta-miR-196b4 586.400±63.340a2 027.800±86.280bbta-miR-200b231.420±9.568a279.770±7.003abta-miR-1197760.670±1.307a10.120±0.909bNovel_445186.650±6.044a49.910±1.311bNovel_130308.790±2.841b1 081.200±45.100aNovel_334157.120±2.664a209.670±2.437a
注:不同小写字母表示差异显著(P <0.05)。
Note:Different lowercase letters indicate significant differences(P<0.05).
2.3 bta-miR-133a的保守性分析
使用miRBase数据库检索出牛在内的6个物种miR-133a的成熟序列,其结果见表3,miR-133a(UUUGGUCCCCUUCAACCAGCUG)在各个物种中高度保守。
表3 不同物种 miR-133a 成熟序列
Tab.3 The mature sequence of
miR-133a in different species
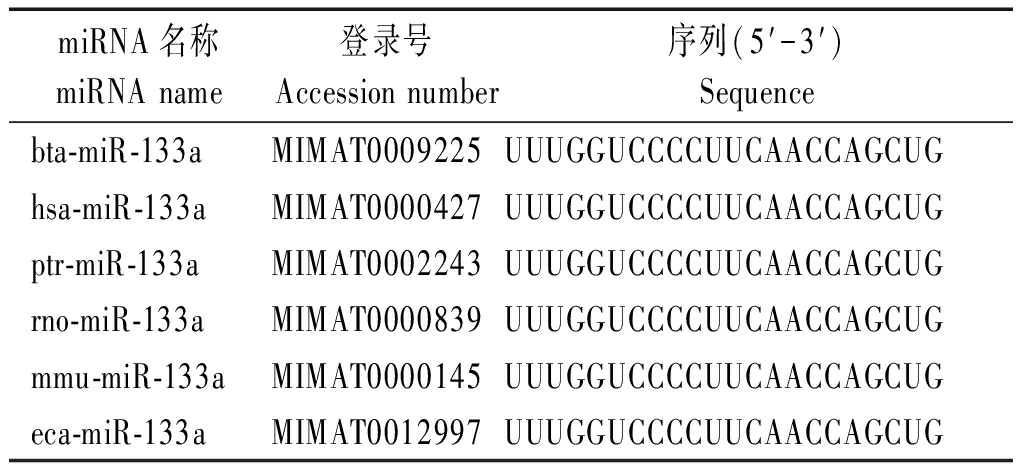
miRNA名称miRNA name登录号Accession number序列(5′-3′)Sequencebta-miR-133aMIMAT0009225UUUGGUCCCCUUCAACCAGCUGhsa-miR-133aMIMAT0000427UUUGGUCCCCUUCAACCAGCUGptr-miR-133aMIMAT0002243UUUGGUCCCCUUCAACCAGCUGrno-miR-133aMIMAT0000839UUUGGUCCCCUUCAACCAGCUG mmu-miR-133aMIMAT0000145UUUGGUCCCCUUCAACCAGCUGeca-miR-133aMIMAT0012997UUUGGUCCCCUUCAACCAGCUG
2.4 bta-miR-133a靶基因预测和富集分析
通过GO富集和KEGG通路富集分析,筛选出与肉牛骨骼肌发育相关的bta-miR-133a,使用
TargetScan 7.2软件进行靶基因预测,其结果见表4,预测到bta-miR-133a的靶基因594个,其中排名前10位的靶基因分别为:GNB4、GPM6A、TMEM167A、PLEKHA3、RAP2C、PAX7、ERI2、MAML1、SGPP1、AFTPH。使用DAVID 6.8(https://david.ncifcrf.gov/)和KOBAS 3.0(http://kobas.cbi.pku.edu.cn/kobas3/)在线软件对预测到的bta-miR-133a靶基因进行GO富集和KEGG通路富集分析。通过GO富集分析可知(图3),bta-miR-133a靶基因显著富集于细胞增殖的负调控、胚胎骨骼系统发育、肌动蛋白细胞骨架组织的调节、肌动蛋白丝结合、细胞连接等与骨骼肌发育相关的GO条目中,其中靶基因PAX7富集于肌动蛋白丝结合条目中。通过KEGG通路富集分析可知(表5),bta-miR-133a靶基因显著富集于黏附连接、肌动蛋白细胞骨架的调节、PI3K-Akt、FoxO、MAPK、Ras、TGF-β等信号通路与骨骼肌生长发育密切相关。使用TargetScan 7.2软件预测到bta-miR-133a与靶基因PAX7的靶标位区2个,2个靶标位区相差683个碱基,详细结合靶标位点见表6。
表4 bta-miR-133a的部分预测靶基因
Tab.4 Some predicted target genes of bta-miR-133a

靶基因Target gene转录本登录号Transcript accession number基因信息Genetic information上下++总分Total context++ score总PCTAggregate PCTGNB4ENST00000232564.3鸟嘌呤核苷酸结合蛋白(G 蛋白),β多肽-1.01> 0.99GPM6AENST00000280187.7糖蛋白M6A-0.64> 0.99TMEM167AENST00000502346.1跨膜蛋白167A-0.63> 0.99PLEKHA3ENST00000234453.5含有 pleckstrin 同源结构域,家族 A(磷酸肌醇结合特异性)成员 3-0.740.99RAP2CENST00000342983.2RAS致癌基因家族的成员-0.560.99PAX7ENST00000420770.2PAX7-0.560.99ERI2ENST00000569729.1ERI1 外切糖核酸酶家族成员 2-0.530.99MAML1ENST00000292599.3MAML1-0.530.98SGPP1ENST00000247225.61-磷酸鞘氨醇磷酸酶 1-0.520.98AFTPHENST00000238856.4AFTPH-0.520.98
表5 bta-miR-133a的靶基因在KEGG通路中富集结果
Tab.5 The enrichment results of bta-miR-133a target genes in the KEGG pathway

KEGG通路KEGG pathwayP值P value基因名称Gene name癌症的途径Pathways in cancer0.000 2CTBP2、HHIP、TCF7、FGF1、EGFR、ADCY6、ADCY5、IGF1R、TFG、MECOM、RUNX1T1、WNT4、TGFB2、PRKCB、TPM3、ZBTB16、VEGFC、RHOA、PML、TGFBR1、TRAF3、GNB4、FGF12、CRK、FGFR1、BCL2L1吗啡成瘾0.000 3GABBR2、GABRB1、PDE1C、PRKCB、GNB4、CACNA1B、ARRB1、PDE7A、PDE8B、ADCY6、ADCY5Morphine addictionPI3K-Akt 信号通路0.000 4ITGB5、INSR、VEGFC、FGF1、EGFR、IGF1R、COL1A1、PPP2R5E、GNB4、SGK1、FGF12、EIF4B、FGFR1、PI3K-Akt signaling pathwayBCL2L1、CREB5、MCL1
表5(续)
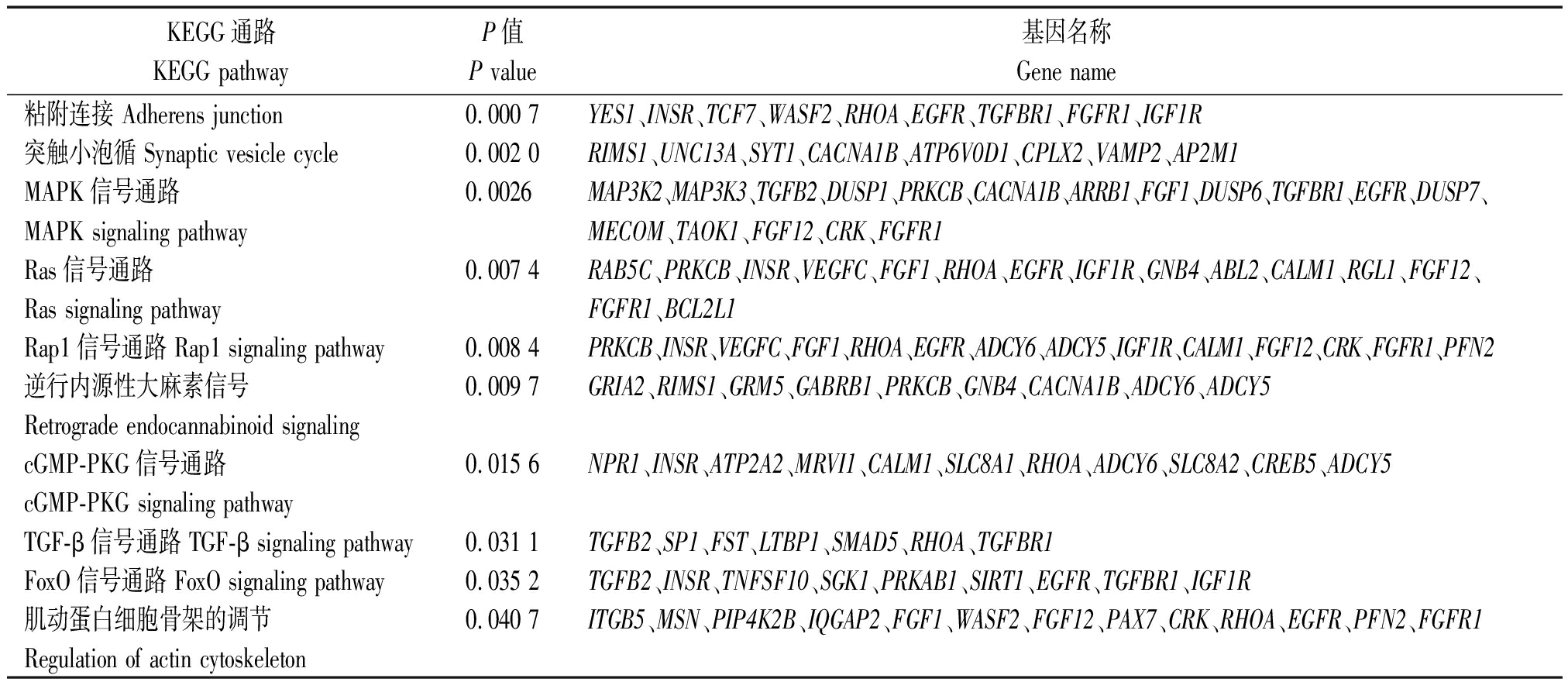
KEGG通路KEGG pathwayP值P value基因名称Gene name粘附连接 Adherens junction0.000 7YES1、INSR、TCF7、WASF2、RHOA、EGFR、TGFBR1、FGFR1、IGF1R突触小泡循 Synaptic vesicle cycle0.002 0RIMS1、UNC13A、SYT1、CACNA1B、ATP6V0D1、CPLX2、VAMP2、AP2M1MAPK信号通路0.0026MAP3K2、MAP3K3、TGFB2、DUSP1、PRKCB、CACNA1B、ARRB1、FGF1、DUSP6、TGFBR1、EGFR、DUSP7、MAPK signaling pathwayMECOM、TAOK1、FGF12、CRK、FGFR1Ras信号通路0.007 4RAB5C、PRKCB、INSR、VEGFC、FGF1、RHOA、EGFR、IGF1R、GNB4、ABL2、CALM1、RGL1、FGF12、Ras signaling pathwayFGFR1、BCL2L1Rap1信号通路 Rap1 signaling pathway0.008 4PRKCB、INSR、VEGFC、FGF1、RHOA、EGFR、ADCY6、ADCY5、IGF1R、CALM1、FGF12、CRK、FGFR1、PFN2逆行内源性大麻素信号0.009 7GRIA2、RIMS1、GRM5、GABRB1、PRKCB、GNB4、CACNA1B、ADCY6、ADCY5Retrograde endocannabinoid signalingcGMP-PKG信号通路0.015 6NPR1、INSR、ATP2A2、MRVI1、CALM1、SLC8A1、RHOA、ADCY6、SLC8A2、CREB5、ADCY5cGMP-PKG signaling pathwayTGF-β信号通路 TGF-β signaling pathway0.031 1TGFB2、SP1、FST、LTBP1、SMAD5、RHOA、TGFBR1FoxO信号通路 FoxO signaling pathway0.035 2TGFB2、INSR、TNFSF10、SGK1、PRKAB1、SIRT1、EGFR、TGFBR1、IGF1R肌动蛋白细胞骨架的调节0.040 7ITGB5、MSN、PIP4K2B、IQGAP2、FGF1、WASF2、FGF12、PAX7、CRK、RHOA、EGFR、PFN2、FGFR1Regulation of actin cytoskeleton
表6 bta-miR-133a与靶基因PAX7预测靶标位区
Tab.6 bta-miR-133a and target gene PAX7 predicted target sites
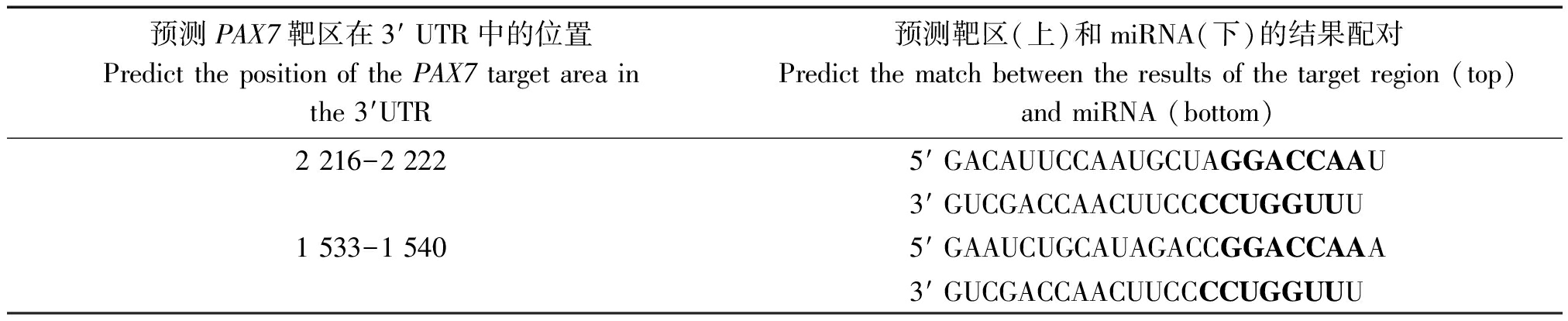
预测PAX7靶区在3′ UTR中的位置Predict the position of the PAX7 target area in the 3′UTR预测靶区(上)和miRNA(下)的结果配对Predict the match between the results of the target region (top) and miRNA (bottom)2 216-2 2225′ GACAUUCCAAUGCUAGGACCAAU3′ GUCGACCAACUUCCCCUGGUUU 1 533-1 5405′ GAAUCUGCAUAGACCGGACCAAA3′ GUCGACCAACUUCCCCUGGUUU
注:靶基因PAX7位于染色体1:18630846-18748866;碱基加粗为预测靶区(上)和miRNA(下)的配对碱基。
Note:The target gene PAX7 is located on chromosome 1:18630846-18748866;Base bold is the paired base of the predicted target region(top)and miRNA(bottom).
2.5 bta-miR-133a与靶基因PAX7的靶标关系验证
通过荧光素酶报告基因系统检测试剂盒检测荧光素酶活性水平,其结果见图4,与转染空白载体组相比,转染bta-miR-133a 组极显著降低荧光素酶活性(P<0.01),转染bta-miR-133a-mu组的荧光素酶活性无显著变化(P>0.05)。表明bta-miR-133a在PAX7 3′-UTR存在结合位点,PAX7是bta-miR-133a的靶基因。
2.6 bta-miR-133a促进肌管形成
由图5可知,诱导分化1 d,各组之间无显著差异(P>0.05)。诱导分化2 d,与空白组相比,bta-miR-133a过表达组出现细胞融合,形成多核细胞;bta-miR-133a抑制组细胞融合数量无明显变化,大部分依然是单核细胞。诱导分化5 d,与空白组相比,bta-miR-133a过表达组有大量肌管形成,肌管直径明显增大;bta-miR-133a抑制组开始出现少量肌管,肌管直径较短。诱导分化6 d,与空白组相比,bta-miR-133a过表达组肌管直径明显增粗,肌管长度增大,数量增多;bta-miR-133a抑制组肌管的直径较短,肌管长度较短,数量无显著差异(P>0.05)。
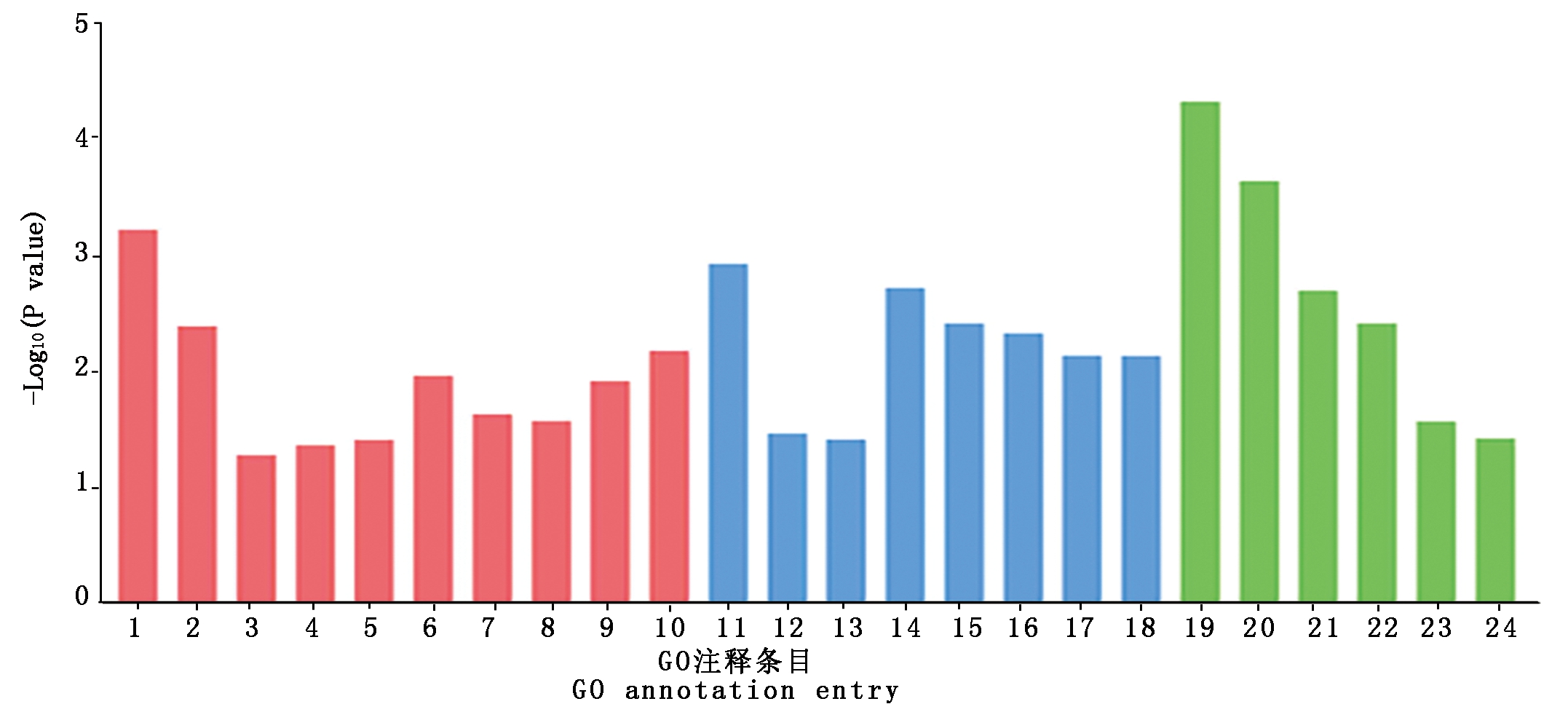
图3 bta-miR-133a预测靶基因的GO注释条目
Fig.3 The GO annotation items of bta-miR-133a predicted target genes
红色柱状图代表生物过程;蓝色柱状图代表分子功能;绿色柱状图代表细胞组成;1.RNA聚合酶Ⅱ启动子转录的正调控;2.转录的正调控;3.细胞增殖的负调控;4.胚胎骨骼系统发育;5.DNA修复的正调控;6.肌动蛋白细胞骨架组织的调节;7.经典Wnt信号通路的负调控;8.生长;9.血管生成;10.转化生长因子β受体信号通路;11.核苷酸结合;12.受体信号蛋白丝氨酸/苏氨酸激酶活性;13.ATP结合14.mRNA结合;15.蛋白质异二聚化活性;16.泛素蛋白连接酶结合;17.蛋白酪氨酸磷酸酶活性;18.肌动蛋白丝结合;19.核;20.神经元投射;21.核质;22.细胞质;23.细胞-细胞粘附连接;24.细胞连接。
The red bar graph represents biological process;The blue bar graph represents molecular function;The green bar graph represents cellular component;1.Positive regulation of transcription from RNA polymerase Ⅱ promoter;2.Positive regulation of transcription;3.Negative regulation of cell proliferation;4.Embryonic skeletal system development;5.Positive regulation of DNA repair;6.Regulation of actin cytoskeleton organization;7.Negative regulation of classical Wnt signaling pathway;8.Growth;9.Angiogenesis;10.Transforming growth factor beta receptor signaling pathway;11.Nucleotide binding;12.Receptor signaling protein serine/threonine kinase activity;13.ATP binding;14.mRNA binding;15.Protein heterodimerization activity;16.Ubiquitin protein ligase binding;17.Protein tyrosine phosphatase activity;18.Actin filament binding;19.Nucleus;20.Neuron projection;21.Nucleoplasm;22.Cytoplasm;23.Cell-cell adherens junction;24.Cell junction.
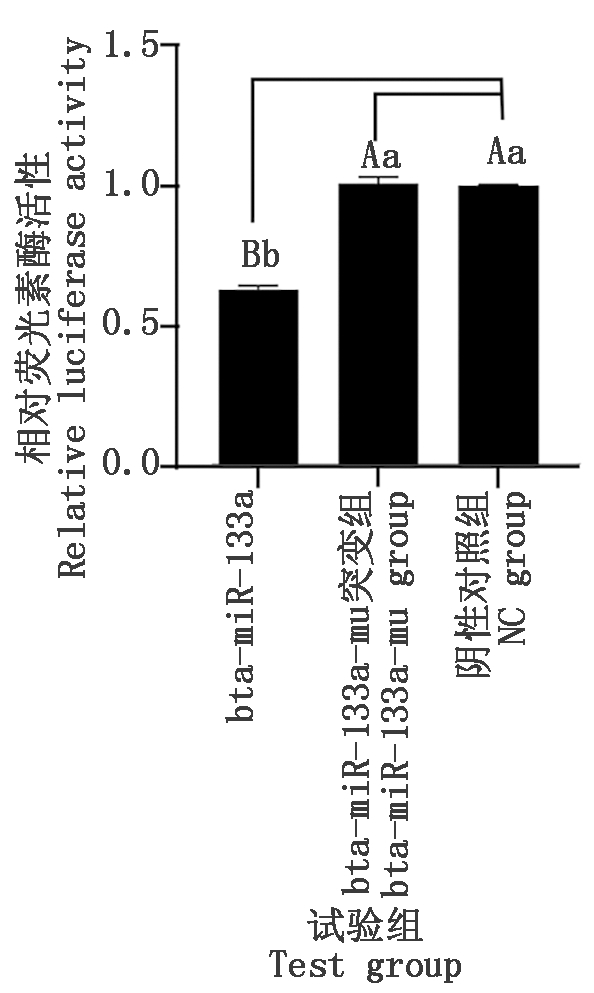
不同小写字母表示差异显著(P<0.05),不同大写字母
表示差异极显著(P<0.01)。图6-7同。
Different lowercase letters indicate significant differences(P<0.05),
different capital letters indicate extremely significant
differences(P<0.01).The same as Fig.6-7.
图4 bta-miR-133a与PAX7靶
向关系的双荧光素酶检测
Fig.4 Dual luciferase detection of the targeting
relationship between bta-miR-133a and PAX7
2.7 成肌细胞增殖率检测
转染后,分别在24,48,72 h进行CCK-8细胞增殖率检测。由图6可知,24 h,各处理组之间无显著差异(P>0.05)。48 h,与空白组相比,bta-miR-133a过表达组显著降低成肌细胞的增殖率(P<0.05);bta-miR-133a抑制组无显著差异(P>0.05)。72 h,与空白组相比,bta-miR-133a过表达组极显著降低成肌细胞的增殖率(P<0.01);bta-miR-133a抑制组显著提高成肌细胞的增殖率(P<0.05)。

竖列代表同一个处理组,横列代表同一个时间点,比例尺=100 μm。
The vertical column represents the same treatment group,the horizontal column represents the same time point,scale bar=100 μm.
图5 1,2,5,6 d成肌细胞的诱导分化过程
Fig.5 The induction and differentiation process of myoblasts at 1,2,5,and 6 d

图6 CCK-8检测在24,48,72 h细胞增殖率
Fig.6 CCK-8 detection of cell proliferation rate at 24,48,72 h
2.8 过表达、敲除bta-miR-133a对靶基因PAX7和增殖分化相关基因mRNA表达水平的影响
转染后,48 h提取细胞RNA,使用qRT-PCR检测PAX7、PCNA、CCND1、Myh1、Myod1的表达量。由图7可知,bta-miR-133a过表达组极显著抑制靶基因PAX7的表达(P<0.01),bta-miR-133a抑制组显著促进PAX7的表达(P<0.05)。在细胞增殖方面(图7-B),bta-miR-133a过表达组极显著抑制PCNA的表达(P<0.01),显著抑制CCND1的表达(P<0.05),bta-miR-133a抑制组显著促进PCNA和CCND1的表达(P<0.05);在细胞分化方面(图7-C),bta-miR-133a过表达组极显著促进Myod1和Myh1的表达(P<0.01),bta-miR-133a抑制组显著抑制Myod1的表达(P<0.01),对Myh1的表达没有显著影响(P>0.05)。

图7 PAX7、PCNA、CCND1、Myh1、Myod1基因表达水平
Fig.7 The expression levels of PAX7,PCNA,CCND1,Myh1,Myod1 genes
3 讨论与结论
骨骼肌的生长发育过程大致分为成肌细胞分化形成阶段、成肌细胞迁移增殖阶段和肌管形成阶段,涉及多基因表达和信号途径调控,过程极其复杂,miRNAs则是该过程的重要调控因子[16-18]。研究表明,miRNAs参与了几乎所有细胞过程的调控,关于miRNAs在骨骼肌发育中的潜在机制和作用的研究也越来越多,大多采用高通量测序建库结合生物信息学分析的方法[19-21]。本研究以动物胚胎工程中心实验室建立的布莱凯特黑牛和鲁西黄牛背最长肌测序小RNA文库为数据背景,采用GO富集和KEGG通路富集分析筛选出与肉牛骨骼肌发育密切相关的差异miRNAs(bta-miR-1、bta-miR-133a、bta-miR-206、bta-miR-26a、bta-miR-27b、bta-miR-143、bta-miR-378、bta-miR-10b、bta-let-7g、bta-miR-499),选取TPM表达量较高的bta-miR-133a作为研究对象。据报道,从人和小鼠肌肉组织中克隆鉴定出肌肉特异性miR-206,可以促进骨骼肌细胞分化,抑制小鼠C2C12细胞增殖[22]。同时在Ling等[23]的研究中发现,miR-27b可通过调控PAX3基因促进山羊骨骼肌卫星细胞分化而抑制细胞增殖。miR-378a的水平升高会抑制成肌抑制因子,进而正反馈调控Myod促进成肌分化[24],敲除miR-378a会影响骨骼肌中脂肪酸和葡萄糖氧化的能量代谢,进而影响肌纤维类型的发育[25]。本研究通过保守性分析,比对不同物种miR-133a成熟序列(UUUGGUCCCCUUCAACCAGCUG)的同源性,发现miR-133a在各个物种中高度保守,表明miR-133a在生物进化过程中可能具有重要功能。通过生物信息学在线网站分析,预测到bta-miR-133a靶基因594个,发现预测结果排名前10的靶基因(GNB4、GPM6A、TMEM167A、PLEKHA3、RAP2C、PAX7、ERI2、MAML1、SGPP1、AFTPH)中,多与癌症相关,例如:GPM6A在肺癌组织中低表达会影响患者预后,GPM6A可作为预测肺癌发生、转移及判断预后的有效分子标记物[26];RAP2C与脑胶质瘤[27]、喉鳞状细胞癌[28]、乳腺癌[29]等癌症发展相关;AFTPH被认为是乳腺浸润癌(BRCA)、肺鳞癌(LUSC)和胰腺癌(PAAD)的潜在治疗靶标和预后生物标志物[30]。靶基因PAX7主要参与心肌与骨骼肌的生长发育,在成年期和肌肉再生过程中起主要作用,对卫星细胞的生物发生和存活至关重要[31-32]。同时也有研究表明,PAX7可通过与肌肉调节因子家族成员之间的交叉抑制,维持着卫星细胞的自我更新,从而决定了骨骼肌维持和修复所必需的增殖、分化和自我更新[33-34]。以上研究分析表明,PAX7与肌肉生长发育调节机制的功能相关性较强,因此,选取PAX7作为bta-miR-133a的靶基因进行下一步的研究。此外,bta-miR-133a的预测靶基因PLEKHA3和ERI2在目前的研究中报道较少,功能尚待进一步研究。
基于本试验是探究不同品种间肉牛骨骼肌生长发育的分子机制,对bta-miR-133a靶基因进行GO富集和KEGG通路富集分析,推测这些靶基因的潜在生物学功能。发现bta-miR-133a靶基因主要显著富集在细胞增殖的负调控、肌动蛋白细胞骨架组织的调节、胚胎骨骼系统开发、肌动蛋白丝结合等GO条目中和MAPK、PI3K-Akt、FoxO、Ras、TGF-beta、肌动蛋白细胞骨架的调节等与骨骼肌发育密切相关的信号通路中。MAPK信号通路由4个亚家族组成,其中细胞外信号调节激酶(ERK)主要调控细胞生长和分化,且Ras信号通路与MAPK信号通路中的信号蛋白存在联系,共同参与调控肌肉的发育过程;IGF1通过介导PI3K-Akt信号通路促进成肌细胞的增殖和骨骼肌生长[35-36];FoxO 信号通路中的FOXO1、FOXO3和FOXO32广泛参与肌肉的生长发育过程[37];TGF-β信号通路中的TGFβ2、TGFβ3可抑制卫星细胞的分化,进而影响骨骼肌的生长发育[38];靶基因PAX7富集在肌动蛋白丝结合的GO条目和肌动蛋白细胞骨架的KEGG通路中,表明bta-miR-133a与骨骼肌的发育过程密切相关,且可能通过靶基因PAX7参与调控该过程。已有研究表明,PAX7是维持静止的卫星细胞和肌肉再生所必需的基因[39-40],可作为关键上游调节因子参与肌肉的生长发育过程[41],发挥着维持骨骼肌再生和修复的作用。进一步研究表明,PAX7与骨骼肌的生长发育调节机制的功能相关性最强,因此,选取PAX7作为bta-miR-133a的靶基因。
基于上述分析讨论,预测bta-miR-133a的种子序列(CCUGGUU)与靶基因PAX7的靶标结合位区有2个,都位于3′ UTR区,中间相差683个碱基。进一步通过双荧光素酶报告试验发现,bta-miR-133a与PAX7存在靶向关系,证明了PAX7是bta-miR-133a的靶基因。在试验开展前考虑到bta-miR-133a运用的局限性,进行了bta-miR-133a的PCR扩增试验,测序结果发现该miRNA在小鼠和牛组织上的片段扩增不受影响,且在前人的相关研究中,已有相类似的细胞模型验证试验,Kong等[42-43]研究发现,miR-17在小鼠C2C12细胞和牛的骨骼肌成肌细胞中,皆发挥着促进成肌细胞分化的相同作用;在miR-143、miR-24-3p等miRNA上也有相类似的研究[44-46]。因此,本试验以小鼠C2C12细胞作为细胞模型,进行细胞功能验证试验。转染Blank、NC mimic、bta-miR-133a mimic、NC inhibitor和bta-miR-133a inhibitor,诱导分化后发现,诱导分化5 d,与空白组相比,bta-miR-133a过表达组的细胞有大量肌管形成,肌管长度较长,而转染bta-miR-133a抑制组形成的肌管较少,大部分成肌细胞还在融合中,表明bta-miR-133a过表达组的成肌细胞向肌管分化的进程加快,而bta-miR-133a抑制组被抑制。在成肌细胞分化的过程中,观察到bta-miR-133a对肌管形成前期的作用效果并不明显,但在诱导分化5 d后,观察到各个处理组之间的细胞形态差异最明显,表明bta-miR-133a对成肌细胞分化的作用明显;在诱导分化6 d后,观察到bta-miR-133a抑制组的肌管直径显著增大、长度增长,表明转染bta-miR-133a inhibitor导致肌管形成的高峰期延后。虽然bta-miR-133a抑制组的成肌细胞分化过程较慢,但各个处理组的成肌细胞均进入肌管形成过程,最终形成肌管,表明bta-miR-133a inhibitor可以在一定程度上抑制成肌细胞分化,减缓其分化速度,但无法直接阻断成肌细胞的融合分化。综上表明,bta-miR-133a促进肌管分化,且主要在肌管形成后期发挥作用,这与邢义珅[47]、秦瑞峰等[48]、陈永乐等[49]研究报道的关于成肌细胞增殖分化的过程相一致。采用CCK-8检测细胞增殖率发现bta-miR-133a过表达组抑制细胞增殖的速率在24~48 h最大,bta-miR-133a抑制组促进细胞增殖的速率也在24~48 h最大,表明bta-miR-133a抑制成肌细胞增殖,且主要在细胞增殖的前期发挥主要作用。
研究表明,关于miRNAs对成肌细胞增殖分化方面的研究多采用增殖标志性基因PCNA、CCND1和分化标志性基因Myod1、Myh1的表达量来反映成肌细胞增殖分化的程度[50-53]。通过qRT-PCR检测转染后48 h成肌细胞中增殖标志性基因PCNA、CCND1和分化标志性基因Myod1、Myh1的表达量,发现与空白组相比,转染bta-miR-133a minic,极显著抑制PCNA,显著抑制CCND1的表达,极显著促进Myod1和Myh1的表达;转染bta-miR-133a inhibitor,显著促进PCNA和CCND1的表达,显著抑制Myod1的表达。表明bta-miR-133a参与调控增殖细胞中DNA的复制过程[54],抑制细胞从G1期到S期的过程;促进肌球蛋白的合成,促进肌卫星细胞向成肌细胞转化,最终融合形成肌管[55]。基于以上试验结果分析表明,bta-miR-133a抑制成肌细胞增殖,促进成肌细胞分化。通过qRT-PCR检测PAX7的表达量,发现bta-miR-133a抑制PAX7表达,与Nguyen等[56]在尼罗罗非鱼骨骼肌发现的miR-133a与假定靶基因PAX7的负调控关系相一致。有的研究报道PAX7基因的表达量下降会促进成肌细胞分化[41],进一步表明bta-miR-133a促进成肌细胞分化。
本研究选取bta-miR-133a的目标靶基因时,结合了生物学信息分析以及相关功能性研究,虽然只选取了PAX7,但是已有研究表明bta-miR-133a对成肌细胞的增殖分化过程的调控可能不仅仅通过靶基因PAX7,也可能通过调控其他调节因子来参与骨骼肌的发育过程。在该过程中,miR-133可通过靶向调控血清反应因子(SRF)[57]、NPTB[58]或者UCP2[59]来参与肌细胞增殖分化过程。因此,bta-miR-133a与PAX7以及其他靶基因的分子调控网络还需要进行深入的探索。
bta-miR-133a可能通过抑制靶基因PAX7的表达,抑制成肌细胞的增殖,促进成肌细胞的分化,进而参与调节肉牛骨骼肌的生长发育。
[1] 范永辉. miR-34b通过靶向SYISL调节成肌细胞增殖和分化[D].武汉:华中农业大学,2019.
Fan Y H. miR-34b regulates myoblast proliferation and differentiation by targeting SYISL[D].Wuhan:Huazhong Agricultural University,2019.
[2] Sweetman D,Goljanek K,Rathjen T,Oustanina S,Braun T,Dalmay T,Münsterberg A. Specific requirements of MRFs for the expression of muscle specific microRNAs,miR-1,miR-206 and miR-133[J].Developmental Biology,2008,321(2):491-499. doi:10.1016/j.ydbio.2008.06.019.
[3] Safa A,Bahroudi Z,Shoorei H,Majidpoor J,Abak A,Taheri M,Ghafouri-Fard S. miR-1:A comprehensive review of its role in normal development and diverse disorders[J].Biomedicine & Pharmacotherapy,2020,132:110903.doi:10.1016/j.biopha.2020.110903.
[4] 于太永.脂肪细胞因子leptin和TNF-α对猪骨骼肌成肌细胞增殖分化的影响及其机制[D].杨凌:西北农林科技大学,2007.
Yu T Y. Effects of leptin and TNF-α on the porcine myoblast proliferation and differentiation and its molecular mechanism[D].Yangling:Northwest Agriculture and Forestry University,2007.
[5] Mok G F,Lozano-Velasco E,Maniou E,Viaut C,Moxon S,Wheeler G,Münsterberg A. miR-133 mediated regulation of the hedgehog pathway orchestrates embryo myogenesis[J].Development,2018:145(12):159657.doi:10.1242/dev.159657.
[6] 王丽娟. MiR-1和miR-133在山羊骨骼肌组织和细胞中的表达规律及功能研究[D].合肥:安徽农业大学,2014.
Wang L J. The expression profiles and function analysis of miR-1 and miR-133 in goat skeletal muscle tissue and cell[D].Hefei:Anhui Agricultural University,2014.
[7] 吴宁昭. miRNA-1和miRNA-133在鸭骨骼肌发育中的表达及功能初步研究[D].扬州:扬州大学,2017.
Wu N Z. The expression of miRNA-1 and miRNA-133 and its function on duck skeletal cell proliferation and differentiation[D].Yangzhou:Yangzhou University,2017.
[8] 王星果,邵芳,龚道清,卢祥云,顾志良. 鸡miR-133a靶向调控BIRC5基因的表达[J].中国农业科学,2013,46(7):1441-1447.doi:10.3864/j.issn.0578-1752.2013.07.015.
Wang X G,Shao F,Gong D Q,Lu X Y,Gu Z L. miR-133a targets BIRC5 to regulate its gene expression in chicken[J].Scientia Agricultura Sinica,2013,46(7):1441-1447.
[9] Huang M B,Xu H,Xie S J,Zhou H,Qu L H. Insulin-like growth factor-1 receptor is regulated by microRNA-133 during skeletal myogenesis[J].PLoS One,2011,6(12):e29173. doi:10.1371/journal.pone.0029173.
[10] Liu R L,Liu X X,Bai X J,Xiao C Z,Dong Y J. Different expression of lipid metabolism-related genes in Shandong black cattle and Luxi cattle based on transcriptome analysis[J].Scientific Reports,2020,10:21915.doi:10.1038/s41598-020-79086-4.
[11] Liu R L,Liu X X,Bai X J,Xiao C Z,Dong Y J. Identification and characterization of circRNA in longissimus dorsi of different breeds of cattle[J].Frontiers in Genetics,2020,11:565085.doi:10.3389/fgene.2020.565085.
[12] 董雅娟. 牛[M].北京:中国农业出版社,2016:20.
Dong Y J. Cattle[M].Beijing:China Agriculture Press,2016:20.
[13] 刘瑞莉,吴磊,袁玮,柏学进,吕娟娟,董雅娟. MYL3基因在肉牛肌肉生长过程中的功能研究[J].华北农学报,2019,34(2):221-228. doi:10.7668/hbnxb.201750982.
Liu R L,Wu L,Yuan W,Bai X J,Lü J J,Dong Y J.Function of MYL3 gene in muscle growth of cattle[J].Acta Agriculturae Boreali-Sinica,2019,34(2):221-228.
[14] 吴磊,刘瑞莉,袁玮,柏学进,刘贤勋,吕娟娟,肖超柱,董雅娟. CRTC2基因在不同品种和月龄肉牛的表达分析[J].畜牧与兽医,2019,51(10):9-14.
Wu L,Liu R L,Yuan W,Bai X J,Liu X X,Lü J J,Xiao C Z,Dong Y J. Analysis of CRTC2 gene expression in cattle of different varieties and months of age[J].Animal Husbandry & Veterinary Medicine,2019,51(10):9-14.
[15] 刘瑞莉,吴磊,袁玮,柏学进,吕娟娟,董雅娟. 基于转录组筛选肉牛骨骼肌差异基因[J].华北农学报,2018,33(S1):64-72.doi:10.7668/hbnxb.2018.S1.011.
Liu R L,Wu L,Yuan W,Bai X J,Lü J J,Dong Y J. Screening of skeletal muscle differential genes based on transcriptome[J].Acta Agriculturae Boreali-Sinica,2018,33(S1):64-72.
[16] Cesana M,de Cacchiarelli D,Legnini I,Santini T,Sthandier O,Chinappi M,Tramontano A,Bozzoni I.A long noncoding RNA controls muscle differentiation by functioning as a competing endogenous RNA[J].Cell,2011,147(2):358-369.doi:10.1016/j.cell.2011.09.028.
[17] Ma X Y,Wei D W,Cheng G,Li S J,Wang L,Wang Y N,Wang X Y,Zhang S,Wang H B,Zan L S. Bta-miR-130a/b regulates preadipocyte differentiation by targeting PPARG and CYP2U1 in beef cattle[J].Molecular and Cellular Probes,2018,42:10-17.doi:10.1016/j.mcp.2018.10.002.
[18] Dey B K,Gagan J,Dutta A. miR-206 and-486 induce myoblast differentiation by downregulating Pax7[J].Molecular and Cellular Biology,2011,31(1):203-214. doi:10.1128/mcb.01009-10.
[19] 孙加节. 秦川牛肌肉与脂肪组织发育相关miRNA鉴定及miR-10020调控机制解析[D].杨凌:西北农林科技大学,2015.
Sun J J. Identification of microRNAs from Chinese Qinchuan bovine muscle and adipose tissues and functional analysis of miR-10020 during myogenesis[D].Yangling:Northwest Agriculture and Forestry University,2015.
[20] 王艳红. 山羊不同生长发育阶段肌肉组织miRNA的鉴定及其调控机制研究[D].杨凌:西北农林科技大学,2015.
Wang Y H. Identification of muscle-related miRNA from skeletal muscle in different development stage goat and analysis on their regulation roles[D].Yangling:Northwest Agriculture and Forestry University,2015.
[21] 郭晓萍. 猪骨骼肌miRNA转录组分析及miR-486功能的初步研究[D].南宁:广西大学,2015.
Guo X P. Transcriptome analysis of porcine skeletal muscle miRNAs and primary study of miR-486 function[D].Nanning:Guangxi University,2015.
[22] 娄秋宏. gga-miR-206在金茅黑鸡胚胎骨骼肌发育中的表达分析及靶基因验证[D].扬州:扬州大学,2019.
Lou Q H. Expression of gga-miR-206 in different stages of skeletal muscle development in Jinmao black chicken embryos and its targets identification[D].Yangzhou:Yangzhou University,2019.
[23] Ling Y H,Sui M H,Zheng Q,Wang K Y,Wu H,Li W Y,Liu Y,Chu M X,Fang F G,Xu L N. miR-27b regulates myogenic proliferation and differentiation by targeting Pax3 in goat[J].Scientific Reports,2018,8:3909. doi:10.1038/s41598-018-22262-4.
[24] Gagan J,Dey B K,Layer R,Yan Z,Dutta A. MicroRNA-378 targets the myogenic repressor MyoR during myoblast differentiation[J].Journal of Biological Chemistry,2011,286(22):19431-19438.doi:10.1074/jbc.M111.219006.
[25] 化朝举. miR-378a对骨骼肌肌纤维类型及其代谢的调控机理[D].北京:中国农业科学院,2016.
Hua C J. Molecular mechanism of miR-378a regulating myofiber type and metabolism of skeletal muscle[D].Beijing:Chinese Academy of Agricultural Sciences,2016.
[26] 庚跃琦,孔令珍,贾垂明,李丹丹. GPM6A在肺癌组织中的表达差异及临床意义[J].现代肿瘤医学,2019,27(9):1546-1550.doi:10.3969/j.issn.1672-4992.2019.09.019.
Geng Y Q,Kong L Z,Jia C M,Li D D. Analysis of expression profile of GPM6A and clinical significance in lung cancer tissues[J].Journal of Modern Oncology,2019,27(9):1546-1550.
[27] Wang X C,Wang C,Xi L,Yu Z Q. Rap2c as a novel biomarker for predicting poor prognosis in glioma[J].OncoTargets and Therapy,2020,13:3073-3083. doi:10.2147/OTT.S247731.
[28] Wang Z X,Huang C P,Zhang A B,Lu C,Liu L F. Overexpression of circRNA_100290 promotes the progression of laryngeal squamous cell carcinoma through the miR-136-5p/RAP2C axis[J].Biomedicine & Pharmacotherapy,2020,125:109874.doi:10.1016/j.biopha.2020.109874.
[29] Zhu X Y,Qiu J X,Zhang T,Yang Y P,Guo S,Li T S,Jiang K F,Zahoor A,Deng G Z,Qiu C W. MicroRNA-188-5p promotes apoptosis and inhibits cell proliferation of breast cancer cells via the MAPK signaling pathway by targeting Rap2c[J].Journal of Cellular Physiology,2020,235(3):2389-2402. doi:10.1002/jcp.29144.
[30] Zhu T J,Chen Y T,Liu Z J,Leng Y X,Tian Y. Expression profiles and prognostic significance of AFTPH in different tumors[J].FEBS Open Biochemistry,2020,10(12):2666-2677.doi:10.1002/2211-5463.13003.
[31] 孙多临,冯汉卿,赵恒,陈胜男,李松美,王春生. 绵羊Pax7基因慢病毒示踪载体的构建与表达检测[J].黑龙江畜牧兽医,2020(19):11-15,165.doi:10.13881/j.cnki.hljxmsy.2019.12.0060.
Sun D L,Feng H Q,Zhao H,Chen S N,Li S M,Wang C S. Construction and expression detection of Pax7 gene lentiviral vector in sheep[J].Heilongjiang Animal Science and Veterinary Medicine,2020(19):11-15,165.
[32] Relaix F,Rocancourt D,Mansouri A,Buckingham M. A Pax3/Pax7-dependent population of skeletal muscle progenitor cells[J].Nature,2005,435(7044):948-953. doi:10.1038/nature03594.
[33] Relaix F,Montarras D,Zaffran S,Gayraud-Morel B,Rocancourt D,Tajbakhsh S,Mansouri A,Cumano A,Buckingham M. Pax3 and Pax7 have distinct and overlapping functions in adult muscle progenitor cells[J].The Journal of Cell Biology,2006,172(1):91-102. doi:10.1083/jcb.200508044.
[34] Oustanina S,Hause G,Braun T. Pax7 directs postnatal renewal and propagation of myogenic satellite cells but not their specification[J].The EMBO Journal,2004,23(16):3430-3439. doi:10.1038/sj.emboj.7600346.
[35] Tureckova J,Wilson E M,Cappalonga J L,Rotwein P. Insulin-like growth factor-mediated muscle differentiation:collaboration between phosphatidylinositol 3-kinase-akt-signaling pathways and myogenin[J].Journal of Biological Chemistry,2001,276(42):39264-39270. doi:10.1074/jbc.M104991200.
[36] Yu M L,Wang H,Xu Y L,Yu D B,Li D F,Liu X H,Du W X. Insulin-like growth factor-1(IGF-1)promotes myoblast proliferation and skeletal muscle growth of embryonic chickens via the PI3K/Akt signalling pathway[J].Cell Biology International,2015,39(8):910-922. doi:10.1002/cbin.10466.
[37] 石斌刚. 天祝白牦牛肌肉生长和肌内脂肪沉积相关基因筛选与鉴定[D].兰州:甘肃农业大学,2020.
Shi B G. Identification of genes associated with muscle growth and intramuscular fat deposition in Tianzhu white yak[D].Lanzhou:Gansu Agricultural University,2020.
[38] 张维娅. 不同年龄骨骼肌卫星细胞的分化能力及调控机制研究[D].武汉:华中农业大学,2018.
Zhang W Y. The differentiation and regulation mechanism of skeletal muscle satellite cells at different ages[D].Wuhan:Huazhong Agricultural University,2018.
[39] von Maltzahn J,Jones A E,Parks R J,Rudnicki M A. Pax7 is critical for the normal function of satellite cells in adult skeletal muscle[J].Proceedings of the National Academy of Sciences of the United States of America,2013,110(41):16474-16479. doi:10.1073/pnas.1307680110.
[40] Günther S,Kim J,Kostin S,Lepper C,Fan C M,Braun T. Myf5-positive satellite cells contribute to Pax7-dependent long-term maintenance of adult muscle stem cells[J].Cell Stem Cell,2013,13(5):590-601.doi:10.1016/j.stem.2013.07.016.
[41] Buckingham M,Relaix F. PAX3 and PAX7 as upstream regulators of myogenesis[J].Seminars in Cell & Developmental Biology,2015,44:115-125.doi:10.1016/j.semcdb.2015.09.017.
[42] Kong D L,He M,Yang L,Zhou R T,Yan Y Q,Liang Y,Teng C B. MiR-17 and miR-19 cooperatively promote skeletal muscle cell differentiation[J].Cellular and Molecular Life Sciences,2019,76(24):5041-5054.doi:10.1007/s00018-019-03165-7.
[43] 孔德麟. miR-17-92家族对骨骼肌成肌细胞分化调节机制的研究[D].哈尔滨:东北林业大学,2019.
Kong D L. Mechanism study of MiR-17-92 in regulating skeletal muscle cell differentiation[D].Harbin:Northeast Forestry University,2019.
[44] 张蔚然. miR-143对牛骨骼肌卫星细胞成肌分化的调控作用研究[D].天津:天津农学院,2017.
Zhang W R. Study on the regulation mechanism of microRNA-143 in the proliferation and myogenic differentiation process of bovine skeletal muscle satellite cells[D].Tianjin:Tianjin Agricultural University,2017.
[45] 代阳. microRNA-128对牛骨骼肌卫星细胞增殖和成肌分化的调控机制研究[D].天津:天津农学院,2016.
Dai Y. Study on the regulation mechanism of microRNA-128 in the proliferation and myogenic differentiation process of bovine skeletal muscle satellite cells[D].Tianjin:Tianjin Agricultural University,2016.
[46] 邬明丽. miR-24-3p调控肌细胞增殖与凋亡的机制研究[D].杨凌:西北农林科技大学,2019.
Wu M L. Regulating mechanism of miR-24-3p in myoblasts proliferation and apoptosis[D].Yangling:Northwest Agriculture and Forestry University,2019.
[47] 邢义珅. NCAGP基因对牛胎儿骨骼肌来源的成肌细胞有丝分裂和成肌分化的影响及机理研究[D].北京:中国农业科学院,2018.
Xing Y S. The effects of NCAPG on the mitosis and myogenic differentiation of myoblasts derived from fetal bovine skeletal muscle and mechanism study[D].Beijing:Chinese Academy of Agricultural Sciences,2018.
[48] 秦瑞峰,顾晓明,秦晓春. 骨骼肌肌细胞增殖及分化过程中凋亡现象[J].第四军医大学学报,2001,22(1):20-22. doi:10.3321/j.issn:1000-2790.2001.01.006.
Qin R F,Gu X M,Qin X C. Apoptosis in proliferation and differentiation of skeletal muscle cells[J].Journal of the Fourth Military Medical University,2001,22(1):20-22.
[49] 陈永乐,周光前,邓宇斌,智伟,谢彗琪,邓力,杨志明,王亚柱. C2C12成肌细胞体外诱导分化为肌管的实验[J].中山大学学报(医学科学版),2008,29(1):10-15.doi:10.3321/j.issn:1672-3554.2008.01.003.
Chen Y L,Zhou G Q,Deng Y B,Zhi W,Xie H Q,Deng L,Yang Z M,Wang Y Z. In vitro inducement of C2C12 myoblasts into differentiation of myotubes[J].Journal of Sun Yat-Sen University(Medical Sciences),2008,29(1):10-15.
[50] Tian H Z,She Z W,Gao X J,Wang W P,Tian H. MicroRNA-31 regulates dental epithelial cell proliferation by targeting Satb2[J].Biochemical and Biophysical Research Communications,2020,532(2):321-328.doi:10.1016/j.bbrc.2020.07.138.
[51] 王萌. miR-34c通过YY1调控骨骼肌细胞增殖及分化的机制研究[D].北京:中国农业大学,2017.
Wang M. miR-34c regulates the proliferation and differentiation of skeletal muscle cells by targeting YY1[D].Beijing:China Agricultural University,2017.
[52] 韩福慧,李倩,栾兆进,王国义,柳楠,贺建宁,薛明. miR-374b及其靶基因Myf6调控成肌细胞增殖分化的研究[J].中国畜牧杂志,2020,56(8):95-100. doi:10.19556/j.0258-7033.20190726-07.
Han F H,Li Q,Luan Z J,Wang G Y,Liu N,He J N,Xue M.Proliferation and differentiation regulation of miR-374b on ovine myoblasts by targeting Myf6 gene[J].Chinese Journal of Animal Science,2020,56(8):95-100.
[53] 侯欣华. 猪骨骼肌相关miRNA的鉴定及miR-378对肌肉发育调控的研究[D].南京:南京农业大学,2011.
Hou X H. Identification of porcine skeletal muscle related miRNA and regulation role of miR-378 in myogenesis[D].Nanjing:Nanjing Agricultural University,2011.
[54] 刘建华,梁煜,车雨彤,王凤,刘旭莹,赵弼时,王强,安丽霞,乔利英,刘文忠. miR-128-1-sp对绵羊前体脂肪细胞增殖和分化调节作用的研究[J].畜牧兽医学报,2020,51(6):1207-1218.doi:10.11843/j.issn.0366-6964.2020.06.005.
Liu J H,Liang Y,Che Y T,Wang F,Liu X Y,Zhao B S,Wang Q,An L X,Qiao L Y,Liu W Z.Study on the regulation of miR-128-1-sp in proliferation and differentiation of ovine preadipocyte[J].Acta Veterinaria et Zootechnica Sinica,2020,51(6):1207-1218.
[55] 宋成创.IGF2基因来源的IGF2 AS和miR-483调控牛骨骼肌细胞增殖分化机制研究[D].杨凌:西北农林科技大学,2019.
Song C C. IGF2 gene-derived IGF2 AS and miR-483 regulate proliferation and differentiation of bovine skeletal muscle cells[D].Yangling:Northwest Agriculture and Forestry University,2019.
[56] Nguyen H T,Frasch M. MicroRNAs in muscle differentiation:Lessons from Drosophila and beyond[J].Current Opinion in Genetics & Development,2006,16(5):533-539. doi:10.1016/j.gde.2006.08.010.
[57] Chen J F,Mandel E M,Thomson J M,Wu Q L,Callis T E,Hammond S M,Conlon F L,Wang D Z.The role of microRNA-1 and microRNA-133 in skeletal muscle proliferation and differentiation[J].Nature Genetics,2006,38(2):228-233. doi:10.1038/ng1725.
[58] Boutz P L,Chawla G,Stoilov P,Black D L. MicroRNAs regulate the expression of the alternative splicing factor nPTB during muscle development[J].Genes & Development,2007,21(1):71-84. doi:10.1101/gad.1500707.
[59] Chen X,Wang K H,Chen J N,Guo J G,Yin Y,Cai X,Guo X,Wang G Q,Yang R,Zhu L Y,Zhang Y,Wang J,Xiang Y,Weng C Y,Zen K,Zhang J F,Zhang C Y. In vitro evidence suggests that miR-133a-mediated regulation of uncoupling protein 2(UCP2)is an indispensable step in myogenic differentiation[J].The Journal of Biological Chemistry,2009,284(8):5362-5369.doi:10.1074/jbc.M807523200.