苎麻是重要的天然纤维原料,其纤维具有防臭抑菌、透气抗电、清凉舒爽等优良品质,其他纤维无法替代。苎麻原料中除含有天然纤维外,还包含许多果胶等“胶质”成分。要获得苎麻中的纤维,必须经过“脱胶”处理[1]。传统的沤麻工艺效率低,对环境污染严重、水资源严重浪费,化学脱胶工艺能耗高、成本高、纤维品质劣化,而生物脱胶具有节能、环保、高效的优点,为苎麻脱胶的发展方向[2-3]。
生物脱胶需要一系列酶协同完成,其中果胶酶是苎麻生物脱胶关键因子之一,而脱胶果胶酶的主要成分是果胶裂解酶(Pectate lyase,PEL,EC 4.2.2.2)[2]。PEL对果胶底物的亲和力与果胶甲酯化程度呈正相关,可直接通过反式消除作用随机切割甲酯化果胶的α-1,4糖苷键,在果胶非还原性末端的C5处消去一个氢原子到还原性末端的C1处生成羟基,非还原性末端生成带不饱和双键的聚半乳糖醛酸[4]。据报道,Bacillus licheniformis HDYM-03、Paenibacillus polymyxa KF-1、Pectobacterium carotovorum HG-49和Dickeya dadantii DCE-01是目前可用于工业化苎麻生物脱胶的微生物[5-8]。从这些苎麻脱胶微生物中发掘优良果胶裂解酶并使其高效表达,是一条提高脱胶果胶裂解酶研究效率的有效途径。多基因家族编码的PEL广泛存在于微生物中,且降解来源不同果胶的功能特性存在明显差异[9-11]。因此,PEL脱胶功能特性与脱胶机制一直是生物脱胶领域关注的热点,但在苎麻生物脱胶功能特征上的研究报道[12]相对较少[12]。本研究拟在筛选得到苎麻高效脱胶菌株D. dadantii DCE-01[1],并将其PEL基因pelG403克隆至质粒pEASY-Blunt E1的基础上,将pelG403基因更换至质粒拷贝数更高的pET28a载体中,诱导其高效表达,同时对其进行相关生物信息学分析,探索苎麻脱胶果胶裂解酶基因pelG403高效表达的方法和途径,并为脱胶果胶裂解酶关键位点分析、分子改造奠定基础。
1 材料和方法
1.1 试验材料
1.1.1 菌株与载体 pEASY-Blunt E1-G403/BL21工程菌由中国农业科学院麻类研究所生物加工研究室构建与保藏;表达载体pET28a、原核克隆感受态E.coli TOP10与原核表达感受态E.coli BL21(DE3)均购自Novagen公司。
1.1.2 主要试剂 超强高保真PCR试剂盒Ultra HiFidelity PCR Kit、DNA Marker Ⅲ、IPTG、卡那霉素(Kan)、DNA回收试剂盒和质粒抽提试剂盒均购自天根生物公司;其余常规化学试剂等商业化产品购自国药集团;引物合成和DNA序列测定由长沙擎科生物公司完成。
1.1.3 主要仪器 PCR仪,BioRad公司;凝胶快速成像仪,Vilber公司;冷冻离心机,Sigma公司;核酸电泳槽,北京六一公司;核酸浓度检测仪,Thermo Fisher公司;酶标仪,Thermo Fisher公司;振荡培养箱,上海知楚仪器公司。
1.2 试验方法
1.2.1 pelG403基因克隆 根据果胶裂解酶基因pelG403的碱基序列(GenBank登录号:JX964998),利用生物信息学软件SnapGene设计引物。正向引物和反向引物分别带入限制性酶切位点Nde Ⅰ和Xho Ⅰ,正向引物F的序列:5′-CGCATATGATGCCCATCTCACA
TTTTTC-3′(划线部分为Nde Ⅰ酶切位点),反向引物R的序列:5′-CCTCGAGTTATTTACAAGCTGAGCTGG-3′(划线部分为XhoⅠ酶切位点)。
以pEASY-Blunt E1-G403重组质粒进行PCR扩增。PCR反应体系:质粒模板1 μL,正、反向引物(10 μmol/L)各0.75 μL,2×UltraHiFi Mix 12.5 μL,用ddH2O补足至25 μL。PCR参数设置:94 ℃ 2 min;98 ℃ 10 s,60 ℃ 30 s,68 ℃ 15 s,35个循环;68 ℃,5 min。凝胶电泳检测PCR 产物后回收目的片段,用限制性内切酶NdeⅠ、XhoⅠ分别对回收的目的片段和pET28a质粒进行酶切;回收酶切后的pelG403目的片段和pET28a载体片段,加入T4 DNA 连接酶过夜,获得重组质粒pET28a-G403(图1)。将重组质粒转化E.coli TOP10克隆感受态细胞,经Kan抗性平板培养16~20 h;挑选典型单菌落,经PCR鉴定后送至长沙擎科公司进行核酸测序验证。
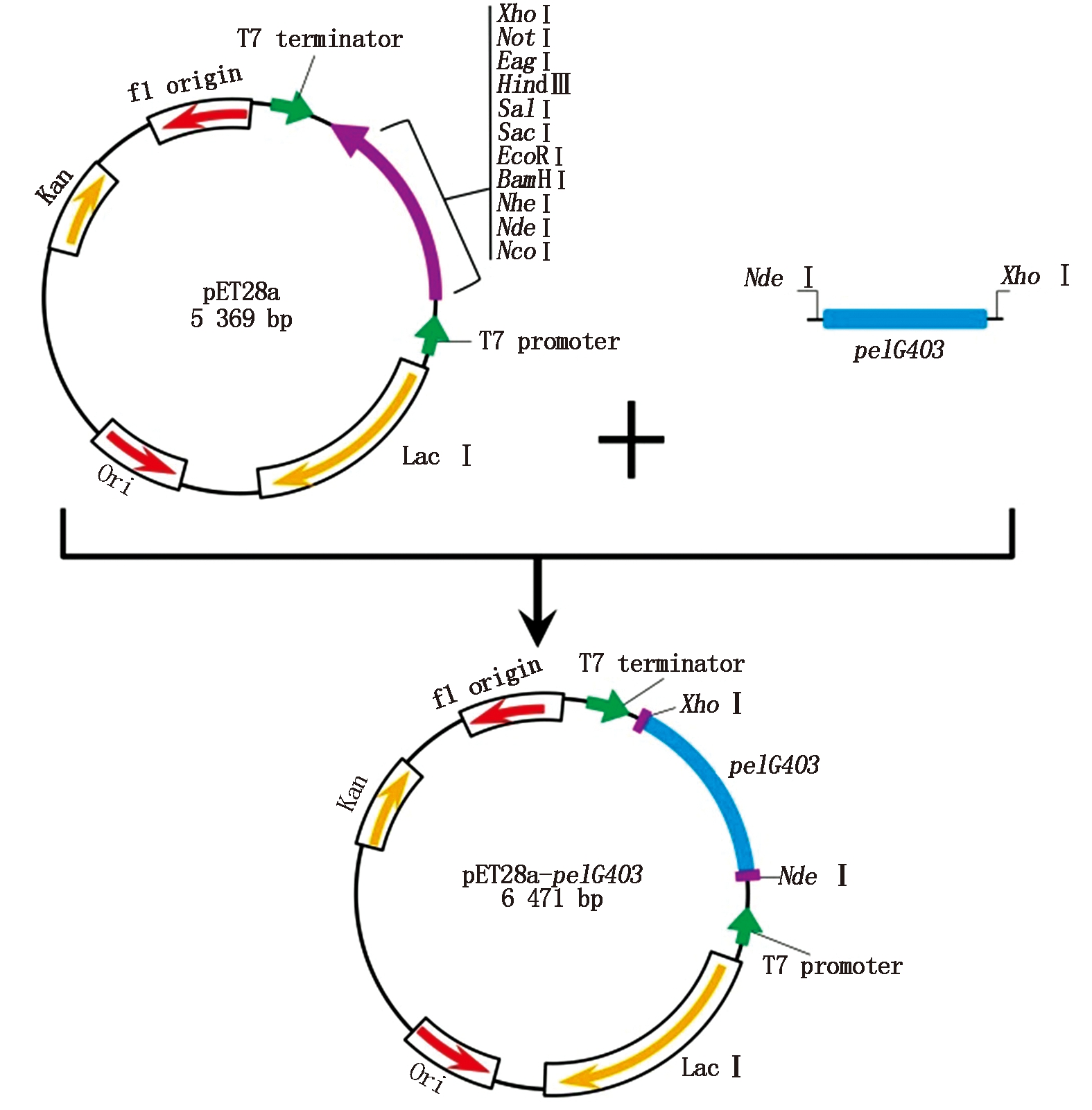
图1 果胶裂解酶重组子构建
Fig.1 Schematic diagram of expression vector for pectate lyase gene
1.2.2 果胶裂解酶PelG403的诱导表达 取测序检验正确的工程菌pET28a-G403/TOP至Kan抗性LB培养基,37 ℃,220 r/min,培养12 h后抽提质粒。将重组质粒pET28a-G403转化E.coli BL21(DE3)表达感受态中,经Kan抗性平板筛选,挑选典型阳性克隆接种于Kan抗性筛选的LB培养基,37 ℃,200 r/min培养至菌体OD600为0.4~0.6,加入IPTG(终浓度为0.5 mmol/L),28 ℃,150 r/min诱导PelG403表达。
1.2.3 重组蛋白的检测分析 SDS-PAGE检测:取 1 mL 的诱导成熟的工程菌菌液,5 000 r/min 10 min,弃废液,生理盐水重悬洗涤菌体后用40 μL灭菌ddH2O充分混匀,加入 10 μL 5×Loading Buffer,煮沸3~5 min,以未导入目的基因的pET28a/BL21菌株做相同处理作阴性对照;各取10 μL上样SDS-PAGE预制胶,电压130 V电泳60 min,考马斯亮蓝染色观察蛋白条带。将染色后的SDS-PAGE胶浸于缓冲液中,剪取NC 膜和滤纸一起放入缓冲液中平衡,湿转,封闭,孵育一抗,孵育二抗,TMB显色进行Western Blot 鉴定[13-14]。
1.2.4 果胶裂解酶活力测定 用0.05 mol/L的Gly-NaOH的缓冲液(pH值9.0)配置聚半乳糖醛酸钠底物溶液(5 mg/mL)。添加适量工程菌发酵液上清至1 mL底物溶液,摇匀后50 ℃酶解反应10 min,加2 mL DNS,沸水浴显色5 min;以灭活相同酶做阴性对照,测定OD520[15]。酶活力定义:底物1 min分解出1 μmol不饱和还原性物质需要的酶量为1个酶活力单位,以U表示。
1.2.5 PelG403生物信息学分析 利用生物信息学在线网站Expasy(https://web.expasy.org/protparam/)分析PelG403序列的基本理化性质;SignalP-5.0(http://www.cbs.dtu.dk/services/SignalP/)预测其信号肽;用Pfam(http://pfam.xfam.org/search/sequence)对PelG403的结构域进行分析。
用生物信息学软件Mega 7.0对来源于DCE-01菌株的PelG403氨基酸序列与其他微生物来源的PEL氨基酸序列进行聚类分析,随后ClustalW软件分别进行双序列比对;用计算机在线软件PSIPRED(http://bioinf.cs.ucl.ac.uk/psipred/)预测果胶裂解酶PelG403的二级结构;利用瑞士生物信息学中心在线工具SWISS-MODEL(https://swissmodel.expasy.org/)对果胶裂解酶PelG403进行同源建模,VMD进行作图分析,并利用Verify-3D对模型进行评分[16]。
2 结果与分析
2.1 pelG403基因扩增
对引入Nde Ⅰ和Xho Ⅰ酶切位点的目的基因进行检验,图2结果表明,其片段约为1 200 bp,这与预期的1 179 bp(pelG403目的基因片段与引入的酶切位点之和)相符。
2.2 菌落PCR鉴定
随机挑取4个单菌落,进行PCR鉴定。图3结果表明:4个转化子的PCR产物均有一特异条带,其分子量(约1 200 bp)与预计大小(1 179 bp)基本一致。
2.3 PelG403表达效果分析
2.3.1 SDS-PAGE分析 将pelG403从重组质粒pEASY-Blunt E1-G403更换至pET28a载体后,收集诱导成熟的pET28a-G403/BL21菌体,对PelG403表达效果进行SDS-PAGE分析。图4结果表明,基因工程菌菌体样品在35 ku上方有较清晰的专一蛋白条带,但与预测带有20个氨基酸的His标签的融合蛋白分子量(43.6 ku)相比略小。
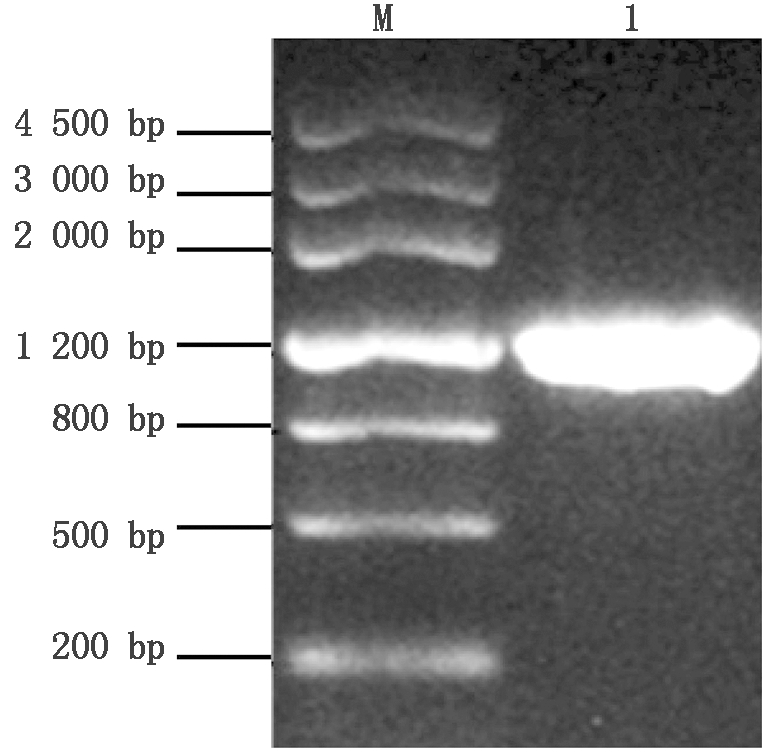
M.DNA Marker Ⅲ;1. 扩增的pelG403基因。
M.DNA Marker Ⅲ;1.Amplified pelG403 gene.
图2 pelG403基因PCR扩增
Fig.2 PCR amplification of pectate gene pelG403
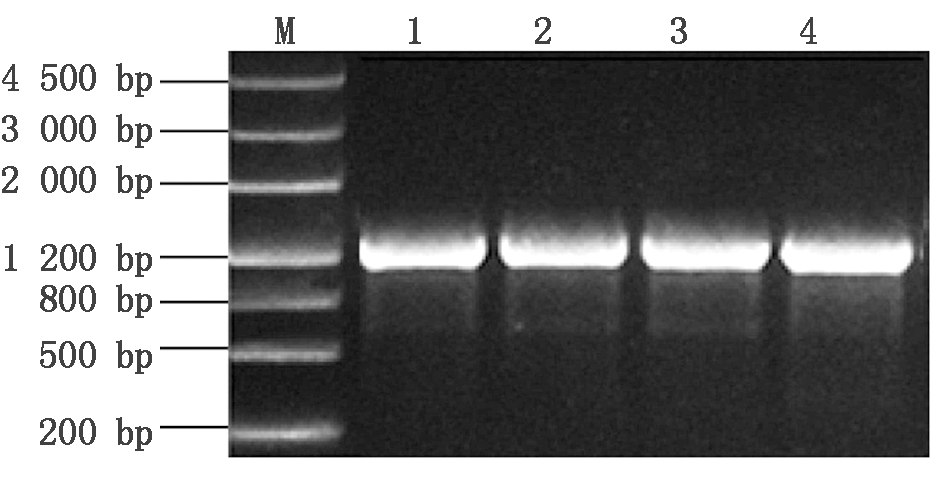
M. DNA Marker Ⅲ;1-4. 4个阳性克隆的PCR产物。
M. DNA Marker Ⅲ;1-4.PCR products of four positive clones.
图3 阳性克隆鉴定
Fig.3 Identification of positive clones
Western Blot对菌体蛋白进一步分析,图4结果表明,在34~43 ku有明显信号条带。其分子量也比预期融合蛋白分子量43.6 ku略小。
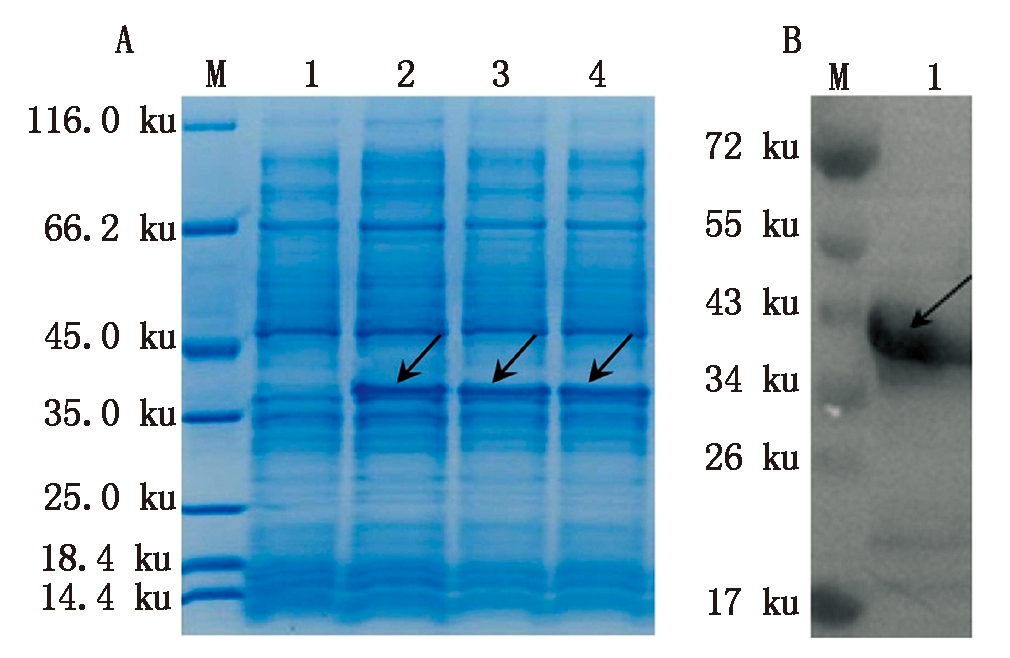
A.SDS-PAGE分析: M.Protein Marker;1.阴性对照;2-4.诱导样品。
B.Western Blot分析: M.Protein Marker;1.诱导样品。
A.SDS-PAGE analysis:M.Protein Marker;1.Negative control;2-4.Induced
samples:B.Western Blot analysis: M.Protein Marker;1.Induced samples.
图4 PelG403表达效果分析
Fig.4 Expression identification of PelG403
2.3.2 酶活力检测 取诱导12 h的pET28a-G403/BL21工程菌发酵液,测定其果胶裂解酶活力,表1结果表明,果胶裂解酶活力达335.8 U/mL,较pEASY-Blunt E1-G403/BL21提高了3.05倍。
表1 工程菌的果胶裂解酶活力
Tab.1 Pectate lyase activity of engineering bacteria

菌株Strains果胶裂解酶活力/(U/mL)Pectate lyase activitypET28a/BL210pET28a-G403/BL21335.8pEASY-Blunt E1-G403S/BL2182.8
2.4 果胶裂解酶PelG403生物信息学分析
2.4.1 基本性质分析 对果胶裂解酶基因及其编码产物进行生物信息学分析,结果表明:该果胶裂解酶基因pelG403序列全长为1 164 bp,编码的果胶裂解酶PelG403含387个氨基酸,其N末端前35个氨基酸为信号肽序列,前体蛋白和成熟蛋白的分子量大小约为41.4,37.8 ku,其理论pI为7.64,半胱氨酸数量为4个,不稳定性指数为27.42;该蛋白含有典型的右手β-螺旋结构,属于Pec lyase C家族。
2.4.2 亲缘关系分析 将来源于DCE-01菌株的苎麻脱胶果胶裂解酶PelG403氨基酸序列与其他微生物来源的果胶裂解酶氨基酸序列进行比较,并用Mega 7.0采用邻接法构建进化树(图5),结果表明:PelG403与同样来源于D.dadantii的果胶裂解酶(GenBank登录号:ADN00346)的亲缘关系最近,进一步用ClustalW软件将PelG403与进化树中亲缘关系较近的果胶裂解酶序列分别进行比对,目的序列与D.dadantii(ADN00346)、D.chrysanthemi(PDB:2EWE)和Pectobacterium versatile(ASN83858)相似性分别为95.9%,82.9%,82.0%。
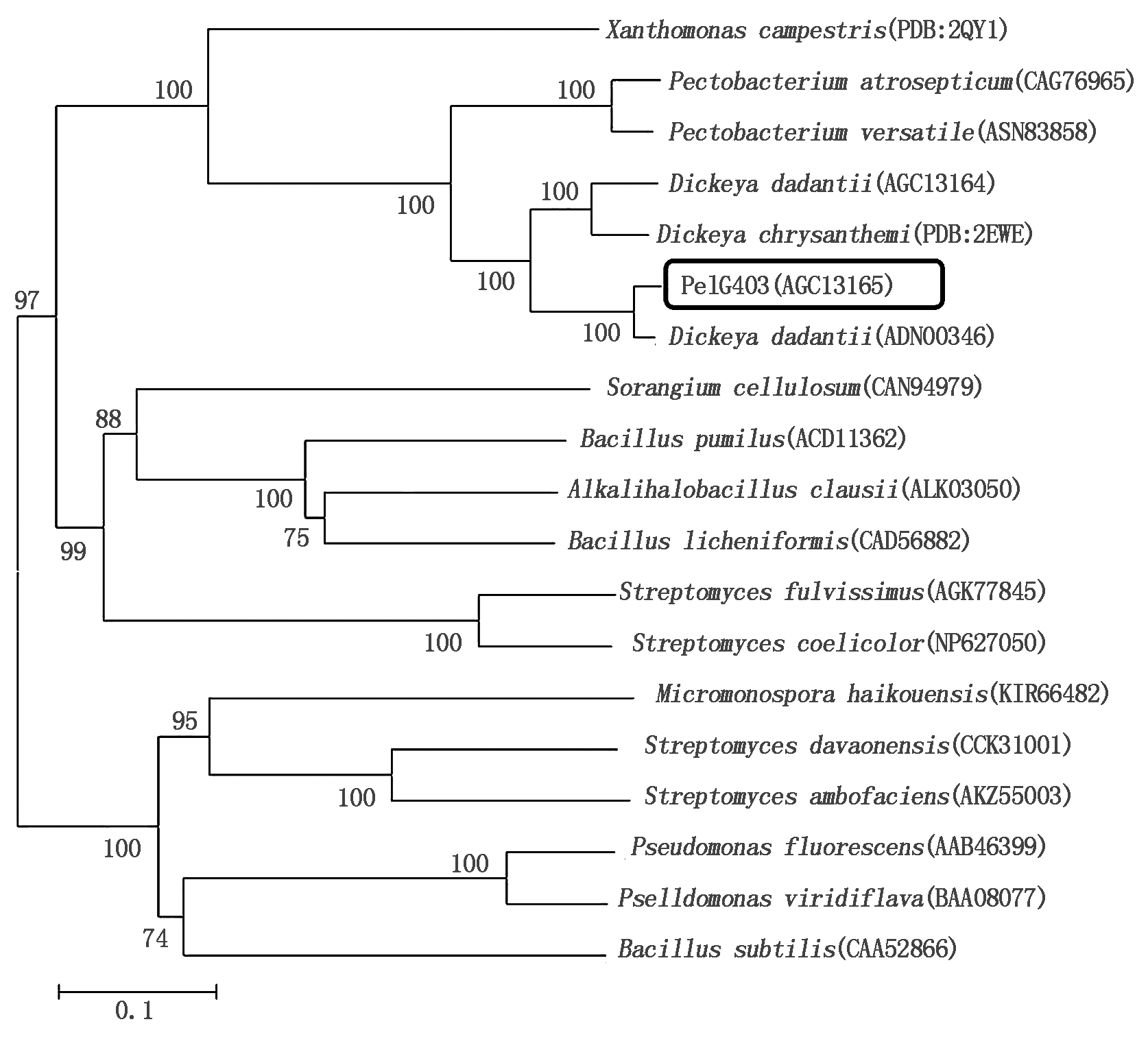
图5 果胶裂解酶氨基酸序列进化树
Fig.5 Evolutionary tree of amino acid sequence of pectate lyase
2.4.3 二级结构分析 利用PSIPRED生物信息工具分析PelG403的二级结构(图6),结果表明:在PelG403中,螺旋占总蛋白的10.2%;折叠占总蛋白的46.0%;卷曲占比43.8%。
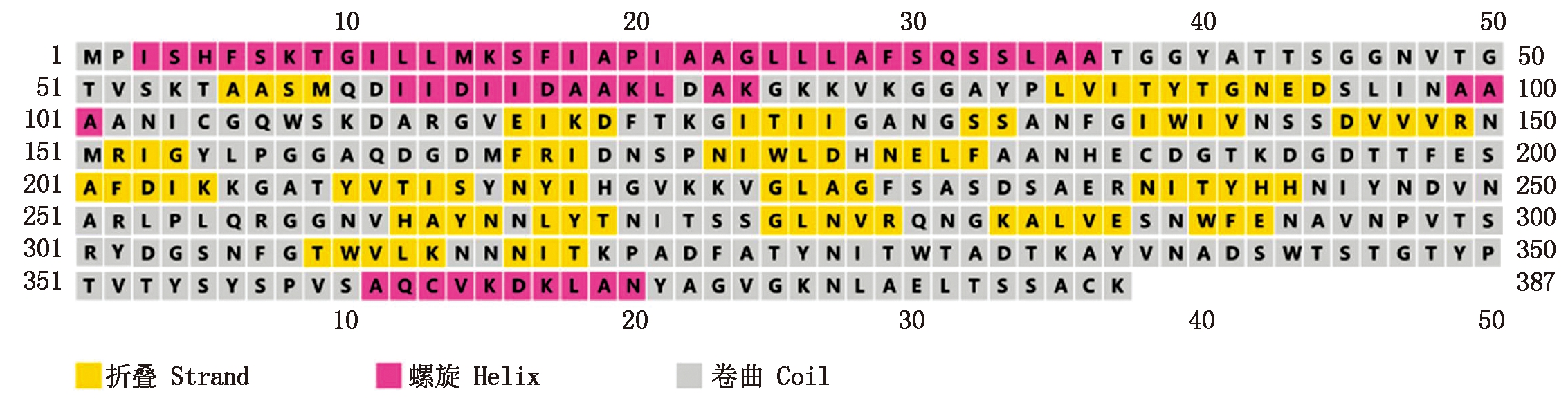
图6 PelG403二级结构预测
Fig.6 Secondary structure prediction of PelG403
2.4.4 高级结构分析 从PDB数据库中选取了同源性最高的细菌来源PEL蛋白空间结构(PDB:2EWE)为模板,对PelG403进行同源建模,结果表明(图7):PelG403主要由螺旋和卷曲构成,与二级结构预测的结果基本相符;且属于典型的右手β-螺旋结构,与Pfam结构域分析一致,表明PelG403经同源建模得到的蛋白三维结构可信度高。
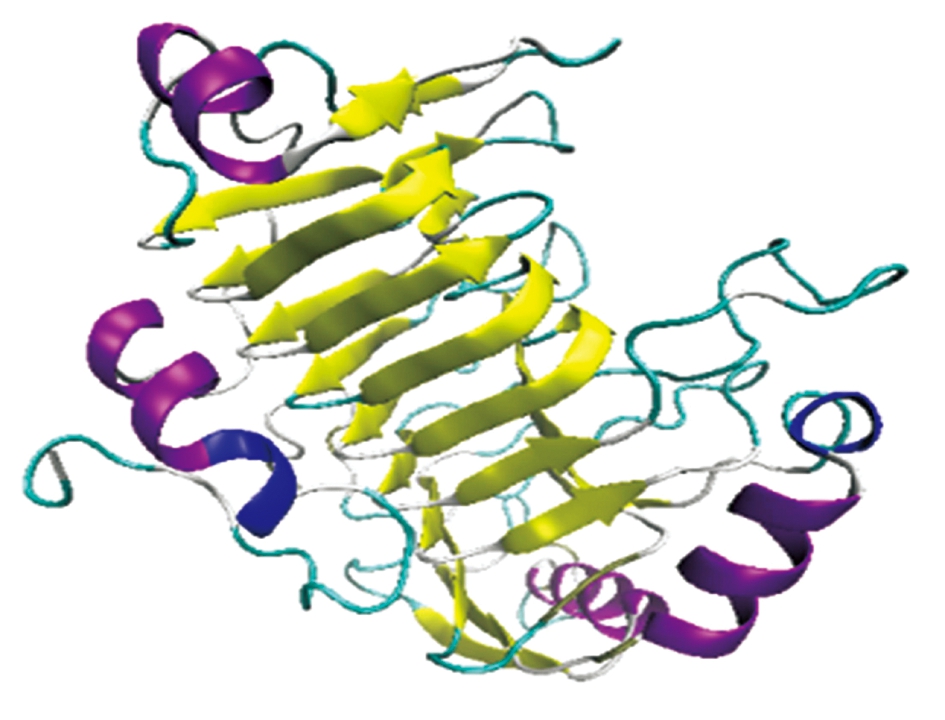
图7 PelG403三维结构预测
Fig.7 Three-dimensional structure prediction of PelG403
3 讨论
果胶裂解酶参与生物脱胶时不需要果胶去酯化,可直接作用于甲酯化果胶,促进半纤维素酶类和其他脱胶因子的有效渗透,其催化能力是苎麻生物脱胶的限速步骤[17-20]。因此,对PEL进行高效表达有助于获得大量优良工业用果胶裂解酶,降低纯化难度,有助于PEL脱胶功能与酶学特性的研究。
构建原核表达系统时,重组蛋白的表达量受诸多因素影响,如密码子偏好、启动子强度和表达质粒的质粒拷贝数等,这些因素对于蛋白的过量表达具有关键作用[21]。贺扬等[22]研究发现,利用pGEX-2T 和pET28a 2种质粒对IGF-2蛋白进行原核表达,虽然两者均表达出具有生物学活性的IGF-2蛋白,但质粒拷贝数更高的pET28a表达体系的产量远高于pGEX-2T,本研究通过更换质粒拷贝数更高的pET28a表达载体,使PelG403高效表达,其酶活力提高了3.05倍。这与前人的研究结果一致。
pET28a-G403/BL21表达产物的分子量比预测带6×His标签的融合蛋白分子量(43.6 ku)略小,而PelG403预测信号肽为35个氨基酸,6×His标签有20个氨基酸,而表达产物的分子量正好与除去这55个氨基酸大小一致;推测可能由于目的蛋白分泌后,其携带His标签序列伴随着信号肽序列一同被降解[23],有待进一步证实。
工业化脱胶加工时,需要相对高温高碱的反应环境,当PEL相应稳定性不够时,PEL的空间结构容易被破坏,果胶裂解酶的活性大幅度降低,甚至完全失活[24]。本研究利用三维结构建模、同源序列建树、氨基酸序列比、结合重要结构域对苎麻脱胶果胶裂解酶PelG403序列与结构进行分析,为该类酶的关键位点分析、分子改造及进一步揭示PEL脱胶功能特性奠定了基础。未来将进一步分析确定PelG403的催化位点及功能域,并结合其关键氨基酸位点进行定点突变,提高PelG403的比酶活和耐热耐碱性,为苎麻脱胶加工的酶制剂产业提供理想材料。
[1] 成莉凤,刘正初,冯湘沅,汪启明,李琦,郑科,杨琦,段盛文. 苎麻脱胶果胶复合酶的优选及其效果分析[J].纺织学报,2017,38(6):64-68. doi:10.13475/j.fzxb.20160601505.
Cheng L F,Liu Z C,Feng X Y,Wang Q M,Li Q,Zheng K,Yang Q,Duan S W. Screening on compound pectinase for ramie degumming and its effect analysis[J].Journal of Textile Research,2017,38(6):64-68.
[2] Guo F F,Zou M Y,Li X Z,Zhao J,Qu Y B. An effective degumming enzyme from Bacillus sp. Y1 and synergistic action of hydrogen peroxide and protease on enzymatic degumming of ramie fibers[J].BioMed Research International,2013,2013:212315. doi:10.1155/2013/212315.
[3] Biswas D,Chakrabarti S K,De S,Paral R. Eco-friendly degumming technology for ramie fiber[J].Journal of Natural Fibers,2016,13(2):227-237. doi:10.1080/15440478.2015.1005327.
[4] Yadav P K,Singh V K,Yadav S,Yadav K D S,Yadav D. In silico analysis of pectin lyase and pectinase sequences[J].Biochemistry (Moscow),2009,74(9):1049-1055. doi:10.1134/S0006297909090144.
[5] Ge J P,Yang Z Y,Du R P,Zhang L,Ping W X,Zhao D. Production of pectinolytic enzymes by two Bacillus spp. strains and their application in flax degumming[J].Transactions of Tianjin University,2019,25(4):413-419. doi:10.1007/s12209-018-0181-3.
[6] Yuan Y,Zhang X Y,Zhao Y,Zhang H,Zhou Y F,Gao J.A novel PL9 pectate lyase from Paenibacillus polymyxa KF-1:cloning,expression,and its application in pectin degradation[J].International Journal of Molecular Sciences,2019,20(12):3060-3077.doi:10.3390/ijms20123060.
[7] Wang Y W,Bai Y,Shu T,Fan P,Zhang H S,Turunen O,Xiong H R,Yu L J.Characterization of a versatile glycoside hydrolase Cel5M from Pectobacterium carotovorum HG-49 for ramie degumming[J].Textile Research Journal,2020,90(13/14):1602-1615.doi:10.1177/0040517519894748.
[8] Cheng L F,Duan S W,Zheng K,Feng X Y,Yang Q,Liu Z Y,Liu Z C,Peng Y D. An alkaline pectate lyase D from Dickeya dadantii DCE-01:clone,expression,characterization,and potential application in ramie bio-degumming [J].Textile Research Journal,2019,89(11):2075-2083. doi:10.1177/0040517518790971.
[9] 董俊帅,秦星,张志伟,张宇宏,张伟.宏基因组来源果胶酸裂解酶在毕赤酵母中的表达及应用[J].生物技术进展,2018,8(3):237-245,277. doi:10.19586/j.2095-2341.2018.0041.
Dong J S,Qin X,Zhang Z W,Zhang Y H,Zhang W. Expression and application of a pectate lyase from metagenome in Pichia pastoris[J].Current Biotechnology,2018,8(3):237-245,277.
[10] Kumar S,Jain K K,Singh A,Panda A K,Kuhad R C.Characterization of recombinant pectate lyase refolded from inclusion bodies generated in E. coli BL21(DE3)[J].Protein Expression and Purification,2015,110:43-51. doi:10.1016/j.pep.2014.12.003.
[11] Wang X W,Lu Z H,Xu T,Selvaraj J N,Yi L,Zhang G M. Improving the specific activity and thermo-stability of alkaline pectate lyase from Bacillus subtilis 168 for bioscouring[J].Biochemical Engineering Journal,2018,129:74-83. doi:10.1016/j.bej.2017.11.001.
[12] 王亚伟. 胡萝卜软腐果胶杆菌苎麻脱胶增效的关键酶及应用研究 [D].武汉:华中科技大学,2019.
Wang Y W. Characterization and application of the key enzymes for efficient ramie degumming by Pectobacterium carotovorum[D].Wuhan:Huazhong University of Science and Technology,2019.
[13] 胡月清,王若仲,黄志刚,萧浪涛.马铃薯转录因子基因StWRKY57的克隆及其蛋白原核表达[J].华北农学报,2021,36(1):10-17.doi:10.7668/hbnxb.20191222.
Hu Y Q,Wang R Z,Huang Z G,Xiao L T. Cloning and prokaryotic expression of transcription factor StWRKY57 gene in potato[J].Acta Agriculturae Boreali-Sinica,2021,36(1):10-17.
[14] 赵丽玲,王会,柴志欣,王吉坤,王嘉博,武志娟,信金伟,钟金城,姬秋梅. 牦牛lncFAM200B的克隆鉴定、表达及生物信息学分析[J].华北农学报,2020,35(5):220-230.doi:10.7668/hbnxb.20191071.
Zhao L L,Wang H,Chai Z X,Wang J K,Wang J B,Wu Z J,Xin J W,Zhong J C,Ji Q M. Cloning,expression and bioinformatics analysis of yak lncFAM200B[J].Acta Agriculturae Boreali-Sinica,2020,35(5):220-230.
[15] 于平,王欣馨,任倩,黄星星,易明花.产碱性果胶酶菌株的筛选和鉴定及其酶学性质[J].中国食品学报,2018,18(9):288-296. doi:10.16429/j.1009-7848.2018.09.037.
Yu P,Wang X X,Ren Q,Huang X X,Yi M H.Screening and identification of a high-yield alkaline pectinase strain and enzymatic properties of alkaline pectinase[J].Journal of Chinese Institute of Food Science and Technology,2018,18(9):288-296.
[16] Manasaryan G,Suplatov D,Pushkarev S,Drobot V,Kuimov ![]() V,Nilov D. Bioinformatic analysis of the nicotinamide binding site in poly(ADP-Ribose)polymerase family proteins[J].Cancers,2021,13(6):1201-1217. doi:10.3390/cancers13061201.
V,Nilov D. Bioinformatic analysis of the nicotinamide binding site in poly(ADP-Ribose)polymerase family proteins[J].Cancers,2021,13(6):1201-1217. doi:10.3390/cancers13061201.
[17] Zhang C J,Yao J,Zhou C,Mao L W,Zhang G M,Ma Y H.The alkaline pectate lyase PEL168 of Bacillus subtilis heterologously expressed in Pichia pastoris is more stable and efficient for degumming ramie fiber[J].BMC Biotechnology,2013,13(1):26. doi:10.1186/1472-6750-13-26.
[18] Yadav S,Dubey A K,Anand G,Kumar R,Yadav D. Purification and biochemical characterization of an alkaline pectin lyase from Fusarium decemcellulare MTCC 2079 suitable for Crotalaria juncea fiber retting[J].Journal of Basic Microbiology,2014,54(S1):S161-S169. doi:10.1002/jobm.201300281.
[19] Jenkins J,Mayans O,Pickersgill R. Structure and evolution of parallel β-helix proteins[J].Journal of Structural Biology,1998,122(1/2):236-246. doi:10.1006/jsbi.1998.3985.
[20] Fan P,He F,Yang Y,Ao M Z,Ouyang J,Liu Y,Yu L J. In-situ microbial degumming technology with Bacillus sp. HG-28 for industrial production of ramie fibers[J].Biochemical Engineering Journal,2015,97:50-58. doi:10.1016/j.bej.2014.12.010.
[21] 杨海泉,刘龙,李江华,堵国成,陈坚.微生物酶高效异源表达策略的最新研究进展[J].食品科学,2013,34(9):351-357.doi:10.7506/spkx1002-6630-201309070.
Yang H Q,Liu L,Li J H,Du G C,Chen J.Recent advance in high-level heterologous expression of microbial enzymes[J].Food Science,2013,34(9):351-357.
[22] 贺扬,余巧玲,王均,覃川杰,李华涛.罗非鱼原核表达基因研究进展[J].生物技术通报,2021,37(2):195-202. doi:10.13560/j.cnki.biotech.bull.1985.2020-0699.
He Y,Yu Q L,Wang J,Qin C J,Li H T.Advances in prokaryotic expression gene of tilapia[J].Biotechnology Bulletin,2021,37(2):195-202.
[23] Paetzel M.Bacterial signal peptidases[J].Sub-Cellular Biochemistry,2019,92:187-219. doi:10.1007/978-3-030-18768-2_7.
[24] Atalah J,C ceres-Moreno P,Espina G,Blamey J M.Thermophiles and the applications of their enzymes as new biocatalysts[J].Bioresource Technology,2019,280:478-488. doi:10.1016/j.biortech.2019.02.008.
ceres-Moreno P,Espina G,Blamey J M.Thermophiles and the applications of their enzymes as new biocatalysts[J].Bioresource Technology,2019,280:478-488. doi:10.1016/j.biortech.2019.02.008.