由于人类长期向大气中大量排放氟化物,大气臭氧层结构受到破坏,导致到达地表的紫外线(UV-B)辐射增强。大气平流层中臭氧每减少1%,到达地球表面的太阳紫外线辐射就增加2%[1-2];北半球因臭氧层破坏而导致的UV-B辐射年增加率将达到14%,南半球则达到40%[3];纬度和海拔是影响UV-B辐射的另一重要因素。研究表明,生活在低纬度地区的生物会比生活于高纬度地区的生物接受更多的UV-B辐射[4],海拔每升高1 000 m,UV-B辐射强度则提升10%~20%[5]。云南(海拔600~3 000 m,21~29°N)为典型的低纬高原,因此,该地区动植物受UV-B辐射增强的影响相对更严重。
紫甘薯为薯肉颜色紫色至深紫色的甘薯(Ipomoea batatas(L.)Lam.),云南境内紫甘薯种植面积已达2.388万hm2,居全国第2位[6]。紫甘薯除含有普通甘薯所具有的全部营养成分外,还富含花青素、类黄酮、绿原酸等药用成分,是一种兼具粮食、经济和药用特性于一身的重要作物[6]。当前,紫甘薯鲜食和加工市场需求因居民对紫甘薯营养保健功能认知度及认可度的不断提高,以及以花青素应用为代表的医疗、工业需求增加而不断扩张[7-9]。故适当发展优质紫甘薯栽培,不仅有利于优化居民的膳食结构,而且是云南发展“高原特色农业”“效益农业”的有效途径。为了适应市场对优质紫甘薯品种的消费需求,云南农业大学薯类作物研究所选育出优质高花色苷(0.59 mg/g)品种-滇紫甘薯24。目前,关于甘薯响应UV-B辐射增强的研究主要集中于农艺性状[10]和生理生化方面[11-12],而就紫甘薯响应UV-B辐射增强的转录组分析的研究则鲜有报道,因此,本研究利用RNA测序(RNA-sequencing,RNA-seq)技术对增强UV-B辐射处理前后的紫甘薯叶片样品进行转录组测序比对,鉴定出样品响应UV-B辐射增强的显著差异表达应答基因,并对其差异表达基因(Differentially expressed genes,DEGs)进行基因本体(Gene ontology,GO)和京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)注释与富集分析,从转录组水平上对紫甘薯响应UV-B辐射增强的分子机制进行深入研究,以期为紫甘薯耐UV-B辐射相关基因的筛选提供科学依据。
1 材料和方法
1.1 材料及处理
试验于云南农业大学试验田内进行(25.04° N,102.73° E,海拔1 950.0 m),采用盆栽(高35 cm,直径40 cm;基质:红壤土∶腐殖土=1∶1),株行距:35 cm×40 cm。于2017年6月15日扦插,发根缓苗后进行UV-B照射处理。将带防雨灯架的紫外灯安装在田间搭建的网架上,用紫外灯管(UVB-40,南京华强电子有限公司,波长280~320 nm)进行人工增补UV-B辐射,辐照强度以灯管至甘薯最高叶面的高度和灯管数量调节,用UV-B紫外辐照计测量辐照强度(北京师范大学光电仪器厂)。以自然光照为对照(CK),自然光照基础上增加7.2 kJ/(m2·d)为UV-B辐射处理(T),每天照射5 h(11:00-16:00),阴雨天除外[13]。灯管高度随植株的生长高度适时调节,同时,为了使对照组与处理组的自然光照条件一致,在其上安装空灯架。于处理第60 天随机选取15株紫甘薯,取倒5叶,3次生物学重复,每个重复5株,将样品迅速放入取样管后液氮速冻,实验室内储存于-80 ℃超低温冰箱待用。
1.2 测序样品总RNA提取
采用TRNzol试剂盒(Invitrogen,Carlsbad,CA,美国)从叶片组织中提取总RNA[14],1%的琼脂糖凝胶电泳(电压180 V,电泳时间16 min)和Nanodrop分光光度计(IMPLEN,CA,美国)检测RNA纯度,Agilent Bioanalyzer 2100(Agilent Technologies,Santa Clara,CA,美国)测定RNA的浓度和完整性。
1.3 转录组测序
将提取的样本总RNA送至北京诺禾致源生物信息科技有限公司测序并进行文库构建。采用Illumina Hiseq 2500平台对质量合格的RNA文库进行测序。同时,使用SolexaQA package对Raw reads按照QPhred≥20和读长≥25 bp进行过滤,以获得高质量的Clean reads,并利用HISAT软件将已获得的Clean reads与甘薯参考基因组(http://public-genomes-ngs.molgen.mpg.de/sweetpotato/)进行序列比对分析。
1.4 差异表达转录因子基因的筛选
基因表达水平用FPKM(Expected number of fragments per kilobase of transcript sequence per millions base pairs sequenced)法计算[15]。借助DESeq(1.10.1)软件进行DEGs的筛选,筛选标准为|log2FC|≥2,padj<0.05,FDR<0.01。为了进一步研究差异表达基因的功能,利用GOSeq、topGO、hmmscan(Release 2.12)进行GO富集分析,显著富集标准为Corrected P-value<0.05;利用KOBAS(V2.0)对差异表达基因进行KEGG Pathway富集分析,显著富集标准为Corrected P-value<0.05。
1.5 qRT-PCR
提取对照组和处理组的叶片总RNA使用TUREscript 1st Stand cDNA Synthesis Kit(北京艾德莱生物科技有限公司)试剂盒合成cDNA。qRT-PCR用SYBR® Green Supermix在analytikjena-qTOWER2.2型荧光定量PCR仪上进行测定。反应条件为:95 ℃,3 min;95 ℃,10 s,58 ℃,30 s,39个循环;溶解曲线60~95 ℃,+1 ℃/cycle,保持时间4 s。基因的相对表达量用2-ΔΔCt方法计算[16],以18S DNA为内参(表1)。
表1 qRT-PCR引物序列
Tab.1 The primer sequences used in qRT-PCR
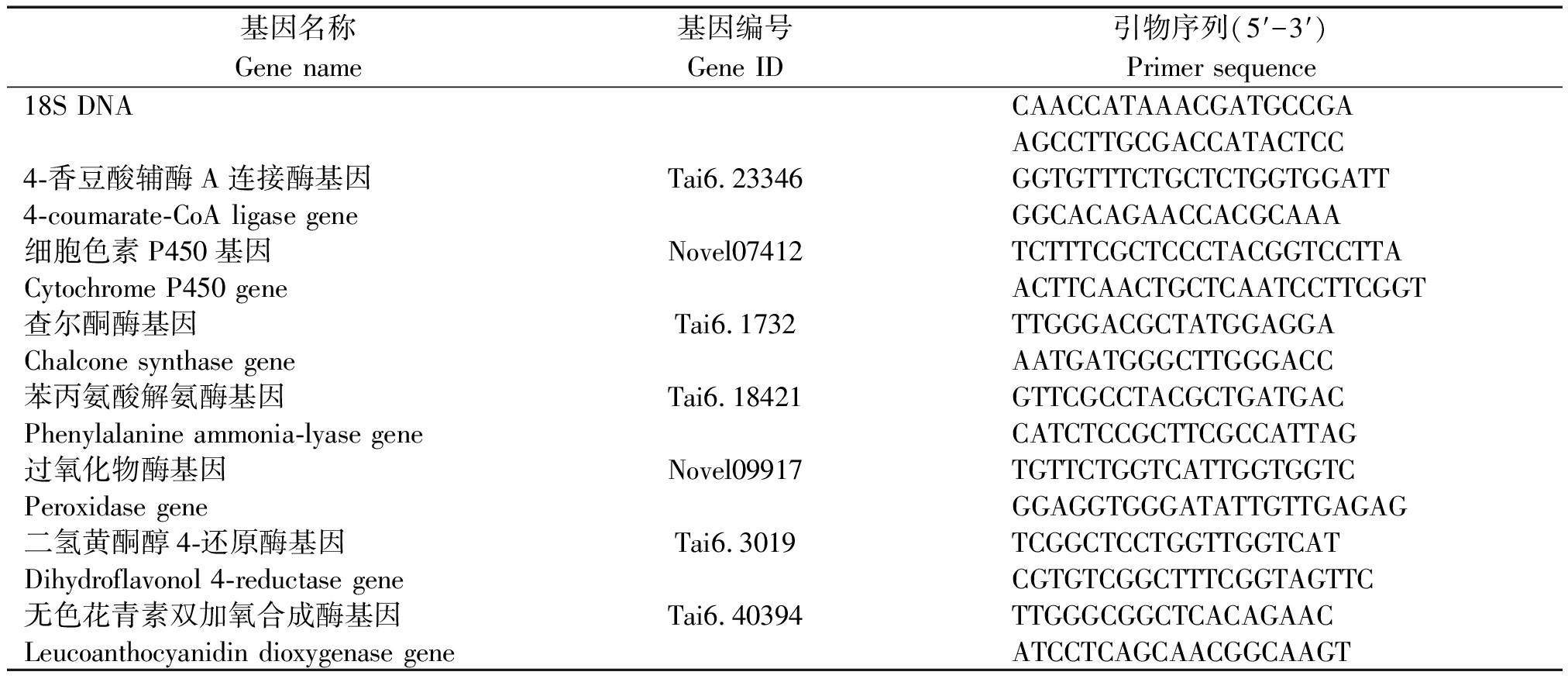
基因名称Gene name基因编号Gene ID引物序列(5′-3′)Primer sequence18S DNACAACCATAAACGATGCCGAAGCCTTGCGACCATACTCC4-香豆酸辅酶A连接酶基因Tai6.23346GGTGTTTCTGCTCTGGTGGATT4-coumarate-CoA ligase geneGGCACAGAACCACGCAAA细胞色素P450基因Novel07412TCTTTCGCTCCCTACGGTCCTTACytochrome P450 geneACTTCAACTGCTCAATCCTTCGGT查尔酮酶基因Tai6.1732TTGGGACGCTATGGAGGAChalcone synthase geneAATGATGGGCTTGGGACC苯丙氨酸解氨酶基因Tai6.18421GTTCGCCTACGCTGATGACPhenylalanine ammonia-lyase geneCATCTCCGCTTCGCCATTAG过氧化物酶基因Novel09917TGTTCTGGTCATTGGTGGTCPeroxidase geneGGAGGTGGGATATTGTTGAGAG二氢黄酮醇4-还原酶基因Tai6.3019TCGGCTCCTGGTTGGTCATDihydroflavonol 4-reductase geneCGTGTCGGCTTTCGGTAGTTC无色花青素双加氧合成酶基因Tai6.40394TTGGGCGGCTCACAGAACLeucoanthocyanidin dioxygenase geneATCCTCAGCAACGGCAAGT
2 结果与分析
2.1 总RNA质量特征
由图1可以看出,各样品总RNA条带清晰无弥散、无降解、无污染。自然光照与增强UV-B辐射下的OD260/280分别为2.000~2.138,OD260/230分别为1.509~2.102,RNA浓度分别为160~252 ng/μL,RIN分别为5.9~7.6,各值均在检测合格范围内,这表明各样品提取的总RNA纯度较高,质量符合上机测序要求(表2)。
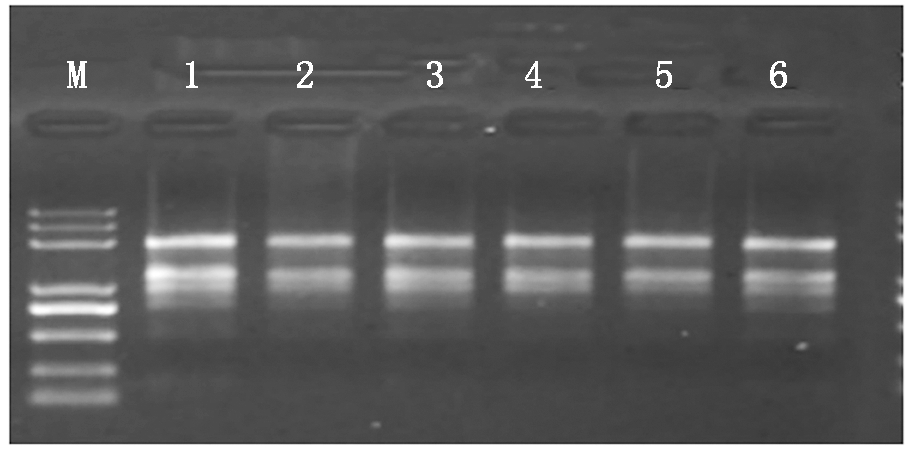
M.Trans 2KPlus;泳道编号对应表2样品序号。
M.Trans 2KPlus;The lane number
corresponded to the Tab.2 sample number.
图1 总RNA琼脂糖凝胶电泳检测
Fig.1 Total RNA agarose gel electrophoresis
表2 样品总RNA质检结果
Tab.2 The sample total RNA quality test results
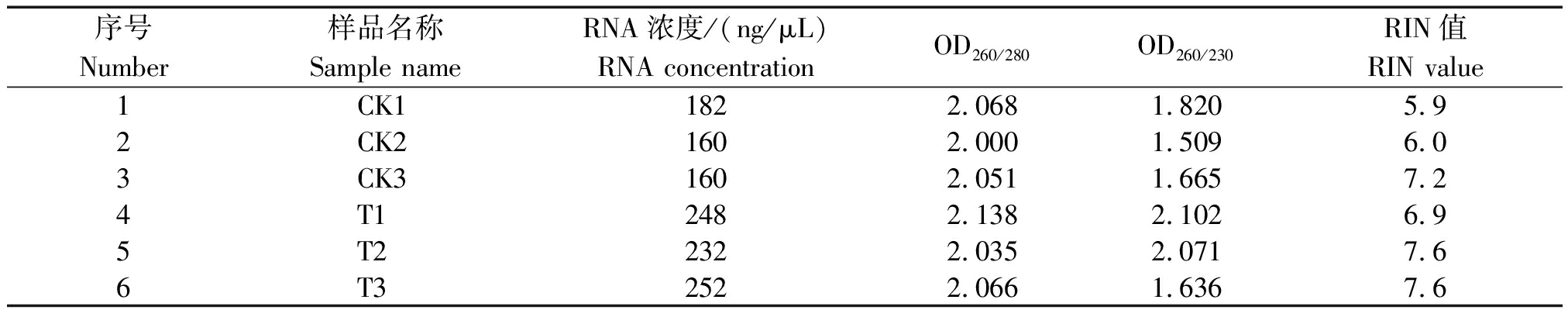
序号Number样品名称Sample nameRNA浓度/(ng/μL)RNA concentrationOD260/280OD260/230RIN值RIN value1CK11822.0681.8205.92CK21602.0001.5096.03CK31602.0511.6657.24T12482.1382.1026.95T22322.0352.0717.66T32522.0661.6367.6
2.2 紫甘薯叶片RNA测序质量分析
将Raw reads和Clean reads比对到甘薯参考序列,CK和T的Raw reads和Clean reads的比例相近,且二者中比对到参考序列的比率分别为72.56%和73.70%,充分说明测序数据质量可信[17](表3)。
表3 样品测序数据统计结果
Tab.3 Statistical results of the sample sequencing data
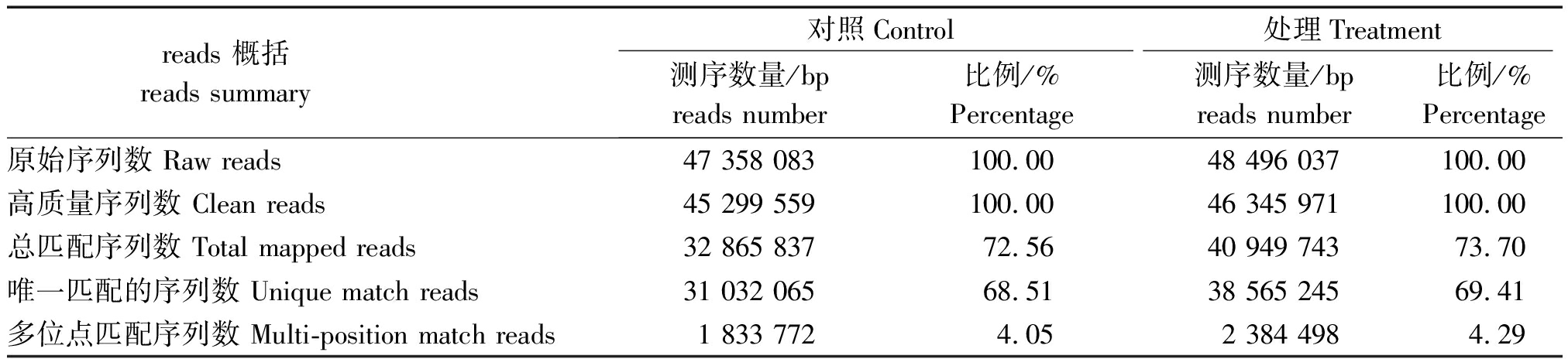
reads 概括 reads summary 对照Control处理Treatment测序数量/bp reads number比例/%Percentage测序数量/bpreads number比例/% Percentage原始序列数 Raw reads47 358 083100.0048 496 037100.00高质量序列数 Clean reads45 299 559100.0046 345 971100.00总匹配序列数 Total mapped reads32 865 83772.5640 949 74373.70唯一匹配的序列数 Unique match reads31 032 06568.5138 565 24569.41多位点匹配序列数 Multi-position match reads1 833 7724.052 384 4984.29
2.3 紫甘薯叶片响应UV-B辐射增强下的差异表达基因分析
UV-B辐射处理后紫甘薯叶片中共有477个基因表达发生显著变化,其中上调表达的基因有382个,占总差异表达基因的80.08%,下调表达基因有95个,占总差异表达基因的19.92%。由图2可以看出,差异表达基因中变化倍数在2~3倍、3~5倍、5倍以上的上(下)调差异表达基因分别占总上(下)调基因总数的57.59%(74.74%),34.55%(24.21%),7.85%(1.05%)。上述结果说明,UV-B辐射增强后紫甘薯的差异表达基因主要以上调表达为主,且变化幅度主要集中在2~3倍。
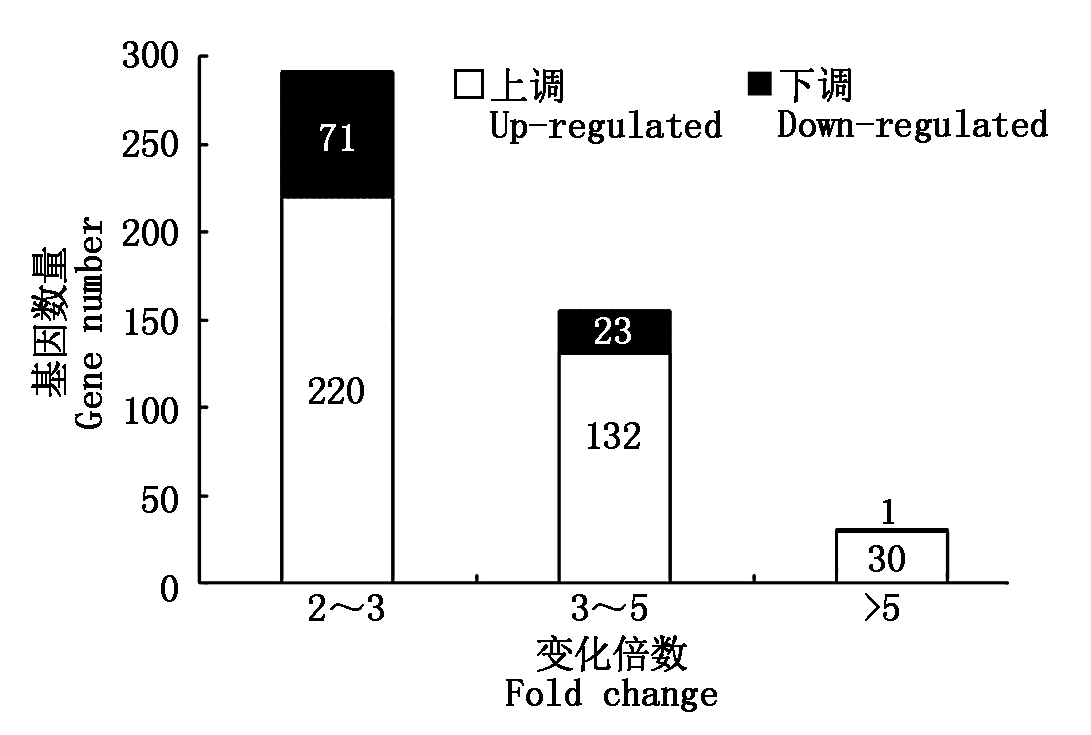
图2 紫甘薯叶片响应UV-B辐射增强下DEGs的统计分析
Fig.2 Statistical analysis of DEGs of purple sweet potato
leaf in response to enhanced the UV-B
2.4 紫甘薯叶片响应UV-B辐射增强下DEGs的GO功能富集分析
差异表达基因行使的主要生物学功能以GO功能显著性富集分析来确定。本研究共显著富集到分属于生物过程和分子功能的16条功能条目,表明UV-B辐射增强下紫甘薯叶片的DEGs主要通过这2个生物功能来提高自身的适应性。生物过程主要为氧化还原过程,其DEGs数占总数的63.92%,分子功能主要富集在氧化还原酶活性中,其DEGs占比为40.91%,表明紫甘薯叶片主要通过生物过程中的氧化还原过程和分子功能中的氧化还原酶活性来抵抗UV-B辐射带来的伤害,暗示UV-B辐射增强对紫甘薯叶片造成氧化伤害(表4)。
表4 紫甘薯叶片响应UV-B辐射增强下DEGs的GO功能分类
Tab.4 GO function categories of DEGs of purple
sweet potato leaf in response to enhanced the UV-B
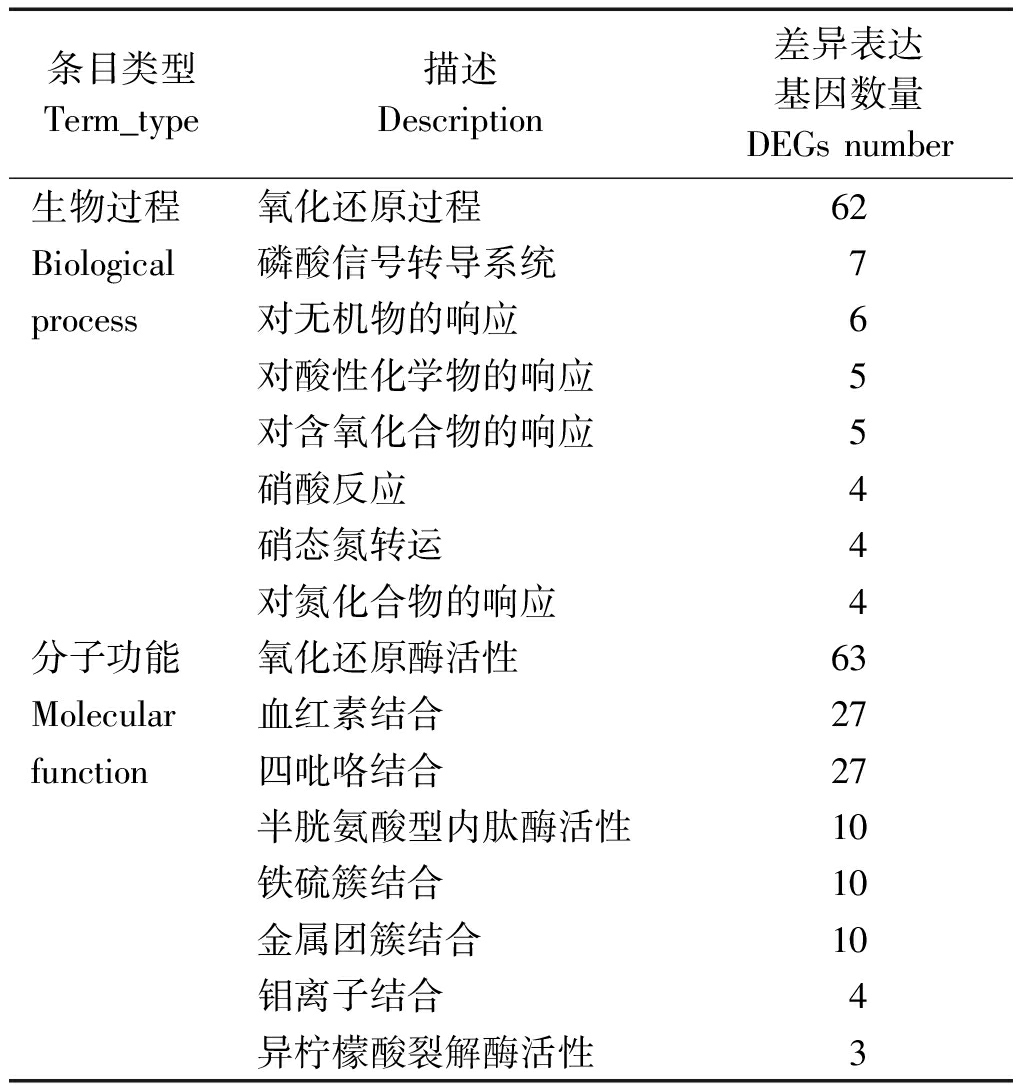
条目类型Term_type描述Description差异表达基因数量DEGs number生物过程氧化还原过程62Biological磷酸信号转导系统7process对无机物的响应6对酸性化学物的响应5对含氧化合物的响应5硝酸反应4硝态氮转运4对氮化合物的响应4分子功能氧化还原酶活性63Molecular血红素结合27function四吡咯结合27半胱氨酸型内肽酶活性10铁硫簇结合10金属团簇结合10钼离子结合4异柠檬酸裂解酶活性3
2.5 紫甘薯叶片响应UV-B辐射增强下DEGs的KEGG代谢通路显著性富集分析
KEGG是系统分析基因功能、基因组信息的数据库,能够较准确的分析生物体内的代谢和研究代谢网络。通过分析可知,DEGs主要分布在56条代谢通路中,其中达到显著差异水平的通路共有9条,分别为次生代谢产物生物合成、植物昼夜节律、苯丙素生物合成、类黄酮生物合成、氮代谢、苯丙氨酸代谢、芥子油苷生物合成、油菜素内酯生物合成、硫胺素代谢,其中次生代谢产物生物合成通路所包含的DEGs数量最多,暗示其为紫甘薯响应UV-B辐射增强的主要代谢通路(表5)。
表5 紫甘薯叶片响应UV-B辐射增强下
DEGs显著性富集的KEGG Pathway
Tab.5 KEGG Pathway with significant enrichment
of DEGs of purple sweet potato leaf in
response to enhanced the UV-B
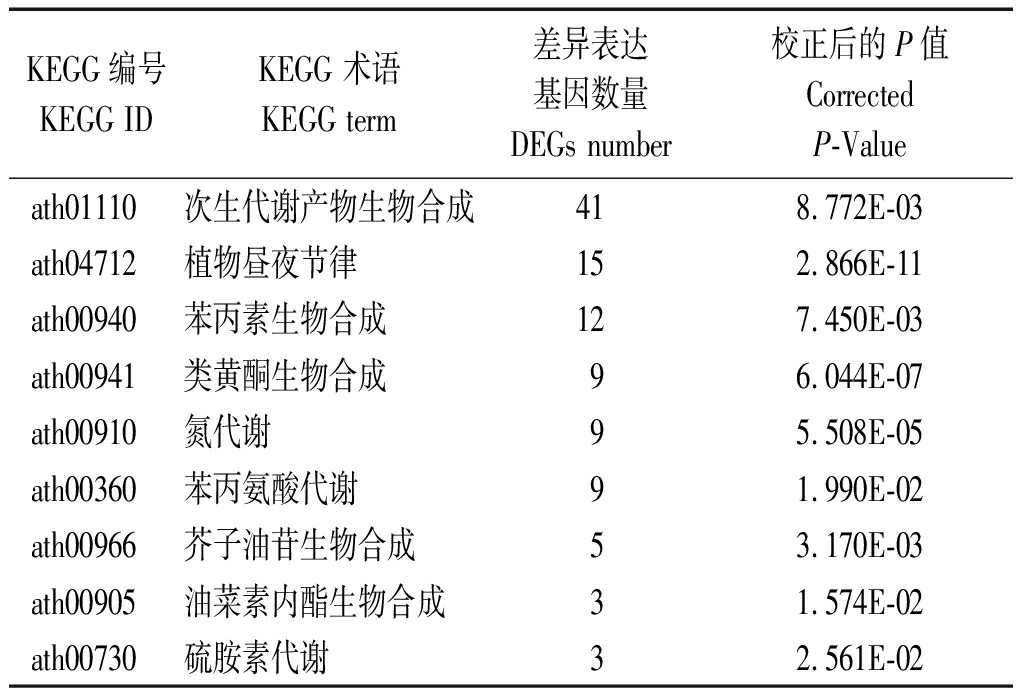
KEGG编号KEGG IDKEGG 术语KEGG term差异表达基因数量DEGs number校正后的P值Corrected P-Valueath01110次生代谢产物生物合成418.772E-03ath04712植物昼夜节律152.866E-11ath00940苯丙素生物合成127.450E-03ath00941类黄酮生物合成96.044E-07ath00910氮代谢95.508E-05ath00360苯丙氨酸代谢91.990E-02ath00966芥子油苷生物合成53.170E-03ath00905油菜素内酯生物合成31.574E-02ath00730硫胺素代谢32.561E-02
对次生代谢产物生物合成通路中的DEGs进行功能注释,结果发现,与类黄酮代谢相关的基因最多,如类苯丙氨酸解氨酶基因(Tai6.36393、Tai6.18421)、4-香豆酸-CoA连接酶6基因(Tai6.23346)、查尔酮合酶基因(CHS)(Tai6.1732、Tai6.53503、Tai6.45381)、查尔酮合酶DIV亚型X1基因(Tai6.1734、Tai6.46186)、二氢黄酮醇4-还原酶基因(DFR)(Tai6.3019)、咖啡酰-CoA O-甲基转移酶基因At4g26220亚型X1基因(Tai6.25039)、花青素合成酶基因(Tai6.42091、Tai6.40394)、类花青素3-O-葡萄糖基转移酶7基因(Tai6.49002);其次为细胞色素P450家族,如类细胞色素P450 71A1基因(Tai6.44113)、类细胞色素P450 82D47(Novel07412)、类细胞色素P450 83B1基因(CYP83B1)(Tai6.44115、Tai6.18064、Tai6.41206)、类细胞色素P450 84A1基因(Novel06436、Novel06172)、类细胞色素P450 85A1基因(CYP85A1)(Tai6.2806、Tai6.19253、Tai6.15801)(表6)。上述结果表明,这些基因可能在响应UV-B辐射中起关键作用。
表6 紫甘薯叶片响应UV-B辐射增强下的主要差异表达基因
Tab.6 Main DEGs of purple sweet potato leaf in response to enhanced the UV-B

基因编码Gene IDLog2值Log2 value基因注释Gene annotation基因编码Gene IDLog2值Log2 value基因注释Gene annotationTai6.22271-2.661-氨基环丙烷-1-羧酸合成酶异构体X1Tai6.474272.28类短链脱氢酶/还原酶2bTai6.213482.133-脱氢喹酸合成酶Tai6.131672.12类甘露醇脱氢酶Tai6.367812.33α-1,4葡聚糖磷酸化酶L-2同工酶Novel079563.36类过氧化物酶P7-likeTai6.236502.00丙酮酸脱羧酶1Novel01630-2.04类环烯醇合酶2Tai6.23346-2.514-香豆酸-CoA连接酶6Tai6.27653.24类阳离子过氧化物酶1Tai6.1732-3.30查尔酮合成酶Novel12255-4.97类异亮氨酸N-单加氧酶1Tai6.53503-2.49查尔酮合成酶Tai6.100974.14类组氨酸类脱羧酶Tai6.45381-2.54查尔酮合成酶Tai6.61352.10葡萄糖-6-磷酸1-脱氢酶Tai6.1734-2.76查尔酮合成酶DIV亚型X1Tai6.46181-2.70无特征蛋白LOC109163763Tai6.46186-2.73查尔酮合成酶DIV亚型X1Tai6.268372.93无特征蛋白LOC116021983Tai6.3019-4.52二氢黄酮醇4-还原酶Tai6.441132.82类细胞色素P450 71A1Tai6.42091-2.00花青素合成酶Novel074123.17类细胞色素P450 82D47Tai6.40394-2.10花青素合成酶Tai6.441152.56类细胞色素P450 83B1Tai6.25039-2.89咖啡酰-CoA O-甲基转移酶At4g26220亚型X1Tai6.180642.38类细胞色素P450 83B1Tai6.363932.14类苯丙氨酸解氨酶Tai6.412062.86类细胞色素P450 83B1Tai6.184212.38类苯丙氨酸解氨酶Novel061723.36类细胞色素P450 84A1Tai6.49002-2.07类花青素3-O-葡萄糖基转移酶7Novel064363.63类细胞色素P450 84A1Novel09917-3.15过氧化物酶3-likeTai6.158012.17类细胞色素P450 85A1Novel06434Inf过氧化物酶72Tai6.192532.47类细胞色素P450 85A1Novel098735.34过氧化物酶72Tai6.28062.56类细胞色素P450 85A1Tai6.421852.12类赤霉素20氧化酶1
2.6 紫甘薯叶片响应UV-B辐射增强下的转录因子分析
转录因子(Transcription factor,TF)为转录调控中的核心功能蛋白,在植物响应逆境胁迫中充当重要角色[18]。UV-B辐射胁迫下,紫甘薯叶片共有43个转录因子基因差异表达,约占差异表达总基因数的9.01%,其分别隶属于16个转录因子家族。这些转录因子家族中转录因子较多的家族分别为C2H2、NAM、bHLH和RR-A-type,其中NAM和RR-A-type基因全为上调表达,C2H2基因以上调表达为主,bHLH基因上、下调数目相等(图3)。
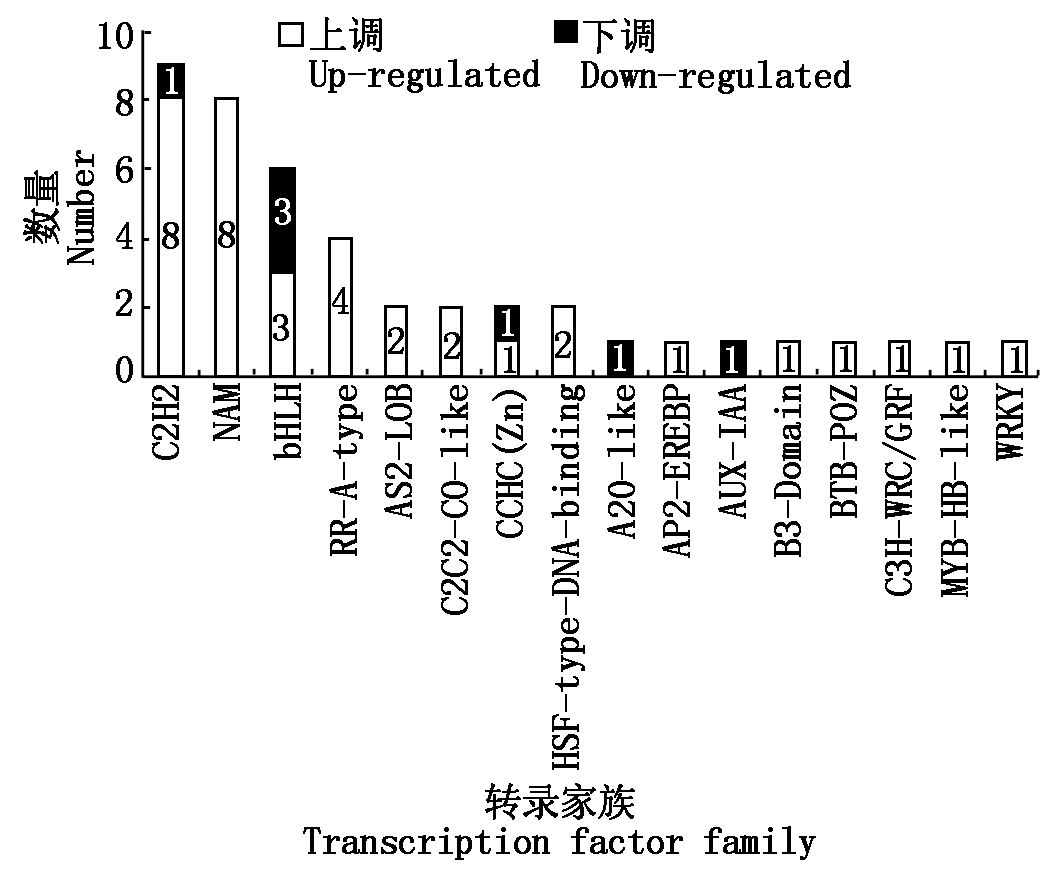
图3 紫甘薯叶片响应UV-B辐射增强下的转录因子
Fig.3 Transcription factors of purple sweet potato
leaf in responsed to enhanced the UV-B
2.7 DEGs的qRT-PCR验证
随机选取7个DEGs以验证测序差异表达基因数据的可靠性,其中上调表达基因2个(PAL(Tai6.18421)和CYP450(Novel07412)),下调表达基因5个(CHS(Tai6.1732)、4CL(Tai6.23346)、DFR(Tai6.3019)、LDOX(Tai6.40394)和peroxidase gene(Novel09917))。以18S DNA为内参,进行qRT-PCR验证。结果显示,选定基因在UV-B辐射增强胁迫下的表达趋势与RNA-seq的检测结果一致(图4),表明转录组测序获得的数据较为可靠[19-20]。
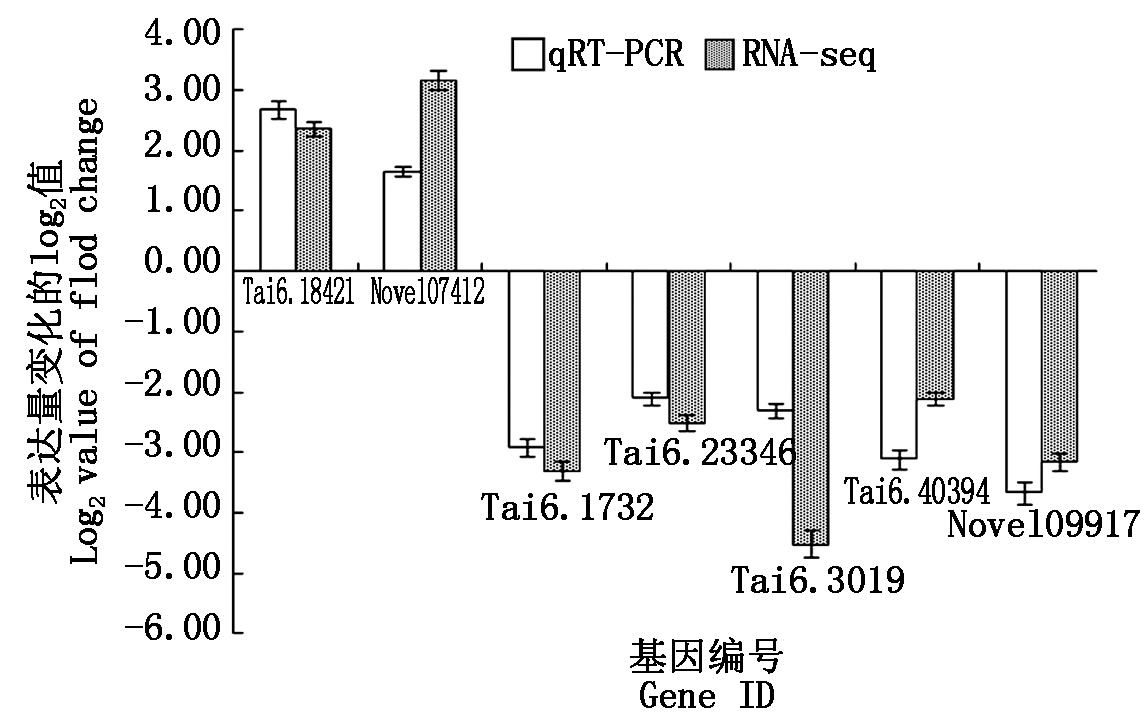
图4 紫甘薯叶片响应UV-B辐射增强的
DEGs的qRT-PCR验证
Fig.4 qRT-PCR validation of DEGs of purple sweet
potato leaf in response to enhanced the UV-B
3 讨论与结论
本研究采用RNA-seq技术研究紫甘薯叶片对UV-B辐射增强的响应,共获得显著响应UV-B辐射增强的差异表达基因477个,其中382个上调表达,95个下调表达,表明这些DEGs对UV-B辐射胁迫的响应主要以上调表达为主。GO功能注释显示,DEGs主要分布在生物过程中的氧化还原过程和分子功能中的氧化还原酶活性中;KEGG Pathway分析表明,DEGs主要富集在次生代谢产物生物合成通路中,且主要分布在类黄酮生物合成过程和细胞色素P450中。
类黄酮作为主要的UV-B吸收化合物,在植物生理生化层面起有效抵御UV-B辐射伤害的作用[21]。高等植物黄酮类化合物经“一般苯丙烷类途径(General phenylpropanoid pathway)”合成,肉桂酸和香豆酰辅酶A是合成途径的中间产物,其合成受苯丙氨酸解氨酶(PAL)、肉桂酸4-羟化酶(C4H)和4香豆酸辅酶A连接酶(4CL)的调控[22-23],随后查尔酮合酶(CHS)将4-香豆酰-辅酶A转化为查尔酮,这一过程是类黄酮生物合成的第一个限速步骤[24]。此后,通过对分子骨架的修饰产生不同的类黄酮亚群,该过程是由CHS、黄烷酮-3-羟化酶(F3H)、类黄酮3′-羟化酶(F3′H)、类黄酮3′,5′羟化酶(F3′,5′H)、类黄酮3-甲基转移酶(F3MT)、类黄酮3′,5′甲基转移酶(F3′,5′MT)、隐色花色素还原酶(LAR)、二氢黄酮醇4-还原酶(DFR)、花色素合成酶(ANS)、3-氧-甲基转移酶(COMT)、CCoAMT和HCT控制的。王霞等[25]以1个紫色甘薯突变体和诱变亲本为试验材料,通过转录组测序分析证实PAL、C4H、CHS、CHI、F3H和DFR可能与紫色甘薯中花青素的合成有关。本研究发现,类PAL(Tai6.36393、Tai6.18421)上调表达了2倍多,4-香豆酸-CoA连接酶6基因(Tai6.23346)下调表达了2倍多,CHS(Tai6.1732、Tai6.53503、Tai6.45381)下调了2.49~3.30倍,DFR(ai6.3019)下调了4.25倍,咖啡酰-CoA O-甲基转移酶At4g26220亚型X1基因(Tai6.25039)、ANS(Tai6.42091、Tai6.40394)及类花青素3-O-葡萄糖基转移酶7基因(Tai6.49002)下调了2.00~2.89倍,表明UV-B辐射增强下类黄酮合成通路中的各关键酶基因主要以下调表达为主,且IbCHS和IbDFR下调幅度最大,暗示其分别为类黄酮和花色甘合成的关键限速酶,致使UV-B辐射增强下紫甘薯叶片中的类黄酮与花色苷含量均下降[26]。
细胞色素P450是高等植物中最大的酶蛋白家族,其在合成植物次生代谢产物(木质素中间物、苯丙烷类的生物合成、芥子油、IAA、萜类、黄酮类、异黄酮等)中具有重要的作用[27]。前人研究发现,CYP83B1为肟的代谢酶,并参与吲哚族芥子油苷核心结构的形成,其能够高效催化来源于色氨酸的吲哚-3-乙醛肟(IAOx),植物体内的CYP83B1缺失后,IAOx就不能生成吲哚族芥子油苷,而是流向IAA合成通路,在植物中积累大量的IAA加重顶端优势,反之则可诱导植物顶端优势减弱[28]。CYP85A1在油菜素内酯(BR)生物合成途径中具有重要作用[29],油菜素内酯可以通过改善相关抗氧化指标来响应非生物胁迫,进而提高植株抗逆性能[30-31]。本研究共发现分属于5个CYP450家族的10个基因,其中IbCYP83B1(Tai6.44115、Tai6.18064、Tai6.41206)和IbCYP85A1(Tai6.2806、Tai6.19253、Tai6.15801)数量最多,且全部基因均上调表达,表明IbCYP83B1和IbCYP85A1在UV-B辐射增强下起关键的正向调控作用,并可促进紫甘薯叶片中芥子油苷和油菜素内酯含量的增加[32-35]。
转录因子在响应由环境因素引起的基因表达调控方面发挥着重要作用[36]。前人研究表明,C2H2为锌指蛋白的一个类型,在调控植物非生物胁迫如干旱、盐胁迫、低温等方面起重要作用[37]。bHLH(碱性螺旋-环-螺旋)类转录因子是植物中第二大转录因子家族,其不仅具有调节植物生长发育、参与花青素合成的功能,还在植物低温胁迫、干旱胁迫、盐害胁迫等非生物胁迫方面起重要作用[38]。NAC是植物特有的转录因子中最大的家族之一,其主要涉及对植物的生长发育调控、植物的逆境胁迫应答、调控植物的抗病性、参与植物次生生长调控以及激素信号转导等过程[39]。RR-A-type(反应调节因子和假定的新型转录因子),其为ARR-B(反应调节因子)的一个亚族,在植物细胞分裂素信号转导中起正调控作用[40]。本研究中C2H2、NAM、bHLH、RR-A-type家族的DEGs数量最多,表明其在紫甘薯响应UV-B辐射中起关键作用,其中NAM和RR-A-type的全部及C2H2的多数基因均为上调表达,暗示其在辐射增强下主要起正向调控作用,bHLH上、下调数目相等,暗示其在辐射增强下既起正向调控作用也具有负向调控作用,这与前人在小麦(Triticum aestivum L.)中的研究结果相似[41]。上述结果表明,UV-B辐射增强下,紫甘薯叶片可能主要通过C2H2、NAM、RR-A-type转录因子编码基因的正向调节来应对辐射增强,同时通过bHLH调控花青素含量,NAC调节ABA、GA等植物激素含量,RR-A-type调控细胞分裂素信号转导等途径来提高紫甘薯叶片对辐射增强的适应性。
综上所述,紫甘薯叶片响应UV-B辐射增强的关键基因可能为类黄酮代谢过程中的IbCHS和IbDFR,以及CYP450家族的IbCYP83B1和IbCYP85A1,起主要调控作用的转录因子可能为C2H2、NAM、bHLH和RR-A-type。本研究不仅可为紫甘薯耐UV-B靶标基因的筛选提供一些候选基因,还可以为揭示紫甘薯响应UV-B胁迫转录调控机制的研究奠定理论基础。
[1] Zhang W L,Jiang W B. UV treatment improved the quality of postharvest fruits and vegetables by inducing resistance[J].Trends in Food Science & Technology,2019,92(3):71-80.doi:10.1016/j.tifs.2019.08.012.
[2] McKenzie R L,Aucamp P J,Bais A F,Björn L O,Ilyas M,Madronich S. Ozone depletion and climate change:Impacts on UV radiation[J].Photochemical & Photobiological Sciences,2011,10(2):182-198. doi:10.1039/c0pp90034f.
[3] Taalas P,Kaurola J,Kylling A,Shindell D,Sausen R,Dameris M,Grewe V,Herman J,Damski J,Steil B. The impact of greenhouse gases and halogenated species on future solar UV radiation doses[J].Geophysical Research Letters,2000,27(8):1127-1130.doi:10.1029/1999GL010886.
[4] Qian G,Ran X,Zhou C X,Deng D Q,Zhang P L,Guo Y,Luo J H,Zhou X H,Xie H,Cai M. Systemic lupus erythematosus patients in the low-latitude plateau of China:altitudinal influences[J].Lupus,2014,23(14):1537-1545. doi:10.1177/0961203314544186.
[5] Caldwell M M,Robberecht R,Billings W D. A steep latitudinal gradient of solar ultraviolet-B radiation in the arctic-alpine life zone[J].Ecology,1980,61(3):600-611. doi:10.2307/1937426.
[6] 陆建珍,徐雪高,汪翔,秦建军,易中懿.中国紫甘薯产业发展分析[J].农业展望,2020,16(8):81-89.doi:10.3969/j.issn.1673-3908.2020.08.012.
Lu J Z,Xu X G,Wang X,Qin J J,Yi Z Y. Analysis of purple sweet potato industry in China [J].Agricultural Outlook,2020,16(8):81-89.
[7] 侯丽娟,李雪英,刘刚,王同勇,朱海波,田孝威. 威海紫甘薯种植加工产业现状和发展对策[J].农产品加工(学刊),2014(2):49-51. doi:10.3969/j.issn.1671-9646(X).2014.02.016.
Hou L J,Li X Y,Liu G,Wang T Y,Zhu H B,Tian X W. Weihai purple sweet potato cultivation and processing industrialization status and development countermeasures [J].Academic Periodical of Farm Products Processing,2014(3):49-51.
[8] 郑丽瑶. 紫薯花青素及其提取工艺的研究进展[J].东南园艺,2019,7(3):56-60.
Zheng L Y. Advances in research on purple sweet potato anthocyanins and their extraction process [J].Southeast Horticulture,2019,7(3):56-60.
[9] 张婷,姚刚,范玉和,程勇杰,陈小伟,张沙沙,崔斌,毛旸晨,蔡海莺,胡东,毛建卫. 紫薯功能性研究进展[C]//中国食品科学技术学会第十四届年会暨第九届中美食品业高层论坛论文摘要集. 无锡:中国食品科学技术学会,美国食品科技学会,2017.
Zhang T,Yao G,Fan Y H,Cheng Y J,Chen X W,Zhang S S,Cui B,Mao Y C,Cai H Y,Hu D,Mao J W. The functional research progress of purple sweet potatoes [C]//Abstracts of Food Summit in China & 14th Annual Meeting of CIFST. Wuxi:Chinese Institute of Food Science and Technology,American Institute of Food Science and Technology,2017.
[10] 孟凡来,白磊,郭华春,赵大伟,王应梅,李玉祥,滕娟. 云南低纬高原不同类型紫甘薯对UV-B辐射增强的响应[J].热带农业科学,2021,41(2):24-32. doi:10.12008/j.issn.1009-2196.2021.02.004.
Meng F L,Bai L,Guo H C,Zhao D W,Wang Y M,Li Y X,Teng J. Response of two types of purple sweet potatoes to UV-B radiation enhancement in Yunnan low latitude plateau[J].Chinese Journal of Tropical Agriculture,2021,41(2):24-32.
[11] 孟凡来,郭华春. 不同甘薯品种抗UV-B辐射增强的效应分析[J].中国农业气象,2019,40(5):293-300.doi:10.3969/j.issn.1000-6362.2019.05.003.
Meng F L,Guo H C. Effect analysis of anti-UV-B enhancement of two sweet potato cultivars[J].Chinese Journal of Agrometeorology,2019,40(5):293-300.
[12] 孟凡来,郭华春. UV-B辐射增强对甘薯光合特性和紫外吸收物质的影响[J].作物杂志,2019(5):114-119. doi:10.16035/j.issn.1001-7283.2019.05.019.
Meng F L,Guo H C. Effects of enhanced UV-B on photosynthetic characteristics and UV-absorbing compounds of sweet potato[J].Crops,2019(5):114-119.
[13] 何永美,湛方栋,祖艳群,徐渭渭,李元. 大田增强UV-B辐射对元阳梯田地方水稻茎秆性状和倒伏指数的影响[J].应用生态学报,2015,26(1):39-45. doi:10.13287/j.1001-9332.20141208.002.
He Y M,Zhan F D,Zu Y Q,Xu W W,Li Y. Effects of enhanced UV-B radiation on culm charateristics and lodging index of two local rice varieties in Yuanyang terraces under field condition[J].Chinese Journal of Applied Ecology,2015,26(1):39-45.
[14] Ji C Y,Bian X F,Lee C J,Kim H S,Kim S E,Park S C,Xie Y Z,Guo X D,Kwak S S. De novo transcriptome sequencing and gene expression profiling of sweet potato leaves during low temperature stress and recovery[J].Gene,2019,700:23-30. doi:10.1016/j.gene.2019.02.097.
[15] Trapnell C,Williams B A,Pertea G,Mortazavi A,Kwan G,Van Baren M J,Salzberg S L,Wold B J,Pachter L. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation[J].Nature Biotechnology,2010,28(5):511-515. doi:10.1038/nbt.1621.
[16] Livak K J,Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J].Methods,2001,25(4):402-408.doi:10.1006/meth.2001.1262.
[17] 马进,郑钢.南方型紫花苜蓿叶片盐胁迫应答基因鉴定与分析[J].农业生物技术学报,2015,23(12):1531-1541.doi:10.3969/j.issn.1674-7968.2015.12.001.
Ma J,Zheng G. Identification and preliminary analysis of salt stress-responsive genes in leaves of Southern Type Alfalfa(Medicago sativa ′millennium′)[J].Journal of Agricultural Biotechnology,2015,23(12):1531-1541.
[18] 张婧蕾,李佳赟,王依纯,裴翠明,马进.南方型紫花苜蓿耐盐突变体叶片盐胁迫应答差异基因鉴定与分析[J].农业生物技术学报,2017,25(10):1588-1599.doi:10.3969/j.issn.1674-7968.2017.10.004.
Zhang J L,Li J Y,Wang Y C,Pei C M,Ma J. Identification and analysis of differentially-expressed genes under salt stress in leaves of southern type Alfalfa(Medicago sativa ′Millennium′)salt tolerant mutant[J].Journal of Agricultural Biotechnology,2017,25(10):1588-1599.
[19] 张鹏钰,王国瑞,曹丽茹,袁珍,库丽霞,王同朝,卫丽.干旱胁迫和复水处理下玉米差异表达转录因子基因分析[J].农业生物技术学报,2020,28(2):211-222.doi:10.3969/j.issn.1674-7968.2020.02.003.
Zhang P Y,Wang G R,Cao L R,Yuan Z,Ku L X,Wang T C,Wei L. Analysis of differentially expressed transcription factor genes in maize(Zea mays)under drought stress and re-watering[J]. Journal of Agricultural Biotechnology,2020,28(2):211-222.
[20] 魏薇,贾彦丽,吴硕,智福军.水分胁迫下枣裂果的全长转录组分析[J].华北农学报,2020,35(S1):63-71.doi:10.7668/hbnxb.20191835.
Wei W,Jia Y L,Wu S,Zhi F J. Full-length transcriptome analysis of fruit cracking of Chinese jujube under water stress[J].Acta Agriculturae Boreali-Sinica,2020,35(S1):63-71.
[21] Frohnmeyer H,Staiger D. Ultraviolet-B radiation-mediated responses in plants.Balancing damage and protection[J].Plant Physiology,2003,133(4):1420-1428. doi:10.1104/pp.103.030049.
[22] Deshmukh A B,Datir S S,Bhonde Y,Kelkar N,Samdani P,Tamhane V A. De novo root transcriptome of a medicinally important rare tree Oroxylum indicum for characterization of the flavonoid biosynthesis pathway[J].Phytochemistry,2018,156:201-213. doi:10.1016/j.phytochem.2018.09.013.
[23] Tao X,Fang Y,Huang M J,Xiao Y,Liu Y,Ma X R,Zhao H. High flavonoid accompanied with high starch accumulation triggered by nutrient starvation in bioenergy crop duckweed(Landoltia punctata)[J].BMC Genomics,2017,18(1):166. doi:10.1186/s12864-017-3559-z.
[24] Tohge T,Yonekura-Sakakibara K,Niida R,Watanabe-Takahashi A,Saito K. Phytochemical genomics in Arabidopsis thaliana:A case study for functional identification of flavonoid biosynthesis genes[J].Pure and Applied Chemistry,2007,79(4):811-823.doi:10.1351/pac200779040811.
[25] 王霞,李恩广,玄曼霖,燕宝会,王晶珊,隋炯明.基于转录组测序的紫色甘薯突变体中花青素合成相关基因的分析[J].青岛农业大学学报(自然科学版),2018,35(1):27-31. doi:10.3969/J.ISSN.1674-148X.2018.01.005.
Wang X,Li E G,Xuan M L,Yan B H,Wang J S,Sui J M. Gene analysis of anthocyanin biosynthesis in purple sweet potato mutant based on transcriptome sequencing [J].Journal of Qingdao Agricultural University (Natural Science),2018,35(1):27-31.
[26] 石晓芳. 甘薯花青素合成途径基因表达差异的研究[D].重庆:西南大学,2013.
Shi X F. Study on the expression differences of anthocyanin biosynthetic pathway genes in sweet potato[D].Chongqing:Southwest University,2013.
[27] 温国琴. 苦荞细胞色素P450基因的克隆、原核表达及逆境条件对其在芽期苦荞中表达的影响[D].成都:四川农业大学,2013.
Wen G Q. Cloning and prokaryotic expression of cytochrome P450 gene from Fagopyrum tataticum,and its expression level in adversity at sprout stage[D].Chengdu:Sichuan Agricultural University,2013.
[28] Bak S,Tax F E,Feldmann K A,Galbraith D W,Feyereisen R. CYP83B1,a cytochrome P450 at the metabolic branch point in auxin and indole glucosinolate biosynthesis in Arabidopsis[J].The Plant Cell,2001,13(1):101-111. doi:10.1105/tpc.13.1.101.
[29] Wang H,Li W Q,Qin Y G,Pan Y P,Wang X F,Weng Y Q,Chen P,Li Y H. The cytochrome P450 gene CsCYP85A1 is a putative candidate for Super Compact-1(Scp-1)plant architecture mutation in Cucumber(Cucumis sativus L.)[J]. Frontiers in Plant Science,2017,8:266. doi:10.3389/fpls.2017.00266.
[30] Soares C,de Sousa A,Pinto A,Azenha M,Teixeira J,Azevedo R A,Fidalgo F. Effect of 24-epibrassinolide on ROS content,antioxidant system,lipid peroxidation and Ni uptake in Solanum nigrum L. under Ni stress[J].Environmental and Experimental Botany, 2016,122:115-125. doi:10.1016/j.envexpbot.2015.09.010.
[31] Kaur H,Sirhindi G,Bhardwaj R,Alyemeni M N,Siddique K H M,Ahmad P. 28-homobrassinolide regulates antioxidant enzyme activities and gene expression in response to salt-and temperature-induced oxidative stress in Brassica juncea[J].Scientific Reports,2018,8(1):8735. doi:10.1038/s41598-018-27032-w.
[32] Sun B,Jiang M,Yuan Q,Zhang F,Tang H R. Bioinformatics analysis of the gene CYP83B1 in cabbage(Brassica oleracea var. capitata)[C]//Proceedings of the 2018 3rd International Workshop on Materials Engineering and Computer Sciences(IWMECS 2018). Ji′nan:Computer Science and Electronic Technology International Society,2018.
[33] 赵桂红,石宏,张妮妮,陆苗,王晶,李焘. 菘蓝CYP83B1基因的克隆与表达分析[J].植物科学学报,2017,35(1):64-72. doi:10.11913/PSJ.2095-0837.2017.10064.
Zhao G H,Shi H,Zhang N N,Lu M,Wang J,Li T. Cloning and expression analysis of CYP83B1 from Isatis indigotica Fort[J].Plant Science Journal,2017,35(1):64-72.
[34] Li X J,Guo X,Zhou Y H,Shi K,Zhou J,Yu J Q,Xia X J. Overexpression of a brassinosteroid biosynthetic gene Dwarf enhances photosynthetic capacity through activation of Calvin cycle enzymes in tomato[J].BMC Plant Biology,2016,16:33. doi:10.1186/s12870-016-0715-6.
[35] 鲁雪莉,段方猛,万芳源,李宝笃,宋雯雯. 过量表达菠菜SoCYP85A1基因增强烟草的耐盐性[J].植物生理学报,2017,53(3):454-460. doi:10.13592/j.cnki.ppj.2016.0551.
Lu X L,Duan F M,Wan F Y,Li B D,Song W W. Overexpression of SoCYP85A1 gene from Spinacia oleracea enhances high-salt tolerance in tobacco[J].Plant Physiology Journal,2017,53(3):454-460.
[36] Chen Y Y,Li M Y,Wu X J,Huang Y,Ma J,Xiong A S. Genome-wide analysis of basic helix-loop-helix family transcription factors and their role in responses to abiotic stress in carrot [J].Molecular Breeding,2015,35(5):125-136. doi:10.1007/s11032-015-0319-0.
[37] 钟婵娟,彭伟业,王冰,刘世名,戴良英,李魏. 植物逆境响应相关的C2H2型锌指蛋白研究进展[J].植物生理学报,2020,56(11):2356-2366. doi:10.13592/j.cnki.ppj.2020.0171.
Zhong C J,Peng W Y,Wang B,Liu S M,Dai L Y,Li W. Advances of plant C2H2 zinc finger proteins in response to stresses[J].Plant Physiology Journal,2020,56(11):2356-2366.
[38] 王力伟,房永雨,刘红葵,郭保民,王蕴华,孙瑞芬,吉鹏华,何江峰.bHLH转录因子的研究进展[J].畜牧与饲料科学,2020,41(1):23-27.doi:10.12160/j.issn.1672-5190.2020.01.005.
Wang L W, Fang Y Y, Liu H K, Guo B M, Wang Y H, Sun R F, Ji P H, He J F. Research progress of bHLH transcription factors[J].Animal Husbandry and Feed Science,2020,41(1):23-27.
[39] 王春雨,张茜. 植物NAC转录因子功能研究进展[J].生物技术通报,2018,34(11):8-14. doi:10.13560/j.cnki.biotech.bull.1985.2018-0443.
Wang C Y,Zhang Q. Research progress on plant NAC transcription factors[J].Biotechnology Bulletin,2018,34(11):8-14.
[40] To J P C,Deruère J,Maxwell B B,Morris V F,Hutchison C E,Ferreira F J,Schaller G E,Kieber J J. Cytokinin regulates type-A Arabidopsis response regulator activity and protein stability via two-component phosphorelay [J].The Plant Cell,2007,19(12):3901-3914. doi:10.1105/tpc.107.052662.
[41] Wang F,Xu Z B,Fan X L,Zhou Q,Cao J,Ji G S,Jing S Z,Feng B,Wang T. Transcriptome analysis reveals complex molecular mechanisms underlying UV tolerance of wheat(Triticum aestivum,L.)[J].Journal of Agricultural and Food Chemistry,2019,67(2):563-577. doi:10.1021/acs.jafc.8b05104.