叶绿体是绿色植物最重要的细胞器之一,涉及包括光合作用在内的多条代谢途径[1-3]。在成熟叶绿体的约3 000种蛋白质中,仅100种左右由叶绿体自身基因编码,其余均由核基因编码,在细胞质合成后,通过叶绿体外膜和内膜上的转运蛋白转运到叶绿体内[4-5]。随着叶绿体的分化发育和外界环境变化,叶绿体蛋白质组也处于动态的变化中[6-7],植物需根据内外环境的变化对叶绿体蛋白质的输入进行调控。
Ling等[8-12]的研究结果显示,SP1(Suppressor of ppi1 locus1)蛋白是拟南芥(Arabidopsis thaliana)叶绿体蛋白质输入调控的关键因子,该蛋白由sp1基因编码,定位于叶绿体外膜,是一种泛肽E3连接酶[12],可催化叶绿体外膜上的TOC33、TOC75、TOC159(Translocon of the outer membrane of chloroplasts)等多种转运蛋白发生泛肽化,再在SP2(Suppressor of ppi1 locus2)蛋白、CDC48(Cell division cycle 48)蛋白以及胞质中的泛肽-蛋白酶体系统(Ubiquitin-proteasome system,UPS)的参与下,将泛肽化蛋白降解清除[11]。通过控制叶绿体的蛋白质输入,sp1基因的表达可影响叶绿体内的活性氧(Reactive oxygen species,ROS)产生水平,从而影响植物的逆境响应能力[9,11]。
尽管sp1基因对植物的叶绿体蛋白输入和抗逆性具有重要影响[8-9],迄今为止,对该基因的研究主要是以拟南芥为材料展开的[8-12]。叶绿体的功能与植物的光合效率直接相关,对于农作物,叶绿体蛋白输入调控直接影响到作物的产量和抗逆性。水稻是全球范围内最重要的粮食作物之一,也是重要的模式植物,研究水稻的sp1同源基因对水稻生长发育和胁迫抗性的影响,对阐明该基因的作用机制具有重要意义,也可以为农作物品种的研发和选育提供参考依据[13]。
笔者在前期工作中[14],采用生物信息学手段和分子生物学技术,确定并克隆了sp1基因在水稻中的同源序列OsSP1(Os07g0647800),该基因的编码产物含343个氨基酸残基,与拟南芥SP1蛋白氨基酸残基数相同,二者序列相似度为71.18%,且信号肽的位置和序列也与拟南芥SP1蛋白相同,亚细胞定位分析显示该蛋白确实定位于水稻叶绿体。转录水平分析显示OsSP1在叶片中表达水平高于叶鞘和根,并且具有明显的干旱响应特性。这些结果显示了OsSP1基因在结构和定位上与拟南芥sp1基因的一致性,但OsSP1基因对水稻生长发育和胁迫耐受性的影响有待进一步验证。
CRISPR/Cas9基因编辑技术作为一种简便、高效的基因编辑工具,自开始报道以来[15-16],在几年的时间里得到了快速发展,技术也日臻成熟完善[17-18],在水稻等多种生物的基因功能研究中得到了广泛应用[19-21]。本研究利用植物基因过表达技术和CRISPR/Cas9基因编辑技术,分别对水稻OsSP1基因进行过量表达和基因编辑,通过对获得的过表达株系和基因编辑株系进行表型对比、叶绿素含量测定和ROS水平测定,确定OsSP1基因对水稻生长发育的影响,通过对纯合过表达株系进行干旱胁迫处理,确定OsSP1基因对水稻胁迫耐受性的影响。
1 材料和方法
1.1 植物材料及栽培条件
本研究以水稻品种中花11为遗传转化受体,转基因材料种植于苗圃温网室,常规种植和管理。
1.2 CRISPR/Cas9基因编辑靶位点的选择和载体构建
在Rice Genome Annotation Project网站(http://rice.plantbiology.msu.edu/)进行OsSP1基因的编码序列分析,根据外显子的位置选择2个20 bp的靶点序列,分别称为靶序列1(target 1)和靶序列2(target 2)。通过NCBI的Blast(https://www.ncbi.nlm.nih.gov/tools/primer-blast/)分析,确保2个靶位点的特异性。
基因编辑载体的构建方法参照Ma等[17]的报道,以pYL-OsU-gRNA质粒为模板,在20 μL的KOD neo反应体系中加入Ufp1-F和gRNA-R引物各1 μL(10 μmol/L),target1-F、target1-R或target2-F、target2-R引物各0.5 μL(10 μmol/L),扩增出pOsU-target片段和target-gRNA片段。取扩增产物0.1 μL作为模板,加入每个对应靶点的U6a-F、g1-R或U6b-F、g2-R引物进行第2轮overlapping PCR,分别扩增出pOsU-target1-gRNA片段和pOsU-target2-gRNA片段。PCR反应条件:94 ℃,15 s,58 ℃,15 s,68 ℃,20 s,28个循环。扩增产物用DNA回收试剂盒回收。取每个靶点扩增产物约15 ng,与酶切后的CRISPR/Cas9载体混合,将sgRNA表达框连入Cas9载体,连接体系总体积为20 μL(Buffer 2.0 μL,Bsa Ⅰ酶切过的CRISPR/Cas9质粒60 ng,T4 DNA连接酶1 μL,2个靶点的sgRNA扩增产物共30 ng,ddH2O补至20 μL),连接条件为:37 ℃,5 min;10 ℃,5 min,20 ℃,5 min,10个循环;37 ℃,5 min。PCR引物见表1。连接产物转化大肠杆菌TOP10菌株,经菌液PCR和核酸序列测定获得阳性克隆,提取质粒转化农杆菌EHA105。
1.3 OsSP1过表达载体的构建
以笔者在前期工作中构建的带有OsSP1编码序列的质粒[14]作为模板,用带有酶切位点的引物sp1-SpeⅠ-F和sp1-HindⅢ-R(表1),扩增OsSP1基因从起始密码子ATG至终止密码子TGA的全长序列,产物经凝胶电泳分离和天根DNA回收试剂盒回收,送上海生工测序确定为正确序列后,采用酶切、连接方法,将该序列连入到过表达载体质粒的35S启动子和nos终止子之间,构建完整的p35S-OsSP1-nos表达框。连接产物转化大肠杆菌TOP10菌株,经菌液PCR和核酸序列测定获得阳性克隆,提取质粒转化农杆菌EHA105。
表1 PCR引物序列
Tab.1 PCR primers

引物名称Primer name引物序列(5′-3′)Primer sequence(5′-3′)Ufp1-FCTCCGTTTTACCTGTGGAATCGgRNA-RCGGAGGAAAATTCCATCCACtarget1-FGAGCACAACGATGCTGGATCAGTTTTAGAGCTAGAAATtarget1-RTGATCCAGCATCGTTGTGCTCTCGGCAGCCAAGCCAGCAtarget2-FGCGAATTCAGCGACCACACAAGTTTTAGAGCTAGAAATtarget2-RTTGTGTGGTCGCTGAATTCGCCAACACAAGCGGCAGCU6a-FACCGGTAAGGCGCGCCGTAGTGCTCGACTAGTATGGAATCGGCAGCAAAGGg1-RCAGGGAGCGGATAACAATTTCACACAGGCACATCCACTCCAAGCTCTTGU6b-FGTGCCTGTGTGAAATTGTTATCCGCTCCCTGGAATCGGCAGCAAAGGg2-RTAGCTCGAGAGGCGCGCCAATGATACCGACGCGTATCCATCCACTCCAAGCTCTTGsp1-SpeⅠ-FTCAGACTAGTATGTTGATCCCATGGGGCsp1-HindⅢ-RTGACAAGCTTTCAATGGCGGAAAGTTCTCAsp1t1-FTCTGCCAACAAAGTGGCATGAsp1t1-RGATTGGACACATAATGGCACTGsp1t2-FGCATATTAGGTTCTTAGATGCTsp1t2-RGAAGCAAGCTGGTATAACCThpt-FCTGAACTCACCGCGACGTCTGTChpt-RTAGCGCGTCTGCTGCTCCATACAsp1-1210-FACTTTCCGCCATTGACAsp1-1379-RTGCTTCGGCGCAATACOsActin1-FCTTCATAGGAATGGAAGCTGCGGGTAOsActin1-RCGACCACCTTGATCTTCATGCTGCTA
1.4 水稻遗传转化
参照Ozawa[22]的方法,通过农杆菌介导法转化水稻愈伤组织,通过潮霉素筛选,分别获得T0 OsSP1基因编辑植株和过表达植株。
1.5 基因编辑植株靶位点序列的检测
T0基因编辑植株采用氢氧化钠法[23]提取叶片DNA作为模板,设计2个目的序列的检测引物sp1t1-F、sp1t1-R和sp1t2-F、sp1t2-R(表1),扩增目的片段后,PCR产物送上海生工测序,根据测序结果分析基因编辑植株中目的基因的突变情况。
1.6 过表达植株纯合后代筛选和Real time PCR检测
T0过表达植株采用氢氧化钠法提取叶片DNA作为模板,通过hpt-F和hpt-R引物进行潮霉素磷酸转移酶基因(hpt)的扩增筛选出阳性株系,种植后获得T1种子,将每个株系分单株收获的T1种子100颗发芽后,用含10 mg/L潮霉素的木村B营养液筛选7 d,统计正常生长与死亡植株的比例,存活与死亡植株比例符合3∶1的即为目的基因单拷贝插入株系,种植后分单株收获T2种子,按T1筛选方法,用潮霉素筛选7 d,所有植株均正常生长的株系即为单拷贝纯合子株系[24]。
野生型和纯合子株系的叶片样品各4片,分别混合、剪碎后经Magen试剂盒提取总RNA,Vazyme反转录试剂盒反转录,cDNA产物用ddH2O稀释10倍后作为模板,以OsActin1(Os03g07181000)作为内参,使用引物sp1-1210-F、sp1-1379-R、OsActin1-F和OsActin1-R(表1)进行Real time PCR扩增,数据处理采用2-ΔΔCt法[25-26],Excel软件和Sigma Plot软件进行分析和作图。Real time PCR反应条件:95 ℃,3 min;95 ℃,15 s,60 ℃,30 s,40个循环。
1.7 叶绿素含量测定
叶绿素含量测定参照李合生 [27]的方法,对分蘖期植株,由上至下取第2片叶片,每个株系平行取样3份,剪去叶尖和叶基部后,剪成细丝,混匀,称取0.01 g,用2 mL叶绿素提取液(V丙酮∶V酒精∶V水=4.5∶4.5∶1.0)萃取出叶绿素,提取液用分光光度计比色,分别测定663(叶绿素a吸收峰),645 nm(叶绿素b吸收峰)波长的OD值,按公式:Chla(叶绿素a)=13.95×OD663-6.88×OD645;Chlb(叶绿素b)=24.96×OD645-7.32×OD663,分别计算叶绿素a和叶绿素b的浓度(mg/L),二者之和为叶绿素总浓度,再根据下式进一步求出叶片中叶绿素的总含量:叶绿素总含量(mg/g鲜质量)=(叶绿素总浓度×提取液体积×稀释倍数)/样品鲜质量(g)。数据处理采用t-检验,Excel软件和Sigma Plot软件进行分析和作图。
1.8 DAB染色
DAB染色参照Thordal-Christensen等[28]的方法,对分蘖期植株,选择晴朗的天气于清晨取样,每株由上至下取第2,3,4片叶片,迅速插入1 mg/L的DAB(二氨基联苯胺,3′3-diaminobenzidine,pH值3.8)溶液中,自然光下反应约3 h,待叶片出现大量暗棕色条纹后,终止反应,用无水乙醇在80 ℃水浴中脱色1 h以上至叶绿素脱尽。
1.9 干旱处理
纯合过表达株系OE3-1-2、OE4-1-6及野生型种子发芽后,点播于水稻种植用土壤中生长至一叶一心期,选择生长一致的幼苗栽种于干旱处理用橡胶桶(桶高:12 cm,桶口直径:25 cm,桶底直径:20 cm,土壤厚度:4 cm),每桶野生型和转基因植株各25株。生长至三叶一心期的植株停止供水,进行干旱处理约7 d,至叶片全部干枯后,恢复供水,继续生长7 d,观察恢复状况。
2 结果与分析
2.1 CRISPR/Cas9基因编辑靶位点的选择和载体构建
通过Rice Genome Annotation Project网站对OsSP1编码区进行结构分析的结果显示,该基因编码区含有11个短序列外显子,分别在第4和第7外显子上选择靶序列1和靶序列2作为基因编辑靶位点(图1-A),经Blast分析,确定2个靶序列均具有高特异性,不会对其他同源基因造成影响。构建成功的CRISPR/Cas9基因编辑载体结构见图1-B。
经目的基因扩增、限制性酶切和T4 DNA连接酶连接,构建成功的过表达载体结构见图1-C。
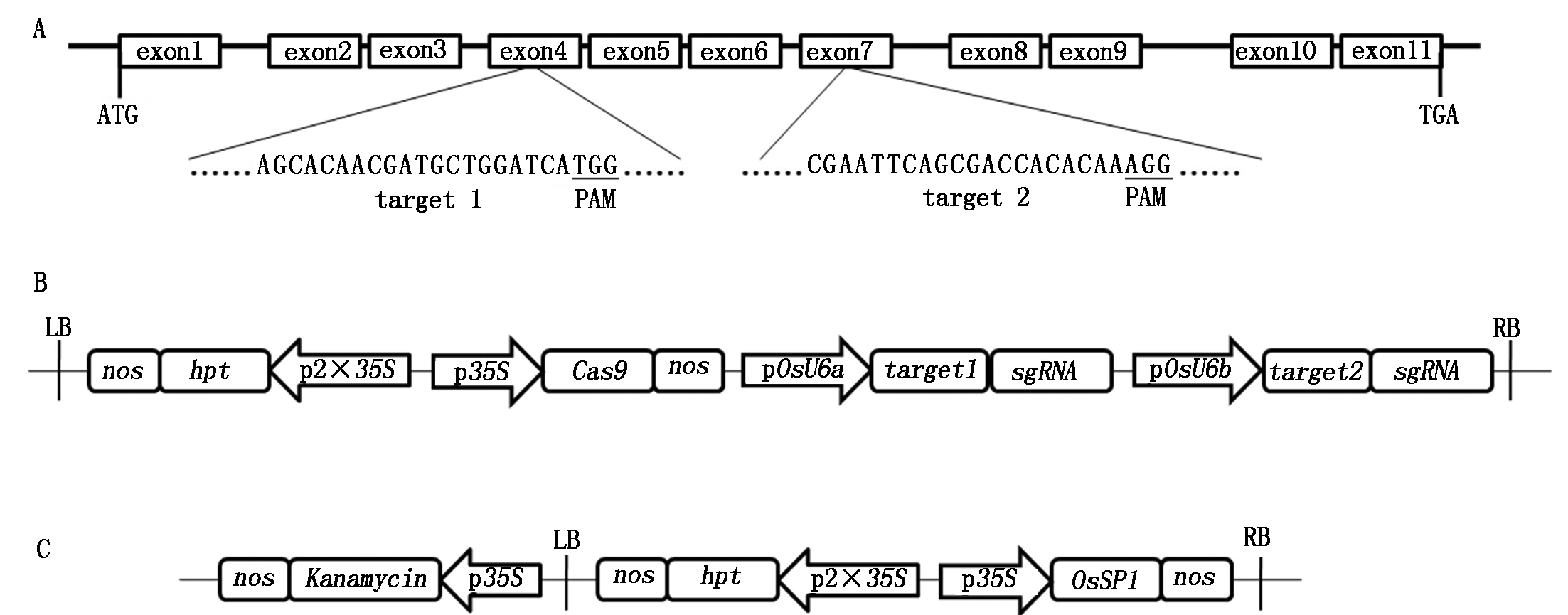
exon1~11.外显子1~11;LB.左边界;nos. nos终止子;hpt.潮霉素B磷酸转移酶基因;p2×35S. 2×35S启动子;p35S.35S启动子;Cas9.Cas9蛋白基因;pOsU6a.OsU6a启动子;target1.靶序列1;sgRNA.gRNA编码序列;pOsU6b.OsU6b启动子;target2.靶序列2;RB.右边界;Kanamycin.卡那霉素抗性基因;OsSP1.OsSP1基因。
exon1-11.exon1-11;LB.Left border;nos.nos terminator;hpt.Hygromycin B phosphotransferase gene;p2×35S.2×35S promoter;p35S.35S promoter;Cas9.Cas9 gene;pOsU6a.OsU6a promoter;target1.target1 sequence;sgRNA.gRNA coding sequence;pOsU6b.OsU6b promoter;target2.target2 sequence;RB.Right border;Kanamycin.Kanamycin resistance gene;OsSP1.OsSP1 gene.
图1 OsSP1基因编辑序列在OsSP1基因中的位置(A)、基因编辑载体结构(B)和过表达载体结构(C)
Fig.1 Position of two targets for gene editing in OsSP1 gene(A),
the structure of gene editing vector(B)and overexpression vector(C)
2.2 T0-基因编辑基因植株的突变类型
通过农杆菌转化水稻愈伤组织,经潮霉素筛选,共获得9个具有潮霉素抗性的阳性T0基因编辑株系。经叶片DNA提取和检测引物扩增目的片段,将扩增产物测序,结果显示(表2),9个株系中,有2个株系突变发生于靶序列1,纯合突变和杂合突变各1个,纯合突变系sp1-1的突变方式为A删除,该位点为基因读码框的第291个核苷酸。有6个株系突变发生于靶序列2,其中双等位突变5个,杂合突变1个,突变方式包括单核苷酸的插入、删除,以及2,3,180 bp的删除。有1个株系sp1-9的1,22个靶序列同时发生了突变,造成1个612 bp的大片段删除和1个5 bp删除,但只有一个等位基因发生了突变,为杂合突变。
表2 T0基因编辑株系中OsSP1基因的突变类型
Tab.2 Mutation type of OsSP1 gene in T0 gene-edited lines

靶序列Target sequence株系名称Line name突变位点Mutated site突变方式Mutation pattern突变类型Mutation type1sp1-1AGCACAACGATGCTGG-TCATGG-A纯合突变1sp1-2AGCACAACGAT-------------------------24 bp杂合突变2sp1-3CGAATTCAGCGAC--CACAAAGG------------------------------ACACAAAGG-CA-180 bp双等位突变2sp1-4CGAATTCAGCGACC--ACAAAGGCGAATTCAGCGACCAACACAAAGG-AC+A双等位突变2sp1-5CGAATTCAGCGACCAC-CAAAGGCGAATTCAGCGACCACAACAAAGG-A+A双等位突变2sp1-6CGAATTCAGCGACC-CACAAAGG-A杂合突变2sp1-7CGAATTCAGC------------GCGAATTCAGCGACC-CACAAAGG-12 bp-A双等位突变2sp1-8CGAATTCAGCGAC--CACAAAGGCGAATTCAGCGA---CACAAAGG-CA-CCA双等位突变1、2sp1-9------AATTCAGCG-----ACAAAGGCG-612 bp,-ACCAC杂合突变
注:横线表示缺失碱基;下划线并加粗字母表示插入碱基。
Note:The horizontal line represents the deleted base;The underlined and bold font represents the inserted base.
2.3 过表达植株中的纯合子筛选和表达分析
通过农杆菌转化水稻愈伤组织获得的T0过表达株系,经叶片DNA提取和hpt序列扩增,共获得12个阳性株系(图2-A),种植后获得的T1和T2种子,发芽后经潮霉素筛选获得单拷贝插入株系。最终获得3个单拷贝纯合子株系命名为OE2-2-1、OE3-1-4和OE4-3-8(分别来自T0的株系2、株系3和株系4)。以水稻OsActin1作为内参,进行Real time PCR分析的结果显示(图2-B),目的基因OsSP1在3个纯合过表达株系中的转录水平均显著高于野生型植株,分别为野生型植株中的3.5,4.6,5.4倍。
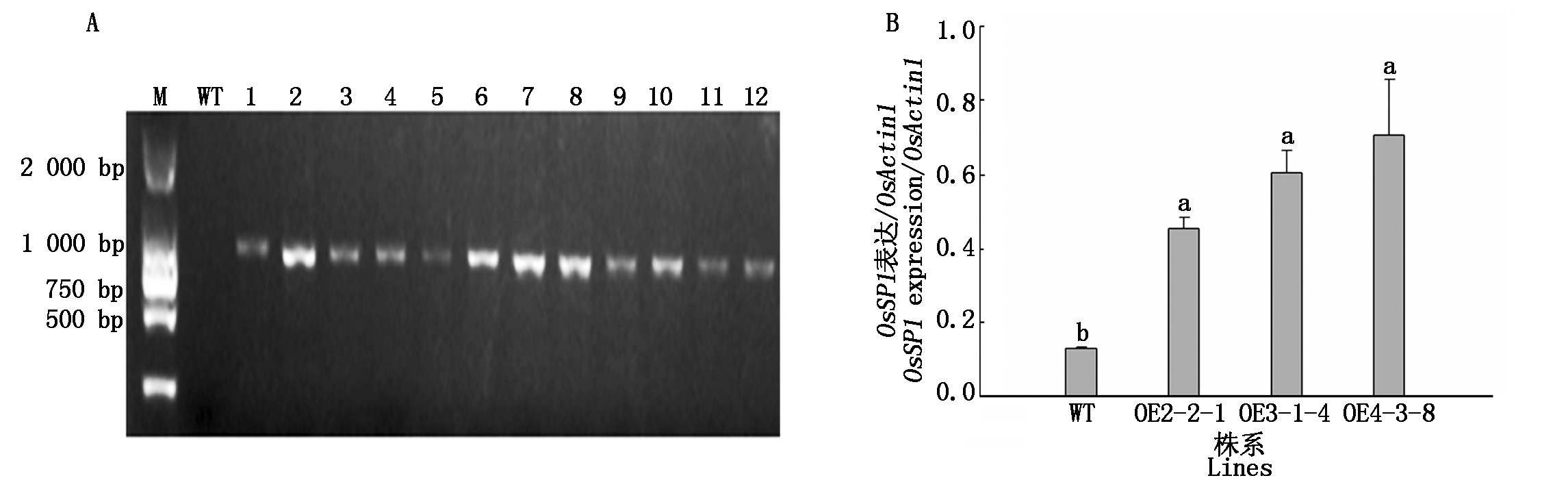
M.Marker;WT.野生型植株;1-12.T0转基因株系。不同的小写字母表示与野生型相比差异显著(P<0.05);
不同的大写字母表示与野生型相比差异极显著(P<0.01)。图4-5同。
M.Marker;WT.Wild type plant;1-12.The T0 transgenic lines.Different lowercase letters indicate significant differences compared with wild type
plants(P<0.05); Different capital letters indicate extremely significant differences compared with wild type plants (P< 0.01).The same as Fig.4-5.
图2 T0过表达植株的hpt基因检测(A)和T2纯合植株的OsSP1转录表达(B)
Fig.2 Evaluation of hpt gene in T0(A)and transcriptional expression of OsSP1 in T2(B)overexpression plants
2.4 基因编辑和过表达植株的表型
纯合突变、双等位突变和杂合突变的T0和T1OsSP1基因编辑植株均表现出明显的生长迟滞表型。植株生长缓慢,矮小,叶片黄化,从幼苗起即出现叶片早衰、早枯现象,不分蘖,老叶较快进入衰老、死亡状态,植株从上至下始终只保留3~4片未干枯叶片(图3)。T0植株在灌浆期后,叶片衰老进一步加快,在种子进入腊熟期前叶片即全部干枯(图3-A),结实率低,每株结实约10颗种子,且种子不饱满(图3-A、B)。T1植株的生长表型与T0相同(图3-C、D),但各种突变类型的T1株系分别在春、夏季种植后均未抽穗结实(图3-E)。由于各类突变的基因编辑植株结实数均较少,未能获得足够种子进行大规模种植和农学表型统计。
纯合过表达植株生长良好,各时期生长状态和结实情况与野生型无明显差异(图3-D、E)。将OE2-2-1、OE3-1-4和OE4-3-8土培种植后,每个株系取20株进行株高、剑叶长、分蘖数、千粒质量表型统计结果显示,纯合过表达植株的农学性状与野生型植株均无显著差异(图4)。

A.T0基因编辑植株乳熟期;B.T1基因编辑植株种子;C.T1基因编辑植株幼苗;D.纯合过表达植株和
T1植株基因编辑植株分蘖期;E.纯合过表达植株和T1植株基因编辑植株乳熟期。
A.T0 gene-edited plant at milk ripe stage;B.Seeds of T1 gene-edited plant;C.Seedlings of T1 gene-edited plants;
D.Overexpression plants and T1 gene-edited plants at tillering stage;E.Overexpression plants and T1 gene-edited plant at milk ripe stage.
图3 过表达和基因编辑植株的表型
Fig.3 Phenotype of overexpression and gene-edited plants
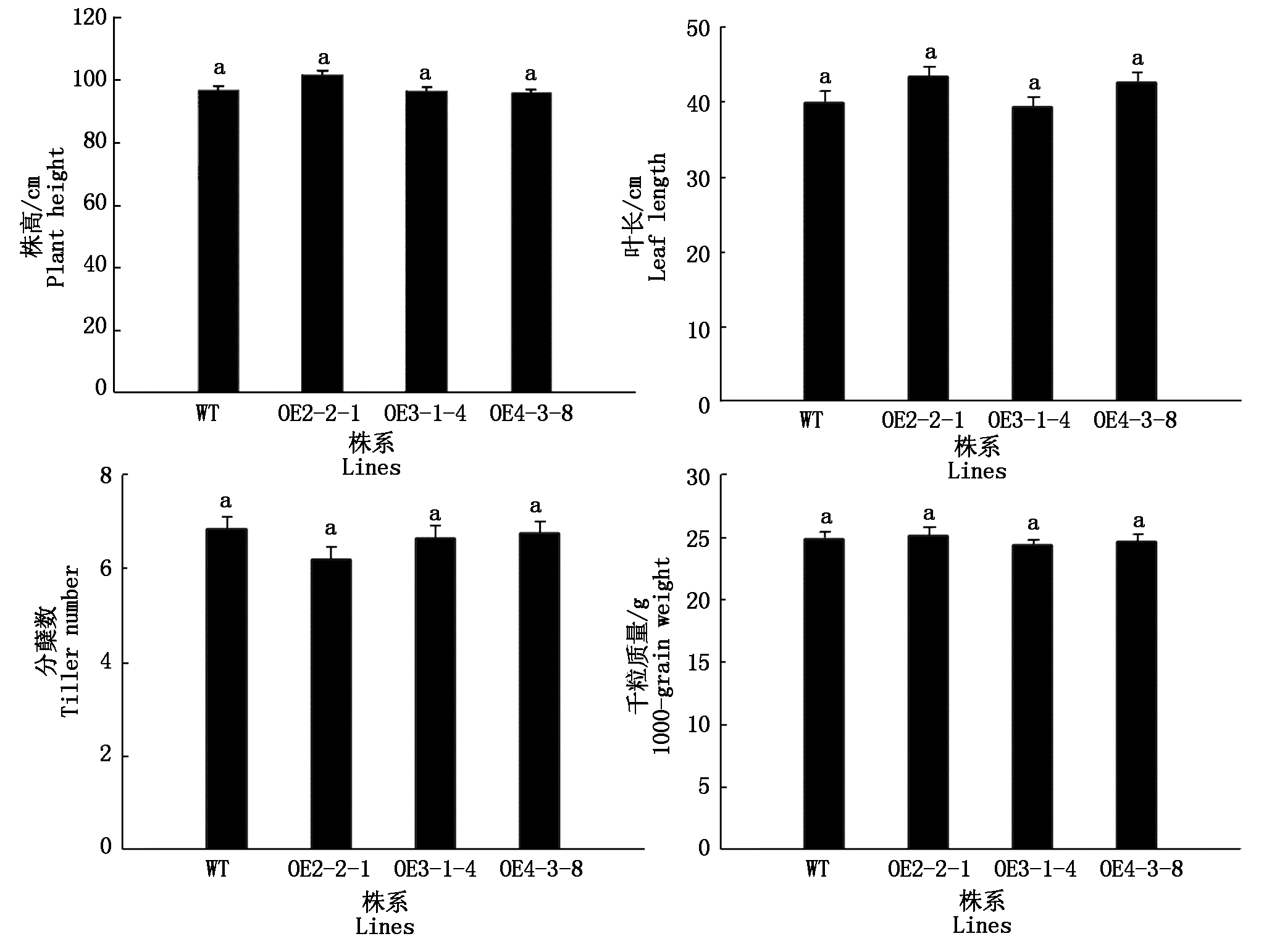
图4 过表达植株的农艺性状
Fig.4 Agronomic traits of overexpression plants
2.5 基因编辑和过表达植株的叶绿素含量和H2O2水平
基因编辑植株具有明显的叶片黄化表型,为确定植株的叶绿素的变化水平,对分蘖期的纯合过表达株系OE3-1-4、OE4-3-8和纯合突变株系sp1-1-2及双等位突变株系sp1-5-1进行了叶绿素含量分析。结果显示,2个过表达株系的叶绿素含量(以鲜质量计)与野生型植株相比无显著变化,而2个基因编辑株系的叶绿素含量均出现显著或极显著下降,与野生型植株比较,分别具有显著差异和极显著差异(图5-A)。
选取分蘖期的过表达植株OE4-3-8和基因编辑植株sp1-1-2,每株从上至下取成熟程度不同的第2,3,4片叶片,采用DAB染色法进行了叶片H2O2检测,结果显示,对比每类植株自身的第2,3,4片叶片,H2O2水平并无明显差别;对比不同类型植株的相同序号叶片,H2O2含量也未见明显差异(图5-B)。
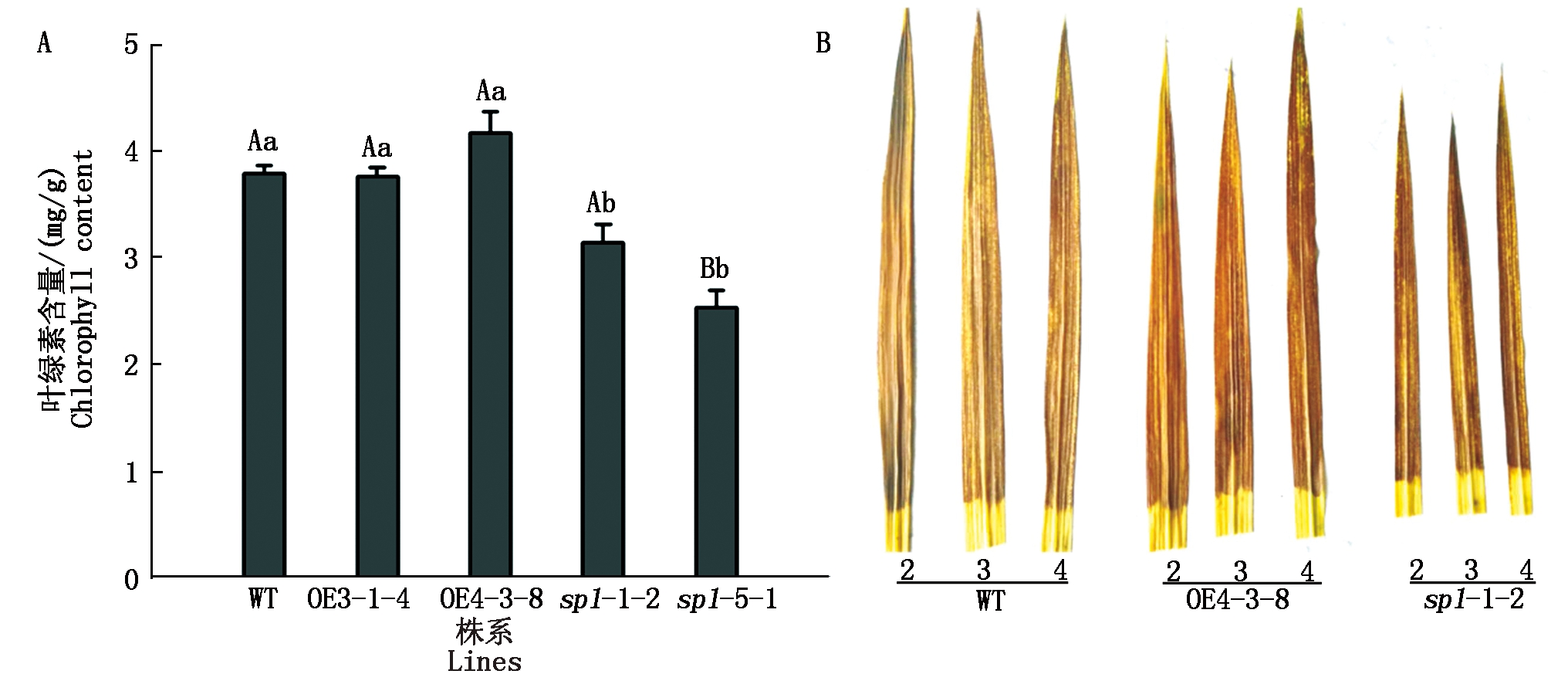
图5 过表达和基因编辑植株的叶绿素含量(A)和H2O2水平(B)
Fig.5 The chlorophyll values(A)and H2O2 level(B)for the overexpression and gene-edited plants
2.6 过表达植株的干旱耐受性
对三叶一心期的纯合过表达植株OE3-1-4和OE4-3-8进行重度干旱处理和复水,复水7 d后观察恢复状态,结果显示,在重度干旱处理和恢复供水后,2个过表达株系的恢复能力与野生型植株相比并无明显差异(图6),干旱胁迫抗性未见增强。

图6 恢复供水后过表达和野生型植株的干旱胁迫抗性
Fig.6 The drought stress tolerance of overexpression
and wild-type plants after rewatering
由于基因编辑植株在正常条件下即已出现严重的生长发育迟滞,且产生的种子量较少,发芽力差,未能对OsSP1基因编辑植株进行干旱处理和耐受性分析。
3 结论与讨论
在拟南芥中,sp1基因是叶绿体蛋白输入的控制基因,并通过该途径控制叶绿体内ROS的产生,进而影响植物的逆境响应能力[9,11]。叶绿体的功能与植物的光合效率直接相关,对于水稻等农作物,叶绿体蛋白质组的稳定性将直接影响到作物的产量和抗逆性[29]。为确定水稻OsSP1基因对水稻生长发育、产量及胁迫抗性的影响,本研究通过基因过表达技术创建了OsSP1基因过表达植株,通过CRISPR/Cas9基因编辑技术,在OsSP1基因编码区选择了双靶点进行编辑,获得了基因编辑植株。水稻SP1蛋白与拟南芥SP1蛋白高度同源[14],属于泛肽E3连接酶家族成员[12]。靶序列1和靶序列2的突变分别会造成SP1蛋白从第97个或第199个氨基酸(或其附近)开始发生移码突变或片段缺失,这2个位点均位于蛋白质分子中2个跨膜区之间的肽段上[14],发生移码突变后,其下游结构,包括第220-250位氨基酸残基区的跨膜序列,以及羧基端非跨膜肽段中的RING(Really interesting new gene)保守结构域,都将受到严重影响,造成SP1不能与泛肽系统中的泛肽结合酶(Ubiquitin-conjugating enzyme,E2)发生识别[11],从而失去既定功能。
在正常生长条件下,拟南芥sp1突变体的生长状态与野生型植株并无差别[9]。与拟南芥不同,水稻OsSP1基因编辑植株在正常生长条件下即表现出叶绿素水平显著或极显著下降、早衰、不分蘖、低结实率或不结实等表型。由于氧化胁迫是引发叶片早衰的重要因素[30-32],并且sp1在拟南芥中的作用机制与氧化胁迫有关[9],本研究也对水稻过表达和基因编辑植株进行了H2O2检测,但过表达和基因编辑植株中H2O2的生成水平相比野生型植株均无明显变化,说明在OsSP1基因编辑植株中,早衰等表型可能并不是氧化胁迫导致。根据OsSP1基因编辑植株的生长状况和显著或极显著降低的叶绿素水平,可确定OsSP1基因突变引发了叶绿体蛋白质组整体水平的变化和植物体的全面代谢失调[8],从而造成了矮小、早衰、减产或失去育性等严重缺陷,而不只局限于与光反应有关的蛋白质的输入失调和氧化胁迫。根据Pan等[33]的报道,拟南芥中存在sp1基因的同源基因,当sp1基因突变后,其部分功能可能由同源基因替代,因此,拟南芥sp1突变体在正常条件下没有出现明显的生长缺陷。根据水稻OsSP1基因编辑植株的表型,结合生物信息学分析结果[14],推测水稻中没有类似的同源基因补充OsSP1突变后缺失的功能,因此,OsSP1突变直接干扰了水稻的生长发育,并且其影响程度远超过sp1突变对拟南芥的影响。
拟南芥的sp1过表达植株在正常生长条件下与野生型植株表型一致,但对渗透胁迫和盐胁迫的抗性明显增强[9]。水稻的OsSP1过表达植株在正常条件下的生长状态和结实情况与野生型植株也无明显差异,但与拟南芥不同的是,在干旱处理后,OsSP1过表达植株的抗性也未见明显变化,表明在水稻中过量表达OsSP1后,对植株的生长发育和干旱抗性均无明显的促进作用,也无明显的干扰。
OsSP1基因编辑植株和过表达植株的表型和生理生化指标显示了OsSP1基因对水稻生长发育的重要调控作用,提高基因表达水平对水稻的生长和干旱抗性无明显影响,但基因突变严重影响水稻植株的生长发育和结实,其详细调控机制有待进一步发掘。
[1] Bose J,Munns R,Shabala S,Gilliham M,Pogson B,Tyerman S D. Chloroplast function and ion regulation in plants growing on saline soils:Lessons from halophytes[J].Journal of Experimental Botany,2017,68(12):3129-3143. doi:10.1093/jxb/erx142.
[2] Sabater B. Evolution and function of the chloroplast. Current investigations and perspectives[J].International Journal of Molecular Sciences,2018,19(10):3095. doi:10.3390/ijms19103095.
[3] Chan K X,Phua S Y,Crisp P,McQuinn R,Pogson B J. Learning the languages of the chloroplast:Retrograde signaling and beyond[J].Annual Review of Plant Biology,2016,67:25-53. doi:10.1146/annurev-arplant-043015-111854.
[4] Jarvis P,Kessler F. Mechanisms of chloroplast protein import in plants[M].//Plastid Biology.New York:Springer,2014.doi:10.1007/978-1-4939-1136-3_9.
[5] Richardson L G L,Small E L,Inoue H,Schnell D J. Molecular topology of the transit peptide during chloroplast protein import[J].The Plant Cell,2018,30(8):1789-1806. doi:10.1105/tpc.18.00172.
[6] Wang L X,Liang W,Xing J H,Tan F L,Chen Y Y,Huang L,Cheng C L,Chen W. Dynamics of chloroplast proteome in salt-stressed mangrove Kandelia candel (L.)Druce[J].Journal of Proteome Research,2013,12(11):5124-5136. doi:10.1021/pr4006469.
[7] Tamburino R,Vitale M,Ruggiero A,Sassi M,Sannino L,Arena S,Costa A,Batelli G,Zambrano N,Scaloni A,Grillo S,Scotti N. Chloroplast proteome response to drought stress and recovery in tomato(Solanum lycopersicum L.)[J].BMC Plant Biology,2017,17(1):40. doi:10.1186/s12870-017-0971-0.
[8] Ling Q H,Huang W H,Baldwin A,Jarvis P. Chloroplast biogenesis is regulated by direct action of the ubiquitin-proteasome system[J].Science,2012,338(6107):655-659. doi:10.1126/science.1225053.
[9] Ling Q H,Jarvis P. Regulation of chloroplast protein import by the ubiquitin E3 ligase SP1 is important for stress tolerance in plants[J].Current Biology,2015,25(19):2527-2534. doi:10.1016/j.cub.2015.08.015.
[10] Ling Q H,Jarvis P. Plant signaling:Ubiquitin pulls the trigger on chloroplast degradation[J].Current Biology,2016,26(1):R38-R40. doi:10.1016/j.cub.2015.11.022.
[11] Ling Q H,Broad W,Trösch R,Töpel M,Sert T D,Lymperopoulos P,Baldwin A,Jarvis R P. Ubiquitin-dependent chloroplast-associated protein degradation in plants[J].Science,2019,363(6429):eaav4467. doi:10.1126/science.aav4467.
[12] Pan R H,Hu J P. Sequence and biochemical analysis of Arabidopsis SP1 protein,a regulator of organelle biogenesis[J].Communicative & Integrative Biology,2017,10(4):e1338991. doi:10.1080/19420889.2017.1338991.
[13] Sadali N M,Sowden R G,Ling Q H,Jarvis R P. Differentiation of chromoplasts and other plastids in plants[J].Plant Cell Reports,2019,38(7):803-818. doi:10.1007/s00299-019-02420-2.
[14] 腾海艳. 水稻Ossp1基因的亚细胞定位及其干旱条件下的表达[J].江苏农业学报,2020,36(3):529-534. doi:10.3969/j.issn.1000-4440.2020.03.001.
Teng H Y. Subcellular localization and expression under drought conditions of rice Ossp1 gene[J].Jiangsu Journal of Agricultural Sciences,2020,36(3):529-534.
[15] Jinek M,East A,Cheng A,Lin S,Ma E B,Doudna J. RNA-programmed genome editing in human cells[J].eLife,2013,2:e00471. doi:10.7554/eLife.00471.
[16] Ran F A,Hsu P D,Wright J,Agarwala V,Scott D A,Zhang F. Genome engineering using the CRISPR-Cas9 system[J].Nature Protocols,2013,8(11):2281-2308. doi:10.1038/nprot.2013.143.
[17] Ma X L,Zhang Q Y,Zhu Q L,Liu W,Chen Y,Qiu R,Wang B,Yang Z F,Li H Y,Lin Y R,Xie Y Y,Shen R X,Chen S F,Wang Z,Chen Y L,Guo J X,Chen L T,Zhao X C,Dong Z C,Liu Y G. A robust CRISPR/Cas9 system for convenient,high-efficiency multiplex genome editing in monocot and dicot plants[J].Molecular Plant,2015,8(8):1274-1284. doi:10.1016/j.molp.2015.04.007.
[18] Li J F,Zhang D D,Sheen J. Cas9-based genome editing in Arabidopsis and tobacco[J].Methods in Enzymology,2014,546:459-472. doi:10.1016/B978-0-12-801185-0.00022-2.
[19] Char S N,Li R Q,Yang B. CRISPR/Cas9 for mutagenesis in rice[J].Methods in Molecular Biology,2019,1864:279-293. doi:10.1007/978-1-4939-8778-8_19.
[20] Wang Y,Geng L Z,Yuan M L,Wei J,Jin C,Li M,Yu K,Zhang Y,Jin H B,Wang E,Chai Z J,Fu X D,Li X G. Deletion of a target gene in Indica rice via CRISPR/Cas9[J].Plant Cell Reports,2017,36(8):1333-1343. doi:10.1007/s00299-017-2158-4.
[21] Zhang J S,Zhang H,Botella J R,Zhu J K. Generation of new glutinous rice by CRISPR/Cas9-targeted mutagenesis of the waxy gene in elite rice varieties[J].Journal of Integrative Plant Biology,2018,60(5):369-375. doi:10.1111/jipb.12620.
[22] Ozawa K. Establishment of a high efficiency Agrobacterium-mediated transformation system of rice(Oryza sativa L.)[J].Plant Science,2009,176(4):522-527. doi:10.1016/j.plantsci.2009.01.013.
[23] 陈文岳,包劲松,周祥胜,舒庆尧. 一种可用于PCR分析的水稻DNA简易提取法[J].中国水稻科学,2005,19(6):561-563. doi:10.16819/j.1001-7216.2005.06.015.
Chen W Y,Bao J S,Zhou X S,Shu Q Y. A simplified rice DNA extraction protocol for PCR analysis[J].Chinese Journal of Rice Science,2005,19(6):561-563.
[24] 腾海艳. 水稻叶绿体中干旱诱导型光呼吸代谢支路的创建[D].广州:华南农业大学,2017.
Teng H Y. Construction of a drought inducible photorespiratory by pass in chloroplasts of rice[D].Guangzhou:South China Agricultural University,2017.
[25] Schmittgen T D,Livak K J. Analyzing real-time PCR data by the comparative CT method[J].Nature Protocols,2008,3(6):1101-1108. doi:10.1038/nprot.2008.73.
[26] Arocho A,Chen B Y,Ladanyi M,Pan Q L.Validation of the 2-ΔΔCt calculation as an alternate method of data analysis for quantitative PCR of BCR-ABL P210 transcripts[J].Diagnostic Molecular Pathology,2006,15(1):56-61. doi:10.1097/00019606-200603000-00009.
[27] 李合生. 植物生理生化实验原理和技术[M].北京:高等教育出版社,2000:167-169.
Li H S. Principles and techniques of plant physiological biochemical experiment[M].Beijing:Higher Education Press,2000:167-169.
[28] Thordal-Christensen H,Zhang Z G,Wei Y D,Collinge D B. Subcellular localization of H2O2 in plants. H2O2 accumulation in papillae and hypersensitive response during the barley-powdery mildew interaction[J].The Plant Journal,1997,11(6):1187-1194.doi:10.1046/j.1365-313x.1997.11061187.x.
[29] Yang X L,Li Y Y,Qi M F,Liu Y F,Li T L. Targeted control of chloroplast quality to improve plant acclimation:From protein import to degradation[J].Frontiers in Plant Science,2019,10:958. doi:10.3389/fpls.2019.00958.
[30] Han M,Kim C Y,Lee J,Lee S K,Jeon J S. OsWRKY42 represses OsMT1d and induces reactive oxygen species and leaf senescence in rice[J].Molecules and Cells,2014,37(7):532-539.doi:10.14348/molcells.2014.0128.
[31] He Y,Li L J,Zhang Z H,Wu J L. Identification and comparative analysis of premature senescence leaf mutants in rice(Oryza sativa L.)[J].International Journal of Molecular Sciences,2018,19(1):140.doi:10.3390/ijms19010140.
[32] Woo H R,Kim H J,Lim P O,Nam H G. Leaf senescence:Systems and dynamics aspects[J].Annual Review of Plant Biology,2019,70:347-376. doi:10.1146/annurev-arplant-050718-095859.
[33] Pan R H,Satkovich J,Chen C,Hu J P. The E3 ubiquitin ligase SPl-like 1 plays a positive role in peroxisome biogenesis in Arabidopsis[J].The Plant Journal,2018,94(5):836-846. doi:10.1111/tpj.13900.