干旱胁迫是所有的非生物胁迫中最复杂,并且是全球规模的胁迫,对水稻产量及质量造成严重威胁[1]。由于C4作物在高温、强光以及干旱等条件下,较C3作物具有明显的生长优势、水分以及营养利用效率,生物学产量也较高[2],因此,科学家利用转基因技术,将玉米等C4植物中C4光合途径关键基因导入C3植物中,达到增强光合效率和提高产量的目的[3]。玉米磷酸烯醇式丙酮酸羧化酶(Phosphoenolpyruvate carboxylase,PEPC,EC 4.1.1.31)是C4光合途径的关键酶[4],已发现,高表达的转玉米C4-PEPC基因水稻在高光强[5]、高温[6]、干旱[7]和低氮[8]等逆境条件下表现出高光合效率和高产量的特性,因此,提高水稻的C4光合特性将是提高水稻抵御非逆境的有效策略之一[9]。
表观遗传机制调控植物应答外界胁迫是近年来发现的重要机制之一,其中DNA甲基化是表观遗传的重要形式之一,在调节应激相关基因表达和植物胁迫耐受中也起重要作用[10]。植物DNA甲基化机制又是非常复杂的,因物种、基因型、组织以及作用元件而异,但是很多研究都来自拟南芥的研究成果[11]。实际上,水稻基因组具有复杂性质,其不连续的异染色质和广泛分布的DNA甲基化也为了解植物DNA甲基化研究提供了更丰富的资源[12]。已绘制了水稻籼粳亚种在基因表达、生长发育以及胁迫响应中的DNA甲基化图谱[13]。前人研究表明,高表达转玉米C4-PEPC水稻植株的外源导入基因C4-PEPC启动子在干旱条件下也存在甲基化现象,而且通过C4-PEPC的去甲基化,上调C4-PEPC的表达和其酶活性,共同参与了转基因水稻植株对干旱的早期响应[7],提示DNA甲基化可能也是水稻响应干旱的机制之一。为此,本研究以高表达转玉米C4-PEPC基因水稻(PC)和野生型水稻(WT)为材料,通过施用不同浓度的DNA甲基化抑制剂5-氮杂胞苷(5-Azacytidine,5-azaC),研究不同生育期DNA甲基化在水稻响应干旱中影响,并研究聚乙二醇模拟的干旱条件下,分析DNA甲基化在水稻干旱响应的品种差异机制,为丰富水稻干旱响应机制提供信息。
1 材料和方法
1.1 种植
本试验使用的材料最初来源于Ku等[14]将玉米C4-PEPC基因导入日本粳稻Kitaake,C4-PEPC基因在野生型中获得高表达,获得的T2转基因材料,并经过江苏省农业科学院光合生理实验室在南京的18代稳定培养选育所得的转基因水稻(PC)以及未转基因原种水稻Kitaake(WT)。盆栽试验于2018年5-9月在江苏省农业科学院粮食作物研究所水稻示范基地网室中进行。5月2日,将水稻种子使用75%的乙醇浸泡5 min,随后使用50%次氯酸钠浸泡15 min,蒸馏水冲洗数次,保证无消毒液残留,消毒后使用蒸馏水浸种,置于30 ℃黑暗条件下泡种3 d,待种子露白,放在湿润的培养皿中催芽,当种子长出约0.5 cm的芽后,选取生长一致的材料,于5月10日播种于江苏省农业科学院网室中,常规水肥和病虫害管理。
1.2 水稻发芽率试验
将消毒的水稻种子分组,试验包括对照组( 清水发芽培养),以下简称处理A,模拟干旱组(10% 聚乙二醇6000 ,10% Polyethylene glycol 6000,简称10% PEG,处理B)以及5-氮杂胞苷(5-Azacytidine,5-azaC)联合模拟干旱组(5-azaC+10% PEG),5-azaC (A2385)购自Sigma公司。芽期5-azaC 联合模拟干旱组设置7 个处理浓度,分别为C:2.5 μmol/L+10% PEG、D:5.0 μmol/L+10% PEG、E:7.5 μmol/L+10% PEG、F: 10.0 μmol/L+10% PEG、G:30.0 μmol/L+10% PEG、H: 50.0 μmol/L+10% PEG和I:100.0 μmol/L+10% PEG。每个处理重复3 次,每次100 粒,试验材料培养4 d,每天记录发芽率(发芽率=当天内总发芽种子数/供试种子数×100%)。
1.3 水培试验处理
消毒的水稻种子浸种催芽,待长出约0.5 cm的芽后,将种子按间隔插入黑色的水稻培养板上培养,在长出第2片叶子之前均用清水培养,水稻长到2叶期时,将幼苗转移到用国际水稻所(International Rice Research Institute)标准营养液中,置于 30 ℃/25 ℃ (昼/夜)、14 h/10 h(光/暗)光照培养箱中培养[15]。待水稻长到 5~6 叶期后,选择株型、长势和叶片均一的植株作为试验材料,将材料分组,以国际标准营养液作为对照,药剂处理采用根吸入法,在处理前一天,将植株移入含不同浓度5-azaC的营养液,暗中预处理12 h,次日再用光下适应2 h,之后将植株转移到含有10%(m/V) PEG-6000的培养液中(以下称模拟干旱胁迫,Drought stress,DS),在光下处理2 h 后。之后取下倒二叶,迅速在-80 ℃液氮中保存,用于测定各项生理指标,同时测定处理前后相对含水量(Relative water content,RWC),在苗期并以RWC 作为耐旱性指标。
1.4 盆栽试验处理
盆栽里装的土为稻田黏壤土,每1种处理的PC和WT分别种植9盆,盆钵提前做好随机区组分布,将发芽的水稻种植在盆钵中,日常水肥和病虫管理。待水稻幼苗长至4~5叶期时,挑选长势均一的幼苗移栽至仅施加了基肥(即复合肥,含15%的N,15%的P,15%的K)的盆中,每盆栽5穴,每穴栽1株,盆栽试验所用的器皿均为底部直径25 cm,高25 cm的陶瓷盆钵。盆栽试验总施氮量为300 kg/hm2,分别以基肥(35% N)、分蘖肥(20% N)和穗肥(45% N)等按时期施加,移栽后所施加的氮肥均为尿素。随机分成2组,一组正常灌溉(对照,CK),一组自然干旱(Drought stress,DS)。其中自然干旱处理在水稻孕穗期对试验组PC和WT停止正常浇灌,进行自然干旱处理,将盆钵排水自然落干,顶部用透明薄膜覆盖防雨水,自然干旱组又随机分为3组,另外2组为5-azaC联合干旱组,其中5-azaC设置了2个浓度,分别为50,100 μmol/L,该处理每5 d分别喷施2种浓度5-azaC溶液1次,共处理5次,每株共喷施50 mL溶液(DS+5-azaC);单独自然干旱组则喷施同样体积的清水(以下称干旱胁迫,DS)。5 次处理结束后,所有处理均恢复正常灌溉,开花后50 d 收获,考察产量及其构成因子。
1.5 光合参数的测定
不同处理的盆栽水稻植株在开花后7~14 d测定剑叶的光合参数,方法参照Li等[16]的方法,使用美国LI-COR公司生产的LI-6400款便携式光合测定仪,选择晴天无云的8:30-11:30室外条件下进行测定,叶片光合参数测定的条件:使用红蓝光源测定,光量子通量密度(PPFD,Photosynthetic photon flux density) 800 μmol/(m2·s),流速500 μmol/s,待CO2和H2O开始稳定后开始测量。测定供试材料的净光合速率(Net photosynthetic rate,Pn)、气孔导度(Stomatal coductence,Gs)、胞间CO2浓度(Intercellular CO2 concentration,Ci)和蒸腾速率(Transpiration rate,Tr),通过WUE=Pn/Tr,计算出水分利用率(Water use efficiency,WUE)。通过CE=Pn/Ci,计算出羧化效率(Carboxylation efficiency,CE)。每个处理测定5张叶片,每个处理5次重复。
1.6 相对含水量的测定
植株的水分状态通过植株相对含水量(Relative water content,RWC)的测定决定[17]。试验包括对照组(清水处理,处理A)、模拟干旱组(10% PEG,处理B)以及5-azaC联合模拟干旱组(5-azaC+10% PEG),苗期5-azaC联合模拟干旱组设置9个处理浓度,分别为C:2.5 μmol/L+10% PEG、D:5.0 μmol/L+10% PEG、E:7.5 μmol/L+10% PEG、F:10.0 μmol/L+10% PEG、G:30.0 μmol/L+10% PEG、H:50.0 μmol/L+10% PEG、I:100.0 μmol/L+10% PEG、J:250.0 μmol/L+10% PEG和K:500.0 μmol/L+10% PEG。植株在光下处理2 h后,剪取第4叶中间6 cm部分,测其鲜质量(Fresh weight,FW),然后将叶切片在室温下漂浮在去离子水中6 h,测定其饱和水质量(Turgid weight ,TW),随后在105 ℃烘箱中杀青15 min,70 ℃的烘箱中干燥过夜至恒质量,再称质量以获得干质量(Drought weight,DW)。RWC=(FW-DW)/(TW-DW)×100%[17],每个处理测定5 张叶片,每个处理5个重复。
1.7 丙二醛含量的测定
根据Velikova等[18]方法,测定样品中丙二醛含量。
1.8 可溶性蛋白和脯氨酸含量的测定
参照考马斯亮蓝G-250法[19]测定苗期叶片的可溶性蛋白含量,脯氨酸含量参考Troll等[20]方法测定。
1.9 可溶性糖和各糖组分含量的测定
样品中可溶性总糖、蔗糖、葡萄糖和果糖含量参照Ambavaram等[21]的方法进行。
1.10 磷酸烯醇式丙酮酸羧化酶活性的测定
磷酸烯醇式丙酮酸羧化酶活性的测定参考唐玉婷等[8]的方法。
1.11 总RNA提取和qRT-PCR
总RNA 的提取和实时荧光定量聚合酶链式反应(Quantitative real-time PCR,qRT-PCR) 根据等报道的方法制备总RNA和反转录[5]。在PCR 仪(ETC811,北京东胜创新生物科技有限公司)上进行。根据制造商的说明书使用SYBR Premix Ex Taq Ⅱ 试剂盒(TaKaRa Biotechnology(大连)有限公司)进行qRT-PCR 分析,用Applied Biosystems Step One 实时PCR 系统(Applied Biosystems,Foster City,CA,USA)分析。qRT-PCR 反应条件为95 ℃ 10 min;94 ℃ 30 s,58 ℃ 40 s 和68 ℃ 各 1 min,共32 次循环。重复3 次。在Primer 3 上设计引物,以水稻组成性表达的Actin 基因为内部参照,引物序列见表1,所有引物均由生工生物工程(上海)有限公司合成。
1.12 产量构成因子的测定
在植株开花后50 d,收获植株,在收获取样时各个处理取长势和穗数平均值相近的10株测定其产量构成因子,收获后自然风干,当籽粒含水量≤14.5%后,再进行室内考种。测定每株株高、穗数、穗长、穗实粒数、穗粒数、结实率、千粒质量以及单株产量。
1.13 统计分析
使用SPSS 19.0软件对数据进行ANOVA分析,采用Excel 2013对数据进行描述和作图,P<0.05。
表1 实时荧光定量聚合酶链式反应的基因引物
Tab.1 Gene primes for qRT-PCR

基因Gene引物序列Primers登录号Login IDActinF:5′-CCCTCTTTCATCGGTATGGA-3′Os01g4349863R:5′-TTGATCTTCATGCTGCTTGG-3′C4-PEPCF:5′-CCCACTATCCTTCGCAAGAC-3′E17154R:5′-CTAGCCAGTGTTCTGCATGCCGG-3′OsMET1aF:5′-GAAGCTGTCCACAGGGCAGA-3′Os03g0798400R:5′-AAATGAGGGCGATGCAGTGAAG-3′OsMET1bF:5′-ACTATCATCTGGCCAACTGGTGG-3′Os07g0182900R:5′-CCCTCGATCATCGGAGAAGG-3′OsK24F:5′-CGTGTTGGCTTCAGTGAAT-3′Os05g4433941R:5′-CCTTCTCTATCTAAGGGCCG-3′OsK35F:5′-TTGTGTTGGCTTCAGTGAAA-3′Os08g4349863R:5′-CCTTCGCTGTCTAAGGACTG-3′OsK1aF:5′-AACCAGAGGTAACAGGCAGG-3′Os03g4332495R:5′-CATCTGTCAAGGAATGCAGG-3′
注:Actin.肌动蛋白基因;OsK1a、OsK24、OsK35.丝氨酸/苏氨酸蛋白激酶基因;C4-PEPC.C4型磷酸烯醇式丙酮酸羧化酶基因;OsMET1.甲基转移酶1基因。
Note:Actin. Actin;OsK1a,OsK24,OsK35. Serine/threonine protein kinase gene;C4-PEPC. C4 phosphoenolpyruvate carboxylase gene;OsMET1. Methyltransferase 1 gene.
2 结果与分析
2.1 不同浓度5-azaC对水稻发芽率的影响
从表2可见,在模拟干旱条件下,小于30.0 μmol/L的5-azaC处理均促进水稻快速萌动,而且在培养第4天具有较高的发芽率,而大于G处理(5-azaC浓度为30.0 μmol/L)缓解干旱抑制的效果不显著,5-azaC对两材料干旱缓解作用浓度有差异,其中WT为C处理、D处理和E处理(2.5~7.5 μmol/L),而PC则在F处理(10.0 μmol/L 5-azaC联合PEG)具有最高的发芽率,低浓度5-azaC对芽期水稻干旱有缓解效果,且材料之间有差异。
2.2 喷施不同浓度5-azaC联合10% PEG-6000 模拟干旱胁迫对水稻苗期的影响
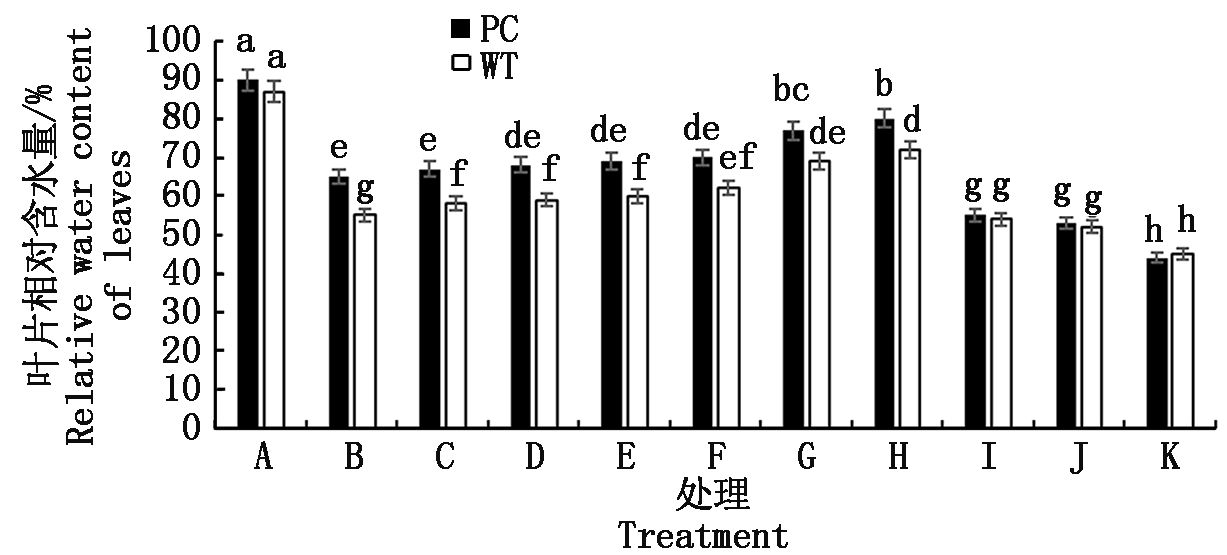
2.2.1 叶片相对含水量 如图1可知,较低浓度5-azaC处理对模拟干旱条件下水稻的保水能力有一定促进效果,其中WT的5-azaC促进作用浓度为2.5~50.0 μmol/L,而PC则为30.0~50.0 μmol/L,也呈现在该促进的作用浓度下,随着浓度的增加,促进作用增强,但是到50.0 μmol/L(H处理)出现阈值,其中苗期两材料DNA抑制剂处理浓度的差异提示两材料DNA甲基化水平不同。当达到100.0 μmol/L 5-azaC时,水稻叶片的RWC则显著下降,两材料的保水能力没有显著差别,继续增加浓度,则与100.0 μmol/L 5-azaC处理均没有显著差异,而500.0 μmol/L 5-azaC处理则抑制更显著,均低于单独模拟干旱处理的水平,提示,DNA甲基化过程也参与水稻苗期干旱响应,其中两材料DNA甲基化水平的差异可能是其耐旱差异表现的内在机制。结合供试水稻芽期和苗期的抑制剂作用浓度,可以看出,芽期<苗期。
表2 不同水稻材料在不同5-azaC浓度处理下发芽率的变化
Tab.2 Germination rates of difference varieties at different concentrations of 5-azaC %
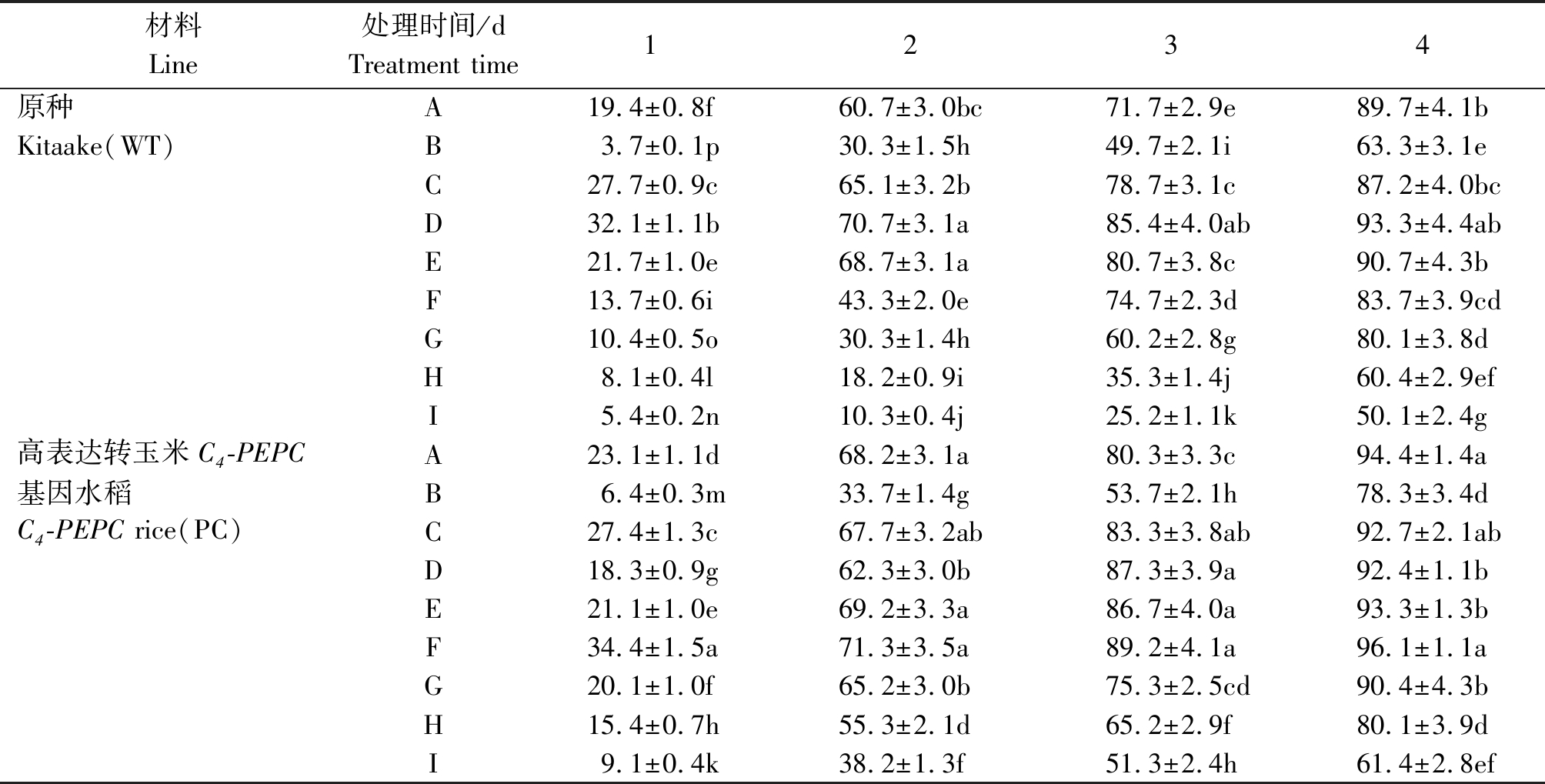
材料Line处理时间/dTreatment time 1234原种A19.4±0.8f60.7±3.0bc71.7±2.9e89.7±4.1bKitaake(WT)B3.7±0.1p30.3±1.5h49.7±2.1i63.3±3.1eC27.7±0.9c65.1±3.2b78.7±3.1c87.2±4.0bcD32.1±1.1b70.7±3.1a85.4±4.0ab93.3±4.4abE21.7±1.0e68.7±3.1a80.7±3.8c90.7±4.3bF13.7±0.6i43.3±2.0e74.7±2.3d83.7±3.9cdG10.4±0.5o30.3±1.4h60.2±2.8g80.1±3.8dH8.1±0.4l18.2±0.9i35.3±1.4j60.4±2.9efI5.4±0.2n10.3±0.4j25.2±1.1k50.1±2.4g高表达转玉米C4-PEPCA23.1±1.1d68.2±3.1a80.3±3.3c94.4±1.4a基因水稻B6.4±0.3m33.7±1.4g53.7±2.1h78.3±3.4dC4-PEPC rice(PC)C27.4±1.3c67.7±3.2ab83.3±3.8ab92.7±2.1abD18.3±0.9g62.3±3.0b87.3±3.9a92.4±1.1bE21.1±1.0e69.2±3.3a86.7±4.0a93.3±1.3bF34.4±1.5a71.3±3.5a89.2±4.1a96.1±1.1aG20.1±1.0f65.2±3.0b75.3±2.5cd90.4±4.3bH15.4±0.7h55.3±2.1d65.2±2.9f80.1±3.9dI9.1±0.4k38.2±1.3f51.3±2.4h61.4±2.8ef
注:不同小写字母表示0.05 水平上差异显著(P<0.05)。表3 同。
Note:The different lowercase letters are significant difference at 0. 05 level(P<0.05).The same as Tab. 3.
不同小写字母表示0.05 水平上差异显著(P<0.05)。图2-8同。
The different lowercase letters are significant difference at 0. 05 level (P<0.05).The same as Fig.2-8.
图1 模拟干旱条件下不同浓度的5-azaC对水稻苗期叶片相对含水量的影响
Fig.1 Effects of different concentrations of 5-azaC on the relative water content of rice leaves at seedling stage under simulated drought conditions
2.2.2 叶片的C4-PEPC基因表达和PEPC酶活性 如图2可知,小于50.0 μmol/L 5-azaC联合模拟干旱处理,与单独干旱处理相比,对PC的C4-PEPC表达没有显著影响,与单独模拟干旱的水平类似,但是高于这个浓度,则显著抑制了该基因的表达水平。而对水稻叶片的PEPC酶活性则也存在浓度阈值,其中PC叶片中PEPC酶活性在30.0 μmol/L 5-azaC联合模拟干旱处理活性显著高于单独干旱的处理,50.0,100.0 μmol/L 5-azaC联合模拟干旱处理的均显著低于其单独干旱处理,但是还是高于未处理的对照,而500.0 μmol/L 5-azaC联合模拟干旱处理则显著抑制了PEPC酶活性;与PC相比,不同处理WT的酶活性均较低,DNA甲基化抑制剂对其影响的趋势与PC的类似,基因表达和酶活性的表现与叶片的含水量有一致的表现。很显然,低浓度的5-azaC联合干旱处理增加了水稻酶活性,而随着处理浓度的增加减少了两材料酶活性差值的幅度,500.0 μmol/L 5-azaC处理的抑制最大,显著低于未处理对照,提示外源导入的C4-PEPC与水稻DNA甲基化水平的差异表现有关。
2.2.3 叶片丙二醛含量 进一步选取了2个导致供试水稻叶片相对含水量差异表现的作用浓度(50.0,100.0 μmol/L 5-azaC)联合模拟干旱处理,测定了植株倒二叶的丙二醛含量(图3)。与B处理相比,H处理(DS+50.0 μmol/L 5-azaC)可以显著降低相同水稻材料的MDA含量,其中PC的含量显著小于WT的,而I处理(DS+100.0 μmol/L 5-azaC)则显著增加了水稻MDA的含量,但两材料含量不存在显著差异,提示不同浓度的5-azaC处理造成了供试水稻膜脂过氧化水平差异,其中低浓度处理下PC的膜脂过氧化较低,伤害较少,有利于保持干旱胁迫下其叶片的水分。
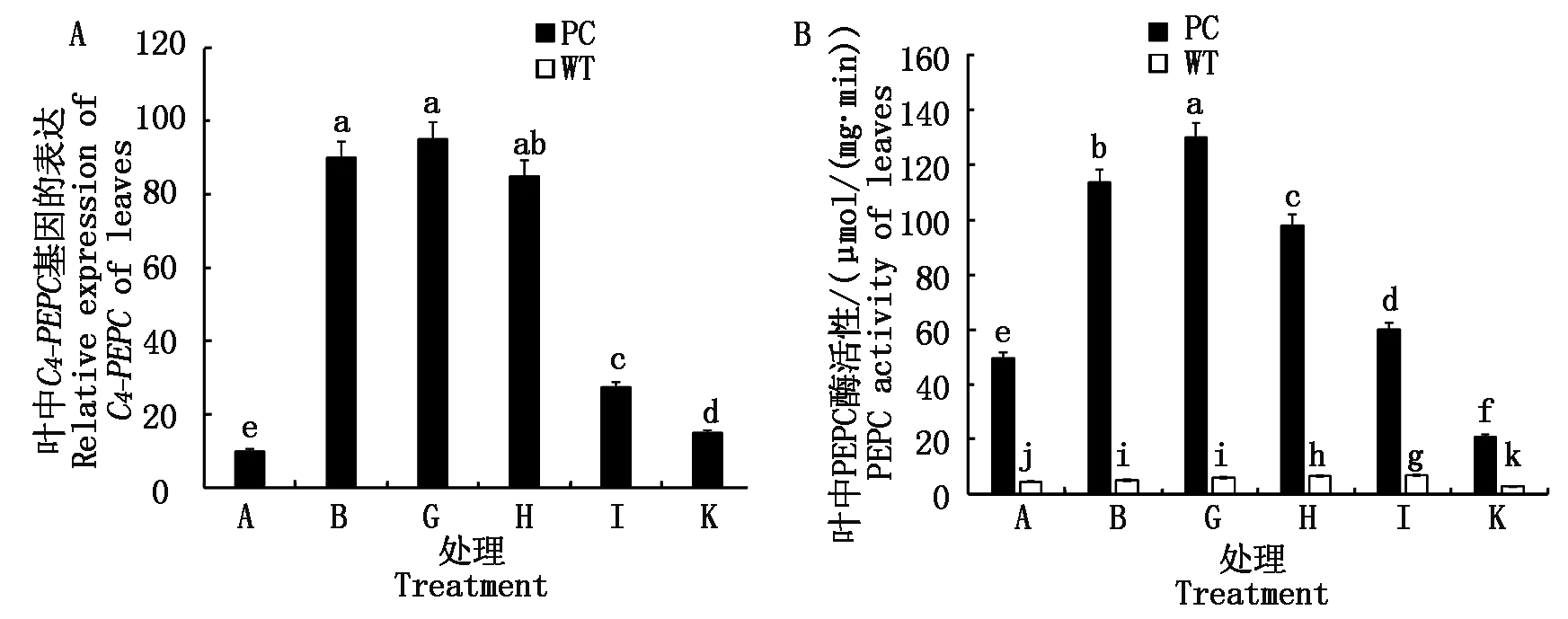
图2 模拟干旱条件下不同浓度5-azaC对水稻苗期叶片C4-PEPC基因表达和PEPC酶活性的影响
Fig.2 Effects of different concentrations of 5-azaC on the relative expression C4-PEPC and PEPC activities of rice leaves at seedling stage under simulated drought conditions
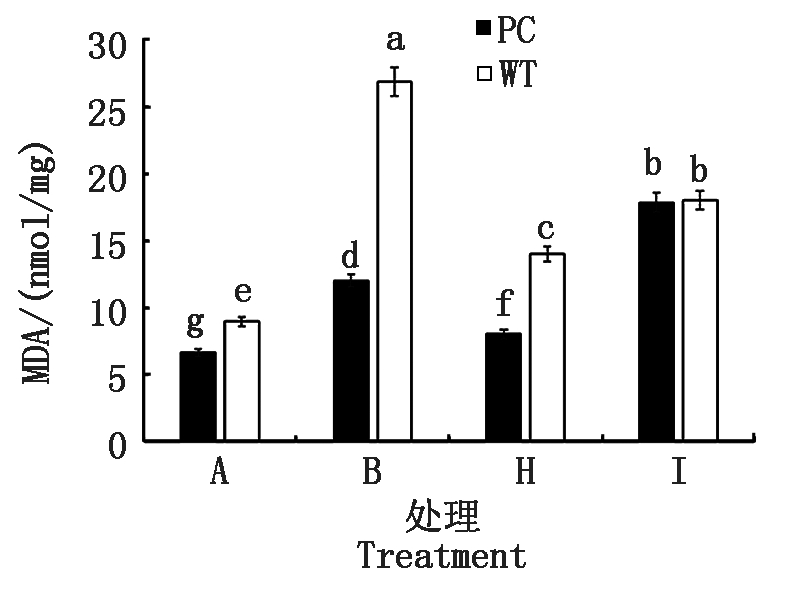
图3 模拟干旱条件下不同浓度5-azaC对水稻苗期叶片丙二醛的影响
Fig.3 Effects of different concentrations of 5-azaC on MDA content of rice leaves at seedling stage under simulated drought conditions
2.2.4 水稻叶片可溶性总糖、可溶性蛋白含量和脯氨酸含量 从图4-A可知,与B处理相比,H处理和I处理均显著降低了PC水稻叶片可溶性糖的含量,但低浓度的H处理没有改变两材料的差异,PC仍大于WT,但是高浓度的I 处理下两材料的含量不存在显著差异;与DS处理相比,两材料在低浓度的5-azaC+DS处理下的可溶性蛋白的含量不存在显著差异,而高浓度则显著降低了两材料叶片的可溶性蛋白,且WT大于PC(图4-B);叶片中脯氨酸含量则与可溶性糖的含量变化趋势相反,与DS处理相比,5-azaC+DS处理均显著增加了脯氨酸含量,且5-azaC的浓度越高,其增幅越显著,但是高浓度5-azaC+DS处理下两材料的含量不存在显著差异(图4-C),提示叶片中可溶性糖和脯氨酸含量的差异可能是两材料干旱条件下DNA甲基化表现差异的重要物质。
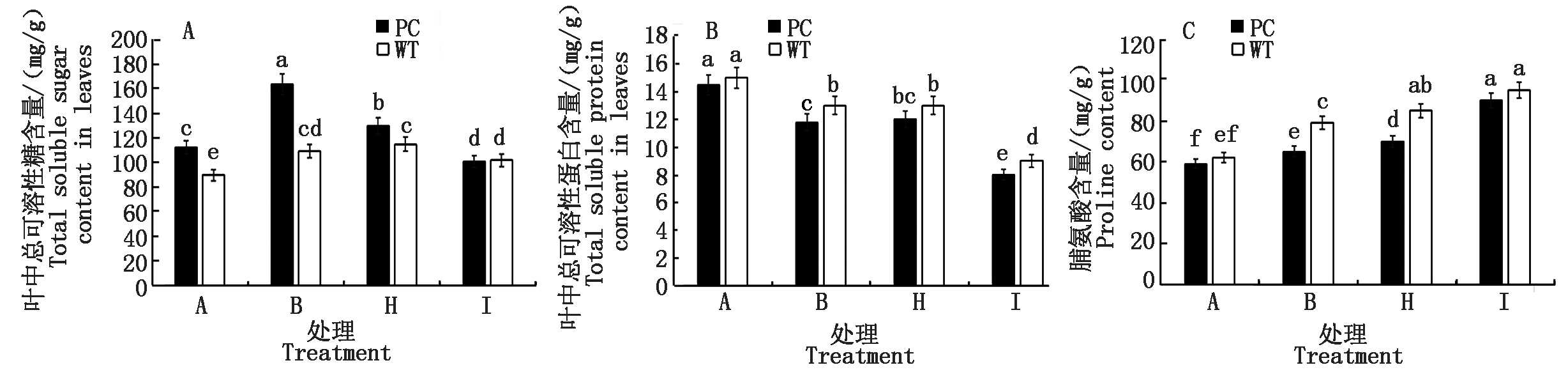
图4 模拟干旱条件下不同浓度5-azaC对水稻苗期叶片可溶性糖、可溶性蛋白和脯氨酸含量的影响
Fig.4 Effects of different concentrations of 5-azaC on soluble sugar,soluble protein and proline content of rice leaves at seedling stage under simulated drought conditions
2.2.5 水稻叶片可溶性糖组分 从图5水稻叶片可溶性糖组分可知,与B处理相比,H处理和I处理均显著提高了PC中蔗糖含量,而低浓度5-azaC+DS处理则显著降低了WT的蔗糖含量,而高浓度则显著增加了WT的蔗糖含量(图5-A);与对照相比,B处理增加了WT的蔗糖含量而降低了PC的蔗糖含量。进一步与B处理相比,2个5-azaC+DS处理均显著增加了两材料的葡萄糖含量,但是品种间没有显著差异,提示导致2个5-azaC浓度差异的水稻干旱表现可能与葡萄糖关系不大(图5-B);与对照相比,B处理,均导致两材料叶片内果糖含量的增加,但两材料含量不存在显著差异,H处理(50.0 μmol/L 5-azaC)则提高了PC果糖含量,而降低了WT的果糖含量,恢复到与未处理对照的水平,而高浓度5-azaC联合干旱处理则显著降低了两材料果糖的含量,而且品种间不存在显著性差异(图5-C)。
2.2.6 水稻叶片SnRK1基因表达 蔗糖非发酵1(Sucrose nonfermenting-1,SNF1)蛋白激酶属于丝氨酸/苏氨酸蛋白激酶,是重要的能量和逆境的枢纽,在水稻中SnRK1亚家族包括3个基因,分别是OsSnRK1A、OsK24和OsK35[22],并且与蔗糖含量密切相关[23]。从图6可以看出,水稻的3个SnRK1基因的表达无论在单独模拟干旱处理还是在50.0 μmol/L 5-azaC联合干旱处理均显示了两材料的差异,且PC大于WT,但在高浓度100.0 μmol/L 5-azaC联合干旱处理则没有显著差异,提示蔗糖和糖信号基因表达的差异可能是导致水稻DNA甲基化水平差异的内在机制。
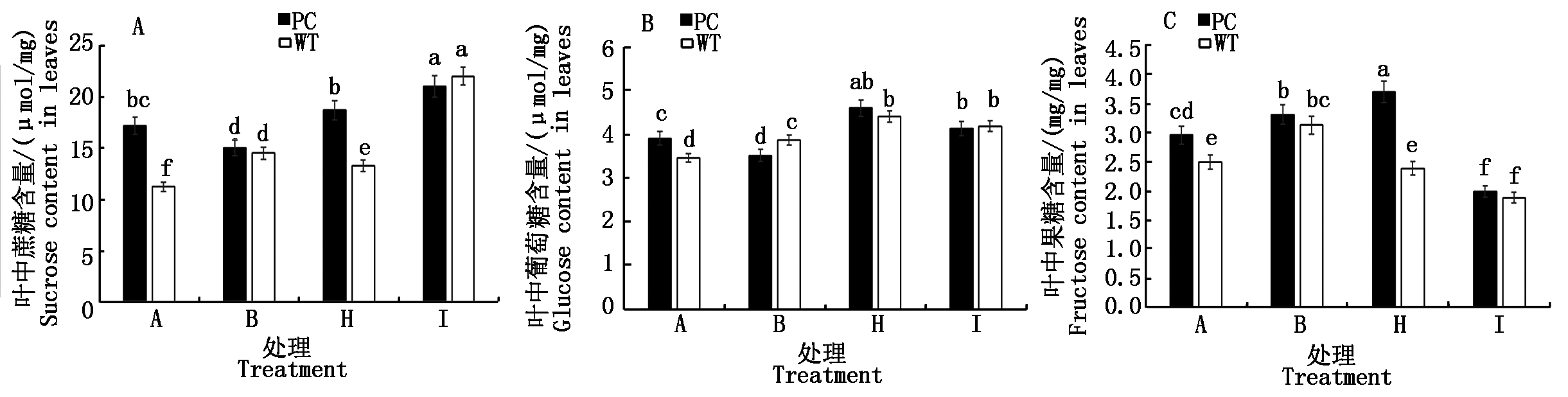
图5 模拟干旱条件下不同浓度5-azaC对水稻苗期叶片蔗糖、葡萄糖和果糖含量的影响
Fig.5 Effects of different concentrations of 5-azaC on sucrose,glucose and fructose contents of rice leaves at seedling stage under simulated drought conditions
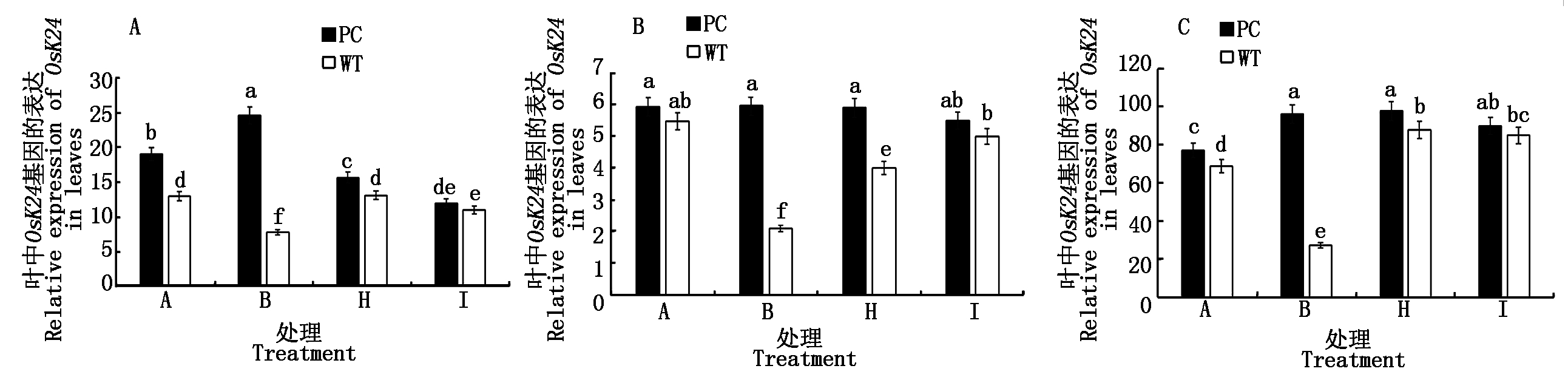
图6 模拟干旱条件下不同浓度5-azaC对水稻苗期叶片SnRK1s基因表达的影响
Fig.6 Effects of different concentrations of 5-azaC on the relative gene expression of SnRK1s of rice leaves at seedling stage under simulated drought conditions
2.2.7 水稻叶片甲基化酶基因的表达 作为最普遍类型的胞嘧啶甲基化,植物CG甲基化由主要的CG甲基化酶和甲基转移酶1(MET1)保持,MET1基因的主要作用是保持CpG的甲基化[24]。水稻基因组中有2个MET1基因,分别为OsMET1a[25]和OsMET1b[26]。如图7可知,与对照相比,DS处理降低了水稻的2个MET1基因的表达,而50 μmol/L 5-azaC+DS处理与DS处理下PC的OsMETIa表达水平没有显著差异,但是进一步降低了WT的基因表达水平,而高浓度5-azaC+DS干旱处理进一步显著降低了两材料的基因表达水平,仍然可以看出,PC的表达大于WT(图7-A),而对于OsMET1b,与其单独干旱处理(B)相比,低浓度5-azaC+DS处理(H)显著诱导了供试水稻的OsMET1b增加,但是对于同一处理(H),PC的诱导水平显著大于WT,而高浓度则显著降低了PC相关的基因表达;而对高浓度WT的表达,与其低浓度的相比,则仍显示显著增加趋势(图7-B),看来,DNA甲基化相关基因的表达水平与其5-azaC处理的表现一致,PC显著高于WT。
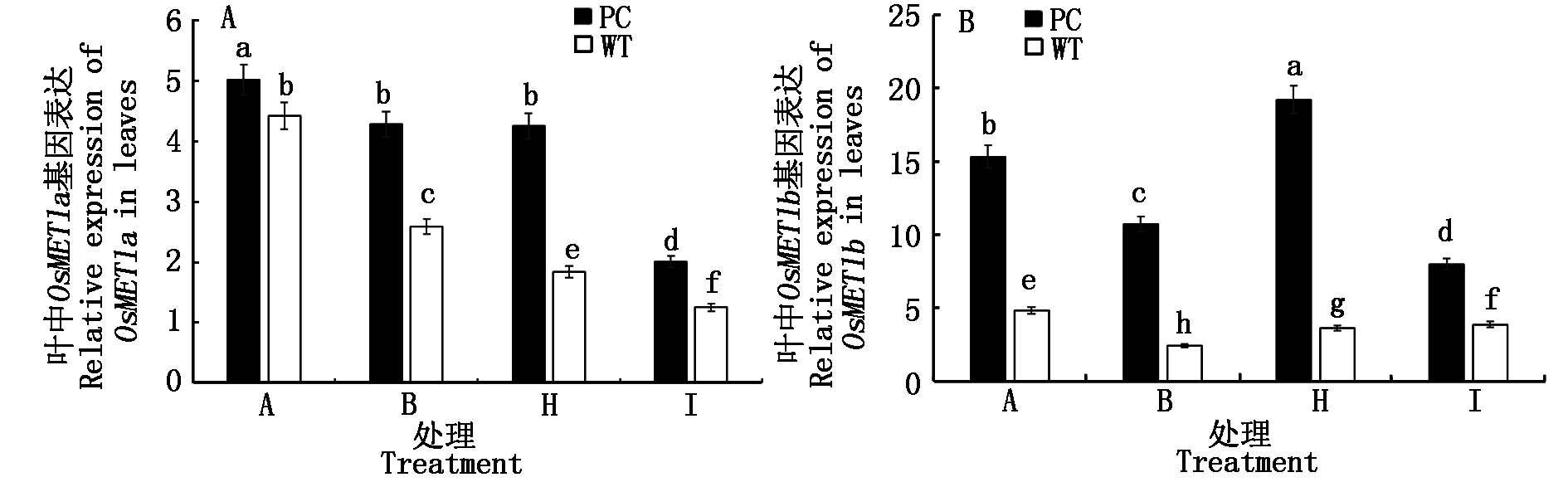
图7 模拟干旱条件下不同浓度5-azaC对水稻苗期叶片OsMET1基因表达的影响
Fig.7 Effects of different concentrations of 5-azaC on the relative gene expression of OsMET1 of rice leaves at seedling stage under simulated drought conditions
2.3 水稻剑叶的光合参数
进一步通过盆栽试验,在生育后期,自然干旱处理并联合喷施50.0,100.0 μmol/L 5-azaC溶液,观察不同处理下水稻剑叶的光合参数的变化。如图8-A可知,两浓度均显著抑制了水稻的净光合速率,并没有观察到与芽期和苗期低浓度促进,高浓度抑制的趋势,但在两浓度下PC的净光合速率数值始终显著高于同样处理下WT的,无论是对照,还是单独或联合干旱处理,PC仍然高于WT;50.0 μmol/L 5-azaC联合模拟干旱处理显著降低了水稻的气孔导度,而100.0 μmol/L 5-azaC则显著提高两材料的气孔导度(图8-B);蒸腾速率也是类似的表现(图8-D);相应地,降低了100.0 μmol/L 5-azaC处理下的水分利用率(图8-E),而只提高WT的羧化效率(图8-F),看来,DNA甲基化抑制剂对生育后期水稻植株光合特性的影响更复杂。
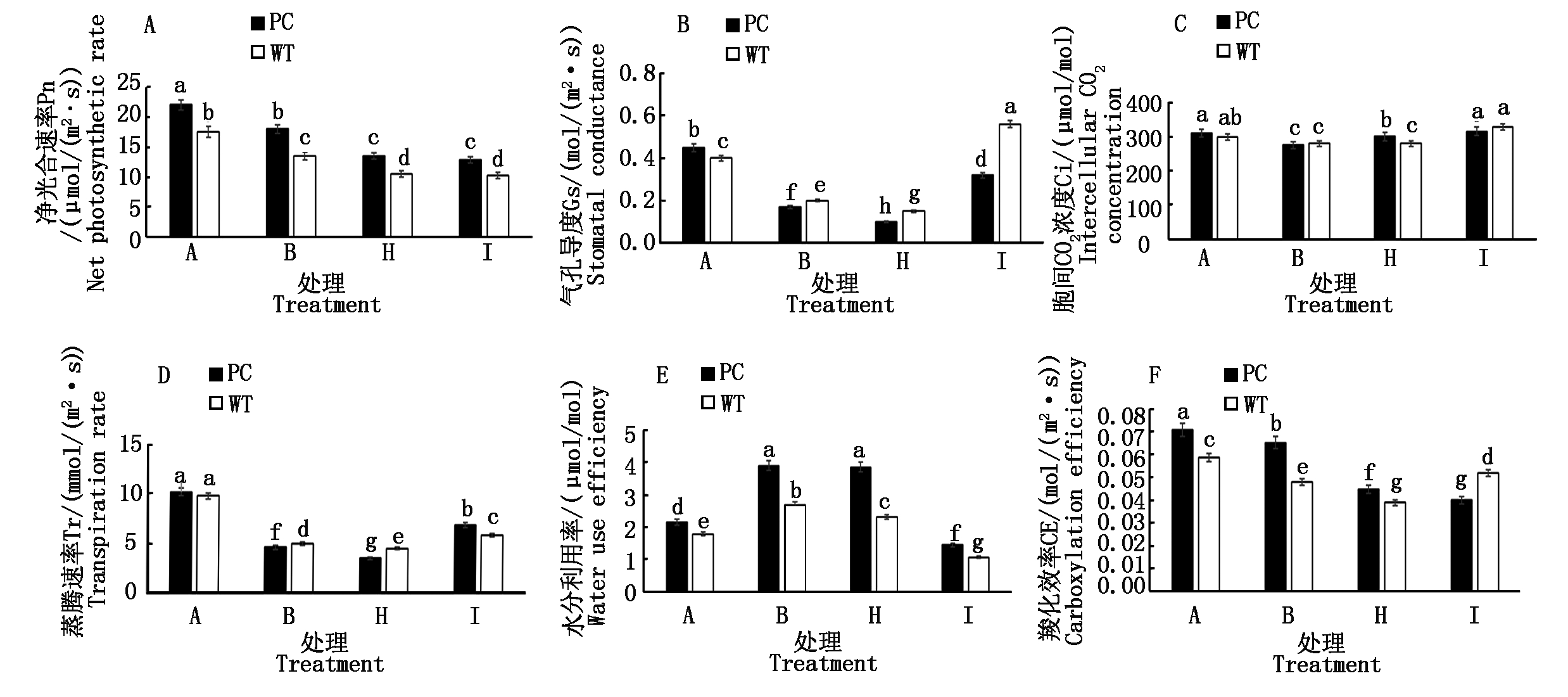
图8 自然干旱条件下不同浓度5-azaC对水稻开花后14 d剑叶的光合参数的影响
Fig.8 Light parameters of the flag leaf in different varieties on 14th day after flowing at different 5-azaC concentrations under natural drought conditions
2.4 5-azaC对水稻产量构成因子的影响
如表3盆栽试验结果所示,单独干旱处理显著降低了水稻的株高,50.0 μmol/L 5-azaC+DS处理对水稻株高没有显著影响,与DS的类似,而高浓度100.0 μmol/L 5-azaC+DS处理显著降低了WT的株高;DS处理也减少了单株穗数,5-azaC+DS处理可以部分缓解单株穗数的减少,其中PC的效果显著,而WT的并不显著;DS和50.0 μmol/L 5-azaC+DS对穗长影响不显著,高浓度5-azaC+DS显著降低了WT的穗长;穗粒数和千粒质量的表现与穗长的类似;DS处理也显著减少了水稻的单穗实粒数,而且WT显著小于PC。值得注意的是,与对照相比,DS和50.0 μmol/L 5-azaC+DS处理降低了WT的结实率,而高浓度的5-azaC+DS处理则显著增加了水稻的结实率,但是由于DNA甲基化抑制剂对产量构成的其他因子均呈负效应,因此,最终的单株产量,2个DNA甲基化抑制剂联合干旱处理对干旱条件下水稻产量的下降没有显著的缓解效果,其中高浓度100.0 μmol/L 5-azaC+DS处理对WT的抑制效果更大,显著低于其DS和50 μmol/L 5-azaC+DS,可见,DNA甲基化水平在生育后期的调控更复杂,产量和剑叶的净光合速率的变化类似,均是负效应。
表3 不同水稻材料在不同处理下产量及其构成因子的变化
Tab.3 Yields and composition of different varieties at different treatments
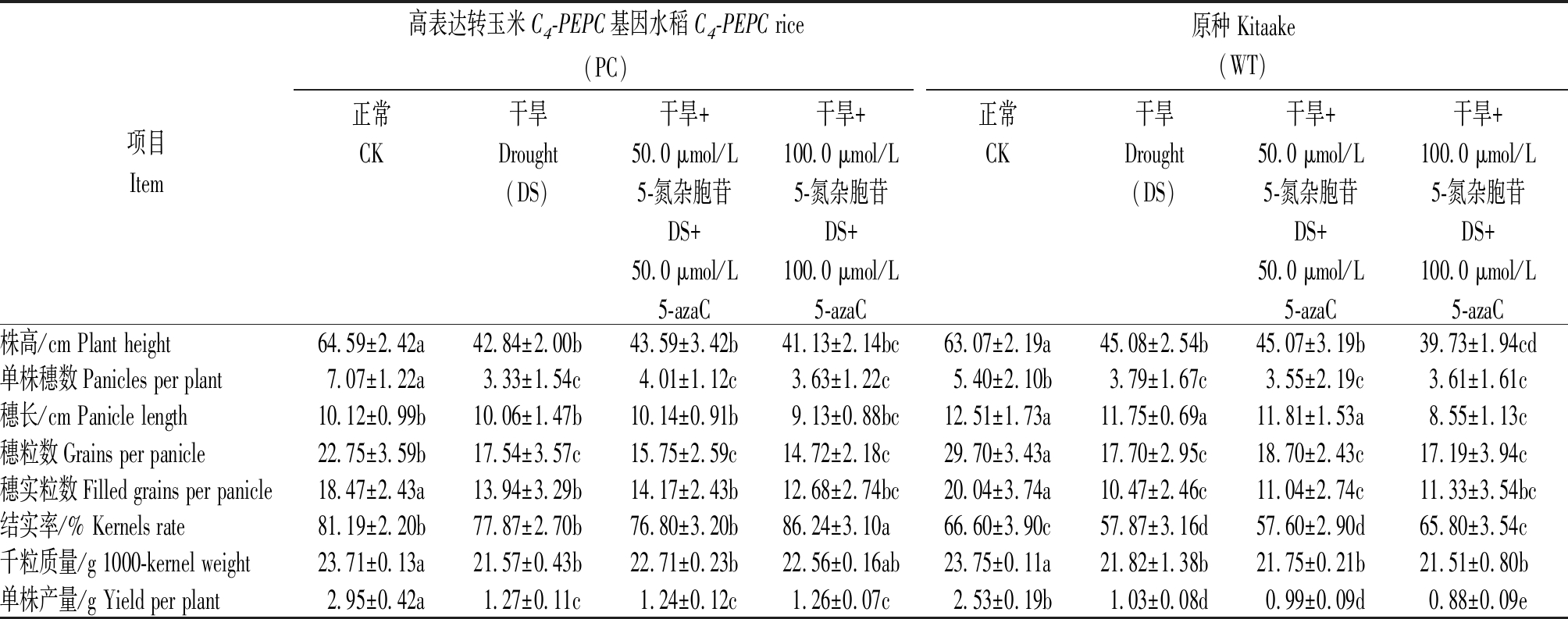
项目Item高表达转玉米C4-PEPC基因水稻 C4-PEPC rice(PC)原种 Kitaake(WT)正常CK干旱Drought(DS)干旱+50.0 μmol/L5-氮杂胞苷DS+50.0 μmol/L5-azaC干旱+100.0 μmol/L5-氮杂胞苷DS+100.0 μmol/L5-azaC正常CK干旱Drought(DS)干旱+50.0 μmol/L5-氮杂胞苷DS+50.0 μmol/L5-azaC干旱+100.0 μmol/L 5-氮杂胞苷DS+100.0 μmol/L5-azaC株高/cm Plant height64.59±2.42a42.84±2.00b43.59±3.42b41.13±2.14bc63.07±2.19a45.08±2.54b45.07±3.19b39.73±1.94cd单株穗数Panicles per plant7.07±1.22a3.33±1.54c4.01±1.12c3.63±1.22c5.40±2.10b3.79±1.67c3.55±2.19c3.61±1.61c穗长/cm Panicle length10.12±0.99b10.06±1.47b10.14±0.91b9.13±0.88bc12.51±1.73a11.75±0.69a11.81±1.53a8.55±1.13c穗粒数Grains per panicle22.75±3.59b17.54±3.57c15.75±2.59c14.72±2.18c29.70±3.43a17.70±2.95c18.70±2.43c17.19±3.94c穗实粒数Filled grains per panicle18.47±2.43a13.94±3.29b14.17±2.43b12.68±2.74bc20.04±3.74a10.47±2.46c11.04±2.74c11.33±3.54bc结实率/% Kernels rate81.19±2.20b77.87±2.70b76.80±3.20b86.24±3.10a66.60±3.90c57.87±3.16d57.60±2.90d65.80±3.54c千粒质量/g 1000-kernel weight23.71±0.13a21.57±0.43b22.71±0.23b22.56±0.16ab23.75±0.11a21.82±1.38b21.75±0.21b21.51±0.80b单株产量/g Yield per plant2.95±0.42a1.27±0.11c1.24±0.12c1.26±0.07c2.53±0.19b1.03±0.08d0.99±0.09d0.88±0.09e
3 讨论
DNA甲基化可通过调控植物生长发育相关基因的表达,从而影响植物生长发育过程[27]。水稻在胚胎发育、种子萌发和苗的生长等不同的生育期均具有不同的DNA甲基化动态变化[28]。水稻不同组织的DNA甲基化水平也有差异,如水稻胚乳中全基因组DNA 甲基化程度比胚中的低,主要表现为胚乳中储存蛋白和淀粉合成相关基因的甲基化水平较低[29]。栽培稻辽粳944、药用野生稻以及它们杂交后代的叶片和小穗DNA甲基化水平出现相同的趋势,且随着生长发育的进行,DNA甲基化水平均呈下降趋势,杂种的总甲基化水平高于双亲[30]。本研究进一步通过DNA甲基化抑制剂(5-azaC)联合干旱处理观察到,在不同生育期,5-azaC对水稻干旱胁迫作用浓度不同,呈现芽期<苗期,且在同一生育期,表现为低浓度缓解干旱的抑制,而高浓度则加重干旱的抑制;本研究设置的5-azaC作用浓度对水稻生育后期效应不显著,高低浓度的5-azaC均显著降低了干旱胁迫下水稻株高、剑叶的净光合速率和单株产量,呈负效应,其中5-azaC 作用于PC浓度均高于WT,显示其具有较高的DNA甲基化水平。
逆境胁迫常常会引发植物DNA甲基化状态的改变,这种改变会启动植物体内与逆境相关基因的表达以及某些转座子的转座,从而使植物能够应对外界不良环境[31]。水稻在干旱胁迫下DNA甲基化动态水平因品种、年份以及组织而异,而且部分基因的甲基化位点可以稳定遗传后代。如以中旱3号、汕优63和矮仔占为材料,在10% PEG6000模拟干旱处理下研究其DNA甲基化动态变化表明,不同水稻品种的DNA甲基化程度存在品种间差异,其中中旱3号和汕优63叶片中活性甲基循环过程受干旱胁迫诱导增加(甲基转移酶基因的表达增加),而矮仔占叶片中这一过程受到干旱胁迫的抑制(抑制了DNA甲基转移酶、组蛋白甲基转移酶基因的表达)[32]。在干旱胁迫处理下,降低水稻叶和根的甲基化程度有利于激活基因的表达,从而提高水稻耐旱性[33]。籼稻多基因聚合系DK151及其轮回亲本IR64进行了连续3个季节的DNA甲基化水平测定,结果表明:干旱胁迫可以使水稻DNA甲基化发生变化,启动抗旱相关基因的表达,使水稻抗旱性增强,这些干旱胁迫诱导的甲基化变化中约有四分之一的去甲基化位点在复水后不能恢复到原始状态,而甲基化位点中约有一半不能恢复,可以遗传给下一代[34]。在真核细胞中,5-氮杂胞苷(5-azaC)是一种抑制DNA甲基转移酶活性到基因组DNA低甲基化的类似物[35]。本研究结果表明:在干旱条件下,降低了水稻苗期叶片的甲基化酶基因的表达,低浓度的5-azaC施用继续降低了内源甲基化酶基因的表达,但是高浓度的施用则进一步降低了水稻苗期叶片的保水能力,显著低于单独干旱处理下叶片的相对含水量,提示该浓度可能严重干扰了水稻内源DNA甲基化的动态平衡。需要注意的是这种浓度的调节效应对水稻生育后期影响不大,表明水稻DNA甲基化水平调节很复杂,短期的干旱响应与长期的干旱耐性的作用也不同。5-azaC在芽期和苗期的干旱胁迫调节效果存在品种间差异,耐旱性较强的PC具有较高的5-azaC增益作用浓度。
磷酸烯醇式丙酮酸羧化酶是C4植物CO2固定的第一个关键酶,在![]() 和Mg2+存在下,催化磷酸烯醇丙酮酸(Phosphoenolpyruvate,PEP)发生不可逆β-羧化反应生成草酰乙酸(Oxaloacetic acid,OAA)和无机磷酸[36],对增强植物光合作用起重要作用。有研究发现,光合作用相关基因的DNA甲基水平和模式在水稻籼和粳亚种中存在差异[13]。科学家通过基因工程获得的高表达转玉米C4型PEPC水稻(以下简称PC)[14]也具有较高的光合能力和籽粒产量,并在高温、干旱、以及低氮下中表现出较好的抗性及耐性等[37-39] 。已观察到PC的 C4-PEPC的启动子在干旱条件下也存在去甲基化现象,促进转基因水稻植株对快速响应干旱[7]。本研究观察到,不同浓度的5-azaC处理,对PC水稻芽期和苗期不同干旱表现,提示了其总的DNA水平也与WT的不同,而且叶片C4-PEPC表达也具有阈值效应,低浓度促进,高浓度抑制,与水稻苗期叶片的相对含水量表现一致,提示了外源C4-PEPC导入可能改变了水稻对5-azaC 的阈值范围,并观察到它的干旱表现差异并与其内源糖信号相关基因的表达同步。
和Mg2+存在下,催化磷酸烯醇丙酮酸(Phosphoenolpyruvate,PEP)发生不可逆β-羧化反应生成草酰乙酸(Oxaloacetic acid,OAA)和无机磷酸[36],对增强植物光合作用起重要作用。有研究发现,光合作用相关基因的DNA甲基水平和模式在水稻籼和粳亚种中存在差异[13]。科学家通过基因工程获得的高表达转玉米C4型PEPC水稻(以下简称PC)[14]也具有较高的光合能力和籽粒产量,并在高温、干旱、以及低氮下中表现出较好的抗性及耐性等[37-39] 。已观察到PC的 C4-PEPC的启动子在干旱条件下也存在去甲基化现象,促进转基因水稻植株对快速响应干旱[7]。本研究观察到,不同浓度的5-azaC处理,对PC水稻芽期和苗期不同干旱表现,提示了其总的DNA水平也与WT的不同,而且叶片C4-PEPC表达也具有阈值效应,低浓度促进,高浓度抑制,与水稻苗期叶片的相对含水量表现一致,提示了外源C4-PEPC导入可能改变了水稻对5-azaC 的阈值范围,并观察到它的干旱表现差异并与其内源糖信号相关基因的表达同步。
糖作为信号分子参与了植物对非生物胁迫的过程,其中SnRK作为联系糖和非生物逆境的枢纽而发挥重要的作用[40]。在植物SnRK家族基因根据结构不同可以分为3个亚家族,分别为SnRK1、SnRK2和SnRK3[22]。干旱条件下,PC水稻叶片中可溶性糖积累增强了其糖代谢和转运[41],并观察到SnRK1[7]、SnRK2[38]和SnRK3[42]的参与,有利于其耐旱性的增强。本研究结果看出,与DS处理相比,50 μmol/L 5-azaC+DS处理使PC的OsMETIb表达显著增加,而且也伴随着其内蔗糖、果糖含量和OSK24表达的显著变化,其中蔗糖和果糖含量增加,OSK24表达显著下降,而WT呈相反的变化趋势,且与PC呈显著差异,提示PC内源可能通过糖信号的差异参与调节DNA甲基化对干旱的响应,低浓度5-azaC处理诱导PC的DNA甲基化增强,但是当5-azaC浓度提高到100 μmol/L,则均显著抑制了两材料的OsMETI基因的表达以及其他内源组分差异,加剧干旱胁迫,且两材料的耐旱表现差异均消除。植物DNA的甲基化和与之拮抗的去甲基化,共同决定了基因组总的甲基化水平及其分布模式,对维持正常的基因转录和基因组稳定性至关重要。而植物中主动去甲基化是通过一个双功能糖基化酶和裂解酶直接将甲基化胞嘧啶从DNA上碱基切除修复来实现的[43]。需要关注的是植物中DNA甲基化过程是可逆的,会因高代而恢复逆境处理前水平。研究表明:去甲基化酶基因ROS1敲除导致子一代整体DNA甲基化的平衡破坏,并出现异常表型,如矮化和叶片变小,但该材料种植到第4代,植株总的DNA甲基化水平又可恢复到未处理水平,提示植物可能存在某种机制修复维持相对稳定的DNA甲基化水平,从而有利于植物基因组和染色体状态的稳定[44]。本研究也观察到无论是低浓度还是高浓度5-azaC均对生育后期水稻产生了不良影响,如矮化、净光合速率下降和单株产量下降等,但对PC的影响似乎小于WT。PC对低浓度DNA甲基化抑制处理有部分耐受性,提示其内源的DNA甲基化水平较高或者存在某种机制维持其体内总DNA甲基化水平稳定。PC糖信号表达差异是否参与水稻ROS1调节,有利于维持其基因组的稳定性,值得深入研究。
低甲基化/去甲基化和诱变试剂5-azaC主要是通过抑制DNA甲基转移酶活性防止DNA甲基化[35],已在不同的植物中应用,但是因物种、剂量、时间和过程等导致了对植物发育的不同影响[45-52],以往研究多用于处理离体细胞[48-49]、种子[28,37,50]或者植株发育早期如小孢子[51]以及愈伤组织[47,52]等,并可建立高频的双单倍体技术[51]、抗病[47,52]、抗除草剂[47]等新资源。本研究进一步研究了其在模拟干旱条件下水稻芽期和苗期的效应,观察到低浓度促进,而高浓度抑制,而且存在品种差异。但是,本研究进一步试图将促进水稻芽期和苗期干旱胁迫缓解的5-azaC浓度处理生育后期水稻,期望获得干旱缓解的外源调节物质,但是在生育后期均是负效应,由此可知,水稻DNA甲基化水平的调节很复杂,未来要实现人工调节,还需要今后更多的浓度和更多的品种进行探索。无论如何,DNA甲基化作为表观遗传标记提供了一种新的变异来源,通过调节其水平增强非生物逆境的潜力已经显现,相关研究将为科学家和育种者从提高作物抗性和减少产量损失方面带来新的研究方向。
综上,DNA甲基化参与了水稻干旱响应,但不同生育期表现不同。其中糖信号在调节DNA甲基化机制以增强PC水稻干旱耐性中起重要作用。
[1] Yusuff O,Rafii M Y,Chukwu S C,Fatai A,Usman M G,Isiaka K,Kamarudin Z F,Muhammad I,Kolapo K. Drought resistance in rice from conventional to molecular breeding:a review[J].International Journal of Molecular Sciences,2019,20(14):3519.doi:10.3390/ijms20143519.
[2] Bailey-Serres J,Parker J E,Ainsworth E A,Oldroyd G E D,Schroeder J I. Genetic strategies for improving crop yields[J]. Nature,2019,575(7781):109-118.doi:10.1038/s41586-019-1679-0.
[3] Ermakova M,Danila F R,Furbank R T,von Caemmerer S. On the road to C4 rice:advances and perspectives[J]. The Plant Journal,2020,101(4):940-950.doi:10.1111/tpj.14562.
[4] Izui K,Matsumura H,Furumoto T,Kai Y. Phosphoenolpyruvate carboxylase:a new era of structural biology[J]. Annual Review of Plant Biology,2004,55:69-84.doi:10.1146/annurev.arplant.55.031903.141619.
[5] Liu X L,Li X,Dai C C,Zhou J Y,Yan T,Zhang J F.Improved short-term drought response of transgenic rice over-expressing maize C4 phosphoenolpyruvate carboxylase via calcium signal cascade[J]. Journal of Plant Physiology,2017,218:206-221.doi:10.1016/j.jplph.2017.08.005.
[6] Liu X L,Li X,Zhang C,Dai C C,Zhou J Y,Ren C G,Zhang J F. Phosphoenolpyruvate carboxylase regulation in C4-PEPC-expressing transgenic rice during early responses to drought stress[J].Physiologia Plantarum,2017,159(2):178-200. doi:10.1111/ppl.12506.
[7] Huo K,Li X,He Y F,Wei X D,Lu W,Zhao C F,Wang C L. Exogenous ATP enhance signal response of suspension cells of transgenic rice(Oryza sativa L.)expressing maize C4-pepc encoded phosphoenolpyruvate carboxylase under PEG treatment[J].Plant Growth Regulation,2017,82(1):55-67.doi:10.1007/s10725-016-0238-z.
[8] 唐玉婷,李霞,陆巍,魏晓东.高表达转C4型PEPC 基因水稻在低氮下诱导碳氮酶稳定光合作用[J]. 华北农学报,2015,30(4):95-100.doi:10.7668/hbnxb.2015.04.017.
Tang Y T,Li X,Lu W,Wei X D. Transgenic rice with high expression of C4-PEPC genes induced higher carbon and nitrogen key enzyme to maintain photosynthesis under low nitrogen conditions[J].Acta Agriculturae Boreali-Sinica,2015,30(4):95-100.
[9] 严婷,李佳馨,李霞,谢寅峰. 转C4型PEPC 基因水稻非生物胁迫耐受性研究进展[J].淮阴工学院学报,2019,28(5):62-68.doi:10.3969/j.issn.1009-7961.2019.05.012.
Yan T,Li J X,Li X,Xie Y F. Research progress on abiotic stress tolerance of transgenic rice with C4-PEPC gene[J]. Journal of Huaiyin Institute of Technology,2019,28(5):62-68.
[10] 杜康兮,沈文辉,董爱武. 表观遗传调控植物响应非生物胁迫的研究进展[J]. 植物学报,2018,53(5):581-593.doi:10.11983/CBB17143.
Du K X,Shen W H,Dong A W. Advances in epigenetic regulation of abiotic stress response in plants[J].Bulletin of Botany,2018,53(5):581-593.
[11] Karim R,Nuruzzaman M,Khalid N,Harikrishna J A. Importance of DNA and histone methylation in in vitro plant propagation for crop improvement:a review[J]. Annals of Applied Biology,2016,169(1):1-16.doi:10.1111/aab.12280.
[12] Li X Y,Wang X F,He K,Ma Y Q,Su N,He H,Stolc V,Tongprasit W,Jin W W,Jiang J M,Terzaghi W,Li S G,Deng X W. High-resolution mapping of epigenetic modifications of the rice genome uncovers interplay between DNA methylation,histone methylation,and gene expression[J]. The Plant Cell,2008,20(2):259-276.doi:10.1105/tpc.107.056879.
[13] Zhang Q,Liang Z,Cui X A,Ji C M,Li Y,Zhang P X,Liu J R,Riaz A,Yao P,Liu M,Wang Y P,Lu T G,Yu H,Yang D L,Zheng H K,Gu X F.N6-Methyladenine DNA methylation in Japonica and Indica rice genomes,and its association with gene expression,plant development and stress responses[J]. Molecular Plant,2018,11(12):1492-1508.doi:10.1016/j.molp.2018.11.005.
[14] Ku M S,Agarie S,Nomura M,Fukayama H,Tsuchida H,Ono K,Hirose S,Toki S,Miyao M,Matsuoka M. High-level expression of maize phosphoenolpyruvate carboxylase in transgenic rice plants[J]. Nature Biotechnology,1999,17(1):76-80.doi:10.1038/5256.
[15] Yoshida S,Forno D A,Cock J H,Gomez K A. Laboratory manual for physiological studies of rice[M]. Philippines:The International Rice Research Institute,1976.
[16] Li X,Wang C,Ren C G. Effects of 1-butanol,neomycin,and calcium on the photosynthetic characteristics of pepc transgenic rice[J].African Journal of Biotechnology,2011,10(76):17466-17476.doi:10.5897/AJB10.1565.
[17] Smart R E,Bingham G E. Rapid estimates of relative water content[J]. Plant Physiology,1974,53(2):258-260.doi:10.1104/pp.53.2.258.
[18] Velikova V,Yordanov I,Edreva A. Oxidative stress and some antioxidant systems in acid rain-treated bean plants:protective role of exogenous polyamines[J]. Plant Science,2000,151(1):59-66.doi:10.1016/S0168-9452(99)00197-1.
[19] Bradford M M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J]. Analytical Biochemistry,1976,72(1/2):248-254.doi:10.1006/abio.1976.9999.
[20] Troll W,Lindsley J. A photometric method for the determination of proline[J].Journal of Biological Chemistry,1955,215(2):655-660.doi:10.1016/S0021-9258(18)65988-5.
[21] Ambavaram M M R,Basu S,Krishnan A,Ramegowda V,Batlang U,Rahman L,Baisakh N,Pereira A. Coordinated regulation of photosynthesis in rice increases yield and tolerance to environmental stress[J].Nature Communications,2014,5:5302. doi:10.1038/ncomms6302.
[22] 张金飞,李霞,谢寅峰. 植物SnRKs家族在胁迫信号通路中的调节作用[J]. 植物学报,2017,52(3):346-357.doi:10.11983/CBB16095.
Zhang J F,Li X,Xie Y F. The function of sucrose nonfermenting-1 related protein kinases in stress signaling[J].Bulletin of Botany,2017,52 (3):346-357.
[23] Horacio P,Martinez-Noel G. Sucrose signaling in plants:A world yet to be explored[J]. Plant Signaling & Behavior,2013,8(3):e23316.doi:10.4161/psb.23316.
[24] Lindroth A M,Cao X F,Jackson J P,Zilberman D,McCallum C M,Henikoff S,Jacobsen S E. Requirement of CHROMOMETHYLASE3 for maintenance of CpXpG methylation[J].Science,2001,292(5524):2077-2080.doi:10.1126/science.1059745.
[25] Yamauchi T,Johzuka-Hisatomi Y,Fukada-Tanaka S,Terada R,Nakamura I,Iida S. Homologous recombination-mediated knock-in targeting of the MET1a gene for a maintenance DNA methyltransferase reproducibly reveals dosage-dependent spatiotemporal gene expression in rice[J]. The Plant Journal,2009,60(2):386-396.doi:10.1111/j.1365-313X.2009.03947.x.
[26] Yamauchi T,Moritoh S,Johzuka-Hisatomi Y,Ono A,Terada R,Nakamura I,Iida S. Alternative splicing of the rice OsMET1 genes encoding maintenance DNA methyltransferase[J].Journal of Plant Physiology,2008,165(17):1774-1782.doi:10.1016/j.jplph.2007.12.003.
[27] 袁超,张少伟,牛义,汤青林,魏大勇,王志敏. 植物DNA 甲基化作用机制的研究进展[J]. 生物工程学报,2020,36(5):838-848.doi:10.13345/j.cjb.190373.
Yuan C,Zhang S W,Niu Y,Tang Q L,Wei D Y,Wang Z M. Advances in research on the mechanism of DNA methylation in plants[J]. Chinese Journal of Biotechnology,2020,36(5):838-848.
[28] Akimoto K,Katakami H,Kim H J,Ogawa E,Sano C M,Wada Y,Sano H. Epigenetic inheritance in rice plants[J]. Annals of Botany,2007,100(2):205-217.doi:10.1093/aob/mcm110.
[29] Zemach A,Kim M Y,Silva P,Rodrigues J A,Dotson B,Brooks M D,Zilberman D. Local DNA hypomethylation activates genes in rice endosperm[J]. Proceedings of the National Academy of Sciences of the United States of America,2010,107(43):18729-18734.doi:10.1073/pnas.1009695107.
[30] 何平,疏冕,蔡晓丹,傅雪琳.栽培稻×药用野生稻种间杂种基因组DNA甲基化的遗传与变异研究[J].华北农学报,2017,32(4):19-31.doi:10.7668/hbnxb.2017.04.004.
He P,Shu M,Cai X D,Fu X L. Inheritance and variations of DNA methylation in interspecific hybrid F1 between O.sativa and O.officinalis[J]. Acta Agriculturae Boreali-Sinica,2017,32(4):19-31.
[31] Zhang H M,Lang Z B,Zhu J K. Dynamics and function of DNA methylation in plants[J]. Nature Reviews Molecular Cell Biology,2018,19(8):489-506.doi:10.1038/s41580-018-0016-z.
[32] 张小丽,刘敏,商奇,葛才林.水稻叶片中活性甲基循环、转移相关基因对干旱胁迫的应答[J]. 中国水稻科学,2011,25(3):236-242.doi:10.3969/j.issn.1001-7216.2011.03.002.
Zhang X L,Liu M,Shang Q,Ge C L. Responses of active methyl cycle and transfer genes to drought stress in rice leaves[J]. Chinese Journal of Rice Science,2011,25(3):236-242.
[33] 潘雅姣,傅彬英,王迪,朱苓华,黎志康. 水稻干旱胁迫诱导 DNA 甲基化时空变化特征分析[J]. 中国农业科学,2009,42(9):3009-3018.doi:10.3864/j.issn.0578-1752.2009.09.001.
Pan Y J,Fu B Y,Wang D,Zhu L H,Li Z K. Spatial and temporal profiling of DNA methylation induced by drought stress in rice[J].Scientia Agricultura Sinica,2009,42(9):3009-3018.
[34] Wang W S,Pan Y J,Zhao X Q,Dwivedi D,Zhu L H,Ali J,Fu B Y, Li Z K. Drought-induced site-specific DNA methylation and its association with drought tolerance in rice (Oryza sativa L.)[J].Journal of Experimental Botany,2011,62(6):1951-1960.doi:10.1093/jxb/erq391.
[35] Friedman S.The inhibition of DNA (cytosine-5) methylases by 5-azacytidine.The effect of azacytosine-containing DNA[J]. Molecular Pharmacology,1981,19(2):314-320.
[36] O′Leary B,Park J,Plaxton W C. The remarkable diversity of plant PEPC (phosphoenolpyruvate carboxylase): recent insights into the physiological functions and post-translational controls of non-photosynthetic PEPCs[J]. The Biochemical Journal,2011,436(1):15-34.doi:10.1042/bj20110078.
[37] 宋凝曦,谢寅峰,李霞. 干旱胁迫下表观遗传机制对转C4型PEPC基因水稻种子萌发的影响[J]. 植物学报,2020,55(6):677-692. doi:10.11983/CBB20048.
Song N X,Xie Y F,Li X. Effects of epigenetic mechanisms on C4 phosphoenolpyruvate carboxylase transgenic rice (Oryza sativa) seed germination under drought stress[J]. Chinese Bulletin of Botany,2020,55(6):677-692.
[38] He Y F,Xie Y F,Li X,Yang J. Drought tolerance of transgenic rice overexpressing maize C4-PEPC gene related to increased anthocyanin synthesis regulated by sucrose and calcium[J]. Biologia Plantarum,2020,64:136-149.doi:10.32615/bp.2020.031.
[39] Tang Y T,Li X,Lu W,Wei X D,Zhang Q J,Lü C G,Song N X. Enhanced photorespiration in transgenic rice over-expressing maize C4 phosphoenolpyruvate carboxylase gene contributes to alleviating low nitrogen stress[J]. Plant Physiology and Biochemistry,2018,130:577-588. doi:10.1016/j.plaphy.2018.08.013.
[40] 何亚飞,李霞,谢寅峰.植物中糖信号及其对逆境调控的研究进展[J].植物生理学报,2016,52(3):241-249.doi:10.13592/j.cnki.ppj.2015.0554.
He Y F,Li X,Xie Y F. Research progress in sugar signal and its regulation of stress in plants[J].Plant Physiology Journal,2016,52(3):241-249.
[41] Zhang C,Li X,He Y F,Zhang J F,Yan T,Liu X L. Physiological investigation of C4-phosphoenolpyruvate-carboxylase-introduced rice line shows that sucrose metabolism is involved in the improved drought tolerance[J]. Plant Physiology and Biochemistry,2017,115:328-342.doi:10.1016/j.plaphy.2017.03.019.
[42] 张金飞,李霞,何亚飞,谢寅峰. 外源葡萄糖增强高表达转玉米C4型PEPC水稻耐旱性的生理机制[J]. 作物学报,2018,44(1):82-94.doi:10.3724/SP.J.1006.2018.00082.
Zhang J F,Li X,He Y F,Xie Y F. Physiological mechanism on drought tolerance enhanced by exogenous glucose in C4-PEPC rice[J]. Acta Agronomica Sinica,2018,44(1):82-94.
[43] Morales-Ruiz T,Ortega-Galisteo A P,Ponferrada-Marín M I,Martínez-Macías M I,Ariza R R,Rold n-Arjona T. DEMETER and REPRESSOR OF SILENCING 1 encode 5-methylcytosine DNA glycosylases[J]. Proceedings of the National Academy of Sciences of the United States of America,2006,103(18):6853-6858. doi:10.1073/pnas.0601109103.
n-Arjona T. DEMETER and REPRESSOR OF SILENCING 1 encode 5-methylcytosine DNA glycosylases[J]. Proceedings of the National Academy of Sciences of the United States of America,2006,103(18):6853-6858. doi:10.1073/pnas.0601109103.
[44] Williams B P,Gehring M. Stable transgenerational epigenetic inheritance requires a DNA methylation-sensing circuit[J].Nature Communications,2017,8(1):2124-2132.doi:10.1038/s41467-017-02219-3.
[45] Morales-Ruiz T, Ortega-Galisteo A P, Ponferrada-Marin M I, Martínez-Macías M I, Ariza R R, Roldán-Arjona T. DEMETER and REPRESSOR OF SILENCING 1 encode 5-methylcytosine DNA glycosylases[J].Proceedings of the National Academy of Sciences of the United States of America,2006,103(18):6853-6858,doi:10.1073/pnas.0601109103.
[46] 石鹏,王永,金龙飞,张大鹏,赵志浩,曹红星,雷新涛. 植物组织培养过程中的 DNA 甲基化研究进展[J].热带作物学报,2019,40(1):199-207.doi:10.3969/j.issn.1000-2561.2019.01.029.
Shi P,Wang Y,Jin L F,Zhang D P,Zhao Z H,Cao H X,Lei X T. Research progress on DNA methylation during plant tissue culture[J]. Chinese Journal of Tropical Crops,2019,40(1):199-207.
[47] Tyunin A P,Kiselev K V,Zhuravlev Y N. Effects of 5-azacytidine induced DNA demethylation on methyltransferase gene expression and resveratrol production in cell cultures of Vitis amurensis[J]. Plant Cell,Tissue and Organ Culture,2012,111(1):91-100.doi:10.1007/s11240-012-0175-0.
[48] Yamamoto N,Kobayashi H,Togashi T,Mori Y,Kikuchi K,Kuriyama K,Tokuji Y. Formation of embryogenic cell clumps from carrot epidermal cells is suppressed by 5-azacytidine,a DNA methylation inhibitor[J]. Journal of Plant Physiology,2005,162(1):47-54.doi:10.1016/j.jplph.2004.05.013.
[49] Castilho A,Neves N,Rufini-Castiglione M,Viegas W,Heslop-Harrison J S.5-methylcytosine distribution and genome organization in triticale before and after treatment with 5-azacytidine[J].Journal of Cell Science,1999,112(23):4397-4404.doi:10.1242/jcs.112.23.4397.
[50] 侯泽豪,杨飞,商水根,方汉顺,张文英,徐延浩. 丁酸钠和5-氮杂胞苷对大麦、水稻、玉米、小麦种子萌发及芽苗期生长的影响[J].江苏农业科学,2017,45(22):73-77. doi:10.15889/j.issn.1002-1302.2017.22.019.
Hou Z H,Yang F,Shang S G,Fang H S,Zhang W Y,Xu Y H. Sodium butyrate and 5-azacytidine on seed germination and growth in seedling stage of barley,rice,maize and wheat[J]. Jiangsu Agricultural Sciences,2017,45(22):73-77.
[51] Solís M T,El-Tantawy A A,Cano ![]() M C,Testillano P S.5-azacytidine promotes microspore embryogenesis initiation by decreasing global DNA methylation,but prevents subsequent embryo development in rapeseed and barley[J]. Frontiers in Plant Science,2015,6:472.doi:10.3389/fpls.2015.00472.
M C,Testillano P S.5-azacytidine promotes microspore embryogenesis initiation by decreasing global DNA methylation,but prevents subsequent embryo development in rapeseed and barley[J]. Frontiers in Plant Science,2015,6:472.doi:10.3389/fpls.2015.00472.
[52] Munsamy A,Rutherford R S,Snyman S J,Watt M P. 5-Azacytidine as a tool to induce somaclonal variants with useful traits in sugarcane (Saccharum spp.)[J].Plant Biotechnology Reports,2013,7(4):489-502.doi:10.1007/s11816-013-0287-y.