植物中有些RNA分子可以通过胞间连丝或植物韧皮部进行长距离移动。在外界环境刺激下,这些可移动的RNA在源(Source)中转录,然后通过韧皮部运输到库(Sink)中并发挥作用[1-4]。目前发现3类可移动的RNA分子存在于韧皮液内,外源类病毒或RNA病毒,植物内源mRNA以及非编码小RNA[5]。植物病毒侵入寄主细胞后,通过胞间连丝和韧皮部筛管分别实现细胞间转运和长距离转运,从而对植物系统实现侵染[6]。病毒RNA是最早被证实可以在植物韧皮部进行长距离运输的RNA,其在韧皮液的运输机制为研究植物内源mRNA提供了重要参考。通过对植物韧皮液转录组分析发现了数千种mRNA,它们所编码的蛋白质参与信号转导等功能[7-8]。然而并非所有韧皮液中发现的RNA均进行长距离移动。目前已在嫁接植物体中发现了些许可移动mRNA,研究表明,这些长距离运输的mRNA可以作为信号分子对植物的生长发育进行调控[9-12]。最近的一项研究通过大豆和菜豆的异缘嫁接试验证明,地上部产生的很大一部分小RNA被运输到根部,然而mRNA在地上部和根部之间的移动非常有限,推测小RNA才是远程通信的“信使”[13]。尽管越来越多的试验表明,可移动RNA分子可作为活跃的信号分子在细胞间转移,但目前对可移动RNA运输的机制了解有限。因此,移动的RNA是如何转移到其他细胞当中还仍待研究。
在RNA实时成像系统中,利用荧光标记的RNA结合蛋白(RNA-binding proteins,RBPs)进行间接RNA标记[14-15]。利用GFP融合MS2噬菌体外壳蛋白(MS2-GFP),将目标RNA融合到MS2识别RNA茎环(Stem loop,SL)的串联重复序列上,MS2-GFP与RNA-SL之间的相互作用可以使活细胞中的RNA可视化。利用来自猿猴病毒40(Simian virus 40,SV40)的核定位信号(Nuclear localization signal,NLS)将MS2-GFP限制在细胞核上,减少了来自MS2-GFP的胞质荧光背景,当MS2SV40-GFP与含有SL的RNA共表达时,MS2SV40-GFP与其RNA识别位点相连,RNA-蛋白复合物被保留在细胞质中从而形成荧光。该系统已成功应用于酵母、果蝇、哺乳动物和植物中mRNA的可视化[16-21]。然而来自SV40的NLS无法有效地将小的GFP融合蛋白限制在细胞核上,导致少量MS2SV40-GFP蛋白在细胞质中积累[22-23]。然而该系统在植物中使用时通常会在细胞质中产生大量的荧光背景,极大地干扰了RNA信号的检测。后来对MS2系统进行改进,用一种特异定位于拟南芥顶端分生组织细胞核的转录因子FD替代SV40作为核定位信号,大大减少了细胞质荧光背景[24-26]。本研究构建了含有RNA茎环串联重复结构的表达载体pSL24UbiK,基于MS2FD-GFP的RNA标记系统,噬菌体外壳蛋白MS2与RNA茎环串联重复结构识别,共聚焦显微镜观察GFP信号的亚细胞定位情况,可以鉴定RNA的可移动性,为今后可移动RNA的研究奠定了基础。
1 材料和方法
1.1 试验材料
野生型拟南芥(Col-0)和野生型本生烟草(Nicotiana benthamiana)均由长江大学农学院湿地作物微嫁接实验室提供,大肠杆菌DH5α、根癌农杆菌GV3101由本实验室保存。质粒p1390-35S-MS2FD-GFP、p1390-35S-FT-SL24由“台湾”中央研究院植微所余天心博士提供,限制性内切酶Mfe Ⅰ、EcoRⅠ购于 New England Biolabs公司。本研究所用引物均由生工生物工程股份有限公司合成。
1.2 试验方法
1.2.1 pSL24UbiK表达载体的构建 利用引物SL24F1:TGATTAACAGCTCGCGGGCCCAAGCCCTAGGTAATACGACTCACTATAGG和SL24R1:CTTACTCAGTTAGGTCAATTGCACACTCGAGAATTAACCCTCACTAAAGG 从pCR4-24xMS2SL-stable质粒中扩增出含有SL24序列的片段,将该片段连入双元载体pUbiK,转化 DH5α感受态细胞,挑取单克隆菌株扩繁,提取质粒,经酶切鉴定,并在测序结果比对正确后,最终得到pSL24UbiK载体。
1.2.2 RNA提取及cDNA合成 取幼嫩的野生型拟南芥叶片剪碎放入液氮预冷的研钵中,加入适量液氮,充分研磨后采用TRIzol法提取RNA,RNA的浓度通过Nano Drop 2000(Thermo)测定,质量和完整性通过1%琼脂糖凝胶电泳进行检测。用反转录试剂盒进行cDNA第一链的合成。
1.2.3 基因克隆 提取拟南芥DNA,以DNA为模板,对GUS基因进行PCR扩增;以上述合成的cDNA作为模板,对FT(FLOWER ING LOCUS T)基因进行CDS 全长序列的扩增(表1)。PCR反应程序:95 ℃预变性3 min;95 ℃变性15 s,59 ℃/ 60 ℃退火15 s,72 ℃延伸30 s,35个循环;72 ℃总延伸5 min,4 ℃保存。用1%琼脂糖凝胶电泳对PCR产物进行检验,然后将PCR产物用纯化试剂盒进行回收。pSL24UbiK经限制性内切酶Mfe Ⅰ 37 ℃反应2 h后纯化备用。利用同源重组酶将克隆的2个基因片段分别连接到线性化载体pSL24UbiK上。取5 μL重组产物转化大肠杆菌DH5α感受态细胞,挑取单克隆菌株扩繁,提取质粒,经酶切(EcoR Ⅰ)鉴定,由公司测序。在测序结果比对正确后,最终得到pSL24UbiK-GUS和pSL24UbiK-FT载体。将构建好的质粒转化农杆菌 GV3101。
表1 本研究中用到的引物
Tab.1 Primer sequence used in this research

引物名称Primer name引物序列(5′-3′)Primer sequenceGUS-FTAATTCTCGAGTGTGCAATTGGTGTGAACAACGAACTGAACTGGCGUS-RCTTACTCAGTTAGGTCACTGCCACTGACCGGATGCFT-FAATTCTCGAGTGTGCACAAATTAAAGAAGCAGAAACFT-RTTACTCAGTTAGGTCAATTGATAGGCATCATCACCGTTC
1.2.4 烟草叶片的瞬时表达及共聚焦激光显微镜成像 将含重组质粒的农杆菌500 μL加入至含有抗生素的20 mL LB液体培养基中,28 ℃过夜振荡培养5 000 r/min离心10 min,收集菌体倒掉上清液,用无菌水重悬菌体,使菌液浓度分别达到OD 600 ≈0.3。将pSL24UbiK-FT和p1390-35S-MSFD-GFP、pSL24UbiK-GUS和p1390-35S-MS2FD-GFP、p1390-35S-FT-SL24和p1390-35S-MS2FD-GFP的菌液分别等体积混匀,选取叶龄在35 d左右的本氏烟草,用2 mL注射器(去掉针头)在叶片背部注射菌液直至叶片组织空隙被液体填满,而后将注射后的烟草放置于25 ℃条件下培养,48 h之后用水将侵染过的烟草叶片清洗一遍,从侵染的叶片区域取出约0.5 cm2的样品制片,在Leica激光共聚焦显微镜TCS-SP8下对荧光信号(GFP)进行观察,激发光波长选择488 nm。
2 结果与分析
2.1 FT、GUS基因扩增
使用引物GUS-F/R、FT-F/R,对目的基因进行序列扩增,得到了大小符合、条带清晰且单一的PCR条带,其中GUS基因长度为430 bp;FT的CDS全长为675 bp(图1)。
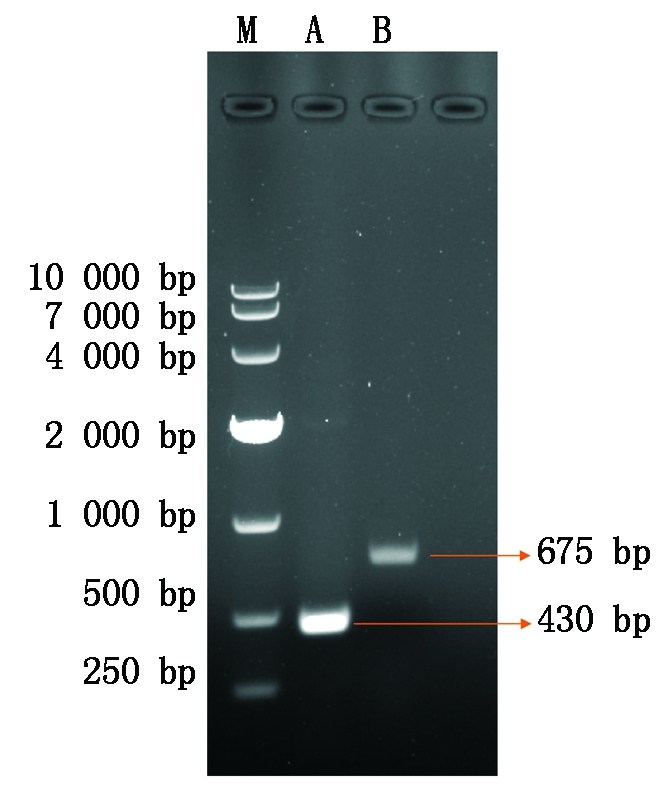
A.GUS;B.FT;M.DL10000 DNA Marker。
图1 GUS、FT基因扩增的琼脂糖凝胶电泳
Fig.1 Agarose gel electrophoresis of GUS and FT gene amplification
2.2 表达载体的构建
重组产物转化DH5α感受态细胞后,在抗性培养基上挑选阳性菌落进行质粒提取,经EcoRⅠ酶切pSL24UbiK、pSL24UbiK-FT、pSL24UbiK-GUS质粒结果如图所示(图2)。条带大小符合预期,初步判定pSL24UbiK、pSL24UbiK-FT、pSL24UbiK-GUS表达载体构建成功。送样测序后,通过序列比对,进一步确认表达载体构建成功。
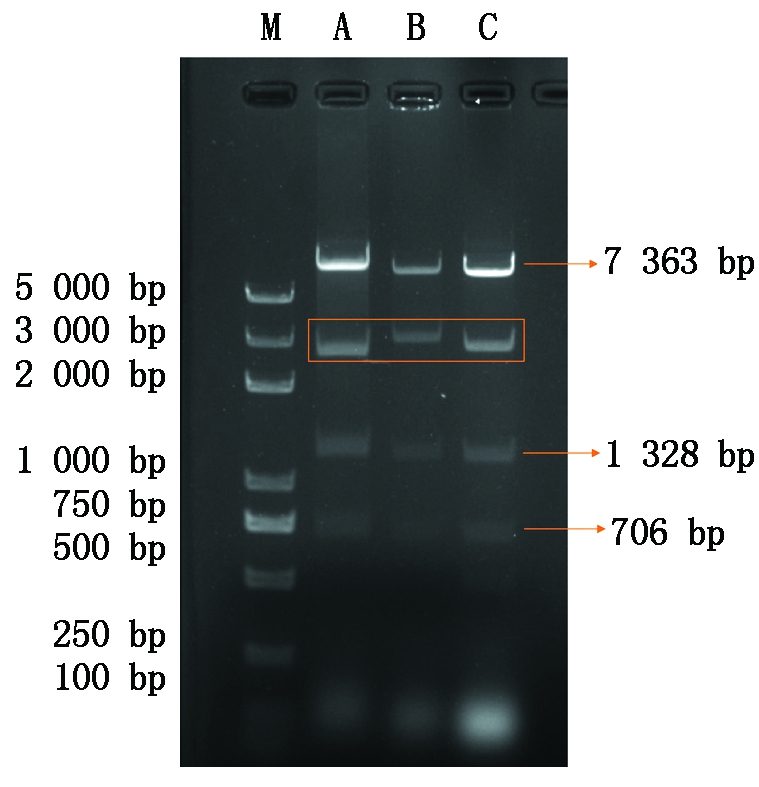
A.pSL24UbiK经EcoR Ⅰ 酶切后产生的7 363,2 606,1 328,706 bp大小的片段; B.pSL24UbiK-FT经EcoR Ⅰ 酶切后产生的7 363,3 281,1 328,706 bp大小的片段; C.pSL24UbiK-GUS经EcoR Ⅰ 酶切后产生的7 363,3 036,1 328,706 bp大小的片段; M.Trans2K Plus DNA Marker。
A.The 7 363,2 606,1 328,706 bp fragments of pSL24UbiK after digestion with EcoR Ⅰ; B.The 7 363,3 281,1 328,706 bp fragments of pSL24UbiK-FT after digestion with EcoR Ⅰ; C.The 7 363,3 036,1 328,706 bp fragments of pSL24UbiK-GUS after digestion with EcoR Ⅰ; M. Trans2K Plus DNA Marker.
图2 pSL24UbiK表达载体的构建
Fig.2 Construction of pSL24UbiK expression vector
2.3 MS2FD-GFP的亚细胞分布
瞬时表达的GFP或MS2-GFP在细胞核和细胞质中均有定位,虽然将SV40 NLS插入到MS2-GFP(MS2SV40-GFP)中大大降低了MS2-GFP的细胞质背景,但仍然可以检测到的MS2-GFP细胞质背景,极大干扰了信号的检测[26]。将改造后的含有p1390-35S-MS2FD-GFP的农杆菌侵染烟草叶片,48 h后用倒置荧光显微镜观察到MS2FD-GFP定位在细胞核中(图3),与MS2SV40-GFP相比大大减少MS2-GFP的细胞质背景影响,为后续试验的验证提供了良好的支撑依据。
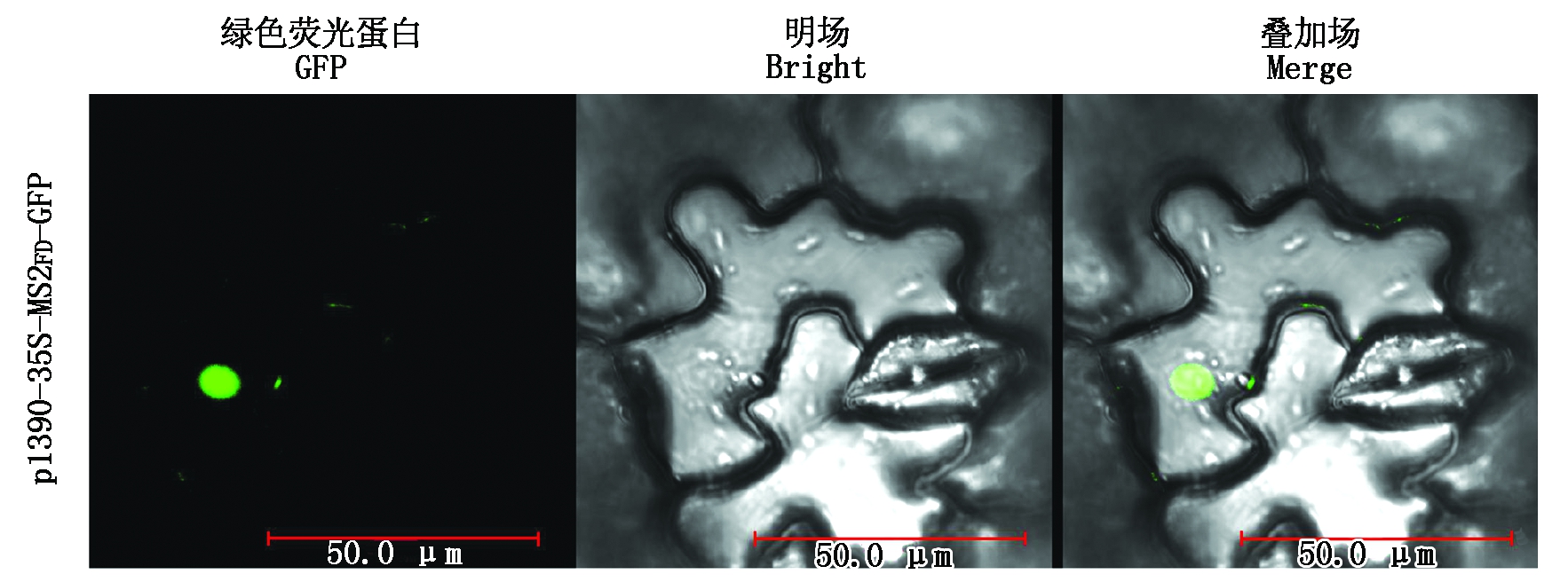
图3 MS2FD-GFP的亚细胞定位
Fig.3 Subcellular localization of MS2FD-GFP
2.4 可移动RNA FT的亚细胞分布
在本氏烟草叶片中,通过农杆菌渗透法将p1390-35S-FT-SL24与p1390-35S-MS2FD-GFP共侵染烟草叶片,MS2FD-GFP可与FT-SL24融合,在细胞核和胞质中均检测到GFP信号,并在细胞质附近显示出点状信号(图4)。与此同时,将成功构建的pSL24UbiK-FT与p1390-35S-MS2FD-GFP共侵染烟草叶片,发现与对照试验中p1390-35S-FT-SL24与p1390-35S-MS2FD-GFP共侵染结果一致(图4)。

图4 植物细胞中可移动RNA的亚细胞分布
Fig.4 Subcellular localization of mobile RNA in plant cells
2.5 不可移动RNA GUS的亚细胞分布
为了解释可移动RNA在细胞质附近检测出的点状信号,将不可移动RNA GUS融合载体pSL24UbiK上,将pSL24UbiK-GUS与p1390-35S-MS2FD-GFP共侵染烟草叶片,48 h后用倒置荧光显微镜观察细胞中 GFP 的表达以及位置。结果发现,不可移动RNA GUS在细胞质附近没有检测到荧光信号,只在细胞核位置有荧光信号的出现(图5)。由此可以看出,通过将RNA融合到pSL24UbiK载体上经过该系统检测,若在细胞质附近检测到荧光信号则说明该RNA是可移动的,反之亦然。
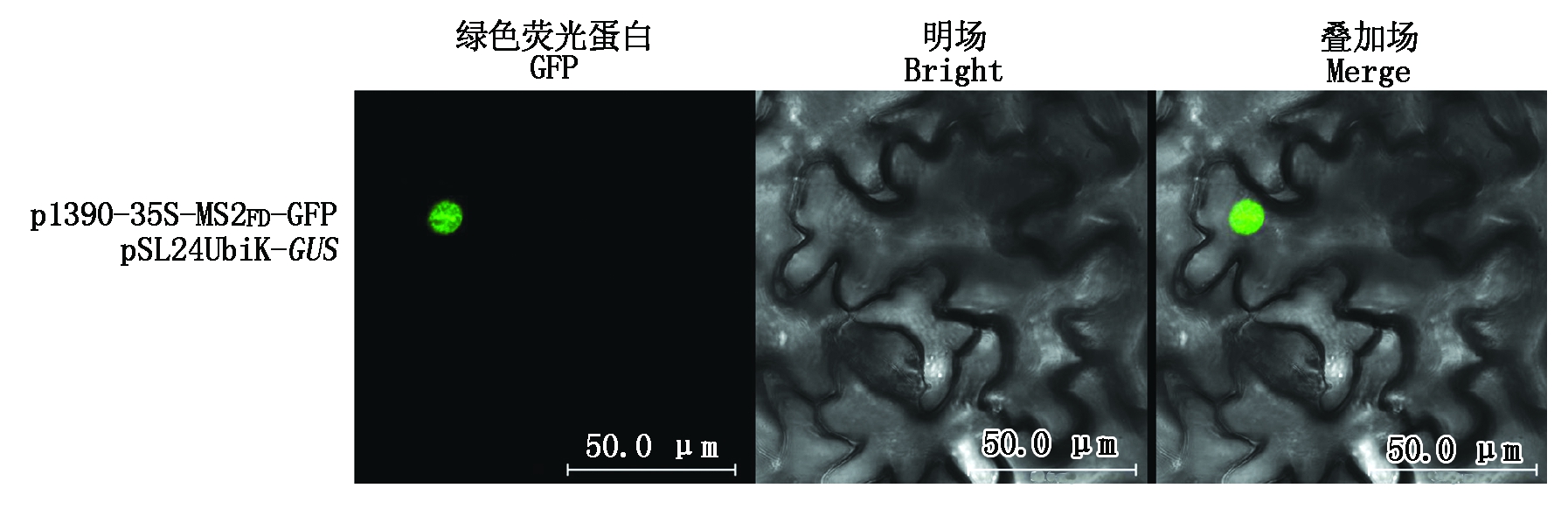
图5 共聚焦显微镜下观察植物细胞中移动RNA和非移动RNA的亚细胞分布差异
Fig.5 The subcellular distribution difference of mobile RNA and non-mobile RNA in plant cells was observed under confocal microscopy
3 结论与讨论
可视化活细胞内RNA的实时运输是了解特定RNA细胞分布最有效的方法。在这些方法中,通过基因编码的荧光RBP进行间接RNA标记使特定组织中的RNA可视化[14-15,27]。尽管许多物种在RNA实时成像方面取得了显著进展,但在植物中的研究相对有限[15,28]。另外,依赖于MS2的RNA可视化系统的缺点之一是需要在目标RNA上多次重复MS2识别的SL结构。检测活植物细胞中的荧光信号至少需要重复24个SL序列,这与哺乳动物细胞的结果一致[29]。在目标RNA上插入大片段的MS2识别SL序列(SL序列的24个重复序列为870 bp)可能会干扰目标RNA的生物学功能。实时RT-qPCR和共聚焦显微镜分析表明,在目标FT RNA上插入SL序列不会极大地干扰RNA的转录和翻译[26]。但是,不排除在其他RNA上插入SL序列导致其功能破坏的可能性。FT RNA在光周期诱导的叶片中转录翻译,并且可以从叶片向茎尖移动,调控光周期敏感植物开花,可被作为典型的可移动的RNA进行研究[30-31]。本研究构建了含有24个茎环串联重复的RNA结构表达载体pSL24UbiK,利用不可移动RNA GUS作为阴性对照,观察了可移动RNA和不可移动RNA在细胞中的定位情况,验证了该方法的可行性,为今后研究可移动RNA的运输奠定了基础。
植物韧皮液内RNA分子长距离移动的发现具有重要意义,可移动RNA作为植物调控模式中的新信号,是植物界研究的新热点。目前,虽然已知植物韧皮液内存在上千种RNA,但是能进行长距离移动的只有小部分被验证,且明确功能的更少,对于其运输调控机制也仅停留在假说阶段。本鉴定方法可以快速验证RNA分子是否可以移动,避免了传统的复杂验证过程,极大地提高了工作效率,为科研人员节省了大量的时间和精力。
[1] Banerjee A K,Chatterjee M,Yu Y,Suh S G,Miller W A,Hannapel D J. Dynamics of a mobile RNA of potato involved in a long-distance signaling pathway[J].The Plant Cell,2006,18(12):3443-3457.doi:10.1105/tpc.106.042473.
[2] Lu K J,Huang N C,Liu Y S,Lu C G,Yu T S. Long-distance movement of Arabidopsis FLOWERING LOCUS T RNA participates in systemic floral regulation[J].RNA Biology,2012,9(5):653-662. doi:10.4161/rna.19965.
[3] Huang N C,Jane W N,Chen J,Yu T S. Arabidopsis thaliana CENTRORADIALIS homologue(ATC)acts systemically to inhibit floral initiation in Arabidopsis[J].The Plant Journal,2012,72(2):175-184. doi:10.1111/j.1365-313x.2012.05076.x.
[4] Lough T J,Lucas W J. Integrative plant biology:Role of phloem long-distance macromolecular trafficking[J].Annual Review of Plant Biology,2006,57:203-232. doi:10.1146/annurev.arplant.56.032604.144145.
[5] Kehr J,Buhtz A. Long distance transport and movement of RNA through the phloem[J].Journal of Experimental Botany,2008,59(1):85-92. doi:10.1093/jxb/erm176.
[6] Jorgensen R A,Atkinson R G,Forster R L S,Lucas W J. An RNA-based information superhighway in plants[J].Science,1998,279(5356):1486-1487. doi:10.1126/science.279.5356.1486.
[7] Gaupels F,Buhtz A,Knauer T,Deshmukh S,Waller F,van Bel A J E,Kogel K H,Kehr J. Adaptation of aphid stylectomy for analyses of proteins and mRNAs in barley phloem Sap[J].Journal of Experimental Botany,2008,59(12):3297-3306. doi:10.1093/jxb/ern181.
[8] Deeken R,Ache P,Kajahn I,Klinkenberg J,Bringmann G,Hedrich R. Identification of Arabidopsis thaliana phloem RNAs provides a search criterion for phloem-based transcripts hidden in complex datasets of microarray experiments[J].The Plant Journal,2008,55(5):746-759. doi:10.1111/j.1365-313X.2008.03555.x.
[9] Guo S G,Zhang J G,Sun H H,Salse J,Lucas W J,Zhang H Y,et al. The draft genome of watermelon(Citrulluslanatus)and resequencing of 20 diverse accessions[J].Nature Genetics,2013,45(1):51-58. doi:10.1038/ng.2470.
[10] Thieme C J,Rojas-Triana M,Stecyk E,Schudoma C,Zhang W N,Yang ![]() M,Walther D,Schulze W X,Paz-Ares J,Scheible W R,Kragler F. Endogenous Arabidopsis messenger RNAs transported to distant tissues[J].Nature Plants,2015,1(4):15025. doi:10.1038/nplants.2015.25.
M,Walther D,Schulze W X,Paz-Ares J,Scheible W R,Kragler F. Endogenous Arabidopsis messenger RNAs transported to distant tissues[J].Nature Plants,2015,1(4):15025. doi:10.1038/nplants.2015.25.
[11] Yang Y Z,Mao L Y,Jittayasothorn Y,Kang Y M,Jiao C,Fei Z J,Zhong G Y. Messenger RNA exchange between scions and rootstocks in grafted grapevines[J].BMC Plant Biology,2015,15(1):251. doi:10.1186/s12870-015-0626-y.
[12] Zhang W N,Thieme C J,Kollwig G,Apelt F,Yang L,Winter N,Andresen N,Walther D,Kragler F. tRNA-related sequences trigger systemic mRNA transport in plants[J].The Plant Cell,2016,28(6):1237-1249. doi:10.1105/tpc.15.01056.
[13] Li S,Wang X T,Xu W Y,Liu T,Cai C M,Chen L Y,Clark C B,Ma J X.Unidirectional movement of small RNAs from shoots to roots in interspecific heterografts[J].Nature Plants,2021,7:50-59. doi:10.1038/s41477-020-00829-2.
[14] Keryer-Bibens C,Barreau C,Osborne H B. Tethering of proteins to RNAs by bacteriophage proteins[J].Biology of the Cell,2008,100(2):125-138. doi:10.1042/BC20070067.
[15] Urbanek M O,Galka-Marciniak P,Olejniczak M,Krzyzosiak W J. RNA imaging in living cells-methods and applications[J].RNA Biology,2014,11(8):1083-1095. doi:10.4161/rna.35506.
[16] Bertrand E,Chartrand P,Schaefer M,Shenoy S M,Singer R H,Long R M. Localization of ASH1 mRNA particles in living yeast[J].Molecular Cell,1998,2(4):437-445. doi:10.1016/s1097-2765(00)80143-4.
[17] Rook M S,Lu M,Kosik K S. CaMKIIalpha 3′untranslated region-directed mRNA translocation in living neurons:Visualization by GFP linkage[J].The Journal of Neuroscience,2000,20(17):6385-6393.doi:10.1523/jneurosci.20-17-06385.2000.
[18] Forrest K M,Gavis E R. Live imaging of endogenous RNA reveals a diffusion and entrapment mechanism for Nanos mRNA localization in Drosophila[J].Current Biology,2003,13(14):1159-1168. doi:10.1016/s0960-9822(03)00451-2.
[19] Hamada S,Ishiyama K,Choi S B,Wang C L,Singh S,Kawai N,Franceschi V R,Okita T W. The transport of prolamine RNAs to prolamine protein bodies in living rice endosperm cells[J].The Plant Cell,2003,15(10):2253-2264. doi:10.1105/tpc.013466.
[20] Sambade A,Brandner K,Hofmann C,Seemanpillai M,Mutterer J,Heinlein M. Transport of TMV movement protein particles associated with the targeting of RNA to plasmodesmata[J].Traffic(Copenhagen, Denmark),2008,9(12):2073-2088. doi:10.1111/j.1600-0854.2008.00824.x.
[21] Park H Y,Lim H,Yoon Y J,Follenzi A,Nwokafor C,Lopez-Jones M,Meng X H,Singer R H. Visualization of dynamics of single endogenous mRNA labeled in live mouse[J].Science,2014,343(6169):422-424. doi:10.1126/science.1239200.
[22] Köhler R H. GFP for in vivo imaging of subcellular structures in plant cells[J].Trends in Plant Science,1998,3(8):317-320.doi:10.1016/S1360-1385(98)01276-X.
[23] Wu B,Chao J A,Singer R H. Fluorescence fluctuation spectroscopy enables quantitative imaging of single mRNAs in living cells[J].Biophys J,2012,102(12):2936-2944. doi:10.1016/j.bpj.2012.05.017.
[24] Abe M,Kobayashi Y,Yamamoto S,Daimon Y,Yamaguchi A,Ikeda Y,Ichinoki H,Notaguchi M,Goto K,Araki T. FD,a bZIP protein mediating signals from the floral pathway integrator FT at the shoot apex[J].Science,2005,309(5737):1052-1056. doi:10.1126/science.1115983.
[25] Wigge P A,Kim M C,Jaeger K E,Busch W,Schmid M,Lohmann J U,Weigel D. Integration of spatial and temporal information during floral induction in Arabidopsis[J].Science,2005,309(5737):1056-1059. doi:10.1126/science.1114358.
[26] Luo K R,Huang N C,Yu T S. Selective targeting of mobile mRNAs to plasmodesmata for cell-to-cell movement[J].Plant Physiology,2018,177(2):604-614. doi:10.1104/pp.18.00107.
[27] Tyagi S. Imaging intracellular RNA distribution and dynamics in living cells[J].Nature Methods,2009,6(5):331-338. doi:10.1038/nmeth.1321.
[28] Tilsner J. Techniques for RNA in vivo imaging in plants[J].Journal of Microscopy,2015,258(1):1-5. doi:10.1111/jmi.12208.
[29] Fusco D,Accornero N,Lavoie B,Shenoy S M,Blanchard J M,Singer R H,Bertrand E. Single mRNA molecules demonstrate probabilistic movement in living mammalian cells[J].Curr Biol,2003,13(2):161-167. doi:10.1016/s0960-9822(02)01436-7.
[30] Turck F,Fornara F,Coupland G. Regulation and identity of florigen:FLOWERINGLOCUS T moves center stage[J].Annual Review of Plant Biology,2008,59:573-594. doi:10.1146/annurev.arplant.59.032607.092755.
[31] Li C Y,Gu M,Shi N N,Zhang H,Yang X,Osman T,Liu Y L,Wang H Z,Vatish M,Jackson S,Hong Y G. Mobile FT mRNA contributes to the systemic florigensignalling in floral induction[J].Scientific Reports,2011,1:73. doi:10.1038/srep00073.