在植物体内,蔗糖主要在细胞质中合成,催化的酶为蔗糖合成酶(Sucrose synthase,SS)与磷酸蔗糖合成酶(Sucrose phosphate synthase,SPS),其葡萄糖基供体都为尿苷二磷酸葡萄糖(UDPG)[1],而淀粉合成主要在叶绿体中完成,催化的酶为淀粉合成酶(Soluble starch synthase,SSS)与淀粉磷酸化酶(Starch phosphorylase,SP),其葡萄糖基供体分别为腺苷二磷酸葡萄糖(ADPG)与1-磷酸葡萄糖(1-P-G)[2]。1-P-G是UDPG与ADPG合成的共同底物,而1-P-G又来自于上游糖酵解(EMP)或光合作用(Photosynthesis)生成的6-磷酸葡萄糖(6-P-G),其中,磷酸葡萄糖变位酶(PGM)可以催化1-P-G与6-P-G的互相转化,并使两者处于动态平衡中[3],以维持1-P-G与6-P-G在细胞质与叶绿体中的浓度平衡以及蔗糖与淀粉的合成。
磷酸葡萄糖变位酶(Phosphoglucomutase,PGM)属磷酸己糖变位酶家族。根据亚细胞定位的不同,PGM分为2种,胞质型PGM(Cytosol PGM,cPGM)定位于细胞质,质体型PGM(Plastid PGM,pPGM)定位于叶绿体[4]。PGM蛋白一般以单亚基存在,少数物种以同型双亚基发挥功能,如豌豆[5],其单亚基分子量约为60 ku,双亚基分子量约为130 ku。对于同一物种,pPGM的氨基酸数量与分子量都较cPGM略大,所有报道的PGM最适pH值都偏碱性,在7.5~9.0。同时,不同来源的PGM对于同一底物的酶促动力学参数Km、Vmax也表现出较大的差异,说明PGM的活性发挥具有组织特异性与底物浓度特异性[6]。
动物与人类PGM基因的研究要早于植物,早在20世纪30年代,就在兔子与青蛙肌肉提取物中发现了PGM[7],人类PGM1基因于1993年被克隆出来[8],而植物中第一个报道的PGM基因是1994年在菠菜(Spinacia oleracea)中克隆获得的[9]。目前,PGM基因已经从拟南芥(Arabidopsis thaliana)[10]、玉米(Zea mays)[11]、马铃薯(Solanum tuberosum)[12]、豌豆(Pisum sativum)[13]等多种植物中克隆、表达及纯化出来。小桐子(Jatropha curcas L.)是木本能源植物,隶属于大戟科(Euphorbiaceae)麻疯树属(Jatropha),其种子含油量高达35%~60%,且流动性、与石化油掺和性好,品质优于国内零号柴油,达到了欧IV标准,是理想的柴油替代品[14]。小桐子是原产于热带及亚热带地区的喜温植物,低温冷害是影响小桐子地域分布、限制小桐子产业发展的主要环境因素。可溶性糖是参与小桐子抗冷性的重要渗透调节物质,而PGM基因是调节可溶性糖代谢途经的关键基因。关于小桐子PGM基因的研究目前还没有报道,本研究通过同源序列比对方法克隆得到小桐子cPGM与pPGM基因的全长编码框序列,利用qRT-PCR技术研究了两者在低温条件下的表达特性,并构建了两者的原核表达载体,在大肠杆菌中成功表达,为小桐子PGM活性分析以及过表达与突变功能鉴定积累了数据。
1 材料和方法
1.1 试验材料及处理
试验所用的小桐子种子来自云南省元谋县干热河谷地区。经过筛选的饱满种子,用1.5% CuSO4消毒20 min,之后用蒸馏水润洗5次去除残余的CuSO4,消毒后的种子置26 ℃的恒温培养箱中,用蒸馏水浸泡吸涨24 h。取白瓷盘垫5层滤纸并用蒸馏水湿润,将吸涨的种子播于其中,在温度26 ℃、相对湿度75%、光周期16 h/8 h的恒温培养箱中萌发5 d。将发芽的种子播种于培养土,放置在同上条件的恒温培养箱中生长15 d直至第2片真叶完全展开[15]。之后将小桐子幼苗置于低温培养箱中进行12 ℃低温处理,分别取低温处理0.5,3,12,24,48 h与对照(CK,正常培养)幼苗的第2片真叶与根,以及吸涨24 h的种子,液氮速冻后保存于1.5 mL离心管中,放置于-80 ℃冰箱中用于后续试验RNA的提取。
1.2 试验方法
1.2.1 小桐子cPGM与pPGM基因的鉴定及序列分析 根据Noir等[10]鉴定的拟南芥2个cPGM基因(At1g70730、At1g23190)与1个pPGM基因(At5g51820),利用其蛋白质序列(NP_177230、NP_173732、NP_199995)对GenBank小桐子蛋白质数据库(GenBank注释版本101)进行BlastP相似性检索,经过去除重复序列,下载得到非冗余的小桐子cPGM与pPGM的基因、mRNA、蛋白质等序列数据。小桐子cPGM与pPGM蛋白的基本参数分子量(Mw)、等电点(pI)利用ProtParam(http://web.expasy.org/protparam/)软件进行解析。通过GenBank数据库下载其他植物的cPGM与pPGM蛋白序列,与鉴定的小桐子cPGM与pPGM蛋白序列共同利用ClustalX软件进行相似性比对,并通过MEGA软件基于邻接法构建系统进化树。同时,比对结果利用GenDOC软件进行cPGM与pPGM蛋白保守结构域的分析。cPGM与pPGM基因结构基于在线软件GSDS(http://gsds.cbi.pku.edu.cn/)进行分析与绘制。染色体定位以Wu等[16]构建的小桐子遗传连锁图谱进行锚定。
1.2.2 小桐子cPGM与pPGM基因的荧光定量表达分析 利用TRIzol试剂(Invitrogen公司)提取对照(CK)与不同低温处理下的叶片、根以及吸涨种子的总RNA,之后利用DNase Ⅰ(TaKaRa公司)去除总RNA中的残余基因组DNA,最终得到纯化的总RNA。经过浓度测定,分别取3 μg总RNA,利用PrimeScript RT Reagent Kit with gDNA Eraser试剂盒(TaKaRa公司)合成第一链cDNA,进行小桐子cPGM与pPGM的qRT-PCR表达分析。仪器为LightCycler96(Roche公司),20 μL反应体系,每个样品重复3次。器官表达以β-Actin为内参基因,低温表达以GAPDH为内参基因[17],所用引物见表1。扩增条件为:94 ℃预变性30 s;94 ℃变性10 s,53.5 ℃退火15 s,72 ℃延伸15 s,45个循环,之后增加溶解曲线程序:95 ℃ 10 s,65 ℃ 60 s,97 ℃ 1 s,连续检测信号。采用2-ΔΔCt方法进行基因表达量的计算。以叶片的表达量为基准进行器官差异表达分析,以对照(CK)的表达量为基准进行低温差异表达分析。
表1 qRT-PCR试验中所用的引物序列
Tab.1 Primers used in the qRT-PCR experiment
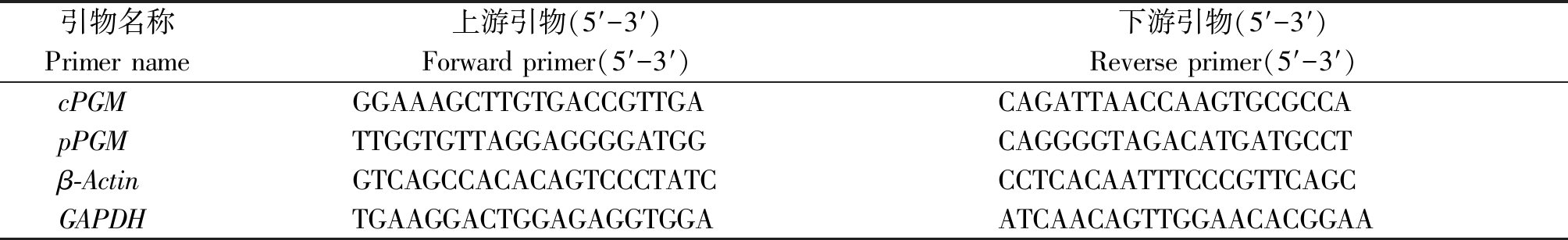
引物名称Primer name上游引物(5′-3′)Forward primer(5′-3′)下游引物(5′-3′)Reverse primer(5′-3′)cPGMGGAAAGCTTGTGACCGTTGACAGATTAACCAAGTGCGCCApPGMTTGGTGTTAGGAGGGGATGGCAGGGGTAGACATGATGCCTβ-ActinGTCAGCCACACAGTCCCTATCCCTCACAATTTCCCGTTCAGCGAPDHTGAAGGACTGGAGAGGTGGAATCAACAGTTGGAACACGGAA
1.2.3 小桐子cPGM与pPGM基因的克隆、原核表达载体构建及目的蛋白诱导表达 根据小桐子cPGM与pPGM基因的mRNA序列,设计带酶切位点的全长编码框Coding sequence(CDS)扩增引物,引物序列为cPGM-F:5′-TGGTTCCGCGTGGATCCCCGGAATTCATGGTAGTGTTCAAGGTTTC-3′(下划线表示EcoR Ⅰ酶切位点),cPGM-R:5′-TCGTGGTGGTGGTGGTGGTGCTCGAGTGTAATAACAGTTGGTGCAG-3′(下划线表示XhoⅠ酶切位点);pPGM-F:5′-TGGTTCCGCGTGGATCCCCGGAATTCATGGCGTCTTCTTCTTGTTTC-3′(下划线表示EcoRⅠ酶切位点),pPGM-R:5′-TCGTGGTGGTGGTGGTGGTGCTCGAGTGTGATGACTGTAGGCTTCTC-3′(下划线表示XhoⅠ酶切位点)。以1.2.2中对照叶片的cDNA为模板,利用KOD FX Neo DNA Polymerase(TOYOBO公司)进行PCR扩增,扩增条件为:98 ℃预变性5 min;98 ℃变性10 s,60 ℃退火30 s,68 ℃延伸2 min,30个循环;68 ℃后延伸5 min。将扩增片段与原核表达载体pGEX-4T-1经EcoR Ⅰ与Xho Ⅰ双酶切,切胶回收后利用T4 DNA连接酶16 ℃过夜连接,得到重组载体,之后通过热激法转化大肠杆菌感受态细胞(TransGene公司Trans1-T1),涂布LB抗性平板(含50 mg/L的Amp),37 ℃过夜培养。经菌落PCR以及EcoRⅠ与XhoⅠ双酶切鉴定阳性的菌落,摇菌提取重组载体分别命名为pGEX-4T-1-JccPGM、pGEX-4T-1-JcpPGM,送华大基因公司进行片段测序。
将pGEX-4T-1空质粒以及构建的小桐子pGEX-4T-1-JccPGM、pGEX-4T-1-JcpPGM原核表达载体转化大肠杆菌BL21(DE3)表达菌株,挑取单克隆接种至5 mL LB液体培养基中(含50 mg/L的Amp),37 ℃ 200 r/min过夜振荡培养。之后按体积比1∶100接种至100 mL LB液体培养基中(含50 mg/L的Amp),37 ℃ 200 r/min振荡培养至OD600值为0.6,取1 mL诱导前菌液至1.5 mL离心管中,置于4 ℃保存。之后,加入异丙基-β-D-硫代半乳糖苷(IPTG)至终浓度为1 mmol/L,于恒温16 ℃振荡诱导表达,分别取诱导1,3,6,12 h的菌液1.0~1.5 mL离心管中于4 ℃保存备用。将以上诱导前与诱导不同时间的菌液12 000 r/min离心5 min,弃上清,菌体沉淀利用200 μL PBS缓冲液(pH值7.4)重悬浮,加入50 μL 5×SDS上样缓冲液,100 ℃沸水中保温10 min,取出冷却至室温,12 000 r/min离心5 min,取20 μL上清蛋白样品上样,进行SDS-PAGE电泳(5%浓缩胶,12%分离胶),检测蛋白质的表达情况。
2 结果与分析
2.1 小桐子cPGM与pPGM基因的鉴定及序列分析
根据同源序列比对,在小桐子基因组中鉴定到1个cPGM基因(GenBank登录号:105634093)、1个pPGM基因(105648538)。基因长度分别为7 608,8 114 bp,mRNA长度分别为2 214,2 347 bp,分别编码582,637 aa的蛋白质。ProtParam预测显示,cPGM与pPGM蛋白分子量分别为63.2,69.5 ku,等电点分别为6.06,6.27。基因结构分析表明,小桐子cPGM基因定位3号染色体,包含19个外显子,且包含207 bp与258 bp的5′-UTR与3′-UTR非编码区域(图1-A);pPGM基因定位5号染色体,包含22个外显子,且包含258 bp与175 bp的5′-UTR与3′-UTR非编码区域(图1-B)。
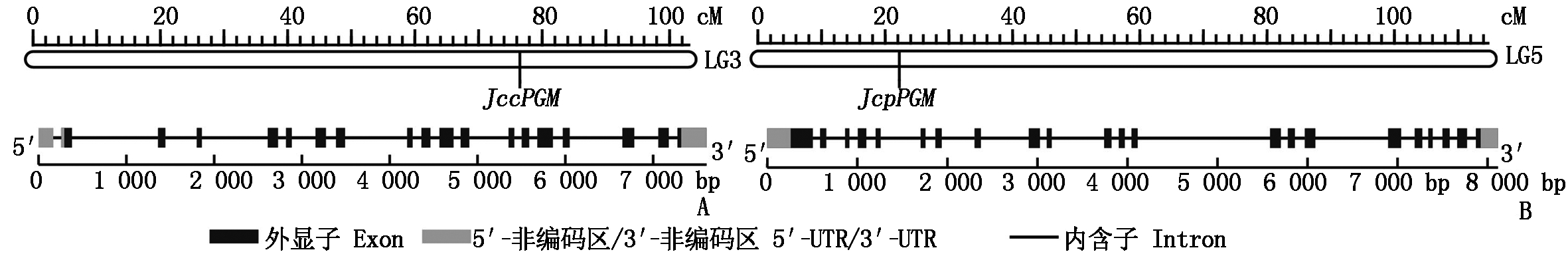
A.cPGM基因;B.pPGM基因。cM.厘摩;LG.染色体。
A.cPGM;B.pPGM.cM.Centi morgan;LG.Chromosome.
图1 小桐子cPGM与pPGM的基因结构
Fig.1 Gene structure of cPGM and pPGM from J.curcas
对72种植物cPGM与pPGM蛋白序列进行多序列比对并构建系统进化树,结果显示,定位细胞质的cPGM与定位叶绿体的pPGM明显聚类为两大分支,且分支内单子叶植物(Monocotyledons)与双子叶植物(Eudicotyledons)也单独聚类(图2),说明单、双子叶植物的PGM是分别进化的。氨基酸序列比对表明,所有pPGM都较cPGM长,且pPGM蛋白都包括长度为60~90 aa的N端叶绿体定位信号肽(图3)。另外,从N端到C端,cPGM蛋白都较pPGM蛋白多4段氨基酸序列-V/S-G-A/V/L-D-G-S-、-S/E-G-P-E-、-G-K-S-N/S/D-S/T-E/N/G-、-S-L/I-G/S/P-E/D-V-N-,且富含侧链R基团较小的氨基酸如Gly、Ala,酸性氨基酸如Asp、Glu以及含有羟基的氨基酸如Ser、Thr,其中,小桐子cPGM蛋白较pPGM蛋白多4段肽链为-108VGVDGS113-、-183SGPE186-、-283GKSNSE288-、-470SLGEVN475-(图4-A)。空间结构分析显示,cPGM较pPGM多余肽段都位于蛋白立体结构的表面β-转角区域,且空间位置一致(图4-B),推测可能是cPGM蛋白的磷酸化/去磷酸化共价修饰调节序列,与4段肽链序列都包含Ser或Thr一致。
2.2 小桐子cPGM与pPGM基因的差异表达分析
通过实时荧光定量表达分析小桐子cPGM与pPGM的器官与低温表达特性。结果显示,cPGM与pPGM基因都在叶片中高表达,而在根与种子中表达量较低(图5-A、C),与已报道PGM主要存在于绿色组织及光合器官的结论一致。低温胁迫条件下,在叶片中,cPGM与pPGM基因都表现出2次降低-升高的表达变化特性,表达量波动幅度较大,且分别在低温处理12,3 h达到最大表达量,较对照分别提高1.69(P<0.05),1.84倍(P<0.05)(图5-B、D)。
2.3 小桐子cPGM与pPGM原核表达载体的构建与蛋白表达
通过EcoR Ⅰ与Xho Ⅰ双酶切,将小桐子cPGM与pPGM基因的编码框序列连接至pGEX-4T-1,构建了两者的原核表达载体pGEX-4T-1-JccPGM、pGEX-4T-1-JcpPGM,通过测序验证均未出现突变。之后,分别将大肠杆菌BL21(DE3)表达菌株、转pGEX-4T-1空质粒的菌株、转pGEX-4T-1-JccPGM与pGEX-4T-1-JcpPGM重组质粒的菌株经裂解后进行SDS-PAGE蛋白电泳检测(图6)。在IPTG诱导的不同时间,cPGM与pPGM分别出现1条约90.7,97.0 ku的融合蛋白条带,且分子量与预期大小一致,说明小桐子cPGM与pPGM基因在大肠杆菌中正确表达,而对照BL21(DE3)表达菌株(CK-1)、转pGEX-4T-1空质粒的菌株(CK-2)以及转pGEX-4T-1-JccPGM与pGEX-4T-1-JcpPGM重组质粒但未经IPTG诱导的菌株(0 h)都没有出现对应的蛋白条带。另外,cPGM与pPGM蛋白都在IPTG诱导1 h后即开始高效表达,随着诱导时间的延长(3,6,12 h),cPGM表达量也在逐渐增加(图6-A),而pPGM表达量则保持稳定(图6-B)。
3 讨论与结论
在植物体中,cPGM与pPGM分别催化细胞质与叶绿体中1-P-G与6-P-G的可逆互变,以控制蔗糖和淀粉的浓度保持动态平衡。以积累蔗糖为主的农作物如甘蔗、甜菜等,光合作用中间产物或淀粉水解产物6-P-G主要由叶绿体转移至细胞质,在cPGM作用下转化为1-P-G,进而形成UDPG,用以合成蔗糖,而以积累淀粉为主的禾本科作物如水稻、小麦等,蔗糖降解产物6-P-G主要由细胞质转移至叶绿体,在pPGM作用下转化为1-P-G,进而形成ADPG,用以合成淀粉[3]。小桐子属多糖多酚类植物,低温条件下,与积累蔗糖和淀粉的农作物不同,碳素同化总量下降,可溶性糖增加,尤其会积累更多的棉子糖系列寡糖(Raffinose family oligosaccharides,RFOs)[18],降低细胞的渗透势以抵抗低温环境,同时,也可作为糖信号参与植物低温信号传导系统。本研究中pPGM较cPGM的低温诱导表达响应更加迅速,在低温胁迫3 h即达到最大表达量,另外,推测小桐子在低温条件下,pPGM较cPGM活性也较高,以加快可溶性寡糖的合成,降低细胞的渗透势,提高抗冷性。
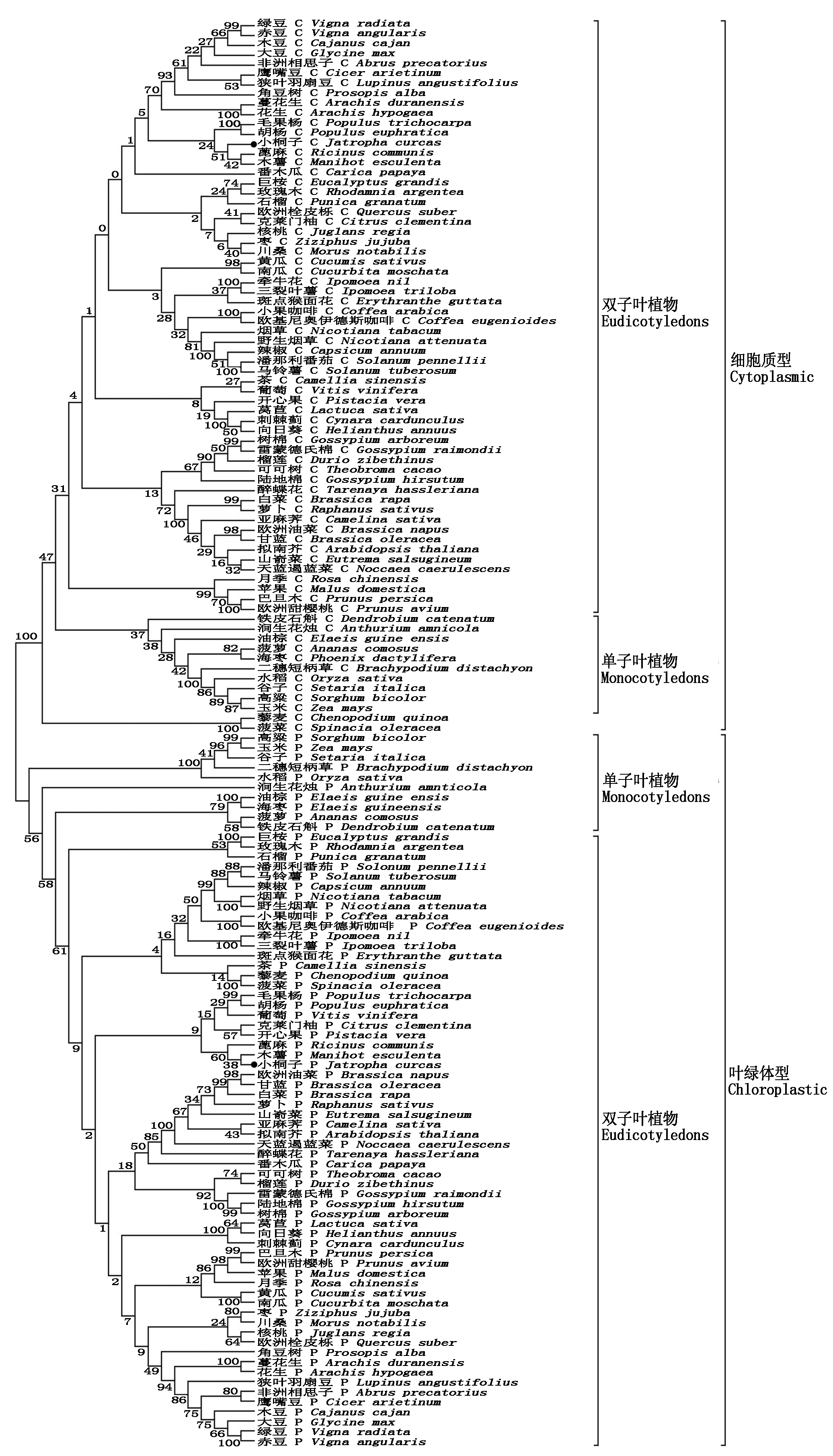
C与 P分别表示细胞质与叶绿体PGM基因。圆点表示小桐子PGM基因。
Cytoplasmic and chloroplastic PGM genes are indicated by C and P,respectively. J.curcas PGM genes are marked by dots.
图2 小桐子与其他物种cPGM与pPGM基因家族的系统进化分析
Fig.2 Phylogenetic relationship of J.curcas with other species cPGM and pPGM gene families

横线表示N端叶绿体定位信号肽。
The line indicates chloroplast localization signal peptide in N-terminal.
图3 小桐子cPGM与pPGM N-端结构域序列比对
Fig.3 Sequence alignment of N-terminal domain in J.curcas cPGM and pPGM
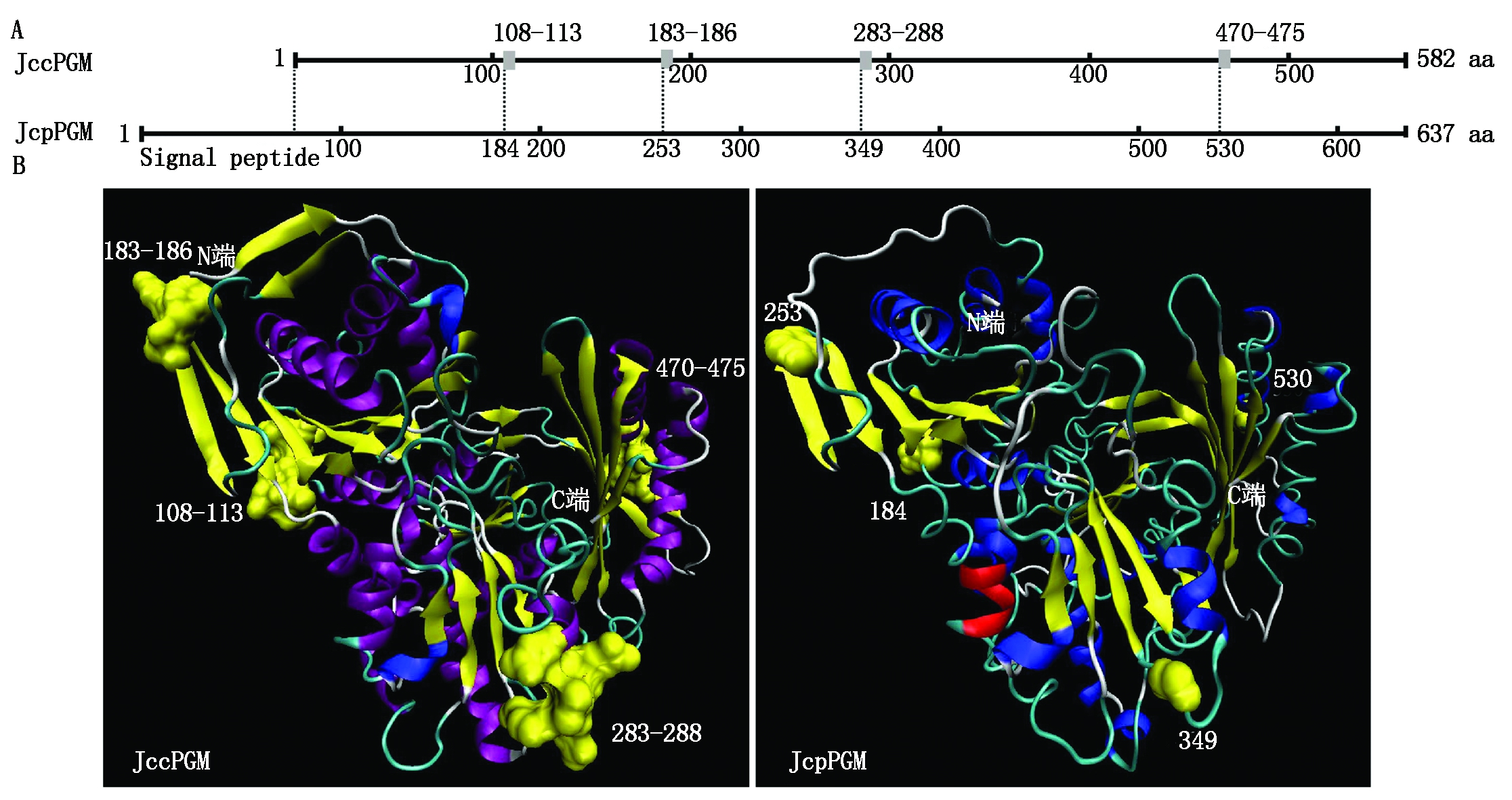
图4 小桐子cPGM与pPGM蛋白序列与空间结构
Fig.4 Protein sequence and 3D-dimensional structure of J.curcas cPGM and pPGM
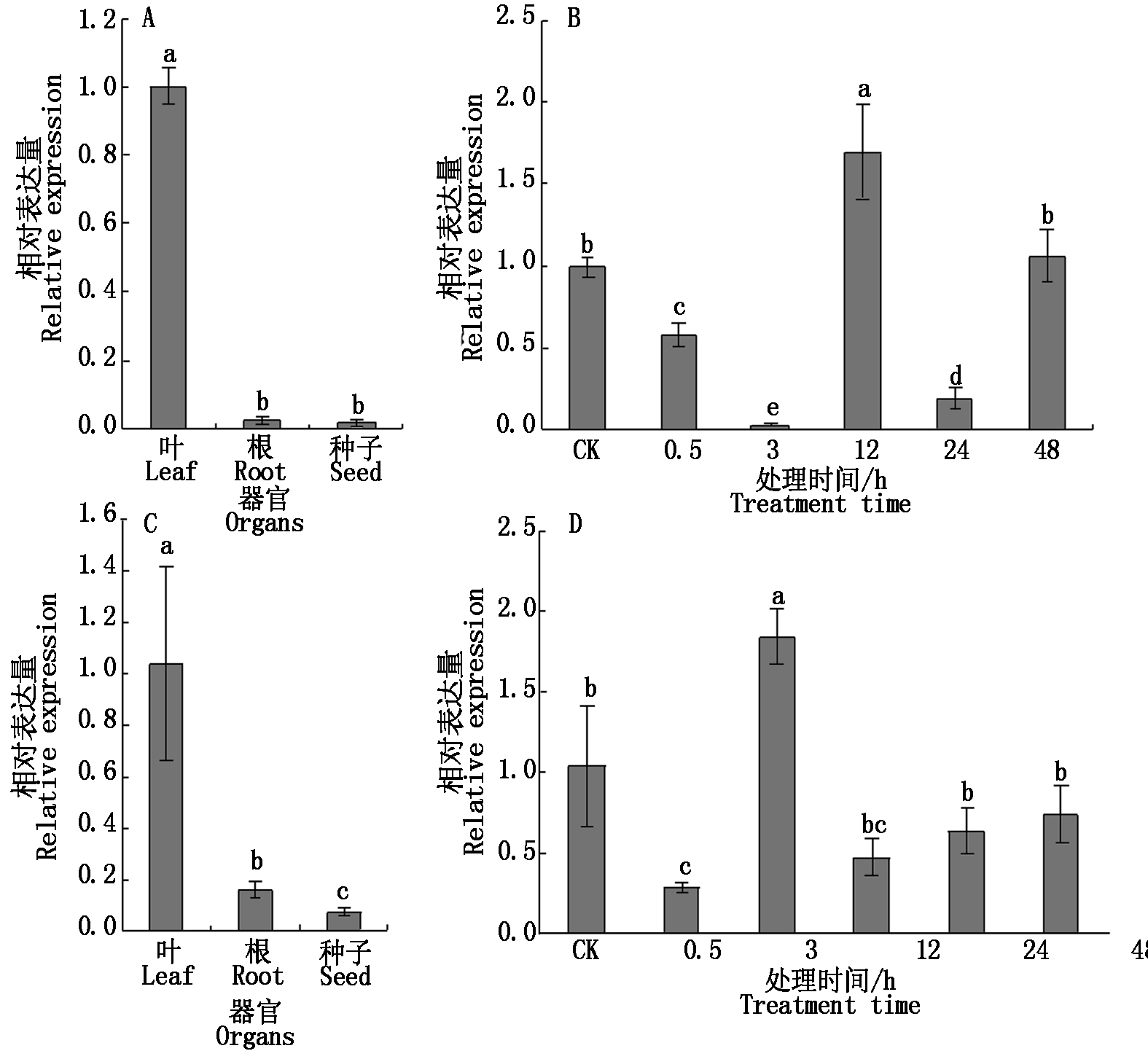
A与C分别表示cPGM与pPGM基因的器官表达特性;B与D分别表示cPGM与pPGM基因在叶片中的低温表达分析。
不同小写字母表示差异显著(P<0.05)。
Organ expression characteristics of cPGM and pPGM are indicated by A and C,respectively;Chilling expression analysis of cPGM and pPGM in leaves are showed by B and D,respectively. Different lowercase letters indicate significant difference(P<0.05).
图5 小桐子cPGM与pPGM基因在不同器官与低温胁迫下的表达分析
Fig.5 Expression analysis of J.curcas cPGM and pPGM genes in different organs and under chilling stress
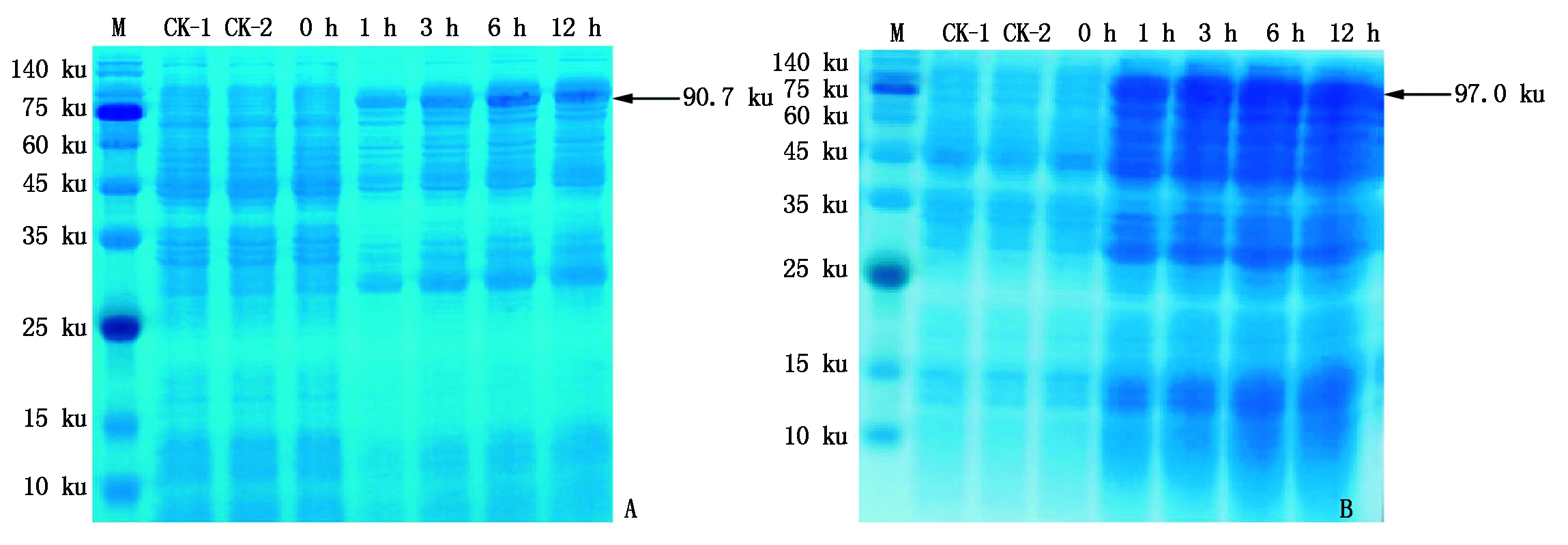
A.cPGM基因;B.pPGM基因。CK-1.大肠杆菌BL21(DE3)表达菌株;CK-2.转pGEX-4T-1空质粒的菌株;转重组质粒未经IPTG诱导的菌株用0 h表示;转重组质粒诱导1,3,6,12 h分别用1,3,6,12 h表示。M.蛋白质Marker。
A and B represent cPGM and pPGM,respectively.CK-1.The supematants from E.coil BL21(DE3)strain;CK-2.The supematants from E.coil BL21(DE3)strain containing pGEX-4T-1;The supematants from E.coil BL21(DE3)strain with recombinant vector but no IPTG induction are represented by 0 h;The supematants from E.coil BL21(DE3)strain with recombinant vector and induced by IPTG for 1,3,6,and 12 h are represented by 1,3,6,and 12 h,respectively. M.Protein Marker.
图6 小桐子cPGM与pPGM基因大肠杆菌表达蛋白的SDS-PAGE电泳检测
Fig.6 SDS-PAGE electrophoresis detection of E.coli expression proteins of J.curcas cPGM and pPGM genes
现有报道表明,单一野生型的等位基因已能够满足糖代谢的需要[19],但从基因型显示,cPGM包含较多等位基因,对应多种同工酶,且功能是冗余的,同时,cPGM的表达与活性具有积累性,共同互补发挥细胞质糖类合成作用。另外,本研究中蛋白序列比对表明,cPGM较pPGM多4段肽链,表现出更多样的活性调节方式如磷酸化共价修饰调节,以适应细胞质中多样糖类合成的需要。Egli等[1]获得了拟南芥pgm2与pgm3单突变体和pgm2与pgm3双突变体,糖类浓度测定表明,单突变体与双突变体的蔗糖、葡萄糖及果糖浓度较野生型没有显著变化,且单突变体的植株表型也与野生型无异,但双突变体的雌雄配子发育及受精能力都受到严重影响。另外,完全丧失cPGM活性的拟南芥的生长也受到显著抑制,植株矮小,根系不发达,生物产量明显降低[20]。在小桐子基因组中仅鉴定到1个cPGM基因,较其他植物如拟南芥少,暗示其表达与功能不可替代,突变将导致严重的生长缺陷[21],但也为后期功能验证提供了理论基础,同时,过表达cPGM基因可提高小桐子蔗糖及其他可溶性糖的含量,对于培育小桐子抗冷新品种也具有潜在的应用价值[19]。pPGM的等位基因则较为单一,同工酶较少,决定其功能具有不可替代性和不积累性。拟南芥pPGM突变体中淀粉的含量较野生型降低了85%,而可溶性糖如淀粉合成底物ADPG的含量则上升显著[22-23]。Tauberger等[12]反义表达pPGM的马铃薯,其块茎中淀粉含量也显著降低,而蔗糖的含量明显上升。另外,pPGM突变百脉根的叶片中根本无法积累淀粉,也不能发生淀粉的碘染色反应[24-25]。以上研究结果表明,突变或反义表达pPGM都可表现出极显著的淀粉含量降低,也暗示以积累淀粉为主的农作物,通过过表达pPGM基因可能会显著提高淀粉的积累和生物学产量。
[1] Egli B,Kölling K,Köhler C,Zeeman S C,Streb S. Loss of cytosolic phosphoglucomutase compromises gametophyte development in Arabidopsis[J].Plant Physiology,2010,154(4):1659-1671. doi:10.1104/pp.110.165027.
[2] Zeeman S C,Smith S M,Smith A M. The diurnal metabolism of leaf starch[J].The Biochemical Journal,2007,401(1):13-28. doi:10.1042/BJ20061393.
[3] Fettke J,Albrecht T,Hejazi M,Mahlow S,Nakamura Y,Steup M. Glucose 1-phosphate is efficiently taken up by potato(Solanum tuberosum)Tuber parenchyma cells and converted to reserve starch granules[J].The New Phytologist,2010,185(3):663-675. doi:10.1111/j.1469-8137.2009.03126.x.
[4] Herbert M,Burkhard C,Schnarrenberger C. A survey for isoenzymes of glucosephosphate isomerase,phosphoglucomutase,glucose-6-phosphate dehydrogenase and 6-Phosphogluconate dehydrogenase in C3-,C4- and crassulacean-acid-metabolism plants,and green algae[J].Planta,1979,145(1):95-104. doi:10.1007/BF00379933.
[5] Galloway C M,Dugger W M. Purification and characterization of phosphoglucomutase from peas[J].Physiologia Plantarum,1994,92(3):479-486. doi:10.1111/j.1399-3054.1994.tb08839.x.
[6] 曹宛虹,王永健. 植物中的葡糖磷酸变位酶[J].生物工程进展,2000,20(5):32-35. doi:10.3969/j.issn.1671-8135.2000.05.010.
Cao W H,Wang Y J. Phosphoglucomutase in plants[J].Progress in Biotechnology,2000,20(5):32-35.
[7] Cori G T,Colowick S T,Cori C F. The formation of glucose-1-phosphoric acid in extracts of mammalian tissues and of yeast [J].Journal of Biological Chemistry,1938,123:375-386. doi:10.1063/1.1710417.
[8] Whitehouse D B,Tomkins J,Lovegrove J U,Hopkinson D A,McMillan W O. A phylogenetic approach to the identification of phosphoglucomutase genes[J].Molecular Biology and Evolution,1998,15(4):456-462. doi:10.1093/oxfordjournals.molbev.a025942.
[9] Penger A,Pelzer-Reith B,Schnarrenberger C. cDNA sequence for the plastidic phosphoglucomutase from Spinacia oleracea (L.)[J].Plant Physiology,1994,105(4):1439-1440. doi:10.1104/pp.105.4.1439.
[10] Noir S,Bräutigam A,Colby T,Schmidt J,Panstruga R. A reference map of the Arabidopsis thaliana mature pollen proteome[J].Biochemical and Biophysical Research Communications,2005,337(4):1257-1266. doi:10.1016/j.bbrc.2005.09.185.
[11] Manjunath S,Lee L H K,Winkle P V,Bailey-Serres J. Molecular and biochemical characterization of cytosolic phosphoglucomutase in maize:Expression during development and in response to oxygen deprivation[J].Plant Physiology,1998,117(3):997-1006. doi:10.1104/pp.117.3.997.
[12] Tauberger E,Fernie A R,Emmermann M,Renz A,Kossmann J,Willmitzer L,Trethewey R N. Antisense inhibition of plastidial phosphoglucomutase provides compelling evidence that potato tuber amyloplasts import carbon from the cytosol in the form of glucose-6-phosphate[J].The Plant Journal,2000,23(1):43-53. doi:10.1046/j.1365-313x.2000.00783.x.
[13] Salvucci M E,Drake R R,Broadbent K P,Haley B E,Hanson K R,McHale N A. Identification of the 64 kilodalton chloroplast stromal phosphoprotein as phosphoglucomutase[J].Plant Physiology,1990,93(1):105-109. doi:10.1104/pp.93.1.105.
[14] Montes J M,Melchinger A E. Domestication and breeding of Jatropha curcas L.[J].Trends in Plant Science,2016,21(12):1045-1057. doi:10.1016/j.tplants.2016.08.008.
[15] Ao P X,Li Z G,Fan D M,Gong M. Involvement of antioxidant defense system in chill hardening-induced chilling tolerance in Jatropha curcas seedlings[J].Acta Physiologiae Plantarum,2013,35(1):153-160. doi:10.1007/s11738-012-1058-z.
[16] Wu P Z,Zhou C P,Cheng S F,Wu Z Y,Lu W J,Han J L,et al. Integrated genome sequence and linkage map of physic nut(Jatropha curcas L.),a biodiesel plant[J].The Plant Journal,2015,81(5):810-821. doi:10.1111/tpj.12761.
[17] Zhang L,He L L,Fu Q T,Xu Z F. Selection of reliable reference genes for gene expression studies in the biofuel plant Jatropha curcas using real-time quantitative PCR[J].International Journal of Molecular Sciences,2013,14(12):24338-24354. doi:10.3390/ijms141224338.
[18] Wang H B,Zou Z R,Wang S S,Gong M. Global analysis of transcriptome responses and gene expression profiles to cold stress of Jatropha curcas L.[J].PLoS One,2013,8(12):e82817. doi:10.1371/journal.pone.0082817.
[19] 李晓屿,李玉花,李晗,李治龙,蓝兴国. 植物葡萄糖磷酸变位酶的研究进展[J].植物生理学报,2015,51(5):617-622.doi:10.13592/j.cnki.ppj.2015.0151.
Li X Y,Li Y H,Li H,Li Z L,Lan X G. Advances in research on phosphoglucomutase in plants[J].Plant Physiology Journal,2015,51(5):617-622.
[20] Fernie A R,Tauberger E,Lytovchenko A,Roessner U,Willmitzer L,Trethewey R N. Antisense repression of cytosolic phosphoglucomutase in potato(Solanum tuberosum)results in severe growth retardation,reduction in Tuber number and altered carbon metabolism[J].Planta,2002,214(4):510-520. doi:10.1007/s004250100644.
[21] Malinova I,Kunz H H,Alseekh S,Herbst K,Fernie A R,Gierth M,Fettke J. Reduction of the cytosolic phosphoglucomutase in Arabidopsis reveals impact on plant growth,seed and root development,and carbohydrate partitioning[J].PLoS One,2014,9(11):e112468. doi:10.1371/journal.pone.0112468.
[22] Andriotis V M E,Pike M J,Schwarz S L,Rawsthorne S,Wang T L,Smith A M. Altered starch turnover in the maternal plant has major effects on Arabidopsis fruit growth and seed composition[J].Plant Physiology,2012,160(3):1175-1186. doi:10.1104/pp.112.205062.
[23] Stitt M,Zeeman S C. Starch turnover:Pathways,regulation and role in growth[J].Current Opinion in Plant Biology,2012,15(3):282-292. doi:10.1016/j.pbi.2012.03.016.
[24] Caspar T,Huber S C,Somerville C. Alterations in growth,photosynthesis,and respiration in a starchless mutant of Arabidopsis thaliana (L.)deficient in chloroplast phosphoglucomutase activity[J].Plant Physiology,1985,79(1):11-17. doi:10.1104/pp.79.1.11.
[25] Vriet C,Welham T,Brachmann A,Pike M,Pike J,Perry J,Parniske M,Sato S,Tabata S,Smith A M,Wang T L. A suite of Lotus japonicus starch mutants reveals both conserved and novel features of starch metabolism[J].Plant Physiology,2010,154(2):643-655. doi:10.1104/pp.110.161844.