牦牛(Bos grunniens)被誉为“高原之舟”和“全能家畜”,不仅是牧民赖以生存的生活和生产资料,同时也是自然选择下具有较强抗逆性的稀有遗传种质资源。我国是世界上牦牛存栏量最多的国家,总数约1 560万头[1],主要分布在海拔4 000~5 000 m的青藏高原及毗邻地区,年均气温≤0 ℃,冷季时间漫长,枯草期长达7个月[2]。研究表明,枯草期牦牛掉膘可达30%,经解剖分析发现皮下脂肪组织被大量消耗[3],表明皮下脂肪组织的沉积及代谢在牦牛抗寒、耐饥饿的适应过程中发挥关键作用。脂肪组织是动物体重要的储能器官,动物长期处于饥饿或冷刺激的状态,储存的脂肪被消耗产生能量和热量,维持机体的基础代谢及体温恒定。然而,牦牛脂肪组织沉积及代谢的相关遗传调控分子尚未完全明晰。
CIDEA基因是诱导细胞凋亡DNA片段化45样效应因子(Cell death-inducing DNA fragmentation factor 45(DFFA)-like effectors,CIDE)家族成员之一,其N端与DFFA高度同源[4]。最初的功能研究发现CIDEA调节Caspase依赖的细胞凋亡及DNA的断裂。随着研究的深入,发现CIDEA在人体和小鼠中与肥胖的发生、胰岛素敏感性及脂滴形成密切相关[4]。敲除CIDEA基因后小鼠的机体能量代谢提高、脂肪水解增加、胰岛素的敏感性提高 [5-6]。研究表明,CIDEA的亚细胞定位主要位于脂滴,其正向调节脂肪细胞内脂滴的融合及增大[7-8]。与白色脂肪组织相比,CIDEA在棕色脂肪组织的表达极高[6],棕色脂肪组织(Brown adiposse tissue,BAT)可消耗脂肪产生热量,维持机体体温的稳定[9]。牦牛具有极强的抗寒适应性,研究表明,牦牛皮下脂肪组织具有棕色组织的特性[10],提示CIDEA基因可能参与调控牦牛脂肪组织的沉积及脂代谢。然而,目前关于牦牛CIDEA基因的序列未知,并且其在牦牛脂肪的时空表达尚不清楚。
因此,本研究以金川牦牛皮下脂肪cDNA为模板,克隆金川牦牛CIDEA基因的CDS区;通过生物信息学的分析方法,分析CIDEA编码蛋白的结构和理化性质、物种间的同源性及进化关系;采用实时荧光定量PCR技术检测CIDEA基因在金川牦牛不同组织及皮下脂肪细胞分化过程中的表达模式,旨在为进一步探索CIDEA基因调控牦牛抗寒适应性提供基础数据。
1 材料和方法
1.1 样品采集
试验所用牦牛共6头,均为年龄4岁左右的金川牦牛,由四川省阿坝羌族藏族自治州金川县牦牛养殖场提供。将其屠宰后取心脏、肝脏、脾脏、肺脏、肾脏、背腰最长肌、腿肌、皮下脂肪等组织置于液氮保存1 d,然后转移至-80 ℃的冰箱长期保存。
1.2 试剂
胶回收试剂盒、pGM-T链接试剂盒、质粒提取试剂盒及大肠杆菌DH5α感受态细胞、PCRMix均购自天根生化科技有限公司;TRIzol试剂、反转录试剂盒、荧光定量试剂盒、DL2000 DNA Marker均购自宝日医生物技术(北京)有限公司(TaKaRa中国)。
1.3 试验方法
1.3.1 RNA提取与cDNA合成 采用TRIzol法提取金川牦牛各组织的总RNA,提取的总RNA用紫外分光光度计测定浓度和质量,OD260nm/280nm=1.8~2.0的样品可用于后续试验。将检测合格的RNA按照反转录试剂盒操作手册,合成cDNA备用。
1.3.2 CIDEA基因T载体构建及测序 以金川牦牛脂肪组织RNA反转录得到cDNA为模板,对CIDEA基因进行PCR扩增。PCR反应体系:cDNA模板(500 ng/μL)1 μL、上下游引物(10 μmol/L)各0.5 μL、PCR Mix 7.5 μL、ddH2O 5.5 μL。PCR反应程序:94 ℃ 预变性4 min;94 ℃ 变性45 s,60 ℃ 退火45 s,72 ℃ 延伸1 min,35个循坏;72 ℃ 延伸6 min。将PCR产物进行1.5%的琼脂糖凝胶电泳35 min,照胶观察是否有目的条带。将通过PCR 扩增所得到的目的基因,用胶回收试剂盒进行纯化。并与PGM-T载体(16 ℃)连接过夜,第2天将连接产物转入DH5α感受态细胞,将菌液置于固体培养基中培养12~16 h,挑取4~6个进行克隆扩大培养。将其他菌液送至成都擎科梓熙生物技术有限公司进行测序,得到CIDEA基因CDS区的全长序列。
1.3.3 CIDEA基因的生物信息学分析 通过NCBI中的ORF Finder寻找牦牛CIDEA基因的开放阅读框,并获取牦牛CIDEA蛋白的氨基酸序列。通过NCBI网站中Blast程序(https://blast.ncbi.nlm.nih.gov/Blast.cgi)将牦牛CIDEA基因的核苷酸序列和蛋白质序列与不同物种进行同源性比对分析,并根据氨基酸序列利用MEGA 5.0软件构建物种间进化树。利用 ProtParam在线程序(https://web.expasy.org/protparam/)分析牦牛CIDEA蛋白理化性质,ProtScale在线程序(https://web.expasy.org/protscale/)分析CIDEA蛋白亲疏水性;同时运用TMHMM在线程序(http://www.cbs.dtu.dk/services/TMHMM-2.0/)与SignalP在线程序(http://www.cbs.dtu.dk/services/SignalP/)预测蛋白的跨膜区和信号肽功能;利用GOR4在线程序(https://npsa-prabi.ibcp.fr/NPSA/npsa_gor4.html)预测牦牛CIDEA蛋白的二级结构;最后利用 SWISS-MODEL 在线程序(https://swissmodel.expasy.org/interactive)预测牦牛CIDEA蛋白的三级结构。牦牛CIDEA基因与不同物种间的同源性比对及蛋白质结构预测等具体方法参照文献 [11-12]。
1.3.4 牦牛前体脂肪细胞的分离与成脂诱导分化 金川牦牛犊牛(<1月龄)屠宰后分离皮下脂肪组织,剪成1 mm3大小,置于15 mL离心管,加入适量的1.5 mg/mL Ⅰ型胶原酶溶液(DMEM配置)在37 ℃水浴锅消化,每隔15 min振荡10 s。消化1 h后,用含10%胎牛血清(FBS)的DMEM终止消化,并用100 μm过滤器去除结缔组织和未消化的组织。滤液离心5 min (1 700 r/min),获得细胞沉淀。用含DMEM、20% FBS、1%青霉素/链霉素(P/S)的生长培养基重悬细胞,并接种于10 cm的培养皿中。在37 °C 、5% CO2的恒温培养箱培养12 h后,用PBS洗涤细胞2~3次,然后每2 d更换为新鲜的培养液。待细胞融合率达到80%,传代至12孔板备用。
待12孔板细胞融合率达到100%,记为第0 天,加入诱导液(Induction medium,IM)孵育3 d,诱导液含DMEM、10% FBS、2.85 μmol/L胰岛素、0.3 μmol/L地塞米松(DEXA)、1 μmol/L罗格列酮和0.63 mmol/L 3-甲基-3-异丁基次黄嘌呤,然后用含DMEM、10% FBS、200 nmol/L 胰岛素和10 nmol/L T3的分化培养基(Differentiation medium,DM)孵育6 d,形成成熟的脂肪细胞。诱导成脂分化过程中,每3 d更换1次培养基。分别于第0,3,6,9 天弃培养基收集细胞样本,并保存于-80 ℃冰箱中待提取RNA。
1.3.5 Bodipy染色 分化成熟的脂肪细胞去除DM,用含Bodipy(终浓度为2 μmol/L)和Hoechst 33342(终浓度为10 μg/mL)染料的DM孵育细胞45 min。然后用PBS洗涤细胞3次,加入新鲜DM,用ZEISSLSM 800共聚焦显微镜拍摄细胞图片。
1.3.6 实时荧光定量PCR技术(qRT-PCR) 针对CIDEA、ADIPOQ和PPARγ基因的CDS序列,采用Primer Premier 5.0软件设计特异性定量引物(表1),以反转录得到的金川牦牛心脏、肝脏、脾脏、肺脏、肾脏、脂肪、腿肌、背肌等组织及脂肪细胞的cDNA为模板,按照TaKaRa荧光定量试剂盒说明书检测上述基因的相对表达量,以环孢素A受体(Peptidylprolyl isomerase A,PPIA)为内参基因。荧光定量PCR反应体系:SYBR Green Master Mix 7.5 μL,cDNA(100 ng/μL)2 μL,上、下游引物(10 μmol/L)各0.4 μL,RNase-free ddH2O 4.7 μL。qRT-PCR反应条件如下:95 ℃ 预变性3 min;95 ℃ 变性10 s,60 ℃退火20 s,72 ℃ 延伸30 s,39个循环。以2-ΔΔCt法分析荧光定量结果。
表1 引物信息
Tab.1 Primer information
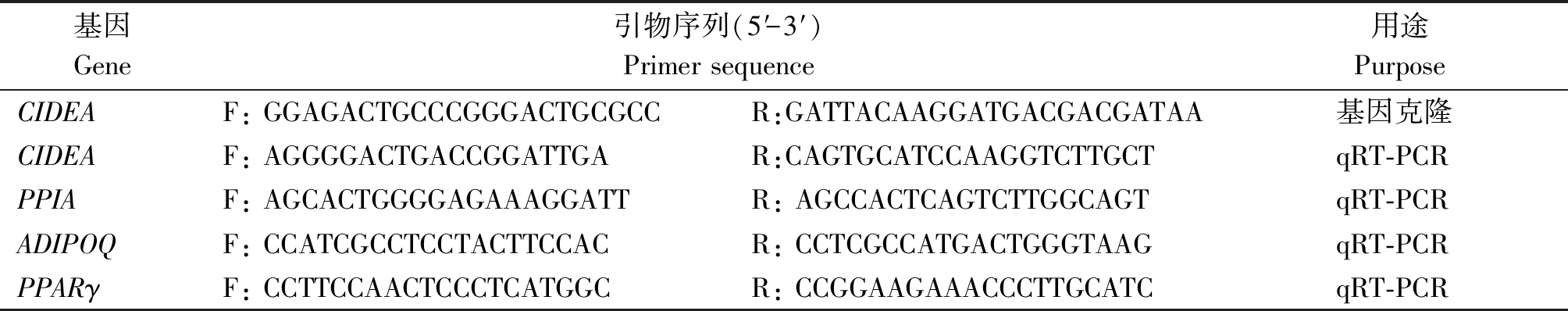
基因Gene引物序列(5′-3′)Primer sequence用途PurposeCIDEAF: GGAGACTGCCCGGGACTGCGCCR:GATTACAAGGATGACGACGATAA基因克隆CIDEAF: AGGGGACTGACCGGATTGA R:CAGTGCATCCAAGGTCTTGCTqRT-PCRPPIAF: AGCACTGGGGAGAAAGGATTR: AGCCACTCAGTCTTGGCAGTqRT-PCRADIPOQF: CCATCGCCTCCTACTTCCACR: CCTCGCCATGACTGGGTAAGqRT-PCRPPARγF: CCTTCCAACTCCCTCATGGCR: CCGGAAGAAACCCTTGCATCqRT-PCR
1.3.7 数据统计与分析 根据上述荧光定量结果,采用2-ΔΔCt法对数据结果进行计算处理。采用SPSS 20.0软件中单因素方差(One-way ANOVA)模块分析不同组织中CIDEA基因表达量的差异显著性;采用SPSS 20.0 软件中独立样本t检验模块分析CIDEA基因在脂肪细胞分化不同时间点的差异显著性。P<0.05为差异显著,P <0.01为差异极显著。
2 结果与分析
2.1 牦牛CIDEA基因的克隆
本试验以金川牦牛皮下脂肪组织的cDNA为模板,设计特异引物,PCR扩增获得696 bp的核苷酸序列(图1-A),进一步将PCR产物进行切胶回收,构建T载体克隆,送成都擎科梓熙生物技术有限公司测序,其长度和碱基组成与预测一致(图1-B)。序列分析发现,CIDEA基因CDS序列长为684 bp,共编码227 个氨基酸。因此,本研究克隆获得了金川牦牛CIDEA基因的CDS序列。
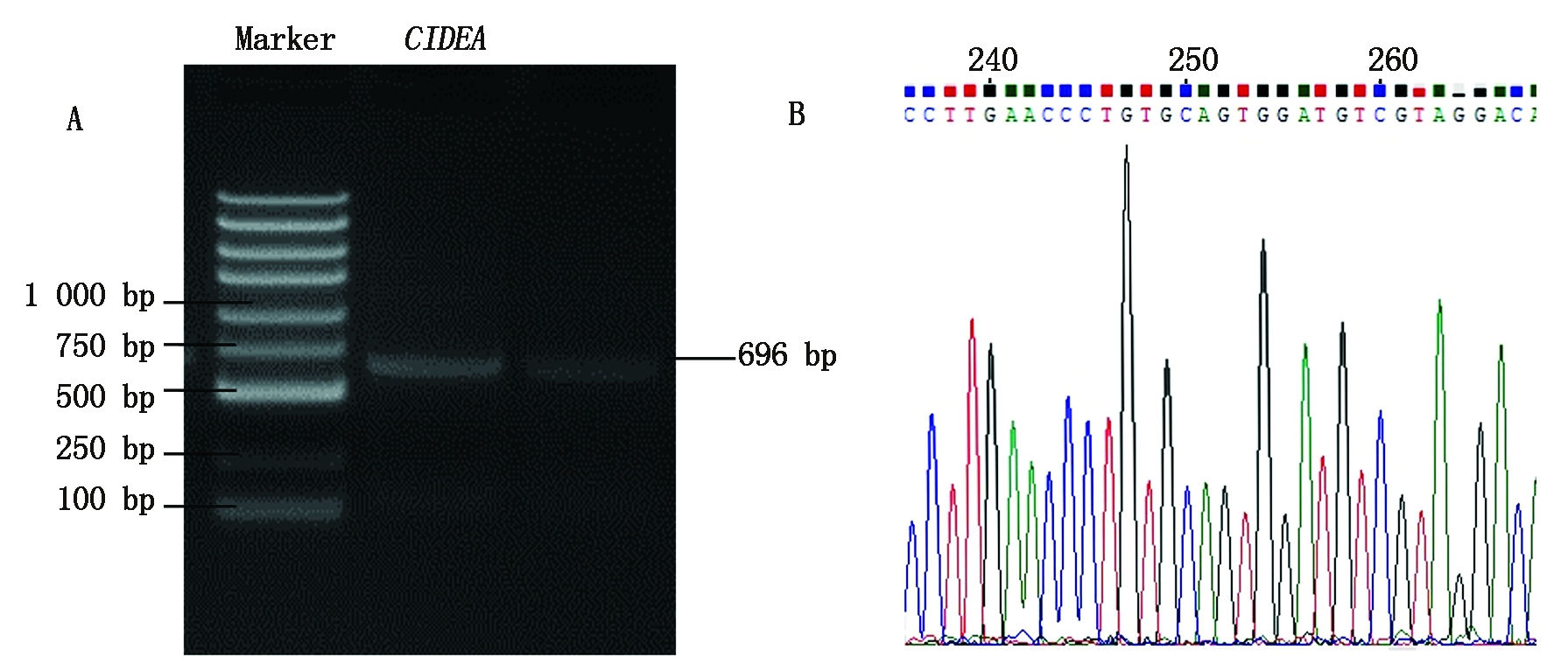
A.金川牦牛CIDEA基因PCR产物琼脂糖凝胶电泳;B.金川牦牛CIDEA基因序列pGM-T载体测序图谱。
A. Agarose gel electrophoresis of CIDEA PCR products in Jinchuan yak;B. The sequencing curve of CIDEA cloned into pGM-T.
图1 金川牦牛CIDEA基因克隆与测序结果
Fig.1 Cloning and sequencing results of CIDEA in Jinchuan yak
2.2 牦牛CIDEA基因在不同物种间的同源性
将金川牦牛CIDEA基因核苷酸及氨基酸序列与GenBank中黄金仓鼠(Mesocricetus auratus)、鸡(Gallus gallus)、马(Equus caballus)、牛(Bos taurus)、大鼠(Rattus norvegicus)、人(Homo sapiens)、家养雪貂(Mustela putorius furo)、猪(Sus scrofa)等物种进行同源性比对,结果显示,牦牛CIDEA基因与牛的同源性最高,核苷酸同源性为94.14%,氨基酸同源性为99.54%;与鸡的同源性最低,核苷酸同源性仅为63.38%,氨基酸同源性为59.05%(图2-A、B)。同时根据氨基酸序列,构建物种进化树(图2-C),与序列比对结果一致,牦牛与普通牛之间的亲缘关系最近,与鸡之间的亲缘关系最远。

A.核苷酸同源性比对;B.氨基酸同源性比对;C.牦牛与其他物种CIDEA氨基酸序列系统进化树。
A.Nucleotide acid homology comparison;B.Amino acid homology comparison;C.CIDEA amino acid sequence phylogenetic tree of yak and other species.
图2 牦牛CIDEA基因在不同物种间同源性比对
Fig.2 Homology comparison of CIDEA in yak among different species
2.3 CIDEA蛋白质理化性质及结构预测分析
经ProtParam在线程序对牦牛CIDEA蛋白的氨基酸组成与理化性质进行预测(表2),结果显示,CIDEA蛋白共由20种氨基酸组成,其中亮氨酸(Leu)含量最高,达11.5%;蛋白质分子质量为16,等电点为8.88,不稳定指数为55.57(>40为不稳定),平均亲水指数-0.292(正数为疏水,负数为亲水蛋白),运用ProtScal在线程序进一步验证该蛋白为不稳定的碱性亲水蛋白(图3-A)。利用TMHMM在线程序预测CIDEA蛋白跨膜结构(图3-B)和SignalP在线程序预测CIDEA蛋白的信号肽(图3-C),经分析得CIDEA蛋白既不是跨膜蛋白也不是分泌蛋白。此外,由DOR4在线程序对牦牛CIDEA蛋白的二级结构进行预测,结果显示,二级结构主要为α螺旋(蓝色长竖线区)和无规则卷曲(紫色短竖线区),分别占比为33.92%,51.98%(图3-D);进一步通过SWISS-MODEL在线程序得到CIDEA蛋白的三级结构,其中GMQE值为0.28,模拟质量相对较好(图3-E)。
表2 氨基酸组成
Tab.2 Amino acid composition
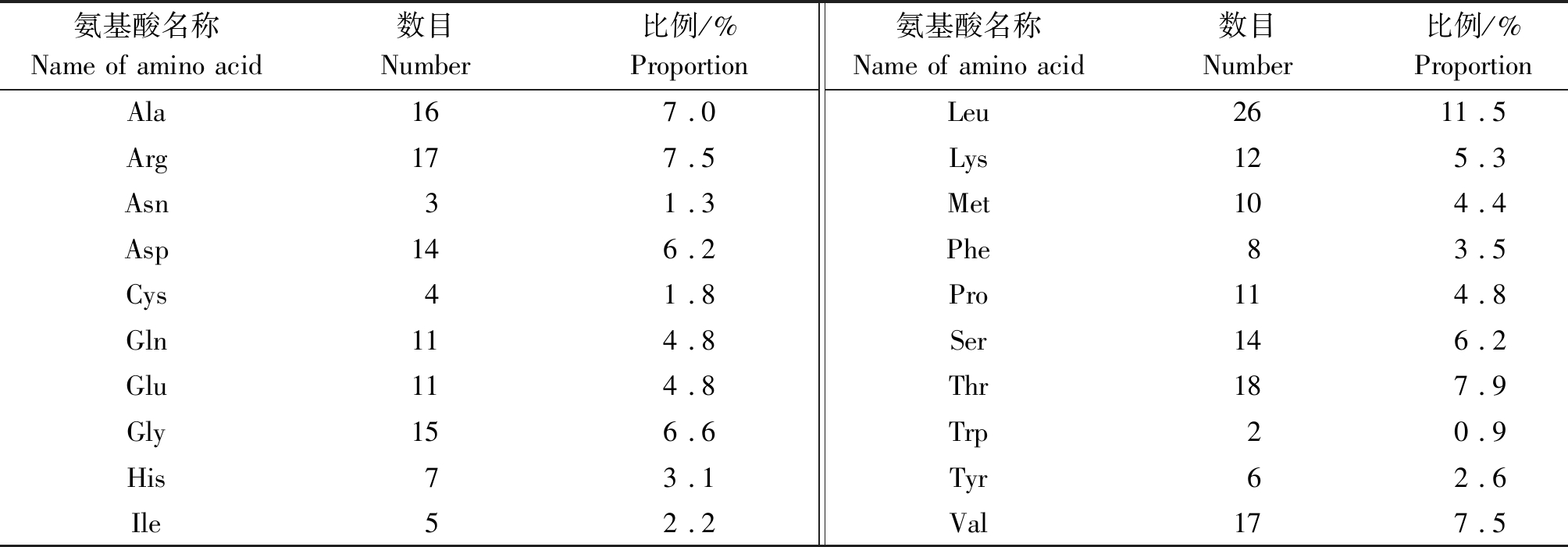
氨基酸名称Name of amino acid数目Number比例/%Proportion氨基酸名称Name of amino acid数目Number比例/%ProportionAla167 .0Leu2611 .5Arg177 .5Lys125 .3Asn31 .3Met104 .4Asp146 .2Phe83 .5Cys41 .8Pro114 .8Gln114 .8Ser146 .2Glu114 .8Thr187 .9Gly156 .6Trp20 .9His73 .1Tyr62 .6Ile52 .2Val177 .5
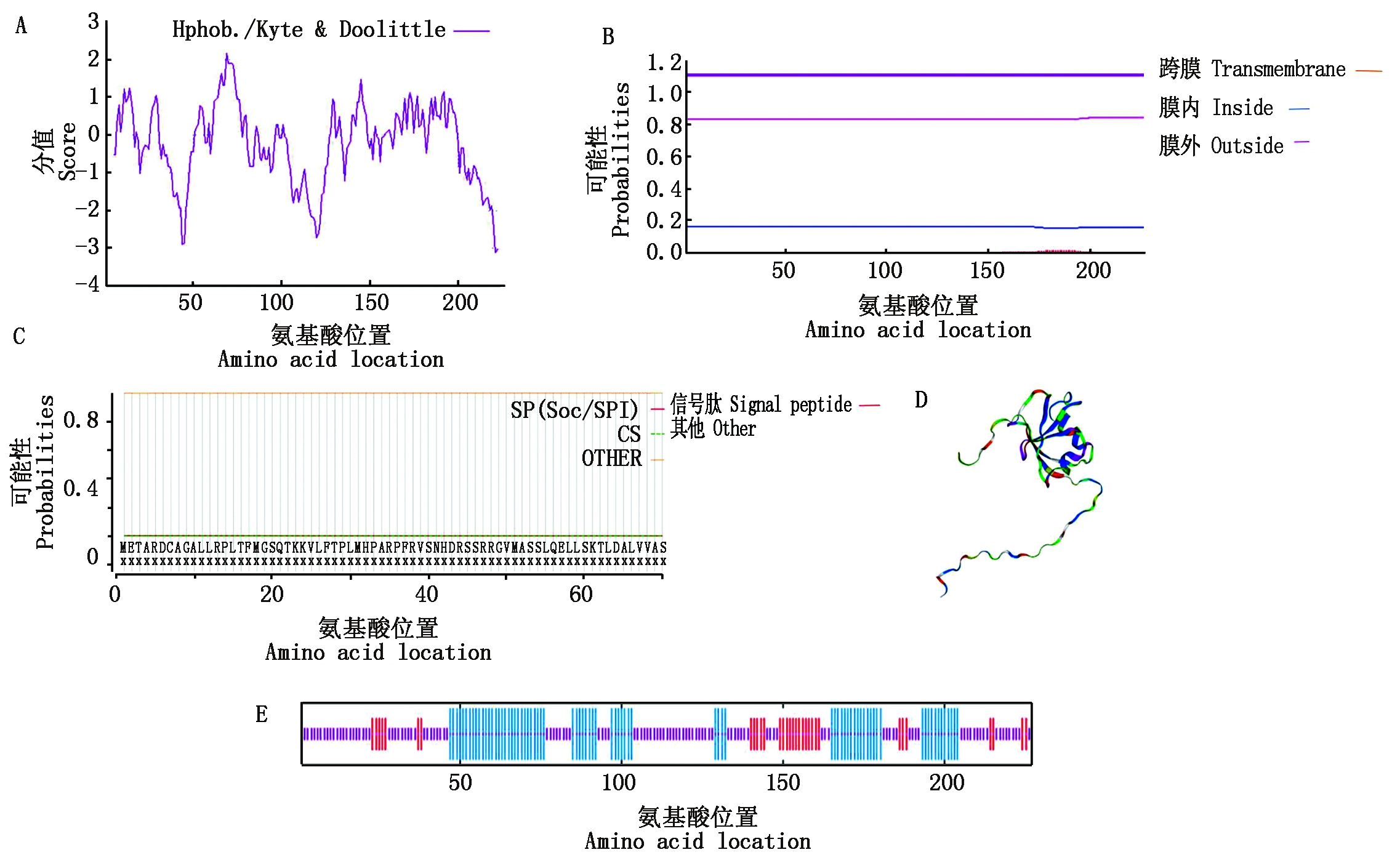
A.蛋白质亲疏水性的预测(以0为界正值表示疏水性,负值表示亲水性);B.蛋白跨膜结构预测;C.蛋白信号肽预测(当SPI值接近1时,存在信号肽);D.CIDEA蛋白三级结构;E.CIDEA蛋白二级结构:紫色竖线表示无规则卷曲,红色竖线表示β折叠,蓝色竖线表示α螺旋。
A. Protein hydrophilic and hydrophobic prediction (A positive value of 0 indicates hydrophobicity and a negative value indicates hydrophilicity); B. Predicted the protein transmembrane structure; C. Signal peptide structure of protein (Signal peptide present when SPI value is close to 1); D. Tertiary structure of CIDEA protein; E. Secondary structure of CIDEA protein: Random coil, β-fold and α-helix are indicated by purple, red and blue bold vertical lines, respectively.
图3 CIDEA生物信息学分析
Fig.3 Bioinformatic analysis of CIDEA
2.4 CIDEA基因的组织表达分析
以PPIA基因为内参基因,通过实时荧光定量PCR技术检测CIDEA基因在金川牦牛不同组织(肺脏、肝脏、心脏、脾脏、肾脏、脂肪组织、背最长肌、腿肌)中的mRNA表达量。研究结果显示,CIDEA基因在金川牦牛脂肪组织中表达量最高,在肺脏组织中的表达量最低;在脂肪组织的表达量约为肺脏组织表达量的2 192倍(P <0.01)。在脂肪组织中CIDEA基因的表达量与其他组织相比较,差异均极显著;此外,CIDEA基因在肝脏的表达量较高,约为肺脏组织表达量的54倍(图4)。以上研究表明,CIDEA基因在脂肪组织丰富表达。
2.5 CIDEA基因时序表达分析
分离的原代脂肪细胞,待细胞融合率达到100%(图5-A),进行成脂诱导分化。在诱导分化过程中,随诱导分化天数增加,包含脂滴的脂肪细胞数量逐渐增加,且随分化维持时间的延长,脂滴逐渐变大(图5-B-E)。在分化第9天,用中性脂质特异染料Bodipy标记脂滴,Hoechst 33342染料标记细胞核,结果显示,在诱导细胞内有大量脂滴形成,并且少量细胞包含一个大脂滴,细胞核被挤压到细胞边缘(图5-F)。与上述细胞形态学观察一致,脂肪细胞分化标志基因表达量如过氧化物酶增殖物激活受体γ基因(PPARγ)、脂联素基因(ADIPOQ)在分化过程中呈现上升的趋势(图5-G-I),表明牦牛脂肪细胞分化模型建立成功。同时用上述样本检测CIDEA基因在脂肪细胞分化中的表达情况,显示CIDEA基因在加入IM后,其mRNA显著上调,到第6天时其表达量上调近200倍(图5-I),与分化前期相比差异极显著(P<0.01);分化第9天时,CIDEA基因与第6天相比略有下降,但差异不显著(P>0.05)。以上研究结果表明,CIDEA基因在前体脂肪细胞生脂过程中呈上升的表达模式。
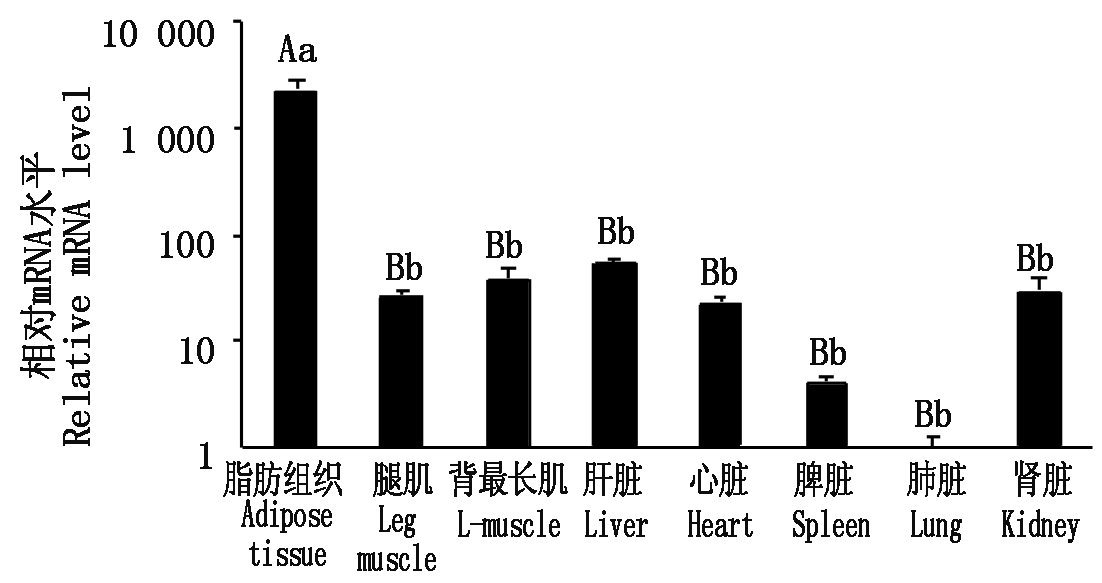
L-muscle. 背最长肌;不同小写字母表示差异显著(P<0.05);不同大写字母表示差异极显著(P<0.01)。图5同。
L-muscle. Longissimus dorsi muscle; The different lowercase letters indicate significant differences(P<0.05). The different capital letters indicate extremely significant differences(P<0.01). The same as Fig.5.
图4 CIDEA基因在金川牦牛不同组织的表达模式
Fig.4 The expression pattern of CIDEA in Jinchuan yak various tissues
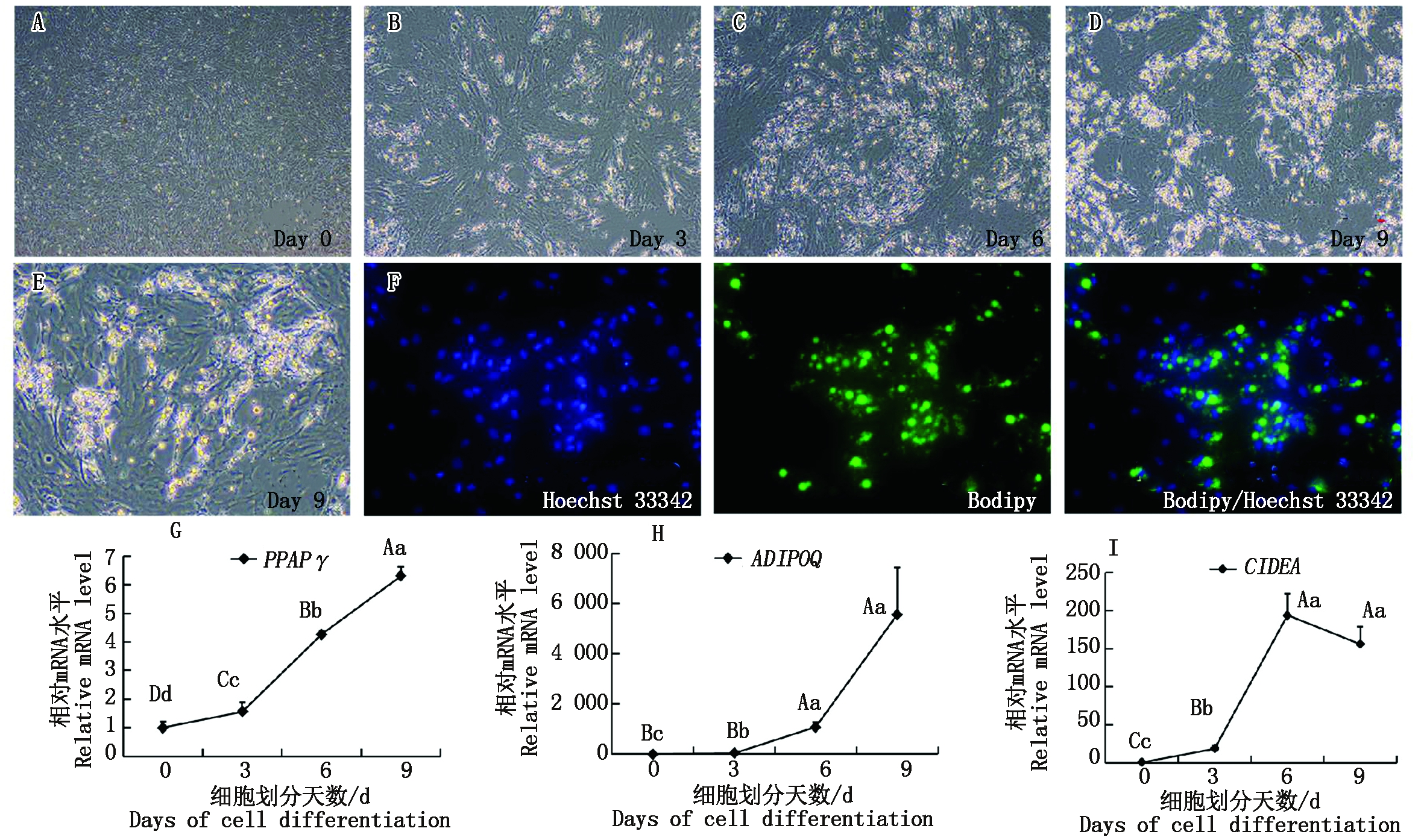
A. 原代脂肪细胞;B-E. 成脂诱导后第3,6,9天细胞形态;F. 分化第9天Bodipy染色:细胞核蓝色荧光标记,脂滴绿色荧光标记;G-I. 脂肪细胞分化过程中PPARγ(G)、ADIPOQ(H)、CIDEA(I)的相对mRNA表达量。
A. Primary preadipocyte;B-E. The cell morphology of adipocytes at day 3,day 6 and day 9 after adipogenic induction;F. The bodipy staining of adipocytes at day 9 after adipogenic induction:Blue fluorescence labeling of nucleus and green fluorescence labeling of lipid droplets;G-I. The relative mRNA expression quantity of PPARγ (G),ADIPOQ (H)and CIDEA (I)during adipocytes differentiation.
图5 CIDEA基因在金川牦牛前体脂肪细胞分化中的表达模式
Fig.5 The expression pattern of CIDEA in differentiation of Jinchuan yak preadipocytes
3 讨论与结论
牦牛分布于我国青藏高原及其毗邻地区,其冷季适应性研究是探索其高原适应性的重点领域之一。脂肪组织作为机体储能的主要形式[13],脂肪细胞及其分泌产物涉及广泛生理过程[14]。研究发现,在饲草丰富的暖季,牦牛皮下脂肪沉积较多;而在冷季枯草期,皮下脂肪组织被大量消耗,表明牦牛皮下脂肪组织的分解代谢是其维持枯草期最基本生命活动的重要能量来源[15]。尽管目前的研究发现了牦牛皮下脂肪组织的沉积与消耗对其冷季适应性极为重要,但关于牦牛皮下脂肪组织脂代谢分子遗传学基础及其调控因子仍不清楚。
本研究采用PCR法扩增得到金川牦牛CIDEA基因完整编码区序列,其中CDS序列长度为684 bp,编码227个氨基酸,是一个碱性亲水蛋白。物种间同源性比对及进化树结果表明,金川牦牛CIDEA基因与普通牛的同源性最高、亲缘关系最近,与鸡的同源性低、亲缘关系最远,表明CIDEA基因在反刍动物和家禽中的功能可能存在差异。
通过实时荧光定量PCR检测发现,金川牦牛CIDEA基因在脂肪组织中表达量最高,肝脏、背腰最长肌也有较高表达量,肺脏中表达量最低。与本研究结果一致,CIDEA 基因在小鼠心脏、肝脏、脾脏、小肠、淋巴结、脂肪、脑、肾脏等组织中均有表达,其中在脂肪组织中表达最高 [16-17] 。在猪中,CIDEA基因在猪脂肪组织中表达量最高,在小肠、肌肉和心脏中表达较低[18]。在肉鸡各组织中检测结果显示,CIDEA基因也在脂肪组织中的表达量最高,其次是肾脏、肝脏和卵巢,在心脏、脾脏、肺脏、胸肌、腿肌中表达量较少[19]。在隆林山羊中,CIDEA基因在脂肪组织的表达量极显著高于其他组织与器官[20]。综上,本研究结果及其他动物中的表达分析显示,CIDEA基因在脂肪组织的表达量最高,在其他组织中的表达量有所差异。因此,推测CIDEA是脂肪组织特异表达基因。
目前的研究仅报道了CIDEA基因在小鼠体内具有调控脂肪沉积及脂代谢的功能。采用aP2启动子构建CIDEA的转基因小鼠,发现小鼠皮下脂肪沉积增加,产生肥胖的表型,但是内脏脂肪组织大小不变,且提高了胰岛素的敏感性[6],表明CIDEA基因在不同类型脂肪组织的功能及作用机制具有差异性。白色脂肪组织和棕色脂肪组织在动物机体能量代谢中的作用差异较大,有研究比较小鼠白色和棕色脂肪组织CIDEA基因的表达,结果显示,其在棕色脂肪组织中有较高的表达量[4,21]。CIDEA基因敲除小鼠棕色脂肪组织肪代谢速率显著提高,脂肪酸氧化速率明显加快,其可能的分子机制为CIDEA与AMPK-β亚基特异性作用形成蛋白复合体,促进AMPK复合体通过泛素化介导蛋白酶体途径的降解[22]。尽管谢凤莲[9]研究报道牦牛的皮下脂肪组织具有棕色脂肪组织的特性,高表达产热相关的标志基因,如解偶联蛋白1(Uncoupling protein 1,UCP1)等,但CIDEA基因在牦牛皮下脂肪沉积的功能及作用机制仍需要进一步研究。
鉴于在牦牛体内研究CIDEA基因的功能存在较大的困难,因此,本研究成功构建了金川牦牛前体脂肪细胞成脂分化的体外模型。采用酶消化法分离的原代前体脂肪细胞,经IM和DM的诱导能分化形成成熟的脂肪细胞,ADIPOQ和PPARγ成脂标志基因均在分化过程中呈现上升的趋势。以此为模型,发现CIDEA基因随着分化天数的增加,表达水平逐渐升高,并且分化末期与前体脂肪细胞相比,差异极显著(P<0.01);分化第9天时,CIDEA基因与第6天相比略有下降,但差异不显著(P>0.05)。因此,CIDEA基因在脂肪组织特异表达以及分化末期高表达的结果提示其参与牦牛的脂肪沉积与脂肪细胞分化,这些数据为进一步解析牦牛CIDEA基因在脂肪代谢中的功能奠定了重要基础。
本研究成功克隆了金川牦牛CIDEA基因完整编码区序列,序列全长696 bp,其中CDS序列长度为684 bp,共编码227个氨基酸。生物信息学分析预测金川牦牛CIDEA蛋白是一个保守的碱性亲水蛋白,无信号肽与跨膜结构域,二级结构主要为α螺旋与无规则卷曲。组织表达分析结果显示,CIDEA基因在脂肪组织的表达量最高,且在脂肪细胞分化过程中呈现上升的趋势。由此可推测,CIDEA基因与牦牛脂肪细胞分化与脂肪沉积密切相关。
[1] 孟庆辉,陈永杏,董红敏,白加德,刘艳菊,郭青云,成振华. 牦牛分布特点及其种群数量[J].家畜生态学报,2017,38(3):80-85. doi:10.3969/j.issn.1673-1182.2017.03.017.
Meng Q H,Chen Y X,Dong H M,Bai J D,Liu Y J,Guo Q Y,Cheng Z H. The distribution characteristics and populations of yak[J].Journal of Domestic Animal Ecology,2017,38(3):80-85.
[2] 王国文. 青藏高原牦牛NPY对其极端生境的适应机制[D].兰州: 西北师范大学,2016.
Wang G W. Mechanism of NPY action on the adaptation to extreme habitats in Qinghai-Tibet plateau yak[D].Lanzhou:Northwest Normal University,2016.
[3] 褚敏. 营养胁迫条件下牦牛皮下脂肪和背肌差异microRNAs的筛选与鉴定[D].北京:中国农业科学院,2015.
Chu M. Identification and analysis of differential expressed microRNAs in subcutaneous and longissimus muscle of yak under nutrition pressure[D].Beijing:Chinese Academy of Agricultural Sciences,2015.
[4] 王瑞. Cidea和Cidec的转录调控以及在细胞脂肪生成中的作用[D].北京:北京协和医学院,2009.
Wang R. Transcriptional regulation of Cidea and Cidec and their function in cell lipogenesis[D].Beijing:Peking Union Medical College Hospital,2009.
[5] Fischer A W,Shabalina I G,Mattsson C L,Abreu-Vieira G,Cannon B,Nedergaard J,Petrovic N. UCP1 inhibition in Cidea-overexpressing mice is physiologically counteracted by brown adipose tissue hyperrecruitment[J].American Journal of Physiology:Endocrinology and Metabolism,2017,312(1):E72-E87. doi:10.1152/ajpendo.00284.2016.
[6] Abreu-Vieira G,Fischer A W,Mattsson C,de Jong J M A,Shabalina I G,Rydén M,Laurencikiene J,Arner P,Cannon B,Nedergaard J,Petrovic N. Corrigendum:Cidea improves the metabolic profile through expansion of adipose tissue[J]. Nature Communications,2016,7:12395. doi:10.1038/ncomms12395.
[7] Barneda D,Planas-Iglesias J,Gaspar M L,Mohammadyani D,Prasannan S,Dormann D,Han G S,Jesch S A,Carman G M,Kagan V,Parker M G,Ktistakis N T,Klein-Seetharaman J,Dixon A M,Henry S A,Christian M. The brown adipocyte protein CIDEA promotes lipid droplet fusion via a phosphatidic acid-binding amphipathic helix[J].eLife,2015,4(4):e07485. doi:10.7554/eLife.07485.
[8] Wu L Z,Zhou L K,Chen C,Gong J Y,Xu L,Ye J,Li D,Li P. Cidea controls lipid droplet fusion and lipid storage in brown and white adipose tissue[J].Science China Life Sciences,2014,57(1):107-116. doi: 10.1007/s11427-013-4585-y.
[9] 谢凤莲. 不同季节牦牛机体棕色脂肪组织相关基因表达差异及其脂肪酸组成成分的比较[D].西宁: 青海大学,2019.
Xie F L. The expression difference of genes related to brown adipose tissue of yak and comparison of fatty acid composition in different seasons[D].Xining:Qinghai University,2019.
[10] 郭一帆,陈佩杰,肖卫华. 肥胖状态下脂肪组织线粒体功能紊乱与运动调控[J].中国生物化学与分子生物学报,2020,36(10):1145-1150. doi:10.13865/j.cnki.cjbmb.2020.08.1196.
Guo Y F,Chen P J,Xiao W H. Mitochondrial dysfunction and exercise regulation in adipose tissue during obesity[J].Chinese Journal of Biochemistry and Molecular Biology, 2020,36(10):1145-1150.
[11] 梁莎莎,庞春英,邓廷贤,马小娅,陆杏蓉,段安琴,梁贤威. 奶水牛固醇携带蛋白2基因克隆、生物信息学分析及其组织表达检测[J].中国畜牧兽医,2018,45(9):2358-2367. doi:10.16431/j.cnki.1671-7236.2018.09.002.
Liang S S,Pang C Y,Deng T X,Ma X Y,Lu X R,Duan A Q,Liang X W. Cloning,bioinformatics and tissue expression analysis of SCP2 gene in dairy buffalo[J].China Animal Husbandry & Veterinary Medicine,2018,45(9):2358-2367.
[12] 黄韵琪,邓廷贤,唐辉,马小娅,梁莎莎,陆杏蓉,段安琴,王文文,庞春英,梁贤威. 水牛SND1基因克隆及生物信息学分析[J].中国畜牧兽医,2018,45(7):1759-1767. doi:10.16431/j.cnki.1671-7236.2018.07.004.
Huang Y Q,Deng T X,Tang H,Ma X Y,Liang S S,Lu X R,Duan A Q,Wang W W,Pang C Y,Liang X W. Cloning and bioinformatics analysis of SND1 gene in buffalo[J].China Animal Husbandry & Veterinary Medicine,2018,45(7):1759-1767.
[13] 龚大为,李平,朱大龙. 脂肪细胞因子研究进展[J].中国糖尿病杂志,2007,15(10):637-638. doi: 10.3321/j.issn:1006-6187.2007.10.025.
Gong D W,Li P,Zhu D L.Advance in study of adipocytokines[J].Chinese Journal of Diabetes,2007,15(10):637-638.
[14] 张一甫.大鼠肝再生中脂肪滴蛋白质组的差异分析[D].上海:复旦大学,2010.
Zhang Y F. Proteomic analysis of lipd droplets during rat liver regeneration[D].Shanghai:Fudan University,2010.
[15] 薛白,赵新全,张耀生.青藏高原天然草场放牧牦牛体重和体成分变化动态[J].动物营养学报,2005,17(2):54-57.doi:10.3969/j.issn.1006-267X.2005.02.011.
Xue B,Zhao X Q,Zhang Y S. Weight and body composition dynamic changes of yaks grazing on nature grassland in Qinghai-Tibetan plateau[J].Chinese Journal of Animal Nutrition,2005,17(2):54-57.
[16] 赵东宇,郁苗,汪文敏,任豪,廖榕玉,徐俐,周林康.Cidea和Cidec促进肝脏中脂类的积累[J].现代生物医学进展,2018,18(5):801-806.doi:10.13241/j.cnki.pmb.2018.05.001.
Zhao D Y,Yu M,Wang W M,Ren H,Liao R Y,Xu L,Zhou L K. Cidea and Cidec promote the lipid storage in liver[J].Progress in Modern Biomedicine,2018,18(5):801-806.
[17] He Q,Diao Y,Zhao T T,Hou B Y,Ngokana L D,Liang H,Nie J H,Tan P Z,Huang H,Li Y Z,Qi L,Zhao Y Y,Liu Y,Gao X,Zhou L Y. SREBP1c mediates the effect of acetaldehyde on Cidea expression in alcoholic fatty liver mice[J]. Scientific Reports,2018,8(1):191-205. doi:10.1038/s41598-018-19466-z.
[18] 李艳华.猪CIDE家族基因的克隆及功能研究[D].武汉:华中农业大学,2008.
Li Y H. Cloning and functional research of porcine CIDEs family[D].Wuhan:Huazhong Agricultural University,2008.
[19] 黄柳梅.CIDEa和CIDEc基因在肉鸡中的表达分析[D].南昌:江西农业大学,2016.
Huang L M. Expression analysis of CIDEa and CIDEc genes in broiler chickens[D].Nanchang:Jiangxi Agricultural University,2016.
[20] 邹菊红,江雨航,张叁保,高小童,申玉建,韦英明,黄大安,黄艳娜,蒋钦杨.山羊CIDEa基因克隆及组织表达分析[J].黑龙江畜牧兽医,2020(21):75-79,176.doi:10.13881/j.cnki.hljxmsy.2019.12.0332.
Zou J H,Jiang Y H,Zhang S B,Gao X T,Shen Y J,Wei Y M,Huang D A,Huang Y N,Jiang Q Y. Cloning and tissue expression analysis of goat CIDEa[J].Heilongjiang Animal Science and Veterinary Medicine,2020(21):75-79,176.
[21] Xu L,Zhou L K,Li P. CIDE proteins and lipid metabolism[J].Arteriosclerosis,Thrombosis,and Vascular Biology,2012,32(5):1094-1098. doi:10.1161/ATVBAHA.111.241489.
[22] 龚婧怡. Cidea以及Fsp27在脂肪代谢中的功能以及分子机制[D].北京: 清华大学,2010.
Gong J Y. Function and molecular mechanism of Cidea and Fsp27 in lipid metabolism[D].Beijing:Tsinghua University,2010.