根肿菌属原生动物界(Protozoa),根肿菌纲(Plasmodiophoromycetes),根肿菌目(Plasmodiophorales),根肿菌属(Plasmodiophora),对油菜、甘蓝等多种十字花科作物危害严重,调查表明田间受根肿病危害的油菜发病较轻减产30%左右,严重田块可造成油菜绝收[1]。根肿菌休眠孢子的抗逆性强,可在土壤中存活数十年之久,主要为害十字花科植物根部,其根表皮薄壁细胞受到根肿菌刺激后大量分裂和膨大,呈大小不一形状不规则的肿瘤[2];从微观层面上讲,寄主植物的一些正常生理代谢受到影响[3],诱导植物产生吲哚乙酸(Indole-3-acetic acid)、细胞分裂素(Cytokinin)、糖类(Carbohydrate)以及蛋白质(Protein),随即产生肿根。研究学者们对芜菁、甘蓝、萝卜等资源进行筛选鉴定后获得部分抗病信息:芜菁(Brassica. rapa,AA,2n=20)根肿病抗性是由单显性基因控制的数量性状遗传[4],具有小种特异性。甘蓝(Brassica oleracea,CC,2n=18)根肿病抗性是由多基因控制的数量遗传性状,且多为隐性遗传,抗感范围广、遗传规律较为复杂,由于不同定位研究使用不同的根肿病抗性源及不同的芸薹根肿菌分离株,导致定位起来比较困难。萝卜(Raphanus sativus)根肿病抗性普遍较强,大部分萝卜品种对根肿病抗病性可能是由单个显性位点控制,受显性主效基因控制,同时还可能存在其他微效抗性基因[5]。胡靖锋等[6]通过抗根肿病萝卜胞质不育系和甘蓝自交系杂交,获得抗根肿病AACC型染色体甘蓝型油菜材料ZZCZ13000。部分抗病基因已在育种中广泛应用:华中农大选育出首批具有应用价值的抗根肿病甘蓝型油菜(B. napus L.AACC,2n=38)杂交新品种华油杂62R(含抗4号生理小种抗病位点CRb)和常规新品种华双5R(含抗4号生理小种抗病位点PbBa8.1),对根肿病4号生理小种具有免疫抗性,为我国育种家提供了新材料。
随着后基因组时代的到来,第三代分子标记SNP应运而生。由LGC(Laboratory of the Government Chemist 英国政府化学家实验室)开发的竞争性等位基因特异性PCR(Kompetitive Allele Specific PCR,KASP)基因分型技术,可以对SNP和特定位点上的InDel进行精准的双等位基因判断,基于自己独特的ARMSPCR原理实现SNP分型,SNP在基因组中具有分布密度高、突变率低、二态性高、稳定性好、易于进行高通量自动化分析等特点[7],在水稻、小麦等作物性状基因的精细定位、分子辅助育种、种子资源鉴定等方面发挥重要作用。Han等[8]利用雄性不育突变体位点Ms-cd1开发的KASP标记,成为甘蓝Ms-cd1开发的首批KASP标记。张强等[9]利用KASP标记鉴定辣椒细胞质类型,为辣椒三系杂交育种的应用奠定了研究基础。Yang等[10]基于KASP标记技术将种质群体划分为籼型和粳型2个亚群体,有效地维护和利用水稻遗传资源。Fang等[11]利用六倍体小麦抗叶锈病基因Lr34第11外显子和第22外显子等位变异SNP位点开发的KASP标记,能加速小麦Lr34的筛选。Devran等[12]针对抗番茄斑萎病原菌Sw-5基因座开发了KASP标记,并在多个具有不同遗传背景的番茄基因型中进行验证,能成功筛选番茄斑萎病纯合子抗性植株、杂合子抗性植株和感病品种植株。
Crr1基因来源于欧洲饲料萝卜Siloga,目前已经从Siloga中鉴定出Crr1、Crr2和Crr4 3个抗病基因,分别位于A08、A01和A06染色体上,对根肿病4号生理小种具有抗性。对Crr1基因精细定位发现:Crr1可能包含2个基因位点,一个主要的根肿病抗性基因位点Crr1a和另一个作用较小的次要基因位点Crr1b[13],其中Crr1a编码TIR-NB-LRR抗病蛋白。Hatakeyama等[14]和Suwabe等[15]研究发现Crr2基因是Crr1和Crr4基因表达的增强剂,也就是说,只有与Crr2基因相互作用时,Crr1和Crr4才能对4号生理小种产生抗性。我国对根肿病的研究起步较晚,基于根肿病基因的KASP标记研究还不多,本试验中抗性亲本华双5R含有从芜菁转育来的抗病位点PbBa8.1,该位点包含一个抗病基因Crr1[16]。甘蓝型油菜(B. napus L.AACC,2n=38)基因组中LOC103834349基因与芜菁Crr1基因有较高的同源性,本研究对抗病亲本华双5R的Crr1基因与感病亲本LOC103834349基因进行克隆测序,发现部分SNP位点,针对第1486-1487上的非同义突变位点,开发了一套精准检测自交后代抗根肿病基因型的KASP分子标记,能够快速精准检测抗感甘蓝型油菜的基因型,大幅提高了甘蓝型油菜根肿病抗性育种效率。
1 材料和方法
1.1 试验材料
抗性亲本材料甘蓝型油菜华双5R(含根肿病4号生理小种抗病位点PbBa8.1,来自华中农业大学作物遗传育种研究所油菜研究室)为供体亲本。25个农艺性状好,含油量在48%~54%,油酸含量在70%~78%的甘蓝型油菜自交系(来自水稻油菜抗病育种湖南省重点实验室)为受体亲本进行杂交获得F1,F1后代经过自交获得F2群体。2020年10月13日将42个F2群体播种在上一年根肿病发病严重的田块中。
1.2 试验方法
1.2.1 田间表型鉴定 材料种植地上一年根肿病发病严重,发病率95%左右。播种前利用根肿菌18S rRNA特异引物对田间土壤病菌孢子含量进行荧光定量PCR测定,孢子数为5.1×103~104个/g。田间鉴定在2020年12月12日进行,将每个单株小心从土壤中拔出,仔细检查根部发病情况,根据发病与否进行登记挂牌。挑选经过田间抗病鉴定的42个F2群体的771个单株进行KASP分型检测。
1.2.2 DNA的提取 取0.1~0.5 g大小的叶片放入96孔板,置于冻干机中抽真空16 h后于研磨仪中研碎。在96孔板中每孔加入500 μL DNA提取液,振荡混匀后于75 ℃烘箱中温浴30 min。温浴结束后,4 000 r/min离心10 min,取96孔板中190 μL上清液至新的96孔板,并在新的96孔板中每孔加入190 μL异丙醇。将96孔板于-20 ℃冰箱中放置1 h,然后4 000 r/min离心10 min,弃上清液,置于50 ℃烘箱中烘干,加入500 μL超纯水溶解DNA,4 ℃保存用于后续的KASP检测。
1.2.3 KASP分子标记设计 在数据库中查找Crr1和LOC103834349序列间的SNP位点(http://www.ncbi.nlm.nih.gov/),选择位于编码区内第1 486-1 487上非同义突变位点,应用引物设计软件Batch Primer3设计KASP标记引物,包括3条引物:K07-X、K07-Y是2条等位基因特异性正向引物,5′端分别加上荧光序列标签VIC和FAM(下划线部分为荧光序列标签),3′末端碱基为SNP位点变异碱基,2条特异性引物末端碱基分别包括SNP位点的等位变异;K07-C为共同的反向引物(图1)。KASP-PCR反应LGC SNP line基因分型平台上进行,反应总体系为0.8 μL,100 μmol/L Primer由浓度为100 μmol/L的K07-X、K07-Y、K07-C与超纯水按12∶12∶30∶46的体积比混合得到。PCR反应混合物(0.8 μL)包含:100 μmol/L Primer 0.005 9 μL、2×KASP Master Mix 0.394 5 μL、超纯水0.399 5μL、DNA(干燥)20~50 ng。PCR程序为:94 ℃预变性15 min;95 ℃变性20 s,56~65 ℃退火延伸60 s,每循环退火延伸温度降低0.8 ℃,共计10个循环;94 ℃变性20 s,57 ℃退火延伸60 s,共30个循环。
KASP-PCR扩增产物反应完成后利用扫描仪Pherastar对KASP反应产物进行荧光数据读取,荧光扫描的结果自动转化成图形。
1.2.4 测序验证 根据LOC103834349和Crr1基因序列设计引物LOC-1(表1),该引物对可以扩增
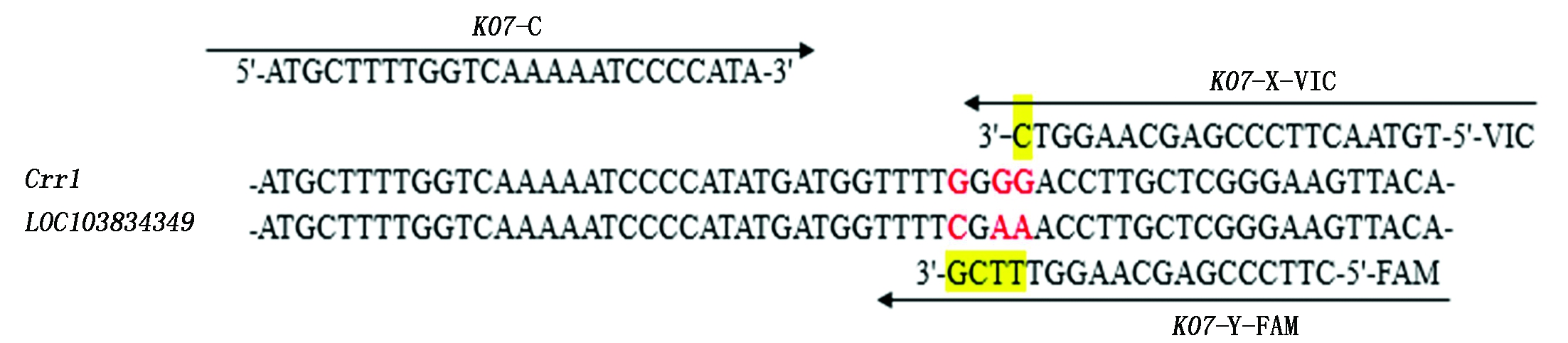
图1 用于开发标记的SNP位点及其引物设计
Fig.1 SNP locus for the development of markers and primer design
表1 KASP标记引物及测序引物
Tab.1 The KASP primers and sequencing primers
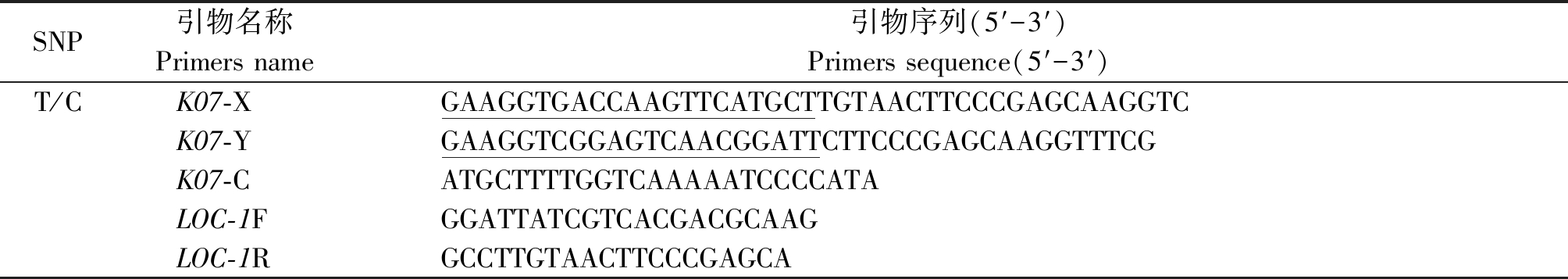
SNP引物名称Primers name引物序列(5′-3′)Primers sequence(5′-3′)T/CK07-XGAAGGTGACCAAGTTCATGCTTGTAACTTCCCGAGCAAGGTCK07-YGAAGGTCGGAGTCAACGGATTCTTCCCGAGCAAGGTTTCGK07-CATGCTTTTGGTCAAAAATCCCCATALOC-1FGGATTATCGTCACGACGCAAGLOC-1RGCCTTGTAACTTCCCGAGCA
注:下划线为荧光序列标签。
Note: The underline is the fluorescent sequence label.
KASP标记SNP位点上下游281 bp的基因序列。取田间鉴定抗病和感病的油菜各6株,提取DNA后用上面引物进行PCR扩增。PCR反应体系25 μL,包括1.1×T3 PCR Mix 22 μL(北京擎科生物科技有限公司)、10 μmol/L上下游引物各1 μL、478 ng/μL基因组DNA 1 μL。扩增程序为98 ℃预变性2 min;98 ℃变性10 s,退火温度60 ℃退火10 s,72 ℃延伸10 s,34个循环;最后72 ℃延伸2 min,于4 ℃保存。PCR产物由北京擎科生物科技有限公司进行测序,测序结果使用软件DNAMAN进行序列比对。
2 结果与分析
2.1 田间鉴定
对种植于大田的F2植株逐一拔出用目测法检查根系生长情况,确定其抗感表型。从42个F2群体中共鉴定了771个单株,其中有456株根系正常,没有染病。有315株根部有肿块,为染病植株(图2)。

左侧为抗性材料;右侧为感病材料。
The left is the clubroot resistant plants;The right is the clubroot susceptible plants.
图2 田间抗感材料对照
Fig.2 Comparison of clubroot resistant and susceptible plants
2.2 KASP标记开发与验证
利用该等位变异SNP位点设计的KASP标记对经过田间鉴定的771个单株进行分型检测。检测结果显示,待检测材料清楚地分为两类(图3):聚合在接近X轴的显示蓝色的样本的基因型为连接VIC荧光标签序列的纯合抗病AA基因型,聚合在接近Y轴上的显示红色的样本的基因型为连接FAM荧光标签序列的纯合感病GG基因型,位于坐标轴接近45度轴心显示紫色的样本基因型表示含有A和G的杂合基因型,左下角显示黑色的样本为空白对照。其中315个单株含有纯合感病基因型GG,322个单株含有纯合抗病基因型AA,134个单株含有基因型GA。对编号为2033的F2群体中125株材料的KASP分型情况(表2)进行χ2检测,其中纯合抗病基因型AA 30株,纯合感病基因型GG 33株,杂合基因型GA 62株,经χ2检测,抗病材料与感病材料符合3∶1理论值,抗病纯合基因型、抗病杂合基因型与感病纯合基因型的比值符合1∶2∶1理论值,说明该抗病位点为显性单基因抗病位点。
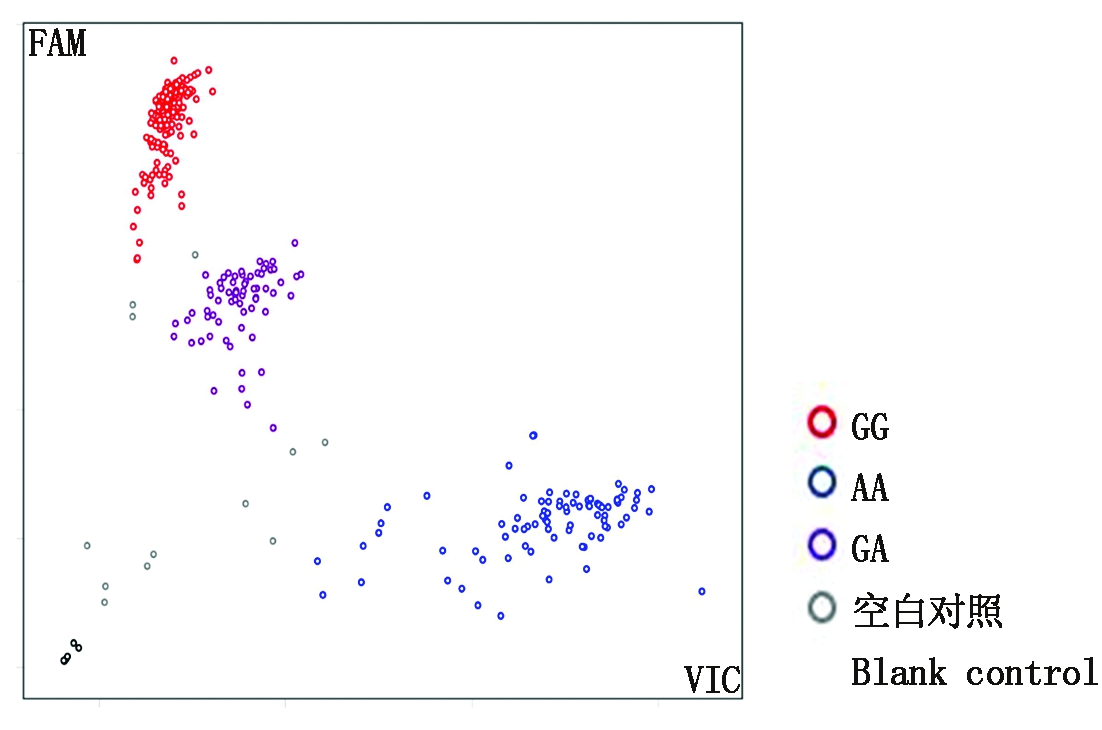
图3 KASP标记对771份油菜单株分型
Fig.3 KASP marking classification map of 771 Brassica napus
表2 编号2033 F2群体KASP分型与田间鉴定结果
Tab.2 KASP typing and field identification results of the No.2033 F2 population
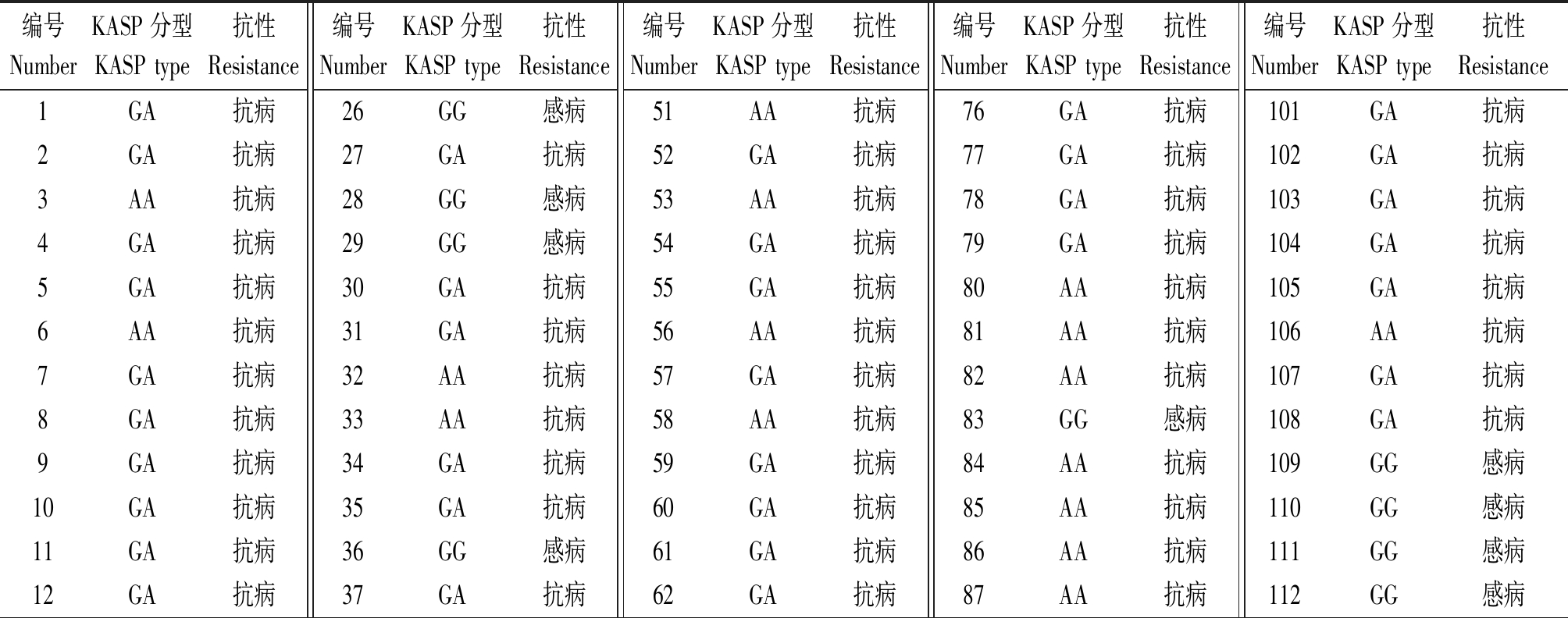
编号NumberKASP分型KASP type抗性Resistance编号NumberKASP分型KASP type抗性Resistance编号NumberKASP分型KASP type抗性Resistance编号NumberKASP分型KASP type抗性Resistance编号NumberKASP分型KASP type抗性Resistance1GA抗病26GG感病51AA抗病76GA抗病101GA抗病2GA抗病27GA抗病52GA抗病77GA抗病102GA抗病3AA抗病28GG感病53AA抗病78GA抗病103GA抗病4GA抗病29GG感病54GA抗病79GA抗病104GA抗病5GA抗病30GA抗病55GA抗病80AA抗病105GA抗病6AA抗病31GA抗病56AA抗病81AA抗病106AA抗病7GA抗病32AA抗病57GA抗病82AA抗病107GA抗病8GA抗病33AA抗病58AA抗病83GG感病108GA抗病9GA抗病34GA抗病59GA抗病84AA抗病109GG感病10GA抗病35GA抗病60GA抗病85AA抗病110GG感病11GA抗病36GG感病61GA抗病86AA抗病111GG感病12GA抗病37GA抗病62GA抗病87AA抗病112GG感病
表2(续)
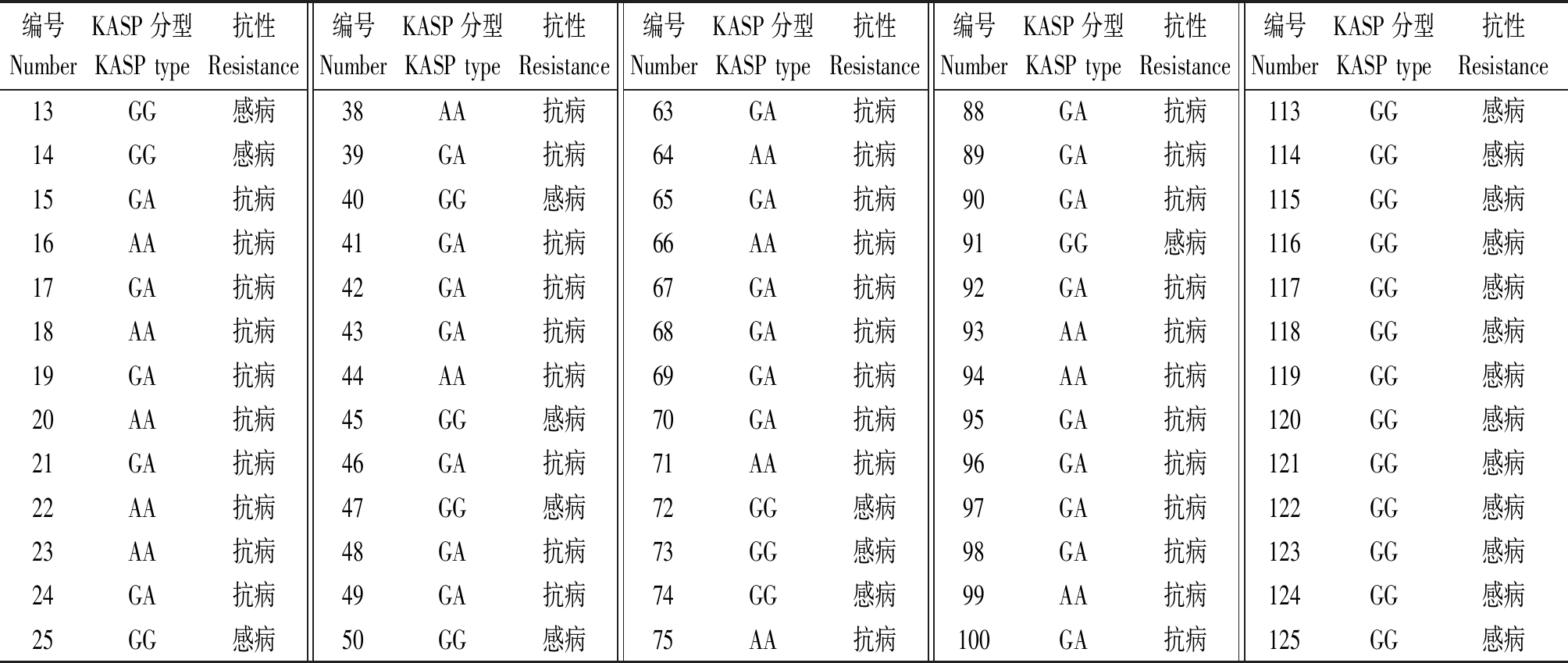
编号NumberKASP分型KASP type抗性Resistance编号NumberKASP分型KASP type抗性Resistance编号NumberKASP分型KASP type抗性Resistance编号NumberKASP分型KASP type抗性Resistance编号NumberKASP分型KASP type抗性Resistance13GG感病38AA抗病63GA抗病88GA抗病113GG感病14GG感病39GA抗病64AA抗病89GA抗病114GG感病15GA抗病40GG感病65GA抗病90GA抗病115GG感病16AA抗病41GA抗病66AA抗病91GG感病116GG感病17GA抗病42GA抗病67GA抗病92GA抗病117GG感病18AA抗病43GA抗病68GA抗病93AA抗病118GG感病19GA抗病44AA抗病69GA抗病94AA抗病119GG感病20AA抗病45GG感病70GA抗病95GA抗病120GG感病21GA抗病46GA抗病71AA抗病96GA抗病121GG感病22AA抗病47GG感病72GG感病97GA抗病122GG感病23AA抗病48GA抗病73GG感病98GA抗病123GG感病24GA抗病49GA抗病74GG感病99AA抗病124GG感病25GG感病50GG感病75AA抗病100GA抗病125GG感病
AA分型和杂合的GA分型单株田间检测均为抗病表型,GG分型均为感病表型,KASP分型结果与田间鉴定结果一致。比对结果说明,该KASP标记对根肿病抗、感植株进行正确分型,可作为新型分子标记应用于根肿病的分子标记辅助选择。
2.3 测序验证
取抗、感亲本材料各6株,提取DNA后扩增KASP标记位点上下游281 bp碱基序列。PCR产物进行测序验证,结果显示,抗病亲本在KASP引物设计位点为AA碱基,感病亲本则为GG碱基(图4),测序结果证明了KASP分型结果准确性。

1~6.感病亲本;7~12.抗病亲本。
1-6.Susceptible parents;7-12.Resistant parents.
图4 抗、感亲本KASP标记位点DNA序列测序结果
Fig.4 Sequencing results of KASP marker site DNA sequence in resistant and susceptible parents
3 讨论与结论
分子标记可以避免环境条件对杂种优势类群分类的影响,对于一些系谱不清或者混合的材料可以有根据性的划分为不同类群。针对杂种优势类群,人们开发了许多标记系统,如RAPD、AFLP、SSR和SNP,以及一些经济有效的基因分型平台来检测SNP位点,如Golden Gate[17]和Infinium[18]平台,TaqMan和KASP平台[19]。KASP分析在用于基因分型的SNP数量方面提供了灵活性,该方法准确度高,成本低,这一特征使KASP标记比其他SNP基因分型分析更具优势。本研究针对与根肿病抗性显著关联的SNP位点开发了KASP标记,能有效将杂交后代的基因型分类,在检测的771个样本中,315个单株为纯合感病基因型GG,322个单株为纯合抗病基因型AA,134个单株为杂合抗病基因型GA,与田间检测结果一致。其中编号2033的F2群体125株材料中纯合抗性基因型AA 30株,纯合感病基因型GG 33株,杂合基因型GA 62株。经χ2检测,抗病材料与感病材料符合3∶1比例,抗病纯合基因型、抗病杂合基因型与感病纯合基因型的比值符合1∶ 2∶1理论值,说明该抗病位点为显性单基因抗病位点,与预期相符。
前人以白菜(B.rapa,AA,2n=20)为根肿病抗性材料,定位了10个CR基因[15,20-28],Crr1、Crr2、Crr3、Crr4、CRa、CRb、CRc、CRd、CRk、CRs,以及5个抗病位点,PbBa1.1、PbBa3.1、PbBa3.2、PbBa3.3、PbBa8.1。其中CRa是首个被人工克隆的根肿病基因[29];在萝卜(R. sativus)根肿病抗性相关的QTL定位检测中,检测出RsCr1、RsCr2、RsCr3、RsCr4和RsCr5 5个抗性基因[30]。华双5R对根肿病抗性的产生是由于从芜菁ECD04中导入了含有PbBa8.1抗性位点的芜菁基因组片段,该片段含有根肿病抗性基因Crr1。根肿病抗性由2种途径控制,一种是在抗性中起共同调控作用的途径,另一种是起增强抗性的额外途径。这2种途径揭示了关于根肿病抗性进化的假说,一是根肿病抗性最初是由单一的主效基因控制,在芸薹属植物基因组的进化过程中,主效基因分化为2个功能不同的重复基因;另一种是抗性基因最初聚集在某一原始基因组区域,由于外界环境等其他因素的影响导致染色体重新排列后分布在不同的基因组区域,在拟南芥(Arabidopsis thaliana)的某些基因组区域已经观察到抗病基因的聚类。其中,Crr1和Crr2 2个主要QTL区域在拟南芥第4号染色体的一个小区域内存在重叠,该区域属于拟南芥基因组抗病基因簇MRCs之一。这些结果表明,根肿病抗性基因起源于一个共同祖先基因组MRC,随后在进化过程中分布到不同区域。甘蓝型油菜(B.napus L.AACC,2n=38)是由白菜(B.rapa,AA,2n=20)与甘蓝(B.oleracea,CC,2n=18)通过自然杂交和染色体组自然加倍后形成,在进化的过程中,芜菁ECD04保留了根肿病抗性,而甘蓝型油菜散失了根肿病抗性,推测可能原因之一是在漫长的进化过程中甘蓝型油菜基因组中LOC103834349基因丢失了部分序列,从而使其根肿病抗性散失了。
本研究针对甘蓝型油菜LOC103834349基因和根肿病抗病亲本Crr1基因开发了一个高通量、低成本的根肿病KASP分子标记。田间表型验证表明该标记准确可靠,可以在短时间内对大量材料进行快速可靠的分析鉴定,提高油菜根肿病抗性育种效率,该标记可作为一个新的高通量、高选择效率的分子标记,能快速准确筛选抗根肿病材料。
[1] 王惟萍,柴阿丽,石延霞,谢学文,李宝聚. 基于傅里叶变换红外光谱的大白菜根肿病定量测评[J].光谱学与光谱分析,2015,35(5):1243-1247. doi:10.3964/j.issn.1000-0593(2015)05-1243-05.
Wang W P,Chai A L,Shi Y X,Xie X W,Li B J. Quantitative detection of Chinese cabbage clubroot based on FTIR spectroscopy[J].Spectroscopy and Spectral Analysis,2015,35(5):1243-1247.
[2] 费维新,HWANG Sheaufang,王淑芬,吴晓芸,高智谋,李强生,侯树敏,荣松柏,江莹芬. 根肿菌生理小种鉴定与甘蓝型油菜品种资源的抗性评价[J].中国油料作物学报,2016,38(5):626-639. doi:10.7505/j.issn.1007-9084.2016.05.013.
Fei W X,Sheaufang H,Wang S F,Wu X Y,Gao Z M,Li Q S,Hou S M,Rong S B,Jiang Y F. Pathotype identification of Plasmodiophora brassicae and resistance of rapeseed variety resources to clubroot disease[J].Chinese Journal of Oil Crop Sciences,2016,38(5):626-639.
[3] 张炜. 甘蓝抗根肿病的指标体系构建[D].重庆:西南大学,2019.
Zhang W. Construction of indicator system for cabbage resistance to clubroot disease[D].Chongqing: Southwest University,2019.
[4] 王秀珍. 利用属间杂交将萝卜属的根肿病抗性转移到甘蓝型油菜[D].北京:中国农业科学院,2019.
Wang X Z.Intergeneric crosses for the transfer of resistance to the clubroot from Raphanus sativus to Brassica napus[D].Beijing: Chinese Academy of Agricultural Sciences,2019.
[5] 洪雅婷,沈向群,陈永浩,马士芳,郭艳峰,李林. 四季萝卜(Raphanus sativus var. radicula)抗根肿病遗传规律[J].西北农业学报,2013,22(7):138-142.
Hong Y T,Shen X Q,Chen Y H,Ma S F,Guo Y F,Li L.The study on inheritance of clubroot resistance in Radish(Raphanus sativus var. radicula)[J].Acta Agriculturae Boreali-Occidentalis Sinica,2013,22(7):138-142.
[6] 胡靖锋,杨红丽,徐学忠,和江明,赵颖. 芸薹属萝卜胞质不育抗根肿病种质创新研究[J].浙江农业学报,2015,27(8):1394-1398. doi:10.3969/j.issn.1004-1524.2015.08.14.
Hu J F,Yang H L,Xu X Z,He J M,Zhao Y. Study on the CMS germplasm innovation of clubroot resistance in Brassica[J].Acta Agriculturae Zhejiangensis,2015,27(8):1394-1398.
[7] Li C T,Zhang S H,Li L,Chen J Z,Liu Y,Zhao S M. Selection of 29 highly informative InDel markers for human identification and paternity analysis in Chinese Han population by the SNPlex genotyping system[J].Molecular Biology Reports,2012,39(3):3143-3152. doi:10.1007/s11033-011-1080-z.
[8] Han F Q,Zhang X L,Yuan K W,Fang Z Y,Yang L M,Zhuang M,Zhang Y Y,Wang Y,Liu Y M,Li Z S,Lü H. A user-friendly KASP molecular marker developed for the DGMS-based breeding system in Brassica oleracea species[J].Molecular Breeding,2019,39(6):1-7.doi:10.1007/s11032-019-0987-2.
[9] 张强,张涛,常晓轲,韩娅楠,程志芳,刘卫,王彬,姚秋菊. 一个辣椒胞质雄性不育SCAR标记的KASP转化及其应用[J].华北农学报,2019,34(5):93-98. doi:10.7668/hbnxb.20190131.
Zhang Q,Zhang T,Chang X K,Han Y N,Cheng Z F,Liu W,Wang B,Yao Q J.Transformation and application of one molecular marker from SCAR to KASP in pepper CMS[J].Acta Agriculturae Boreali-Sinica,2019,34(5):93-98.
[10] Yang G L,Chen S P,Chen L K,Sun K,Huang C H,Zhou D H,Huang Y T,Wang J F,Liu Y Z,Wang H,Chen Z Q,Guo T. Development of a core SNP arrays based on the KASP method for molecular breeding of rice[J].Rice,2019,12(1):1-18. doi:10.1186/s12284-019-0272-3.
[11] Fang T L,Lei L,Li G Q,Powers C,Hunger R M,Carver B F,Yan L L. Development and deployment of KASP markers for multiple alleles of Lr34 in wheat[J].Theoretical and Applied Genetics,2020,133(7):2183-2195. doi:10.1007/s00122-020-03589-x.
[12] Devran Z,Kahveci E. Development and validation of a user-friendly KASP marker for the Sw-5 locus in tomato[J].Australasian Plant Pathology,2019,48(5):503-507. doi:10.1007/s13313-019-00651-1.
[13] Suwabe K,Suzuki G,Nunome T,Hatakeyama K,Mukai Y,Fukuoka H,Matsumoto S. Microstructure of a Brassica rapa genome segment homoeologous to the resistance gene cluster on Arabidopsis chromosome 4[J].Breeding Science,2012,62(2):170-177. doi:10.1270/jsbbs.62.170.
[14] Hatakeyama K,Suwabe K,Tomita R N,Kato T,Nunome T,Fukuoka H,Matsumoto S. Identification and characterization of Crr1a,a gene for resistance to clubroot disease(Plasmodiophora brassicae Woronin)in Brassica rapa L.[J].PLoS One,2013,8(1): e54745. doi:10.1371/journal.pone.0054745.
[15] Suwabe K,Tsukazaki H,Iketani H,Hatakeyama K,Kondo M,Fujimura M,Nunome T,Fukuoka H,Hirai M,Matsumoto S. Simple sequence repeat-based comparative genomics between Brassica rapa and Arabidopsis thaliana:The genetic origin of clubroot resistance[J].Genetics,2006,173(1):309-319. doi:10.1534/genetics.104.038968.
[16] 战宗祥,江莹芬,朱紫媛,张春沙,杨庆勇,李倩,侯照科,龚建芳,程雨贵,吴江生,傅廷栋,周永明,朴钟云,张椿雨. 与位点PbBa8.1紧密连锁分子标记的开发及甘蓝型油菜根肿病抗性育种[J].中国油料作物学报,2015,37(6):766-771.doi:10.7505/j.issn.1007-9084.2015.06.005.
Zhan Z X,Jiang Y F,Zhu Z Y,Zhang C S,Yang Q Y,Li Q,Hou Z K,Gong J F,Cheng Y G,Wu J S,Fu T D,Zhou Y M,Piao Z Y,Zhang C Y.Development of molecular markers closely linked to PbBa8.1 and breeding for resistance to root swelling of Brassica napus[J].Chinese Journal of Oil Crop Sciences,2015,37(6):766-771.
[17] Morbitzer R,Elsaesser J,Hausner J,Lahaye T. Assembly of custom TALE-type DNA binding domains by modular cloning[J].Nucleic Acids Research,2011,39(13):5790-5799. doi:10.1093/nar/gkr151.
[18] Dedeurwaerder S,Defrance M,Calonne E,Denis H,Sotiriou C,Fuks F. Evaluation of the infinium methylation 450K technology[J].Epigenomics,2011,3(6):771-784. doi:10.2217/epi.11.105.
[19] Steele K A,Quinton-Tulloch M J,Amgai R B,Dhakal R,Khatiwada S P,Vyas D,Heine M,Witcombe J R. Accelerating public sector rice breeding with high-density KASP markers derived from whole genome sequencing of indica rice[J].Molecular Breeding,2018,38(4):1-13. doi:10.1007/s11032-018-0777-2.
[20] Matsumoto E,Yasui C K,Ohi M,Tsukada M. Linkage analysis of RFLP markers for clubroot resistance and pigmentation in Chinese cabbage(Brassica rapa ssp. pekinensis)[J].Euphytica,1998,104(2):79-86. doi:10.1023/A:1018370418201.
[21] Suwabe K,Tsukazaki H,Iketani H,Hatakeyama K,Fujimura M,Nunome T,Fukuoka H,Matsumoto S,Hirai M. Identification of two loci for resistance to clubroot(Plasmodiophora brassicae Woronin)in Brassica rapa L.[J].Theoretical and Applied Genetics,2003,107(6):997-1002. doi:10.1007/s00122-003-1309-x.
[22] Hirai M,Harada T,Kubo N,Tsukada M,Suwabe K,Matsumoto S. A novel locus for clubroot resistance in Brassica rapa and its linkage markers[J].Theoretical and Applied Genetics,2004,108(4):639-643. doi:10.1007/s00122-003-1475-x.
[23] Piao Z Y,Deng Y Q,Choi S R,Park Y J,Lim Y P. SCAR and CAPS mapping of CRb,a gene conferring resistance to Plasmodiophora brassicae in Chinese cabbage(Brassica rapa ssp. pekinensis)[J].Theoretical and Applied Genetics,2004,108(8):1458-1465. doi:10.1007/s00122-003-1577-5.
[24] Sakamoto K,Saito A,Hayashida N,Taguchi G,Matsumoto E. Mapping of isolate-specific QTLs for clubroot resistance in Chinese cabbage(Brassica rapa L. ssp. pekinensis)[J].Theoretical and Applied Genetics,2008,117(5):759-767. doi:10.1007/s00122-008-0817-0.
[25] Chen J J,Jing J,Zhan Z X,Zhang T,Zhang C Y,Piao Z Y. Identification of novel QTLs for isolate-specific partial resistance to Plasmodiophora brassicae in Brassica rapa[J].PLoS One,2013,8(12):e85307. doi:10.1371/journal.pone.0085307.
[26] Pang W X,Liang S,Li X N,Li P P,Yu S,Lim Y P,Piao Z Y. Genetic detection of clubroot resistance loci in a new population of Brassica rapa[J].Horticulture, Environment, and Biotechnology,2014,55(6):540-547. doi:10.1007/s13580-014-0079-5.
[27] Pang W X,Fu P Y,Li X N,Zhan Z X,Yu S,Piao Z Y. Identification and mapping of the clubroot resistance gene CRd in Chinese cabbage(Brassica rapa ssp. pekinensis)[J].Frontiers in Plant Science,2018,9:653. doi:10.3389/fpls.2018.00653.
[28] Hatakeyama K,Niwa T,Kato T,Ohara T,Kakizaki T,Matsumoto S. The tandem repeated organization of NB-LRR genes in the clubroot-resistant CRb locus in Brassica rapa L.[J].Molecular Genetics and Genomics,2017,292(2):397-405. doi:10.1007/s00438-016-1281-1.
[29] 付蓉.分子标记辅助选择在油菜抗根肿病和高油酸育种中的应用[D].武汉:华中农业大学,2019.
Fu R. Application of molecular marker assisted selection clubroot in resistance and high oleic acid breeding Brassica napus [D].Wuhan:Huazhong Agricultural University,2019.
[30] 甘彩霞.萝卜高密度遗传图谱的构建及抗根肿病的QTL定位[C]//中国园艺学会. 中国园艺学会2018年学术年会论文摘要集.青岛:中国园艺学会,2018:1.
Gan C X.Construction of high-density genetic map of radish and QTL mapping of resistance to root swelling[C]//Abstracts of Papers of the 2018 Annual Conference of the Chinese Horticultural Society.Qingdao:Chinese Horticultural Society,2018:1.