氮(N)是植物生长发育和产量形成所必需的大量营养元素。然而,由于在土壤中的有效性较低,N常常是陆地生态系统中最有限的养分之一。氮肥的过量施用不仅造成能源浪费,而且增加了农业成本,同时造成了严重的环境污染[1-2]。在大多数耕地土壤中,硝态氮![]() 和铵态氮
和铵态氮![]() 是高等植物吸收氮的主要形式。氮同化的第一步是植物从土壤中吸收铵盐或硝酸盐进入到根细胞中。
是高等植物吸收氮的主要形式。氮同化的第一步是植物从土壤中吸收铵盐或硝酸盐进入到根细胞中。![]() 的同化比
的同化比![]() 需要更少的能量,因此当植物缺氮时,
需要更少的能量,因此当植物缺氮时,![]() 首先被吸收[3-4]。然而,植物过度吸收
首先被吸收[3-4]。然而,植物过度吸收![]() 会对自身产生毒害作用[5],因此,必须严格调控
会对自身产生毒害作用[5],因此,必须严格调控![]() 的吸收和代谢过程。
的吸收和代谢过程。
植物![]() 的吸收和转运过程受到质膜铵转运蛋白(AMT)的严格调控。AMT基因家族系统发育分析研究表明,该家族可以分为2个亚家族:AMT1亚家族(AMT1簇)和AMT2亚家族(AMT2/3/4簇)[6-7]。目前,拟南芥和水稻AMT家族基因研究得比较透彻。拟南芥AMT家族共包含2个亚家族(AMT1和AMT2),其中AMT1亚家族包含5个成员:AtAMT1;1~AtAMT1;5;AMT2亚家族仅包括AMT2;1。根据动力学研究,植物有2种不同类型的
的吸收和转运过程受到质膜铵转运蛋白(AMT)的严格调控。AMT基因家族系统发育分析研究表明,该家族可以分为2个亚家族:AMT1亚家族(AMT1簇)和AMT2亚家族(AMT2/3/4簇)[6-7]。目前,拟南芥和水稻AMT家族基因研究得比较透彻。拟南芥AMT家族共包含2个亚家族(AMT1和AMT2),其中AMT1亚家族包含5个成员:AtAMT1;1~AtAMT1;5;AMT2亚家族仅包括AMT2;1。根据动力学研究,植物有2种不同类型的![]() 吸收体系。一种是高亲和转运系统(HATS),该系统主要用于铵离子浓度较低(μmol/L级别)时;另一种是低亲和转运系统(LATS),主要作用在铵离子浓度较高(mmol/L级别)时[8]。AtAMT1;1~AtAMT1;3在拟南芥中具有高亲和力[9]。拟南芥AMT2;1属于低亲和
吸收体系。一种是高亲和转运系统(HATS),该系统主要用于铵离子浓度较低(μmol/L级别)时;另一种是低亲和转运系统(LATS),主要作用在铵离子浓度较高(mmol/L级别)时[8]。AtAMT1;1~AtAMT1;3在拟南芥中具有高亲和力[9]。拟南芥AMT2;1属于低亲和![]() 转运体,而酵母的AMT2则是高亲和力
转运体,而酵母的AMT2则是高亲和力![]() 转运体[10]。AMT家族成员各自行使不同的功能,拟南芥AMT1;1~AMT1;3和AMT1;5主要在根部表达,而AMT1;4在花药中特异表达;AMT2;1主要功能是负责铵从根向地上部的转运[11-13]。缺氮时,AtAMT1;1和AtAMT1;3占根吸收
转运体[10]。AMT家族成员各自行使不同的功能,拟南芥AMT1;1~AMT1;3和AMT1;5主要在根部表达,而AMT1;4在花药中特异表达;AMT2;1主要功能是负责铵从根向地上部的转运[11-13]。缺氮时,AtAMT1;1和AtAMT1;3占根吸收![]() 能力的30%~35%,而AtAMT1;2则占18%~26%[14]。水稻中共有10个AMT成员,其中的OsAMT1;1、OsAMT1;2和OsAMT1;3负责根系铵的吸收。在缺氮条件下,OsAMT1;3在根中表达量最强[9,15]。除了拟南芥和水稻,AMT基因已经在多种植物中被鉴定和克隆,其中包括番茄[16-17]、日本莲[18]、玉米[19]、高粱[7]、小麦[20]、棉花[21]、杨树[22-23]、生菜[24]和松树[25]等。
能力的30%~35%,而AtAMT1;2则占18%~26%[14]。水稻中共有10个AMT成员,其中的OsAMT1;1、OsAMT1;2和OsAMT1;3负责根系铵的吸收。在缺氮条件下,OsAMT1;3在根中表达量最强[9,15]。除了拟南芥和水稻,AMT基因已经在多种植物中被鉴定和克隆,其中包括番茄[16-17]、日本莲[18]、玉米[19]、高粱[7]、小麦[20]、棉花[21]、杨树[22-23]、生菜[24]和松树[25]等。
甘蓝型油菜(Brassica napus L.,AnAnCnCn,2n=4x=38)是由二倍体的白菜(Brassica rapa L.,ArAr,2n=2x=20)和二倍体的甘蓝(Brassica oleracea L.,CoCo,2n=2x=18)经过天然的远缘杂交形成的异源四倍体,其基因组内部存在大量的重复序列和同源染色体片段,这导致油菜基因组普遍存在基因多拷贝现象[26-27]。目前,甘蓝型油菜AMT家族基因尚未得到全面鉴定和系统解析,2014年甘蓝型油菜基因组测序的完成使AMT家族基因的鉴定工作成为可能。油菜是我国重要的油料作物,需氮量较大;同时油菜对过量![]() 非常敏感[28]。生态系统中的
非常敏感[28]。生态系统中的![]() 水平主要受土壤的化学性质、pH值、温度、有机质、氧化还原状态、光照和CO2影响[29]。我国油菜的主产区位于长江中下游,降雨量大易造成渍水,在硝酸还原酶的作用下产生
水平主要受土壤的化学性质、pH值、温度、有机质、氧化还原状态、光照和CO2影响[29]。我国油菜的主产区位于长江中下游,降雨量大易造成渍水,在硝酸还原酶的作用下产生![]() 整体土壤偏酸性;局部地区过量施用铵态氮肥使得一段时期内农田土壤中的
整体土壤偏酸性;局部地区过量施用铵态氮肥使得一段时期内农田土壤中的![]() 浓度过高[30]。在这些因素的作用下,实际生产中可能出现油菜铵毒害现象。旱地作物小麦耐铵毒的机制已有报道[31],但在油菜中尚无相关研究。本研究首先在异源四倍体甘蓝型油菜中鉴定AMT家族基因,然后全面系统地解析BnaAMTs基因的基本信息、保守结构域、进化关系、染色体定位等信息,并进行表达分析,为进一步克隆和利用基因改良甘蓝型油菜的氮素利用效率和铵毒抗性提供一定的理论基础。
浓度过高[30]。在这些因素的作用下,实际生产中可能出现油菜铵毒害现象。旱地作物小麦耐铵毒的机制已有报道[31],但在油菜中尚无相关研究。本研究首先在异源四倍体甘蓝型油菜中鉴定AMT家族基因,然后全面系统地解析BnaAMTs基因的基本信息、保守结构域、进化关系、染色体定位等信息,并进行表达分析,为进一步克隆和利用基因改良甘蓝型油菜的氮素利用效率和铵毒抗性提供一定的理论基础。
1 材料和方法
1.1 试验材料
供试材料为冬油菜品种湘油15,由湖南农业大学官春云院士提供。
1.2 试验方法
1.2.1 甘蓝型油菜AMT家族基因的鉴定 利用拟南芥AMT氨基酸序列为种子序列进行Blast分析,在白菜、甘蓝、甘蓝型油菜等植物中寻找AMT基因。在本研究中,使用了以下数据库:拟南芥信息资源数据库(TAIR)(https://www.arabidopsis.org/)用于拟南芥氨基酸序列查找,芸薹属数据库(BRAD)v.1.1(http://brassicadb.org/brad/)[32]用于白菜、甘蓝以及甘蓝型油菜氨基酸序列搜索。根据以下标准对芸薹属植物中的基因进行了命名:种名+染色体+同源基因的名称。例如,BnaA1.AMT1;4是指甘蓝型油菜A1染色体上拟南芥AMT1;4的同源基因。
1.2.2 AMT蛋白的分子特性鉴定 使用ExPASy ProtoParam(http://www.expasy.org/tools/protparam.html)[33]计算了各AMT蛋白的氨基酸数目和组成、分子质量(MW)、理论等电点(pI)、平均亲水系数(GRAVY)和不稳定性指数。利用THMMM(http://www.cbs.dtu.dk/services/TMHMM/)和TMpred(https://embnet.vital-it.ch/software/TMPRED_form.html)[34]预测氨基酸序列的跨膜结构域(TM)数目。
1.2.3 AMT家族基因进化树的构建 利用MEGA 7中的ClustalW功能,对20个甘蓝型油菜AMT蛋白序列、6个拟南芥AMT蛋白序列、9个白菜AMT蛋白序列和10个甘蓝的AMT蛋白序列进行比对,根据序列比对的结果,采用邻近法(NJ)构建系统进化树,Bootstrap值设定为1 000[35-36]。
1.2.4 AMT家族基因的结构分析 为了研究拟南芥和芸薹属作物AMT蛋白的结构差异,将所有的AMT蛋白序列置于MEME v5.1.1[37](http://meme-suite.org/tools/meme)中比对,以表征保守结构域。除了下列参数外,其余参数使用默认设置:最佳图案宽度设置为6~50 bp,最大保守结构域数量设置为10。使用2种不同的工具分析蛋白质的二级结构:GOR4[38](https://npsa-prabi.ibcp.fr/cgibin/page=npsagor4.html/)和PSIPRED[39](http://bioinf.cs.ucl.ac.uk/PSIPRED/)。利用在线网站Phyre2[40](http://www.sbg.bio.ic.ac.uk/phyre2/html)进行同源建模,以构建蛋白质的三维结构。
1.2.5 AMT家族基因的染色体定位分析 为了确定AMT基因在染色体上的位置和方向,将完整的AMT基因核苷酸序列进行BlastTn搜索,从而确定了AMT基因在每个染色体上的起始位置。使用MapInspect v.2010(http://www.soft.com/./MapInspect.html)作图,描绘AMT基因的基因组位置。串联重复基因是指100 kb基因组区域内的2个或更多基因的阵列[41]。
1.2.6 胁迫处理 营养液培养试验在郑州大学农学院光照培养室进行,参数设置如下:温度为22 ℃,光照周期为14 h(光照)/10 h(黑暗),光照强度为300~320 μmol/(m2·s),湿度为60%~75%。选取大小一致的油菜种子,用1%的NaClO灭菌10 min,冲洗干净后,用灭菌纯水于4 ℃下浸泡过夜,然后将种子播种到育苗盘固定着的纱布上,育苗盘中加满纯水。7 d后将长势一致的幼苗移栽至营养液培养,营养液采用Hoagland配方[42]。进行如下试验:①不同氮形态处理:幼苗在2 mmol/L NH4NO3中生长10 d后,氮饥饿3 d,然后分别转移到含硝态氮(N,9 mmol/L ![]() 和铵态氮(A,9 mmol/L
和铵态氮(A,9 mmol/L ![]() 的营养液中处理6 h。②低磷胁迫处理:幼苗在0.25 mmol/L KH2PO4中生长10 d后,一部分转移到5 μmol/L KH2PO4 (低磷)营养液中处理3 d,另一部分仍在0.25 mmol/L KH2PO4(高磷)中生长作为对照。③低硼胁迫处理:幼苗分别在0.25 μmol/L H3BO3 (低硼)和10 μmol/L H3BO3 (高硼,对照)的营养液中培养10 d。④镉胁迫处理:幼苗在无镉营养液中培养10 d后,一部分转移到10 μmol/L CdCl2中培养12 h,另一部分仍在无镉营养液中生长作为对照。⑤盐胁迫处理:幼苗在不含NaCl的营养液中培养10 d后,一部分转移到200 mmol/L NaCl营养液中培养12 h,另一部分仍在不含NaCl的营养液中生长作为对照。所有试验地上部与根系样品均包含3个生物学重复。
的营养液中处理6 h。②低磷胁迫处理:幼苗在0.25 mmol/L KH2PO4中生长10 d后,一部分转移到5 μmol/L KH2PO4 (低磷)营养液中处理3 d,另一部分仍在0.25 mmol/L KH2PO4(高磷)中生长作为对照。③低硼胁迫处理:幼苗分别在0.25 μmol/L H3BO3 (低硼)和10 μmol/L H3BO3 (高硼,对照)的营养液中培养10 d。④镉胁迫处理:幼苗在无镉营养液中培养10 d后,一部分转移到10 μmol/L CdCl2中培养12 h,另一部分仍在无镉营养液中生长作为对照。⑤盐胁迫处理:幼苗在不含NaCl的营养液中培养10 d后,一部分转移到200 mmol/L NaCl营养液中培养12 h,另一部分仍在不含NaCl的营养液中生长作为对照。所有试验地上部与根系样品均包含3个生物学重复。
1.2.7 高通量转录组测序 对上述胁迫处理(1.2.6)的样品进行转录组测序,测序平台为Illumina Hiseq 2000,每个样品生成6.0 Gb测序数据,测序方式为双末端(PE)测序,读取长度为150 bp。以错误发现率(FDR)≤0.05和表达变化倍数≥2为鉴定差异表达基因的阈值。
1.2.8 组织表达分析 组织表达数据来源于甘蓝型油菜转录组信息资源数据库(BnTIR,http://yanglab.hzau.edu.cn/BnTIR)。数据包括甘蓝型油菜中双11的90个不同样品,覆盖了油菜生长过程不同阶段的不同器官。利用GraphPad Prism软件绘制箱型图。
2 结果与分析
2.1 二倍体白菜与甘蓝以及四倍体甘蓝型油菜AMT家族基因的鉴定
为了鉴定甘蓝型油菜中的AMT基因,使用拟南芥AMT的氨基酸序列作为参照,从芸薹属数据库(BRAD)中提取基因,并用油菜转录组数据检验这些基因的注释。进一步用SMART[43]软件对所有潜在基因所编码蛋白质的保守结构域进行校验,以保证每个蛋白质中都含有完整的AMT保守结构域,经过筛选,最终鉴定出20个BnaAMT基因:其中14个为AMT1家族成员:3个BnaAMT1;1s,2个BnaAMT1;2s、3个BnaAMT1;3s、3个BnaAMT1;4s、3个BnaAMT1;5s,其余6个为AMT2亚家族成员BnaAMT2;1s;并基于芸薹属植物基因命名标准进行了命名:BnaA5.AMT1;1~BnaCnn.AMT2;1。其中,Cnn表示该基因锚在C亚基因组上,但具体染色体信息未知;Unn表示该基因在油菜基因组的染色体信息完全未知。用同样的方法找到了白菜和甘蓝的AMT家族基因,白菜中共有9个AMT成员,甘蓝中共有10个AMT成员。分析了这39个基因所编码蛋白质的氨基酸数量、分子质量、等电点、不稳定系数、脂肪指数以及平均亲水系数。所有AMT基因编码蛋白质的氨基酸数量为330~669,其中33个介于475~512,差异较小(表1)。不稳定性指数>40表明蛋白质不稳定[44],分析表明这39个AMT蛋白均为稳定蛋白。
表 1 甘蓝型油菜、白菜、甘蓝AMT家族基因信息
Tab.1 Basic information of AMT family genes in Brassica napus,Brassica rapa and Brassica oleracea
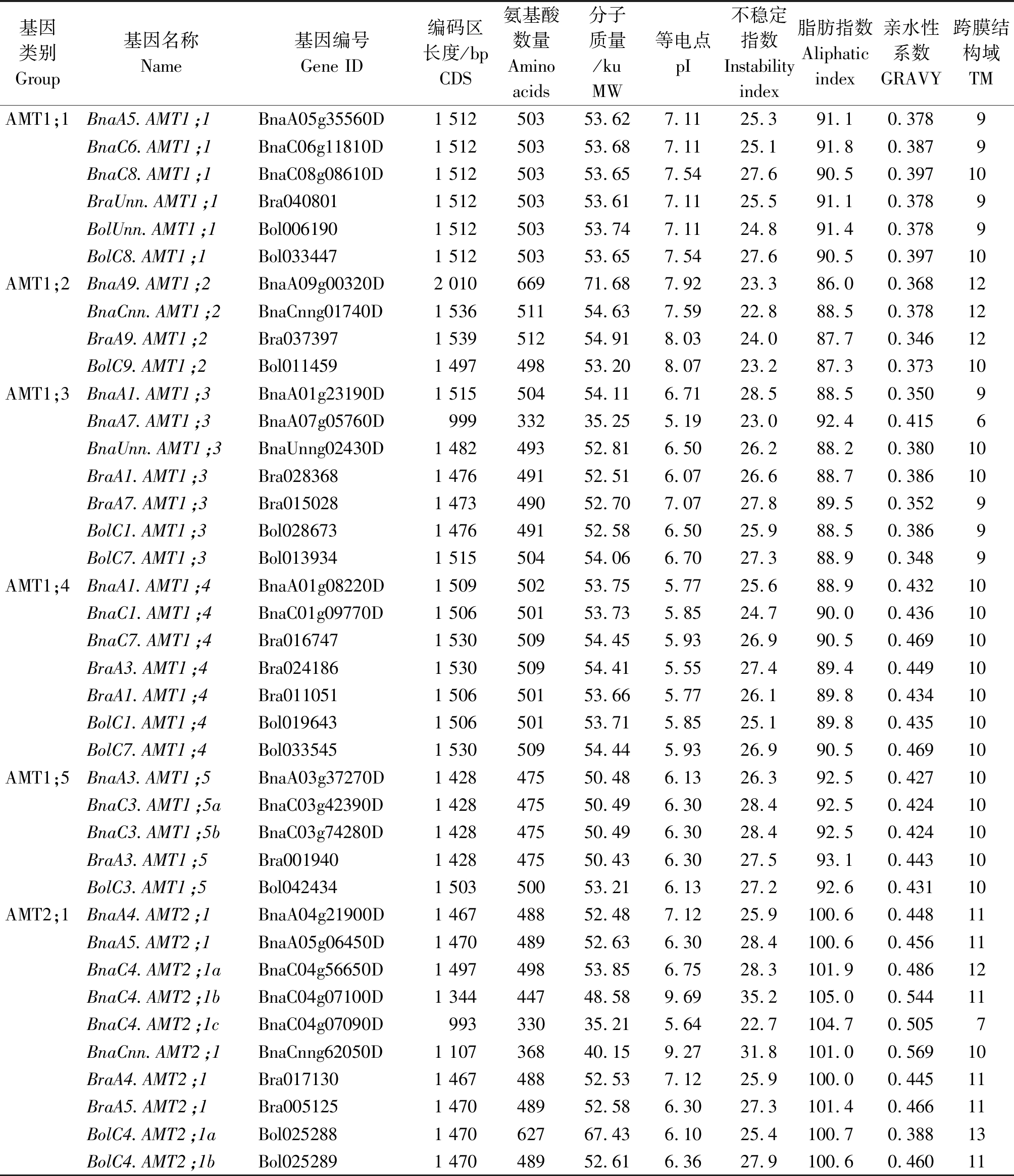
基因类别Group基因名称Name基因编号Gene ID编码区长度/bpCDS 氨基酸数量Aminoacids分子质量/kuMW 等电点pI不稳定指数Instabilityindex脂肪指数Aliphaticindex亲水性系数GRAVY跨膜结构域TMAMT1;1BnaA5.AMT1;1BnaA05g35560D1 51250353.627.1125.391.10.3789BnaC6.AMT1;1BnaC06g11810D1 51250353.687.1125.191.80.3879BnaC8.AMT1;1BnaC08g08610D1 51250353.657.5427.690.50.39710BraUnn.AMT1;1Bra0408011 51250353.617.1125.591.10.3789BolUnn.AMT1;1Bol0061901 51250353.747.1124.891.40.3789BolC8.AMT1;1Bol0334471 51250353.657.5427.690.50.39710AMT1;2BnaA9.AMT1;2BnaA09g00320D2 01066971.687.9223.386.00.36812BnaCnn.AMT1;2BnaCnng01740D1 53651154.637.5922.888.50.37812BraA9.AMT1;2Bra0373971 53951254.918.0324.087.70.34612BolC9.AMT1;2Bol0114591 49749853.208.0723.287.30.37310AMT1;3BnaA1.AMT1;3BnaA01g23190D1 51550454.116.7128.588.50.3509BnaA7.AMT1;3BnaA07g05760D99933235.255.1923.092.40.4156BnaUnn.AMT1;3BnaUnng02430D1 48249352.816.5026.288.20.38010BraA1.AMT1;3Bra0283681 47649152.516.0726.688.70.38610BraA7.AMT1;3Bra0150281 47349052.707.0727.889.50.3529BolC1.AMT1;3Bol0286731 47649152.586.5025.988.50.3869BolC7.AMT1;3Bol0139341 51550454.066.7027.388.90.3489AMT1;4BnaA1.AMT1;4BnaA01g08220D1 50950253.755.7725.688.90.43210BnaC1.AMT1;4BnaC01g09770D1 50650153.735.8524.790.00.43610BnaC7.AMT1;4Bra0167471 53050954.455.9326.990.50.46910BraA3.AMT1;4Bra0241861 53050954.415.5527.489.40.44910BraA1.AMT1;4Bra0110511 50650153.665.7726.189.80.43410BolC1.AMT1;4Bol0196431 50650153.715.8525.189.80.43510BolC7.AMT1;4Bol0335451 53050954.445.9326.990.50.46910AMT1;5BnaA3.AMT1;5BnaA03g37270D1 42847550.486.1326.392.50.42710BnaC3.AMT1;5aBnaC03g42390D1 42847550.496.3028.492.50.42410BnaC3.AMT1;5bBnaC03g74280D1 42847550.496.3028.492.50.42410BraA3.AMT1;5Bra0019401 42847550.436.3027.593.10.44310BolC3.AMT1;5Bol0424341 50350053.216.1327.292.60.43110AMT2;1BnaA4.AMT2;1BnaA04g21900D1 46748852.487.1225.9100.60.44811BnaA5.AMT2;1BnaA05g06450D1 47048952.636.3028.4100.60.45611BnaC4.AMT2;1aBnaC04g56650D1 49749853.856.7528.3101.90.48612BnaC4.AMT2;1bBnaC04g07100D1 34444748.589.6935.2105.00.54411BnaC4.AMT2;1cBnaC04g07090D99333035.215.6422.7104.70.5057BnaCnn.AMT2;1BnaCnng62050D1 10736840.159.2731.8101.00.56910BraA4.AMT2;1Bra0171301 46748852.537.1225.9100.00.44511BraA5.AMT2;1Bra0051251 47048952.586.3027.3101.40.46611BolC4.AMT2;1aBol0252881 47062767.436.1025.4100.70.38813BolC4.AMT2;1bBol0252891 47048952.616.3627.9100.60.46011
2.2 AMT家族基因系统发育分析
用20个甘蓝型油菜BnaAMTs蛋白序列、9个白菜BraAMTs蛋白序列、10个甘蓝BolAMTs蛋白序列和6个拟南芥AtAMTs蛋白序列构建系统进化树。AMT的每个亚家族成员都与拟南芥中的相应同源基因紧密聚集(图1),暗示甘蓝型油菜AMT家族基因与拟南芥相应同源基因具有相似功能。同时,甘蓝型油菜BnaAMTs均与白菜或者甘蓝的AMT成簇出现,拟南芥单独成一个分支(图1),这说明甘蓝型油菜与白菜和甘蓝基因编码的蛋白质在序列上的相似程度高于拟南芥,亲缘关系更近。
2.3 AMT家族基因的染色体定位
通过下载甘蓝型油菜AMT基因相关染色体定位信息,利用软件MapInspect进行染色体定位并进行可视化分析(图2)。结果显示,甘蓝型油菜的20个AMT基因不均匀地分布在12条染色体上:C4染色体上分布3个(BnaC4.AMT2;1a、BnaC4.AMT2;1b、BnaC4.AMT2;1c),A1、A5和C3染色体上均分布2个(BnaA1.AMT1;3、BnaA1.AMT1;4、BnaA5.AMT1;1、BnaA5.AMT2;1、BnaC3.AMT1;5a、BnaC3.AMT1;5b),A4、A7、A9、C1、C6、C7和C8染色体上均分布1个AMT基因(分别是:BnaA4.AMT2;1、BnaA7.AMT1;3、BnaA9.AMT1;2、BnaC1.AMT1;4、BnaC6.AMT1;1、BnaC7.AMT1;4、BnaC8.AMT1;1)。BnaCnn.AMT1;2 (仅知定位在C亚基因组,具体染色体信息未知)、BnaUnn.AMT1;3 (所在亚基因组和染色体信息未知)和BnaCnn.AMT2;1 (仅知定位在C亚基因组,具体染色体信息未知)由于具体位置不详,无法进行染色体定位。其中,BnaC4.AMT2;1b和BnaC4.AMT2;1c在染色体上的相对位置仅为4 412 bp,推断它们为串联重复基因簇。
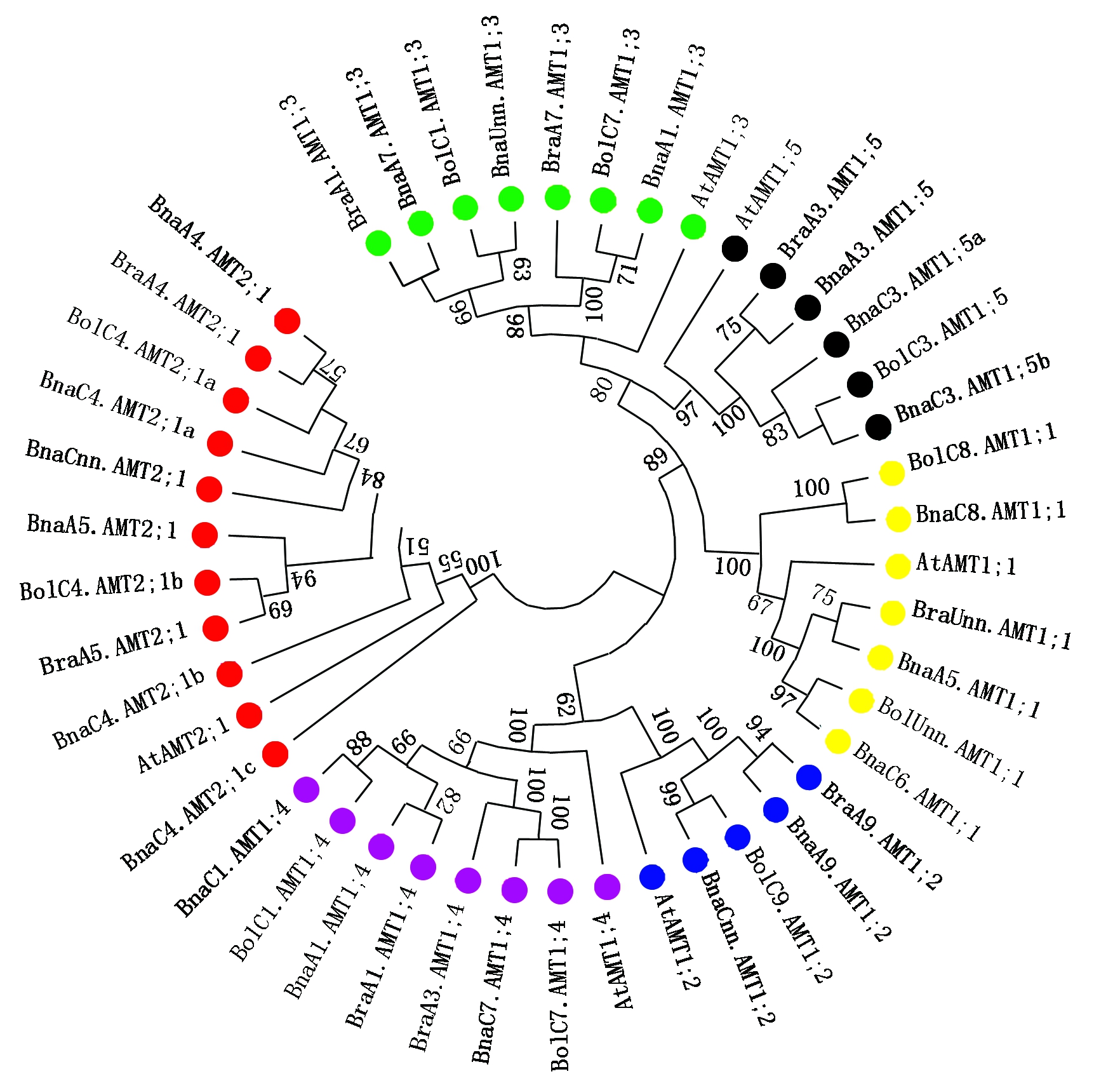
图1 甘蓝型油菜、白菜、甘蓝和拟南芥AMT家族蛋白质的系统进化分析
Fig.1 Phylogenetic tree of AMT proteins in Brassica napus, Brassica rapa, Brassica oleracea and Arabidopsis
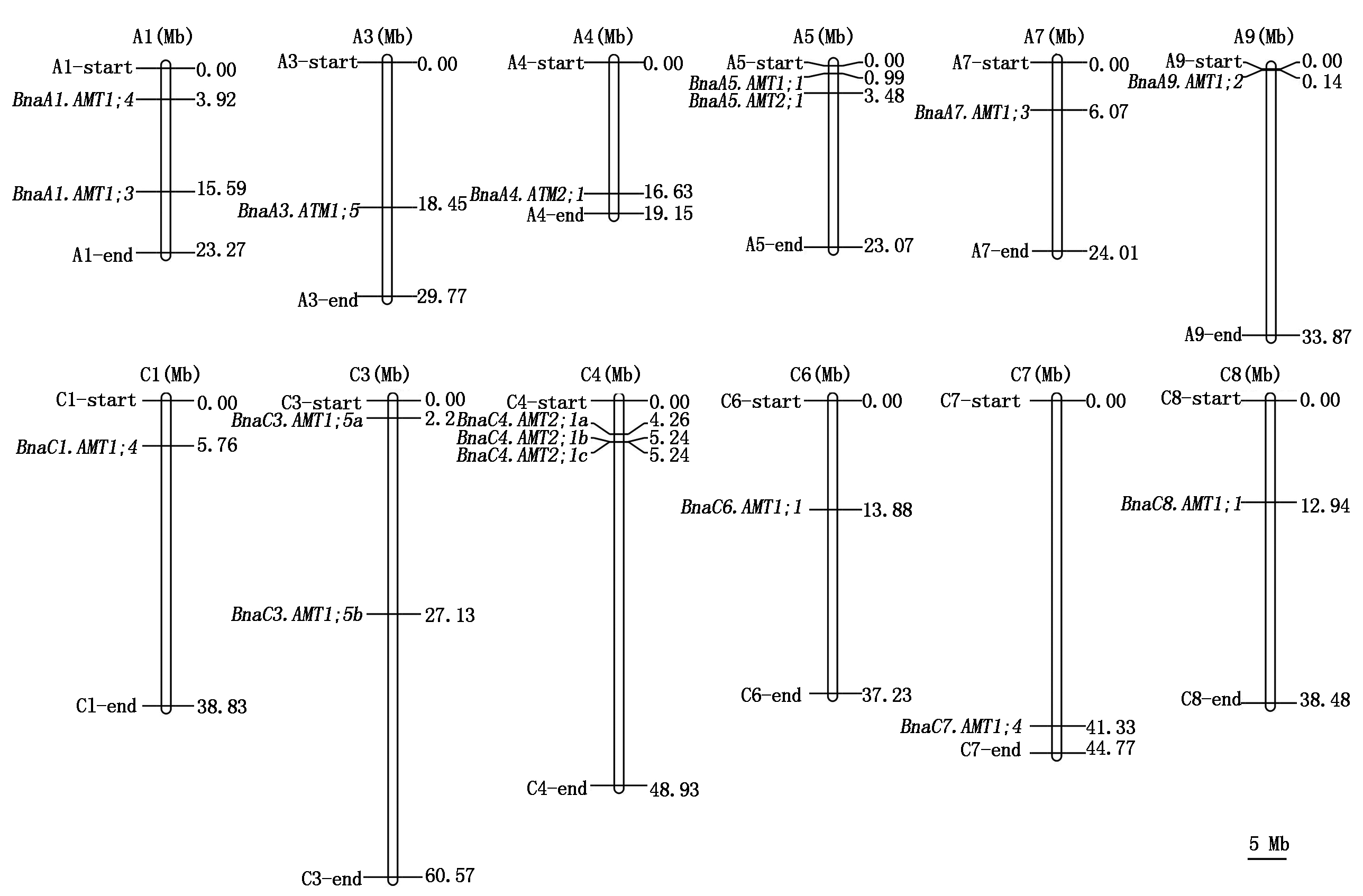
图2 甘蓝型油菜AMT家族基因在染色体上的分布
Fig.2 Chromosomal location of Brassica napus AMT family genes
2.4 多序列比对和保守结构域分析
从分子进化的角度来看,如果氨基酸残基进化保守,则认为它们在功能上或结构上具有重要意义。序列同源性分析表明,甘蓝型油菜与拟南芥AMT蛋白具有高度相似性,表明AMT在异源多倍化过程中是保守的。为了揭示芸薹属植物和拟南芥AMT家族基因之间的内在联系,将45个AMT基因的氨基酸序列提交到MEME网站进行保守基序预测和注释。MEME分析表明,BnaAMT家族基因编码的氨基酸序列在芸薹属植物中基本含有相同的保守结构域(图3),表明它们高度保守。Motif是蛋白质分子中具有特定空间构象和特定功能的结构成分。它是结构域的亚单位,与特定的功能有关,推测具有不同Motif的AMT成员可能在进化和功能上存在差异。如,所有的BnaAMT2;1s基因均没有Motif1、Motif5和Motif6(图3),这可能是AMT2家族功能不同于AMT1家族的结构上的原因。AMT1和AMT2亚家族结构差异巨大,进化的过程中可能分开较早。

图3 甘蓝型油菜AMT家族基因及保守基序分析
Fig.3 Diagram of structure of Brassica napus AMT family genes and their conserved Motifs
2.5 AMT家族蛋白的二级与三维结构的预测
BnaAMT蛋白的二级结构预测见图4。由于多重序列比对显示,AMT1亚家族和AMT2亚家族序列差异较大(图3),分别以BnaA5.AMT1;1和BnaA4.AMT2;1为例进行了二级与三级结构预测。GOR4和PSIPRED的二维结构预测结果相似。AMT家族主要由α-螺旋、延伸链与随机螺旋构成,AMT1家族α螺旋比例高于AMT2家族,而AMT2家族的延伸链与随机螺旋比例高于AMT1家族(图4)。3D轮廓突出了AMT蛋白的保守结构域,可以直观看出AMT蛋白结构中的α-螺旋、延伸链与随机螺旋(图5)。这些结果表明,随机螺旋和α-螺旋是这些AMT蛋白结构的重要组成部分。
2.6 BnaAMT基因的表达特性分析
2.6.1 BnaAMT基因的组织特异性表达 通过对甘蓝型油菜转录组信息资源库中AMT基因的分析,得到17个AMT基因(缺少BnaC3.AMT1;5b、BnaC4.AMT2;1c和BnaCnn.AMT2;1基因,在BnPIR(http://cbi.hzau.edu.cn/bnapus/)甘蓝型油菜泛基因组数据库中检索发现,这些基因在中双11油菜基因组中缺失)在8个组织中的表达数据。结果显示,不同基因在甘蓝型油菜不同器官的表达存在差异,进化树中处于同一分支的基因表达情况相似。例如,BnaA5.AMT1;1和BnaC6.AMT1;1基因在甘蓝型油菜除籽粒外的其他器官中表达量都相对较高,而BnaC8.AMT1;1基因在甘蓝型油菜所有器官都处于低表达状态(图 6)。根器官中表达较高的基因有BnaA5.AMT1;1、BnaC6.AMT1;1、BnaA9.AMT1;2、BnaA7.AMT1;3和BnaC3.AMT1;5a;茎器官中表达较高的基因有BnaA5.AMT1;1、BnaC6.AMT1;1、BnaA9.AMT1;2、BnaA1.AMT1;4和BnaC7.AMT1;4;叶器官中表达较高的基因有BnaA5.AMT1;1、BnaC6.AMT1;1、BnaA9.AMT1;2、BnaA1.AMT1;4、BnaC7.AMT1;4和BnaA5.AMT2;1;蕾器官中表达较高的基因有BnaA1.AMT1;4、BnaC1.AMT1;4、BnaC7.AMT1;4和BnaC4.AMT2;1b;花器官中表达较高的基因有BnaA5.AMT1;1、BnaC6.AMT1;1、BnaA1.AMT1;4、BnaC1.AMT1;4、BnaC7.AMT1;4、BraA3.AMT1;5和BnaC4.AMT2;1b;角果器官中表达较高的基因有BnaA5.AMT1;1、BnaC6.AMT1;1、BnaA1.AMT1;4和BnaC7.AMT1;4;角果壳器官中表达较高的基因有BnaA5.AMT1;1、BnaC6.AMT1;1、BnaA1.AMT1;4、BnaC7.AMT1;4、BnaA5.AMT2;1和BnaC4.AMT2;1b,籽粒中所有基因均呈低表达状态(图 6)。

图4 甘蓝型油菜BnaA5.AMT1;1和BnaA4.AMT2;1蛋白二级结构分析
Fig.4 Secondary structure analysis of BnaA5.AMT1;1 and BnaA4.AMT2;1 proteins
2.6.2 BnaAMT基因对不同氮处理的响应 将油菜幼苗在2 mmol/L NH4NO3条件下培养10 d,然后转移到无氮营养液中饥饿培养3 d,再分别转移到含9 mmol/L ![]() 和0.3 mmol/L
和0.3 mmol/L ![]() 的营养液中培养6 h,取样进行转录组测序。拟南芥AMT家族编码主动转运
的营养液中培养6 h,取样进行转录组测序。拟南芥AMT家族编码主动转运![]() 的载体蛋白,并受硝态氮调控。
的载体蛋白,并受硝态氮调控。
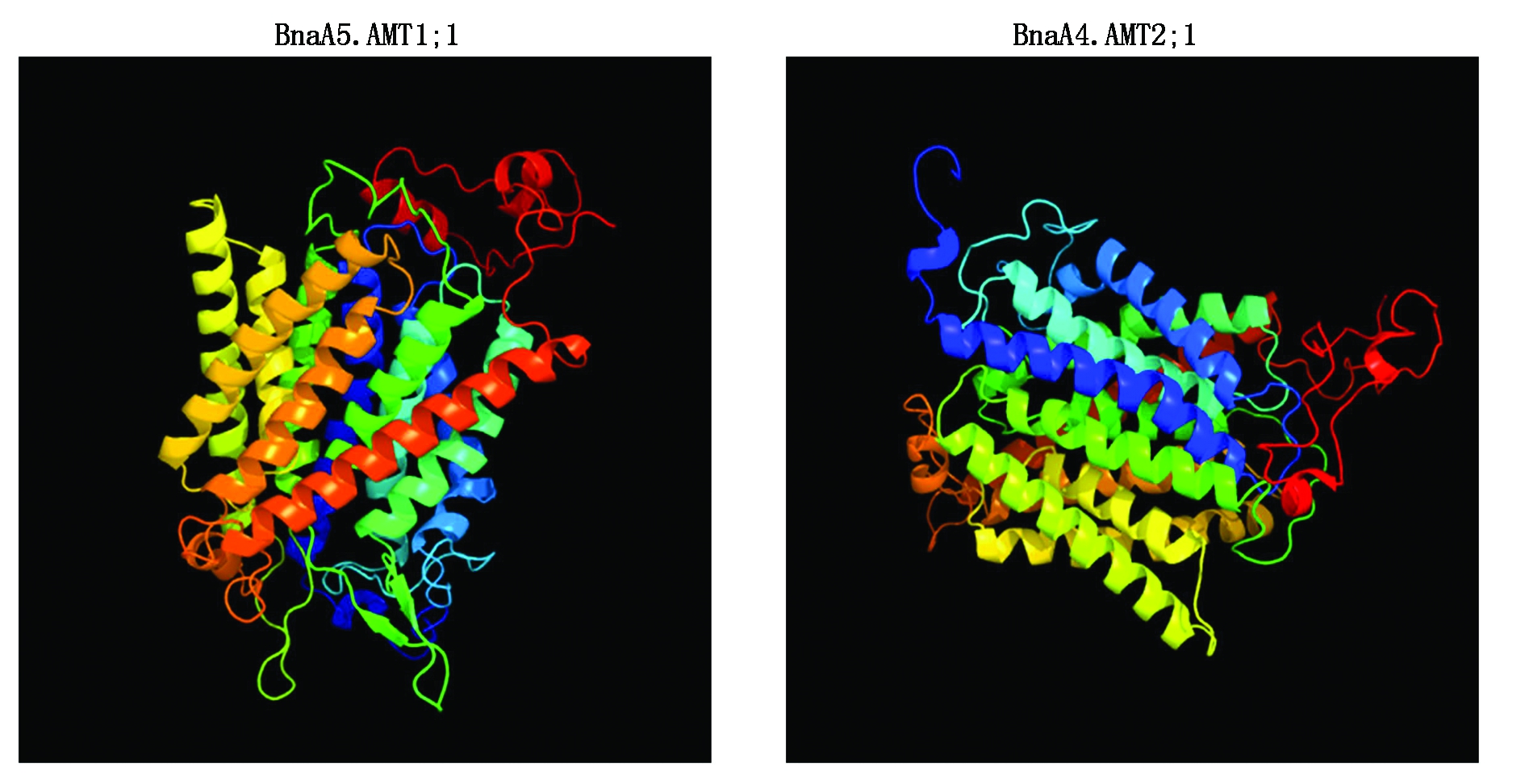
图5 甘蓝型油菜BnaA5.AMT1;1和BnaA4.AMT2;1蛋白三级结构预测
Fig.5 Prediction of three-dimensional structure of BnaA5.AMT1;1 and BnaA4.AMT2;1 proteins

图6 甘蓝型油菜AMT家族基因在不同组织中的表达情况
Fig.6 The expression of BnaAMT family genes in different tissues
AtAMT1;1~AtAMT1;3和AtAMT1;5负责根部铵的吸收[45],本研究发现,根部BnaAMT1;1s和BnaAMT 1;5s均被低![]() 诱导表达,然而BnaAMT1;2s、BnaAMT1;3s和BnaAMT2;1s则被高
诱导表达,然而BnaAMT1;2s、BnaAMT1;3s和BnaAMT2;1s则被高![]() 和高
和高![]() 诱导表达(图7)。拟南芥AMT1;1和AMT2;1可以将
诱导表达(图7)。拟南芥AMT1;1和AMT2;1可以将![]() 转移到地上部,本研究中,地上部BnaAMT1;1s和BnaAMT2;1s的表达与根中的表达模式相似。拟南芥AMT1;4是花粉特异表达基因,通过
转移到地上部,本研究中,地上部BnaAMT1;1s和BnaAMT2;1s的表达与根中的表达模式相似。拟南芥AMT1;4是花粉特异表达基因,通过![]() 的吸收对花粉的氮素营养有贡献[12], BnaC7.AMT1;4在地上部受低
的吸收对花粉的氮素营养有贡献[12], BnaC7.AMT1;4在地上部受低![]() 显著诱导,在高浓度
显著诱导,在高浓度![]() 和
和![]() 的条件下表达水平显著下降,尤其是受到
的条件下表达水平显著下降,尤其是受到![]() 的抑制作用更显著,其他2个BnaAMT1;4s拷贝在9 mmo/L
的抑制作用更显著,其他2个BnaAMT1;4s拷贝在9 mmo/L ![]() 和0.3 mmol/L
和0.3 mmol/L ![]() 这3种培养条件下均几乎不表达(图7)。
这3种培养条件下均几乎不表达(图7)。
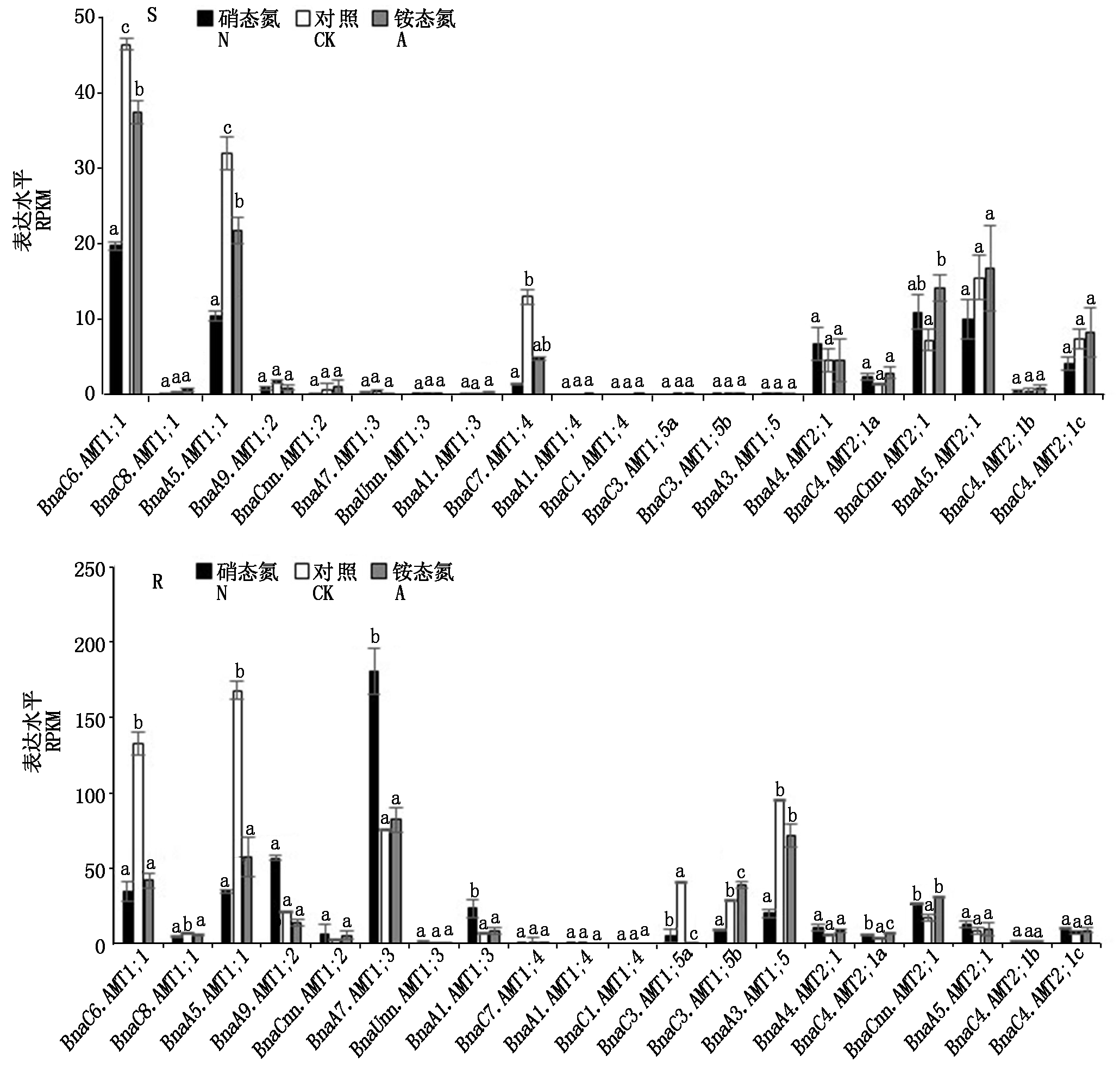
S.地上部;R.根;差异显著性分析采用单因素方差分析,Tukey检验,图中不同字母表示处理之间差异显著(P<0.05)。图8同。
S.Shoot;R.Root;The significance of difference was evaluated using one-way ANOVA,Tukey test,different letters in the same group indicate significant differences among treatments(P<0.05).The same as Fig.8.
图7 甘蓝型油菜BnaAMT家族基因对硝态氮和铵态氮的转录响应
Fig.7 Transcriptome responses of Bna AMT family genes to nitrate and ammonium
2.6.3 BnaAMT对其他非生物胁迫的响应 随后,进一步分析了甘蓝型油菜AMT家族基因对其他营养胁迫与重金属胁迫,以及盐胁迫的转录响应情况,其中包括磷胁迫(缺磷)、硼胁迫(缺硼)、镉胁迫(镉毒害)以及盐胁迫(盐毒害)。在缺磷条件下,BnaC6.AMT1;1和BnaA9.AMT1;2在油菜地上部和根系中的表达量均显著上升,地上部BnaA7.AMT1;3、BnaA5.AMT2;1和BnaC4.AMT2;1c表达量显著上升,只有BnaC7.AMT1;4表达量显著下降,其他基因的表达量无显著变化(图8-A)。在缺硼条件下,根系中的BnaAMT1s 3个成员的表达水平均显著上升;地上部BnaC6.AMT1;1、BnaA9.AMT1;2的表达量显著上升,只有BnaC7.AMT1;4表达量显著下降(图8-B)。无论根部还是地上部,镉毒对AMT家族基因的表达影响较小(图8-C)。油菜受到盐胁迫时,地上部BnaA9.AMT1;2和BnaC4.AMT2;1c表达量显著上升,BnaC4.AMT2;1和BnaCnn.AMT2;1表达量显著下降,其他基因表达无显著变化;根部AMT家族基因整体上则呈现显著下降的趋势(图8-D)。总之,甘蓝型油菜在遭受缺硼或者缺磷胁迫时,AMT家族部分基因呈现上升表达趋势;在盐胁迫条件下,根系AMT家族基因整体表达量下降;镉毒对AMT家族基因的表达无明显影响。

图8 磷、硼、镉、盐胁迫下甘蓝型油菜BnaAMT家族基因的转录组分析
Fig.8 Transcriptome responses of BnaAMT family genes to phosphorus,boron,cadmium and salt stresses
3 结论与讨论
本研究通过数据库比对,在甘蓝型油菜全基因组中发现了20个铵转运蛋白,可分为2个亚家族。其中14个为AMT1家族成员,其余6个为AMT2亚家族成员。由保守基序预测结果可知,甘蓝型油菜铵转运蛋白亚家族内的蛋白质序列保守性强,但亚家族间的差异较大。油菜在进化过程中,基因组发生了多次多倍化事件进而导致油菜内部普遍存在多拷贝基因家族[26-27],类似作物包括小麦(Triticum aestivum L.)[46]和棉花 (Gossypium hirsutum L.) [47]。因此,拟南芥中的每个基因在油菜中应有6个拷贝[48]。一个基因的多个拷贝的存在释放了任何单个拷贝以及自然和人为的选择压力,这促进了新基因功能的出现[49]。由此产生的变异的增加可以用于培育具有理想表型的作物品种[50]。因此,异源四倍体油菜AMT家族成员是否具有不同于拟南芥的新基因功能还有待进一步研究。
水稻和拟南芥中的各个AMT蛋白分别位于植物不同器官,在同一氮素条件下呈现不同的表达活性[4,9],AMT成员间协同作用来满足植物吸收氮素的生理需求。拟南芥负责根系吸收铵离子的主要是AMT1;1~AMT1;3和AMT1;5,它们都是高亲和转运子[11]。其中,AMT1;1、AMT1;2和AMT1;3对铵离子吸收的总贡献率高达95%[51]。AMT1;1和AMT2;1主要负责叶片铵的贮存,AMT2;1也属于高亲和的铵转运子[10]。高亲和的铵转运子AMT1;4研究较少,主要负责花粉中铵的吸收或者运出[12]。在本研究中,BnaAMT1;1s所有拷贝的表达水平均受高浓度![]() 和
和![]() 的抑制,根部BnaAMT1;2s受高浓度
的抑制,根部BnaAMT1;2s受高浓度![]() 诱导,BnaAMT1;3s在根部受高浓度
诱导,BnaAMT1;3s在根部受高浓度![]() 和
和![]() 诱导,BnaAMT1;4s各个拷贝只有BnaC7.AMT1;4在地上部表达量较高,该基因受高浓度
诱导,BnaAMT1;4s各个拷贝只有BnaC7.AMT1;4在地上部表达量较高,该基因受高浓度![]() 和
和![]() 的抑制,其他2个拷贝基本不表达。因此,甘蓝型油菜AMT基因和对应的拟南芥AMT同源基因的亲和性、表达模式既有相同,又有区别,这暗示了拟南芥的AMT基因与其在油菜中对应的同源基因存在一定程度的功能保留和分化。Yuan等[52]曾报道,AtAMT1;1转基因的烟草植株对铵的吸收增加了30%。水稻AMT1;1的超表达植株在低氮条件下可以增加超过30%的籽粒产量[53]。在油菜AMT的多个拷贝中,根部BnaA5.AMT1;1和BnaA7.AMT1;3,叶片BnaC7.AMT1;4和BnaA5.AMT2;1,角果壳BnaA5.AMT2;1表达量高,同时对不同氮水平响应显著,有望利用它们进行转基因操作以培育具有氮素利用效率高或者耐铵毒的油菜品种。
的抑制,其他2个拷贝基本不表达。因此,甘蓝型油菜AMT基因和对应的拟南芥AMT同源基因的亲和性、表达模式既有相同,又有区别,这暗示了拟南芥的AMT基因与其在油菜中对应的同源基因存在一定程度的功能保留和分化。Yuan等[52]曾报道,AtAMT1;1转基因的烟草植株对铵的吸收增加了30%。水稻AMT1;1的超表达植株在低氮条件下可以增加超过30%的籽粒产量[53]。在油菜AMT的多个拷贝中,根部BnaA5.AMT1;1和BnaA7.AMT1;3,叶片BnaC7.AMT1;4和BnaA5.AMT2;1,角果壳BnaA5.AMT2;1表达量高,同时对不同氮水平响应显著,有望利用它们进行转基因操作以培育具有氮素利用效率高或者耐铵毒的油菜品种。
近年来,植物营养元素之间以及植物营养与其他逆境之间互作的研究很多。BcAMT1;2介导了![]() 与
与![]() 互作时铵的高效吸收,是白菜型油菜在
互作时铵的高效吸收,是白菜型油菜在![]() 与
与![]() 配比条件下比在单独的
配比条件下比在单独的![]() 或
或![]() 处理下长得更好的原因之一[54]。然而,目前还没有
处理下长得更好的原因之一[54]。然而,目前还没有![]() 与其他营养的互作研究。氮与磷的互作研究主要集中在
与其他营养的互作研究。氮与磷的互作研究主要集中在![]() 与磷之间,水稻
与磷之间,水稻![]() 可以激活磷信号,是通过NRT1.1B介导磷的转录抑制子SPX4的降解来实现的[55]。本研究发现,甘蓝型油菜缺磷可以显著提高AMT家族部分基因的表达,由此推测,缺磷可以激活铵信号。在缺硼胁迫下,烟草硝转运子NRT2和硝酸还原酶基因NIA的表达量显著下降[56]。然而本研究发现,甘蓝型油菜在缺硼条件下,AMT家族基因整体呈现上升表达趋势,可能是植物逆境时选择更经济的营养吸收方式(植物吸收
可以激活磷信号,是通过NRT1.1B介导磷的转录抑制子SPX4的降解来实现的[55]。本研究发现,甘蓝型油菜缺磷可以显著提高AMT家族部分基因的表达,由此推测,缺磷可以激活铵信号。在缺硼胁迫下,烟草硝转运子NRT2和硝酸还原酶基因NIA的表达量显著下降[56]。然而本研究发现,甘蓝型油菜在缺硼条件下,AMT家族基因整体呈现上升表达趋势,可能是植物逆境时选择更经济的营养吸收方式(植物吸收![]() 比
比![]() 耗能少)。研究表明,镉胁迫可以增强盐芥根系木栓化[57],木栓化影响铵离子的吸收[14]。然而,镉毒对甘蓝型油菜AMT家族基因的表达影响较小。水稻在盐胁迫时,AMT表达量有所上升,从而保证了氮代谢的稳定性[58]。在盐胁迫下,甘蓝型油菜根系AMT基因的表达量却显著下降,这可能是Na+与
耗能少)。研究表明,镉胁迫可以增强盐芥根系木栓化[57],木栓化影响铵离子的吸收[14]。然而,镉毒对甘蓝型油菜AMT家族基因的表达影响较小。水稻在盐胁迫时,AMT表达量有所上升,从而保证了氮代谢的稳定性[58]。在盐胁迫下,甘蓝型油菜根系AMT基因的表达量却显著下降,这可能是Na+与![]() 的离子半径较为接近,过量的Na+对
的离子半径较为接近,过量的Na+对![]() 的吸收产生了竞争,从而抑制了AMT基因的表达。这些结果为植物营养元素之间以及植物营养与其他非生物逆境之间的互作研究提供了参考思路。
的吸收产生了竞争,从而抑制了AMT基因的表达。这些结果为植物营养元素之间以及植物营养与其他非生物逆境之间的互作研究提供了参考思路。
综上,本研究在甘蓝型油菜基因组中共发现了20个铵转运蛋白,其中14个为AMT1亚家族成员,6个为AMT2亚家族成员。由保守基序预测结果可知,铵转运蛋白亚家族内的蛋白质序列保守性强,但亚家族之间的差异较大。甘蓝型油菜AMT基因与模式植物拟南芥AMT的同源基因的表达模式既有相似之处,又存在差异。这表明在基因组多倍化过程中,AMT基因发生了功能分化,产生了适应油菜的新的基因功能。根部BnaA5.AMT1;1和BnaA7.AMT1;3,叶片BnaC7.AMT1;4和BnaA5.AMT2;1,角果壳BnaA5.AMT2;1表达量高,同时对不同氮水平响应显著,可以利用这些基因进行遗传操作以培育具有理想表型的油菜品种。在缺硼或者缺磷胁迫时,BnaAMT家族部分基因呈现上升表达的趋势;在盐毒害胁迫下,根系AMT家族基因整体表达量下降;镉毒对AMT家族基因的表达无明显影响。这些结果为进一步深入解析甘蓝型油菜AMT家族基因的分子功能奠定了基础。
[1] Liu X J,Zhang Y,Han W X,Tang A H,Shen J L,Cui Z L,Vitousek P,Erisman J W,Goulding K,Christie P,Fangmeier A,Zhang F S. Enhanced nitrogen deposition over China[J].Nature,2013,494(7438):459-462.doi:10.1038/nature11917.
[2] Zhang W F, Cao G X, Li X L, Zhang H Y, Wang C, Liu Q Q, Chen X P, Cui Z L, Shen J B, Jiang R F, Mi G H, Miao Y X, Zhang F S, Dou Z X. Closing yield gaps in China by empowering smallholder farmers[J].Nature,2016,537(7622):671-674.doi:10.1038/nature19368.
[3] Bloom A J,Sukrapanna S S,Warner R L. Root respiration associated with ammonium and nitrate absorption and assimilation by barley[J].Plant Physiology,1992,99(4):1294-1301. doi:10.1104/pp.99.4.1294.
[4] Gazzarrini S,Lejay L,Gojon A,Ninnemann O,Frommer W B,von Wir n N. Three functional transporters for constitutive,diurnally regulated,and starvation-induced uptake of ammonium into Arabidopsis roots[J].The Plant Cell,1999,11(5):937-947. doi:10.1105/tpc.11.5.937.
[5] Britto D T,Kronzucker H J. ![]() toxicity in higher plants:a critical review[J].Journal of Plant Physiology,2002,159(6):567-584. doi:10.1078/0176-1617-0774.
toxicity in higher plants:a critical review[J].Journal of Plant Physiology,2002,159(6):567-584. doi:10.1078/0176-1617-0774.
[6] Hao D L,Zhou J Y,Yang S Y,Qi W,Yang K J,Su Y H. Function and regulation of ammonium transporters in plants[J].International Journal of Molecular Sciences,2020,21(10):3557. doi:10.3390/ijms21103557.
[7] Koegel S,Ait Lahmidi N,Arnould C,Chatagnier O,Walder F,Ineichen K,Boller T,Wipf D,Wiemken A,Courty P E. The family of ammonium transporters(AMT)in Sorghum bicolor:Two AMT members are induced locally,but not systemically in roots colonized by arbuscular mycorrhizal fungi[J].The New Phytologist,2013,198(3):853-865. doi:10.1111/nph.12199.
[8] Rawat S R,Silim S N,Kronzucker H J,Siddiqi M Y,Glass A D M. AtAMT1 gene expression and ![]() uptake in roots of Arabidopsis thaliana:Evidence for regulation by root glutamine levels[J].The Plant Journal,1999,19(2):143-152. doi:10.1046/j.1365-313x.1999.00505.x.
uptake in roots of Arabidopsis thaliana:Evidence for regulation by root glutamine levels[J].The Plant Journal,1999,19(2):143-152. doi:10.1046/j.1365-313x.1999.00505.x.
[9] Sonoda Y,Ikeda A,Saiki S,Wirén N V,Yamaya T,Yamaguchi J. Distinct expression and function of three ammonium transporter genes(OsAMT1;1-1;3)in rice[J].Plant and Cell Physiology,2003,44(7):726-734. doi:10.1093/pcp/pcg083.
[10] Sohlenkamp C,Wood C C,Roeb G W,Udvardi M K. Characterization of Arabidopsis AtAMT2,a high-affinity ammonium transporter of the plasma membrane[J].Plant Physiology,2002,130(4):1788-1796.doi:10.1104/pp.008599.
[11] Yuan L X,Loqué D,Kojima S,Rauch S,Ishiyama K,Inoue E,Takahashi H,von Wireén N. The organization of high-affinity ammonium uptake in Arabidopsis roots depends on the spatial arrangement and biochemical properties of AMT1-type transporters[J].The Plant Cell,2007,19(8):2636-2652. doi:10.1105/tpc.107.052134.
[12] Yuan L X,Graff L,Loqu D,Kojima S,Tsuchiya Y N,Takahashi H,von Wirén N. AtAMT1;4,a pollen-specific high-affinity ammonium transporter of the plasma membrane in Arabidopsis[J].Plant and Cell Physiology,2009,50(1):13-25. doi:10.1093/pcp/pcn186.
[13] Giehl R F H,Laginha A M,Duan F Y,Rentsch D,Yuan L X,von Wirén N. A critical role of AMT2;1 in root-to-shoot translocation of ammonium in Arabidopsis[J].Molecular Plant,2017,10(11):1449-1460. doi:10.1016/j.molp.2017.10.001.
[14] Duan F Y,Giehl R F H,Geldner N,Salt D E,von Wirén N. Root zone-specific localization of AMTs determines ammonium transport pathways and nitrogen allocation to shoots[J].PLoS Biology,2018,16(10):e2006024. doi:10.1371/journal.pbio.2006024.
[15] Li C,Tang Z,Wei J,Qu H Y,Xie Y J,Xu G H. The OsAMT1.1 gene functions in ammonium uptake and ammonium-potassium homeostasis over low and high ammonium concentration ranges[J].Journal of Genetics and Genomics,2016,43(11):639-649. doi:10.1016/j.jgg.2016.11.001.
[16] Lauter F R,Ninnemann O,Bucher M,Riesmeier J W,Frommer W B. Preferential expression of an ammonium transporter and of two putative nitrate transporters in root hairs of tomato[J].Proceedings of the National Academy of Sciences of the United States of America, 1996,93(15):8139-8144. doi:10.1073/pnas.93.15.8139.
[17] Von Wirén N,Lauter F R,Ninnemann O,Gillissen B,Walch-Liu P,Engels C,Jost W,Frommer W B. Differential regulation of three functional ammonium transporter genes by nitrogen in root hairs and by light in leaves of tomato[J].The Plant Journal,2000,21(2):167-175. doi:10.1046/j.1365-313x.2000.00665.x.
[18] Simon-Rosin U,Wood C,Udvardi M K. Molecular and cellular characterisation of LjAMT2;1,an ammonium transporter from the model legume Lotus japonicus[J].Plant Molecular Biology,2003,51(1):99-108. doi:10.1023/a:1020710222298.
[19] Gu R L,Duan F Y,An X,Zhang F S,von Wirén N,Yuan L X. Characterization of AMT-mediated high-affinity ammonium uptake in roots of maize(Zea mays L.)[J].Plant & Cell Physiology,2013,54(9):1515-1524. doi:10.1093/pcp/pct099.
[20] Bajgain P,Russell B,Mohammadi M. Phylogenetic analyses and in-seedling expression of ammonium and nitrate transporters in wheat[J].Scientific Reports,2018,8(1):7082. doi:10.1038/s41598-018-25430-8.
[21] Sun Y C,Sheng S,Fan T F,Liu L,Ke J,Wang D B,Hua J P,Liu L H,Cao F Q. Molecular identification and functional characterization of GhAMT1.3 in ammonium transport with a high affinity from cotton(Gossypium hirsutum L.)[J].Physiologia Plantarum,2019,167(2):217-231. doi:10.1111/ppl.12882.
[22] Wu X Y,Yang H,Qu C P,Xu Z R,Li W,Hao B Q,Yang C P,Sun G Y,Liu G J. Sequence and expression analysis of the AMT gene family in poplar[J].Front Plant Sci,2015,6:337. doi:10.3389/fpls.2015.00337.
[23] 李磊,罗杰,李红,罗志斌. 毛果杨全基因组铵转运蛋白家族成员及其序列分析[J].西北农林科技大学学报(自然科学版),2011,39(2):133-142. doi:10.13207/j.cnki.jnwafu.2011.02.023.
Li L,Luo J,Li H,Luo Z B. Genome-wide analysis of the ammonium transporter gene family in Populus trichocarpa [J].Journal of Northwest A&F University(Nat Sci Ed),2011,39(2):133-142.
[24] 汪玉洁,陈日远,刘厚诚,宋世威,孙光闻. 生菜AMT1基因克隆及其在TiO2/ZnO纳米材料处理下的表达[J].核农学报,2018,32(4):665-672. doi:10.11869/j.issn.100-8551.2018.04.0665.
Wang Y J,Chen R Y,Liu H C,Song S W,Sun G W. Molecular cloning and expression analysis of LsAMT1 gene from lettuce under TiO2/ZnO nanomaterials[J].Journal of Nuclear Agricultural Sciences,2018,32(4):665-672.
[25] Castro-Rodríguez V,Assaf-Casals I,Pérez-Tienda J,Fan X,Avila C,Miller A,C novas F M. Deciphering the molecular basis of ammonium uptake and transport in maritime pine[J].Plant Cell and Environment,2016,39(8):1669-1682. doi:10.1111/pce.12692.
novas F M. Deciphering the molecular basis of ammonium uptake and transport in maritime pine[J].Plant Cell and Environment,2016,39(8):1669-1682. doi:10.1111/pce.12692.
[26] Chalhoub B,Denoeud F,Liu S,Parkin I A,Tang H,Wang X,et al. Plant genetics. early allopolyploid evolution in the post-neolithic Brassica napus oilseed genome[J].Science,2014,345(6199):950-953. doi:10.1126/science.1253435.
[27] Song J M,Guan Z L,Hu J L,Guo C C,Yang Z Q,Wang S,Liu D X,Wang B,Lu S P,Zhou R,Xie W Z,Cheng Y F,Zhang Y T,Liu K D,Yang Q Y,Chen L L,Guo L. Eight high-quality genomes reveal Pan-genome architecture and ecotype differentiation of Brassica napus[J].Nature Plants,2020,6(1):34-45. doi:10.1038/s41477-019-0577-7.
[28] Bouchet A S,Laperche A,Bissuel-Belaygue C,Snowdon R,Nesi N,Stahl A. Nitrogen use efficiency in rapeseed. A review[J].Agronomy for Sustainable Development,2016,36(2):38. doi:10.1007/s13593-016-0371-0.
[29] Esteban R,Ariz I,Cruz C,Moran J F. Review:Mechanisms of ammonium toxicity and the quest for tolerance[J].Plant Science,2016,248:92-101. doi:10.1016/j.plantsci.2016.04.008.
[30] Wang F, Gao J W, Liu Y, Tian Z W, Muhammad A, Zhang Y X, Jiang D, Cao W X, Dai T B. Higher ammonium transamination capacity can alleviate glutamate inhibition on winter wheat(Triticum aestivum L.)root growth under high ammonium stress[J].PLoS One,2016,11(8):e0160997. doi:10.1371/journal.pone.0160997.
[31] Wang F,Gao J W,Tian Z W,Liu Y,Abid M,Jiang D,Cao W X,Dai T B. Adaptation to rhizosphere acidification is a necessary prerequisite for wheat(Triticum aestivum L.)seedling resistance to ammonium stress[J].Plant Physiology and Biochemistry,2016,108:447-455. doi:10.1016/j.plaphy.2016.08.011.
[32] Gasteiger E, Hoogland C, Gattiker A, Duvaud S, Wilkin M R, Appel R, Bairoch A. Protein identification and analysis tools on the ExPASy server[M]//Walker J M.The proteomics protocols handbook.Totowa,NJ:Humana Press,2005:571-607.
[33] Wang X B,Wu J,Liang J L,Cheng F,Wang X W. Brassica database(BRAD)version 2.0:integrating and mining Brassicaceae species genomic resources[J].Database,2015,20:1-8. doi:10.1093/database/bav093.
[34] Hofmann K,Stoffel W. TMbase-A database of membrane spanning protein segments[J].Biological Chemistry Hoppe-Seyler,1993,374(166):166. doi:10.1056/NEJM199001043220121.
[35] Larkin M A,Blackshields G,Brown N P,Chenna R,McGettigan P A,McWilliam H,Valentin F,Wallace I M,Wilm A,Lopez R,Thompson J D,Gibson T J,Higgins D G. Clustal W and Clustal X version 2.0[J].Bioinformatics,2007,23(21):2947-2948.doi:10.1093/bioinformatics/btm404.
[36] Tamura K,Stecher G,Peterson D,Filipski A,Kumar S. MEGA6:molecular evolutionary genetics analysis version 6.0[J].Molecular Biology and Evolution,2013,30(12):2725-2729. doi:10.1093/molbev/mst197.
[37] Bailey T L,Boden M,Buske F A,Frith M,Grant C E,Clementi L,Ren J,Li W W,Noble W S. MEME SUITE:tools for motif discovery and searching[J].Nucleic Acids Research,2009,37:W202-W208. doi:10.1093/nar/gkp335.
[38] Combet C,Blanchet C,Geourjon C,Deléage G. Network protein sequence analysis[J].Trends in Biochemical Sciences,2000,25(3):147-150. doi:10.1016/s0968-0004(99)01540-6.
[39] Buchan D W A,Minneci F,Nugent T C O,Bryson K,Jones D T. Scalable web services for the PSIPRED protein analysis workbench[J].Nucleic Acids Research,2013,41:W349-W357. doi:10.1093/nar/gkt381.
[40] Mezulis S,Sternberg M J E,Kelley L A. PhyreStorm:A web server for fast structural searches against the PDB[J].Journal of Molecular Biology,2016,428(4):702-708. doi:10.1016/j.jmb.2015.10.017.
[41] Smith T F,Waterman M S. Identification of common molecular subsequences[J].Journal of Molecular Biology,1981,147(1):195-197. doi:10.1016/0022-2836(81)90087-5.
[42] Hoagland D R,Arnon D I. The water culture method for growing plants without soil[J].California Agricultural Experimental Station Bulletin,1950,347:32. doi:10.1016/S0140-6736(00)73482-9.
[43] Letunic I,Copley R R,Schmidt S,Ciccarelli F D,Doerks T,Schultz J,Ponting C P,Bork P. SMART 4.0:Towards genomic data integration[J].Nucleic Acids Research,2004,32(Database issue):D142-D144. doi:10.1093/nar/gkh088.
[44] Guruprasad K,Reddy B V B,Pandit M W. Correlation between stability of a protein and its dipeptide composition:A novel approach for predicting in vivo stability of a protein from its primary sequence[J].Protein Engineering, Design and Selection,1990,4(2):155-161. doi:10.1093/protein/4.2.155.
[45] von Wirén N,Gazzarrini S,Gojon A,Frommer W B. The molecular physiology of ammonium uptake and retrieval[J].Current Opinion in Plant Biology,2000,3(3):254-261.
[46] The International Wheat Genome Sequencing Consortium(IWGSC). A chromosome-based draft sequence of the hexaploid bread wheat(Triticum aestivum)genome[J].Science,2014,345(6194):1251788. doi:10.1126/science.1251788.
[47] Li F G,Fan G Y,Lu C R,Xiao G H,Zou C S,Kohel R J,et al.Genome sequence of cultivated upland cotton(Gossypium hirsutum TM-1)provides insights into genome evolution[J].Nature Biotechnology,2015,33(5):524-530. doi:10.1038/nbt.3208.
[48] Jones D M,Wells R,Pullen N,Trick M,Irwin J A,Morris R J. Spatio-temporal expression dynamics differ between homologues of flowering time genes in the allopolyploid Brassica napus[J].The Plant Journal,2018,96(1):103-118. doi:10.1111/tpj.14020.
[49] Conant G C,Wolfe K H. Turning a hobby into a job:how duplicated genes find new functions[J].Nature Reviews Genetics,2008,9(12):938-950. doi:10.1038/nrg2482.
[50] Otto S P. The evolutionary consequences of polyploidy[J].Cell,2007,131(3):452-462. doi:10.1016/j.cell.2007.10.022.
[51] Tegeder M,Masclaux-Daubresse C. Source and sink mechanisms of nitrogen transport and use[J].The New Phytologist,2018,217(1):35-53. doi:10.1111/nph.14876.
[52] Yuan L X,Loqueé D,Ye F H,Frommer W B,von Wirén N. Nitrogen-dependent posttranscriptional regulation of the ammonium transporter AtAMT1;1[J].Plant Physiology,2007,143(2):732-744. doi:10.1104/pp.106.093237.
[53] Ranathunge K,El-Kereamy A,Gidda S,Bi Y M,Rothstein S J. AMT1;1 transgenic rice plants with enhanced ![]() conditions[J].Journal of Experimental Botany,2014,65(4):965-979. doi:10.1093/jxb/ert458.
conditions[J].Journal of Experimental Botany,2014,65(4):965-979. doi:10.1093/jxb/ert458.
[54] Zhu Y N,Huang X M,Hao Y W,Su W,Liu H C,Sun G W,Chen R Y,Song S W. Ammonium transporter(BcAMT1.2)mediates the interaction of ammonium and nitrate in Brassica campestris[J].Frontiers in Plant Science,2019,10:1776. doi:10.3389/fpls.2019.01776.
[55] Hu B,Jiang Z M,Wang W,Qiu Y H,Zhang Z H,Liu Y Q,Li A F,Gao X K,Liu L C,Qian Y W,Huang X H,Yu F F,Kang S,Wang Y Q,Xie J P,Cao S Y,Zhang L H,Wang Y C,Xie Q,Kopriva S,Chu C C. Nitrate-NRT1.1B-SPX4 cascade integrates nitrogen and phosphorus signalling networks in plants[J].Nature Plants,2019,5(4):401-413. doi:10.1038/s41477-019-0384-1.
[56] Camacho-Cristóbal J J,Rexach J,Herrera-Rodríguez M B,Navarro-Gochicoa M T,Gonz lez-Fontes A. Boron deficiency and transcript level changes[J].Plant Science,2011,181(2):85-89. doi:10.1016/j.plantsci.2011.05.001.
lez-Fontes A. Boron deficiency and transcript level changes[J].Plant Science,2011,181(2):85-89. doi:10.1016/j.plantsci.2011.05.001.
[57] 陈宁美,欧阳舒毓,徐维烈,唐帅,韦善君,冯金朝,徐小静. 镉胁迫对盐芥根木栓质代谢的影响[J].河南农业科学,2018,47(10):105-110. doi:10.15933/j.cnki.1004-3268.2018.10.019.
Chen N M,Ouyang S Y,Xu W L,Tang S,Wei S J,Feng J C,Xu X J. Effect of cadmium stress on metabolism of root suberin in Thellungiella salsuginea[J].Journal of Henan Agricultural Sciences,2018,47(10):105-110.
[58] 杨德翠,徐青,赵方贵,柳冕,刘新. NaCl对不同抗性水稻氮代谢及相关基因表达的影响[J].华北农学报,2020,35(2):187-195. doi:10.7668/hbnxb.20190507.
Yang D C,Xu Q,Zhao F G,Liu M,Liu X. Effects of NaCl stress on nitrogen metabolism and related gene expression of rice with different resistance[J].Acta Agriculturae Boreali-Sinica,2020,35(2):187-195.