水稻是世界上最重要的喜温粮食作物之一,低温冷害是水稻整个生长发育过程中经常遭遇的一种自然灾害,是限制其生长发育、地理分布以及产量品质形成的主要环境因子之一。水稻因低温冷害而造成的减产已经是世界范围内普遍存在的问题,据统计中国每年因低温损失稻谷约3×109~5×109 kg。水稻芽期冷害影响水稻成活率、根和芽生长,导致幼苗活力降低[1]。其中根系是评判水稻能否继续生长的重要因素之一[2],因为根系活力是水稻生长情况的一个重要指标,根系活力越高,水稻地下部分对矿物质元素的吸收能力越强,地上部分生长也越好[3]。水稻苗期冷害会影响根系发育,导致根系降低矿物质吸收不足,影响水稻苗期的活力,进而导致水稻产量减少。多数研究结果表明,水稻苗期根数耐冷受数量性状(QTL)控制[4],因此,寻找水稻苗期根数耐冷QTL对于解析水稻耐冷分子遗传基础,选育耐冷新品种有重要的意义。
随着分子标记技术的发展,定位了多个低温胁迫下影响水稻幼苗发育的QTL。比如,以水稻芽期发芽率为耐冷鉴定标准,王棋等[5]以籼稻品种泸恢 99和粳稻品种沈农265杂交所产生的144个家系为材料共检测到了3个发芽期耐冷性QTLs分别位于第 3,5,9染色体上命名为 qLTG-3、qLTG-5、qSCT-9;纪素兰等[6]以Kinmaze/DV85重组自交系 (RILs) 群体为材料检测到11个QTL,其中qLTG-7和qLTG-11可在 3个环境中稳定表达。林静等[7]利用以籼稻9311为受体、粳稻日本晴为供体的染色体片段置换系(CSSLs),在5,7号染色体上鉴定出4个芽期耐冷QTL。
死苗率作为水稻耐冷评判标准,严长杰等[8]以籼稻品种南京11和粳稻品种巴利为亲本构建了67个家系,并在第7号染色体G379b~RG4区间发现存在耐冷有关的基因Cts7。Liu等[9]以存活率为指标,检测到了分布在6条染色体上的6个QTLs。陈亮等[10]以发芽力作为水稻芽期耐冷评判标准,以珍汕97B与多年生稻 AAV002863为亲本的198个家系,构建了140个SSR标记的连锁图谱,并检测到2个低温发芽力相关的基因座;姜树坤等[11]以芽期冷处理后恢复状态为鉴定水稻耐冷评判标准,并把秧苗的恢复状况分为5个等级;在水稻的1,3,9,11号染色体中检测到5个芽期耐冷的QTL。韩龙植等[12]以籼粳交密阳23/吉冷1号的F2∶3 200个家系作为作图群体,以水稻根长、根质量为指标在自然条件和12 ℃冷水胁迫下,检测到在第1,2,6,11,12染色体上17个与根系性状相关的QTL,对表型变异的解释率为5.8%~15.2%,其中与最大根的根径相关,位于第2染色体RM263~RM6区间的qCRD2和位于第11染色体RM21~RM206区间的qCRD11,以及与根干质量相关,位于第2染色体RM262~RM263区间的qCRWT2和位于第11染色体RM229~RM21区间的qCRWT11贡献率较大,分别为15.0%,15.2%,10.6%和12.2%。
虽然对水稻苗期耐冷性进行了较多的QTL定位,这些QTLs在水稻的12条染色体均有分布[13],但是检测到的水稻芽期耐冷QTL大多与发芽率或根长、根质量相关,与芽期根数相关的QTL很少。另外,不同遗传群体得出的结果也不尽相同,因此,有必要利用更多的遗传群体对水稻芽期耐冷QTL进行分析,发掘更多的根数耐冷QTL,为水稻冷胁迫根数分子育种提供参考。基于此,本试验以新构建的9311(受体)/日本晴(供体)染色体片段置换系群体为材料,对低温冷胁迫下控制水稻芽期根数QTL进行定位分析,以期为克隆水稻低温胁迫下控制根数发育的基因奠定基础。
1 材料和方法
1.1 试验材料
该试验材料是以籼稻9311作为轮回亲本,粳稻日本晴作为供体亲本构建获得的包含121个株系群体。于2019年7月采集水稻叶片组织样本,DNA提取、文库构建和全基因组测序由华大基因科技有限公司协助完成并得出高质量bin标记。
1.2 发芽期耐冷性鉴定
苗期水稻耐冷鉴定采取的是李太贵[14]的方法并微做改动。从亲本9311和日本晴以及CSSLs群体中选取30粒籽粒饱满的种子先经过5.5%的次氯酸钠消毒后,用蒸馏水漂洗干净。准备无菌培养皿,并将种子放置于铺有滤纸的培养皿中,在培养皿中加入蒸馏水至刚好浸没种子,待种子在常温25 ℃环境下萌发后,置于恒温培养箱15 ℃低温条件下处理7 d,最后在25 ℃恢复生长7 d,测量根数。根数测定标准:每个家系选择其中整齐一致的10个种子测定其根数。重复3次,计算其平均值用于后期计算分析。
1.3 数据分析
IciMapping V4.2软件对CSSLs群体进行bin标记分析和QTLs定位。以LOD=2.5为检测阈值,当QTL的LOD值大于2.5时,认为存在QTL。QTL命名见参考文献[15]。用SPSS 22软件与Microsoft Excel 2017内置公式进行显著性分析。候选基因分析通过查阅http://rice.plantbiology.msu.edu/网站输入QTLs所在的物理位置找到基因名称并在http://www.ricedata.cn/gene/分析其功能。
2 结果与分析
2.1 根数分析
低温胁迫后,对9311和日本晴根数进行了调查,发现日本晴平均根数为4.50,9311平均根数为7.20,二者在P=0.01的水平上达到极显著差异。CSSLs群体根数平均值为4.84,最高值为7.20,最低值为3.13,CSSLs群体根数表现为连续正态分布,其中水稻根数在3~4的为8个;4~5的为69个;5~6的为35个;6~7的为4个;7~8的为2个,存在明显的超亲分离现象,符合QTL定位要求(表1、图1,2)。
表1 冷胁迫下亲本及CSSLs群体根数
Tab.1 The root number of the two parents and CSSLs population under cold stress

亲本Parents置换系群体 CSSLs population日本晴Nipponbare9311平均值Mean最高值Maximum最低值Minimum偏度Skewness峰度Kurtosis方差Variance4.50±0.21A7.20±0.75B4.847.203.130.571.030.51
注:A和B表示P=0.01水平差异显著。
Note: A and B mean statistically significant difference at P=0.01.
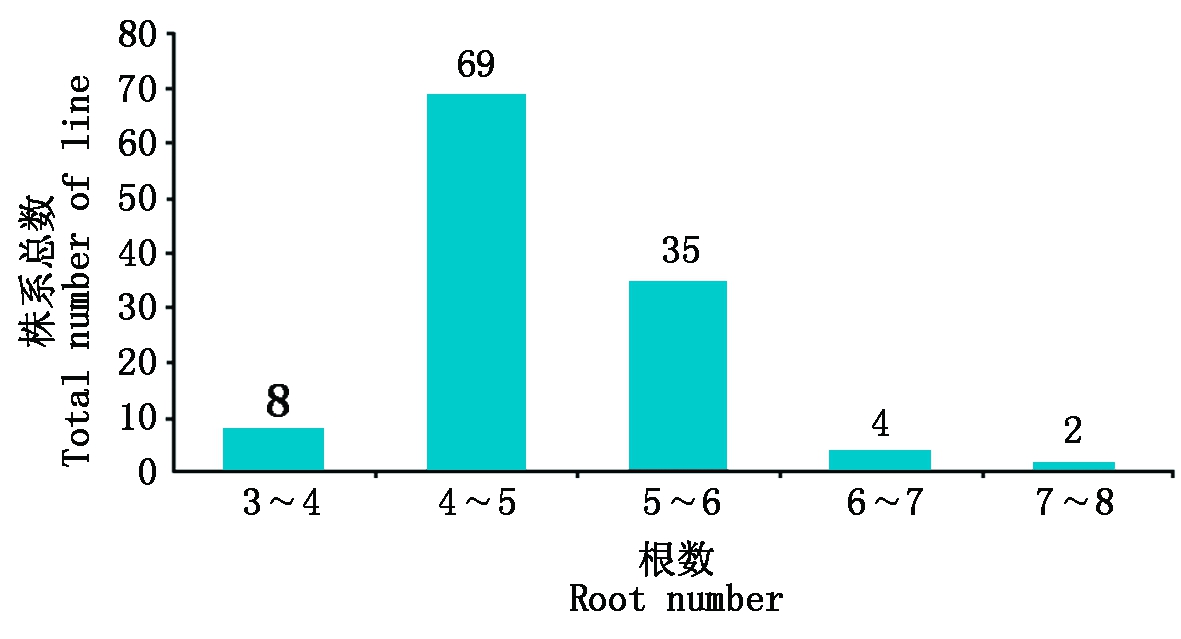
图1 CSSLs群体幼芽期15 ℃处理后根数频数分布
Fig.1 Frequency distribution of root number after 15 ℃ treatment at the sprout period using CSSLs population
2.2 QTL定位
采用IciMapping V4.2软件对低温胁迫下影响水稻根数的QTL进行分析。在15 ℃低温胁迫环境下共检测到2个与水稻根数相关的QTLs(表2),分别命名为qCSRN1.1和qCSRN3。这2个QTLs分别位于水稻第1号染色体chr1~bin43标记(31.75~33.15 Mb区间)和第3号染色体上chr3~bin179标记处(32.2~32.7 Mb区间),加性效应值分别为0.78和-0.67,可解释表型变异的16.26%,9.06%。其中qCSRN1.1增效等位基因来自供体亲本日本晴,qCSRN3增效等位基因来自受体亲本9311。
2.3 上位性QTL分析
本研究共检测出2对低温胁迫下影响水稻根数的上位性互作QTLs(表3)。QTL qCSRN1.1(第1号染色体chr1~bin43标记处)与QTL qCSRN1.2(第1号染色体chr1~bin52标记处)存在互作,LOD值为5.60,互作效应为-0.34;QTL qCSRN1.1(第1号染色体chr1~bin43标记处)与QTL qCSRN9(第9号染色体chr9~bin449标记处)存在互作,LOD值为5.75,互作效应为-0.43。
2.4 候选基因分析
对qCSRN1.1和qCSRN3候选基因分析发现,qCSRN1.1区间内包含138个基因,68个与蛋白合成相关的基因,42个与酶合成相关的基因,3个转录因子(图3);其中LOC_Os01g56200[16]、LOC_Os01g56470[17]、LOC_Os01g57340[18]已被克隆,这3个基因与稻瘟病、白叶枯病相关。qCSRN3区间内包含88个基因,62个与蛋白合成相关的基因,9个与酶合成相关的基因,3个转录因子(图3),LOC_Os03g56950[19]、LOC_Os03g57190[20]、LOC_Os03g57220[21]、LOC_Os03g57240[22]已被克隆;LOC_Os03g57190与冷胁迫相关,是OsPCF6的候选基因,其负向调节水稻耐冷性,可能为qCSRN3的候选目标基因。

图2 染色体置换系图谱基因型图示
Fig.2 Genotype diagram of chromosome segment substitution lines
表2 检测到的耐冷性QTL位点
Tab.2 QTL for clod resistant detected at germination stage and seedling stage

标记名称Mark位点Locus染色体ChromosomeLOD值LOD value加性效应值Additive effect贡献率/%PVEchr1~bin43qCSRN1.114.910.7816.26chr3~bin179qCSRN332.86-0.679.06
表3 水稻芽期耐冷性的上位性效应位点
Tab.3 Epistasis effect of locus for cold tolerance at germination stage in rice

QTL标记名称Marker nameQTL标记名称Marker nameLOD值加性效应Additive effectqCSRN1.1chr1~bin43qCSRN1.2chr1~bin525.60-0.34qCSRN1.1chr1~bin43qCSRN9chr9~bin4495.75-0.43
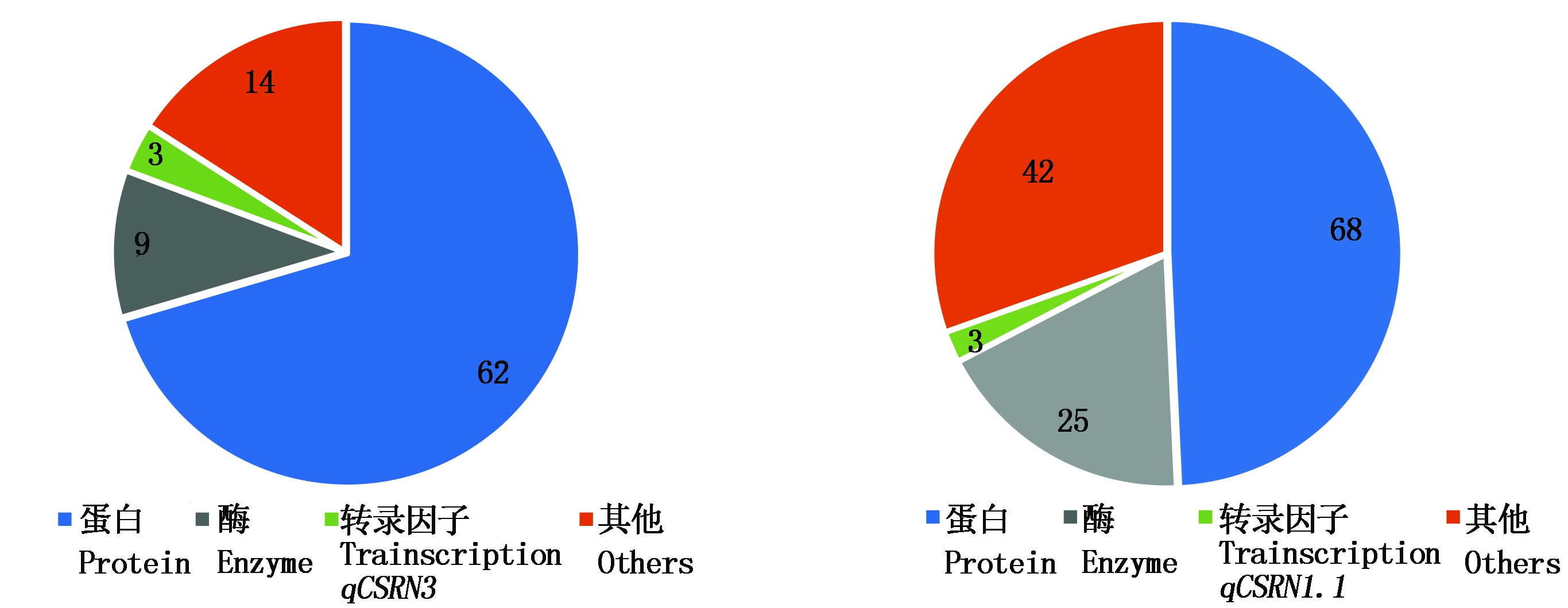
图3 候选基因分析
Fig.3 The analysis of candidate genes
3 讨论
水稻根系既是吸收养分和水分的重要器官又是多种激素、有机酸和氨基酸合成的重要场所,其形态和生理特征与地上部的生长发育、产量和品质形成均有着密切的联系[22]。2011年袁隆平院士将“根系发达”作为水稻新株型的主要性状之一,根系是否发达与水稻品种的好坏直接相关。根数是水稻根系的一个重要组成因素,有研究表明,水稻根数多少会影响水稻的产量与品质[23]。因此,对水稻根系的研究是非常必要的。而温度的变化对水稻根数影响较大。为研究低温胁迫下水稻芽期根数遗传表现,本研究利用9311/日本晴衍生的CSSLs群体对低温(15 ℃)条件下控制根数进行了遗传分析。结果显示低温胁迫下,水稻苗期根数生长除受QTL控制的同时还受到上位性QTL互作的影响,表明水稻上位性互作位点对于可能调控苗期根数冷胁迫起到了重要作用。
根数作为评判水稻苗期耐冷性的一个重要指标研究相对较少。前人在水稻根系研究中由于选用品种不同,处理方法不同,因此,检测到的水稻根数QTL位置、数目、贡献率都存在着较大差异。本研究利用9311(受体)和日本晴(供体)的染色体置换系群体,在15 ℃条件下检测出2个根数QTLs(qCSRN1.1和qCSRN3)。通过与前人根数QTL比较发现,qCSRN1.1与前人定位耐冷相关QTL(基因)位置不同,表明qCSRN1.1可能是一个新的耐冷QTL。qCSRN3与 Andaya等[24]定位的QTL qPSST-3(Chr3,32.0~35.1 MB)位置重叠,qPSST-3是控制水稻芽期耐冷的QTL,表明qCSRN3可能为一个冷胁迫控制水稻的主效位点。
水稻苗期耐冷性状属于数量性状,易受环境影响,不同环境、不同栽培条件、不同群体定位到的QTL不同[25],使用遗传背景复杂的重组自交系群体为后续克隆和解析这些耐冷基因增加了难度。本研究采用染色体置换系进行了苗期耐冷试验,该群体只存在群体之间片段代换的差异,并且该群体遗传背景简单,QTL定位消除了其他背景的干扰,可以通过进一步杂交形成F2次级分离群体把QTL定位到更小的区间,从而实现基因的克隆。并且本研究的优势在于前人对于水稻芽期根数耐冷研究较少,定位出的QTL可以补充苗期根系耐冷QTL数据,为今后聚合耐冷基因,提升育种效率打下坚实基础。
[1] 杨川航,王开,杨航,周大宾,李建修.水稻耐寒育种研究进展[J].中国农学通报,2009,25(6):113-116.
Yang C H, Wang K, Yang H, Zhou D B, Li J X. Advances in breeding of cold-tolerance rice[J].Chinese Agricultural Science Bulletin,2009,25(6):113-116.
[2] 李香玲,冯跃华.水稻根系生长特性及其与地上部分关系的研究进展[J].中国农学通报,2015,31(6):1-6.
Li X L,Feng Y H.Research advance on relation of aerial part and root traits of rice[J].Chinese Agricultural Science Bulletin,2015,31(6):1-6.
[3] 蔡志欢,张桂莲.水稻低温冷害研究进展[J].作物研究,2018,32(3):249-255.doi:10.16848/j.cnki.issn.1001-5280.2018.03.18.
Cai Z H,Zhang G L Research progress of low temperature and chilling injury of rice[J].Crop Research,2018,32(3):249-255.
[4] 刘友发,常硕其,常天根,欧阳翔,邓启云,夏石头,朱新广.超级杂交稻Y两优2号耐冷性QTL定位与杂种优势分析[J].分子植物育种,2020,18(7):2233-2243. doi:10.13271/j.mpb.018.002233.
Liu Y F,Chang S Q ,Chang T G,Ouyang X,Deng Q Y, Xia S T, Zhu X G. QTL mapping of cold tolerance and analysis of heterosis of super hybrid rice Y Liangyou 2[J].Molecular Plant Breeding,2020,18(7):2233-2243.
[5] 王棋,范淑秀,郭江华,陈兆赫,梁银培,刘振宇,殷业超,王嘉宇.利用籼粳交RIL群体对水稻发芽期和苗期耐冷性的QTL分析[J].华北农学报,2019,34(1):83-88.doi:10.7668/hbnxb.201751291.
Wang Q, Fan S X, Guo J H, Chen Z H, Liang Y P, Liu Z Y, Yin Y C ,Wang J Y. QTL analysis of cold tolerance at germination and seedling stages of rice[J].Acta Agriculturae Boreali-Sinica,2019,34(1):83-88.
[6] 纪素兰,江玲,王益华,刘世家,刘喜,翟虎渠,吉村醇,万建民.水稻种子耐低温发芽力的QTL定位及上位性分析[J].作物学报,2008,34(4):551-556. doi:10.3321/j.issn:0496-3490.2008.04.003.
Ji S L,Jiang L, Wang Y H, Liu S J, Liu X, Zhai H Q,Yoshimura A, Wan J M.QTL and Epistasis for low temperature germinability in rice[J].Acta Agronomica Sinica,2008,34(4):551-556.
[7] 林静,朱文银,张亚东,朱镇,赵凌,陈涛,赵庆勇,周丽慧,方先文,王艳平,王才林.利用染色体片段置换系定位水稻芽期耐冷性QTL[J].中国水稻科学,2010,24(3):233-236. doi:10.3969/j.issn.1001-7216.2010.03.004.
Lin J, Zhu W Y,,Zhang Y D,Zhu Z, Zhao L, Chen T, Zhao Q Y, Zhou L H, Fang X W,Wang Y P, Wang C L.Detection of quantitative trait loci for cold tolerance at the bud bursting stage by using chromosome segment substitution lines in rice (Oryza sativa)[J].Chinese Journal of Rice Science 2010, 24(3): 233-236.
[8] 严长杰,李欣,程祝宽,于恒秀,顾铭洪,朱立煌.利用分子标记定位水稻芽期耐冷性基因[J].中国水稻科学,1999,13(3):134-138.doi:10.16819/j.1001-7216.1999.03.002.
Yan C J,Li X,Cheng Z K,Yu H X,Gu M H,Zhu L H.Identifieation of QTL for cold tolerance at early seedling stage in rice[J].Chinese Journal of Rice Science,1999,13(3):134-138.
[9] Liu W Q, Pan X W, Xiong H B,Huang F L,Duan Y H,Li Y C,Min J,Liu S X,Liu L C,Wei X C,Zhao W J,Li X X. QTL mapping and validation of a segment responsible for early-seedling cold tolerance in rice [J].Euphytica,2019,216(1):7.doi:10.1007/s10681-019-2524-0.
[10] 陈亮,楼巧君,孙宗修,邢永忠,余新桥,罗利军.水稻低温发芽力的QTL定位[J].中国水稻科学,2006,20(2):159-164.doi:10.3321/j.issn:1001-7216.2006.02.008.
Chen L,Lou Q J ,Sun Z X,Xing Y Z,Yu X Q,Luo L J.QTL Mapping of low temperature germinability in rice[J].Chinese Journal of Rice Science,2006,20(2):159-164.
[11] 姜树坤,王立志,杨贤莉,李波,母伟杰,董世晨,车韦才,李忠杰,迟力勇,李明贤,张喜娟,姜辉,李锐,赵茜,李文华.基于高密度SNP遗传图谱的粳稻芽期耐低温QTL鉴定[J].作物学报,2020,46(8):1174-1184.doi:10.3724/SP.J.1006.2020.92066.
Jiang S K,Wang L Z, Yang X L, Li B,Mu W J, Dong S C, Che W C, Li Z J, Chi L Y, Li M X, Zhang X J, Jiang H, Li R, Zhao Q, Li W H.Detection of QTLs controlling cold tolerance at bud bursting stage by using a high-density SNP linkage map in japonica rice[J].Acta Agronomica Sinica,2020,46(8):1174-1184.
[12] 韩龙植,张三元,乔永利,阮仁超,张俊国,曹桂兰,高熙宗.冷水胁迫下水稻幼苗期根系性状的QTL分析[J].作物学报,2005,31(11):1415-1421. doi:10.3321/j.issn:0496-3490.2005.11.005.
Han L Z,Zhang S Y, Qiao Y L, Ruan R C, Zhang J G, Cao G L,Gao X Z.QTL analysis of root traits at the seedling stage in rice under cold water irrigation[J].Acta Agronomica Sinica,2005,31(11):1415-1421.
[13] 饶玉春,杨窑龙,黄李超,潘建伟,马伯军,钱前,曾大力.水稻耐冷胁迫的研究进展[J].分子植物育种,2013,11(3):443-450. doi:10.3969/mpb.011.000443.
Rao Y C,Yang Y L,Huang L C,Pan J W,Ma B J,Qian Q,Zeng D L.Research progress on cold stress in rice[J].Molecular Plant Breeding,2013,11(3):443-450.
[14] 李太贵. 水稻开花期的低温对结实率的影响[J]. 作物学报, 1988, 14(1):66-70.
Li T G.Effect of low temperature on seed setting rate in flowering stage of rice [J].Acta Agronomica Sinica,1988,14(1):66-70.
[15] McCouch S R. Gene nomenclature system for rice[J].Rice,2008, 1(1):72-84.doi:10.1007/s12284-008-9004-9.
[16] Chen X W,Chern M S, Canlas P E,Ruan D L,Jiang C Y, Ronald P C,David C B. An ATPase promotes autophosphorylation of the pattern recognition receptor XA21 and inhibits XA21-mediated immunity[J]. Proceedings of the National Academy of Sciences of the United States of Armerica,2010,107(17):8029-8034.doi:10.1073/pnas.0912311107.
[17] Koide Y, Kawasaki A, Telebanco-Yanoria M J,Hairmansis A,Nguyet N T M,Bigirimana J,Fujita D,Kobayashi N,Fukuta Y.Development of pyramided lines with two resistance genes, Pish and Pib, for blast disease (Magnaporthe oryzae B. Couch) in rice (Oryza sativa L.)[J]. Plant Breeding, 2010, 129(6):670-675.doi:10.1111/j.1439-0523.2010.01781.x.
[18] Yuan Y X,Zhong S H, Li Q,Zhu Z R,Lou Y G,Wang L Y,Wang J J,Wang M Y,Li Q L,Yang D L,He Z H. Functional analysis of rice NPR1-like genes reveals that OsNPR1/NH1 is the rice orthologue conferring disease resistance with enhanced herbivore susceptibility[J]. Plant Biotechnology Journal, 2007, 5(2):313-324.doi: 10.1111/j.1467-7652.2007.00243.x.
[19] Todaka D, Nakashima K, Maruyama K,Kidokoro S,Osakabe Y,Ito Y,Matsukura S,Fujita Y,Yoshiwara K,Takagi M O,Kojima M,Sakakibara H,Yamaguchi-Shinozaki K,Shinozaki K Y. Rice phytochrome-interacting factor-like protein OsPIL1 functions as a key regulator of internode elongation and induces a morphological response to drought stress[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(39):15947-15952.doi: 10.1073/pnas.1207324109.
[20] Wang S T,Sun X L, Hoshino Y, Yu Y,Jia B,Sun Z W,Sun M Z,Duan X B,Zhu Y M.MicroRNA319 positively regulates cold tolerance by targeting OsPCF6 and OsTCP21 in rice (Oryza sativa L.)[J].PLoS One, 2014, 9(3):e91357.doi: 10.1371/journal.pone.0091357.
[21] Zhang Z S, Xu Y Y,Xie Z W,Li X Y,He Z H,Peng X X.Association-dissociation of glycolate oxidase with catalase in rice: a potential switch to modulate intracellular H2O2 levels[J]. Molecular Plant, 2016, 9(5):737-748.doi: 10.1016/j.molp.2016.02.002.
[22] Guo T, Lu Z Q, Shan J X, Ye W W,Dong N Q,Lin H X. ERECTA1 acts upstream of the OsMKKK10-OsMKK4-OsMPK6 cascade to control spikelet number by regulating cytokinin metabolism in rice[J]. The Plant Cell, 2020,32(9):2763-2779.doi: 10.1105/tpc.20.00351.
[23] 章怡兰,林雪,吴仪,李梦佳,张晟婕,路梅,饶玉春,王跃星.水稻根系遗传育种研究进展[J].植物学报,2020,55(3):382-393.doi:10.11983/CBB20021.
Zhang Y L, Lin X, Wu Y,Li M J, Zhang S J, Lu M,Rao Y C,Wang Y X.Research progress on genetics and breeding of rice roots[J].Chinese Bulletin of Botany,2020,55(3):382-393.
[24] Andaya V, Mackill D. QTLs conferring cold tolerance at the booting stage of rice using recombinant inbred lines from a japonica×indica cross[J]. Theoretical and Applied Genetics, 2003, 106(6):1084-1090.doi: 10.1007/s00122-002-1126-7.
[25] Ranawake A L, Nakamura C. Cold tolerance of an inbred line population of rice (Oryza sativa L.) at different growth stages[J].Tropical Agricultural Research & Extension,2012,14(2):25-33.doi:10.4038/tare.V14i2.4838.