棉花种子的品质不仅影响棉花的播种品质(发芽势及发芽率)[1],而且影响附着其上纤维的品质,因为棉纤维是由棉种子的表层细胞突起发育而成的,其优劣很大程度上依赖于棉籽的营养供给[2]。此外,棉仁中含有20%~36%的脂肪酸和21%~45%的蛋白质,是重要的油料和蛋白资源[3]。因此,研究棉花种子发育的调控机理对于改良纤维品质、保障棉花高产稳产及提高棉花的附加值具有重要意义。
种子发育涉及细胞分裂、细胞分化、器官发生和胞间通信等过程。研究表明,基因印记、代谢途径、转录因子和激素信号在内的复杂信号通路和调控网络参与了种子的发育过程调控[4-8]。基因印记是影响种子发育最常见的因素,Costa等[9]研究表明,玉米中母系印记基因Meg1在胚乳转移细胞中表达,与种子的生长发育存在直接关系,证明该印迹基因在控制种子大小发育方面起着重要作用 。植物生长发育代谢途径也是影响种子发育的重要因素,其中细胞周期调控、泛素-蛋白酶体降解途径、ABC运输蛋白和碳/氮代谢途径等因素都会影响种子的发育[10-14]。转录因子参与调控种子发育各个时期,WOX、HAP3等转录因子参与调控种子形态发育[15-17],B3、bZIP等转录因子参与调控种子成熟[18-20],AP2/EREBP、bHLH等转录因子参与调控植物种子大小[21-22]。另外,植物激素是影响种子生长发育的重要因素之一,许多植物激素相关基因被证实在调控植物种子发育中发挥关键作用。生长素在启动种子的早期发育过程中具有重要作用,生长素信号途径通过调控珠被细胞分裂调控种子的发育,并对决定种子的最终大小具有重要作用[23-24]。油菜素内酯能够促进籽粒的发育,正调控籽粒大小的发育过程,但有关油菜素内酯调控种子大小的分子机制尚不清楚[25-26]。脱落酸调控种子发育的许多过程,胚胎营养物质的合成、种子休眠、抑制种子萌发等,对调控种子大小可能也有重要作用,对种子的大小起负调控作用[13,27]。赤霉素对植物种子萌发、打破种子休眠以及植物的花和种子的发育具有重要作用,赤霉素可能也调控种子大小[28-30]。miRNA可以调控ABA、生长素和油菜素甾醇类的信号传导,从而可能影响种子发育[31]。虽然植物种子发育相关机理已有大量的研究结果,但棉花作为纤维作物,研究多集中在棉纤维发育方面,对于种子发育方面的研究较少。本研究通过测定棉花种子发育相关突变体ims-15和其近等基因系Ji737系种子发育中期的转录组,筛选差异表达基因,分析ims-15突变体中发生改变的代谢通路,为解析棉花种子发育的调控机制提供依据。
1 材料和方法
1.1 试验材料
陆地棉品系Ji737系,由河北省农林科学院棉花研究所利用远缘杂交技术选育而成。
种子质量突变体ims-15,是由Ji737系中自然突变经8代自交纯化而来。
1.2 取样及数据调查
开花后,将Ji737系、ims-15分别挂牌标记开花时间并自交,取开花后30 d的棉桃,3个生物学重复,剥取胚珠,用液氮速冻后保存于-80 ℃冰箱,用于后续的转录组测序。吐絮后收获自交铃,考种计算千粒质量、衣分等。
1.3 种子发育期转录组分析
委托上海派森诺生物科技有限公司完成RNA提取、文库构建和转录组测序。以30DPA(Day past anthesis) 的Ji737系、ims-15的种子为转录组分析材料,采用Illumina平台进行双末端测序,以陆地棉TM-1的Ghirsutum_genome_HAU_v1.1基因组为参考基因组进行有参转录组分析,对获得的差异表达基因进行GO富集分析、KEGG富集分析、转录因子分析。
1.4 实时荧光定量PCR验证
选择与种子发育相关的30个差异表达基因进行实时荧光定量PCR(qRT-PCR)验证。采用RNAprep Pure 多糖多酚植物总RNA 提取试剂盒提取Ji737系、ims-15的30DPA种子的RNA,然后采用Fast King RT Kit(With gDNase)反转录试剂盒进行反转录合成cDNA后作为模板;最后采用Fast Fire qRT-PCR PreMix(SYBR Green)试剂,以棉花的Histon3基因作为内参基因,在Bio-Rad CFX96实时荧光定量PCR仪上进行qRT-PCR,每个基因设置3次生物学重复,3次技术重复,采用2-ΔΔCt法分析基因相对表达量(以上所用试剂均购于天根生化科技(北京)有限公司)。qRT-PCR用引物序列见表1。
2 结果与分析
2.1 ims-15突变体与Ji737系主要性状比较
ims-15与Ji737系最直观的差异是吐絮后ims-15的纤维不蓬松似僵瓣,种仁变小(图1);考种结果表明,千粒质量降低了20.1%,衣分降低了27.92百分点,均存在极显著差异(表2)。
表1 qRT-PCR引物
Tab.1 The primers used for qRT-PCR

基因Gene上游引物(5′-3′)Forward primer下游引物(5′-3′)Reverse primerGhir_D02G004190GGAGAATCTTCGGACCAGCCACAAGCTTTCCAGTCACAGCACGAGhir_D03G018560CAACGTCTCCAAGCTCTCCTCGATGCTGTTGCTCGGCGATTGTAGhir_A05G010930GCCAATAGCCCAGTCTCATGCCCCAATTCATGGCCGACACTTGCGhir_A01G016820TTTGGAAGGACATCGTGGTCGGCCGGGTCAACAGGATAAACCCCGhir_A10G011470ACCTCTTGCAGGCTCTAGAGAACACCTAAGCCCCTGGCTTCTGAACGhir_A07G007230GACGTTGGGCTATGTTGGGTGTCGGCTTTACCGGCATCATACCAGhir_A05G029380TTTGTTTGAGGAGCCAGGAGCCCTGAGGAAGCATCAACGCAAGCGhir_A01G006300GGGTTCAAAAACAATGCACCGCGCCAATGTTGCATCAAAGGGCAGhir_A05G015460GTGAGCCTCCATCTTACTTGACGGTTCCTGGCAAATGTCTCTGhir_A06G006190CTCATGTCAACACCGTTTGCGGTACGCAATCGTGACCGTCGAAAGhir_A02G003190GCCGTGTTACGGAAACAACCACTCTTCGGTTGCTTCGGCTTTGAGhir_D01G006310GGGTTCCAGGTTGTCCTCATGGTTCCAGTGGACCTTTGCCTGTGGhir_D01G021490 CCAGCTGCACCTACTTTTCCGTGTGGGTCCGAGTTTTGTTCCCAGhir_D12G023960GTGCATATTCAGCCAGGGTGGTAGGGGTCATTGCCAAACCGhir_D04G002380GTGAGGAAGGTGGTGGATAAACGTGGACATGGCGGATAATAGhir_D06G006280TCTCAAAAGCCGAACCACCGTTTGCCGGACATTTTATCGACGCTGhir_A11G026900CCATGGGACATCACCAACAAGCAACTGTGGGGGTTCACAAGGAAGhir_A10G006240TAGCAGCCTTGTGAGTTGGTAAAGAACTGCTCCTGGTCCAGhir_D12G028380ACCGGAAGTTCATGGTGTACGGGTTCGTTGCTCTTTTCGCCGTTGhir_D12G019690ACGGCACACTCGTTGTGTTACCACCAACCATTCCCCATTTCCTGGGhir_D12G026790GAGGAACCCACCATTAGAGAACCTCTTTGGTCCTCTCCAAGAACGhir_D12G026620CCAGGTGGAAAACAGCTTCGGTTGCTGTTGTGCTTGCTGGTACTGhir_A12G028210GGGAGTGCCCAGTTTTTGGTCTCGCCGGCAAATGGAATCAACTCGhir_A13G000130GCGAGCAGGAGAGAAAAGCCATTTTGCATCCACCGCTCTCTTCGGhir_D12G026630GATGGGTGCATTCGGTAGTTTCTCCACCATTTCCTCCTCTATGhir_D12G022560GTTAGCCCTGTTGGCTTCACCAGCCGTGGCCAGAAGTTTTACAGGhir_D13G016330CACCGCCACTACCAGTATCAGCCCAGCCCTTTAGCTGGCTTTGAGhir_D10G012620TGTGACTGGCAACTCGTTTAGCGCGAGACTCCCATAACATAACGhir_A03G019040CCCTACTGCTGGAACTAGAAAGGAAGTTGCCACCACCTCTAAGhir_A07G024770ATCGTCGTCGTCGTCATCATCGGACAACGATTCCTCCGTCCCTGHiston3TCAAGACTGATTTGCGTTTCCAGCGCAAAGGTTGGTGTCTTC

A.ims-15、Ji737吐絮对比;B.ims-15、Ji737种仁对比。
A.Comparison of boll opening between ims-15 and Ji737;B.Comparison of seed kernel of ims-15 and Ji737.
图1 ims-15与Ji737的性状表现
Fig.1 The characterization of ims-15 and Ji737
表2 ims-15与Ji737的表型数据
Tab.2 The trait data of ims-15 and Ji737

材料名称Sample千粒质量/gThousand kernel weight衣分/%Lint percentims-1585.31±0.45A12.82±0.30AJi737106.83±0.21B40.74±0.45B
注:数据后不同大写字母表示数据间极显著差异(P<0.01)。
Note:The different capital letters after data indicates highly significant difference between data at P<0.01.
2.2 测序结果及质量评估
本试验6个测序样品共获得39 275 600 400 bp的原始数据(Raw bases),碱基识别准确率在99.9%以上(Q30)的碱基占比为92.57%~93.47%;过滤后分别产生38 685 856,39 997 916,41 578 188,41 658 750,39 432 722,40 942 408个干净序列,共36 344 376 000 bp 干净数据;比对到参考基因组上的序列占比大于95%,其中只比对到一个位置的序列总数和比对到外显子区域的序列占比均高于95%,表明测序结果良好,数据可用于后续分析(表3)。
表3 测序数据统计及质量检测
Tab.3 Statistics and quality testing of sequencing data
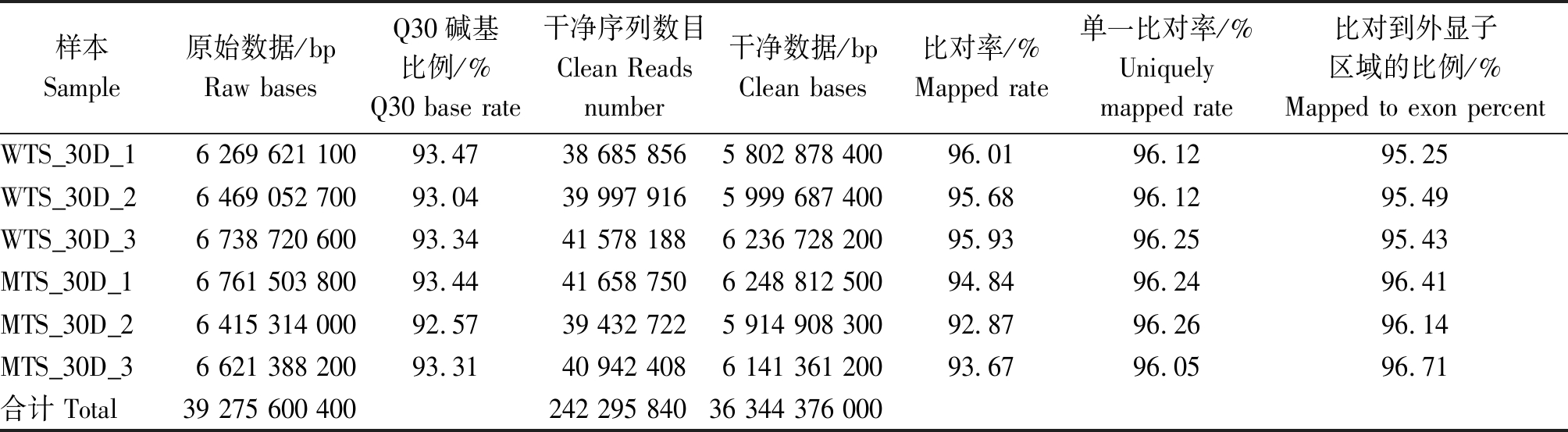
样本 Sample原始数据/bpRaw bases Q30碱基比例/%Q30 base rate干净序列数目Clean Reads number干净数据/bpClean bases 比对率/%Mapped rate单一比对率/%Uniquely mapped rate比对到外显子区域的比例/%Mapped to exon percentWTS_30D_16 269 621 10093.4738 685 8565 802 878 40096.0196.1295.25WTS_30D_26 469 052 70093.0439 997 9165 999 687 40095.6896.1295.49WTS_30D_36 738 720 60093.3441 578 1886 236 728 20095.9396.2595.43MTS_30D_16 761 503 80093.4441 658 7506 248 812 50094.8496.2496.41MTS_30D_26 415 314 00092.5739 432 7225 914 908 30092.8796.2696.14MTS_30D_36 621 388 20093.3140 942 4086 141 361 20093.6796.0596.71合计 Total39 275 600 400242 295 84036 344 376 000
2.3 基因表达数据分析
采用DESeq对基因表达进行差异分析,以表达差异倍数绝对值|Fold change|>2,显著性P-value<0.05作为筛选差异表达基因的阈值,从ims-15和Ji737系30DPA的种胚中筛选到4 239个差异表达基因,其中ims-15相比Ji737上调表达基因2 229个(52.6%),下调表达基因2 010个(47.4%)。分析上、下调基因的差异表达倍数(Fold change),差异倍数2~10倍的差异基因在上、下调基因中均占比最高,分别为76%,91%;差异倍数大于20的上调基因数目多于下调表达的基因数目(图2)。
2.4 差异表达基因的GO富集分析
利用topGO对获得的差异表达基因进行GO富集分析,4 329个差异表达基因共富集到了375个条目,包含生物过程分类的228个条目、分子功能分类的112个条目、细胞组分分类的35个条目。根据GO富集结果,通过Rich factor、FDR值和富集到此GO 条目上的基因个数来衡量富集的程度,筛选出FDR值最小的即富集最显著的前20个条目(图3)。
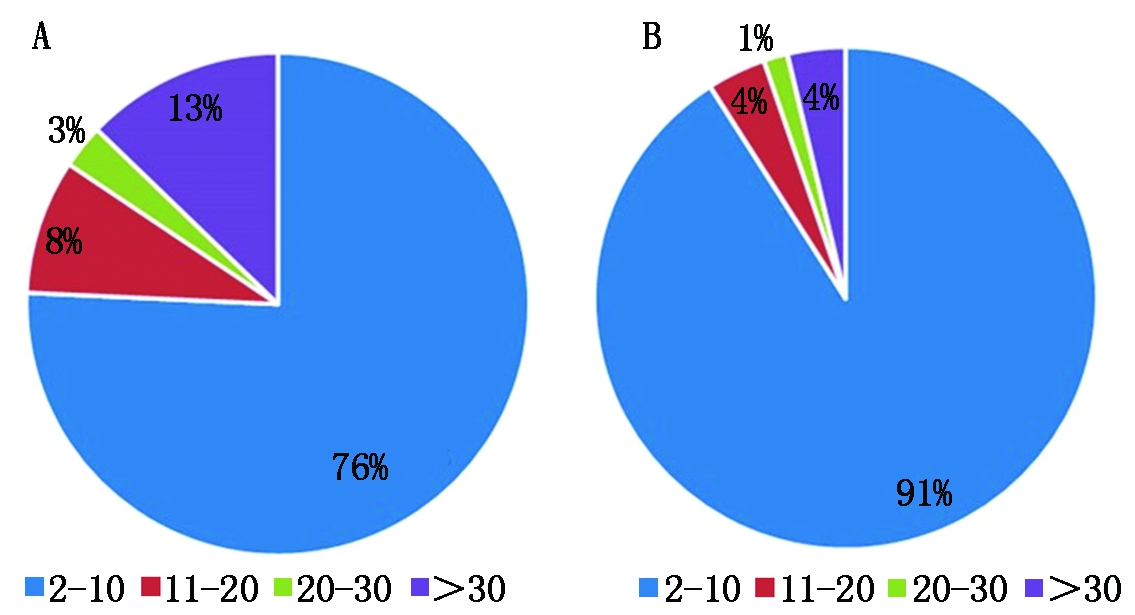
A.上调表达基因差异倍数分布;B.下调表达基因差异倍数分布。
A.Multiple distribution of upregulated genes;
B.Multiple distribution of downregulated genes.
图2 差异表达基因差异倍数分布
Fig.2 The multiple distribution of DEGs
其中包含于生物过程类别的7个条目是细胞壁高分子分解代谢过程、光合作用-光能捕获、细胞壁高分子代谢过程、光合作用、细胞碳水化合物代谢过程和氧化还原过程;属于分子功能类别的4个条目为叶绿素结合、四吡咯结合、铁离子结合和水解酶活性,作用于糖基键;属于细胞组分类别的9个条目为光系统Ⅰ、光系统、光合膜、类囊体部分、类囊体、膜的整体组成、膜固有组成、膜和膜部分。三大类别中共有8个光合作用相关条目,依据富集程度由大到小依次为光合作用-光能捕获、光系统Ⅰ、叶绿素结合、光系统、光合膜、光合作用、类囊体部分和类囊体。富集于这8个条目的差异基因共47个,其中在突变体中下调表达基因40个,占85.11%,且富集到光能捕获、叶绿素结合和光系统Ⅰ等条目的差异基因在突变体中均下调表达(表4)。
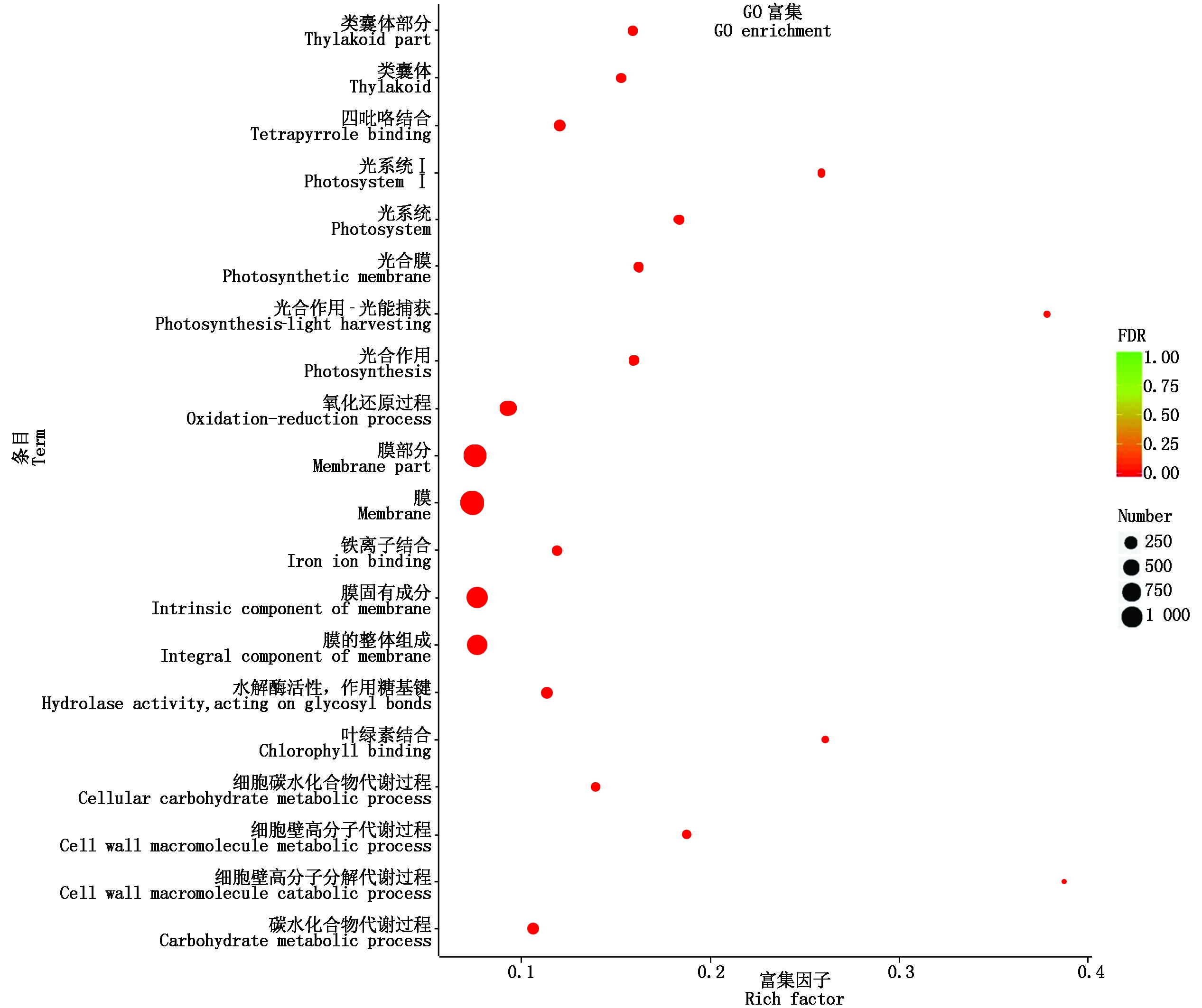
图3 差异表达基因的GO富集分析
Fig.3 GO enrichment analysis of DEGs
表4 光合作用相关条目中的差异表达基因
Tab.4 The DEGs in photosynthesis-related terms
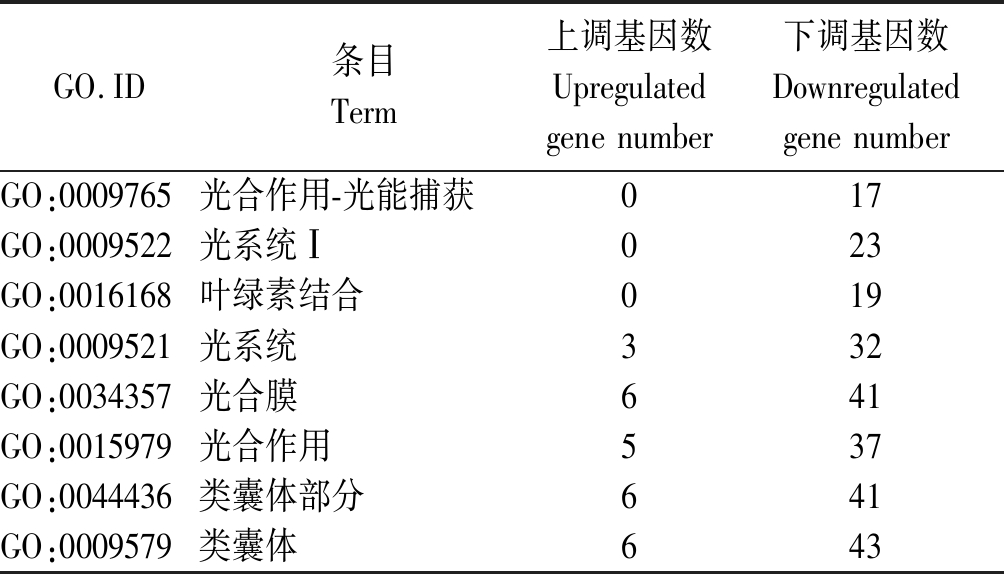
GO.ID条目Term上调基因数Upregulated gene number下调基因数Downregulated gene numberGO:0009765光合作用-光能捕获017GO:0009522光系统Ⅰ023GO:0016168叶绿素结合019GO:0009521光系统332GO:0034357光合膜641GO:0015979光合作用537GO:0044436类囊体部分641GO:0009579类囊体643
2.5 差异表达基因的KEGG富集分析
差异表达基因被注释到117个代谢通路中,主要涉及细胞过程、环境信息处理、遗传信息处理、代谢和有机系统5个大类18个小类。进一步从117个代谢途径中筛选出光合作用-天线蛋白、类黄酮生物合成、植物激素信号转导、苯丙素的生物合成、丝裂原活化蛋白激酶信号途径(MAPK信号途径)、糖酵解/糖异生、淀粉和蔗糖代谢等显著富集的代谢通路20个(图4),其中脂质代谢、氨基酸代谢的相关通路分别有6,5个,占显著富集代谢通路的55%;能量代谢、碳代谢、其他次级代谢产物生物合成、信号转导相关通路各2个;辅助因子和维生素代谢相关通路1个(表5)。
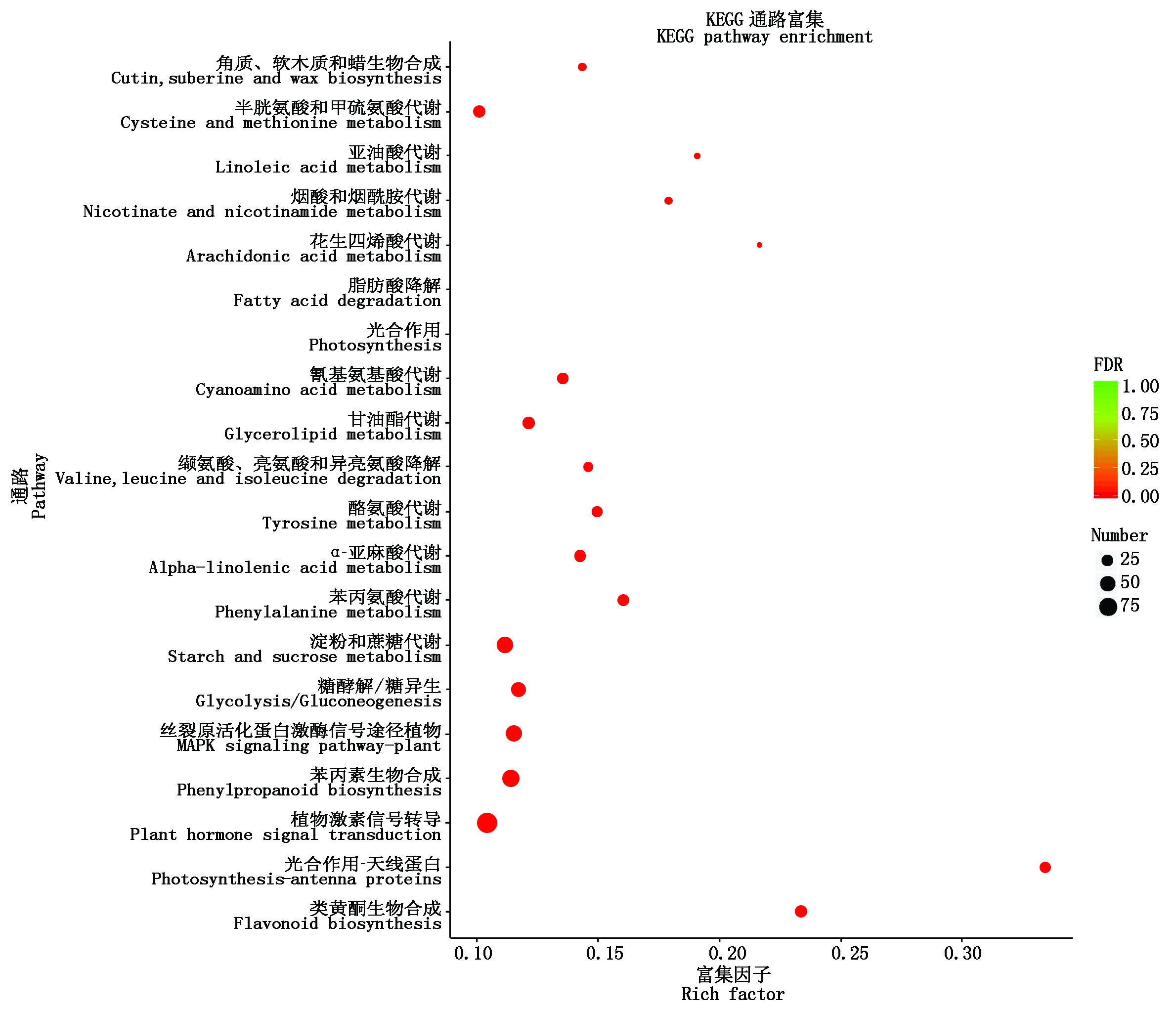
图4 差异表达基因显著富集生物过程
Fig.4 The bubble diagram of significant enrichment biological processes
表5 显著富集生物过程的功能分类
Tab.5 Functional classification of significantly enriched biological processes
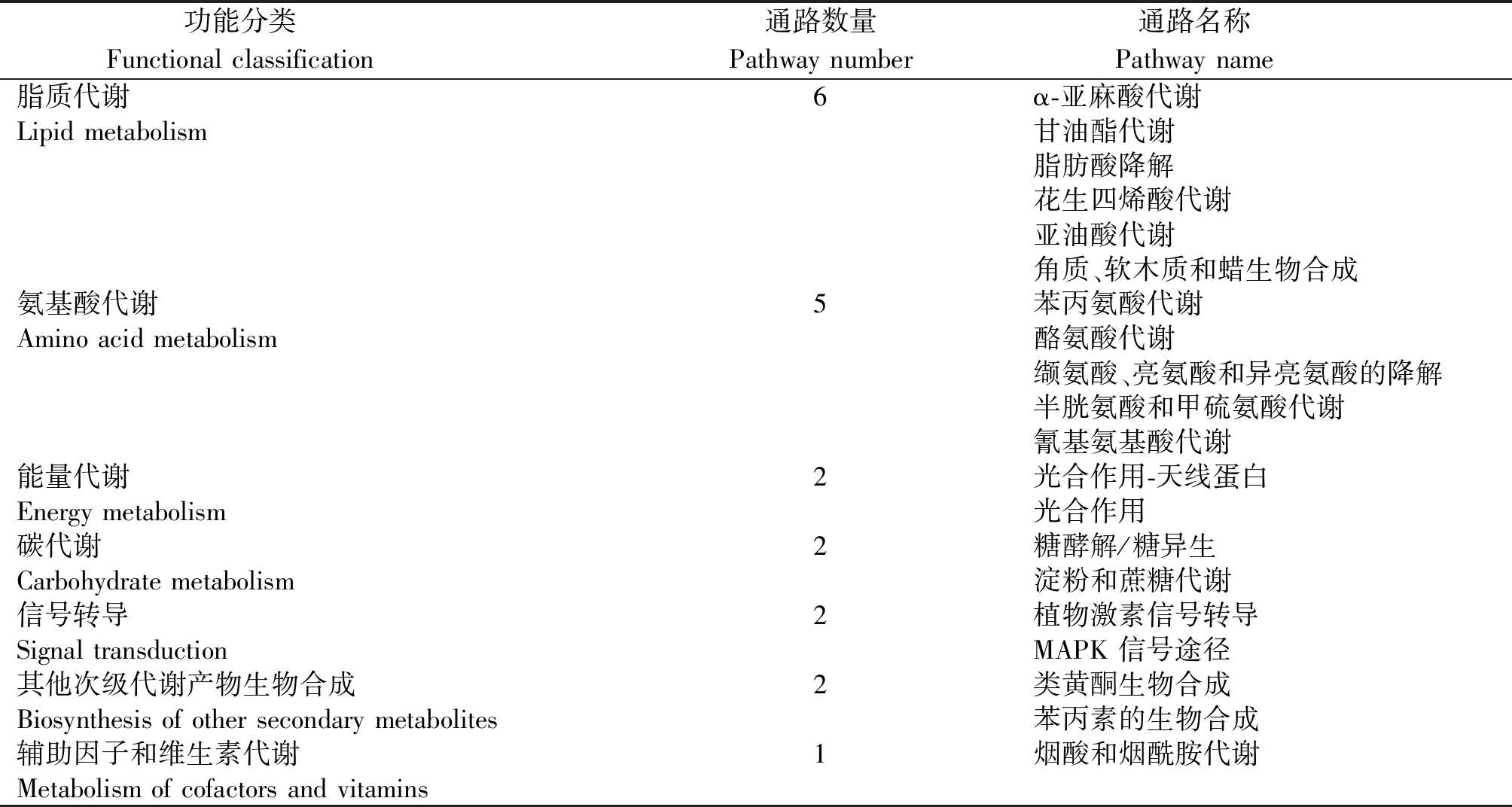
功能分类 Functional classification 通路数量Pathway number通路名称 Pathway name 脂质代谢6α-亚麻酸代谢Lipid metabolism甘油酯代谢脂肪酸降解花生四烯酸代谢亚油酸代谢角质、软木质和蜡生物合成氨基酸代谢5苯丙氨酸代谢Amino acid metabolism酪氨酸代谢缬氨酸、亮氨酸和异亮氨酸的降解半胱氨酸和甲硫氨酸代谢氰基氨基酸代谢能量代谢2光合作用-天线蛋白Energy metabolism光合作用碳代谢2糖酵解/糖异生Carbohydrate metabolism淀粉和蔗糖代谢信号转导2植物激素信号转导Signal transductionMAPK 信号途径其他次级代谢产物生物合成2类黄酮生物合成Biosynthesis of other secondary metabolites苯丙素的生物合成辅助因子和维生素代谢1烟酸和烟酰胺代谢Metabolism of cofactors and vitamins
光合作用-天线蛋白是富集程度最高的通路,参与该通路的18个差异基因在ims-15中均下调表达。另外,显著富集的光合作用通路中共有26个差异基因,其中23个在ims-15中下调表达(表6)。
表6 光合作用相关通路中差异表达基因
Tab.6 DEGs in photosynthesis-related pathways

途径编号Pathway ID途径名称Pathway name上调基因数目UP-regulation gene number 下调基因数目Down-regulation gene number ghi00196光合作用-天线蛋白018ghi00195光合作用323
植物激素信号转导途径是显著富集的代谢通路中注释到差异基因数目最多的通路,说明植物激素信号转导在种子发育中起着重要作用。分析不同激素通路发现,油菜素内脂信号通路中富集到的6个差异基因均下调表达;乙烯信号通路中富集到上调基因15个,下调基因1个;细胞分裂素信号通路中富集到上调基因8个,下调基因1个;生长素信号通路中富集到下调基因22个,上调基因9个;脱落酸信号通路中富集到上调基因14个,下调基因5个;赤霉素信号通路中富集到上调基因4个,下调基因3个;茉莉酸信号通路中富集到上调基因6个,下调基因2个;水杨酸信号通路中富集到上、下调基因各1个(图5)。
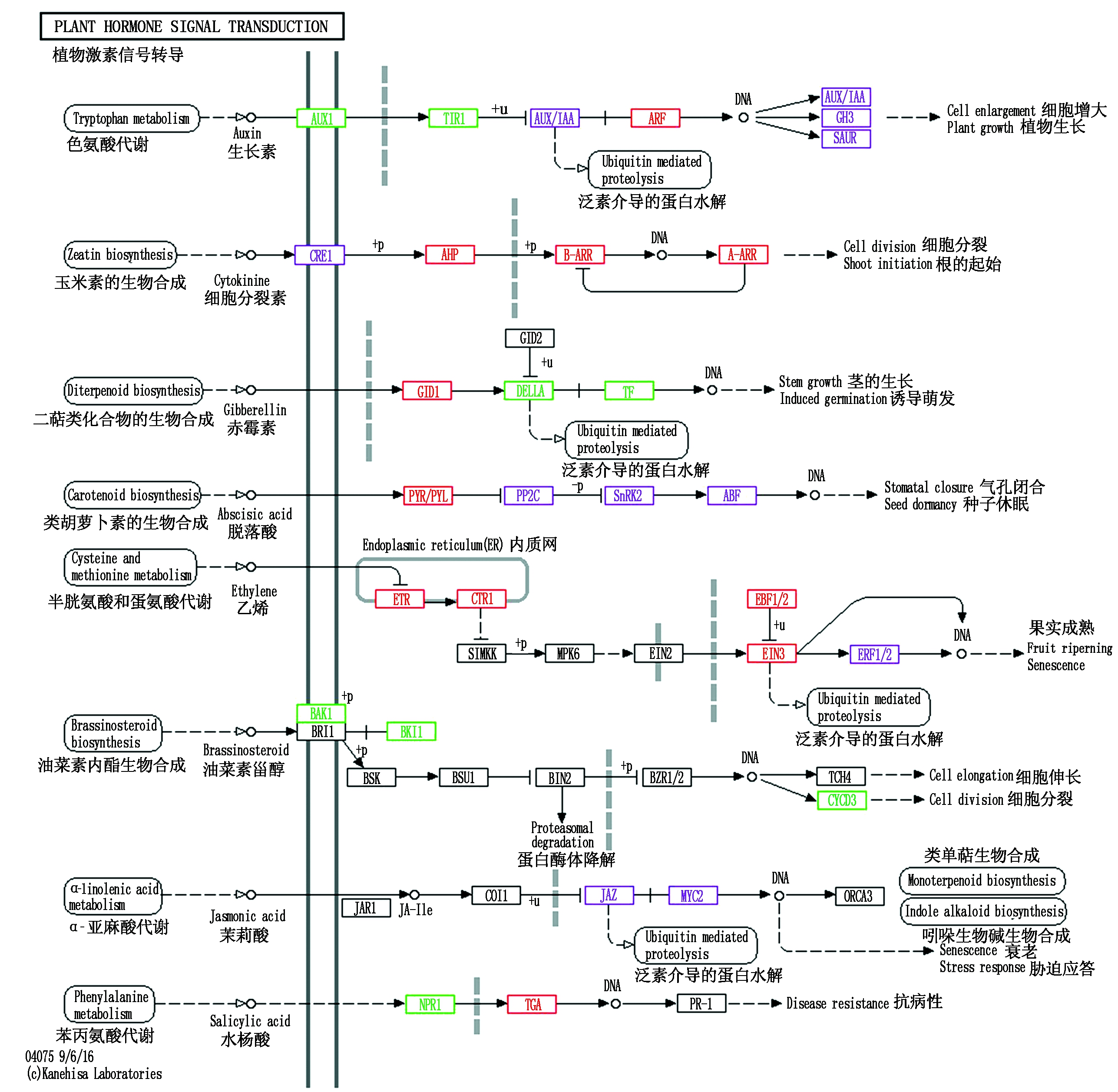
红色表示富集到的差异基因表达上调,绿色表示基因下调,紫色表示基因既有上调也有下调。
Red indicates up regulation of DEGs,green indicates down regulation of DEGs,and purple indicates both up regulation and down regulation of DEGs.
图5 植物激素信号转导途径中的差异表达基因
Fig.5 DEGs in plant hormone signal transduction pathways
2.6 转录因子分析
种子发育受一系列转录因子调控,通过转录因子分析,筛选到448个差异表达的转录因子,分属于41个转录因子家族,包含bHLH、MYB、WRKY、NAC、bZIP、MADS-box、Dof、B3、WOX、AP2/EREBP等已被证实参与种子发育调控的转录因子家族(图6)。其中,ERF、bHLH、MYB 3个转录因子家族包含的差异表达基因明显多于其他家族,分别包含59,56,49个差异表达基因。
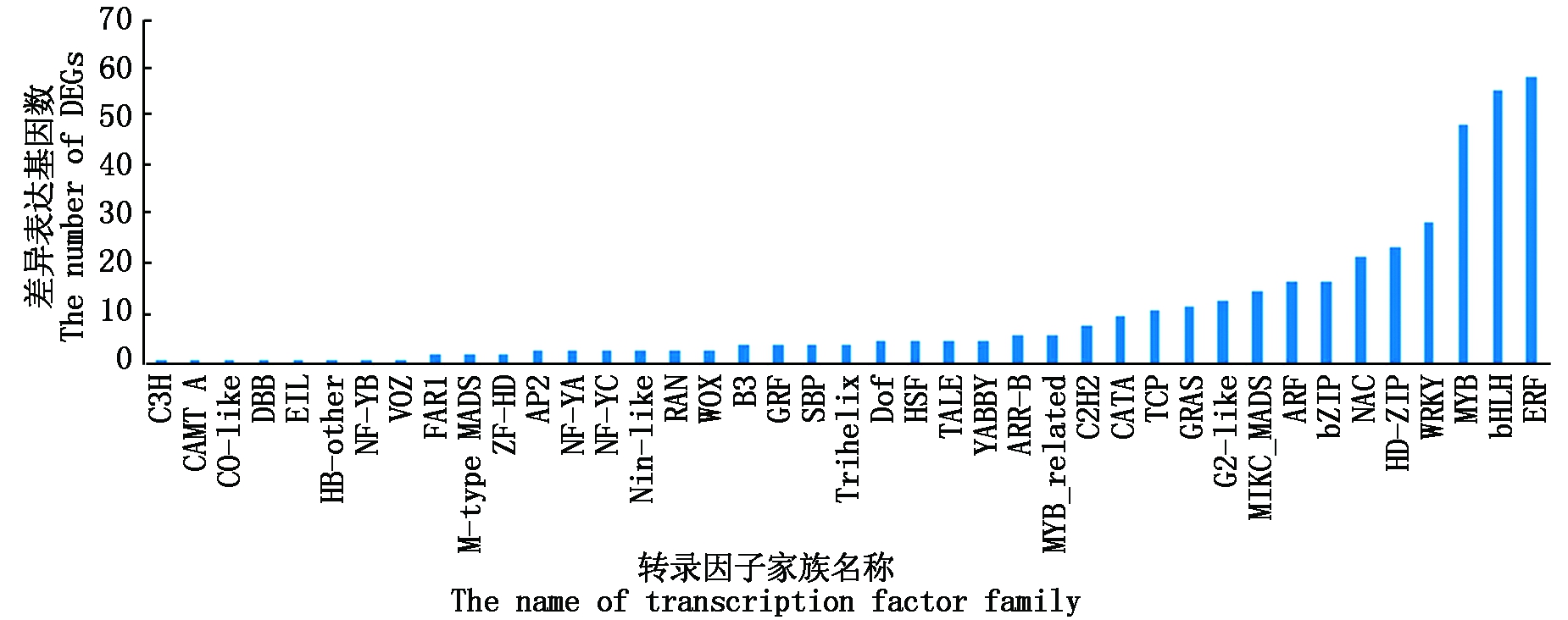
图6 差异表达的转录因子
Fig.6 The differentially expressed transcription factors
2.7 荧光定量验证RNA-seq结果
为了验证RNA-seq差异分析的准确性,从富集程度最高的光合作用-天线蛋白通路、注释到差异基因最多的植物激素信号转导通路及起调控种子发育作用的转录因子家族中筛选出30个差异表达基因进行了qRT-PCR分析,并对RNA-seq和qRT-PCR的结果进行了相关性分析,两者的相关系数R2=0.955 9,说明RNA-seq的结果准确可靠(图7)。验证基因中包含4个光合作用-天线蛋白通路的基因,分别为Ghir_A03G019040(CAP10A)、Ghir_A05G015460(CAB21)、Ghir_A07G007230(LHCA4)、Ghir_D01G006310(CAB13); qRT-PCR的结果显示该4个基因在ims-15中的表达量较Ji737分别降低4.25,14.33,7.31,5.91倍;这与RNA-seq结果中富集到光合作用-天线蛋白通路的基因均下调表达的结果一致。
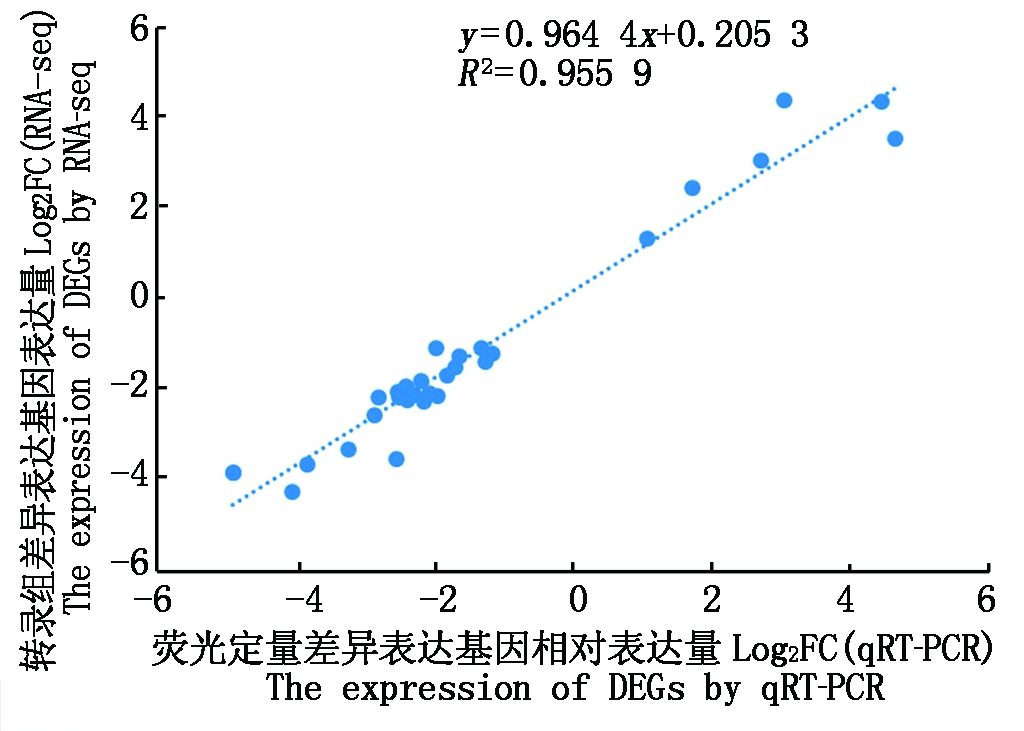
图7 RNA-seq和qRT-PCR相关性分析结果
Fig.7 The correlation analysis results between RNA-seq and qRT-PCR
3 结论与讨论
本研究利用RNA-seq分析了棉花种子发育异常突变体(ims-15)及其近等基因系(Ji737系,种子发育正常)发育中期种胚的转录本差异。与发育正常种胚相比,在ims-15种胚中共有2 229个基因上调表达,2 010个基因下调表达。GO功能富集分析发现差异基因显著富集在光合作用相关的条目; KEGG富集分析表明光合作用-天线蛋白是富集程度最高的通路,富集到该通路的差异表达基因在ims-15中均下调表达;植物激素信号转导途径是富集到差异表达基因最多的通路;转录因子分析表明,多个基因家族参与种子发育调控。
胚性光合作用在合成种子贮存物质中起重要作用。许多植物的种子在胚胎发生期间是绿色的,虽然种子主要是库组织,但它们含有叶绿体,具有典型光合作用机制的类囊体结构和酶[32]。种子中的光合作用至少可以通过3种方式增加生物合成通量:光反应可以产生NADPH和ATP,用于能量需求大的脂肪酸生物合成;氧气的释放可能有助于防止种子内缺氧[22];暗反应可以通过固定呼吸产生的CO2为新陈代谢提供中间体来提高种子的生物合成效率[33]。本研究中Ji737和ims-15种子发育期差异表达的基因显著地富集于光合作用,且差异基因大多在ims-15中下调表达,尤其是参与光合作用-光能捕获的基因在ims-15均下调表达。因此,推测ims-15光捕获能力下降,致使胚性光合作用能力降低,是造成种子质量降低的直接原因。
植物激素是影响种子生长发育的重要因素之一,不同植物激素在种子发育的不同阶段发挥着不同的作用[34]。本研究中植物激素信号转导途径是注释到差异基因数目最多的代谢途径,其中ims-15中起正调控种子大小发育功能的信号转导途径中下调基因的数目显著多于上调基因数目,包括生长素信号转导途径中下调基因22个、上调基因9个,油菜素内酯途径中6个差异表达基因均为下调[23-26]。相反,起负调控作用的信号转导途径中上调基因数目显著多于下调基因数,其中细胞分裂素转导途径中上调基因8个、下调基因仅1个,脱落酸转导途径中上调基因14个、下调基因5个[27-28]。这些结果说明植物激素变化对ims-15种子质量降低起着重要作用。
种子发育同时受一系列转录因子调控[35],它们与植物激素信号转导等信号转导途径结合,构成一个调控种子发育调控网络来调控和监管整个发育过程。在本研究中筛选获得了大量的影响种子质量形成的转录因子家族成员,包括调控胚乳发育、贮藏蛋白合成和脂肪含量的Dof家族[36],调控种子体积大小的WRKY 家族[37],调控种子大小、种子贮藏物质积累的AP2/EREBP家族等[38],说明转录因子活性的变化对ims-15种子质量降低起着重要作用。
本研究通过对种子质量突变体ims-15和其正常种子质量近等基因系Ji737种子发育期转录水平的差异分析,发现了一些与种子质量发育密切相关的代谢途径,尤其是胚性光合作用途径在种子质量形成中起重要作用;发现了植物激素信号转导途径和转录因子在种子质量形成中起着重要调控作用。
[1] 鲁黄均,马家璋.不同环境条件下生产的棉花种子品质的差异及对后代的影响[J].种子,1990(6):17-21.doi:10.16590/j.cnki.1001-4705.1990.06.053.
Lu H J ,Ma J Z. The quality differences of cotton seeds(Gossypium hirsutum L.)produced under different environmental conditions and effects on the growth and development of progeny[J]. Seed,1990(6):17-21.
[2] 郭婷婷,徐鹏,张香桂,沈新莲.陆地棉棉籽营养品质性状与主要农艺性状、纤维品质性状的相关分析[J].江苏农业科学,2012,40(3):79-81. doi:10.3969/j.issn.1002-1302.2012.03.028.
Guo T T,Xu P,Zhang X G,Shen X L. Correlation analysis between nutritional quality characters and main agronomic characters and fiber quality characters of upland cotton seed[J]. Jiangsu Agricultural Sciences,2012,40(3):79-81.
[3] 孙善康,陈建华,项时康,魏守军.棉花种子营养品质研究[J].中国农业科学,1987,20(5):12-16.
Sun S K,Chen J H,Xiang S K,Wei S J. Study on the nutritional quality of cotton seeds [J]. Scientia Agricultura Sinica,1987,20(5):12-16.
[4] Xu W,Dai M Y,Li F,Liu A Z. Genomic imprinting,methylation and parent-of-origin effects in reciprocal hybrid endosperm of castor bean [J]. Nucleic Acids Research,2014,42(11):6987-6998. doi:10.1093/nar/gku375.
[5] Ruan Y L. Sucrose metabolism:gateway to diverse carbon use and sugar signaling[J]. Annual Review of Plant Biology,2014,65:33-67. doi:10.1146/annurev-arplant-050213-040251.
[6] Weber H,Borisjuk L,Wobus U. Molecular physiology of legume seed development[J]. Annual Review of Plant Biology,2005,56:253-279. doi:10.1146/annurev.arplant.56.032604.144201.
[7] Le B H,Cheng C,Bui A Q,Wagmaister J A,Henry K F,Pelletier J,Kwong L,Belmonte M,Kirkbride R,Horvath S,Drews G N,Fischer R L,Okamuro J K,Harada J J,Goldberg R B. Global analysis of gene activity during Arabidopsis seed development and identification of seed-specific transcription factors[J]. Proceedings of the National Academy of Sciences of the United States of America,2010,107(18):8063-8070. doi:10.1073/pnas.1003530107.
[8] Orozco-Arroyo G,Paolo D,Ezquer I,Colombo L. Networks controlling seed size in Arabidopsis[J]. Plant Reproduction,2015,28(1):17-32. doi:10.1007/s00497-015-0255-5.
[9] Costa L M,Marshall E,Tesfaye M,Silverstein K A T,Mori M,Umetsu Y,Otterbach S L,Papareddy R,Dickinson H G,Boutiller K,VandenBosch K A T,Ohki S,Gutierrez-Marcos J F. Central cell-derived peptides regulate early embryo patterning in flowering plants[J]. Science,2014,344(6180):168-172. doi:10.1126/science.1243005.
[10] Tian S J,Wu J J,Li F,Zou J W,Liu Y W,Zhou B,Bai Y,Sun M X. NtKRP,a kinesin-12 protein,regulates embryo/seed size and seed germination via involving in cell cycle progression at the G2/M transition[J]. Scientific Reports,2016,6(1):35641. doi:10.1038/srep35641.
[11] Song X J,Huang W,Shi M,Zhu M Z,Lin H X. A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase[J]. Nature Genetics,2007,39(5):623-630. doi:10.1038/ng2014.
[12] Wang X B,Li Y H,Zhang H W,Sun G L,Zhang W M,Qiu L J. Evolution and association analysis of GmCYP78A10 gene with seed size/weight and pod number in soybean[J]. Molecular Biology Reports,2015,42(2):489-496. doi:10.1007/s11033-014-3792-3.
[13] Xia T,Li N,Dumenil J,Li J,Kamenski A,Bevan M W,Gao F,Li Y H. The ubiquitin receptor DA1 interacts with the E3 ubiquitin ligase DA2 to regulate seed and organ size in Arabidopsis[J].The Plant Cell,2013,25(9):3347-3359. doi:10.1105/tpc.113.115063.
[14] Orsi C H,Tanksley S D. Natural variation in an ABC transporter gene associated with seed size evolution in tomato species [J].PLoS Genetics,2009,5(1):e1000347.doi:10.1371/journal.pgen.1000347.
[15] Viola I L,Gonzalez D H. Structure and evolution of plant homeobox genes[M].Elsevier:Plant Transcription Factors,2016:101-112.doi:10.1016/b978-0-12-800854-6.00006-3.
[16] Zhu T Q,Moschou P N,Alvarez J M,Sohlberg J J,von Arnold S. WUSCHEL-RELATED HOMEOBOX 2 is important for protoderm and suspensor development in the gymnosperm Norway spruce[J]. BMC Plant Biology,2016,16(1):6-14. doi:10.1186/s12870-016-0706-7.
[17] Kagaya Y,Toyoshima R,Okuda R,Usui H,Yamamoto A,Hattori T. LEAFY COTYLEDON1 controls seed storage protein genes through its regulation of FUSCA3 and ABSCISIC ACID INSENSITIVE3[J]. Plant and Cell Physiology,2005,46(3):399-406. doi:10.1093/pcp/pci048.
[18] Roscoe T T,Guilleminot J,Bessoule J J,Berger F,Devic M. Complementation of seed maturation phenotypes by ectopic expression of ABSCISIC ACID INSENSITIVE3,FUSCA3 and LEAFY COTYLEDON2 in Arabidopsis[J]. The Plant and Cell Physiology,2015,56(6):1215-1228. doi:10.1093/pcp/pcv049.
[19] Finkelstein R R,Gampala S S,Rock C D. Abscisic acid signaling in seeds and seedlings[J]. The Plant Cell,2002,14(S):15-45. doi:10.1105/tpc.010441.
[20] Gacek K,Bartkowiak-Broda I,Batley J. Genetic and molecular regulation of Seed Storage Proteins (SSPs) to improve protein nutritional value of oilseed rape (Brassica napus L.) seeds[J]. Frontiers in Plant Science,2018,9:890. doi:10.3389/fpls.2018.00890.
[21] Jiang L Y,Ma X,Zhao S S,Tang Y Y,Liu F X,Gu P,Fu Y C,Zhu Z F,Cai H W,Sun C Q,Tan L B. The APETALA2-Like transcription factor SUPERNUMERARY BRACT controls rice seed shattering and seed size[J].The Plant Cell,2019,31(1):17-36. doi:10.1105/tpc.18.00304.
[22] 张雪晶,江文波,庞永珍. 植物种子大小调控机制的研究进展[J].植物生理学报,2016,52(7):998-1010.doi:10.13592/j.cnki.ppj.2016.0159.
Zhang X J,Jiang W B,Pang Y Z. Advances in the regulation mechanism of plant seed size[J]. Plant Physiology Journal,2016,52(7):998-1010.
[23] Uchiumi T,Okamoto T. Rice fruit development is associated with an increased IAA content in pollinated ovaries[J].Planta,2010,232(3):579-592. doi:10.1007/s00425-010-1197-7.
[24] Schruff M C,Spielman M,Tiwari S,Adams S,Fenby N,Scott R J. The Auxin response factor 2 gene of Arabidopsis links auxin signalling,cell division,and the size of seeds and other organs [J]. Development,2006,133(2):251-261.doi:10.1242/dev.02194.
[25] Jiang W B,Huang H Y,Hu Y W,Zhu S W,Wang Z Y,Lin W H. Brassinosteroid regulates seed size and shape in Arabidopsis[J]. The Plant Physiology,2013,162(4):1965-1977. doi:10.1104/pp.113.217703.
[26] Vriet C,Russinova E,Reuzeau C. Boosting crop yields with plant steroids[J]. The Plant Cell,2012,24(3):842-857. doi:10.1105/tpc.111.094912.
[27] Audran C,Liotenberg S,Gonneau M,North H,Frey A,Tap-Waksman T K,Vartanian N,Marion-Poll A.Localisation and expression of zeaxanthin epoxidase mRNA in Arabidopsis in response to drought stress and during seed development[J]. Functional Plant Biology,2001,28(12):1161-1173. doi:10.1071/PP00134.
[28] 宋松泉,刘军,黄荟,伍贤进,徐恒恒,张琪,李秀梅,梁娟. 赤霉素代谢与信号转导及其调控种子萌发与休眠的分子机制[J].中国科学(生命科学),2020,50(6):599-615. doi:10.1360/SSV-2019-0289.
Song S Q,Liu J,Huang H,Wu X J,Xu H H,Zhang Q,Li X M,Liang J. Gibberellin metabolism and signaling and its molecular mechanism in regulating seed germination and dormancy[J]. Science in China(Series C),2020,50(6):599-615.
[29] 徐平珍,刘涛,杨莹,胡运乾.脱落酸在植物花发育过程中的作用[J].云南植物研究,2007,29(2):215-222. doi:10.3969/j.issn.2095-0845.2007.02.016.
Xu P Z,Liu T,Yang Y,Hu Y Q. The role of abscisic acid in plant flowering[J]. Acta Botanica Yunnanica,2007,29(2):215-222.
[30] Hong Z,Ueguchi-Tanaka M,Fujioka S,Takatsuto S,Yoshida S,Hasegawa Y,Ashikari M,Kitano H,Matsuoka M. The Rice brassinosteroid-deficient dwarf2 mutant,defective in the rice homolog of Arabidopsis DIMINUTO/DWARF1,is rescued by the endogenously accumulated alternative bioactive brassinosteroid,dolichosterone[J]. The Plant Cell,2005,17(8):2243-2254.doi:10.1105/tpc.105.030973.
[31] 龚淑敏,丁艳菲,朱诚. miRNA 在植物种子发育过程中的作用[J]. 遗传,2015,37(6):554-560. doi:10.16288/j.yczz.15-020.
Gong S M,Ding Y F,Zhu C. Role of miRNA in plant seed development[J]. Hereditas,2015,37(6):554-560.
[32] Ruuska S A,Schwender J,Ohlrogge J B. The capacity of green oilseeds to utilize photosynthesis to drive biosynthetic processes[J]. Plant Physiology,2004,136(1):2700-2709. doi:10.1104/pp.104.047977.
[33] Rolletschek H,Borisjuk L,Koschorreck M,Wobus U,Weber H. Legume embryos develop in a hypoxic environment [J].Journal of Experimental Botany,2002,53(371):1099-1107.doi:10.1093/jexbot/53.371.1099.
[34] 董庆坤,刘慧丽.植物激素调控籽粒大小的研究进展[J].华南师范大学学报(自然科学版),2015,47(6):72-78.doi:10.6054/j.jscnun.2015.08.003.
Dong Q K,Liu H L. Progress on the plant hormone regulation related to seed development[J]. Journal of South China Normal University (Natural Science Edition),2015,47(6):78.
[35] 吴玉,沈永宝,史锋厚. 调控植物种子发育的转录因子研究进展[J]. 生物技术通报,2019,35(11):150-159. doi:10.13560/j.cnki.biotech.bull.1985.2019-0043.
Wu Y,Shen Y B,Shi F H. Research progress on transcription factors regulating plant seed development[J]. Biotechnology Bulletin,2019,35(11):150-159.
[36] 张雪,尹悦佳,范贝,李慧杰,费小钰,崔喜艳.植物Dof转录因子的结构特点及功能研究进展[J].作物杂志,2016(2):14-20. doi:10.16035/j.issn.1001-7283.2016.02.003.
Zhang X,Yin Y J,Fan B,Li H J,Fei X Y,Cui X Y. Advances on the structural characteristics and function of Dof transcription factors in plant[J]. Crops,2016(2):14-20.
[37] Garcia D,Fitz Gerald J N,Berger F. Maternal control of integument cell elongation and zygotic control of endosperm growth are coordinated to determine seed size in Arabidopsis[J]. The Plant Cell,2005,17(1):52-60. doi:10.1105/tpc.104.027136.
[38] Wang C M,Wang H W,Zhang J S,Chen S Y. A seed-specific AP2-domain transcription factor from soybean plays a certain role in regulation of seed germination[J]. Science in China Series C,2008,51(4):336-345. doi:10.1007/s11427-008-0044-6.