尖缘螺属Oxyloma Westerlund,1885隶属于腹足纲(Gastropoda),柄眼目(Pulmonata),琥珀螺科(Succineidae)。广泛分布于非洲、欧洲、北美洲及亚洲等地。在我国尖缘螺属仅报道有印度尖缘螺(Oxyloma indica(Pfeiffer,1849))、狭长尖缘螺(Oxyloma pfeifferi(Rossmaessler,1835))[1-2]和五家渠尖缘螺(Oxyloma wujiaquensis Li,Guo & Zhang,2017)[3]。五家渠尖缘螺已知仅分布于新疆。
线粒体基因组具有自己特殊的遗传物质,呈母系遗传,结构简单。作为高频率应用于生物系统进化研究的分子标记,线粒体基因组具有编码区高度保守、拷贝数高、重组率低等特点[4-5]。目前已广泛应用于动物系统发育、物种分类、群体遗传进化、种质鉴定等领域[6]。近年来腹足纲类群线粒体基因组测序数据的增加,促进了其系统演化问题的研究[7-9]。琥珀螺科的线粒体基因组目前报道仅有一种,White等[10]对肺螺亚纲10个种类的线粒体基因组进行测序和研究,首次获得了琥珀螺科的模式种腐败琥珀螺(Succinea putris Linneaus,1758)的线粒体全基因序列。利用高通量测序技术对动植物基因组进行测序现在成为新的基因组研究趋势[11-13],本研究使用高通量测序Illumina Hiseq及Sanger技术,对五家渠尖缘螺线粒体全基因组进行研究。研究结果可为我国琥珀螺科的系统分类学研究提供数据基础。
1 材料和方法
1.1 试验材料
五家渠尖缘螺样本(XJU S15001)于2015年6月14日采集于新疆五家渠市青格达湖湖坝边(N44°07.736′E08°33.678′)。-20 ℃存放于无水乙醇中。
1.2 DNA提取及检测
剪取软体腹足肌肉组织约30 mg,用天根公司提供的DNA提取试剂盒提取基因组DNA。DNA经Nanodrop 2000(Thermo Fisher Scientific,USA)检测浓度及纯度。
1.3 高通量测序
检测合格的DNA样品,通过转座酶打断DNA,两端加上接头构建测序文库。对文库应用高保真的聚合酶进行扩增,对获取的产物进行纯化,同时利用两步法筛选,最终筛选出片段峰值在300 bp的可测序文库。通过Invitrogen Qubit Spectrophotometer(Thermo Fisher Scientific,USA)确定文库的质量,用Agilent 2100 Bioanalyzer (Agilent Technologies,USA)确定文库的大小是否适合于测序平台。对质检合格的文库片段,在Illumina Hiseq 2500测序平台用2×150 的双端测序方法进行测序。测序结果中出现一些缺口,因无法正确测序这些片段,因此,利用Sanger测序技术,设计9对特异引物对其进行补gap测序。高通量测序与Sanger测序由上海天昊生物有限公司测序完成,引物信息见表1。
表1 五家渠尖缘螺基因组测序引物
Tab.1 Primers for sequencing of the mitochondrial genome of Oxyloma wujiaquensis

序号Number引物名称Primer name引物序列(5′-3′)Primer sequences长度/bpLength119B0102A-1FGCTGTCGTTGTCTTTGTATTCGTAGTCT28 19B0102A-1RCGGCTATTTAATACTCCATTGAGCAGAAC29219B0102A-2FGCTGTCGTTGTCTTTGTATTCGTAGTCT2819B0102A-2RCAGGTAATCATGAACTAAAAGGATATTGAGC31319B0102A-3FGTTACTGCCGGTATTTACTTAGTGATTCG2919B0102A-3RGCCCTAAACGAATTTGACAATATCCTAA28419B0102A-4FGGTTTAATATTGAATACCAAGGTGGCTTATT3119B0102A-4RTCCAGTTAAAATCTGAAAACCTAATATTACACC33519B0102A-5FAATGCAAATCCTATAGTTACCCCTACACAC3019B0102A-5RCCCATAATAGCCACAAGAGTAAAAATTCC29619B0102A-6FAATTTGATTAGCACTTCCTAGTTTACGTCTTT3219B0102A-6RTGTAGCTATCATTCAGGGTTATATTTTCACA31719B0102A-7FGCACTAATGTTTGCTATCAGTCGTACAG2819B0102A-7RTACAAAAGCATGTGCAGTAACGATAAC27819B0102A-8FACTCGCCTAATTTCCAAAAAATGCT2519B0102A-8RTTGTAATCGCTCAGGTTGATAGCC24919B0102A-7F2GGGATTTACCCATTTCATTTCTGAGTA2719B0102A-8SEQFTTGGATTATGAGTCCAATAGCTTTGTT27
PCR反应程序为:95 ℃预变性2 min;94 ℃变性20 s,62~64 ℃退火40 s,72 ℃延伸1~3 min,共11个循环;56~58 ℃退火30 s,72 ℃延伸2 min,共24个循环;72 ℃终延伸2 min,4 ℃冷却。PCR扩增体系包括1×GC/HotStarTaq 缓冲液1.5 μL、MgCl2(1.5 mmol/L) 1 μL、dNTP(2.5 mmol/L) 1 μL、上下游引物(10 μmol/L)各1.5 μL、HotStarTaq聚合酶(1 U/μL) 1 μL、模板DNA 1 μL、最终加ddH2O补足至20 μL。扩增产物经1%琼脂糖凝胶电泳检测后,在ABI3730XL测序仪上进行测序。
1.4 序列拼接、基因组装
通过测序平台获得的原始序列Raw reads,利用Trim galore在线过滤软件删除低质量、含有带接头的序列,最终得到高质量可组装的序列Clean reads。使用BWA软件比对至肺螺亚纲类群,按无脊椎动物组装策略使用mito Marker组装软件进行序列组装。
1.5 基因组注释
通过与近缘种序列进行比较后,选定琥珀螺科模式种腐败琥珀螺的线粒体全基因组序列为参考基因组,应用Genious R11.0 (Biomatters Ltd,Auckland,New Zealand)软件及在线注释软件MITOS Web Server [14],对线粒体全基因组序列进行注释,预测tRNA的二级结构、非编码区域的位置等。注释结果经OGORAW在线作图软件进行作图,最后将注释成功的线粒体基因组数据提交到GenBank。
1.6 序列分析及系统进化树
利用Genious R11.0软件对O. wujiaquensis线粒体全基因组的核苷酸序列进行分析,计算AT和 GC偏移值[15]。通过MEGA 7.0软件对五家渠尖缘螺密码子使用情况进行分析,并与腐败琥珀螺的线粒体基因组序列相比较。以新进腹足目Caenogastropoda的Lophinotoma cerithiformis作为外群[10],选择腹足纲的肺螺亚纲及后鳃亚纲共6个种的线粒体基因组序列(表2),用最大似然法(Maximum likelihood,ML)基于13个蛋白质编码基因构建系统进化树,从基因组水平上初步探讨五家渠尖缘螺与其他类群之间的系统关系。
表2 腹足纲部分种类线粒体基因组序列GenBank登录号
Tab.2 GenBank accession number mitochondrial genome sequences of some species in Gastropoda

分类 Classification 种名Species name GenBank登录号GenBank accession 后鳃亚纲侧鳃科PleurobranchidaeBerthellina ilisimaDQ991929OpisthobranchiaChromodorididaeChromodoris magnificaDQ991931捻螺科ActeonidaePupa strigosaAB028237肺螺亚纲耳螺科EllobiidaeMyosotella myosotisAY345053Pulmonata石磺科 OnchidiidaeOnchidella celticaAY345048琥珀螺科SuccineidaeSuccinea putrisJN627206新进腹足目Caenogastropoda塔螺科 TurridaeLophiotoma cerithiformisDQ284754
2 结果与分析
2.1 五家渠尖缘螺线粒体基因组序列分析
通过高通量测序获得原始序列Raw reads为31 259 236 bp,过滤后得到序列Clean reads为27 485 002 bp。经过补gap测序、基因拼接和组装最终得到了14 086 bp的五家渠尖缘螺线粒体全基因组序列(GenBank 登录号:MT670402)。基因注释结果显示,其基因组共有37个基因组成,包括了13个蛋白质编码基因,22个tRNA基因,2个rRNA基因(图1)。 其线粒体全基因组序列与腐败琥珀螺的相似性为76.1%。
2.2 五家渠线粒体基因组基因组成
五家渠尖缘螺线粒体基因组各基因的序列长度、方向及密码子等情况见表3。在37个基因中重链(H)编码15个基因,轻链(L)编码22个基因,基因的排列顺序与腐败琥珀螺基因排列顺序一致。五家渠尖缘螺线粒体基因组序列中有14处存在基因间隔,长度在1~237 bp;12处存在基因重叠,长度在1~59 bp。腐败琥珀螺线粒体全基因序列15处存在基因间隔,长度在1~48 bp;12处存在基因重叠,长度在5~38 bp[10]。二者有所不同。
2.3 核苷酸分析
表4显示了2种琥珀螺线粒体基因组序列的核苷酸特征。五家渠尖缘螺线粒体基因组序列AT含量为75.64%、GC含量为24.36%;腐败琥珀螺的AT含量为77.00%,GC含量为23.00%,具有明显的A、T偏向性。五家渠尖缘螺的线粒体全基因组序列的AT skew为负值,腐败琥珀螺的为正值,GC skew均为正值,说明总体来看二者T、G碱基的使用率较高。但五家渠尖缘螺tRNA基因和rRNA基因的AT skew分别是0.03和0.01,为正数,五家渠尖缘螺这两类基因A碱基的使用频率高于T碱基。
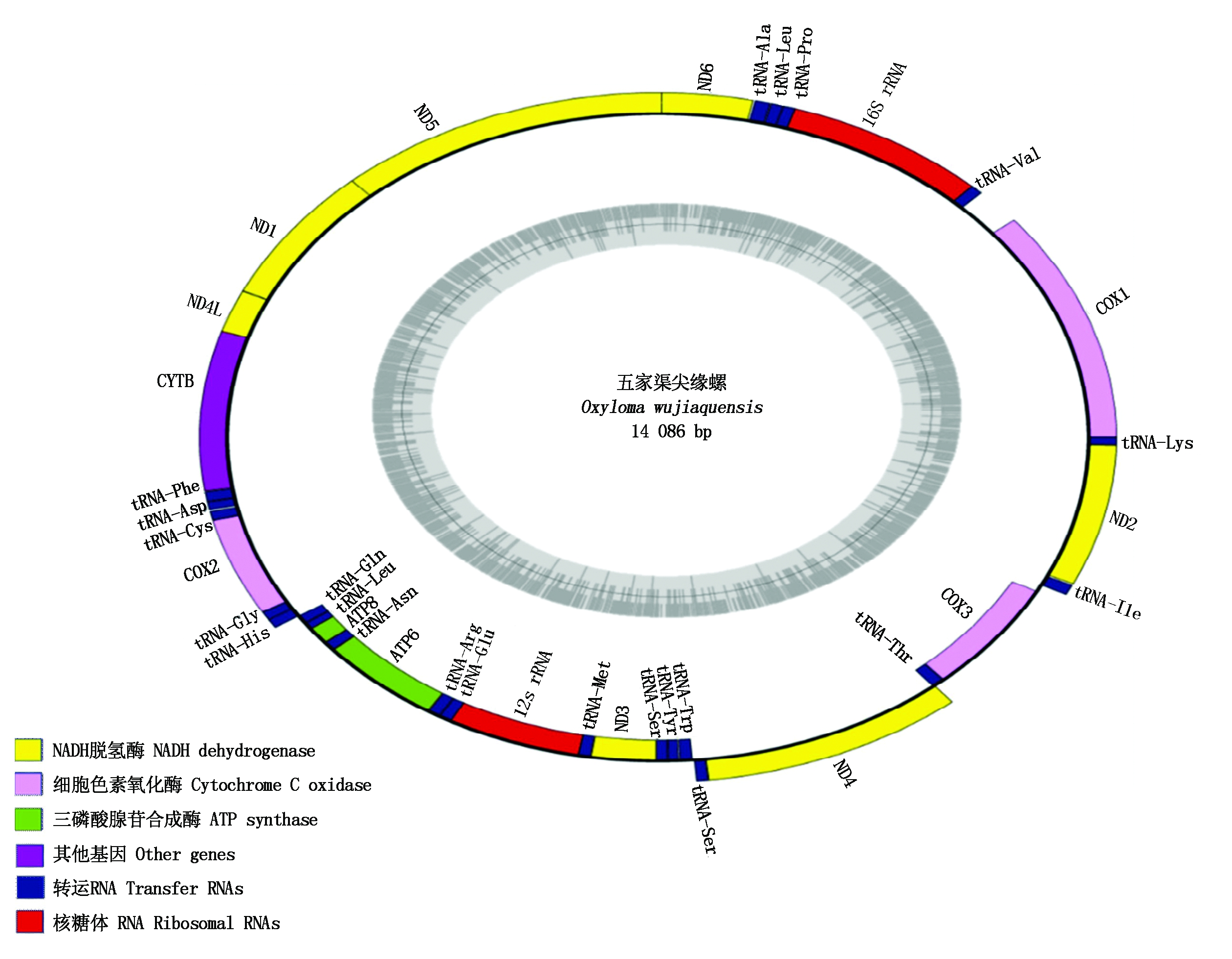
图1 五家渠尖缘螺线粒体基因组结构
Fig.1 Circular map of mitochondrial genome of Oxyloma wujiaquensis
表3 五家渠尖缘螺线粒体基因组组成
Tab.3 Organization of the mitochondrial genome of Oxyloma wujiaquensis
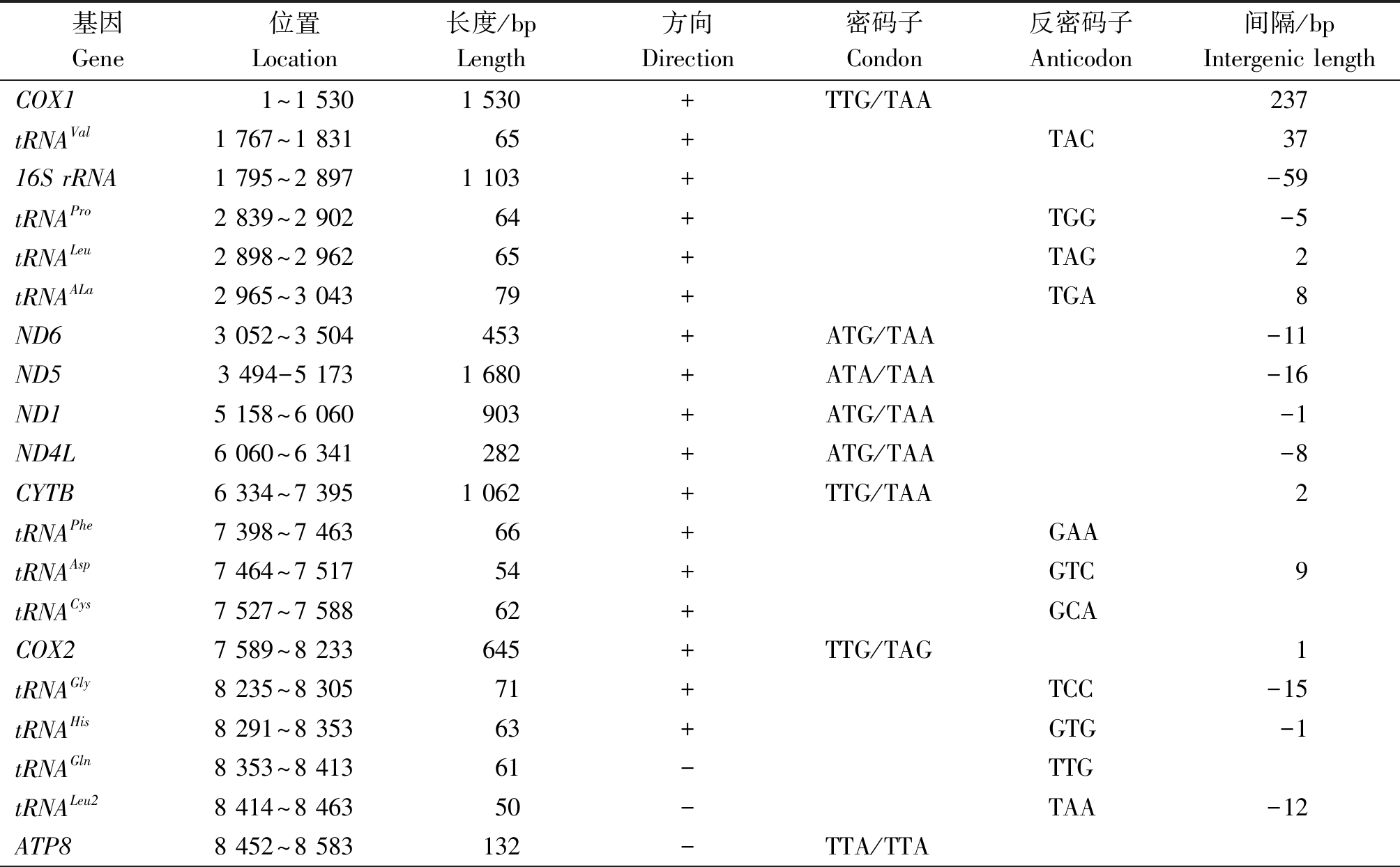
基因Gene位置Location长度/bpLength方向Direction密码子Condon反密码子Anticodon间隔/bpIntergenic lengthCOX1 1~1 5301 530+TTG/TAA237tRNAVal1 767~1 83165+TAC3716S rRNA1 795~2 8971 103+-59tRNAPro2 839~2 90264+TGG-5tRNALeu2 898~2 96265+TAG2tRNAALa2 965~3 04379+TGA8ND63 052~3 504453+ATG/TAA-11ND53 494-5 1731 680+ATA/TAA-16ND15 158~6 060903+ATG/TAA-1ND4L6 060~6 341282+ATG/TAA-8CYTB6 334~7 3951 062+TTG/TAA2tRNAPhe7 398~7 46366+GAAtRNAAsp7 464~7 51754+GTC9tRNACys7 527~7 58862+GCACOX27 589~8 233645+TTG/TAG1tRNAGly8 235~8 30571+TCC-15tRNAHis8 291~8 35363+GTG-1tRNAGln8 353~8 41361-TTGtRNALeu28 414~8 46350-TAA-12ATP88 452~8 583132-TTA/TTA
表3(续)
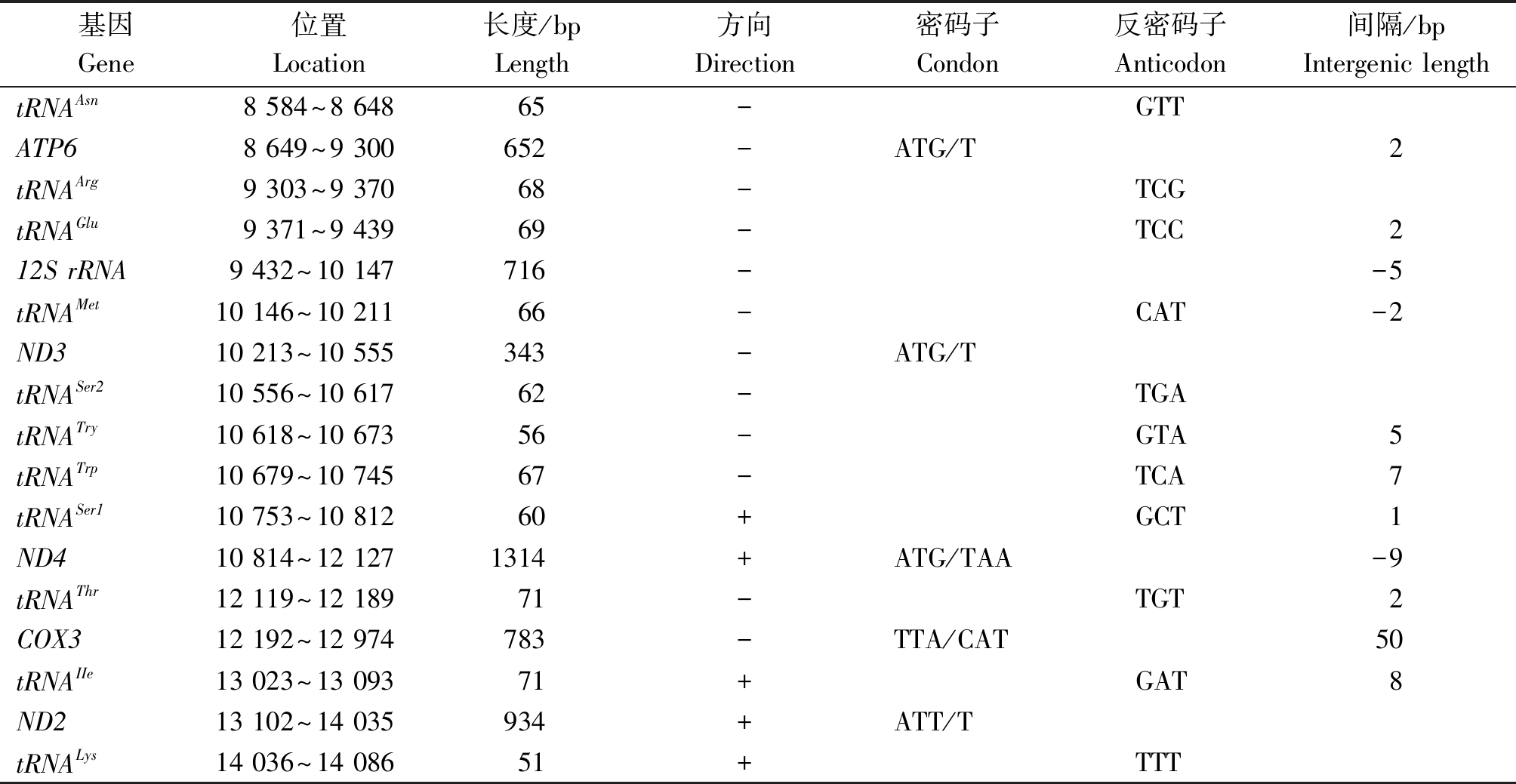
基因Gene位置Location长度/bpLength方向Direction密码子Condon反密码子Anticodon间隔/bpIntergenic lengthtRNAAsn8 584~8 64865-GTTATP68 649~9 300652-ATG/T2tRNAArg9 303~9 37068-TCGtRNAGlu9 371~9 43969-TCC212S rRNA9 432~10 147716--5tRNAMet10 146~10 21166-CAT-2ND310 213~10 555343-ATG/TtRNASer210 556~10 61762-TGAtRNATry10 618~10 67356-GTA5tRNATrp10 679~10 74567-TCA7tRNASer110 753~10 81260+GCT1ND410 814~12 1271314+ATG/TAA-9tRNAThr12 119~12 18971-TGT2COX312 192~12 974783-TTA/CAT50tRNAIIe13 023~13 09371+GAT8ND213 102~14 035934+ATT/TtRNALys14 036~14 08651+TTT
注:负数为基因重叠区;正数为基因间隔区。
Note:Negative numbers are gene overlap regions;Positive numbers are gene gap regions.
表4 2种琥珀螺线粒体基因组核苷酸组成分析
Tab.4 Nucleotide composition of mitochondrial genome of two species of Succineidae
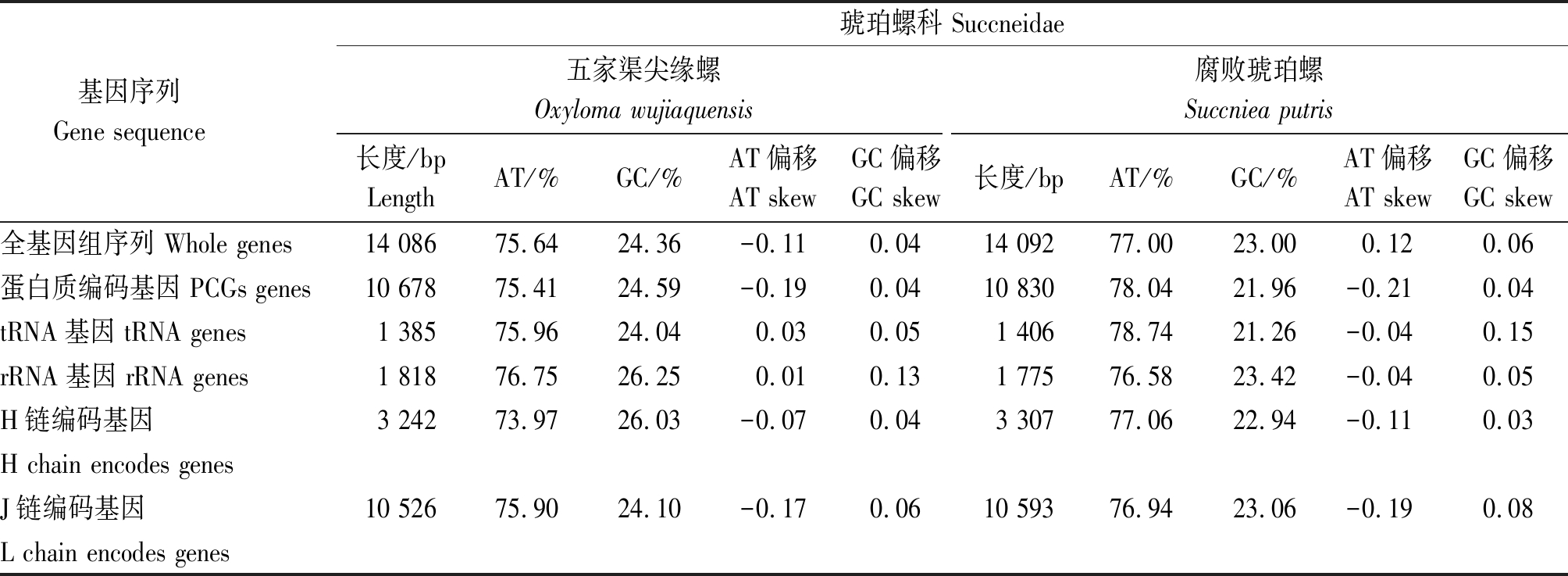
基因序列 Gene sequence 琥珀螺科 Succneidae五家渠尖缘螺Oxyloma wujiaquensis腐败琥珀螺Succniea putris长度/bpLengthAT/%GC/%AT偏移AT skewGC偏移GC skew长度/bpAT/%GC/%AT偏移AT skewGC偏移GC skew全基因组序列 Whole genes14 08675.6424.36-0.110.0414 09277.0023.000.120.06蛋白质编码基因 PCGs genes10 67875.4124.59-0.190.0410 83078.0421.96-0.210.04tRNA基因 tRNA genes1 38575.9624.040.030.051 40678.7421.26-0.040.15rRNA基因 rRNA genes1 81876.7526.250.010.131 77576.5823.42-0.040.05H链编码基因3 24273.9726.03-0.070.043 30777.0622.94-0.110.03H chain encodes genesJ链编码基因 10 52675.9024.10-0.170.0610 59376.9423.06-0.190.08L chain encodes genes
注:AT偏移=(A-T)/(A+T);GC偏移=(G-C)/(G+C)。
Note:AT skew=((A-T)/(A+T);GC skew=(G-C)/(G+C).
2.4 蛋白质编码基因
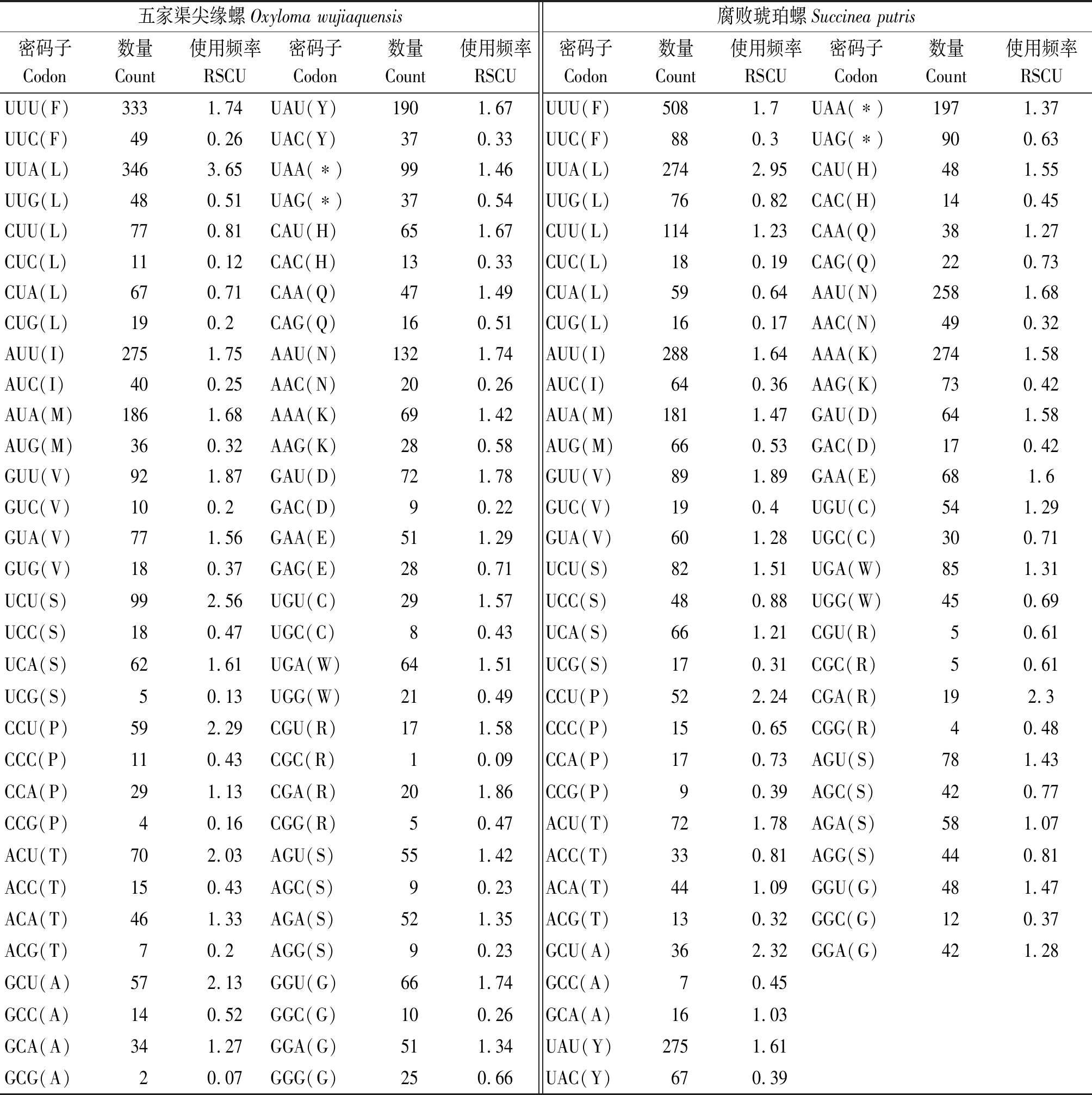
五家渠尖缘螺线粒体基因组中13个蛋白质编码基因(Protein coding genes,PCGs)的总长为10 678 bp,AT含量75.41%,各基因的长度在132~1 680 bp。从表3得知,13个蛋白质编码基因使用ATG(6个)、TTG(3个)、TTA(2个)和ATT(1个) 为起始密码子;使用TAA(8个)、T(3个)、CAT(1个)、TAG(1个)为终止密码子。使用的密码子类型是肺螺亚纲类群中常见的起始和终止密码子。分析琥珀螺科2个种的线粒体基因组序列同义密码子使用频率发现(表5),五家渠尖缘螺线粒体全基因组序列共编码3 435个密码子,腐败琥珀螺共编码4 285个密码子(不含终止密码子),在2个线粒体基因序列中密码子UUA(Leu)、UUU(Phe)、AUU(Ⅱe)等均有较高使用次数。根据图2可知,甲硫氨酸(Met)在2个线粒体基因组序列中使用的密码子有所不同,在五家渠尖缘螺基因组序列中Met使用ATA、ATG、TTG、TCT等4种密码子,其中ATA的数量最多;在腐败琥珀螺基因组序列中Met使用ATA、ATG、TTG等3种密码子,其中ATA的数量最多。
2.5 tRNA、rRNA 基因及控制区
五家渠尖缘螺线粒体基因组中tRNA基因数目为22个,基因总长为1 818 bp,AT为75.96%,GC为24.04%。通过MITOS预测tRNA基因的二级结构,发现tRNASer1、tRNASer2缺少DHU臂。其他基因均能形成经典的三叶草结构,这与腐败琥珀螺和其他肺螺亚纲类群tRNA基因二级结构基本一致。多数后生动物线粒体基因组中tRNASer1基因的二级结构缺失DHU臂,成为典型的基因结构特征之一[15-18]。
五家渠尖缘螺线粒体基因组中的rRNA基因的位置与腐败琥珀螺的一样,16S rRNA位于tRNAVal、tRNAPro之间,12S rRNA位于tRNAGlu、tRNAMet之间。16S rRNA的长度为1 103 bp,12S rRNA为713 bp。rRNA基因序列的AT含量为76.75%,与腐败琥珀螺76.58%的含量相近,具有明显的A、T偏向性。
表5 2种琥珀螺线粒体基因组密码子使用情况 Tab.5 Codon usage in the mitochondrial genome of two species of Succineidae
五家渠尖缘螺Oxyloma wujiaquensis腐败琥珀螺Succinea putris密码子Codon数量Count使用频率RSCU密码子Codon数量Count使用频率RSCU密码子Codon数量Count使用频率RSCU密码子Codon数量Count使用频率RSCUUUU(F)3331.74UAU(Y)1901.67UUU(F)5081.7UAA(∗)1971.37UUC(F)490.26UAC(Y)370.33UUC(F)880.3UAG(∗)900.63UUA(L)3463.65UAA(∗)991.46UUA(L)2742.95CAU(H)481.55UUG(L)480.51UAG(∗)370.54UUG(L)760.82CAC(H)140.45CUU(L)770.81CAU(H)651.67CUU(L)1141.23CAA(Q)381.27CUC(L)110.12CAC(H)130.33CUC(L)180.19CAG(Q)220.73CUA(L)670.71CAA(Q)471.49CUA(L)590.64AAU(N)2581.68CUG(L)190.2CAG(Q)160.51CUG(L)160.17AAC(N)490.32AUU(I)2751.75AAU(N)1321.74AUU(I)2881.64AAA(K)2741.58AUC(I)400.25AAC(N)200.26AUC(I)640.36AAG(K)730.42AUA(M)1861.68AAA(K)691.42AUA(M)1811.47GAU(D)641.58AUG(M)360.32AAG(K)280.58AUG(M)660.53GAC(D)170.42GUU(V)921.87GAU(D)721.78GUU(V)891.89GAA(E)681.6GUC(V)100.2GAC(D)90.22GUC(V)190.4UGU(C)541.29GUA(V)771.56GAA(E)511.29GUA(V)601.28UGC(C)300.71GUG(V)180.37GAG(E)280.71UCU(S)821.51UGA(W)851.31UCU(S)992.56UGU(C)291.57UCC(S)480.88UGG(W)450.69UCC(S)180.47UGC(C)80.43UCA(S)661.21CGU(R)50.61UCA(S)621.61UGA(W)641.51UCG(S)170.31CGC(R)50.61UCG(S)50.13UGG(W)210.49CCU(P)522.24CGA(R)192.3CCU(P)592.29CGU(R)171.58CCC(P)150.65CGG(R)40.48CCC(P)110.43CGC(R)10.09CCA(P)170.73AGU(S)781.43CCA(P)291.13CGA(R)201.86CCG(P)90.39AGC(S)420.77CCG(P)40.16CGG(R)50.47ACU(T)721.78AGA(S)581.07ACU(T)702.03AGU(S)551.42ACC(T)330.81AGG(S)440.81ACC(T)150.43AGC(S)90.23ACA(T)441.09GGU(G)481.47ACA(T)461.33AGA(S)521.35ACG(T)130.32GGC(G)120.37ACG(T)70.2AGG(S)90.23GCU(A)362.32GGA(G)421.28GCU(A)572.13GGU(G)661.74GCC(A)70.45GCC(A)140.52GGC(G)100.26GCA(A)161.03GCA(A)341.27GGA(G)511.34UAU(Y)2751.61GCG(A)20.07GGG(G)250.66UAC(Y)670.39
注: *.终止密码子。
Note:*.Stop codon.
后生动物线粒体全基因组序列中存在长度及位置不同的非编码区域,由于该区的A+T含量较高,又被称为控制区或A+T富集区,该区域负责调控线粒体基因组的复制与转录过程[19-20]。通过MITOS注释软件发现,在五家渠尖缘螺线粒体基因组中存在控制区,长度在42 bp左右,位于COX1和tRNAVal之间。腐败琥珀螺线粒体基因组序列中存在的控制区长度在50 bp左右,位于COX3与tRNAⅡe之间。2个线粒体基因组序列控制区的长度与其他肺螺亚纲类群出现差异[8,10,21-23]。在线粒体基因组序列中,控制区片段的A+T含量较高,易发生碱基突变,属于高度可变区。该区域会影响不同种的动物线粒体基因组长度的大小,甚至会导致线粒体基因组序列的测序失败[24-26]。
2.6 系统发育分析
系统进化树结果显示(图3),五家渠尖缘螺与同科的腐败琥珀螺紧密地聚在一起,形成一支。后鳃亚纲类群聚为一支,肺螺亚纲类群没有聚在一起。结果表明,肺螺亚纲类群并非单系类群,琥珀螺科和后鳃亚纲分别为单系类群。该结果与White等[10]的系统发育分析结果一致。
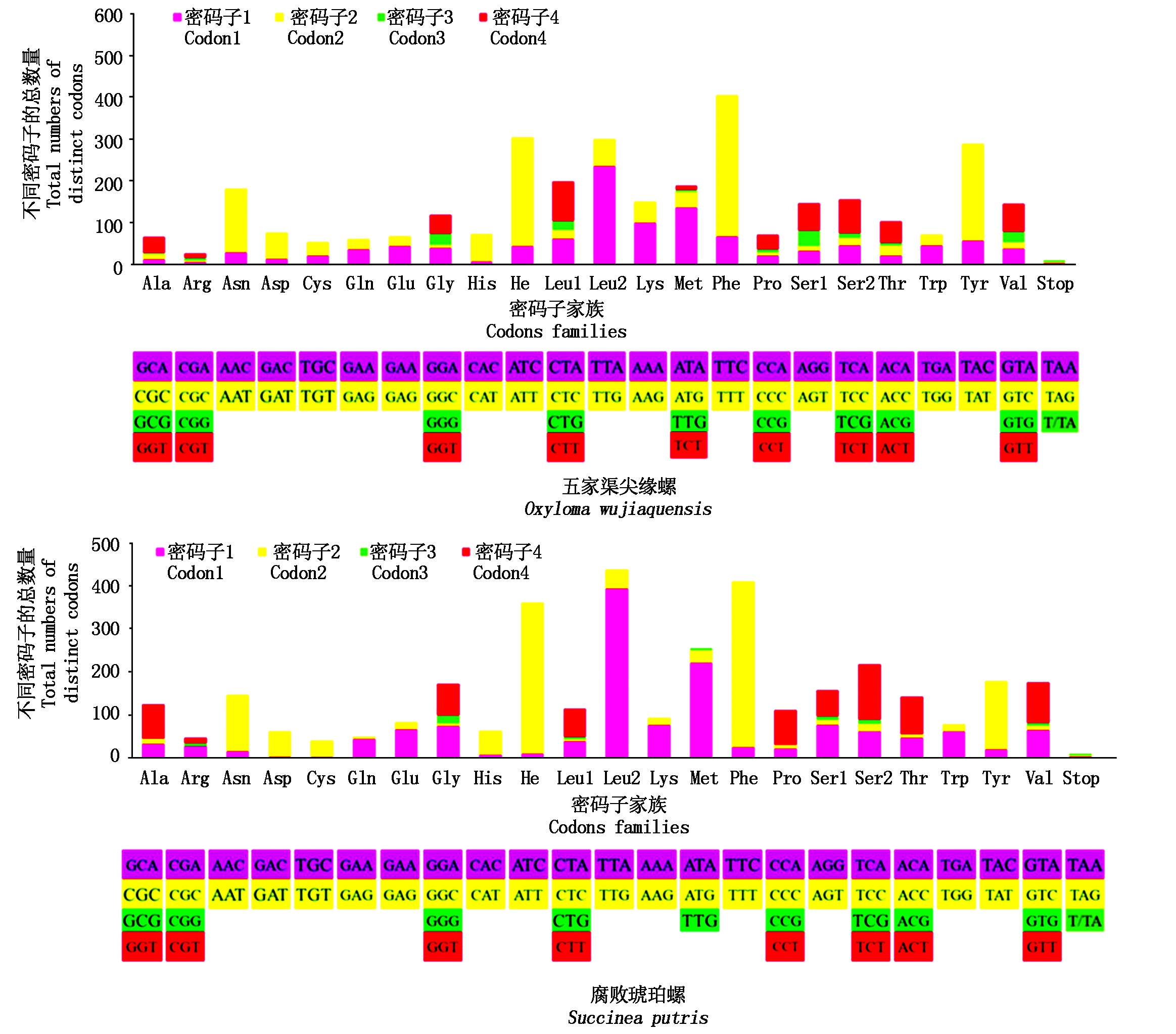
图2 2种琥珀螺线粒体基因组密码子分布
Fig.2 Codon distribution in the mitochondrial genomes of two Succneidae species
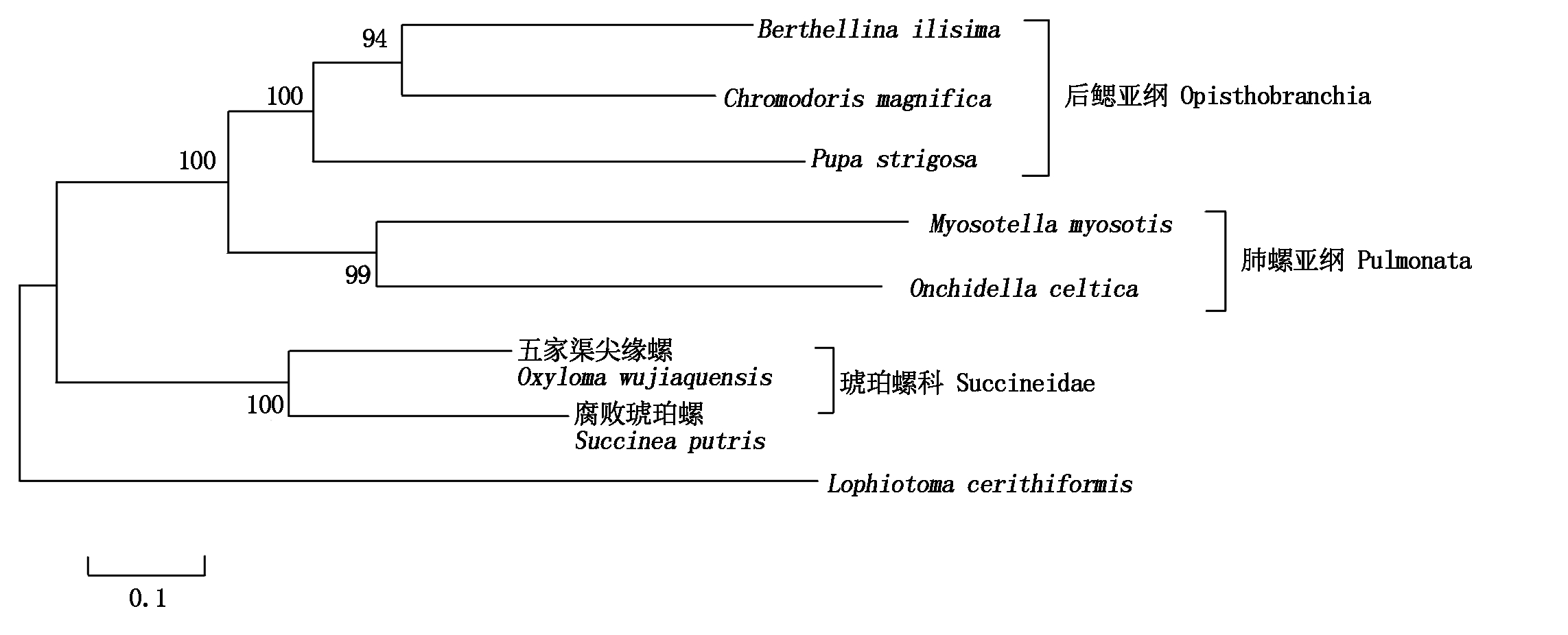
图3 基于线粒体基因组蛋白质编码基因序列构建肺螺亚纲部分种类ML树
Fig.3 Maximum likelihood tree based on protein-coding gene sequences of
mitochondrial genome of some species of Pulmonata
3 讨论
本研究通过高通量测序获得了五家渠尖缘螺线粒体基因组全序列,全长为14 086 bp。通过基因注释后得到了37个基因,基因排列顺序与同科的腐败琥珀螺线粒体基因组序列完全一致,但是在基因序列的长度、碱基含量、密码子使用情况及非编码区的位置和长度上存在差异。五家渠尖缘螺线粒体基因组序列中有14处存在基因间隔,长度在1~237 bp,腐败琥珀螺线粒体全基因组序列15处存在基因间隔,长度在1~48 bp,明显短于五家渠尖缘螺。五家渠尖缘螺线粒体基因组全序列及各基因序列的AT含量均少于腐败琥珀螺,全序列AT含量前者为75.64%,后者为77.00%。在编码蛋白质的基因中,五家渠尖缘螺线粒体全基因组序列共编码3 435个密码子,腐败琥珀螺共编码4 285个密码子。密码子的使用情况基本相同,但编码甲硫氨酸(MET)的密码子有所不同,前者使用ATA、ATG、TTG、TCT等4种密码子,后者则使用ATA、ATG、TTG等3种密码子。终止密码子也有所不同,五家渠尖缘螺COX3基因使用CAT为终止密码子,腐败琥珀螺则没有。线粒体基因组的非编码区域在五家渠尖缘螺中位于COX1和tRNAVal之间,长度为42 bp,腐败琥珀螺及其他的肺螺亚纲类群的非编码区域位于COX3与tRNAⅡe之间[10],长度为50 bp。在测序过程中发现五家渠尖缘螺的非编码区域的AT含量很高,导致全序列测序很困难。以上表明,五家渠尖缘螺与腐败琥珀螺线粒体基因组之间存在一定的差异,这些差异可能是测序过程的不足,或者是物种本身的基因序列特点引起的。由于在NCBI上琥珀螺科线粒体基因全组序列只有一条,因此无法更加系统地分析其在不同属种间的差异性。有关研究有待更多数据的补充。
基因组学及后基因组学的不断发展,对生物学的更深层研究带来便利。线粒体基因组学研究作为其中的重要领域,广泛应用于物种进化、遗传、基因流等研究中。目前,琥珀螺科中只有腐败琥珀螺的线粒体全基因组序列已经完成测序,本次获得的五家渠尖缘螺线粒体全基因组序列将增加琥珀螺科类群基因组数据,为琥珀螺科的资源保护、遗传多样性和系统发育研究提供基础数据。
[1] Pfeiffer L. Descriptions of ten new species of Bulimus,from the collection of H.Cuming,Esq[J]. Proceedings of the Zoological Society of London,1858,26(1):256-258. doi:10.1111/j.1469-7998.1858.tb06372.x.
[2] Roβmäβler,Kobelt E A. Iconographie der Landund Süsswasser-Mollusken,mit vorzüglicher Berück sichtigung dereuropäischen noch nicht abgebildeten Arten[M]. Dresden,Arnold:Band I,Heft I,1835:92.
[3] 李成有,郭阳阳,张卫红. 中国尖缘螺属一新种记述(柄眼目,琥珀螺科)[J]. 海洋科学,2017,41(11):130-133.
Li C Y,Guo Y Y,Zhang W H. A new species of Oxyloma from Xinjiang (Stylommatophora:Succineidae)[J]. Marine Sciences,2017,41(11):130-132.
[4] Boore J L. Animal mitochondrial genomes[J]. Nucleic Acids Research,1999,27(8):1767-1780. doi:10.1093/nar/27.8.1767.
[5] 杨婧,黄原. 线粒体基因组的高通量测序策略[J]. 生命科学,2016,28(1):112-117. doi:10.13376/j.cbls/2016015.
Yang J,Huang Y. Strategy of high-throughput sequencing technology in the mitochondrial genome sequencing[J]. Chinese Bulletin of Life Sciences,2016,28(1):112-117.
[6] Wang P,Yang H F,Zhou W C,Hwang C C,Zhang W H,Qian Z X. The mitochondrial genome of the land snail Camaena cicatricosa (Müller,1774) (Stylommatophora,Camaenidae):the first complete sequence in the family Camaenidae[J]. Zookeys,2014(451):33-48. doi:10.3897/zookeys.451.8537.
[7] Groenenberg D S J,Harl J,Duijm E,Gittenberger E. The complete mitogenome of Orcula dolium (Draparnaud,1801);ultra-deep sequencing from a single long-range PCR using the Ion-Torrent PGM[J]. Hereditas,2017,154(1):7. doi:10.1186/s41065-017-0028-2.
[8] Grande G,Templado J,Zardoya R. Evolution of gastropod mitochondrial genome arrangements[J]. BMC Evolutionary Biology,2008,8(1):61. doi:10.1186/1471-2148-8-61.
[9] Groenenberg D S,Pirovano W,Gittenberger E,Schilthuizen M. The complete mitogenome of Cylindrus obtusus (Helicidae,Ariantinae) using Illumina next generation sequencing[J]. BMC Genomics,2012,13(1):114. doi:10.1186/1471-2164-13-114.
[10] White T R,Conrad M M,Tseng R,Balayan S,Golding R,de Frias Martins A M,Dayrat B A. Ten new complete mitochondrial genomes of pulmonates (Mollusca:Gastropoda) and their impact on phylogenetic relationships[J]. BMC Evolutionary Biology,2011,11(1):295. doi:10.1186/1471-2148-11-295.
[11] 姜志磊,王大铭,王柏凤,宋新元.双斑萤叶甲基因组调研及线粒体基因组分析[J].环境昆虫学报,2019,41(6):1287-1296.doi:10.3969/j.issn.1674-0858.2019.06.18.
Jiang Z L,Wang D M,Wang B F,Song X Y. Genome survey and mitochondrial genome analysis of Monolepta hieroglyphica[J].Journal of Environmental Entomology,2019,41(6):1287-1296.
[12] 刘菲. 贺氏东栖螽线粒体基因组序列分析(直翅目:螽斯科)[J]. 基因组学与应用生物学,2017,36(8):3194-3199.doi:10.13417/j.gab.036.003194.
Liu F. Mitogenome sequence analysis of Xizicus howardi (Orthoptera:Tettigoniidae)[J]. Genomics and Applied Biology,2017,36(8):3194-3199.
[13] 张冬杰,何鑫淼,王文涛,汪亮,刘娣. 民猪全基因组序列测定与分析[J]. 东北农业大学学报,2018,49(11):9-17.doi:10.19720/j.cnki.issn.1005-9369.2018.11.0002.
Zhang D J,He X M,Wang W T,Wang L,Liu D. Determination and analysis of the whole genome sequence of Min pig[J].Journal of Northeast Agricultural University,2018,49(11):9-17.
[14] Bernt M,Donath A,Jühling F,Externbrink F,Florentz C,Fritzsch G,Pütz J,Middendorf M,Stadler P F. MITOS:Improved de novo metazoan mitochondrial genome annotation[J]. Molecular Phylogenetics and Evolution,2013,69(2):313-319.doi:10.1016/j.ympev.2012.08.023.
[15] Perna N T,Kocher T D. Patterns of nucleotide composition at fourfold degenerate sites of animal mitochondrial genomes[J].Journal of Molecular Evolution,1995,41(3):353-358.doi:10.1007/BF00186547.
[16] 钟健,刘增虎,杨伟克,朱峰,董占鹏.琥珀蚕线粒体全基因组测序及序列分析[J]. 昆虫学报,2017,60(8):936-949. doi:10. 16380/j. kcxb. 2017. 08. 011.
Zhong J,Liu Z H,Yang W K,Zhu F,Dong Z P. Sequencing and analysis of the complete mitochondrial genome of Antheraea assama (Lepidoptera:Saturniidae)[J].Acta Entomologica Sinica,2017,60(8):936-949.
[17] 孙丽冉,齐迎菊,田晓轩. 哈氏蜈蚣线粒体基因组全序列分析[J]. 天津中医药,2018,35(3):225-229.doi:10.11656/j.issn.1672-1519.2018.03.19.
Sun L R,Qi Y J,Tian X X. Analysis of mitochondrial genome of Scolopendra subspinipes dehaani[J].Tianjin Journal of Traditional Chinese Medicine,2018,35(3):225-229.
[18] 张玉波,史娅琴,孙翠英.长尾管蚜蝇Eristalis tenax (Linnaeus,1758) 线粒体基因组测序及分析[J].基因组学与应用生物学,2019,38(12):5371-5378. doi:10.13417/j.gab.038.005371.
Zhang Y B,Shi Y Q,Sun C Y. Sequencing and analysis of the mitochondrial genome of Eristalis tenax (Linnaeus,1758)[J]. Genomics and Applied Biology,2019,38(12):5371-5378.
[19] Taanman J W. The mitochondrial genome:structure,transcription,translation and replication [J].Biochim Biophys Acta,1999,1410(2):103-123.doi:10.1016/S0005-2728(98)00161-3.
[20] 李爱玲,张志芳,潘沈元.昆虫线粒体DNA A+T富集区的研究及其在分子进化中的应用[J].中国蚕业,2004,25(1):70-72.doi:10.3969/j.issn.1007-0982.2004.01.036.
Li A L,Zhang Z F,Pan S Y. Study on A+T enrichment region of insect mitochondrial DNA and its application in molecular evolution [J].China Sericulture,2004,25(1):70-72.
[21] 李石柱,王艺秀,刘琴,吕山,王强,吴缨,张仪,周晓农.湖北钉螺线粒体基因组全序列测定研究[J].中国寄生虫学与寄生虫病杂志,2009,27(4):291-296.
Li S Z,Wang Y X,Liu Q,Lü S,Wang Q,Wu Y,Zhang Y,Zhou X N. Complete mitochondrial genome sequence of Oncomelania hupensis(Gastropoda:Rissooidea)[J].Chin J Parasitol Parasit Dis,2009,27(4):291-296.
[22] Lin J H,Zhou W C,Ding H L,Wang P,Ai H M. The mitochondrial genome of the land snail Cernuella virgata (Da Costa,1778):the first complete sequence in the family Hygromiidae (Pulmonata,Stylommatophora)[J].Zookeys,2016(589):55-69. doi:10.3897/zookeys.589.7637.
[23] 魏峦峦,沈斌,沈和定,陈诚,方磊,吴文健. 一种石磺科贝类线粒体基因组全序列分析[J].生物技术通报,2010(8):188-194,213.doi:10.13560/j.cnki.biotech.bull.1985.2010.08.006.
Wei L L,Shen B,Shen H D,Chen C,Fang L,Wu W J. Analysis of complete mitochondrial genome of platevindex mortoni of Onchidiidae (Mollusca:Gastropoda:Pulmonata)[J]. Biotechnology Bulletin,2010(8):188-194,213.
[24] Harrison R G. Animal mitochondrial DNA as a genetic marker in population and evolutionary biology[J]. Trends Ecol Evol,1989,4(1):6-11. doi:10.1016/0169-5347(89)90006-2.
[25] Moszczynska A,Locke S A,Mclaughlin J D,Marcogliese D J,Crease T J. Development of primers for the mitochondrial cytochrome c oxidase I gene in digenetic trematodes (Platyhelminthes) illustrates the challenge of barcoding parasitic helminths [J].Molecular Ecology Resources,2009,9(S1):75-82. doi:10.1111/j.1755-0998.2009.02634.x.
[26] Hu M,Jex A R,Campbell B E,Gasser R B. Long PCR amplification of the entire mitochondrial genome from individual helminths for direct sequencing [J].Nature Protocols,2007,2(10):2339-2344. doi: 10.1038/nprot.2007.358.