我国是世界上最大的小麦生产国,也是最大的消费国,小麦生产在保障国家粮食安全和提升居民生活水平中具有举足轻重的地位[1-2]。随着科学技术的发展,小麦遗传育种研究取得了丰硕的成果,向着优质、抗病、高产、广适的方向发展[3]。同时,每年有大量的小麦良种进入销售市场,随之而来的种子质量问题成为市场和种子企业关注的重要问题,例如发芽率、净度及种子纯度等指标。其中,种子纯度鉴定是种子生产企业必需具备的检验技术,因此,如何快速、高效、准确的鉴定种子纯度,成为摆在育种家、种子销售企业面前的重要课题。
目前,农作物种子纯度检验方法为国标GB/T 3543.5-1995,主要包括外观鉴定和生理生化鉴定。随着分子时代的到来也出现了DNA水平上的“分子鉴定”。外观鉴定简单、经济、快速,但准确性较差,且不同品种间种子外观形态的差异越来越小,因此,靠区别种子形态上的差异来鉴定种子纯度可靠性较低[4]。DNA鉴定没有器官的特异性,不受环境的影响,有较高的准确性、稳定性和重复性。该技术灵敏度高、快速、多态性强、DNA的用量少、具有较好的重复性[5]。但是DNA鉴定需要育苗提取DNA、用特异引物进行PCR检测及电泳等过程,需要进行数以千计的引物设计筛选[6-7]。且上述方法均只能鉴定当代种子纯度,不能鉴定下一代种子的纯度,而商业化育种是要鉴定下一代种子纯度,确保农民种植不出问题,确保粮食安全。
醇溶蛋白在多种作物的籽粒中均有表达,同时其具有品种间多态性,被称为植物的“指纹”[8-10]。在麦类作物中,醇溶蛋白的积累几乎不受环境因素的影响,而且具有品种间异质性,因此,可通过醇溶蛋白的多态性进行品种的纯度鉴定[11]。1986年国际种子检验协会(ISTA)颁布了用于小麦、大麦品种鉴定的酸性聚丙烯酰胺凝胶电泳(A-PAGE)标准程序,用于检测作物的纯度及亲缘关系。
研究表明,不同大麦品种的谷蛋白和醇溶蛋白在不同品种间存在总量、各个组分的含量的多态性,可以为群体结构划分和纯度鉴定提供可靠数据支撑[12-14]。同时,谷子醇溶蛋白的研究也表明,不同材料间,谷子醇溶蛋白的多态性明显,说明醇溶蛋白也可作为谷子不同品种的“指纹”[15]。醇溶蛋白在小麦不同品种、材料中具有广泛的等位变异,影响醇溶蛋白积累的QTL位点分布于1B、2A、2B、2D、3A、3B、4A、5A、6A、6B、7A、7B上[16-17]。换言之,影响和参与小麦醇溶蛋白积累的基因在染色体上均有分布,这也是小麦醇溶蛋白可作为“小麦指纹”的重要原因之一。
本研究中,通过标记不同小麦品种开花日期,提取开花后不同天数的小麦籽粒醇溶蛋白,明确醇溶蛋白的积累模式,找到小麦籽粒可以进行醇溶蛋白鉴定的最早籽粒发育时间,实现收获前的纯度鉴定,为鉴定种子纯度提供技术支持。
1 材料和方法
1.1 试验材料
试验材料为济麦31、津强11号、津强13号,种植于天津市农业科学院农作物研究所实验田,试验材料由天津市农业科学院农作物研究所保存。
1.2 试验方法
对小麦开花日期进行标记,收取济麦31、津强11号、津强13号开花后5,10,15,20,25,30,35 d[18]籽粒,自然风干,备用。
小麦育种基地,随机选取来自不同小麦株的185个穗子,置于烘箱,60 ℃烘干,脱粒,每穗检测1粒种子。由实验员随机插入25粒不同小麦品种的籽粒(包含津强11号),由另外的实验员进行醇溶蛋白提取及检测与杂株分析,分析后与最初实验人员进行核对,剔除混入杂株后,计算鉴定种子纯度,以确保试验准确性。
使用研钵将收获的籽粒研磨成粉末状,进行醇溶蛋白的提取。将粉末放置于2.0 mL离心管,向每个样品即离心管中加入200 μL蛋白提取液(70%乙醇,20%蔗糖),盖好盖子,置于摇床,100 r/min振荡提取2 h。取出上步中的离心管,加入150 μL A-PAGE上样缓冲液,80 mL丙三醇,0.02 g甲基绿,20 mL去离子水,上下颠倒混匀,常温离心10 min (转速12 000 r/min)。进行A-PAGE凝胶电泳检测[19-20]。
2 结果与分析
2.1 小麦籽粒不同发育阶段醇溶蛋白表达分析
对济麦31、津强11号、津强13号小麦进行开花标记,提取小麦不同发育阶段籽粒的醇溶蛋白,分别对开花后5,10,15,20,25,30,35 d和完全成熟的小麦籽粒醇溶蛋白进行A-PAGE电泳。发现小麦开花后5,10 d时醇溶蛋白电泳图谱没有清晰明显的条带,此时籽粒醇溶蛋白的含量几乎为零;济麦31 15 d时开始出现条带,津强11号未见明显条带,津强13号条带明显;开花后20 d,3个品种的条带基本稳定,并与完全成熟籽粒基本一致(图1)。
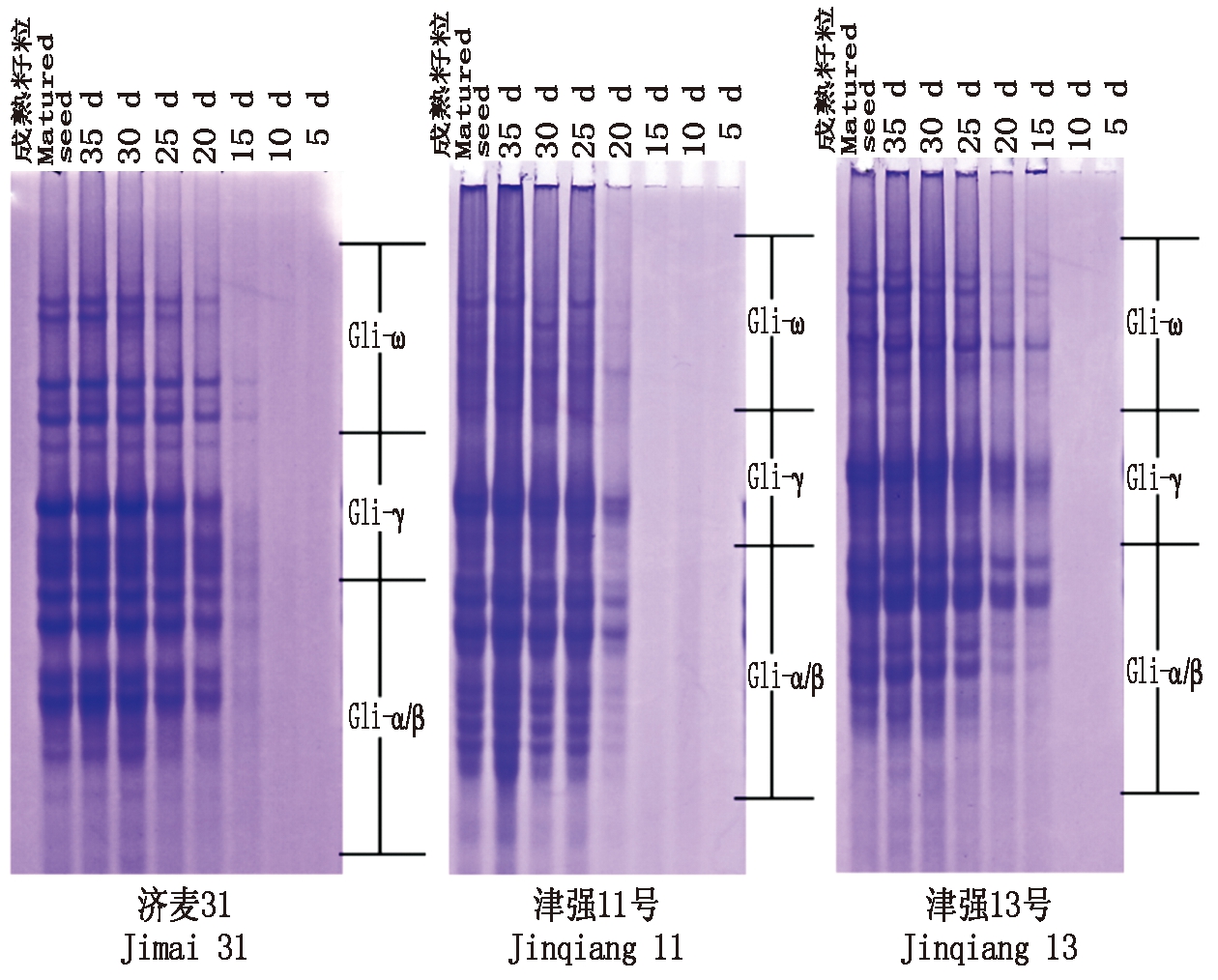
图1 不同发育时期醇溶蛋白表达谱
Fig.1 The expression pattern of gliadin of different stage
2.2 津强11号纯度鉴定
津强11号取株高约75 cm,籽粒红色,田间长势基本一致(图2)。对津强11号收获前的材料进行醇溶蛋白单粒鉴定,在第1个重复中发现14个与

A.单株; B.籽粒;C.大田。
A.Single plant;B.Seed;C.Field.
图2 津强11号田间表型
Fig.2 The phenoltype of Jinqiang 11

图3 津强11号醇溶蛋白纯度鉴定
Fig.3 The seed quality testing based on
gliadin of Jinqiang 11
津强11号带谱不一致的电泳孔道,认为是杂株;在第2个重复中发现14个与津强11号带谱不一致的电泳孔道,认为是杂株;在第3个重复中发现13个与津强11号带谱不一致的电泳孔道,认为是杂株(图3)。
插入的籽粒位于第1,23,27,32,37,42,48,70,82,88,96,100,122,126,134,138,145,148,161,174,177,181,185,188,210孔道(表1),结果发现,与电泳检测结果位置吻合。实验员随机插入25粒,检测出25粒,吻合度为100%。
2.3 纯度鉴定分析
统计分析表明,去掉人为插入杂株后,第1个重复共检测63株,其中杂株7株,纯度为88.89%;第2重复检测62株,其中杂株6株,纯度为90.32%;第3重复检测61株,其中杂株4株,纯度为93.44%。本试验田津强11号平均纯度为90.88%(图4)。
表1 津强11号醇溶蛋白品种纯度鉴定插入品种及位置
Tab.1 The insert variety and location in seed quality testing based on gliadin bands of Jinqiang 11
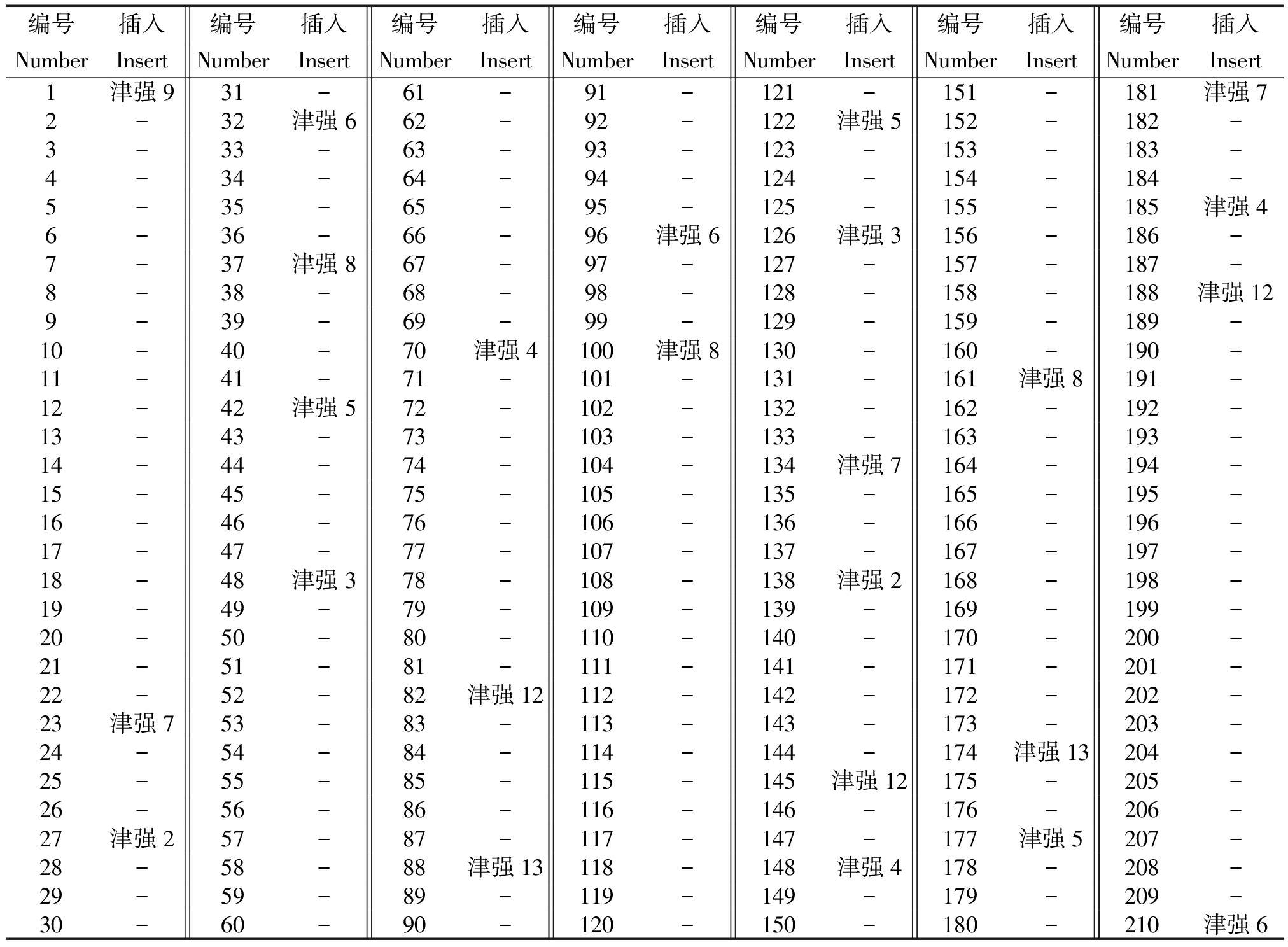
编号插入编号插入编号插入编号插入编号插入编号插入编号插入NumberInsertNumberInsertNumberInsertNumberInsertNumberInsertNumberInsertNumberInsert1津强931-61-91-121-151-181津强72-32津强662-92-122津强5152-182-3-33-63-93-123-153-183-4-34-64-94-124-154-184-5-35-65-95-125-155-185津强46-36-66-96津强6126津强3156-186-7-37津强867-97-127-157-187-8-38-68-98-128-158-188津强129-39-69-99-129-159-189-10-40-70津强4100津强8130-160-190-11-41-71-101-131-161津强8191-12-42津强572-102-132-162-192-13-43-73-103-133-163-193-14-44-74-104-134津强7164-194-15-45-75-105-135-165-195-16-46-76-106-136-166-196-17-47-77-107-137-167-197-18-48津强378-108-138津强2168-198-19-49-79-109-139-169-199-20-50-80-110-140-170-200-21-51-81-111-141-171-201-22-52-82津强12112-142-172-202-23津强753-83-113-143-173-203-24-54-84-114-144-174津强13204-25-55-85-115-145津强12175-205-26-56-86-116-146-176-206-27津强257-87-117-147-177津强5207-28-58-88津强13118-148津强4178-208-29-59-89-119-149-179-209-30-60-90-120-150-180-210津强6
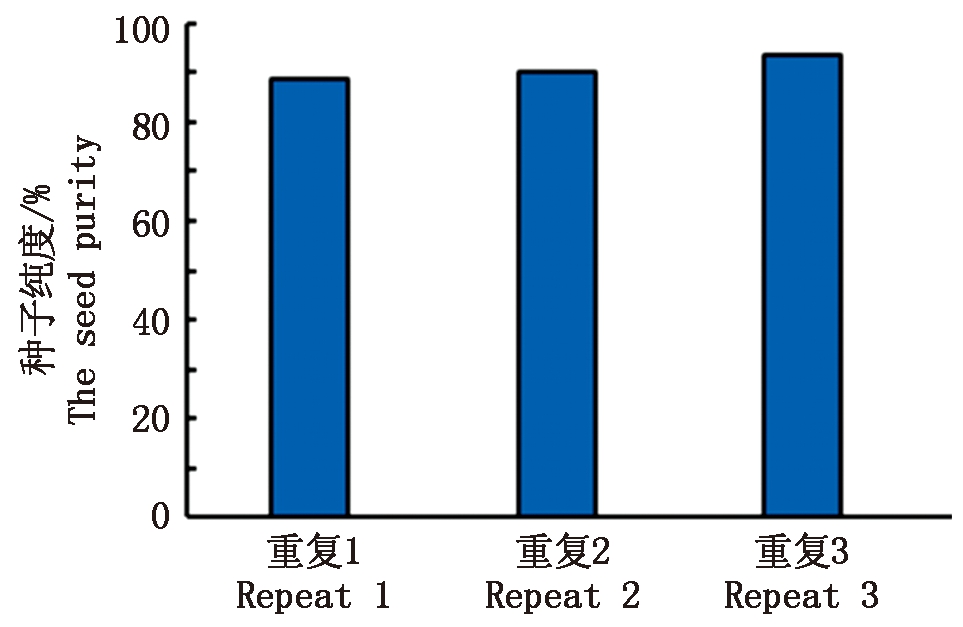
图4 津强11号种子纯度鉴定
Fig.4 The seed purity identification of Jinqiang 11
3 讨论
为了简化良种繁育过程中种子纯度鉴定费时、成本高等困境,本研究通过研究醇溶蛋白表达模式,明确可鉴定醇溶蛋白的最早发育时期,从而在收获前实现种子纯度鉴定。
前人研究表明,编码麦谷蛋白和醇溶蛋白的基因主要在开花后的10~20 d表达,且不同基因组的表达量及表达模式略有差异[21]。编码α/β醇溶蛋白的基因主要在开花后10 d达到表达高峰,随后表达量逐渐下降[22-23]。编码γ型的醇溶蛋白的基因在不同品种间存在差异,如在中国春(Chinese Spring, CS)及中国春的1B染色体代换系(CS-1S1)呈现了不同的表达模式。在CS-1S1中编码γ型的醇溶蛋白的基因从开花后第5天开始表达,开花后10 d表达量迅速上升,在开花后第15天表达出现峰值,而中国春中编码γ型的醇溶蛋白的基因在开花后第10天就达到了表达峰值[24-26]。总之,醇溶蛋白编码基因组高丰度表达,主要集中的开花后的10~20 d。
开花后5,10,15,20,25,30,35 d到完全成熟的小麦籽粒提取醇溶蛋白发现,开花后10 d内的籽粒,均未检测到醇溶蛋白条带,说明醇溶蛋白在开花前期并未大量积累,与基因表达模式相吻合。济麦31开花后第15天出现醇溶蛋白条带,但条带较弱;津强11号在开花后第20天出现可见条带,津强13号在开花后第15天即可出现较明显的醇溶蛋白条带,说明不同品种醇溶蛋白积累速度存在一定差异,但是均呈现出开花后20~25 d可检出明显、完整条带,因此,可在小麦开花后第20~25天至收获前均可进行醇溶蛋白鉴定,完成纯度检测。
目前,DNA指纹纯度检测方法对于种子企业而言存在时间长、工作量大、成本高等诸多问题。同时,单纯依靠外观鉴定种子纯度具有不确定性和风险性,通过对小麦开花后醇溶蛋白的鉴定,能够提早准确的鉴定良种田的种子纯度,为良种繁育企业提供一套新的种子纯度检测技术。
[1] Athwal D S. Semidwarf rice and wheat in global food needs[J].The Quarterly Review of Biology, 1971, 46(1):1-34.doi: 10.1086/406754.
[2] Baar A, Pahr S, Constantin C, Scheiblhofer S,Thalhamer J,Giavi S,Papadopoulos N G,Ebner C,Mari A,Vrtala S,Valenta R. Molecular and immunological characterization of tri a 36, a low molecular weight glutenin, as a novel major wheat food allergen[J]. The Journal of Immunology, 2012, 189(6):3018-3025.doi: 10.4049/jimmunol.1200438.
[3] 刘新月,裴磊,刘莉,张松令,卫云宗,满强,张正斌,徐萍. 优质高产抗旱小麦晋麦101号的选育研究[J]. 分子植物育种, 2019, 17(12):4016-4024.doi:10.13271/j.mpb.017.004016.
Liu X Y,Pei L,Liu L,Zhang S L, Wei Y Z, Man Q, Zhang Z B, Xu P.Breeding of new wheat variety Jinmai 101 with strong gluten, drought resistant and high yield[J].Molecular Plant Breeding, 2019, 17(12):4016-4024.
[4] Bora A, Choudhury P R, Pande V, Mandal A B.Assessment of genetic purity in rice (Oryza sativa L.) hybrids using microsatellite markers[J]. 3 Biotech, 2016,6(1):50. doi: 10.1007/s13205-015-0337-y.
[5] Terryana R T, Rijzaani H, Priyatno T P, Manzila I, Lestari P. Construction of DNA fingerprint for chili pepper varieties using SNAP markers[C]//The 1st International Conference on Genetic Resources and Biotechnology 20-21 August 2018.Bogor:IOP Conference Series Earth and Environmental Science, 2020, 482:012038.doi: 10.1088/1755-1315/482/1/012038.
[6] 李竟才,李世升,张娜娜,徐建波,方元平,项俊.白萝卜纯度鉴定的SSR分子标记筛选与鉴定[J]. 分子植物育种, 2018,16(8):2525-2532.doi:10.13271/j.mpb.016.002525.
Li J C, Li S S, Zhang N N, Xu J B, Fang Y P, Xiang J, Screening and identification of SSR molecular markers for purity identification of white radish[J].Molecular Plant Breeding, 2018, 16(8):2525-2532.
[7] Wang H H, Wijeratne A, Wijeratne S,Lee S,Taylor C G,St Martin S K,Mchale L, Dorrance A E.Dissection of two soybean QTL conferring partial resistance to Phytophthora sojae through sequence and gene expression analysis[J]. Bmc Genomics, 2012, 13(1):428.doi: 10.1186/1471-2164-13-428.
[8] 杨瑞武,魏秀华,周永红,郑有良.赖草属植物醇溶蛋白的遗传多态性[J].云南植物研究,2004,26(1):103-110.doi:10.3969/j.issn.2095.0845.2004.01.011.
Yang R W, Wei X H,Zhou Y H, Zheng Y L. Genetic polymorphism of gliadin in Leymus[J]. Acta Botanica Yunnanica, 2004,26(1):103-110.
[9] Lookhart G L, Bean S R, Culbertson C. Wheat quality and wheat varietal identification[M]//Using cereal ence and technology for the benefit of consumers.Amsterdam:Elsevier, 2005:293-297.doi: 10.1533/9781845690632.8.293.
[10] Mamone G, Addeo F, Chianese L, di Luccia A D,Martino A D, Nappo A,Formisano A, Vivo P D, Ferranti P. Characterization of wheat gliadin proteins by combined two-dimensional gel electrophoresis and tandem mass spectrometry[J]. Proteomics,2005,5(11):2859-2865.doi: 10.1002/pmic.200401168.
[11] Djukic'N, Kneževic'D, Horvat D, Živancˇev D, Torbica A. Similarity of cultivars of wheat (Triticum durum) on the basis of composition of Gliadin alleles[J]. Genetika, 2011, 43(3):527-536.doi:10.2298/GENSR1103527D.
[12] Alvarez J B, Martín L M. Genetic diversity and structure in a natural Hordeum chilense population based on gliadin analysis[J]. Plant Systematics & Evolution, 2006, 261(1-4):11-18.doi:10.1007/s00606-006-0436-7.
[13]  an S K,Özbek Ö,Eser V,Ta
an S K,Özbek Ö,Eser V,Ta kin G B.Polymorphism in seed endosperm proteins (gliadins and glutenins) of turkish cultivated einkorn wheat[Triticum monococcum ssp. monococcum]landraces[J]. Cereal Research Communications,2015,43(1):108-122.doi:10.1556/CRC.2014.0028.
kin G B.Polymorphism in seed endosperm proteins (gliadins and glutenins) of turkish cultivated einkorn wheat[Triticum monococcum ssp. monococcum]landraces[J]. Cereal Research Communications,2015,43(1):108-122.doi:10.1556/CRC.2014.0028.
[14] Utebayev M,Dashkevich S,Bome N,Bulatova K,Shavrukov Y. Genetic diversity of gliadin-coding alleles in bread wheat (Triticum aestivum L.) from northern kazakhstan[J]. PeerJ,2019,7:e7082.doi:10.7717/peerj.7082.
[15] 刘丹,赵子龙,梁丹,曹婷婷,李素敏,王从磊,时晓伟,杨晓然,张明,朱铭,王建贺,冯刚,李素英.谷子、小麦籽粒蛋白、淀粉构成及结构差异分析[J].华北农学报,2020,35(2):72-78.doi:10.7668/hbnxb.20190775.
Liu D,Zhao Z L,Liang D,Cao T T, Li S M, Wang C L, Shi X W, Yang X R, Zhang M, Zhu M, Wang J H,Feng G, Li S Y. Analysis on protein and starch composition and structure of foxtail millet and wheat[J]. Acta Agriculturae Boreali-Sinica,2020,35(2):72-78.
[16] Perretant M R,Cadalen T, Charmet G, Sourdille P, Nicolas P, Boeuf C, Tixier M H, Branlard G, Bernard S. QTL analysis of bread-making quality in wheat using a doubled haploid population[J]. Theoretical and Applied Genetics,2000,100(8):1167-1175.doi:10.1007/s001220051420.
[17] Johnson M,Kumar A,Oladzad-Abbasabadi A,Salsman E,Aoun M,Manthey F A,Elias E M. Association mapping for 24 traits related to protein content, gluten strength, color, cooking, and milling quality using balanced and unbalanced data in durum wheat[Triticum turgidum L. var. durum (Desf).][J]. Frontiers in Genetics,2019,10:717.doi: 10.3389/fgene.2019.00717.
[18] Destro D, Carpentieri-Pípolo V, Kiihl R A D S, AlmeidaL A D. Photoperiodism and genetic control of the long juvenile period in soybean: a review[J]. Crop Breeding &Applied Biotechnology, 2001, 1(1):71-92.doi: 10.13082/1984-7033.v01n01a10.
[19] Ram S, Jain N, Dawar V, Singh R P, Shoran J. Analyses of Acid-PAGE gliadin pattern of indian wheats (Triticum aestivum L.) representing different environments and periods[J]. Crop Science,2005,45(4):1256-1263.doi:10.2135/cropsci2004.0334.
[20] Barak S, Mudgil D, Khatkar B S. Biochemical and functional properties of wheat gliadins: a review[J]. Critical Reviews in Food Science and Nutrition, 2015, 55(3):357-368.doi: 10.1080/10408398.2012.654863.
[21] Wang D W, Li D, Wang J J, Zhao Y, Wang Z J, Yue G D, Liu X, Qin H J, Zhang K P, Dong L L, Wang D W. Genome-wide analysis of complex wheat gliadins, the dominant carriers of celiac disease epitopes[J]. Scientific Reports,2017,7: 44609.doi:10.1038/srep44609.
[22] Kawaura K,Mochida K,Ogihara Y.Expression profile of two storage-protein gene families in hexaploid wheat revealed by large-scale analysis of expressed sequence tags[J]. Plant Physiology, 2005,139(4):1870-1880.doi: 10.1104/pp.105.070722.
[23] Li X H, Wang K, Wang S L, Gao L Y, Xie X X, Hsam S L K, Zeller F J, Yan Y M. Molecular characterization and comparative transcriptional analysis of LMW-m-type genes from wheat (Triticum aestivum L.) and Aegilops species[J]. Theoretical and Applied Genetics, 2010,121(5):845-856.doi:10.1007/s00122-010-1354-1.
[24] Pistón F, Dorado G, Martín A, Barro F.Cloning of nine γ-gliadin mRNAs (cDNAs) from wheat and the molecular characterization of comparative transcript levels of γ-gliadin subclasses[J]. Journal of Cereal Science, 2006,43(1):120-128.doi: 10.1016/j.jcs.2005.07.002.
[25] Wang S L, Shen X X, Ge P, Li J, Subburaj S, Li X H, Zeller F J, Hsam S L K, Yan Y M. Molecular characterization and dynamic expression patterns of two types of γ-gliadin genes from Aegilops and Triticum species[J].Theoretical and Applied Genetics, 2012,125(7):1371-1384.doi: 10.1007/s00122-012-1917-4.
[26] Von Büren M, Lüthy J,Hübner P. A spelt-specific γ-gliadin gene: discovery and detection[J]. Theoretical and Applied Genetics, 2000,100(2):271-279.doi:10.1007/s001220050036.