甜瓜蔓枯病(Gummy stem blight,GSB)是目前全球范围内重要的真菌病害,其病原菌为Stagonosporopsis cucurbitacearum。该病主要通过土壤进行传播,茎蔓和叶片是最主要的发病部位,该病发病极快,一旦发生很难控制,严重影响瓜类的产量和品质,制约了我国瓜类产业的健康发展[1]。选育抗病品种是防治蔓枯病最有效的途径,但由于蔓枯病抗性遗传机制复杂,缺乏高抗病的种质资源,通过常规育种手段选育抗病品种存在耗时长、效果差等问题[2]。因此,明确甜瓜蔓枯病抗病基因对甜瓜蔓枯病抗病育种工作具有重要的意义。
SLAF-Seq(Specific length amplified fragment se-quencing)技术是一种简化的高通量测序技术,其具有效率高、成本低、数量丰富、周期短等优势,该项技术已被广泛应用于构建高密度遗传图谱、高通量分子标记开发、群体进化分析及目标性状的基因定位等方面[3]。QTL(Quantitative trait locus)定位研究为开展分子抗病育种提供了基因资源,国内外已有众多学者利用SLAF-Seq技术结合BSA(Bulked Segregant Analysis)的方法,在不同的植物上获得了与目标性状显著相关的候选基因和分子标记。该技术在包括拟南芥疫霉病[4]、小麦条锈病[5]、玉米大斑病[6]、苹果锈病[7]、黄瓜白粉病[8]、烟草花叶病[9]等许多病害上得到应用。近年来甜瓜全基因组测序的完成为研究甜瓜相关性状的遗传机制奠定了生物信息学的基础,张红等[10]采用SLAF-seq技术结合BSA的方法,结合甜瓜全基因组参考序列,在甜瓜中获得了与甜味和酸味性状相关的基因区域,精细定位了1个与甜瓜含糖量相关基因和3个酸度性状相关基因。
甜瓜蔓枯病抗性遗传机制十分复杂,目前甜瓜对蔓枯病的抗性遗传规律还不是非常明确。通过甜瓜抗感材料杂交后代性状分离的方法,在甜瓜上发现了多个与蔓枯病抗性相关的基因,包括 gsb1、gsb2、gsb3、gsb4和gsb5等[11-13],但目前并未分析这些基因在甜瓜染色体的定位和相关基因功能,因此明确甜瓜蔓枯病抗性基因的定位对甜瓜蔓枯病抗病育种工作有重要的作用。本研究以甜瓜蔓枯病抗病材料P181和感病材料P01为亲本,基于其F2单株接种蔓枯病菌后鉴定结果,分别构建F2极端抗病池和感病池,并利用SLAF-seq和BSA技术对蔓枯病抗性性状进行关联分析,获得GSB主效QTL位点,根据已公布的甜瓜全基因组序列,结合QTL区域内候选基因的功能注释,预测与甜瓜蔓枯病抗病相关的功能基因,为甜瓜蔓枯病相关基因的精细定位和抗病育种的选育提供理论依据。
1 材料和方法
1.1 试验材料
以甜瓜高抗蔓枯病材料P181和高感蔓枯病材料P01为双亲本,与其F2分离群体(180株)共同作为试验群体。甜瓜蔓枯病菌菌株编号14mk020,于2014年在宁波市甜瓜蔓枯病病株中分离并纯化,为宁波市农业科学研究院蔬菜所实验室保存。
1.2 蔓枯病菌接种及病情指数分级
蔓枯病孢子诱导参考张立杰等[14]的方法并略有修改。将蔓枯病菌接种到 PDA培养基活化,在25 ℃黑暗条件下培养5 d。随后挑取菌丝块在燕麦琼脂培养基(艾礼生物科技有限公司,上海)上培养7 d,待白色菌落形成后置于紫外光下进行孢子囊的诱导,诱导条件为紫外时间/黑暗时间=12 h/12 h, 诱导4 d后将菌丝上形成的孢子囊用玻璃棒轻轻刮到无菌水中,用血球计数板在显微镜下计数,并用蒸馏水将孢子悬浮液终浓度调节为1×105个/mL备用。
待甜瓜苗长至三叶一心后,采用喷雾接种法对2个亲本及180株甜瓜F2进行蔓枯病菌孢子液接种处理,喷雾至叶片有滴水时用塑料薄膜覆膜保湿,并放置于人工气候箱中进行培养。接种4 d后统计F2发病情况。病害分级标准参照Zhang等[15]的方法,依据茎蔓病斑发病程度,分级标准如下:1级:无病斑;2级:单个病斑 (1~10 mm) 或几个连续的病斑 (1~20 mm),但未环绕茎部一周;3级:几个连续的病斑 (21~80 mm) 或病斑环绕茎部一周;4级:茎部失水萎缩但植株未死亡;5级:植株死亡。具体如图1所示。

图1 甜瓜蔓枯病不同病害等级
Fig.1 Different disease scales of GSB
1.3 SLAF文库构建及测序
依据F2蔓枯病抗病性鉴定结果,选取F2群体中极端抗病的30个单株和极端感病的30个单株,按照CTAB方法提取单株DNA,并分别构建抗病和感病的DNA混池。利用SLAF技术对合格的DNA样品建库测序,以甜瓜基因组(DHL92)v3.6.1作为参考基因组(http://www.cucurbitgenomics.org/organism/18)。将各样品基因组DNA超声破碎,对得到的片段依次进行3′端加polyA处理、连接测序接头、PCR富集、纯化去接头污染,文库质量检测,最后使用Illumina HiSeq 2000平台进行测序。
1.4 SLAF-seq 测序数据分析及候选基因的筛选
对甜瓜参考基因组进行酶切,将酶切长度在264~414 bp的序列定义为SLAF标签,统计SLAF标签在各染色体的数量。在亲本中搜索多态性的SLAF标签和有reads覆盖区域的SNP。使用GTAK软件对亲本与子代混池进行SNP开发,得到SNP位点集,在关联分析前对SNP进行过滤,依次过滤掉有多个基因型的SNP位点、read支持度小于4的SNP位点、混池间基因型一致的SNP位点及隐性混池基因不是来自于隐性亲本的SNP位点,最终得到高质量的可信SNP位点。采用SNP-index方法计算Δ(SNP-index)指数,取95%置信水平为筛选阈值,对目标性状进行定位,将置信水平线以上的区间定为候选区域。以甜瓜基因组(DHL92)v3.6.1作为参考基因组进行基因注释,利用NR、NT、GO、KEGG和COG等数据库信息,对候选区域内的基因进行功能分析,寻找与甜瓜蔓枯病抗病相关的基因。
2 结果与分析
2.1 亲本及F2蔓枯病抗病性鉴定
在接种后第5天进行蔓枯病抗病性鉴定,亲本发病情况如图2所示,抗病甜瓜亲本P181接种蔓枯病菌后部分叶片略有黄化,出现轻微褐色病斑。感病甜瓜亲本P01接种蔓枯病菌后发病严重,叶片萎蔫,茎部腐烂,整株枯死。
F2发病情况如图3所示,在180株F2群体中,发病等级为1的甜瓜幼苗有40株,等级为2的幼苗有44株,等级为3的幼苗有45株,等级为4的幼苗有35株,等级为5的幼苗有16株。F2群体蔓枯病抗病性状整体呈现连续分布,表明抗病亲本为数量遗传性状,能够进行QTL分析。

图2 感病亲本P01和抗病亲本P181接种蔓枯病后的表型
Fig.2 Phenotype of susceptible parent P01 and resistant
parent P181 with inoculation of S. cucurbitacearum
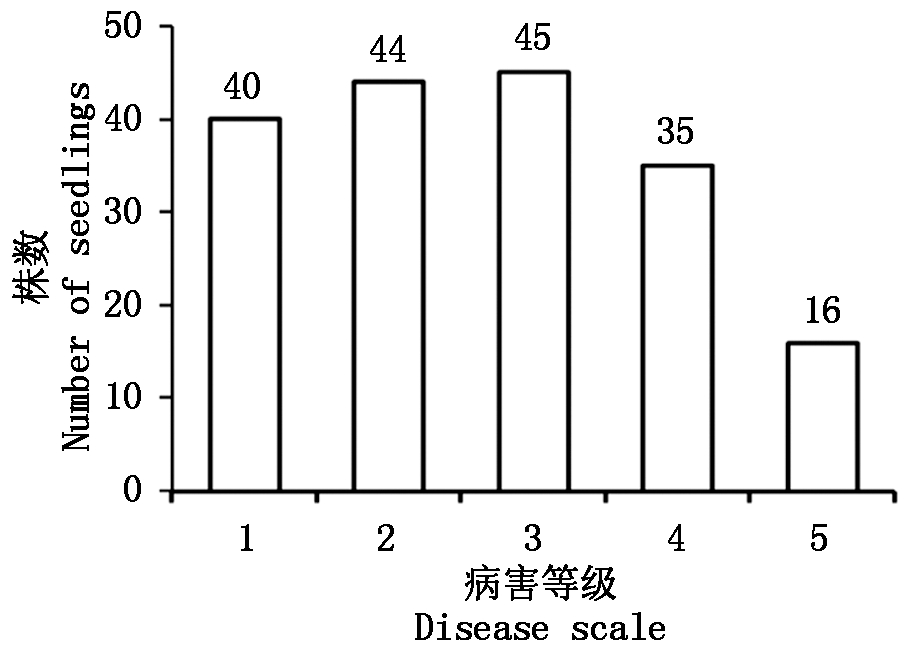
图3 F2群体蔓枯病病级分布
Fig.3 Frequency distribution of disease
scale in the F2 progeny
2.2 SLAF 标记开发和多态性分析
通过SLAF-seq 技术对亲本和极端DNA池进行测序,共获得了126 280 064条 reads 数据,其中父本和母本分别为109 559 650,5 798 772条,抗病池和感病池分别为4 449 302,6 472 340条。测序数据的Q30均大于92%,各样品的GC 含量差别较小,均在37.50%~37.87%,覆盖率均在97%以上(表1),以上结果表明,数据质量高,可用于下一步分析。根据SLAF标签在染色体上的分布,绘制SLAF标签的染色体分布图,具体分布情况如图4所示,开发的SLAF标签在基因组各染色体上分布基本均匀,共获得188 022个SLAF标签。SLAF标签亲本平均测序深度为14.55×,混池平均深度测序深度为13.01×。
表1 各样品SLAF 测序数据评估及比对分析
Tab.1 Statistics of the SLAF sequencing data of each sample
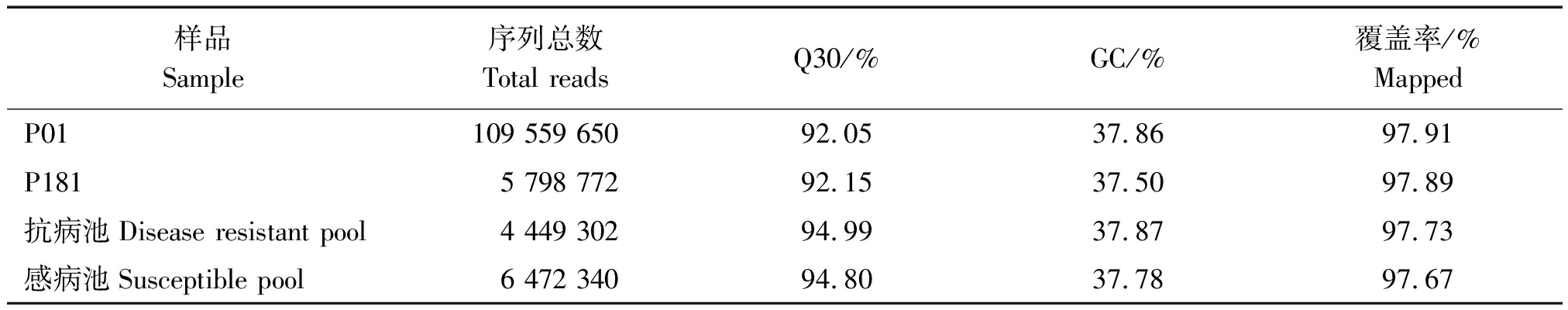
样品Sample序列总数Total readsQ30/%GC/%覆盖率/%MappedP01109 559 65092.0537.8697.91P1815 798 77292.1537.5097.89抗病池Disease resistant pool4 449 30294.9937.8797.73感病池Susceptible pool6 472 34094.8037.7897.67
按照 1 Mb 的窗口对染色体划分。SLAF标签数越多,颜色越深,SLAF标签数越少,颜色越浅;图中颜色越深的区域即SLAF标签集中分布的区域。
The chromosome is divided according to the size of 1 Mb windows. The more the number of SLAF tags in each window, the color is darker, the fewer the number, the color is lighter; the SLAF labels is concentrated on the darker regions in the figure.
图4 候选多态性 SLAF 标签在染色体上的分布
Fig.4 SLAF distribution on genome
2.3 主效QTL定位与分析
在SNP-index关联分析前,先对得到的1 485 054个SNP位点进行过滤,过滤掉有多个等位基因SNP位点共1 358个,再过滤掉混池中reads支持度小于4的位点1 471 553个和亲本中不存在的SNP位点共6 467个,其中由于9号染色体至12号染色体的SNP位点reads值均小于4,因此,在关联分析时这4条染色体上的SNP位点被全部过滤,最终获得了在8条染色体上的高质量SNP位点5 676个,通过SNP_index算法,利用拟合后的ΔSNP_index的99百分位数,得到ΔSNP_index的关联阈值为0.44(图5)。在此阈值上共定位到甜瓜染色体Chr01和Chr04的2个区间,总长度为2.05 Mb,包含218个基因,其中Chr01区域长度为1.19 Mb,位于9 837 447~11 028 838 bp区间,包含60个基因,Chr04区域长度为0.86 Mb,位于33 135 224~33 993 540 bp区间,共包含158个基因(表2)。

图5 SNP-index关联值在染色体上的分布
Fig.5 Distribution of SNP-index association analysis on chromosomes
表2 关联区域信息统计
Tab.2 Statistic information on the association region
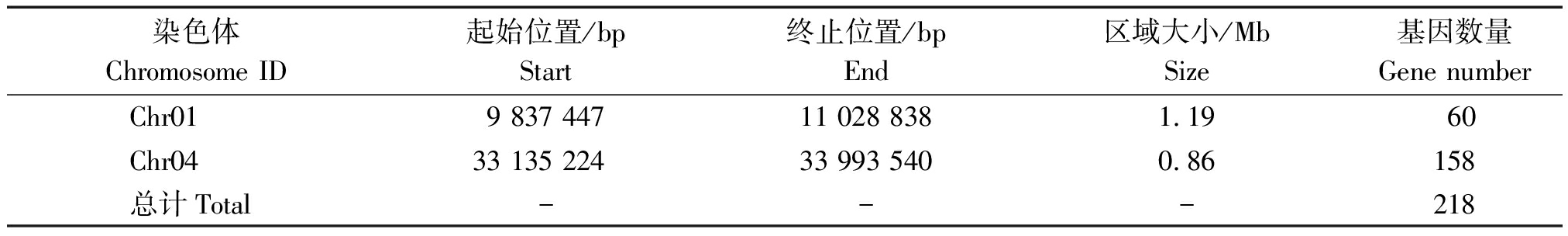
染色体Chromosome ID起始位置/bpStart终止位置/bpEnd区域大小/MbSize基因数量Gene numberChr019 837 44711 028 8381.19 60Chr0433 135 22433 993 5400.86158总计Total---218
2.4 候选区域基因功能分析
利用甜瓜基因组数据库信息,应用Blast软件对候选区域内的基因进行NR、NT、GO、KEGG、COG的深度注释,筛选候选基因,注释结果如表3所示,共在候选区域内注释到189个基因,其中在NR、NT数据库中均注释到189个基因,GO数据库中注释到96个基因,KEGG数据库中注释到64个基因,COG数据库中注释到67个基因。
通过对数据库中的基因进行逐条注释分析,推测与甜瓜蔓枯病抗病性相关的候选基因,分析结果如表4所示,根据前人报道在植物中与抗病性相关的基因,预测共有15个基因与甜瓜蔓枯病抗性相关,包括1个NBS-LRR基因,5个E3泛素蛋白连接酶合成基因,1个RPM1相关蛋白基因,1个锌指蛋白相关基因,1个NAC结构域蛋白基因,2个钙依赖蛋白激酶基因,3个凝集素受体蛋白基因,1个RING-H2指蛋白基因。
表3 候选区域内基因功能注释结果统计
Tab.3 Gene function annotation of candidate regions
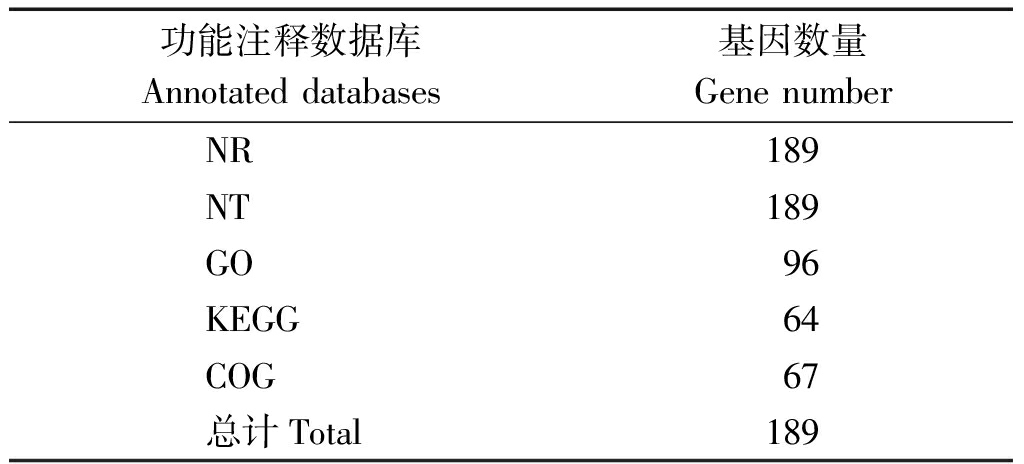
功能注释数据库Annotated databases基因数量Gene numberNR189NT189GO96KEGG64COG67总计Total189
表4 预测甜瓜蔓枯病抗性相关的基因
Tab.4 Predictive genes associated with
GSB resistant of melon
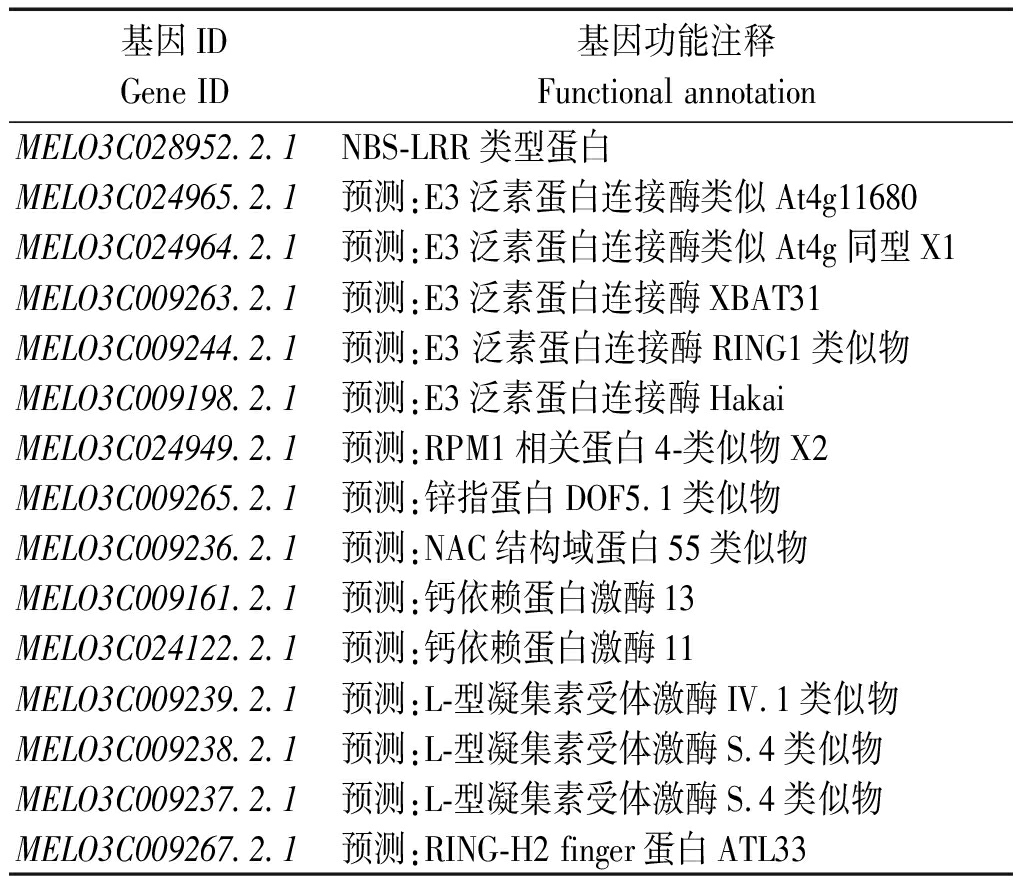
基因IDGene ID基因功能注释Functional annotationMELO3C028952.2.1NBS-LRR类型蛋白MELO3C024965.2.1预测:E3泛素蛋白连接酶类似At4g11680 MELO3C024964.2.1预测:E3泛素蛋白连接酶类似At4g同型X1MELO3C009263.2.1预测:E3泛素蛋白连接酶XBAT31MELO3C009244.2.1预测:E3 泛素蛋白连接酶RING1类似物MELO3C009198.2.1预测:E3泛素蛋白连接酶Hakai MELO3C024949.2.1预测:RPM1相关蛋白4-类似物X2MELO3C009265.2.1预测:锌指蛋白DOF5.1类似物MELO3C009236.2.1预测:NAC结构域蛋白55类似物 MELO3C009161.2.1预测:钙依赖蛋白激酶13MELO3C024122.2.1预测:钙依赖蛋白激酶11MELO3C009239.2.1预测:L-型凝集素受体激酶IV.1类似物MELO3C009238.2.1预测:L-型凝集素受体激酶S.4类似物MELO3C009237.2.1预测:L-型凝集素受体激酶S.4类似物MELO3C009267.2.1预测:RING-H2 finger蛋白ATL33
3 结论与讨论
抗病品种的选育是防治病害最主要的途径,对植物抗病基因的筛选能够从分子层面明确植物的抗病机制。本研究通过 SLAF-BSA技术对甜瓜抗蔓枯病性状进行基因定位,开发 SLAF标签188 022个,高质量SNP标记位点5 676个,这些信息为甜瓜抗病基因的关联定位奠定了基础,在甜瓜基因组中共获得了2个与蔓枯病抗病相关QTL区域。分别定位在染色体Chr01和Chr04,总区间为2.05 Mb。通过筛选得到15个可能与蔓枯病抗病性相关的候选基因,其功能注释涉及NBS-LRR蛋白,E3泛素蛋白连接酶,RPM1相关蛋白、锌指蛋白、NAC结构域蛋白、钙依赖蛋白激酶等。这些基因在植物中均与抗病性密切相关。目前研究结果表明,NBS-LRR结构域能够结合ATP或GTP,参与植物抗病信号的传递[16]。E3泛素蛋白连接酶基因能够调节植物的免疫反应,在番茄叶霉病[17]、小麦白粉病[18]等病害抗病性方面均有报道,RPM1效应蛋白[19]、锌指蛋白[20]、NAC结构域蛋白[21]、钙依赖蛋白激酶[22-23]、凝集素受体激酶[24]、RING-H2 finger蛋白[25]等在也已明确许多植物能够起到抗病调控作用。
目前,瓜类蔓枯病抗病基因的研究主要集中在甜瓜和黄瓜上,不同瓜类不同种系间的抗性遗传特性均不一致[26]。研究结果发现,蔓枯病抗性遗传包括显性遗传和隐性遗传,且大部分为显性遗传。在甜瓜不同的蔓枯病抗性材料中,已发现5个单独作用的抗性基因(gsb-1、gsb-2、gsb-3、gsb-4、gsb-5),但这些研究并明确抗病基因的定位位点,对甜瓜蔓枯病QTL区域内抗病基因功能也未分析。直至2018年,Hu等[27]对甜瓜抗感材料杂交后的F2抗病性状分离,结合基因BSA方法,首先明确了甜瓜蔓枯病的抗感遗传为单基因显性遗传。并将甜瓜抗蔓枯病基因定位在了位于4号染色体的3.94~4.05 Mb的区域内,最终确定MELO03C012987基因与蔓枯病抗病性相关,该基因为编码类似于ACRE146的蛋白。但甜瓜蔓枯病抗性基因定位相关报道仍然较少。在黄瓜蔓枯病抗病基因定位研究方面,目前已发现7个基因与黄瓜蔓枯病的抗性相关,定位在Chr01和Chr04之间,包括LRR基因、Toll蛋白结构域、锌指结构域等[28]。本研究通过对甜瓜蔓枯病进行QTL进行定位,利用甜瓜全基因组信息对候选基因进行注释。研究结果发现,在甜瓜上的定位与前人报道甜瓜蔓枯病和黄瓜蔓枯病的抗病基因定位区域相似,均在Chr01和Chr04的区域内,2个作物间的蔓枯病抗病基因及抗病机制是否具有相似性值得探讨。
通过前人对不同作物不同病害抗病基因的鉴定结果,本研究初步预测上述15个基因可能为参与甜瓜抗蔓枯病调控的候选基因。但具体哪些基因对蔓枯病抗性起到关键的主效作用仍需进一步进行功能及表达验证。在本研究基础上,未来期待通过进一步开展抗病基因克隆技术,对基因功能进行验证,同时开发相应的分子标记。这些成果将对阐明甜瓜与蔓枯病菌的互作机制及蔓枯病分子标记辅助育种具有重要意义。
[1] 高天一,郝芳敏,臧全宇,马二磊,黄芸萍,王毓洪.瓜类蔓枯病研究进展[J].中国瓜菜,2020,33(6):1-5.doi:10.16861/j.cnki.zggc.2020.0128.
Gao T Y, Hao F M, Zang Q Y, Ma E L, Huang Y P, Wang Y H.Research progress on gummy stem blight of cucurbitaceae crops[J].China Cucurbits and Vegetables, 2020,33(6):1-5.
[2] 毕研飞,徐兵划,郭静,张永兵,伊鸿平,钱春桃,陈劲枫.分子标记辅助甜瓜抗蔓枯病基因聚合及白皮脆品种改良[J].南京农业大学学报,2015,38(3):375-380. doi:10.7685/j.issn.1000-2030.2015.03.004.
Bi Y F,Xu B H, Guo J, Zhang Y B, Yi H P, Qian C T, Chen J F.Pyramiding disease resistance genes by marker-assisted selection in melon (Cucumis melo L.) and Baipicui breed improvement[J].Journal of Nanjing Agricultural University, 2015, 38(3):375-380.
[3] 代慧英,张玉琮,单文娟.基于SLAF-seq技术的动物基因组应用及进展[J].生物技术,2020,30(3):290-294.doi:10.16519/j.cnki.1004-311x.2020.03.0047.
Dai H Y,Zhang Y C,Shan W J.Development and application of animals genome based on SLAF-seq technology[J].Biotechnology, 2020, 30(3):290-294.
[4] Meng Y L, Huang Y H, Wang Q H, Wen Q J, Jia J B, Zhang Q, Huang G Y, Quan J L, Shan W X.Phenotypic and genetic characterization of resistance in Arabidopsis thaliana to the oomycete pathogen Phytophthora parasitica[J].Frontiers in Plant Science, 2015, 6:318. doi:10.3389/fpls.2015.00378.
[5] Yin J L, Fang Z W, Sun C, Zhang P, Zhang X, Lu C, Wang S P, Ma D F, Zhu Y X.Rapid identification of a stripe rust resistant gene in a space-induced wheat mutant using specific locus amplified fragment (SLAF) sequencing[J].Scientific Reports, 2018, 8(1):3086.doi:10.1038/s41598-018-21489-5.
[6] 马骏,刘欣芳,齐欣,弓雪,李明,姜敏.基于SALF-seq的玉米抗大斑病基因QTL分析[J]. 辽宁农业科学,2018(2):8-13.doi:10.3969/j.issn.1002-1728.2018.02.002.
Ma J,Liu X F,Qi X,Gong X,Li M,Jiang M.QTL analysis of north corn leaf blight by SALF-seq[J]. Liaoning Agricultural Sciences, 2018(2):8-13.
[7] 张朝红,陈东玫,杨凤秋,赵同生,李扬,赵国栋,赵永波.苹果SLAF图谱构建及果锈基因QTL分析[J].华北农学报,2019,34(5):37-44.doi:10.7668/hbnxb.201751695.
Zhang C H, Chen D M, Yang F Q, Zhao T S, Li Y, Zhao G D, Zhao Y B.Genetic mapping and QTL analysis of fruit russeting in Malus domestica Borkh.[J].Acta Agriculturae Boreali-Sinica, 2019, 34(5):37-44.
[8] Zhang P, Zhu Y Q, Wang L L, Chen L P, Zhou S J.Mining candidate genes associated with powdery mildew resistance in cucumber via super-BSA by specific length amplified fragment (SLAF) sequencing[J].BMC Genomics, 2015, 16:1058.doi:10.1186/s12864-015-2041-z.
[9] 潘旭浩,程立锐,陈小翠,蒋彩虹,任民,杨爱国.基于SLAF-seq技术的烟草抗CMV主效QTL定位[J].中国烟草科学,2018,39(5):1-8.doi:10.13496/j.issn.1007-5119.2018.05.001.
Pan X H, Cheng L R, Chen X Q, Jiang C H, Ren M, Yang A G.CMV resistance QTL analysis in tobacco based on SALF-seq technique[J].Chinese Tobacco Science, 2018, 39(5):1-8.
[10] 张红,吴海波,李寐华,熊韬,王广智,伊鸿平.基于super-BSA技术开发甜瓜含糖量和酸度性状的功能性分子标记[J].中国瓜菜,2015,28(6):21-25.doi:10.16861/j.cnki.zggc.2015.06.005.
Zhang H, Wu H B, Li M H, Xiong T, Wang G Z, Yi H P.Super-BSA-based development of functional molecular markers of sugar content and acidity of melon[J].China Cucurbits and Vegetables, 2015, 28(6):21-25.
[11] Wolukau J N, Zhou X H, Chen J F.Identification of amplified fragment length polymorphism markers linked to gummy stem blight (Didymella bryoniae) resistance in melon (Cucumis melo L.) PI420145[J].Hortscience, 2009, 44(1):32-34.doi:10.21273/hortsci.44.1.32.
[12] Zhang N, Xu B H, Bi Y F, Lou Q F, Chen J F, Qian C T, Zhang Y B, Yi H P.Development of a muskmelon cultivar with improved resistance to gummy stem blight and desired agronomic traits using gene pyramiding[J].Czech Journal of Genetics and Plant Breeding, 2017, 53(1):23-29.doi:10.17221/84/2016-CJGPB.
[13] Frantz J D, Jahn M M. Five independent loci each control monogenic resistance to gummy stem blight in melon (Cucumis melo L.)[J].Theoretical and Applied Genetics, 2004, 108(6):1033-1038.doi:10.1007/s00122-003-1519-2.
[14] 张立杰,张慧玲,哈矿武,王建设.甜瓜蔓枯病病原菌分离与分生孢子的产孢诱导[J].宁夏大学学报(自然科学版),2015,36(4):382-385.doi:10.3969/j.issn.0253-2328.2015.04.020.
Zhang L J, Zhang H L, Ha K W, Wang J S.Isolation and identification of pathogen of Didymella bryoniae and spore production induction of condium[J]. Journal of Ningxia University (Natural Science Edition), 2015, 36(4):382-385.
[15] Zhang Y P, Kyle M, Anagnostou K,Zitter T A.Screening Melon (Cucumis melo) for resistance to gummy stem blight in the greenhouse and field[J].Hort Science, 1997, 32(1):117-121.doi:10.21273/HORTSCI.32.1.117.
[16] Saraste M, Sibbald P R, Wittinghofer A.The P-loop-a common motif in ATP-and GTP-binding proteins[J].Trends Biotechnol,1990, 15(11):430-434.doi:10.1016/0968-0004(90)90281-f.
[17] Chen S C, Zhao H J, Wang M M, Li J D, Wang Z H, Wang F H, Liu A R, Ahammed G J.Overexpression of E3 ubiquitin ligase gene AdBiL contributes to resistance against chilling stress and leaf mold disease in tomato[J].Frontiers in Plant Science, 2017, 8: 1109.doi:10.3389/fpls. 2017.01109.
[18] Zhu Y F, Li Y B, Fei F, Wang Z K, Wang W, Cao A Z, Liu Y, Han S, Xing L P, Wang H Y, Chen W, Tang S Y, Huang X H, Shen Q H, Xie Q, Wang X E.E3 ubiquitin ligase gene CMPG1-V from Haynaldia villosa L. contributes to powdery mildew resistance in common wheat (Triticum aestivum L.)[J]. The Plant Journal, 2015, 84(1):154-168.doi:10.1111/tpj.12966.
[19] Nie Y B, Ji W Q.Cloning and characterization of disease resistance protein RPM1 genes against powdery mildew in wheat line N9134[J].Cereal Research Communications, 2019,47(3):473-483.doi:10.1556/0806.47.2019.27.
[20] Yu Y H, Li X Z, Wu Z J, Chen D X, Li G R, Li X Q, Zhang G H.VvZFP11, a Cys2His2-type zinc finger transcription factor, is involved in defense responses in Vitis vinifera[J].Biologia Plantarum 2016, 60(2):292-298.doi:10.1007/S10535-016-0598-2.
[21] Chang Y N, Yu R M, Feng J L, Chen H Z, Eri H M, Gao G.NAC transcription factor involves in regulating bacterial wilt resistance in potato[J].Functional Plant Biology, 2020, 47(10):925-936.doi:10.1071/FP19331.
[22] Bundó M, Coca M.Enhancing blast disease resistance by overexpression of the calcium-dependent protein kinase OsCPK4 in rice[J].Plant Biotechnology Journal, 2016, 14(6):1357-1367.doi:10.1111/pbi.12500.
[23] Fantino E, Segretin M E, Santin F, Mirkin F G, Ulloa R M.Analysis of the potato calcium-dependent protein kinase family and characterization of StCDPK7, a member induced upon infection with Phytophthora infestans[J].Plant Cell Reports, 2017, 36(7):1137-1157.doi:10.1007/s00299-017-2144-x.
[24] Wang Y, Bouwmeester K, Beseh P, Shan W X, Govers F.Phenotypic analyses of Arabidopsis T-DNA insertion lines and expression profiling reveal that multiple L-type lectin receptor kinases are involved in plant immunity[J].Molecular Plant-Microbe Interactions, 2014, 27(12):1390-1402.doi:10.1094/MPMI-06-14-0191-R.
[25] Ni X M, Tian Z D, Liu J, Song B T, Xie C H.Cloning and molecular characterization of the potato RING finger protein gene StRFP1 and its function in potato broad-spectrum resistance against Phytophthora infestans[J].Journal of Plant Physiology, 2010, 167(6):488-496.doi:10.1016/j.jplph.2009.10.019.
[26] Keinath A P.Reproduction of Didymella bryoniae on nine species of cucurbits under field conditions[J].Plant Disease, 2014, 98(10):1379-1386.doi:10.1094/PDIS-02-14-0139-RE.
[27] Hu Z Y, Deng G C, Mou H P, Xu Y H, Li C, Yang J H, Zhang M F.A re-sequencing-based ultra-dense genetic map reveals a gummy stem blight resistance-associated gene in Cucumis melo[J].DNA Research, 2018, 25(1):1-10.doi: 10.1093/dnares/dsx033.
[28] Liu S L, Shi Y X, Miao H, Wang M, Li B J, Gu X F, Zhang S P.Genetic analysis and QTL mapping of resistance to gummy stem blight in Cucumis sativus seedling stage[J].Plant Disease, 2017, 101(7):1145-1152.doi:10.1094/PDIS-08-16-1116-RE.