TCP(TEOSINTE BRANCHED 1/CYCLOIDEA/PROLIFERATING CELL FACTOR)转录因子由Cubas等[1]在1999年正式提出,该类型转录因子在植物中广泛存在。其中,Teosinte branched 1(TB1)基因最开始在玉米中发现,能抑制腋芽分生组织的生长并能促进雌性花序的形成[2-3]。Cycloidea(CYC)基因来源于金鱼草,该基因控制着金鱼草花的对称性[4]。PROLIFERATING CELL FACTOR(PCF)是在水稻中发现的转录因子,研究发现PCF1和PCF2能结合水稻增殖细胞核抗原(PCNA)基因的启动子,从而调控分生组织的特异性表达[5]。TCP转录因子是植物所特有的一类蛋白质,在多细胞藻类中也有TCP蛋白的存在,仅在单细胞藻类中未发现[6]。TCP转录因子家族保守结构域是一个非典型的碱性氨基酸-螺旋-环-螺旋(basic-Helix-Loop-Helix, bHLH)结构,根据该结构域的差异可分为classⅠ和classⅡ 2个亚家族,classⅠ和classⅡ识别不同的顺式作用元件site IIa (GGNCCCAC)和site IIb (GTGGNCCC)[1, 7]。
TCP转录因子广泛参与植物生长发育调控和环境信号应答的过程。在拟南芥和玉米中发现,与TB1s同源的AtBRC1(AtTCP18)和ZmTB1对分枝的形成表现为负调控作用,通过抑制腋芽分生组织的生长从而抑制分枝的形成[2, 8-9]。菊花CmTCP7基因属CYC类TCP转录因子,研究发现,该基因在菊花小花花芽分化时期和舌状花花瓣中高表达,表明该基因可能参与形成两侧对称的舌状花[10]。水稻OsTB1是一个ZmTB1同源基因,研究发现在Ostb1突变体中水稻分蘖数增加,在过表达OsTB1水稻中分蘖数减少[11-12]。在金鱼草和拟南芥中发现,一些TCP基因能抑制叶片生长,抑制这些基因的表达后出现叶片边缘过渡生长而产生褶皱、锯齿的表型[13-14]。研究发现,拟南芥转录因子TCP14与TCP15参与GA3信号转导过程,响应GA信号后激活相关基因的表达从而正向调控种子萌发[15]。此外,ⅰlhan等[16]在菜豆中鉴定出了27个TCP基因,并通过启动子分析和荧光定量PCR技术发现,有10个菜豆Pvul-TCP基因表达受盐胁迫调控。还有研究表明,转BpTCP7基因能提高白桦的耐盐碱能力[17]。综上所述,TCP转录因子参与了植物分枝形成、花形态建成、叶片生长、种子萌发、逆境响应等过程。
目前,前人分析鉴定出了31个马铃薯TCP转录因子成员,发现在200 mmol/L NaCl胁迫条件下有6个StTCPs基因成员表达差异显著,但其功能尚不明确[18]。因此,本研究基于转录组数据,采用同源克隆的方法克隆出1个马铃薯TCP基因,根据同源性将其命名为StTCP13,实时荧光定量PCR技术(qRT-PCR)测定该基因的表达模式,并构建原核表达载体,在大肠杆菌中表达融合蛋白,通过观察大肠杆菌在不同浓度NaCl盐胁迫条件下的生长表型,初步探究该基因的功能。
1 材料和方法
1.1 试验材料
1.1.1 植物材料 供试材料为马铃薯品种费乌瑞它(Solanum tuberosum L. Favorita),无菌苗由四川农业大学马铃薯研究与开发中心提供。取(21±1) ℃光照培养箱(16 h光/8 h暗)中培养30 d的费乌瑞它无菌苗,整株液氮速冻后于-80 ℃保存,用于总RNA提取。
1.1.2 主要试剂 DNA凝胶纯化回收试剂盒、质粒小量抽提试剂盒、TRIzol、2× Taq PCR Master Mix、感受态大肠杆菌DH5α和BL21等购自天根公司;限制性内切酶、T4连接酶、cDNA合成试剂盒、克隆载体pMD19-T等购自TaKaRa;DNA Marker、蛋白Marker、Loading Buffer等购自Solarbio;原核表达载体pGEX-6P-1由本实验室提供。
1.2 试验方法
1.2.1 马铃薯StTCP13基因克隆 采用同源克隆法,参考NCBI收录的马铃薯(S. tuberosum)TCP13基因CDS序列,登录号为XM_006365599.2,设计基因5′端为BamH Ⅰ、3′端为Xho Ⅰ 酶切位点,Primer 5.0软件设计引物(5′-3′)为:F-CGGGGATCCATGATA CAAAAGATGATTACTAGAGAAG;R-CGGCTCGAGCT AATTATGTGGATTGAAAAAATATTGT。PCR扩增程序为95 ℃预变性3 min; 95 ℃变性30 s,58 ℃退火30 s,72 ℃延伸50 s,35个循环; 72 ℃延伸2 min 30 s,12 ℃保存。琼脂糖凝胶电泳检测结果,目的片段纯化回收后连接T载体,连接产物转化感受态大肠杆菌DH5α,菌落PCR法筛选阳性单克隆送至生工生物测序。
1.2.2 马铃薯StTCP13蛋白结构域分析与系统进化树 采用DNAMAN软件进行氨基酸序列分析与同源性比对,NCBI 数据库的CDD软件(https://www.ncbi.nlm.nih.gov/cdd)分析结构域,MEGA10和iTOL软件构建系统进化树。
1.2.3 马铃薯StTCP13基因的表达模式分析 取样费乌瑞它原原种愈伤后的顶芽芽眼、常温(16±2)℃储藏至发芽芽长为2 mm的顶芽,以及出苗后30 d植株的根、直立茎、叶片进行取样,液氮速冻后-80 ℃保存备用。TRIzol法提取RNA并进行反转录,qRT-PCR测定其相对表达量。从数据库Spud DB(http://potato.plantbiology.msu.edu/)获得StTCP13基因在不同胁迫条件下的FPKM值,Excel整理、TBtool软件分析作图。
1.2.4 马铃薯StTCP13-pGEX6P1原核表达载体的构建 使用BamHⅠ和XhoⅠ 2种限制性内切酶将目的片段从重组T载体上切下,并将pGEX-6P-1载体双酶切线性化,分别纯化回收后用T4连接酶将目的片段与6P-1载体进行连接,连接产物转化感受态大肠杆菌DH5α,随机挑选单克隆菌落扩大培养后提取质粒,双酶切鉴定法筛选阳性单克隆送至生工生物测序。
1.2.5 马铃薯StTCP13-GST标签融合蛋白的原核表达 提取StTCP13-pGEX6P1重组质粒转化感受态大肠杆菌BL21,菌落PCR法筛选阳性单克隆。取1个新鲜的阳性单克隆接种于10 mL含氨苄抗性(Amp+)的LB培养液中,37 ℃摇床200 r/min培养至OD600为0.5,加入终浓度为0.5 mmol/L的IPTG诱导剂诱导0,2,4,6 h,每次取样1 mL菌液于4 ℃保存。样品于12 000 r/min离心集菌1 min,加入80 μL PBS和20 μL 5×Loading Buffer混匀并煮沸8 min,再次12 000 r/min离心1 min,取上清进行SDS-PAGE凝胶电泳。
1.2.6 转StTCP13大肠杆菌BL21盐胁迫表型分析 设置阴性对照为转pGEX-6P-1空载体大肠杆菌BL21,分别挑取阴性对照和转StTCP13大肠杆菌BL21的1个新鲜单克隆,接种至5 mL含Amp+的LB培养液中,37 ℃摇床200 r/min培养至OD600为0.5。4 ℃低温12 000 r/min离心1 min集菌,1 mL无菌超纯水重悬洗涤,重复2次。用无菌超纯水调OD600为0.03,稀释倍数1×、10×、100×、1 000×、10 000×,分别取3 μL在空白对照和盐胁迫处理培养基上点斑。盐胁迫处理培养基为额外添加200,250 mmol/L NaCl的含Amp+LB培养基,空白对照为不额外添加NaCl的含Amp+LB培养基,设置3次重复。37 ℃恒温培养箱倒置培养8 h观察表型。
2 结果与分析
2.1 马铃薯StTCP13基因克隆
1%琼脂糖凝胶电泳检测PCR扩增产物,结果见图1。在500~750 bp,有单一清晰条带。将该片段回收纯化后连接T载体并转化大肠杆菌DH5α,菌落PCR法鉴定阳性单克隆,随机挑选3个阳性单克隆测序。单克隆测序结果一致,测序得到马铃薯StTCP13基因CDS全长669 bp。
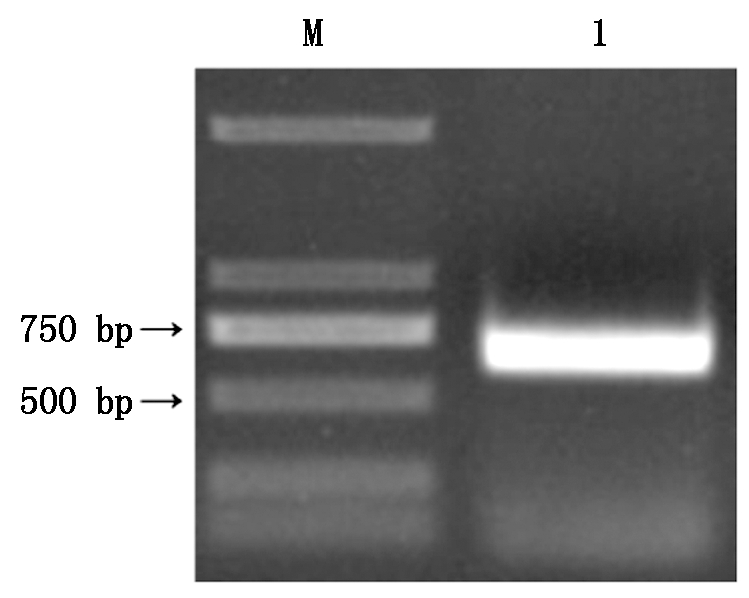
M.2000 bp Marker; 1.StTCP13基因。
M.2000 bp Marker; 1.StTCP13 gene.
图1 StTCP13基因克隆
Fig.1 Cloning of StTCP13 gene
2.2 马铃薯StTCP13蛋白结构域分析
马铃薯StTCP13蛋白结构域预测结果如图2-A所示,在第39-105位氨基酸发现一个典型的TCP超家族结构域。玉米ZmTB1、水稻OsPCF1和OsPCF2是早期发现的TCP转录因子,从NCBI数据库获取ZmTB1(NM_001382583.1)、OsPCF1(XM_015778101.2)、OsPCF2(XM_015792844.2)氨基酸序列,利用DNAMAN软件进行多序列比对,结果如图2-B所示,StTCP13氨基酸序列存在一个非典型的bHLH结构,说明StTCP13可能编码一个由222个氨基酸组成的TCP类蛋白。

图2 StTCP13蛋白结构域分析
Fig.2 Structure domain analysis of StTCP13 protein
2.3 马铃薯StTCP13蛋白系统进化树构建
运用NCBI网站的BlastP程序,检索StTCP13同源性的氨基酸序列,选择相似度不同20个物种的氨基酸序列,MEGA10和iTOL软件构建系统进化树,结果见图3。马铃薯StTCP13与潘那利番茄SpTCP13(XP_006365661.1)、甜椒CaTCP13(XP_016561903.1)、烟草NtTCP13(XP_016491074.1)等氨基酸序列相似度较高,与阔叶栎QlTCP13(XP_030953332.1)、相思子ApTCP13(XP_027358156.1)等氨基酸序列相似度较低。结果表明,StTCP13氨基酸序列保守性与物种进化关系一致。
2.4 马铃薯StTCP13基因的表达模式分析
利用qRT-PCR技术测定StTCP13基因在马铃薯根、茎、叶、芽、芽眼各组织的相对表达量,结果采用-2ΔΔCt法计算,LSD法进行显著性检验。组织表达模式见图4-A,该基因主要在马铃薯的根、叶片和芽中表达,在根中的相对表达量最高、芽眼中相对表达量最低,在根中的表达量是茎中表达量的57.28倍、是芽眼中的224.93倍。整理Spud DB数据库中StTCP13基因的FPKM值,对StTCP13基因的胁迫条件下的表达模式进行分析,结果如图4-B。在150 mmol/L NaCl、50 μmol/L ABA、260 μmol/L甘露醇处理条件下,该基因相对表达量不同程度上调,甘露醇处理条件下表达上调显著,说明StTCP13基因的表达受高盐、ABA、干旱等非生物胁迫条件的诱导。
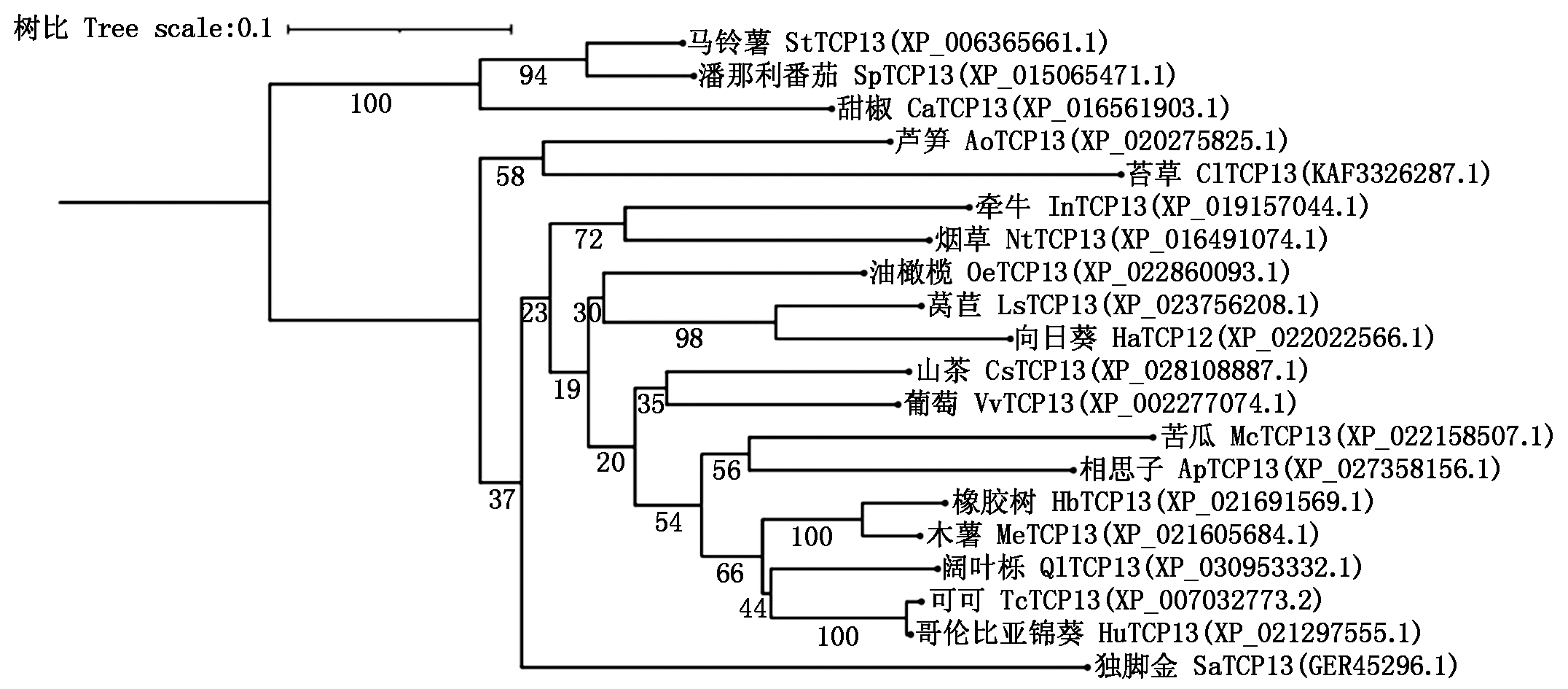
图3 StTCP13蛋白系统进化树
Fig.3 Phylogenetic tree of StTCP13 protein
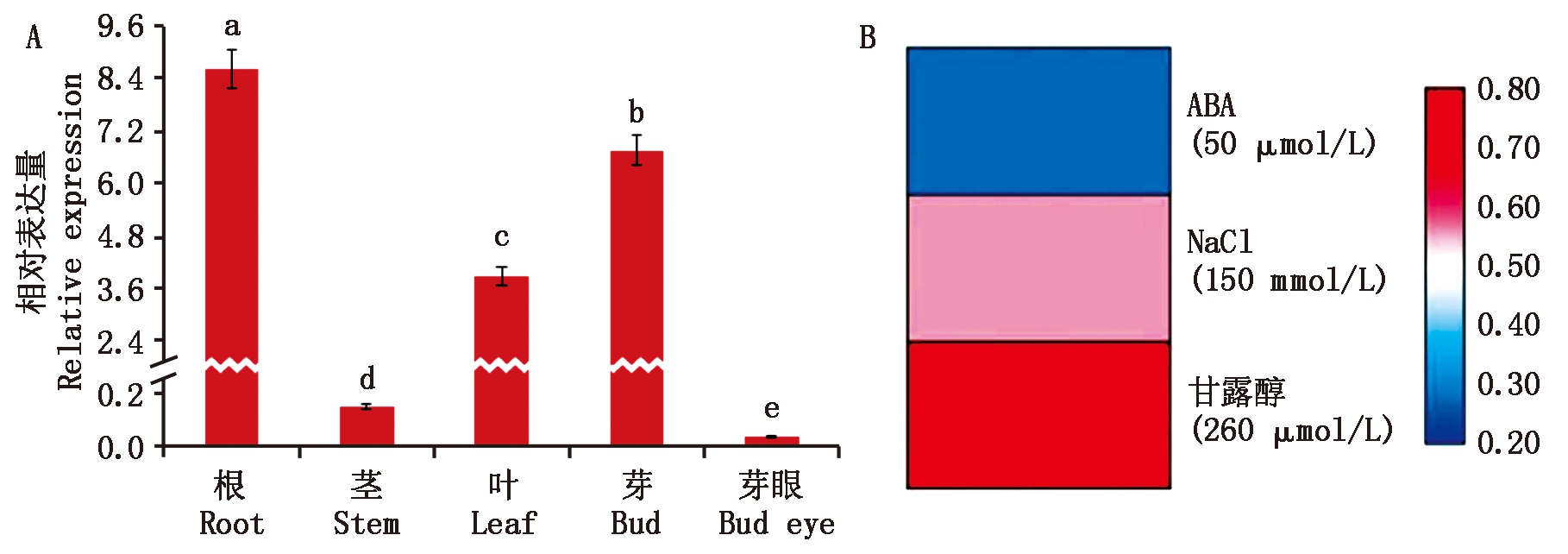
不同小写字母差异显著(P<0.05)。
Significant difference in different lowercase letters (P<0.05).
图4 StTCP13基因的表达模式分析
Fig.4 Analysis of StTCP13 expression pattern
2.5 马铃薯StTCP13-pGEX6P1原核表达载体的构建
设计BamHⅠ和XhoⅠ酶切位点,将 StTCP13连接到pGEX-6P-1原核表达载体上,重组载体转化大肠杆菌,双酶切法鉴定阳性单克隆,结果如图5所示,在500~750 bp处有单一条带,将鉴定的阳性克隆送至测序,测序结果证明StTCP13-pGEX6P1原核表达载体正向构建成功。
2.6 马铃薯StTCP13-GST标签融合蛋白的表达
将构建成功的重组StTCP13-pGEX6P1原核表达载体导入大肠杆菌BL21中,以IPTG(终浓度0.5 mmol/L)为诱导剂,诱导StTCP13-GST标签融合蛋白表达,诱导0,2,4,6,8 h后取样进行SDS-PAGE电泳,预测StTCP13-GST融合蛋白大小约为51 ku,检测结果如图6所示。诱导2 h后,检测到StTCP13-GST融合蛋白的表达,且随诱导时间的增加,目的蛋白表达量上升,结果表明在大肠杆菌BL21中诱导表达了StTCP13-GST融合蛋白,蛋白大小约为51 ku。
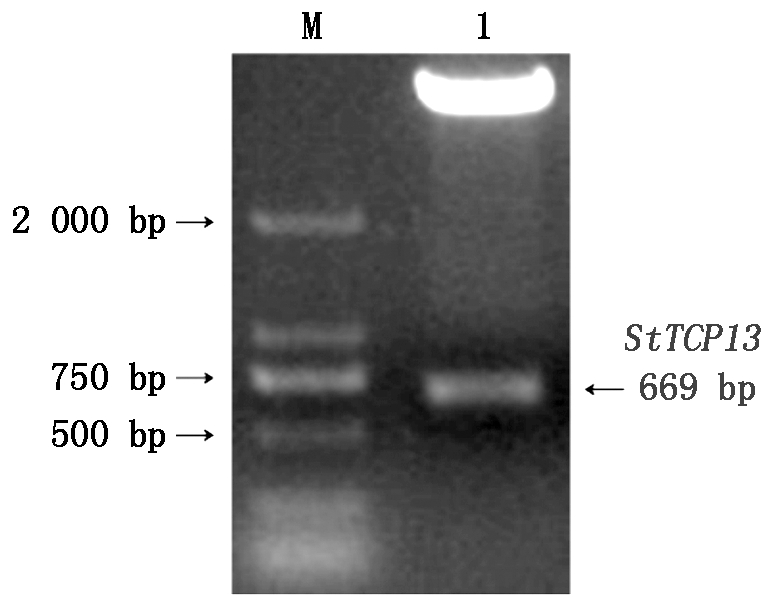
M.2000 bp Marker; 1.pGEX-6P-1和StTCP13基因。
M.2000 bp Marker; 1.pGEX-6P-1 vector and StTCP13 gene.
图5 StTCP13-pGEX6P1载体构建
Fig.5 Vector construction of StTCP13-pGEX6P1
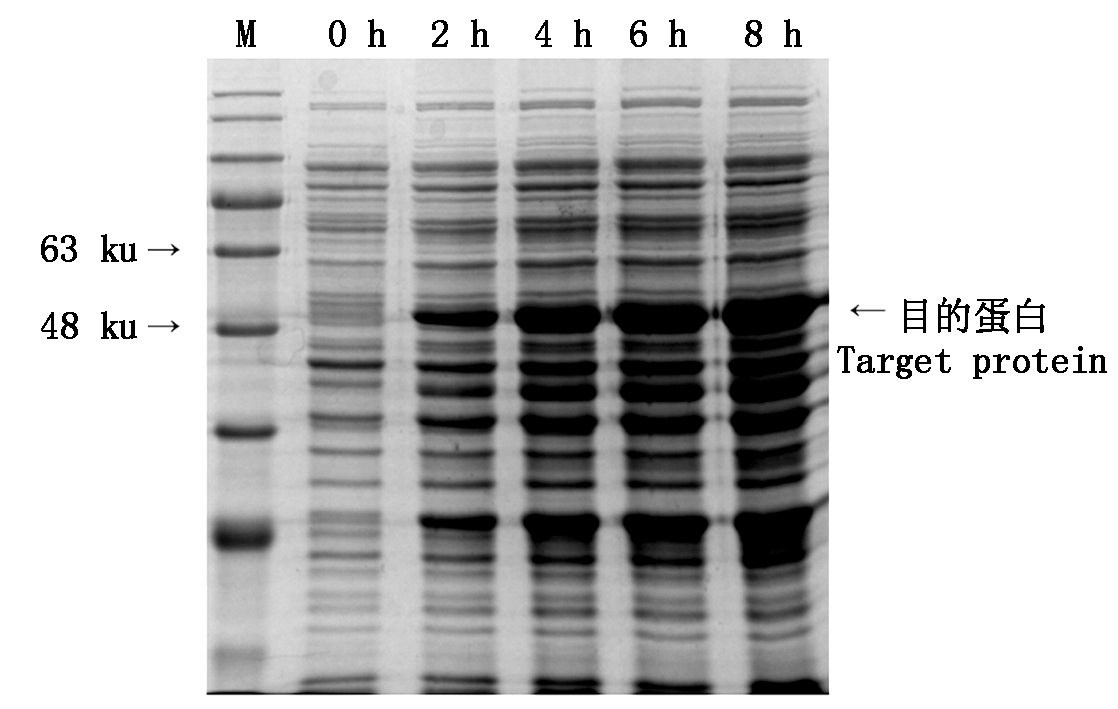
M.180 ku Marker; 0~8 h.诱导时间。
M.180 ku Marker; 0-8 h.Induction time.
图6 StTCP13-GST融合蛋白的原核表达
Fig.6 Prokaryotic expression of
StTCP13-GST tag fusion protein
2.7 转StTCP13大肠杆菌BL21盐胁迫表型分析
盐胁迫处理培养基为额外添加200,250 mmol/L NaCl的含Amp+ LB培养基,空白对照为不额外添加NaCl的含Amp+LB培养基,阴性对照为转pGEX-6P-1空载体大肠杆菌BL21,调OD600为0.03后稀释倍数1×,10×,100×,1 000×,10 000×,取3 μL在培养基上点斑,试验设置3次重复,37 ℃恒温培养箱倒置培养8 h观察表型。结果见图7,LB+200 mmol/L NaCl和LB+250 mmol/L NaCl试验组与对照组相比,表现为抑制大肠杆菌BL21生长,在LB+250 mmol/L NaCl条件下抑制更为明显;在空白对照培养基上(图7-A),转StTCP13大肠杆菌BL21与阴性对照的生长状况无显著差异;在LB+200 mmol/L NaCl和LB+250 mmol/L NaCl培养基上(图7-B、C),随稀释倍数的增加,转StTCP13大肠杆菌BL21表现为生长状况要优于对照组,当盐浓度为250 mmol/L时,表型更为明显。
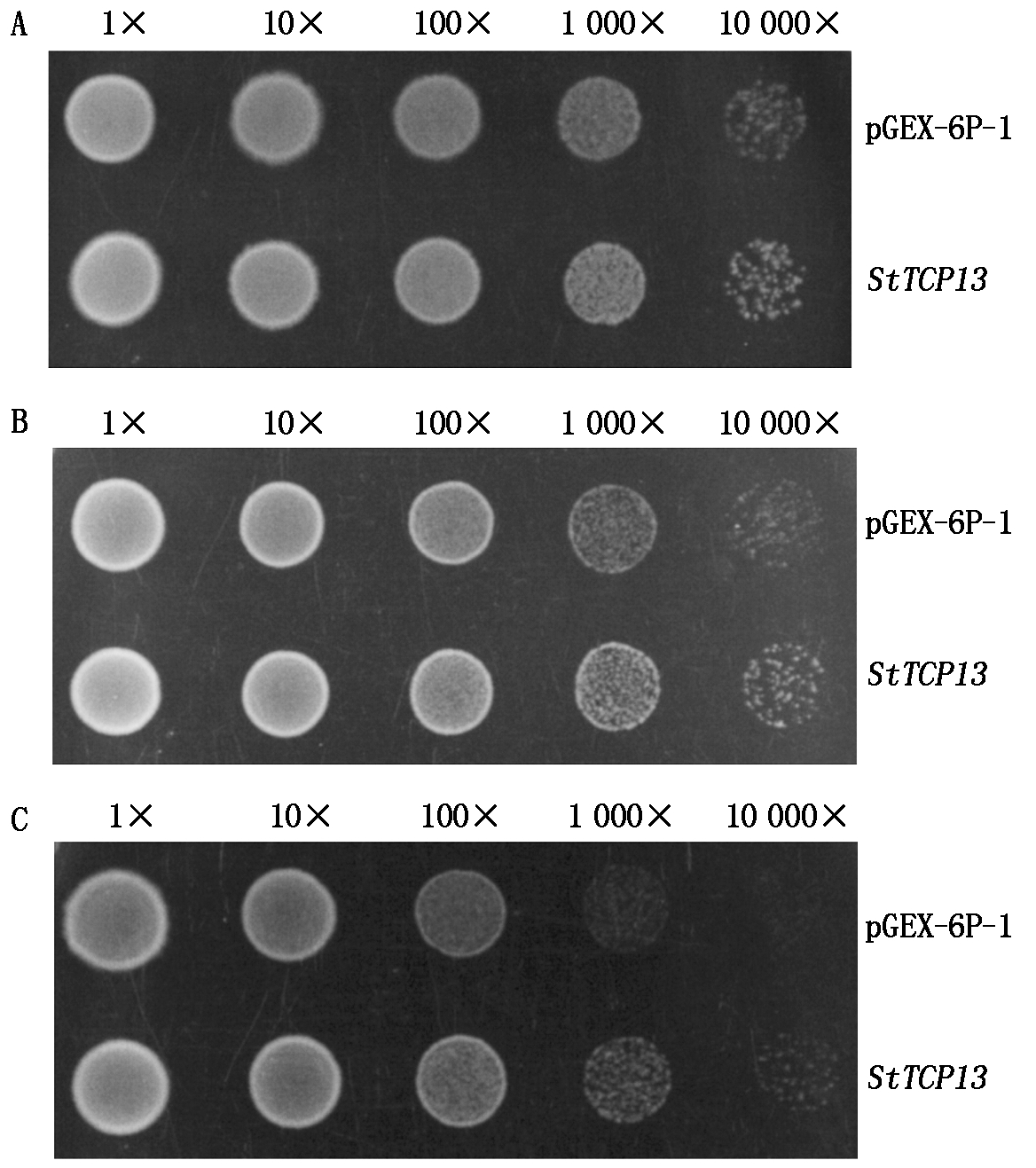
A.LB培养基; B.LB+200 mmol/L NaCl; C.LB+250 mmol/L NaCl。
A.Medium of LB; B.LB+200 mmol/L NaCl; C. LB+250 mmol/L NaCl.
图7 转StTCP13大肠杆菌BL21盐胁迫表型分析
Fig.7 Phenotypic analysis of transgenic E.coli BL21
of StTCP13 gene in salt stress
3 讨论
TCP是植物所特有的一类转录因子,调控植物细胞分裂、影响分生组织的生长[1]。近年,随着测序技术的发展,有更多植物的TCP转录因子被鉴定出来,在烟草中有61个[19],辣椒30个[20],高粱27个[21],芥菜53个[22],葡萄18个[23]。本研究从马铃薯费乌瑞它中克隆出一个TCP转录因子,依据同源性将其命名为StTCP13,该基因编码区全长669 bp,与预测结果一致,可能编码一个由222个氨基酸残基组成的蛋白质。TCP结构域为一个非典型的bHLH结构域,大约由60个氨基酸组成,根据该结构域的差异又分为classⅠ和classⅡ 2个亚家族,与classⅡ相比classⅠ的TCP结构域缺少了4个氨基酸[1,5,7]。蛋白结构域分析结果表明,StTCP13蛋白的第39-105位氨基酸为一个典型的TCP结构域,该结构域是TCP转录因子的重要功能域,识别并结合基因启动子序列[5]。进一步分析其氨基酸序列发现,该TCP结构域不存在4个氨基酸的缺失,说明StTCP13编码蛋白为TCP转录因子超家族classⅡ亚族成员。系统进化树分析结果表明,马铃薯StTCP13与潘那利番茄、甜椒、烟草等TCP13蛋白在同一进化分支上,说明TCP13蛋白在物种亲缘关系上的保守性,同时也暗示了其功能相似性。
研究表明,番茄SlTCP7基因在根、茎、叶、花、果实等组织中均有表达,但表达丰度存在差异,该基因在番茄茎中表达量最高[24]。在对海岛棉TCP转录因子的研究中发现,海岛棉GbTCP14和GbTCP27基因存在组织表达特异性,在花瓣和花萼中表达量较高,并且在开花后5~20 d其表达量存在差异,该时期为棉纤维伸长分化的关键期,表明该基因与棉花纤维伸长分化有关[25-26]。还有研究发现,马铃薯StBRC1a属CYC/TB1类TCP转录因子,该基因在腋芽部位特异高表达,在纯合突变体中植株表现为分枝增多的表型[27]。组织表达模式结果与前人研究一致,StTCP13基因具有组织表达特异性,在马铃薯根、茎、叶、芽、芽眼中的相对表达丰度有显著差异,主要在根、叶片和芽中表达,而在块茎愈伤后的芽眼中相对表达量极低,表明该基因可能与马铃薯根系、叶片和芽生长过程相关。TCP转录因子主要参与调控植物生长和信号响应等多种生理生化过程,包括植物对非生物胁迫逆境的响应。在马铃薯TCP转录因子家族的鉴定中发现,200 mmol/L NaCl处理条件下StTCP20、StTC21等基因表达量显著上调[18]。番茄SlTCP7基因在100 mmol/L NaCl处理3 h后,表达量显著上升,随后下降并维持在一个相对稳定水平,表明该基因可能参与番茄盐胁迫响应过程[24]。还有研究表明,大豆TCPs基因能响应热、干旱、高盐、ABA等非生物胁迫[28]。相关研究表明,TCP转录因子的表达与盐胁迫等逆境有关,基于FPKM值分析马铃薯StTCP13胁迫表达模式发现,该基因可能响应高盐、ABA、干旱等非生物胁迫。
为了进一步探究StTCP13基因响应盐胁迫逆境的功能,本研究构建了StTCP13-GST标签融合蛋白的pGEX-6P-1原核表达载体,将重组载体导入大肠杆菌BL21中,并成功诱导表达了StTCP13-GST融合蛋白。SDS-PAGE电泳结果表明,在诱导8 h后StTCP13蛋白表达量较高。转StTCP13大肠杆菌BL21盐胁迫表型分析结果表明,在LB+200 mmol/L NaCl和LB+250 mmol/L NaCl盐胁迫条件下,大肠杆菌的生长受到一定抑制,而导入了StTCP13基因的大肠杆菌生长状况要优于对照,结果表明,高盐胁迫可能诱导StTCP13的表达,其表达能一定程度上提高大肠杆菌对盐胁迫的耐受性,说明StTCP13在响应盐胁迫逆境中发挥一定功能。
从马铃薯品种费乌瑞它中克隆得到StTCP13基因,成功构建了StTCP13-GST标签融合蛋白的pGEX-6P-1原核表达载体,并在大肠杆菌BL21诱导表达了目的蛋白。该基因CDS区全长669 bp,编码由222个氨基酸组成的蛋白质,StTCP13-GST融合蛋白大小约为51 ku。蛋白结构域分析结果表明,StTCP13蛋白属于TCP超家族中classⅡ亚族成员,系统进化树表明StTCP13蛋白与潘那利番茄SpTCP13和甜椒CaTCP13同源性较高。表达分析结果显示,马铃薯StTCP13基因在根中相对表达量最高,在芽眼中相对表达量最低,并且其表达受盐胁迫诱导。转StTCP13在一定程度上提高了大肠杆菌BL21对高盐胁迫的耐受性。
[1] Cubas P, Lauter N, Doebley J, Coen E. The TCP domain: a motif found in proteins regulating plant growth and development[J]. The Plant Journal, 1999, 18(2):215-222. doi: 10.1046/j.1365-313x.1999.00444.x.
[2] Doebley J, Stec A, Gustus C. Teosinte branched1 and the origin of maize: evidence for epistasis and the evolution of dominance[J]. Genetics, 1995, 141(1):333-346.
[3] Doebley J, Stec A, Hubbard L. The evolution of apical dominance in maize[J]. Nature, 1997, 386(6624):485-488. doi: 10.1038/386485a0.
[4] Carpenter R, Coen E S. Floral homeotic mutations produced by transposon-mutagenesis in Antirrhinum majus[J]. Genes Development, 1990, 4(9):1483-1493.doi: 10.1101/gad.4.9.1483.
[5] Kosugi S, Ohashi Y. PCF1 and PCF2 specifically bind to cis elements in the rice proliferating cell nuclear antigen gene[J]. The Plant Cell, 1997, 9(9):1607-1619.doi: 10.2307/3870447.
[6] 冯志娟, 徐盛春, 刘娜, 张古文, 胡齐赞, 龚亚明. 植物TCP转录因子的作用机理及其应用研究进展[J]. 植物遗传资源学报, 2018, 19(1):112-121.doi: 10.13430/j.cnki.jpgr.2018.01.013.
Feng Z J, Xu S C, Liu N, Zhang G W, Hu Q Z, Gong Y M.Molecular mechanisms and applications of TCP transcription factors in plants[J].Journal of Plant Genetic Resources, 2018, 19(1):112-121.
[7] Kosugi S, Ohashi Y. DNA binding and dimerization specificity and potential targets for the TCP protein family[J]. The Plant Journal, 2002, 30(3):337-348.doi: 10.1046/j.1365-313x.2002.01294.x.
[8] Aguilar-Martínez J A, Poza-Carri n C, Cubas P. Arabidopsis BRANCHED1 acts as an integrator of branching signals within axillary buds[J]. The Plant Cell, 2007, 19(2):458-472.doi:10.1105/tpc.106.048934.
n C, Cubas P. Arabidopsis BRANCHED1 acts as an integrator of branching signals within axillary buds[J]. The Plant Cell, 2007, 19(2):458-472.doi:10.1105/tpc.106.048934.
[9] Seale M, Bennett T, Leyser O.BRC1 expression regulates bud activation potential but is not necessary or sufficient for bud growth inhibition in Arabidopsis[J]. Development, 2017, 144(9):1661-1673.doi:10.1242/dev.145649.
[10] 杨懿, 杨宇杰, 袁存权, 张启翔. 菊花CmTCP7基因的克隆及表达分析[J]. 西北植物学报, 2019,39(4):595-602.doi: 10.7606/j.issn.1000-4025.2019.04.0595.
Yang Y, Yang Y J, Yuan C Q, Zhang Q X. Cloning and expression analysis of CmTCP7 in Chrysanthemum morifolium[J]. Acta Botanica Boreali-Occidentalia Sinica, 2019,39(4):595-602.
[11] Takeda T, Suwa Y, Suzuki M, Kitano H, Ueguchi-Tanaka M, Ashikari M, Matsuoka M, Ueguchi C. The OsTB1 gene negatively regulates lateral branching in rice[J]. The Plant Journal, 2003, 33(3):513-520. doi: 10.1046/j.1365-313X.2003.01648.x.
[12] Choi M S, Woo M O, Koh E B, Lee J, Ham T H, Seo H S, Koh H J. Teosinte Branched 1 modulates tillering in rice plants[J]. Plant Cell Reports, 2012, 31(1):57-65.doi: 10.1007/s00299-011-1139-2.
[13] Nath U, Crawford B C W, Carpenter R, Coen E. Genetic control of surface curvature[J]. Science, 2003, 299(5611):1404-1407.doi: 10.1126/science.1079354.
[14] Palatnik J F, Allen E, Wu X L, Schommer C, Schwab R, Carrington J C, Weigel D. Control of leaf morphogenesis by microRNAs[J]. Nature, 2003, 425(6955):257-263.doi: 10.1038/nature01958.
[15] Resentini F, Felipo-Benavent A, Colombo L, Blázquez M A, Alabadi D, Masiero S. TCP14 and TCP15 mediate the promotion of seed germination by gibberellins in Arabidopsis thaliana[J]. Molecular Plant, 2015, 8(3):482-485.doi: 10.1016/j.molp.2014.11.018.
[16] ![]() E, Büyük
E, Büyük ![]() B. Transcriptome-scale characterization of salt responsive bean TCP transcription factors[J]. Gene, 2018, 642(9):64-73.doi:10.1016/j.gene.2017.11.021.
B. Transcriptome-scale characterization of salt responsive bean TCP transcription factors[J]. Gene, 2018, 642(9):64-73.doi:10.1016/j.gene.2017.11.021.
[17] 张园, 李芳蕊, 李洋, 许思佳, 安琳君, 李慧玉. 转BpTCP7白桦耐盐碱能力分析[J]. 福建林业科技,2019, 46(1):12-15, 61.doi:10.13428/j.cnki.fjlk.2019.01.003.
Zhang Y, Li F R, Li Y, Xu S J, An L J, Li H Y. Analysis of salt-alkali tolerance of BpTCP7 transgenic birch[J]. Journal of Fujian Forestry Science and Technology, 2019, 46(1):12-15, 61.
[18] Wang Y P, Zhang N, Li T, Yang J W, Zhu X, Fang C X, Li S G, Si H J.Genome-wide identification and expression analysis of StTCP transcription factors of potato (Solanum tuberosum L.)[J]. Computational Biology and Chemistry, 2019, 78:53-63.doi:10.1016/j.compbiolchem.2018.11.009.
[19] Chen L, Chen Y Q, Ding A M, Chen H, Xia F, Wang W F, Sun Y H. Genome-wide analysis of TCP family in tobacco[J]. Genetics and Molecular Research, 2016, 15(2):1-14.doi: 10.4238/gmr.15027728.
[20] 冯羽娥. 辣椒全基因组TCP转录因子家族生物信息学分析[J]. 种子科技, 2017, 35(8):152-154. doi:10.3969/j.issn.1005-2690.2017.08.104.
Feng Y E. Genome-wide analysis of TCP transcription factorsin pepper[J]. Seed Science & Technology, 2017, 35(8):152-154.
[21] 郑玲, 白雪婷, 李会云. 高粱TCP基因家族全基因组鉴定及表达分析[J]. 河南农业科学, 2019, 48(10):30-36.doi: 10.15933/j.cnki.1004-3268.2019.10.005.
Zheng L, Bai X T, Li H Y. Genome-wide identification and expression analysis of TCP gene family in Sorghum bicolor L.[J]. Journal of Henan Agricultural Sciences, 2019, 48(10):30-36.
[22] 李坤杰, 谭杉杉, 孙勃, 饶丹, 何琦, 闵爱玲, 李梦瑶. 芥菜TCP转录因子家族全基因组鉴定及表达分析[J]. 四川农业大学学报, 2019, 37(4):459-468.doi: 10.16036/j.issn.1000-2650.2019.04.005.
Li K J, Tan B B, Sun B, Rao D, He Q, Min A L, Li M Y.Genome-wide identification and analysis of TCP transcription factor family in Brassica juncea[J]. Journal of Sichuan Agricultural University,2019, 37(4):459-468.
[23] Leng X P, Wei H R, Xu X Z, Ghuge S A, Jia D J, Liu G S, Wang Y Z, Yuan Y B. Genome-wide identification and transcript analysis of TCP transcription factors in grapevine[J]. BMC Genomics, 2019, 20(1):786.doi:10.1186/s12864-019-6159-2.
[24] 雷豆, 苏周, 吴雨, 韦小英, 何卓远, 邹建, 杨军. 番茄SlTCP7转录因子的克隆及表达分析[J]. 分子植物育种, 2019, 17(5):1446-1453.doi:10.13271/j.mpb.017.001446.
Lei D, Su Z, Wu Y, Wei X Y, He Z Y, Zou J, Yang J. Cloning and expression analysis of a transcription factor SlTCP7 in tomato[J]. Molecular Plant Breeding, 2019,17(5):1446-1453.
[25] 蔡永生, 郑凯, 王怡, 贺宏伟, 曲延英, 倪志勇, 陈家全. 海岛棉GbTCP14基因的克隆及表达分析[J]. 西北植物学报, 2018, 38(3):401-408.doi:10.7606/j.issn.1000-4025.2018.03.0401.
Cai Y S, Zheng K, Wang Y, He H W, Qu Y Y, Ni Z Y, Chen J Q. Cloning and expression analysis of GbTCP14 gene in Gossypium barbadense[J]. Acta Botanica Boreali-Occidentalia Sinica, 2018, 38(3):401-408.
[26] 郑凯, 曲延英, 倪志勇, 蔡永生, 石颖颖, 陈全家.海岛棉转录因子基因GbTCP27的克隆及表达特性分析[J]. 中国农业大学学报, 2020, 25(2):24-33.doi:10.11841/j.issn.1007-4333.2020.02.03.
Zheng K, Qu Y Y, Ni Z Y, Cai Y S, Shi Y Y, Chen Q J. Cloning and expression characterization analysis of the Gossypium barbadense transcription factor gene GbTCP27[J]. Journal of China Agricultural University, 2020, 25(2):24-33.
[27] 冯爽爽, 罗嘉翼, 朱曦鉴, 蒋继滨, 黄三文, 张金喆. 二倍体马铃薯StBRC1a功能缺失突变体的获得及其功能分析[J]. 园艺学报, 2020, 47(1):63-72.doi: 10.16420/j.issn.0513-353x.2019-0275.
Feng S S, Luo J Y, Zhu X J, Jiang J B, Huang S W, Zhang J Z. Homozygous mutant construction and function analysis of TCP transcription factor StBRC1a in diploid potato[J]. Acta Horticulturae Sinica, 2020, 47(1):63-72.
[28] Feng Z J, Xu S C, Liu N, Zhang G W, Hu Q Z, Gong Y M. Soybean TCP transcription factors: evolution, classification, protein interaction and stress and hormone responsiveness[J].Plant Physiology and Biochemistry, 2018, 127:129-142.doi: 10.1016/j.plaphy.2018.03.020.