DOF转录因子家族是植物特有的转录因子家族,参与植物生长发育等诸多生物过程,诸如生长代谢调控、植物激素反应、光周期形态建成以及逆境胁迫等诸多方面。一些研究表明,DOF蛋白可作为转录激活因子或抑制因子在调控下游基因的表达方面发挥着作用[1]。第一个DOF基因是在玉米中发现的,在其基序中含有保守的DNA结合DOF结构域[2]。DOF结构域一般由50~52个氨基酸和含有4个半胱氨酸的锌指结构构成, 这个保守的结构域可以特异的结合核心序列为5′-T/AAAG-3′的顺式元件。DOF保守结构域一般位于DOF蛋白的N端, 和其他的调控蛋白结合形成复合体,共同调控下游基因的表达[3]。DOF基因在植物的生长发育进程中发挥着多方面的作用,诸如植物对逆境胁迫的防御[4-8]、种子储藏蛋白的合成 [9-10]、激素反应[11-12]、碳水化合物代谢[13]、叶片保卫细胞的调控[14-15]、种子萌发[16-17]、光周期调控开花[18-19]等。
近些年来,DOF基因家族在不同植物中的分布进行了广泛的研究。从低等单细胞藻类、苔藓植物[20]到高等植物拟南芥和水稻[21]、大豆[22]、西红柿[23]、玉米[24]、木薯[25]、蓖麻[9]、西瓜[5]等植物中进行了大量的研究。玉米是世界上最重要的作物之一,是粮经饲综合利用的大宗作物,为世界的粮食安全发挥着基石作用。有关DOF基因在玉米的生长发育进程中发挥的生物学功能报道的还不多。ZmDOF1是第一个发现的DOF基因,在花粉粒中有较高的表达水平[26]。同时玉米DOF1和DOF2参与调控一系列下游基因的表达,共同参与调控玉米碳氮代谢的平衡[27-28]。 逆境胁迫例如盐或干旱胁迫是影响玉米生长以及产量的重要影响因子之一。植物也进化了一套精细的分子生理调节机制来适应逆境胁迫环境。本研究选用10个玉米DOF基因作为研究对象,利用定量PCR技术,分析了其在玉米不同组织中以及应答不同非生物胁迫条件下的表达模式,以期为进一步研究其生物学功能提供有价值的信息。
1 材料和方法
1.1 植物材料
本研究中的系列试验材料为玉米自交系B73,为了探测玉米ZmDOF基因在不同组织器官中的表达模式,B73自交系种植于中国热带农业科学院玉米育种基地(广东湛江),生长至扬花授粉期,采集玉米的根系、茎、叶、雄花、授粉14 d的幼穗和苞叶等组织器官,液氮冷冻后,置于-80 ℃冰箱保存。对于胁迫试验,B73的种子经过5%次氯酸钠溶液消毒后,置于黑暗环境下萌发,将发芽一致的种子转移到营养液中进行后续的各种试验,营养液的配制以及各种胁迫处理试验见前人的报告[29]。为了分析这10个基因对硝态氮或铵态氮饥饿的响应模式,将生长在含2 mmol/L KNO3或者NH4Cl营养液中生长14 d的玉米苗转移到含0.02 mmol/L的KNO3或者NH4Cl营养液中,处理0,1,6,24 h后分别取玉米叶片,低氮冷冻,-80 ℃冰箱保存。为了分析10个ZmDOF基因应答干旱和盐胁迫的响应表达模式,将生长14 d长势一致的玉米苗转移到20%的PEG6000溶液和200 mmol/L的NaCl溶液中,分别在0,1,6,24 h 分别取玉米叶片,低氮冷冻,-80 ℃冰箱保存。每个取样时间点设置3个重复。
1.2 RNA提取与定量PCR
取0.1 g嫩叶组织,利用RNeasy FFPE Kit提取试剂盒(217004-50,GIAGEN,德国)提取组织总RNA,RNA浓度与纯度用NanoDrop2000 (ThermoScientific, Massachusetts, 美国)进行测定。RNA提取的完整度用1% (m/V)琼脂胶进行验证。5 μg总RNA用M-MLV反转录酶(TaKaRa Bio Inc., Otsu, 日本)进行反转录生成cDNA。实时荧光定量PCR反应用的是罗氏LightCycler 480 实时定量 PCR仪以及2-ΔΔCT算法。采用Oligo7设计引物,玉米Actin引物用作内参基因,引物序列见表1。基因的相对表达水平用ZmDOF/Actin来表示。每个PCR数据重复4次并取其平均值。
表1 10个ZmDOF基因的实时荧光定量引物
Tab.1 qRT-PCR primers for expression on analysis of ZmDOF

基因名称Gene name基因位点Gene locus上游引物(5′-3′) Forward 下游引物(5′-3′) Reverse ZmDOF3Zm00001d029512ATCCAATGCCGTTTCAGCTCCAGTCCCCGCCTCTTTGTTCTCGTCZmDOF13Zm00001d005100GCTGCGACTCCAACAACACCAAGGTTTGTTCTTGCGACACCCACCACZmDOF20Zm00001d042736GACCAGAACTGCTTCGACTCGCTAGGCTGAGGGCGTCATCTGGGAACZmDOF23Zm00001d051439GCTCTTCCCCTTCGAGGACTTGTGCTGCCATTTCCGATCAAGCTAZmDOF24Zm00001d051577CCAAAACCCGCCTGCTAATCACTCCGCAGCGGCAAACAAACCGGAAGZmDOF31Zm00001d017575TCTTGGATACCGCCATGGCGAACAGTCCCCCCAGCTCAAAGCCAATGZmDOF33Zm00001d017788AAGGCAAAGGCACCGGCAGAGCAGATCGACGAGCCACACGCAAAAZmDOF35Zm00001d038281GCTGCTACGACTCTCTGGGTTTGCGTCCTCAGGTAGCGGATCGAACZmDOF43Zm00001d012280GAGTTCAACTTCGCCAAGGACCAGGAACGCCACGACGACGAAGAGAGZmDOF46Zm00001d026096GCGGTAAACATAAGCGAAGCGGTGCGGCGGGCGAATGGTAGATGAC
1.3 ZmDOFs鉴定、序列比对及进化树分析
10个玉米DOF基因和蛋白序列及染色体位置等信息来自于Phytozome在线数据资源(https://jgi.doe.gov/data-and-tools/phytozome/),拟南芥相关的信息来源于Tair在线数据资源(https://www.arabidopsis.org/)。对于进化树分析,10个ZmDOF蛋白序列和36个拟南芥的AtDOFs蛋白序列构成一个数据矩阵,利用MAFFT在线软件中的L-INS-i算法进行序列比对 (https://mafft.cbrc.jp/alignment/software/),去除不保守的序列后,利用MEGA软件中的NJ算法(http://www.magasoftware.net/),参数设定为P-distance, Pairwise deletion of gaps, Bootstrap值设定为1 000,进行NJ进化树的构建。
1.4 ZmDOFs染色体定位,基因结构及保守结构域分析
根据ZmDOFs基因在玉米基因组中的物理位置,用MapInspect软件绘制ZmDOFs在染色体中的位置(http://www.plantbreeding.wur.nl/uk/software-mapinspect.html)。ZmDOFs的外显子-内含子基因结构通过比较cDNA序列与对应的基因组序列,利用在线网站GSDS (http://gsds.cbi.pku.edu.cn)进行比对绘制。保守结构域分析利用在线软件MEME分析(http://meme-suite.org/tools/meme)。
2 结果与分析
2.1 10个玉米转录因子ZmDOFs的生物信息学分析
10个ZmDOF蛋白序列从Phytozome在线网站下载,其对应蛋白的长度为297(ZmDOF24)~420 aa (ZmDOF46),预测的分子质量和等电点分别为31.44(ZmDOF24)~42.10 ku(ZmDOF46)和4.57 (ZmDOF43)~9.63(ZmDOF13)(表2)。根据这10个DOF基因在染色体上的位置,利用软件MapInspect软件进行了染色体定位,结果显示,这10个ZmDOF基因位于Chr1~Chr6以及Chr8和Chr10染色体上,其中Chr1, Chr2, Chr3, Chr6, Chr8和Chr10各有1个ZmDOF基因,Chr4和Chr5上各分布有2个ZmDOF基因 (图1-A)。这10个ZmDOF蛋白序列的N端都包含保守的DOF结构域,由大约52个氨基酸组成的C2-C2锌指结构构成(图1-B)。通过比对这10个ZmDOF基因的CDS序列,绘制了基因结构,结果显示,这10个ZmDOF基因没有或只有一个内含子,4个ZmDOF基因 (ZmDOF20、ZmDOF31、ZmDOF35和ZmDOF43)没有内含子,其余的只含有1个内含子 (图1-C)。进化树分析结果表明,这10个ZmDOF基因分属于不同的亚家族,亚家族Ⅰ包含ZmDOF23、ZmDOF31和ZmDOF46 3个基因,亚家族Ⅲ包含ZmDOF20、ZmDOF35和ZmDOF43 3个基因,亚家族Ⅳ包含有另外4个基因 (图1-D),表明这3个亚家族基因在玉米的历史进化进程中很早就发生分离,其所发挥的生物学功能各异。
2.2 10个ZmDOF蛋白保守功能基序分析
利用在线的MEME软件进行功能基序分析,共探测到6个保守的功能基序 (图2-A)。其中Motif1预测为保守的DOF结构域,分布于全部的10个ZmDOF中。其他的功能基序散布于不同的蛋白中,8个ZmDOF蛋白含有Motif3基序,7个ZmDOF蛋白的N端含有Motif2基序,5个ZmDOF蛋白含有Motif5基序,3个ZmDOF蛋白含有Motif6,而ZmDOF24和ZmDOF33蛋白的C端分别含有Motif4基序(图2-B)。
表2 10个ZmDOF的生物信息学分析
Tab.2 The bioinformatic analysis of 10 ZmDOFs

基因位点Gene locus基因名称Gene name大小/aaSize分子量/kuMolecular mass等电点PI染色体位置Chromosome location剪切体Splice variantsZm00001d029512ZmDOF335136.734.80Chr1:73971569-739739651Zm00001d005100ZmDOF1332535.059.63Chr2:154144472-1541480291Zm00001d042736ZmDOF2033434.944.59Chr3:176010296-1760117261Zm00001d051439ZmDOF2336737.718.86Chr4:155035798-1550381401Zm00001d051577ZmDOF2429731.448.19Chr4:160304743-1603078852Zm00001d017575ZmDOF3135936.218.55Chr5:200157732-2001599351Zm00001d017788ZmDOF3328830.438.03Chr5:201437666-2014417631Zm00001d038281ZmDOF3532734.615.13Chr6:149318311-1493197451Zm00001d012280ZmDOF4333836.144.57Chr8:166129975-1661313711Zm00001d026096ZmDOF4642042.108.45Chr10:137230411-1372320132
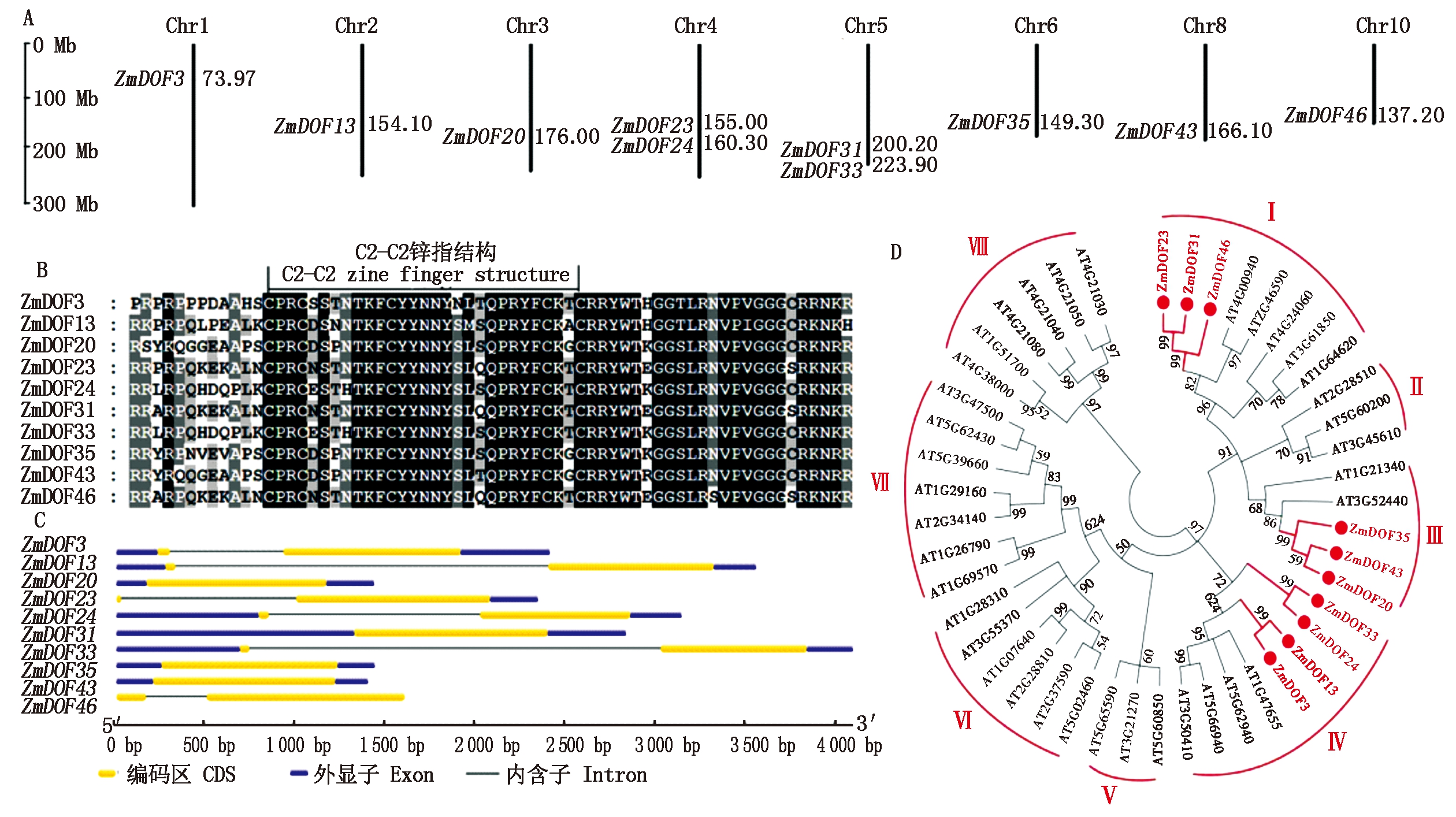
A.10个ZmDOF基因的染色体定位;B.保守的DOF结构域序列分析;C.10个ZmDOF的基因结构分析;D.10个ZmDOF蛋白的进化树分析。
A.Chromosomal localization of 10 ZmDOF genes; B.Multiple sequence alignment of conserved DOF domain;
C.Gene structure analysis of 10 ZmDOF genes; D.The phylogenetic tree analysis of 10 ZmDOF proteins.
图1 10个ZmDOF基因的生物信息学分析
Fig.1 The bioinformatics analysis of 10 ZmDOFs
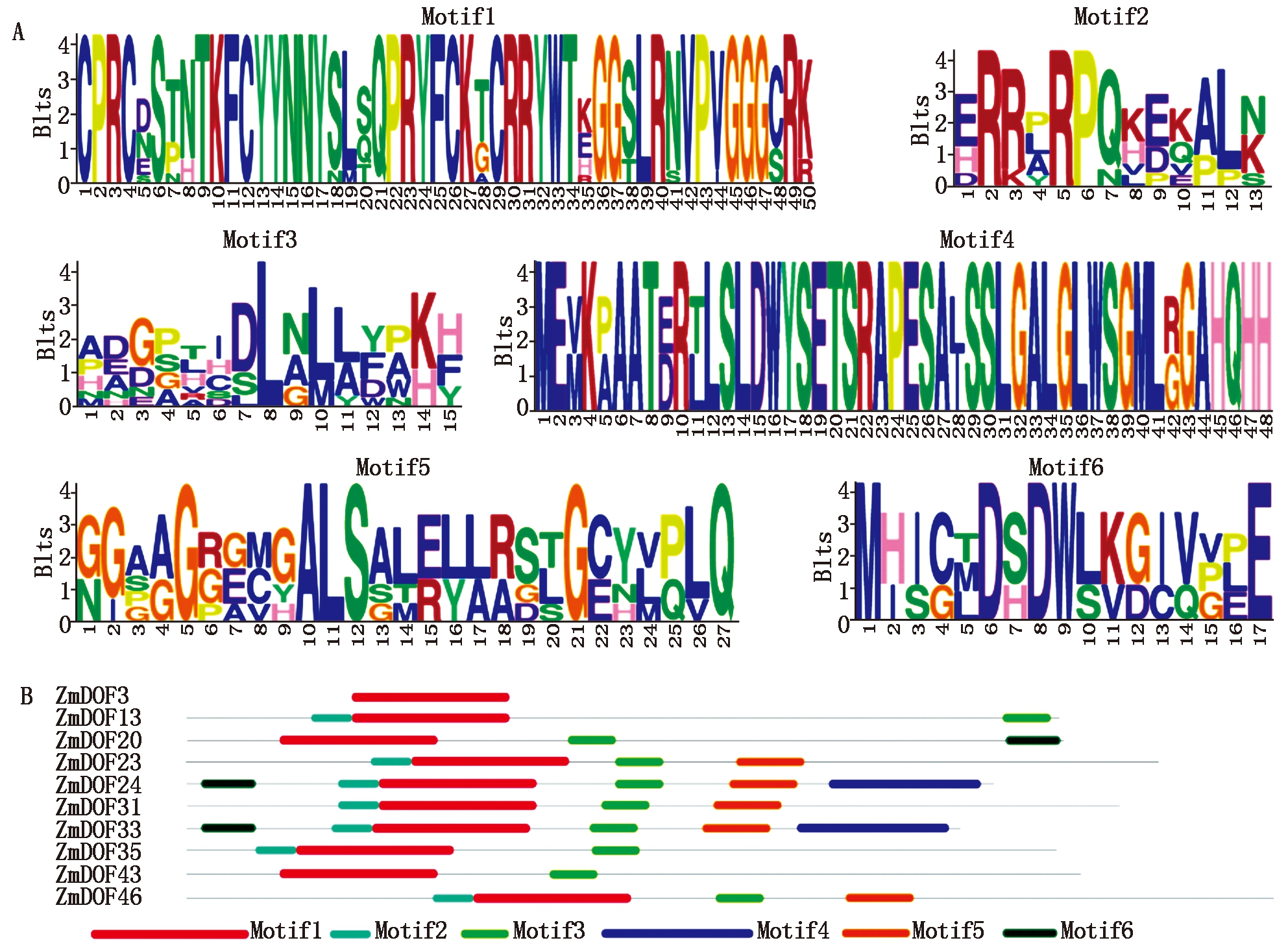
A.10个ZmDOF蛋白的基序分析;B.10个ZmDOF蛋白的基序分布示意图。
A.Motif analysis of 10 ZmDOF proteins; B.Motif distribution pattern of 10 ZmDOF proteins.
图2 10个ZmDOF基因保守结构域分析
Fig.2 Conservation domain analysis of 10 ZmDOFs
2.3 10个玉米DOF基因的组织表达分析
选取玉米B73杨花期的根部、茎部、成熟叶、雄花、授粉15 d的小穗和玉米苞叶为试验材料,利用荧光定量PCR技术,探测了ZmDOF基因在这6个器官中的表达模式,结果表明,10个ZmDOF具有明显的组织特异性表达模式 (图3)。 其中7个基因(ZmDOF20、ZmDOF23、ZmDOF24、ZmDOF31、ZmDOF33、ZmDOF35和ZmDOF43)主要在扬花期的雄花中表达,与其他组织的表达量存在显著差异,表明这7个基因可能参与玉米雄花的生长发育进程。ZmDOF31和ZmDOF33在雄花中的表达量是其他器官的1 000倍以上,表明这2个基因主要在玉米雄花中表达,可能参与玉米雄花的生长发育进程。ZmDOF20、ZmDOF24和ZmDOF35除了主要在雄花中表达外,还在玉米苞叶中表达,表明这3个基因可能参与玉米雄花和苞叶的生长发育。2个基因ZmDOF3和ZmDOF13主要在幼穗中表达,并且其表达量超过其他器官50倍以上,预示着这2个基因在玉米穗的生长发育进程中可能发挥作用。 ZmDOF46主要在玉米的根系中表达,其在根部的表达量是其他器官表达量至少3倍以上。这些结果预示着这10个ZmDOF基因在玉米的生长发育进程中发挥着重要的生物学功能。

R.根;S.茎;L.叶;CB.苞叶;T.雄花;E.幼穗。不同小写字母代表差异性显著(P<0.05)。图4-5同。
R.Root; S.Stem; L.Leaf; CB.Corn bract; T.Tassel; E.Ear.Different small letters indicates significant
difference among treatments at 0.05 level. The same as Fig.4-5.
图3 10个ZmDOF基因的组织特异性表达模式
Fig.3 The tissue expression pattern of 10 ZmDOFs
2.4 10个ZmDOF基因对PEG6000和盐胁迫响应分析
为了探测这10个ZmDOFs在干旱和盐胁迫处理下的应答模式,为这10个基因的功能研究提供有用的信息,用20%的PEG6000和200 mmol/L的NaCl溶液模拟干旱和盐胁迫处理,分别在胁迫处理的0, 1, 6,24 h取叶片,利用荧光定量PCR检测了这10个基因的表达模式。结果表明,在不同胁迫处理1 h时,大部分基因的表达都发生了明显的变化 (图4)。在NaCl胁迫处理下1 h时,ZmDOF46的表达无显著差异,表明ZmDOF46基因可能不参与玉米应答盐胁迫信号途径。ZmDOF3和ZmDOF13的表达下调,其中ZmDOF3表达下降了至少90%。ZmDOF23和ZmDOF31的表达在胁迫处理期间一直上调,其中ZmDOF31的表达上升了至少5倍以上。 在NaCl胁迫处理1 h时,有5个基因(ZmDOF20、ZmDOF24、ZmDOF33、ZmDOF35和ZmDOF43)的表达上调,与处理前相比存在显著差异,其中ZmDOF43表达上调了至少10倍以上,之后这5个基因的表达量又恢复到原来的水平。这些结果表明,有9个ZmDOF基因在应答盐胁迫时,其表达模式发生了改变,预示着这些基因参与调控玉米幼苗应答盐胁迫响应途径。在20%的PEG溶液整个胁迫处理期间,ZmDOF24的表达明显下调 (>90%),而ZmDOF43在胁迫处理早期,其表达显著下调(6 h),之后又出现明显的上调(24 h)。其他8个基因 (ZmDOF3、ZmDOF13、ZmDOF20、ZmDOF23、ZmDOF31、ZmDOF33、ZmDOF35和ZmDOF46)表达一直上调,而ZmDOF46的表达至少上升了10倍以上。这些数据表明,这10个ZmDOF基因的表达模式在干旱胁迫条件下发生了明显的改变,预示着这10个基因可能参与玉米应答干旱胁迫响应机制,结果为后续的ZmDOF基因的生物学功能研究提供了有用的线索。

图4 10个ZmDOF应答20%PEG6000溶液胁迫和200 mmol/L氯化钠胁迫表达模式分析
Fig.4 The expression pattern of 10 ZmDOFs under 200 mmol/L NaCl and 20% PEG stress
2.5 10个ZmDOF基因对不同氮形态的响应分析
为了探测这10个基因对硝态氮或铵态氮胁迫的响应模式,生长至三叶期的玉米幼苗转到硝态氮和铵态氮胁迫条件下,利用qPCR技术分析了这10个基因的表达模式(图5)。 结果表明,在硝态氮或铵态氮胁迫处理下,大部分基因的表达都受到了不同程度的影响。在铵态氮胁迫处理下,有7个基因的表达发生明显的上调,其中ZmDOF3上调的幅度超过5倍,表明这7个基因可能参与玉米幼苗应答铵态氮胁迫响应机制。在硝态氮胁迫处理下,这10个基因的表达都发生了明显的改变。ZmDOF23和ZmDOF46的表达都受到了显著的抑制,其表达量下调了至少90%。其他8个基因的表达在胁迫处理1 h时明显的上调,其中ZmDOF3、ZmDOF13、ZmDOF20、ZmDOF24、ZmDOF35和ZmDOF43的表达量上调了至少10倍。其后随着胁迫时间的延长,这些基因的表达量逐渐恢复到处理前的水平,甚至更低,表明这10个基因是玉米幼苗应答硝态氮胁迫的早期响应基因,可能参与玉米幼苗抵御硝态氮胁迫的响应途径。这些结果表明,ZmDOF基因可能参与调控玉米幼苗不同氮形态的代谢平衡或应答不同氮形态胁迫响应机制。
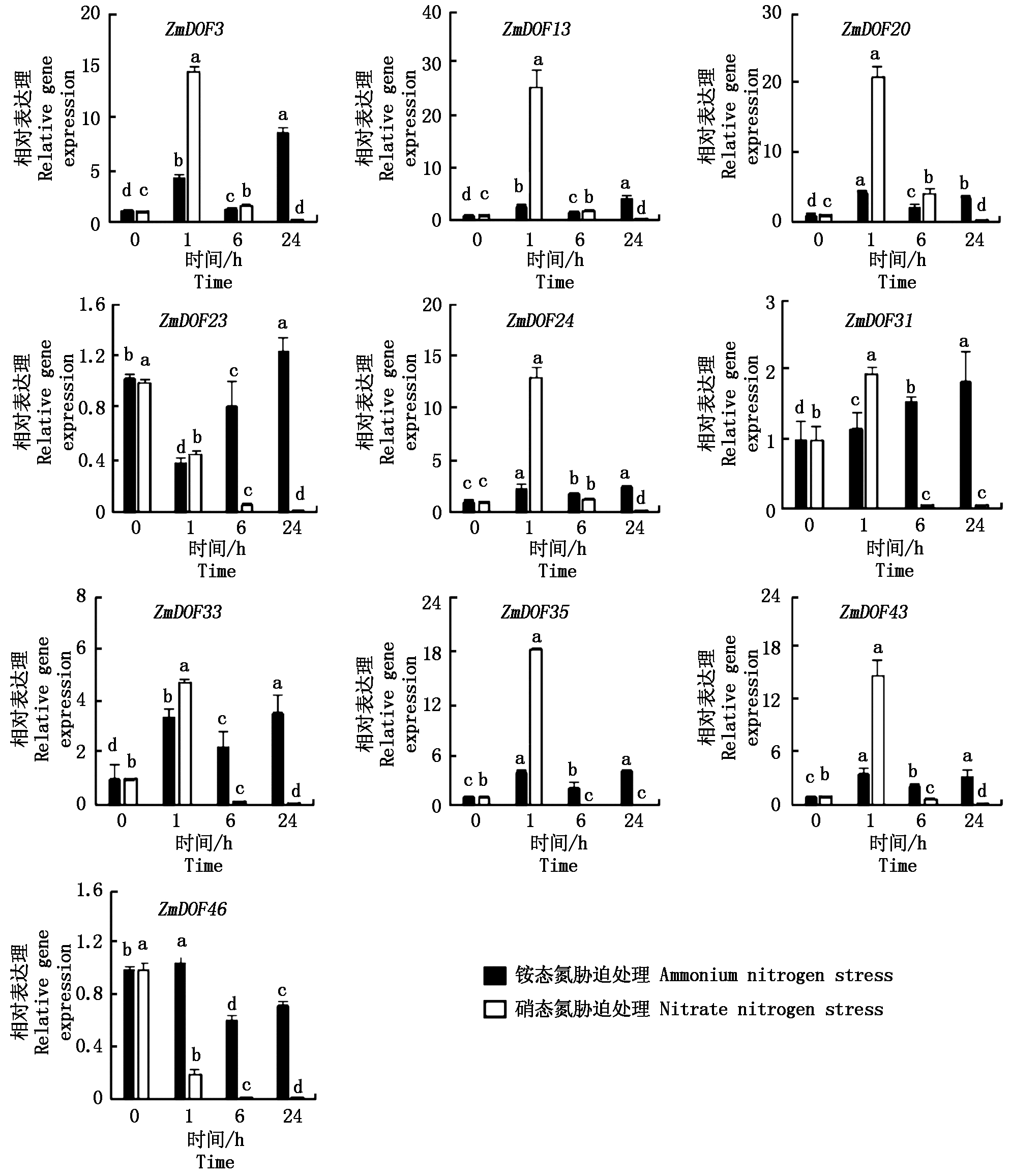
图5 10个ZmDOF基因应答不同氮形态胁迫的响应表达分析
Fig.5 The expression pattern of 10 ZmDOFs in response to nitrogen stress
3 讨论与结论
已有研究表明,植物DOF基因家族在植物的生长发育进程中发挥着重要的作用,诸如种子的生长发育、次生物质的代谢、植物分蘖、维管束的形成和花的发育等。基于基因的表达模式能为功能基因的研究提供有用的信息,利用qRT-PCR技术,探测了这10个基因在玉米6个不同器官组织的表达模式。ZmDOF3和ZmDOF13主要在玉米授粉15 d后的幼嫩的穗中表达,其表达量超过了其他器官50倍以上,表明这2个基因主要在玉米幼嫩的种子中表达。这一结果佐证了前人的研究结果[24],支持这2个基因的功能可能参与玉米幼胚、胚乳和种子的生长发育进程。已有诸多研究结果支持植物DOF基因家族在种子的生长发育进程中发挥着重要的作用[30]。同时进化树的结果表明,这2个基因聚合在一起 (Bootstrap=99%),预示着在玉米的进化历史事件进程中,这2个基因发生分离的时间不长,保留着类似的表达模式和生物学功能。在进化树分析中,聚合在一起的同源基因具有共同的祖先,具有类似的基因和蛋白结构,呈现出相似的时空表达模式和保守的生物学功能。诸如亚家族Ⅰ中的ZmDOF23和ZmDOF31聚合在一起 (Bootstrap=99%),它们的亲缘关系比ZmDOF46要近,ZmDOF23和ZmDOF31的表达模式基本一致,主要在雄花中表达,而ZmDOF46主要在根部表达。亚家族Ⅱ中的3个基因也具有类似的结果,ZmDOF20和ZmDOF43聚合在一起(Bootstrap=99%),其表达模式类似,都主要在雄花中表达,而ZmDOF35除了在雄花表达以外,在苞叶以及幼嫩的穗中也表达。众多植物DOF基因主要在植物的花器官中表达已有报道,表明植物DOF基因家族在花的生长发育进程中也发挥着重要的作用[31-32]。
非生物逆境胁迫诸如干旱和盐等会严重地影响植物正常的光合作用,以及其他重要的代谢活动,比如氮素的代谢平衡等。挖掘及鉴定植物抗逆境胁迫的基因以及阐述其分子生理机理对于保持作物在逆境胁迫下的高产和稳产,保障我国粮食安全显得尤为重要。最近的研究结果表明,调控植物开花的DOF基因家族成员,CDFs的表达受到干旱和盐胁迫的诱导。番茄基因组中编码有5个CDFs基因,SICDF1-5,这些基因的表达受到干旱和盐胁迫的强烈诱导,表明这些基因可能参与番茄抵御逆境胁迫的响应机制。在拟南芥中超表达这些基因,获得的转基因株系具有明显的抗干旱和盐胁迫的能力[31]。研究结果表明,大多数ZmDOFs基因的表达受到盐或干旱胁迫的诱导或抑制,比如在盐胁迫条件下,ZmDOF3的表达下降了90%,而ZmDOF31的表达至少上调了5倍,而在干旱胁迫条件下,ZmDOF24的表达下降90%,而ZmDOF46上调了至少10倍。研究结果预示着这10个ZmDOFs可能广泛的参与调控植物应答盐或干旱胁迫响应途径,并且各个ZmDOF所发挥的生物学功能是不同的。
维持植物体内氮素的代谢平衡对植物的生长发育至关重要。挖掘氮高效吸收利用的分子标记和基因资源,阐明其分子生理机制对于作物的高产稳产,减少资源浪费和环境污染是十分必要的。已有报道玉米DOF1和DOF2基因的表达量与水稻、高粱和小麦的氮吸收效率以及产量呈正相关。在低氮胁迫环境下,ZmDOF1和ZmDOF2通过调控超表达ZmDOF1基因能显著地促进这些作物的光合效率,提高氮素吸收,最终显著的提高这些作物的产量[33]。在低氮环境下,ZmDOF1和ZmDOF2基因通过影响C4循环中的PEPC羧化酶基因的活性,从而影响植物的碳氮代谢平衡,增进植物对氮素的吸收,促进植物生长。小麦Tadof1基因的超量表达可促进谷氨酰胺合成酶和谷氨酸酶的表达,进而影响小麦氮利用效率[27]。ZmDOF1的同源基因,水稻OsDOF18基因具有类似的功能,通过调控氨氮的运输和氮元素的分布,进而影响植物氮素的使用效率[28]。已报道的这些调控氮代谢平衡的基因都受到氨态氮胁迫的明显诱导,而结果中只有ZmDOF3受到氨态氮胁迫诱导的幅度超过5倍。大部分ZmDOF基因的表达水平受到硝态氮胁迫的抑制,至少有6个基因的表达水平下降了至少90%。这些基因在维持玉米硝态氮代谢平衡时发挥的作用还有待进一步的研究。
鉴定了10个ZmDOFs, 并对其理化性质、染色体定位、基因结构和蛋白功能结构域进行分析,利用qRT-PCR技术,检测了这10个ZmDOFs在玉米不同组织中的表达模式,以及应答盐、干旱、铵态氮和硝态氮逆境胁迫条件下的表达响应模式,为后续研究ZmDOFs在玉米生长发育进程以及抵御非生物逆境胁迫的分子生理机制提供有价值的信息。
[1] Yanagisawa S. Dof domain proteins: plant-specific transcription factors associated with diverse phenomena unique to plants[J]. Plant Cell Physiol,2004,45(4):386-391. doi: 10.1093/pcp/pch055.
[2] Yanagisawa S, Izui K. Molecular cloning of two DNA-binding proteins of maize that are structurally different but interact with the same sequence motif[J]. J Biol Chem,1993,268(21):16028-16036. doi:10.1016/S0021-9258(18)82353-5.
[3] Yang G Y, Gao X Q, Ma K H, Li D P, Jia C X, Zhai M Z, Xu Z G. The walnut transcription factor JrGRAS2 contributes to high temperature stress tolerance involving in Dof transcriptional regulation and HSP protein expression[J]. BMC Plant Biology, 2018,18(1):367-370. doi: 10.1186/s12870-018-1568-y.
[4] Qin H, Wang J, Chen X B, Wang F F, Peng P, Zhou Y, Miao Y C, Zhang Y Q, Gao Y D, Qi Y D, Zhou J H, Huang R F. Rice OsDOF15 contributes to ethylene-inhibited primary root elongation under salt stress[J]. The New Phytologist, 2019,223(2):798-813. doi: 10.1111/nph.15824.
[5] Zhou Y, Cheng Y, Wan C P, Li J W, Yang Y X, Chen J J. Genome-wide characterization and expression analysis of the Dof gene family related to abiotic stress in watermelon[J]. Peer J, 2020,8:e8358. doi: 10.7717/peerj.8358.
[6] Liu Y, Liu N N, Deng X, Liu D M, Li M F, Cui D D, Hu Y K, Yan Y M. Genome wide analysis of wheat DNA-binding with one finger (Dof) transcription factor genes: evolutionary characteristics and diverse abiotic stress responses[J]. BMC Genomics,2020,21(1):276-289. doi: 10.1186/s12864-020-6691-0.
[7] Liu J, Cheng Z C, Xie L H, Li X Y, Gao J. Multifaceted role of Phedof12-1 in the regulation of flowering time and abiotic stress responses in Moso bamboo (Phyllostachys edulis)[J]. International Journal of Molecular Sciences,2019,20(2):424-430. doi: 10.3390/ijms20020424.
[8] Ruta V, Longo C, Lepri A, De Angelis V, Occhigrossi S, Costantino P, Vittorioso P. The DOF transcription factors in seed and seedling development[J]. Plants,2019,9(2):218-228. doi: 10.3390/plants9020218.
[9] Zou Z, Zhang X C. Genome-wide identification and comparative evolutionary analysis of the Dof transcription factor family in physic nut and castor bean[J]. Peer J,2019,7(17):e6354. doi:10.7717/peerj.6354.
[10] Washio K. Identification of Dof proteins with implication in the gibberellin-regulated expression of a peptidase gene following the germination of rice grains[J]. Biochim Biophys Acta, 2001,1520(1): 54-62. doi: 10.1016/s0167-4781(01)00251-2.
[11] Mena M, Cejudo F J, Isabel-Lamoneda I, Carbonero P. A role for the DOF transcription factor BPBF in the regulation of gibberellin-responsive genes in barley aleurone[J]. Plant Physiol,2002,130(1):111-119.doi: 10.1104/pp.005561.
[12] Khaksar G, Sangchay W, Pinsorn P, Sangpong L, Sirikantaramas S. Genome-wide analysis of the Dof gene family in durian reveals fruit ripening-associated and cultivar-dependent Dof transcription factors[J]. Scientific Reports ,2019,9(1):12109-12111. doi: 10.1038/s41598-019-48601-7.
[13] Plesch G, Ehrhardt T, Mueller-Roeber B. Involvement of TAAAG elements suggests a role for Dof transcription factors in guard cell-specific gene expression[J].The Plant Journal,2001,28(4):455-464.doi: 10.1046/j.1365-313x.2001.01166.x.
[14] Negi J, Moriwaki K, Konishi M, Yokoyama R, Nakano T, Kusumi K, Hashimoto-Sugimoto M, Schroeder J I, Nishitani K, Yanagisawa S, Iba K. A Dof transcription factor, SCAP1, is essential for the development of functional stomata in Arabidopsis[J]. Curr Biol,2013,23(6):479-484. doi: 10.1016/j.cub.2013.02.001.
[15] Papi M, Sabatini S, Altamura M M, Hennig L, Schäfer E, Costantino P, Vittorioso P. Inactivation of the phloem-specific Dof zinc finger gene DAG1 affects response to light and integrity of the testa of Arabidopsis seeds[J]. Plant Physiol,2002,128(2):411-417. doi: 10.1104/pp.010488.
[16] Gualberti G, Papi M, Bellucci L, Ricci I, Bouchez D, Camilleri C, Costantino P, Vittorioso P. Mutations in the Dof zinc finger genes DAG2 and DAG1 influence with opposite effects the germination of Arabidopsis seeds[J]. The Plant Cell,2002,14(6):1253-1263. doi: 10.1105/tpc.010491.
[17] Huang Y, Han Z M, Cheng N N, Luo M F, Bai X F, Xing Y Z. Minor effects of 11 Dof family genes contribute to the missing heritability of heading date in rice (Oryza sativa L.)[J]. Frontiers in Plant Science,2020,10:1739.doi: 10.3389/fpls.2019.01739.
[18] Skirycz A, Radziejwoski A, Busch W, Hannah M A, Czeszejko J, Kwas'niewski M, Zanor M, Lohmann J U, De Veylder L, Witt I, Mueller-Roeber B. The DOF transcription factor OBP1 is involved in cell cycle regulation in Arabidopsis thaliana[J]. Plant J,2008,56(5):779-792. doi: 10.1111/j.1365-313X.2008.03641.x.
[19] Gupta S, Malviya N, Kushwaha H, Nasim J, Bisht N C, Singh V K, Yadav D. Insights into structural and functional diversity of Dof (DNA binding with one finger) transcription factor[J]. Planta,2015,241(3):549-562. doi: 10.1007/s00425-014-2239-3.
[20] Moreno-Risueno M A, Martínez M, Vicente-Carbajosa J, Carbonero P. The family of DOF transcription factors: from green unicellular algae to vascular plants[J]. Mol Genet Genomics, 2007,277(4):379-390. doi: 10.1007/s00438-006-0186-9.
[21] Lijavetzky D, Carbonero P, Vicente-Carbajosa J. Genome-wide comparative phylogenetic analysis of the rice and Arabidopsis Dof gene families[J]. BMC Evol Biol,2003,23(3):7-13. doi: 10.1186/1471-2148-3-17.
[22] Guo Y, Qiu L J. Genome-wide analysis of the Dof transcription factor gene family reveals soybean-specific duplicable and functional characteristics[J]. PLoS One,2013,8(9):e76809.doi:10.1371/journal.pone.0076809.
[23] Cai X F, Zhang Y Y, Zhang C J, Zhang T Y, Hu T X, Ye J, Zhang J H, Wang T T, Li H X, Ye Z B. Genome-wide analysis of plant-specific Dof transcription factor family in tomato[J]. J Integr Plant Biol, 2013, 55(6):552-566. doi: 10.1111/jipb.12043.
[24] Chen Y Z, Cao J. Comparative analysis of dof transcription factor family in maize[J]. Plant Mol Biol Rep,2015,33(5):1245-1258. doi: 10.1007/s11105-014-0835-9.
[25] Zou Z, Zhu J L, Zhang X C. Genome-wide identification and characterization of the Dof gene family in cassava (Manihot esculenta)[J]. Gene, 2019,687:298-307. doi: 10.1016/j.gene.2018.11.053.
[26] Chen X Y, Wang D X, Liu C, Wang M Z, Wang T, Zhao Q, Yu J J. Maize transcription factor Zmdof1 involves in the regulation of Zm401 gene[J]. Plant Growth Regulation, 2012,66(3):271-284. doi:10.1007/s10725-011-9651-5.
[27] Kumar R, Taware R, Gaur V S, Guru S K, Kumar A. Influence of nitrogen on the expression of TaDof1 transcription factor in wheat and its relationship with photosynthetic and ammonium assimilating efficiency[J]. Molecular Biology Reports,2009,36(8):209-221. doi: 10.1007/s11033-008-9436-8.
[28] Wu Y F, Yang W Z, Wei J H, Yoon H, An G. Transcription factor OsDOF18 controls ammonium uptake by inducing ammonium transporters in rice roots[J]. Molecules and Cells, 2017,40(3):178-185. doi: 10.14348/molcells.2017.2261.
[29] Jue D J, Sang X L, Lu S Q, Dong C, Zhao Q F, Chen H L, Jia L Q. Genome-wide identification, phylogenetic and expression analyses of the Ubiquitin-conjugating enzyme gene family in maize[J]. PLoS One, 2015,10(11):e0143488.doi: 10.1371/journal.pone.0143488.
[30] da Silva D C, da Silveira Falavigna V, Fasoli M, Buffon V, Porto D D, Pappas G J, Pezzotti M, Pasquali G, Revers L F. Transcriptome analyses of the Dof-like gene family in grapevine reveal its involvement in berry, flower and seed development[J]. Horticulture Research,2016,3:16042. doi: 10.1038/hortres.2016.42.
[31] Corrales A R, Carrillo L, Lasierra P, Nebauer S G, Dominguez-Figueroa J, Renau-Morata B, Pollmann S, Granell A, Molina R V, Vicente-Carbajosa J, Medina J. Multifaceted role of cycling DOF factor 3 (CDF3) in the regulation of flowering time and abiotic stress responses in Arabidopsis[J]. Plant Cell and Enviroment, 2017,40(5):748-764. doi: 10.1111/pce.12894.
[32] Corrales A R, Nebauer S G, Carrillo L, Fernández-Nohales P, Marqués J, Renau-Morata B, Granell A, Pollmann S, Vicente-Carbajosa J, Molina R V, Joaquín M. Characterization of tomato cycling dof factors reveals conserved and new functions in the control of flowering time and abiotic stress responses[J]. Journal of Experimental Botany,2014,65(4):995-1012. doi: 10.1093/jxb/ert451.
[33] Pe a P A, Quach T, Sato S, Ge Z X, Nersesian N, Changa T, Dweikat I, Soundararajan M, Clemente T E. Expression of the maize dof1 transcription factor in wheat and Sorghum[J]. Frontiers in Plant Science,2017,8:434-439. doi: 10.3389/fpls.2017.00434.
a P A, Quach T, Sato S, Ge Z X, Nersesian N, Changa T, Dweikat I, Soundararajan M, Clemente T E. Expression of the maize dof1 transcription factor in wheat and Sorghum[J]. Frontiers in Plant Science,2017,8:434-439. doi: 10.3389/fpls.2017.00434.