盐胁迫会抑制植物体内的蛋白质合成、降低光合效率、造成离子失衡以及高渗透胁迫、导致植株萎蔫和细胞凋亡[1],使作物大幅度减产,已成为影响作物生长发育的重要环境因素。近年来,伴随灌溉方式不当、过度施用化肥与工业污染等问题的出现,土壤盐渍化程度不断加重[2]。据统计,全球已有约6%的土地遭受盐害侵袭[3]。我国盐渍土总面积约为0.36亿hm2,占全国可利用土地总面积的4.88%,并以每年1%的速度增加[4]。
水稻(Oryza sativa)是世界上主要的粮食作物之一,全球有超过一半的人口以稻米为主食[5-6]。水稻是一种盐敏感作物,在遭受盐胁迫后会导致产量急剧下降[7]。因此,提高水稻耐盐性,创制耐盐性水稻种质是水稻育种的一个重要目标。近些年来,水稻中已经有很多盐胁迫相关的基因被克隆,如OsDST、OsSOS1、OsHKT、OsWRKY13、OsSNAC1、OsMYB、OsNAC等[8-14]。OsEIL1和OsEIL2基因是拟南芥EIN3在水稻中的同源基因,二者均为转录因子[15]。Yang等[16]研究表明,OsEIL1基因突变会导致水稻根生长对乙烯钝感,OsEIL2基因突变后水稻胚芽鞘生长对乙烯钝感,此外,OsEIL1和OsEIL2基因会结合OsHKT2;1基因启动子的EBS(EIN3-bindingsites)区域,促进OsHKT2;1基因的转录,OsEIL1和OsEIL2基因突变后会导致OsHKT2;1基因表达量下调,从而减少盐胁迫下根对钠离子的吸收,进而增强水稻的耐盐性。
CRISPR/Cas9技术是继ZFN技术和TALEN技术之后的第三代基因编辑技术[17-19]。CRISPR/Cas9系统由sgRNA(Single guide RNA)和Cas9(CRISPR associated protein 9)核酸酶2个部分构成,其中,sgRNA是一个具有颈环结构的单链RNA,起识别靶序列和连接Cas9核酸酶的作用,Cas9是一种限制性核酸内切酶,具有RuvC和HNH 2个核酸酶结构域,分别起到切割DNA正反双链的作用。Cas9核酸酶的识别位点被称为PAM(Protospacer adjacent motif),序列为NGG,Cas9在识别到PAM位点之后会在其上游第3或第4个碱基处造成DNA双链断裂[20],DNA双链断裂后会触发生物体的同源重组(HDR,Homologous direct recombination)修复和非同源末端连接修复(NHEJ,Nonhomologous end joining),其中,同源重组修复会导致碱基的插入或替换,非同源末端连接修复会导致碱基的插入或缺失,从而达到基因敲除的目的[21]。
目前,CRIPSR/Cas9技术已经在多种作物中进行应用,如水稻[22-23]、小麦[24-25]、大豆[26-27]、玉米[28-29]、高粱[30]等。在水稻中,Wang等[31]利用CRISPR/Cas9技术对水稻3个减数分裂相关基因:OsREC8、OsPAIR1、OsOSD1和一个单倍体诱导基因OsMTL进行敲除并获得了水稻无性繁殖的后代,实现了杂交水稻F1杂合性状的固定。Tang等[32]利用CRISPR/Cas9技术敲除水稻中镉离子运输基因OsNramp5降低了水稻镉离子含量。Zhang等[33]利用CRISPR/Cas9技术敲除水稻OsRR22基因获得了耐盐性增强的水稻突变体。
本研究以粳稻品种东农427为研究材料,利用CRISPR/Cas9技术同时对耐盐负调控基因OsEIL1和OsEIL2进行敲除以期获得耐盐性提高的水稻突变体材料,为粳稻耐盐性种质资源改良提供理论指导。
1 材料和方法
1.1 试验材料
本研究以粳稻品种东农427为试验材料,试验所用菌株大肠杆菌(Escherichia coli)感受态Mach1-T1和农杆菌(Agrobacterium tumefaciens)感受态EHA105均购自上海唯地生物技术有限公司,PrimerStarGXL、BsaⅠ核酸内切酶购自NEB公司,T4 DNA ligase 购自宝生物(大连)有限公司。试验所用载体pYL-U3-gRNA、pYL-U6a-gRNA和pYLCRISPR/Cas9-MTmono双元载体由华南农业大学刘耀光院士惠赠。
1.2 敲除靶点设计及载体构建
靶点设计:在RAP(https://rapdb.dna.affrc.go.jp/index.html)数据库下载OsEIL1(Os03g0324300)和OsEIL2(Os07g0685700)基因序列,序列比对发现OsEIL1和OsEIL2的CDS序列存在较高的相似度,因此,利用CRISPR-GE在线工具包(http://skl.scau.edu.cn)[34]在OsEIL1和OsEIL2基因靠近5′端的高度同源区域设计一个敲除靶点,保存接头引物序列。本研究中使用的所有引物均由哈尔滨博仕生物技术有限公司合成(表1)。
参照Ma等[35]的试验方法进行载体构建。
接头制备:将合成好的接头引物原液(100 μmoL/L)各取1 μL混合,加98 μL 0.5×TE Buffer稀释至1 μmoL/L,90 ℃ 30 s后移至室温冷却退火,进行连接头反应。
gRNA表达盒制备:利用酶切法将退火后的双链接头连接到pYL-U3-gRNA载体上,反应体系(10 μL):pYL-U3-gRNA 1 μL,退火后的接头产物1 μL,10×DNA ligase Buffer 1 μL, Bsa Ⅰ限制性内切酶(10 000 U/mL)0.5 μL,T4连接酶(400 000 U/mL) 0.1 μL,ddH2O 6.4 μL。反应程序:37 ℃ 5 min,20 ℃ 5 min,共5个循环。取1.5 μL反应产物作为PCR模板,用上游引物U-F和下游引物gRNA-R进行一轮PCR扩增,反应体系(10 μL):连接头产物1.5 μL,5×PS Buffer 2 μL,U-F/gRNA-R引物(10 μmol/L)各0.2 μL,dNTP(2 mmol/L) 0.6 μL,PrimeStarGXL 0.1 μL, ddH2O 5.4 μL。反应程序:98 ℃ 预变性2 min;98 ℃ 10 s,60 ℃ 10 s,72 ℃ 20 s 共25个循环。将1轮PCR产物稀释20倍后取1 μL为模板,用B1′和BL引物进行第2轮PCR扩增。反应体系(20 μL):稀释后的一轮PCR产物1 μL,5×PS Buffer 2 μL dNTP(2 mmol/L) 1 μL,B1′/BL引物(10 μmol/L)各0.3 μL,PrimeStarGXL 0.2 μL,ddH2O 12.2 μL。反应程序:98 ℃预变性2 min;98 ℃ 10 s,60 ℃ 10 s,72 ℃ 30 s 共25个循环。琼脂糖凝胶电泳检测PCR产物,切胶回收g-RNA表达盒。
CRISPR/Cas9表达载体构建:将二轮PCR胶回收产物克隆到pYL-CRISPR/Cas9-MTmono载体,反应体系(15 μL):二轮PCR胶回收产物2.5 μL,pYL-CRISPR/Cas9-MTmono质粒0.6 μL,Bsa Ⅰ核酸内切酶1 μL,10×cutsmart Buffer 1.5 μL,ddH2O 8.8 μL,PCR反应37 ℃ 15 min后暂停程序,加入T4 Ligase Buffer 1.5 μL,T4 Ligase 0.1 μL,PCR反应 37 ℃ 5 min,10 ℃ 5 min,20 ℃ 5 min,共15个循环。
表1 本研究中使用的引物
Tab.1 Primers used in this study

引物名称Primername引物序列(5′-3′)Primersequence(5′-3′)用途ApplicationT1-FGGCAGTCGCAGGAGCAGGCGCGGCGG接头引物T1-RAAACCCGCCGCGCCTGCTCCTGCHyg-FAGAAGAAGATGTTGGCGACCTHyg特异性引物Hyg-RGTCCTGCGGGTAAATAGCTNOS-FGCGGTGTCATCTATGTTACTAG载体通用引物OsEIL1-T1-FAGCAGTGCGTGATGGGGGAOsEIL1基因测序引物OsEIL1-T1-RACGATATGGCGCTGGTGATGCOsEIL2-T1-FTTTGGATTCTTCGGAGGCGAOsEIL2基因测序引物OsEIL2-T1-RCGGACCTTCTCCTTCCACCAU-FCTCCGTTTTACCTGTGGAATCG一轮PCR引物gRNA-RCGGAGGAAAATTCCATCCACB1′TTCAGAGGTCTCTCTCGCACTGGAATCGGCAGCAAAGG二轮PCR引物BLAGCGTGGGTCTCGACCGGGTCCATCCACTCCAAGCTCEOT1-FGTGCCCACATTTGATTTGAC脱靶检测引物1EOT1-RTTGATCGAGGCGTAGAGTAGAEOT2-FCCACTTCTGCACGATCAGCT脱靶检测引物2EOT2-RCTACGTCAACATCCGCAAGGEOT3-FACAAGCAGTGTTTGGGTTT脱靶检测引物3EOT3-RCTCCTCGACTCTTTCTTATCTCEOT4-FTGAAGGAGTCGGTCAAGTT脱靶检测引物4EOT4-RATGGAGCAGTGGAAGGGAEOT5-FTCCTCGGTGGAGAAGTAGCGG脱靶检测引物5EOT5-RCAGGTTCGTGGCTCGTGTTTG
1.3 质粒扩繁及阳性克隆筛选
将连接产物通过热激法转化至Mach1-T1感受态大肠杆菌中,在含有卡那霉素(50 mg/L)的LB培养基上涂板培养,挑取单菌落,用NOS-F和T1-R引物进行PCR检测阳性菌落,阳性菌落接种于含有卡那霉素(50 mg/L)的LB液体培养基,37 ℃,160 r/min过夜摇菌,用康为世纪公司(北京)的高纯度小量质粒提取试剂盒抽提质粒。抽提好的质粒用AscⅠ内切酶进行酶切检测,将酶切检测正确的质粒通过热激法转入到EHA105感受态农杆菌中。
1.4 T0植株获得及突变情况、潜在脱靶效应检测
用携带载体的农杆菌侵染水稻品种东农427的愈伤组织,潮霉素筛选出阳性愈伤组织并分化成T0植株。采用CTAB法[36]提取T0植株全基因组DNA,以潮霉素基因特异性引物Hyg-F和Hyg-R进行PCR扩增,检测阳性植株。设计引物OsEIL1-T1-F/R和OsEIL2-T1-F/R扩增OsEIL1和OsEIL2基因靶点及其附近的序列,对所有转基因阳性植株进行分析,PCR产物送至擎科生物技术有限公司(北京)进行测序,测序结果使用兼并序列解码(DSDecode)方法进行分析[37]。
利用CRISPR-GE在线工具包预测潜在脱靶位点,选择分值最高的5个位点作为潜在脱靶位点,利用PCR扩增测序分析所有T0突变植株的脱靶效应。
1.5 T1纯合突变植株及无T-DNA元件植株的筛选
在田间选取T1植株50株挂牌,取叶片提取全基因组DNA,利用OsEIL1-T1-F/R和OsEIL2-T1-F/R引物PCR测序,筛选出纯合双基因纯合突变的植株,再利用Hyg-F和Hyg-R引物在纯合的植株中筛选无T-DNA元件插入的植株,将这些植株自交繁殖至T2。
1.6 T2植株盐胁迫鉴定
取野生型种子与突变体种子于28 ℃浸种萌发3 d后加入Hoagland营养液,转移至培养箱中培养,培养温度为26 ℃,设置光周期为16 h光照(3 000 lx)8 h黑暗。幼苗培养28 d后加入200 mmol/L的NaCl溶液进行盐处理,同时设置对照,处理5 d后分别测量突变植株和野生型植株的鲜质量和干质量,并收取处理条件下突变植株和野生型植株的地上部分和地下部分,利用火焰光度法[38]测量Na+、K+离子含量。将盐处理后的植株换成营养液进行恢复处理,7 d后观察处理前后野生型幼苗和突变体幼苗的长势,统计幼苗存活率。
1.7 T2植株农艺性状考察
在大田环境下种植东农427野生型和T2无T-DNA元件插入的纯合突变体材料,成熟时期测量株高、有效穗数、穗粒数、结实率和千粒质量。
2 结果与分析
2.1 水稻OsEIL1和OsEIL2敲除靶点设计及载体构建
根据水稻OsEIL1和OsEIL2基因序列比对结果(图1),在两基因高相似度区域设计1个敲除位点(图2),利用单个sgRNA同时敲除2个基因,将构建好的载体命名为pYLCRISPR/Cas9-OsEIL1-OsEIL2,载体图谱如图3所示。

图1 OsEIL1和OsEIL2基因序列比对
Fig.1 Sequence alignment of OsEIL1 and OsEIL2
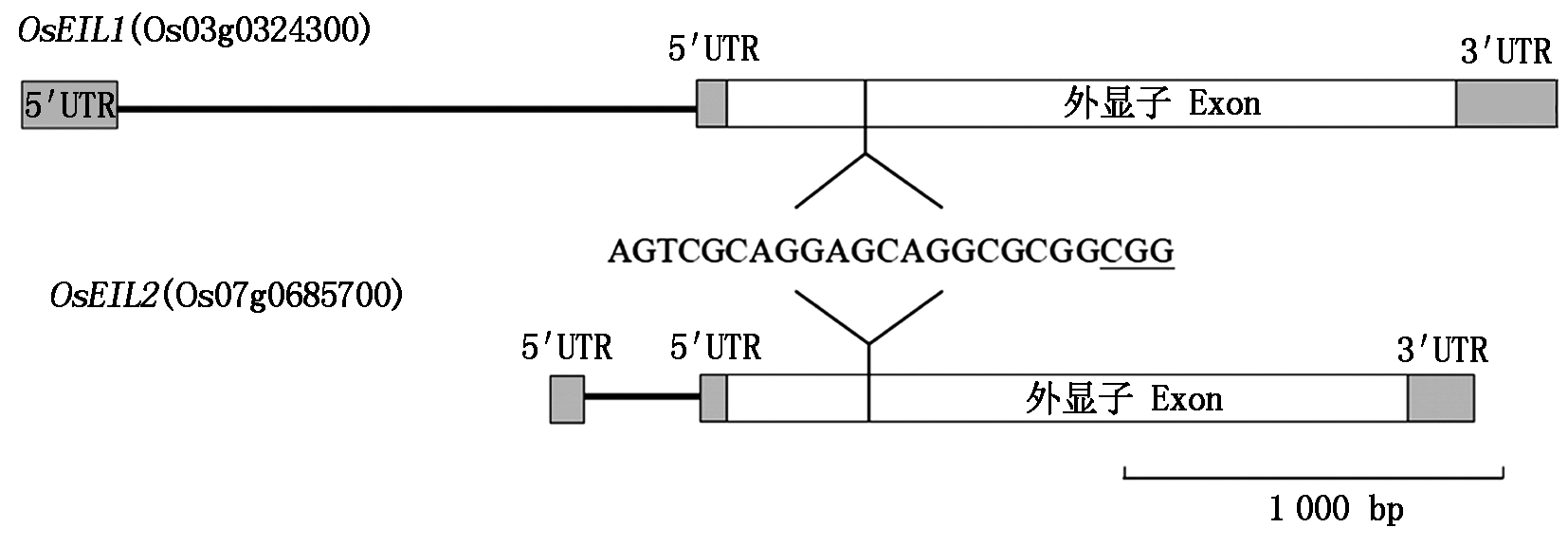
图2 OsEIL1和OsEIL2基因结构和靶点位置
Fig.2 Gene structure and target site of OsEIL1 and OsEIL2
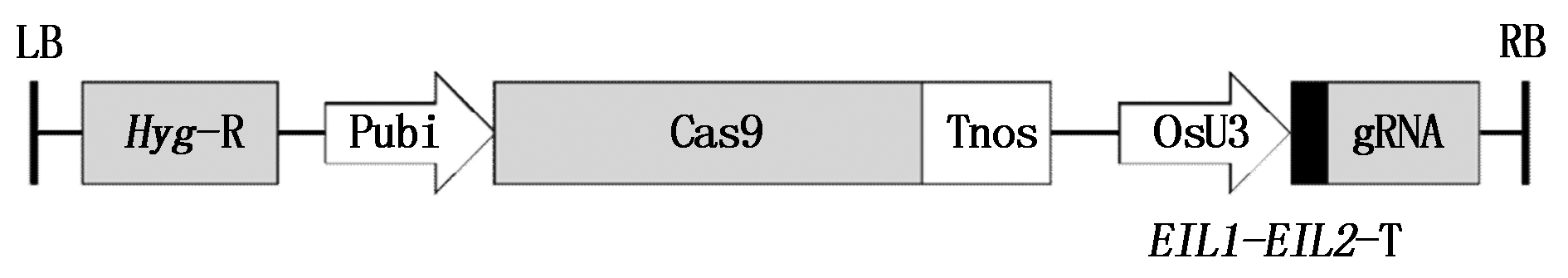
图3 pYLCRISPR/Cas9-OsEIL1-OsEIL2载体主要元件
Fig.3 pYLCRISPR/Cas9-OsEIL1-OsEIL2 main element of vector
2.2 T0转基因阳性苗检测及突变情况、脱靶情况的分析
通过农杆菌介导法将基因编辑载体转入到受体材料东农427中,在T0共获得30株再生植株,用载体特异性引物Hyg-F/R进行PCR检测,筛选阳性植株,结果表明,30株再生植株中有25株为阳性苗,阳性率为83.3%(图4)。
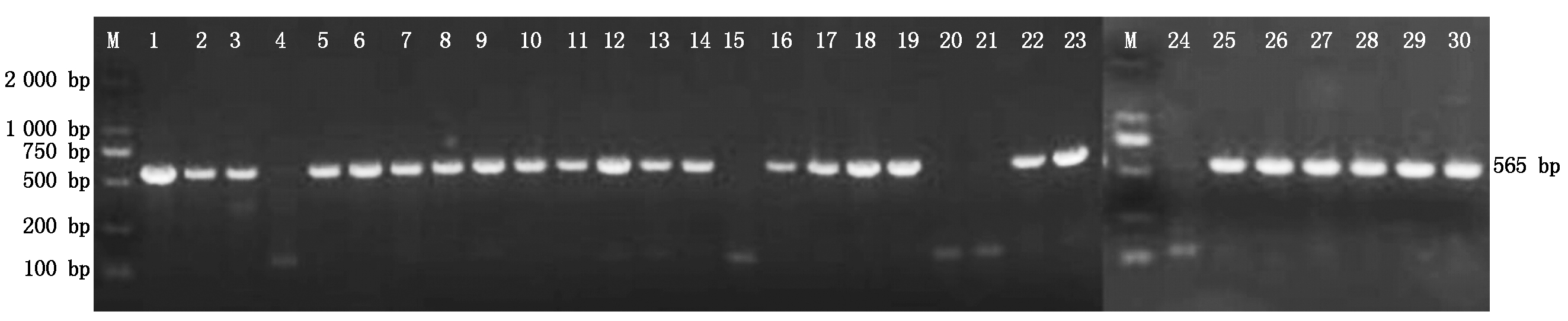
M.DM2000 DNA 标记; 1-30. T0植株。
M.DM2000 DNA Marker; 1-30. T0 plants.
图4 T0植株转基因检测
Fig.4 Transgenic detection of T0 plants
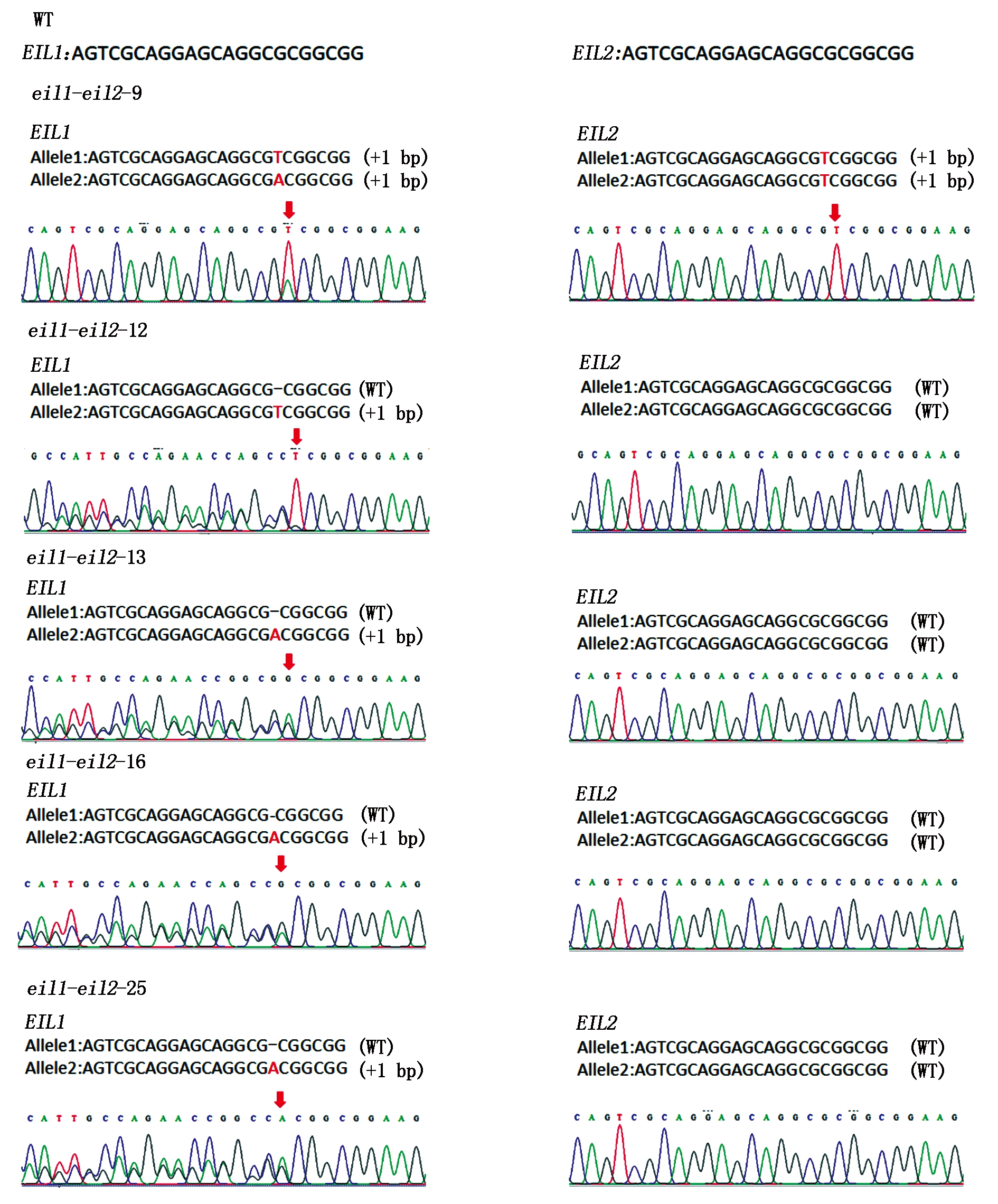
箭头.突变位置;WT.野生型。
Arrow. Mutation site; WT.Wild type.
图5 T0植株突变检测结果
Fig.5 Detection of mutation in T0 plants
利用特异性引物OsEIL1-T1-F/R和OsEIL2-T1-F/R扩增25株阳性植株的靶点区域序列并将PCR产物进行测序,测序结果表明,25株阳性植株中有5株(eil1-eil2-9、eil1-eil2-12、eil1-eil2-13、eil1-eil2-16、eil1-eil2-25)发生了突变(图5),突变率为20.0%,其中,OsEIL1基因突变植株有5株,1株为双等位突变,4株为杂合突变,OsEIL2基因突变植株有1株,突变类型为纯合突变(表2),eil1-eil2-9植株为OsEIL1和OsEIL2基因同时发生突变。对5株T0突变植株的潜在脱靶位点进行了分析,测序结果表明,5株T0突变植株均未发生脱靶效应(表3)
表2 T0转基因植株突变检测
Tab.2 Mutation detection of T0 plants
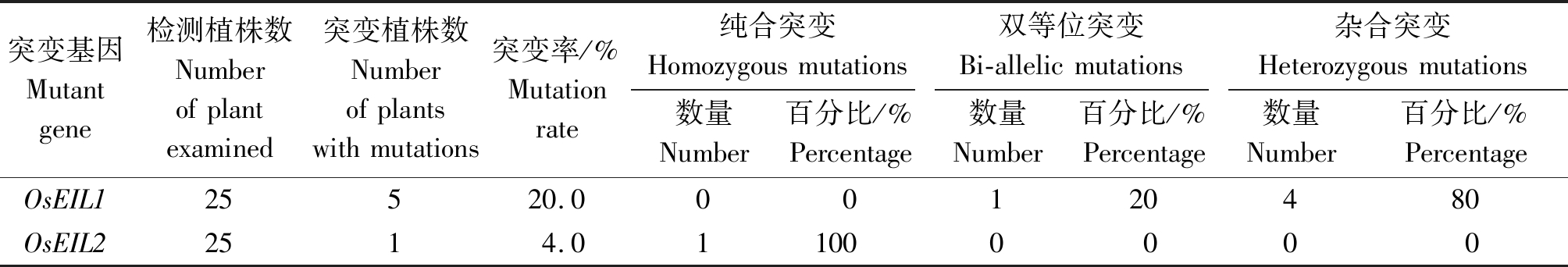
突变基因Mutantgene检测植株数Numberofplantexamined突变植株数Numberofplantswithmutations突变率/%Mutationrate纯合突变Homozygousmutations双等位突变Bi-allelicmutations杂合突变Heterozygousmutations数量Number百分比/%Percentage数量Number百分比/%Percentage数量Number百分比/%PercentageOsEIL125520.000120480OsEIL22514.011000000
表3 潜在脱靶位点检测
Tab.3 Mutation detections in the putative off-target site

潜在脱靶位点Putativeoff-targetsite所在染色体Chromosome位置Position序列Sequence基因Gene区域Region检测植株Detectedplantnumber突变株数Mutantplantnumber1Chr119198047TGGCGCAGGAGGAGGCGCGGGGGOs11g0267400CDS502Chr0822780498CGCCGCAGGGGCAGGCGCGGTGGOs08g0464000CDS503Chr0413820297GGGCGCAGGCGCAGGCGCGGGGGOs04g0306750CDS504Chr0229474266AGTCGCAGCAGCAGCAGCGGCGGIntergenic505Chr031780362AGTCGCAGTCGCAGGCGCAGCGGOs03g0131100CDS50
注:下划线.与sgRNA不匹配的碱基;双下划线.PAM序列。
Note: Underline.The bases that mismatched with sgRNA;Double underline.The PAM sequence.
2.3 无T-DNA元件的双基因纯合突变植株的筛选
将双基因突变植株eil1-eil2-9自交繁殖获得T1种子,将T1种子种成株行,随机挑选50株挂牌提取全基因组DNA,利用OsEIL1-T1-F/R和OsEIL2-T1-F/R筛选出双基因纯合突变植株16株,其中,OsEIL1和OsEIL2基因的突变方式均为单碱基插入,OsEIL1基因起始密码子(ATG)后第372和第373个碱基之间插入“A”导致该蛋白在第278个氨基酸处翻译提前终止,OsEIL2基因起始密码子(ATG)后第384和第385个碱基之间插入“T”导致该蛋白在第535个氨基酸处翻译提前终止。进一步利用Hyg-F/R引物在16株纯合突变植株中筛选出4株无T-DNA元件插入的植株,留种,自交加代,获得T2突变株系,将该株系命名为eil1-eil2HM(图6,7)。
2.4 T2植株耐盐性鉴定
将eil1-eil2HM突变株系进行苗期盐处理,处理前,突变体与野生型植株的长势、鲜质量和干质量与野生型差异不大。用含有200 mmol/L NaCl的营养液处理5 d后,野生型植株的长势受到了明显的抑制,而突变体植株的长势受到部分抑制,大部分植株仍继续存活(图8-A)。在鲜质量和干质量方面,盐处理后突变体植株的鲜质量和干质量均显著高于野生型植株(图8-B,C);在Na+离子含量方面,突变体植株的地上部及地下部Na+含量均显著低于野生型植株(图8-D);在K+离子含量方面,突变体植株地上部及地下部钾离子含量均与野生型植株差异不显著(图8-E);在换成正常营养液恢复7 d后,野生型植株基本全部死亡,存活率为8.3%,而突变体植株仍有大部分存活,存活率为75.0%(图8-F)。
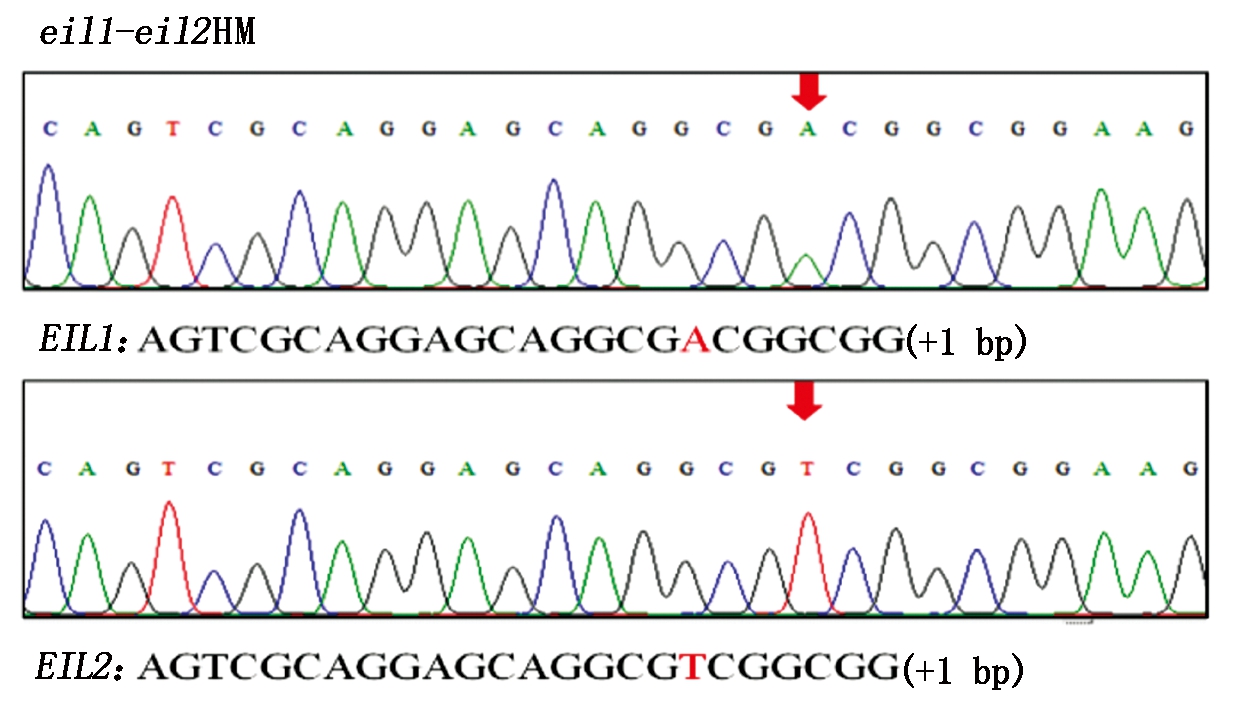
箭头.突变位置。
Arrow. Mutation site.
图6 双基因纯合突变植株测序结果
Fig.6 Sequencing of result of plant with double gene mutation

图7 eil1-eil2HM氨基酸序列分析
Fig.7 Amino acid sequence analysis of eil1-eil2HM
2.5 T2植株农艺性状考察
在成熟期对野生型和突变体植株的农艺性状进行调查,结果表明,野生型植株与突变体植株在株高、有效穗数、穗粒数、结实率和千粒质量上无显著差异(图9-A-F)
3 结论与讨论
CRISPR/Cas9被称为继ZFN和TALEN之后的第3代基因编辑技术,因其具有构建简单、高效定点编辑等优势而广泛应用于作物种质改良,与传统育种相比,CRISPR/Cas9系统可高效快速定点编辑目标基因,改良受体材料的性状,通过自交,在T1或T2即可获得纯合突变且不含T-DNA元件插入的突变株系,极大地缩短了水稻育种的周期。
本研究以耐盐负调控基因OsEIL1和OsEIL2为目标基因,通过序列比对的方式,在OsEIL1和OsEIL2高相似度区域设计一个sgRNA同时对这2个基因进行敲除。在T0 25株转基因苗中获得了5株突变植株(突变率为20.0%),该频率显著低于前人的研究结果[35, 39],推测可能与本研究所用的受体材料和sgRNA序列位点有关。有研究表明,CRISPR/Cas9系统在动物或植物中均可能发生脱靶效应[40-41],选择了5个与sgRNA序列最相近的位点作为潜在脱靶位点,并对T0的5株突变植株进行PCR测序,结果表明,5株突变植株均未发生脱靶效应。将T0双基因突变的植株进行自交,在T1筛选出纯合突变,且不含T-DNA元件插入的植株。纯合突变植株的OsEIL1和OsEIL2基因的突变类型均为单碱基插入,OsEIL1基因为“A”碱基插入,OsEIL2基因为碱基“T”插入。氨基酸序列比对结果显示,OsEIL1和OsEIL2基因单碱基插入后均引起了移码突变,导致翻译提前终止进而引起该蛋白不能发挥正常的生物学功能,由此可导致OsHKT2;1基因表达量下调,从而减少盐胁迫下根对钠离子的吸收,进而增强水稻的耐盐性。
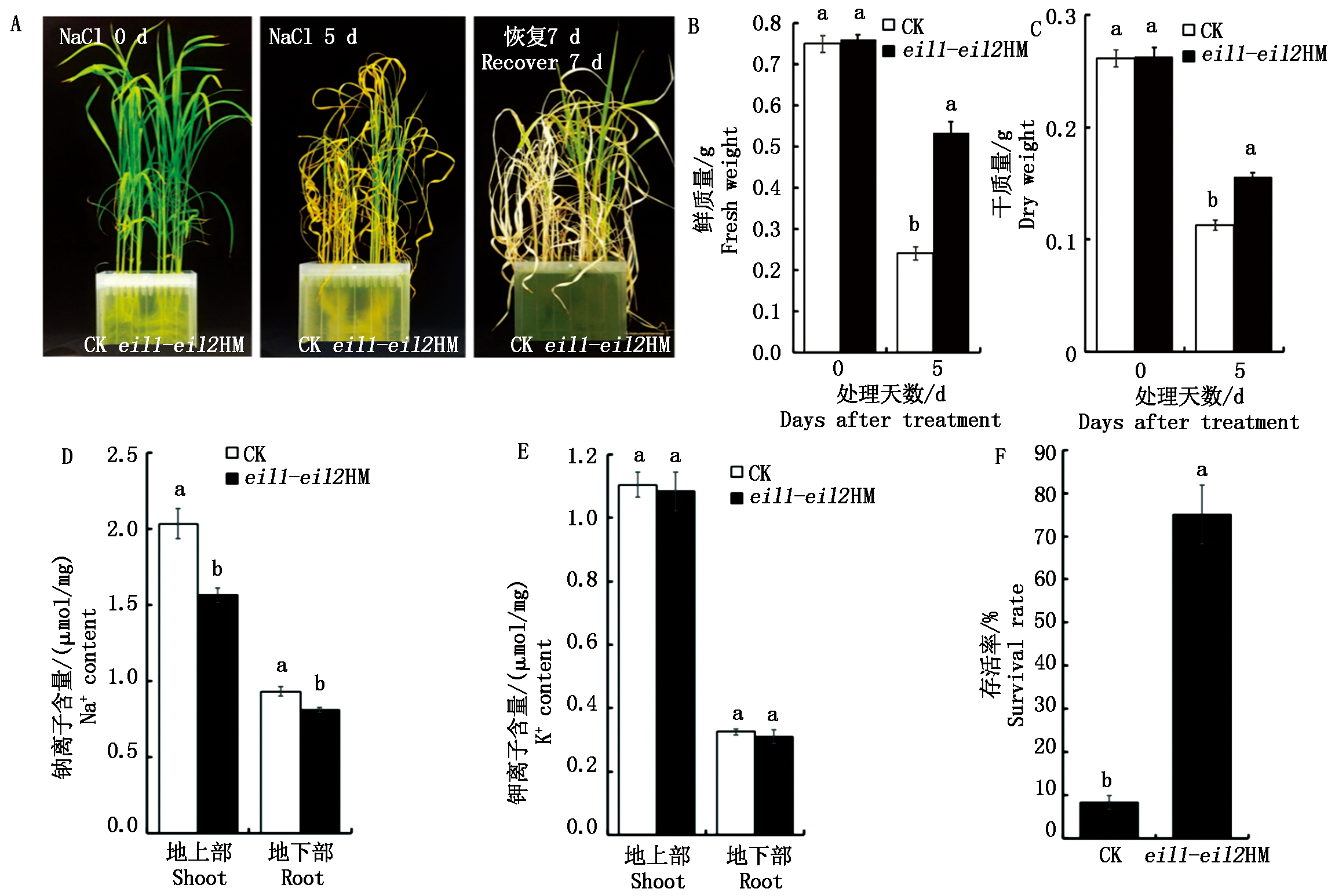
A.野生型与纯合突变体植株盐处理前后、恢复7 d表型观测;B.盐处理5 d后鲜质量变化;C.盐处理5 d后干质量变化;D.野生型和纯合突变体植株盐处理后地上部和地下部中的钠离子含量;E.野生型和纯合突变体植株盐处理后地上部和地下部中的钾离子含量;F.恢复7 d后植株存活率比较。不同小写字母表示在0.05水平差异显著。图9同。
A.Phenotype observation of wild type and homozygous mutant plants before and after salt treatment, and recover 7 days; B.Variation of fresh weight after 5 days of salt treatment; C.Variation of dry weight after 5 days of salt treatment; D. Na+ contents in the roots and shoots of wild type and homozygous mutant plants after salt treatment; E. K+ content in the shoots and roots of wild type and homozygous mutant plants after salt treatment; F.Comparison of plant survival rate after recover 7 days. Different small letters show significantly different at 0.05. The same as Fig.9.
图8 盐处理及指标测定
Fig.8 Salt treatment and related metrics measurements
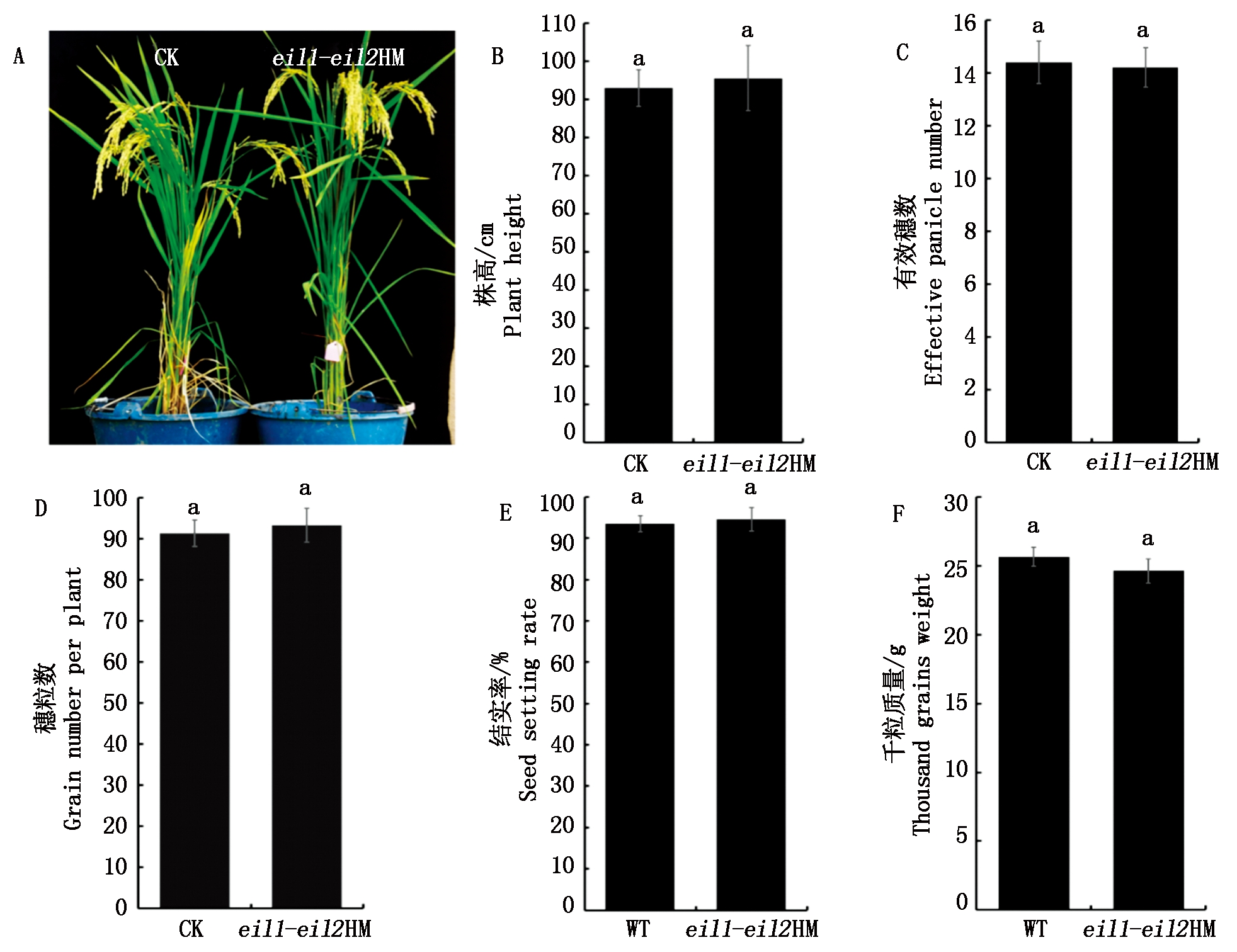
A-F.T2野生型与纯合突变体植株表型观测及株高、有效穗数、穗粒数、结实率、千粒质量分析。
A-F.Wild type and homozygous mutant plants phenotype observation and plant height, effective panicle, grain number per plant, seed setting rate, thousand grains weight analysis of T2 plants.
图9 T2植株农艺性状考察
Fig.9 Agronomic traits of T2 plants
将T2纯合突变株系进行苗期耐盐性鉴定及农艺性状调查,结果表明,OsEIL1和OsEIL2基因突变后,植株对Na+的吸收减弱,从而减轻了Na+毒害,最终提高了水稻的耐盐性。在成熟期对野生型植株和突变体植株的农艺性状进行考察,OsEIL1和OsEIL2基因突变后植株的株高、有效穗数、穗粒数、结实率和千粒质量均未发生显著变化,说明OsEIL1和OsEIL2基因突变并未对植株的主要农艺性状造成影响。
综上所述,本研究以粳稻品种东农427为材料,通过CRISPR/Cas9技术对OsEIL1和OsEIL2基因进行定点敲除并获得了双基因纯合突变且不含T-DNA元件的突变体材料,突变体材料的耐盐性明显提升且主要农艺性状未受到影响。该结果为耐盐水稻品种的培育提供了理论和材料基础。
[1] Ma C Q, Wang Y G, Gu D, Nan J D, Chen S X, Li H Y. Overexpression of S-Adenosyl-l-Methionine synthetase 2 from sugar beet M14 increased Arabidopsis tolerance to salt and oxidative stress[J].International Journal of Molecular Science, 2017,18(4):847. doi:10.3390/ijms18040847.
[2] Ouhibi C, Attia H, Rebah F, Msilini N, Chebbi M, Aarrouf J, Urban L,Lachaal M.Salt stress mitigation by seed priming with UV-C in lettuce plants: growth, antioxidant activity and phenolic compounds[J].Plant Physiology and Biochemistry, 2014,83: 126-133.doi:10.1016/j.plaphy.2014.07.019.
[3] Munns R, Tester M.Mechanisms of salinity tolerance[J].Annual Review of Plant Biology, 2008,59: 651-681. doi:10.1146/annurev.arplant.59.032607.092911.
[4] 杨劲松.中国盐渍土研究的发展历程与展望[J].土壤学报,2008,45(5):837-845. doi:10.3321/j.issn:0564-3929.2008.05.010.
Yang J S. Development and prospect of the research on salt affected soils in China[J].Acta Pedologica Sinica,2008,45(5): 837-845.
[5] Khush G S. What it will take to feed 5.0 billion rice consumers in 2030[J].Plant Molecular Biology, 2005,59(1): 1-6. doi:10.1007/s11103-005-2159-5.
[6] Li Y, Xiao J H, Chen L L, Huang X H, Cheng Z K, Han B, Zhang Q F, Wu C Y. Rice functional genomics research: past decade and future[J].Molecular Plant, 2018,11(3): 359-380. doi:10.1016/j.molp.2018.01.007.
[7] Lutts S, Kinet J M, Bouharmont J. Changes in plant response to NaCl during development of rice(Oryza sativa L.) varzieties differing in salinity resistance[J]. Journal of Experimental Botany, 1995,46(293): 1843-1852. doi:10.1093/jxb/46.12.1843.
[8] Huang X Y, Chao D Y, Gao J P, Zhu M Z, Shi M, Lin H X. A previously unknown zinc finger protein, DST, regulates drought and salt tolerance in rice via stomatal aperture control[J].Genes Development, 2009,23(15): 1805-1817. doi:10.1101/gad.1812409.
[9] El Mahi H, Pérez-Hormaeche J, De Luca A, Villalta I, Espartero J, G mez-Arjona F, Fern
mez-Arjona F, Fern ndez J L, Bundó M, Mendoza I, Mieulet D, Lalanne E, Lee S Y, Yun D J, Guiderdoni E, Aguilar M, Leidi E O, Pardo J M, Quintero F J. A critical role of sodium flux via the plasma membrane Na+/H+ exchanger SOS1 in the salt tolerance of rice[J].Plant Physiology, 2019,180(2):1046-1065. doi:10.1104/pp.19.00324.
ndez J L, Bundó M, Mendoza I, Mieulet D, Lalanne E, Lee S Y, Yun D J, Guiderdoni E, Aguilar M, Leidi E O, Pardo J M, Quintero F J. A critical role of sodium flux via the plasma membrane Na+/H+ exchanger SOS1 in the salt tolerance of rice[J].Plant Physiology, 2019,180(2):1046-1065. doi:10.1104/pp.19.00324.
[10] Suzuki K, Costa A, Nakayama H, Katsuhara M, Shinmyo A, Horie T. OsHKT2;2/1-mediated Na+ influx over K+ uptake in roots potentially increases toxic Na+ accumulation in a salt-tolerant landrace of rice Nona Bokra upon salinity stress[J].Journal of Plant Research, 2016,129(1): 67-77. doi:10.1007/s10265-015-0764-1.
[11] Qiu D Y, Xiao J, Xie W B, Liu H B, Li X H, Xiong L Z,Wang S P. Rice gene network inferred from expression profiling of plants overexpressing OsWRKY13, a positive regulator of disease resistance[J].Molecular Plant, 2008,1(3): 538-551. doi:10.1093/mplant/ssn012.
[12] Hou X, Xie K B, Yao J L, Qi Z Y, Xiong L Z.A homolog of human ski-interacting protein in rice positively regulates cell viability and stress tolerance[J].Proceedings of the National Academy of Sciences of the United States of America, 2009,106(15): 6410-6415. doi:10.1073/pnas.0901940106.
[13] Zhu J K. Salt and drought stress signal transduction in plants[J].Annual Review of Plant Biology,2002,53:247-273. doi:10.1146/annurev.arplant.53.091401.143329.
[14] Erpen L, Devi H S, Grosser J W, Dutt M. Potential use of the DREB/ERF, MYB, NAC and WRKY transcription factors to improve abiotic and biotic stress in transgenic plants[J].Plant Cell, Tissue and Organ Culture, 2017,132(1):1-25. doi:10.1007/s11240-017-1320-6.
[15] Mao C Z, Wang S M, Jia Q J, Wu P.OsEIL1, a rice homolog of the Arabidopsis EIN3 regulates the ethylene response as a positive component[J].Plant Molecular Biology,2006,61(1-2):141-152.doi:10.1007/s11103-005-6184-1.
[16] Yang C, Ma B, He S J, Xiong Q, Duan K X, Yin C C, Chen H, Lu X, Chen S Y, Zhang J S. MAOHUZI6/ETHYLENE INSENSITIVE3-LIKE1 and ETHYLENE INSENSITIVE3-LIKE2 regulate ethylene response of roots and coleoptiles and negatively affect salt tolerance in rice[J]. Plant Physiology, 2015,169(1): 148-165. doi:10.1104/pp.15.00353.
[17] Cermak T, Doyle E L, Christian M, Wang L, Zhang Y, Schmidt C, Baller J A, Somia N V, Bogdanove A J, Voytas D F.Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting[J].Nucleic Acids Research, 2011,39(17): 7879-7879. doi:10.1093/nar/gkr739.
[18] Lloyd A, Plaisier C L, Carroll D, Drews G N.Targeted mutagenesis using zinc-finger nucleases in Arabidopsis[J].Proceedings of the National Academy of Sciences of the United States of America, 2005,102(6): 2232-2237. doi:10.1073/pnas.0409339102.
[19] Cong L, Ran F A, Cox D, Lin S L, Barretto R, Habib N, Hsu P D, Wu X B, Jiang W Y, Marraffini L A, Zhang F.Multiplex genome engineering using CRISPR/Cas systems[J].Science, 2013,339(6121):819-823.doi:10.1126/science.1231143.
[20] Barrangou R,Fremaux C,Deveau H,Richards M,Boyaval P,Moineau S, Romero D A, Horvath P.CRISPR provides acquired resistance against viruses in prokaryotes[J].Science, 2007,315(5819):1709-1712.doi:10.1126/science.1138140.
[21] Puchta H. The repair of double-strand breaks in plants: mechanisms and consequences for genome evolution[J].Journal of Experimental Botany, 2005,56(409): 1-14. doi:10.1093/jxb/eri025.
[22] Huang L Y, Zhang R, Huang G F, Li Y X, Melaku G, Zhang S L, Chen H T, Zhao Y J, Zhang J, Zhang Y S, Hu F Y. Developing superior alleles of yield genes in rice by artificial mutagenesis using the CRISPR/Cas9 system[J].The Crop Journal, 2018,6(5):475-481. doi:10.1016/j.cj.2018.05.005.
[23] Miao C B, Xiao L H, Hua K, Zou C S, Zhao Y, Bressan R A, Zhu J K. Mutations in a subfamily of abscisic acid receptor genes promote rice growth and productivity[J].Proceedings of the National Academy of Sciences of the United States of America, 2018,115(23):6058-6063. doi:10.1073/pnas.1804774115.
[24] Shan Q W, Wang Y P, Li J, Zhang Y, Chen K L, Liang Z, Zhang K, Liu J X, Xi J J, Qiu J L, Gao C X. Targeted genome modification of crop plants using a CRISPR-Cas system[J].Nature Biotechnology, 2013,31(8): 686-688. doi:10.1038/nbt.2650.
[25] Wang Y P, Cheng X, Shan Q W, Zhang Y, Liu J X, Gao C X, Qiu J L. Simultaneous editing of three homoeoalleles in hexaploid bread wheat confers heritable resistance to powdery mildew[J].Nature Biotechnology, 2014,32(9): 947-951. doi:10.1038/nbt.2969.
[26] Do P T, Nguyen C X, Bui H T, Tran L T N, Stacey G, Gillman J D, Zhang Z J, Stacey M G. Demonstration of highly efficient dual gRNA CRISPR/Cas9 editing of the homeologous GmFAD2-1A and GmFAD2-1B genes to yield a high oleic, low linoleic and alpha-linolenic acid phenotype in soybean[J].BMC Plant Biology, 2019,19(1):311. doi:10.1186/s12870-019-1906-8.
[27] Jacobs T B, LaFayette P R, Schmitz R J, Parrott W A. Targeted genome modifications in soybean with CRISPR/Cas9[J]. BMC Biotechnology, 2015,15: 16. doi:10.1186/s12896-015-0131-2.
[28] Liang Z, Zhang K, Chen K L, Gao C X. Targeted mutagenesis in Zea mays using TALENs and the CRISPR/Cas system[J].Journal of Genetics and Genomics, 2014,41(2): 63-68. doi:10.1016/j.jgg.2013.12.001.
[29] Svitashev S, Young J K, Schwartz C, Gao H R, Falco S C, Cigan A M. Targeted mutagenesis, precise gene editing, and site-specific gene insertion in maize using Cas9 and guide RNA[J].Plant Physiol, 2015,169(2):931-945. doi:10.1104/pp.15.00793.
[30] Li A X,Jia S G,Yobi A,Ge Z X,Sato S J,Zhang C,Angelovici R,Clemente T E,Holding D R.Editing of an alpha-kafirin gene family increases, digestibility and protein quality in sorghum[J].Plant Physiology, 2018, 177(4): 1425-1438. doi:10.1104/pp.18.00200.
[31] Wang C, Liu Q, Shen Y, Hua Y F, Wang J J, Lin J R, Wu M G, Sun T T, Cheng Z K, Mercier R, Wang K J.Clonal seeds from hybrid rice by simultaneous genome engineering of meiosis and fertilization genes[J].Nat Biotechnol, 2019,37(3): 283-286. doi:10.1038/s41587-018-0003-0.
[32] Tang L, Mao B G, Li Y K, Lü Q M, Zhang L P, Chen C Y, He H J, Wang W P, Zeng X F, Shao Y, Pan Y L, Hu Y Y, Peng Y, Fu X Q, Li H Q, Xia S T, Zhao B R.Knockout of OsNramp5 using the CRISPR/Cas9 system produces low Cd-accumulating indica rice without compromising yield[J]. Scientific Reports, 2017,7(1): 14438. doi:10.1038/s41598-017-14832-9.
[33] Zhang A N, Liu Y, Wang F M, Li T F, Chen Z H, Kong D Y, Bi J G, Zhang F Y, Luo X X, Wang J H, Tang J J, Yu X Q, Liu G L, Luo L J. Enhanced rice salinity tolerance via CRISPR/Cas9-targeted mutagenesis of the OsRR22 gene[J]. Molecular Breeding, 2019,39(3):47. doi:10.1007/s11032-019-0954-y.
[34] Xie X R, Ma X L, Zhu Q L, Zeng D C, Li G S, Liu Y G. CRISPR-GE: a convenient software toolkit for CRISPR-based genome editing[J].Molecular Plant, 2017,10(9):1246-1249. doi:10.1016/j.molp.2017.06.004.
[35] Ma X L, Zhang Q Y, Zhu Q L, Liu W, Chen Y, Qiu R, Wang B, Yang Z F, Li H Y, Lin Y R, Xie Y Y, Shen R X, Chen S F, Wang Z, Chen Y L, Guo J X, Chen L T, Zhao X C, Dong Z C, Liu Y G. A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants[J].Molecular Plant, 2015,8(8):1274-1284. doi:10.1016/j.molp.2015.04.007.
[36] Rowland L J, Nguyen B. Use of polyethylene glycol for purification of DNA from leaf tissue of woody plants[J].Biotechniques, 1993,14(5):734-736. doi:10.1002/bip.360330513.
[37] Liu W Z,Xie X R,Ma X L,Li J,Chen J H,Liu Y G.DSDecode: a web-based tool for decoding of sequencing chromatograms for genotyping of targeted mutations[J].Molecular Plant, 2015,8(9):1431-1433.doi:10.1016/j.molp.2015.05.009.
[38] 方玉洁,覃永华,熊立仲.水稻叶片Na+,K+含量测定(火焰光度法)[J]. Bio-Protocol, 2018:Bio-101 e1010149. doi:10.21769/BioProtoc.1010149.
Fang Y J, Qin Y H, Xiong L Z.Determination of Na+, K+ content in rice leaf(flame photometric method)[J]. Bio-Protocol, 2018:Bio-101e1010149.
[39] Li M R, Li X X, Zhou Z J, Wu P Z, Fang M C, Pan X P, Lin Q P, Luo W B, Wu G L, Li H Q. Reassessment of the four yield-related genes Gn1a, DEP1, GS3, and IPA1 in rice using a CRISPR/Cas9 system[J].Frontiers in Plant Science, 2016,7: 377. doi:10.3389/fpls.2016.00377.
[40] Tang X, Liu G Q, Zhou J P, Ren Q R, You Q, Tian L, Xin X H, Zhong Z H, Liu B L, Zheng X L, Zhang D W, Malzahn A, Gong Z Y, Qi Y P, Zhang T, Zhang Y. A large-scale whole-genome sequencing analysis reveals highly specific genome editing by both Cas9 and Cpf1(Cas12a) nucleases in rice[J].Genome Biology, 2018,19:84. doi:10.1186/s13059-018-1458-5.
[41] Tsai S Q, Joung J K. Defining and improving the genome-wide specificities of CRISPR Cas9 nucleases[J].Nature Reviews Genetics, 2016,17(5): 300-312. doi:10.1038/nrg.2016.28.
