水稻是世界上重要的粮食作物,20世纪60年代的“绿色革命”提高了全球稻米的产量,开辟了水稻研究的新领域[1-4]。但是,为了实现绿色革命的最大生产潜力,需要大量的氮肥[5]。然而大量的氮肥施用会造成氮素的流失[6],同时引起湖泊和水库的富营养化[7]、地下水污染[8]和大气污染[9]等环境问题,而不同品种间水稻氮肥利用率差异可达到79.6%[10]。因此,筛选氮素应答基因来提高氮素利用率将成为今后科研的重点。
氮素作为一种水稻生长发育必需的营养元素,会显著影响水稻分蘖数目[11]、淀粉品质[12]、糖类[13-14]和氨基酸含量[15-16]等生理特性。长期以来,氮素管理作为栽培调控的重要手段,对于水稻的生长发育产生的影响一直是研究的重点。尽管目前一些关于水稻的氮素应答基因NRT1.1B[17]、OsNRT1.1A/OsNPF6.3[18]、OsNRT2.1[19]、OsNRT2.3[20]、OsPTR9[21]等已被发现,但氮素应答机制并没有完全解释清楚,在栽培上的应用也不够充分。
转录组分析是通过测序技术阐述同一细胞在不同生长时期及生长环境下基因差异表达的情况应用最为广泛的技术[22]。鉴于目前关于水稻分蘖期氮素应答的研究还不够完善,在本研究中,笔者开展了氮处理下水稻分蘖期基因表达的转录组动态分析,目的是确定调控分蘖期水稻叶片的氮素响应基因,为利用氮素应答模式精准调控氮素影响水稻栽培管理提供新思路。
1 材料和方法
1.1 植物生长条件
本试验在哈尔滨东北农业大学进行。首先将水稻品种五优稻4号(WYD4)种子用30%次氯酸钠灭菌30 min后放入锥形瓶中,然后用0.6%的硝酸在室温下处理打破休眠状态,移入30 ℃培养箱催芽,在温室中播种。三叶一心期移栽至0.24 m2的白盆中,插秧规格为3株苗/穴,插秧方式为10 cm×10 cm。
1.2 氮素处理
盆栽土壤的基础肥力为全氮1.51 g/kg、速效氮158.76 mg/kg、速效磷50.65 mg/kg、速效钾177.68 mg/kg、pH值7.52、有机质3.11%。处理组(Nitrogen treatment,Nt)基肥和分蘖肥分别为纯氮72,54 kg/hm2,对照组(Control,CK)不施氮,其他养分含量相同,水分管理为常规管理。每个时间点设置3次生物重复,以确保试验的可靠性。
1.3 样品制备
在氮素处理7,14,21 d后拍照,分别收集叶片在液氮保存30 min后放入试管中,并相应地命名为CK7d、CK14d、CK21d、Nt7d、Nt14d、Nt21d,然后干冰保存下快递至安诺公司进行RNA提取和转录组分析,使用NanoPhotometer®分光光度计(Implen,CA,USA)和Qubit RNA分析试剂盒,在Qubit 3.0荧光仪(Life Technologies,CA,USA)中测定RNA的纯度和浓度。使用Agilent 2100 RNA Nano 6000检测试剂盒(Agilent Technologies,CA,USA)检测RNA样品的完整性和浓度。库成簇(Clustering)与测序使用 HiSeq PE Cluster Kit v4-cBot-HS(Illumia)试剂在cBot上生成簇。随后在 HiSeq 测序平台上运行双端测序程序(PE),得到 150 bp 的双端测序 reads。GffCompare可用于比较装配有RNA-seq数据的转录组和参考转录组。通过HTSeqv 0.6.0对每个样本中每个基因的Reads计数(http://www-huber.embl.de/users/anders/HTSeq/doc/overview)。然后计算FPKM(每千碱基每米的片段映射读数)来估计每个样本中基因的表达水平。
1.4 差异基因表达分析
DESeq2 针对2组之间有生物学重复样本的差异表达分析,比较处理组与参考组,并选取|log2Ratio|≥1和q<0.05的基因作为差异显著表达基因,得到上下调基因个数。DESeq2 理论基础为Count值服从负二项分布假设,主要考虑相同表达水平的基因可能具有相似的离差以及每个基因的表达特性。利用线性回归估计基因在各样品中的表达强度,并用Wald test 计算每个基因在2组样品中不差异表达的概率值P-value,并通过BH方法对P-value进行校正得到q-value。
1.5 功能富集分析
对于差异基因的GO(Gene Ontology,http://geneontology.org/)富集分析根据计算每个GO Term的基因数目,然后应用超几何检验,找出与整个基因组背景相比,在差异表达基因中显著富集的GO Term。计算得到的P-value通过校正之后,以q≤0.05为阈值,满足此条件的GO term定义为在差异表达基因中显著富集的GO term。通过GO功能显著性富集分析能确定差异表达基因行使的主要生物学功能。KEGG(Kyoto Encyclopedia of Genes and Genomes,http://www.kegg.jp/)是基因组破译方面的数据库。在给出染色体中一套完整基因的情况下,它可以对蛋白质交互(互动)网络在各种细胞活动起的作用做出预测。对KEGG 中每个Pathway应用超几何检验进行富集分析,找出差异表达基因中显著性富集的Pathway。
1.6 蛋白互作网络
应用 STRING 蛋白质互作数据库(http://string-db.org/)中的互作关系,针对数据库中包含的物种,直接将目标基因集(比如差异基因list)映射到物种的蛋白互作网络。蛋白互作网络数据文件可以直接导入Cytoscape软件,并根据目标基因集中的基因属性对网络进行可视化编辑。
1.7 数据处理与分析
图表和图像的数据分析利用WPS Office(11.1.0.9339)和Adobe Photoshop(13.0.20120315)进行处理。
2 结果与分析
2.1 氮处理引起分蘖期WYD4植株表型变化
氮素处理后7,14,21 d表型可以看出,施氮后WYD4植株颜色明显比未施氮植株深(图1-A-F),说明叶片颜色群体长势差异与氮素处理后不同时间点不同代谢途径对氮素应答有关。采集分蘖期WYD4叶片进行了转录组分析,期望发现水稻对氮素的应答模式来进行氮素管理,为水稻栽培提供参考。
2.2 氮处理引起分蘖期水稻基因表达变化
对照组和处理组在3个时间点(7,14,21 d)分别提取WYD4嫩绿叶片RNA,得到纯化的mRNA并生成cDNA用于转录组分析。结果发现,与对照相比,施氮的WYD4叶片有3 870个差异表达基因(DEGs,Differential expression genes)(图1-G)。

A、B、C.不施氮条件下7,14,21 d WYD4表型;D、E、F.施氮条件下7,14,21 d WYD4表型;G.DEGs的数量。
A,B,C.WYD4 phenotypes at 7,14,21 d without nitrogen application;D,E,F. 7,14,21 d WYD4 phenotypes under nitrogen application;G.The number of DEGs.
图1 氮素处理后分蘖期WYD4表型及DEGs的数量
Fig.1 WYD4 phenotype and number of DEGs at tillering stage after nitrogen treatment
结果表明,在氮处理的早期(7 d),存在大量的DEGs(2 278,图1-G),但随着处理时间的延长,DEGs的数目反而在不断减少,在这3个时间点上,上调基因的数目比下调基因的数目少,说明氮素早期应答的基因较多,而且氮素处理抑制了氮素应答的基因表达。
通过层次聚类分析并绘制了Venn 图和Volcano图(图2),展示了不同时间氮处理下DEGs表达模式的异同。这些图表显示7,14,21 d的DEGs有相似的表达模式但也有所不同(图2-A)。7 d时,部分DEGs上调,部分DEGs下调,且差异基因数目最大。可能与氮处理的短期反应有关。此外,25个DEGs在3个时间点都出现了转录变化,表明这些基因可能在整个氮处理期间都发挥作用(图2-A),14 d是基因表达差异最大的时间点,这可能与氮素发挥作用的时间有关(图2-B)。
2.3 氮素应答DEGs的GO和KEGG富集分析
GO分析发现氮应答的DEGs主要富集在代谢过程、细胞过程、细胞组分、催化活性和结合等方面(图3),这些结果对于氮素在水稻分蘖期介导氧化还原、物质代谢和酶活性调节等方面具有重要的指导意义。进一步通过KEGG分析发现,大多数DEGs(q_value均小于0.05)显著富集在半乳糖代谢、氨基糖和核苷酸糖代谢、丙氨酸、天冬氨酸和谷氨酸代谢等代谢途径(图4),共有22个DEGs与这些途径显著相关(图5,6)。
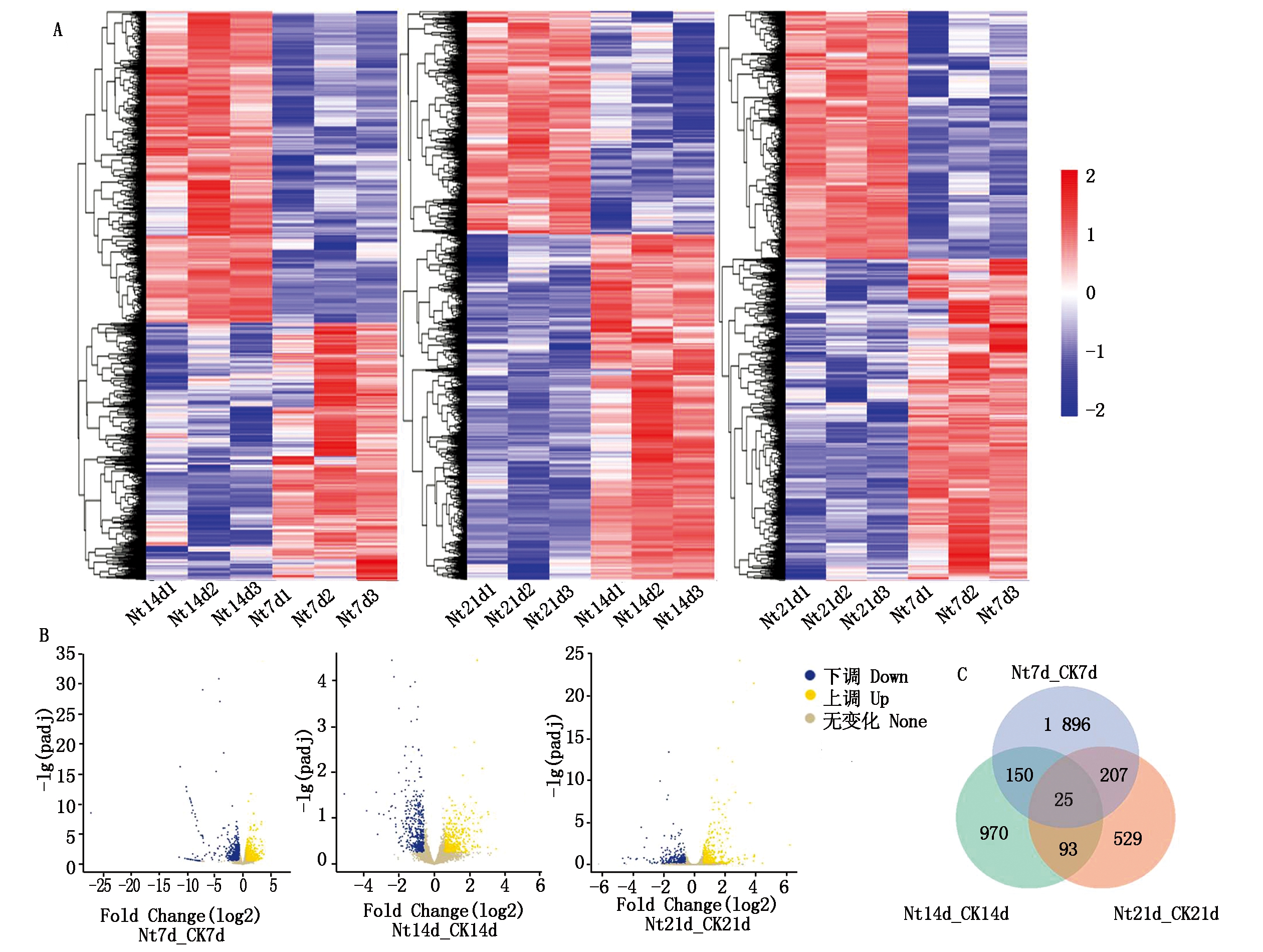
A.氮处理7,14,21 d差异基因层次聚类分析。B.氮处理7,14,21 d差异基因韦恩图。C.氮处理7,14,21 d差异基因火山图。黄色.表达上调基因;蓝色.表达下调基因。
A.Hierarchical clustering analysis of DEGs at 7,14,21 d after nitrogen treatment;B.Venn diagram of DEGs at 7,14,21 d after nitrogen treatment;C.Volcano map of DEGs at 7,14,21 d after nitrogen treatment. Yellow.Up-regulated genes;Blue.Down-regulated genes.
图2 不同氮处理下水稻叶片的聚类分析
Fig.2 Cluster analysis of DEGs in rice leaves under different nitrogen treatments
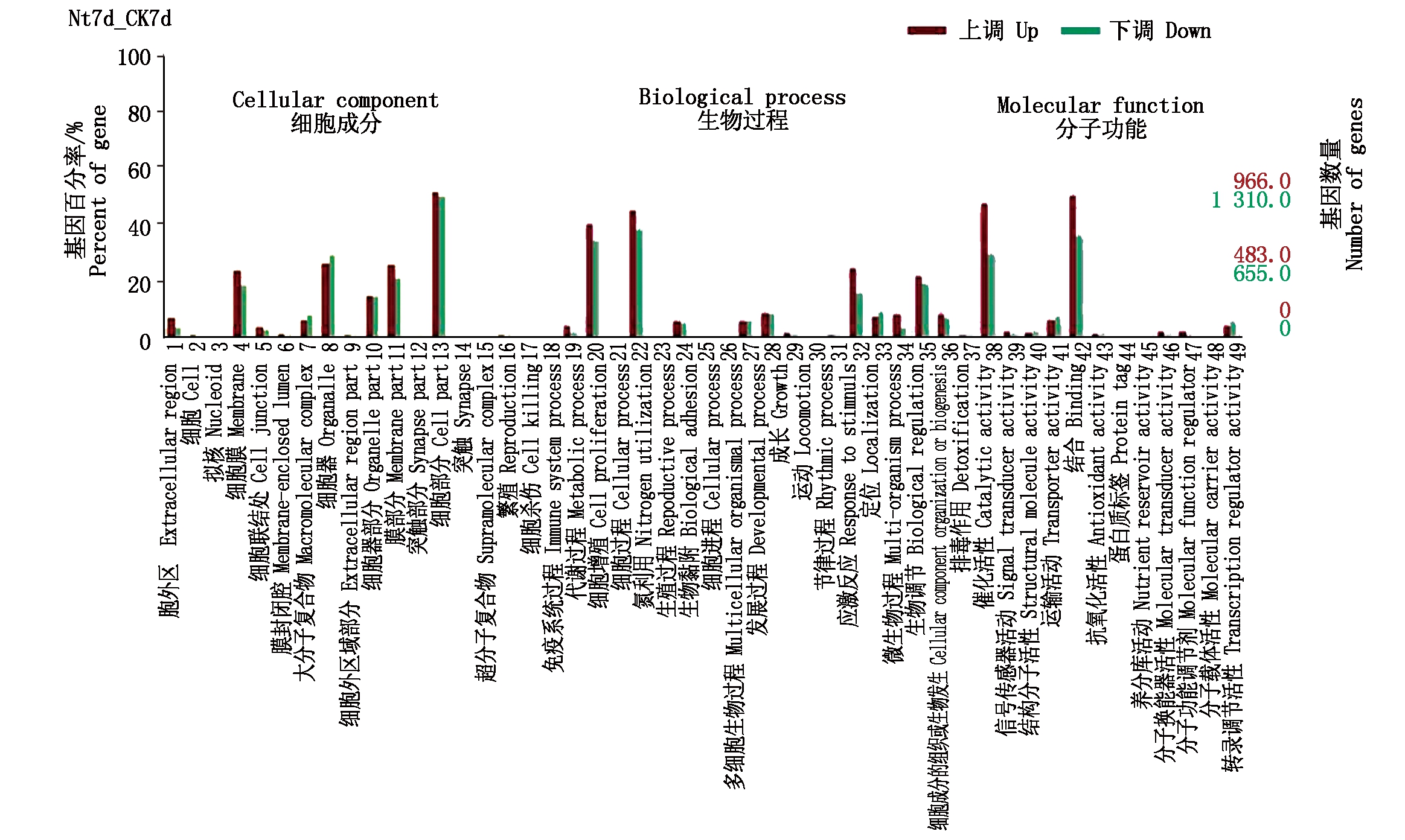
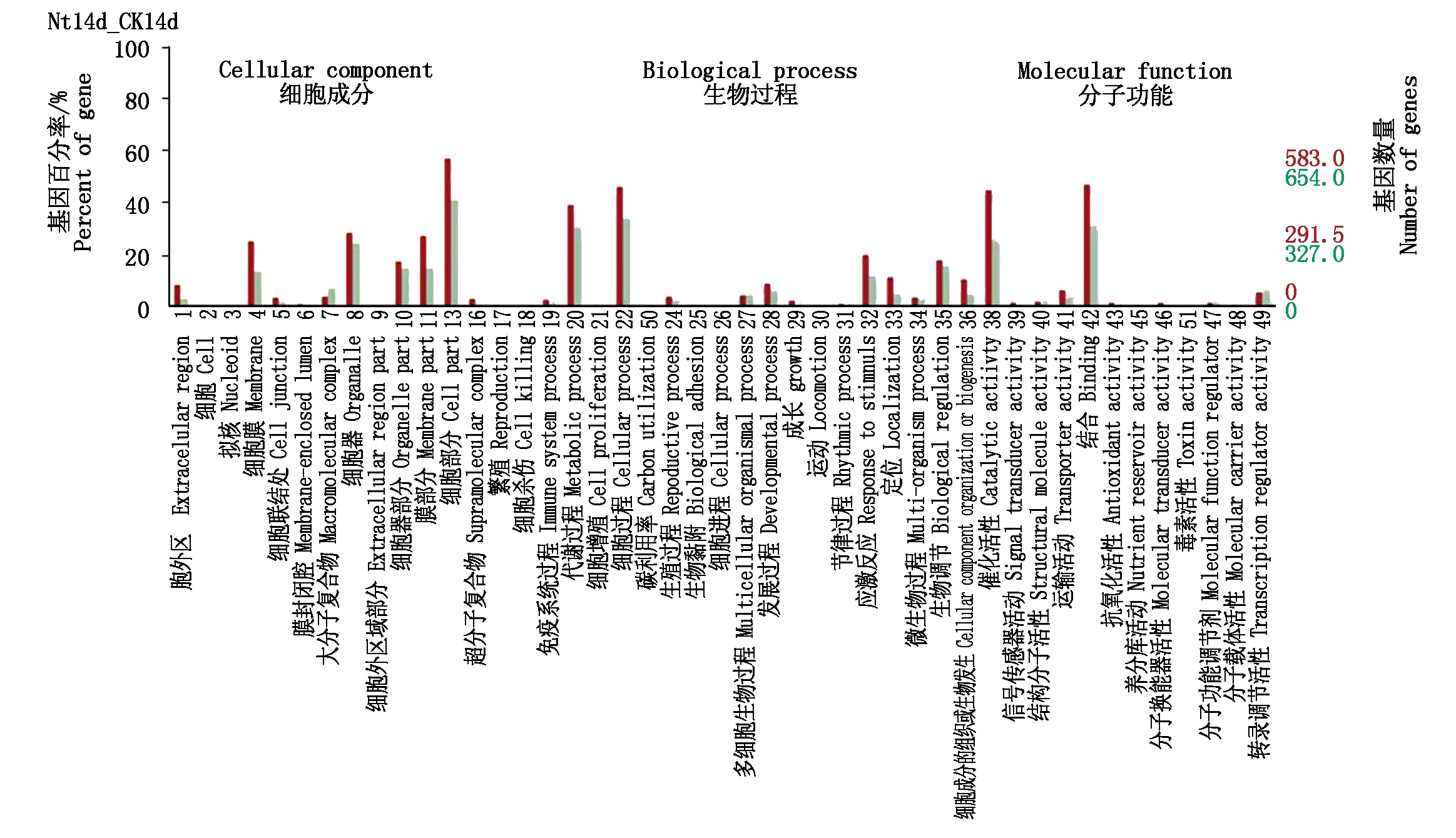

图3 DEGs的GO统计
Fig.3 GO statistics histogram of DEGs
2.4 氮素对氨基酸途径的影响
氨基酸是一种重要的生物分子,是蛋白质合成的基本单元。发现氮素处理会显著影响很多与氨基酸途径有关的基因表达,这些差异表达的基因显著影响丙氨酸、天冬氨酸和谷氨酸的代谢,因为氮元素对氨基酸尤为重要,所以进一步分析了分蘖期WYD4叶片的相关基因表达(图5)。
研究发现,AsnB、ASL、NadB、GOT2、GLT1和GLnA的显著差异表达主要表现在氮素处理后期21 d,说明水稻可能是通过上述6个基因氮素处理后期的应答来调控丙氨酸、天冬氨酸和谷氨酸的代谢途径。
2.5 氮素对糖类代谢的影响
碳氮循环一直是光合植物研究的重点,氮素如何具体调控糖类代谢还需完善。研究结果表明,氮素在初期对半乳糖代谢有显著影响,而在中期对氨基糖和核苷酸糖的代谢有显著影响(图4),对以上2种代谢途径的差异表达进行了进一步的研究。
研究发现,galA、HK、galE(LOC_Os05g51670)、malZ的差异表达主要表现在氮处理前期7 d,HEXA_B、GNPNAT1、GlmS、UAP1、RGP、XYL4、AXS、UXS1、UGDH、RHM、galE(LOC_Os09g15420)和GAUT的差异表达主要表现在氮处理中期14 d,而且氮处理前期(7 d)和氮处理中期(14 d)差异表达上调的基因数远多于差异表达下调的基因数(图6),说明水稻可能是通过上述16个基因氮素处理前中期的应答来调控半乳糖、氨基糖和核苷酸糖的代谢途径的。
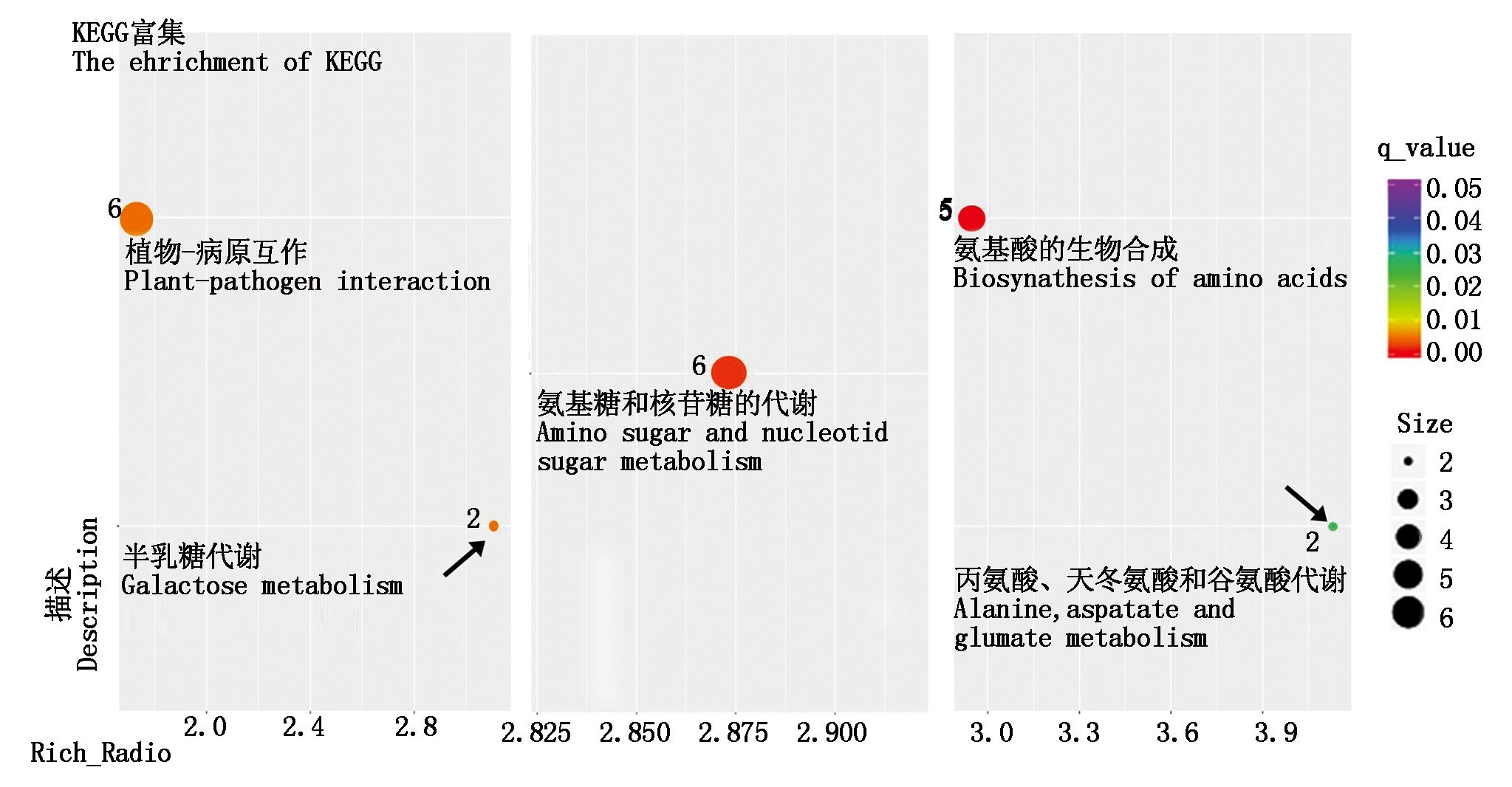
图4 KEGG富集统计
Fig.4 KEGG enrichment statistics
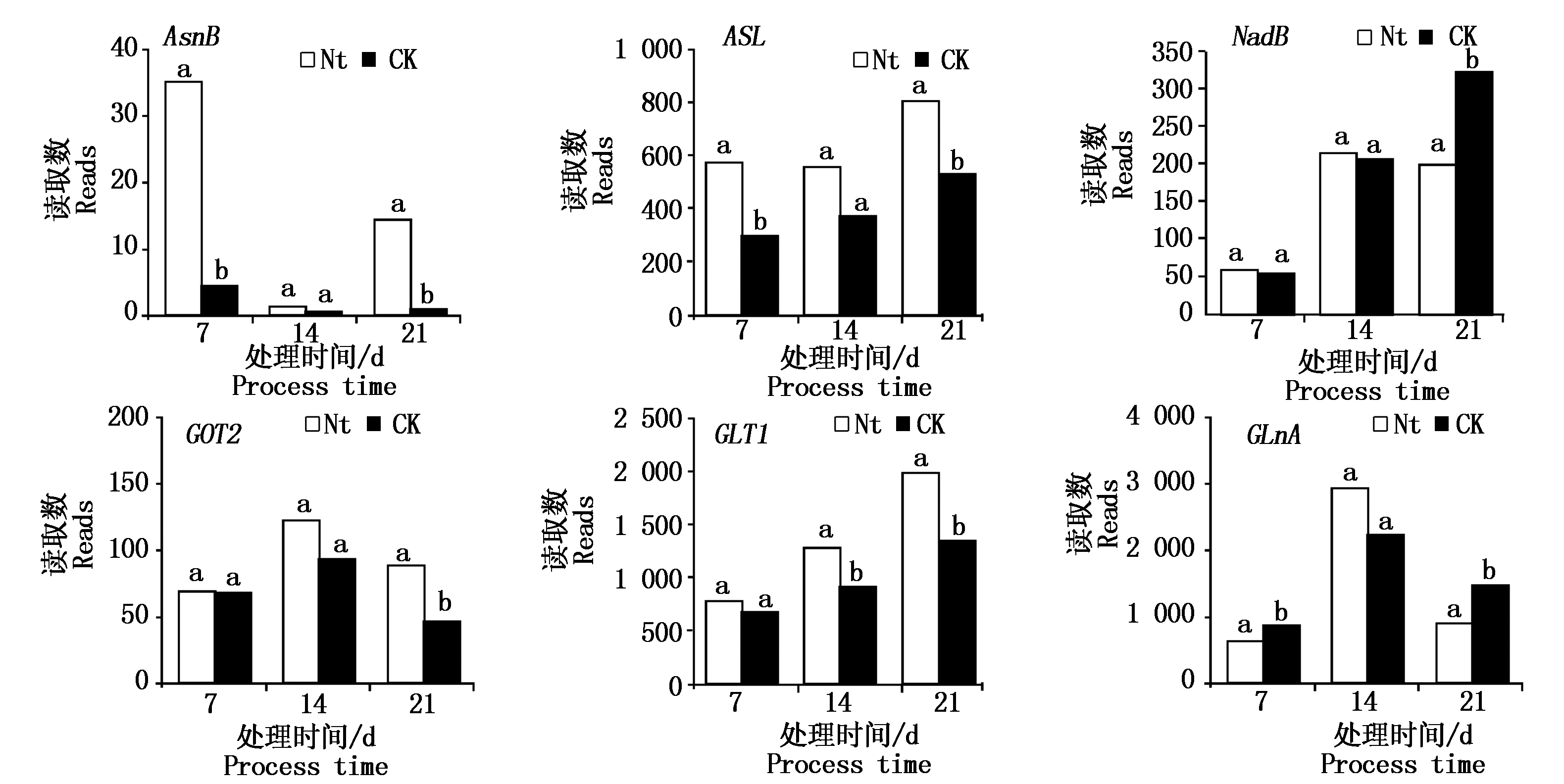
不同小写字母表示差异显著水平(P<0.05)。图6同。
Different lowercase letters indicate significantly different level(P<0.05). The same as Fig.6.
图5 氮处理下丙氨酸、天冬氨酸和谷氨酸的代谢途径差异基因表达
Fig.5 DEGs in the metabolic pathways of alanine,aspartic acid and glutamic acid under nitrogen
2.6 氮素应答的互作蛋白
STRING是收录多个物种预测的和试验验证的蛋白质-蛋白质互作的数据库,通过数据库对氮处理下分蘖期水稻叶片进行互作蛋白预测,发现了35种相互作用的蛋白质,其中11种与氨基酸糖和核苷酸糖途径相互作用,24种与丙氨酸、天冬氨酸和谷氨酸代谢途径相互作用(表1-2)。
发现与UXS1、AsnB和ASL相互作用的蛋白数量最多,这可能是碳氮循环的关键基因。这些结果有助于研究氮对水稻生长发育的影响,为水稻的栽培和调控提供参考。
3 讨论与结论
氮素作为植物生长发育的必需元素,与植物体内的蛋白质、核酸、磷脂和激素等含氮物质密切相关。但是,土壤中的氮肥只有约35%能被植物吸收,氮素的吸收、转运、同化和再分配等过程受到多个基因和环境因子的共同控制[23]。在本研究中,氮处理引起了分蘖期水稻叶片中大量涉及代谢过程、细胞过程、细胞组分、催化活性和结合等方面的基因发生转录变化。
氮的同化过程是指氮素在根和叶片中经系列酶促反应转变成谷氨酰胺、天冬酰胺、谷氨酸、天冬氨酸等有机氮的过程。在植物体中,大部分的氨通过GLT1(LOC_Os01g48960.1,谷氨酸合成酶基因)和谷氨酰胺-2-氧戊二酸氨基转移酶的共同作用被同化为有机形态,通过ASN合成酶作用Gln与Asp反应生成Asn和Glu,目前已经发现抑制GLT1会降低氨基酸含量[24],而ASN合成酶在调节氮向有机氮池的流动方面起着重要的作用,ASN合成酶催化反应的所有底物和产物都是植物在韧皮部运输氮的代谢过程中主要的氮载体[25]。同时Asn易受不同生理和环境条件导致剧烈变化[26-29]。在拟南芥中过表达ASN合成酶会导致植株内ASN含量升高同时在限氮培养基上幼苗的耐受性提高[30],但在水稻中的报道还很少。在本研究中,发现分蘖期水稻叶片在氮处理后期AsnB(ASN合成酶基因)显著升高,GLT1也显著升高,这可能导致ASN含量的升高。同时笔者还发现在氮处理下ASL(精氨琥珀酸裂解酶基因)和GOT2(天冬氨酸转氨酶基因)显著升高,而NadB(天冬氨酸氧化酶基因)和GLnA(谷氨酰氨合成酶基因)显著下降,说明氮素通过这些基因在后期的差异表达来影响丙氨酸、天冬氨酸和谷氨酸的代谢,因为这些酶对氮素的响应相对迟缓,所以可以考虑通过长期缓释的氮素处理来调控氮代谢途径酶活性。
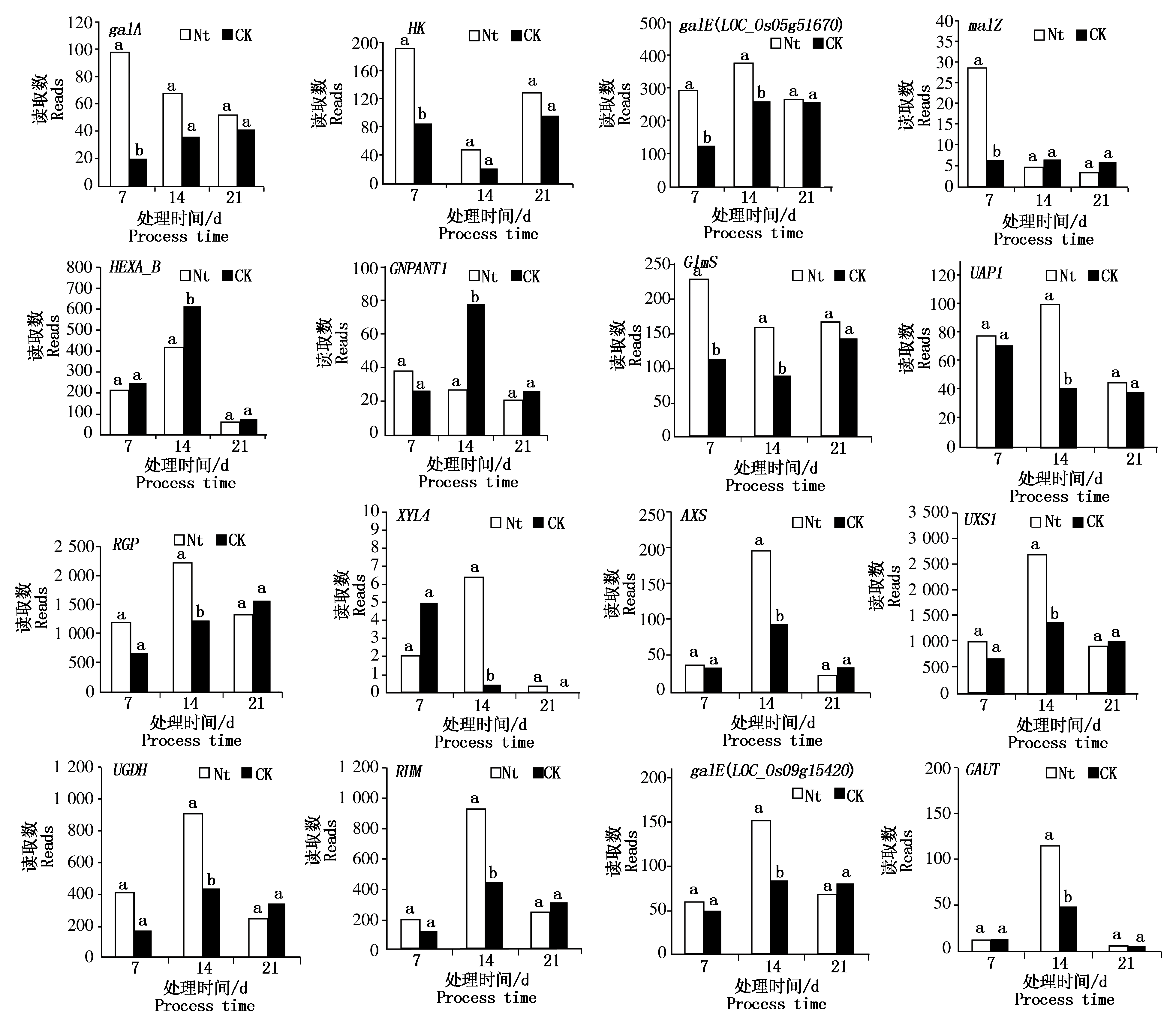
图6 氮处理下半乳糖、氨基糖和核苷酸糖代谢途径差异基因表达
Fig.6 DEGs in the metabolic pathways of galactose,aminoglycan and nucleotide sugar under nitrogen treatment
表1 预测的氨基糖和核苷酸糖代谢互作蛋白
Tab.1 Candidate protein interacting with proteins in amino sugar and nucleotide sugar pathway
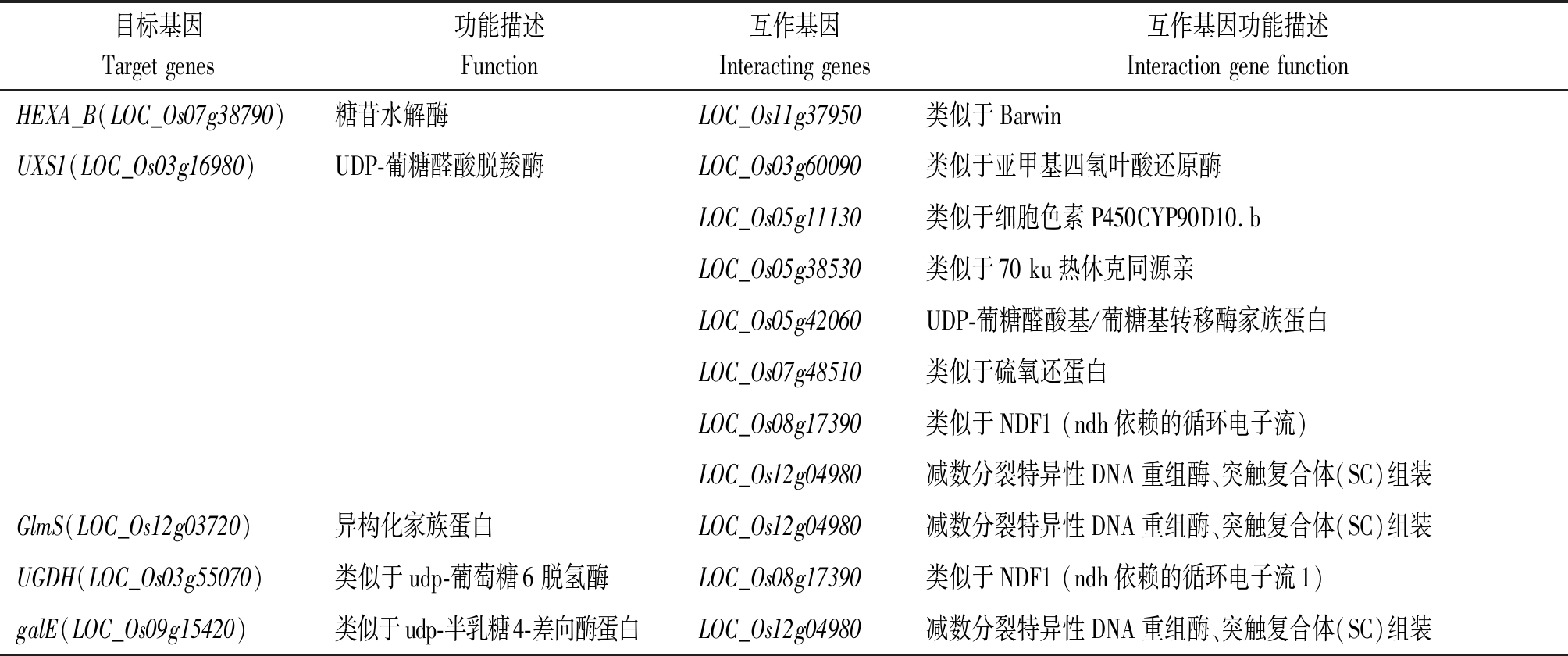
目标基因Targetgenes功能描述Function互作基因Interactinggenes互作基因功能描述InteractiongenefunctionHEXA_B(LOC_Os07g38790)糖苷水解酶LOC_Os11g37950类似于BarwinUXS1(LOC_Os03g16980)UDP-葡糖醛酸脱羧酶LOC_Os03g60090类似于亚甲基四氢叶酸还原酶LOC_Os05g11130类似于细胞色素P450CYP90D10.bLOC_Os05g38530类似于70ku热休克同源亲LOC_Os05g42060UDP-葡糖醛酸基/葡糖基转移酶家族蛋白LOC_Os07g48510类似于硫氧还蛋白LOC_Os08g17390类似于NDF1(ndh依赖的循环电子流)LOC_Os12g04980减数分裂特异性DNA重组酶、突触复合体(SC)组装GlmS(LOC_Os12g03720)异构化家族蛋白LOC_Os12g04980减数分裂特异性DNA重组酶、突触复合体(SC)组装UGDH(LOC_Os03g55070)类似于udp-葡萄糖6脱氢酶LOC_Os08g17390类似于NDF1(ndh依赖的循环电子流1)galE(LOC_Os09g15420)类似于udp-半乳糖4-差向酶蛋白LOC_Os12g04980减数分裂特异性DNA重组酶、突触复合体(SC)组装
注:基因功能描述参考The Rice Annotation Project Database(https://rapdb.dna.affrc.go.jp/index.html)。表2同。
Note:The small letter a means gene function description from The Rice Annotation Project Database (https://rapdb.dna.affrc.go.jp/index.html). The same as Tab.2.
表2 预测的丙氨酸、天冬氨酸和谷氨酸代谢互作蛋白
Tab.2 Candidate proteins interacting with proteins in alanine,aspartic acid and glutamate metabolism
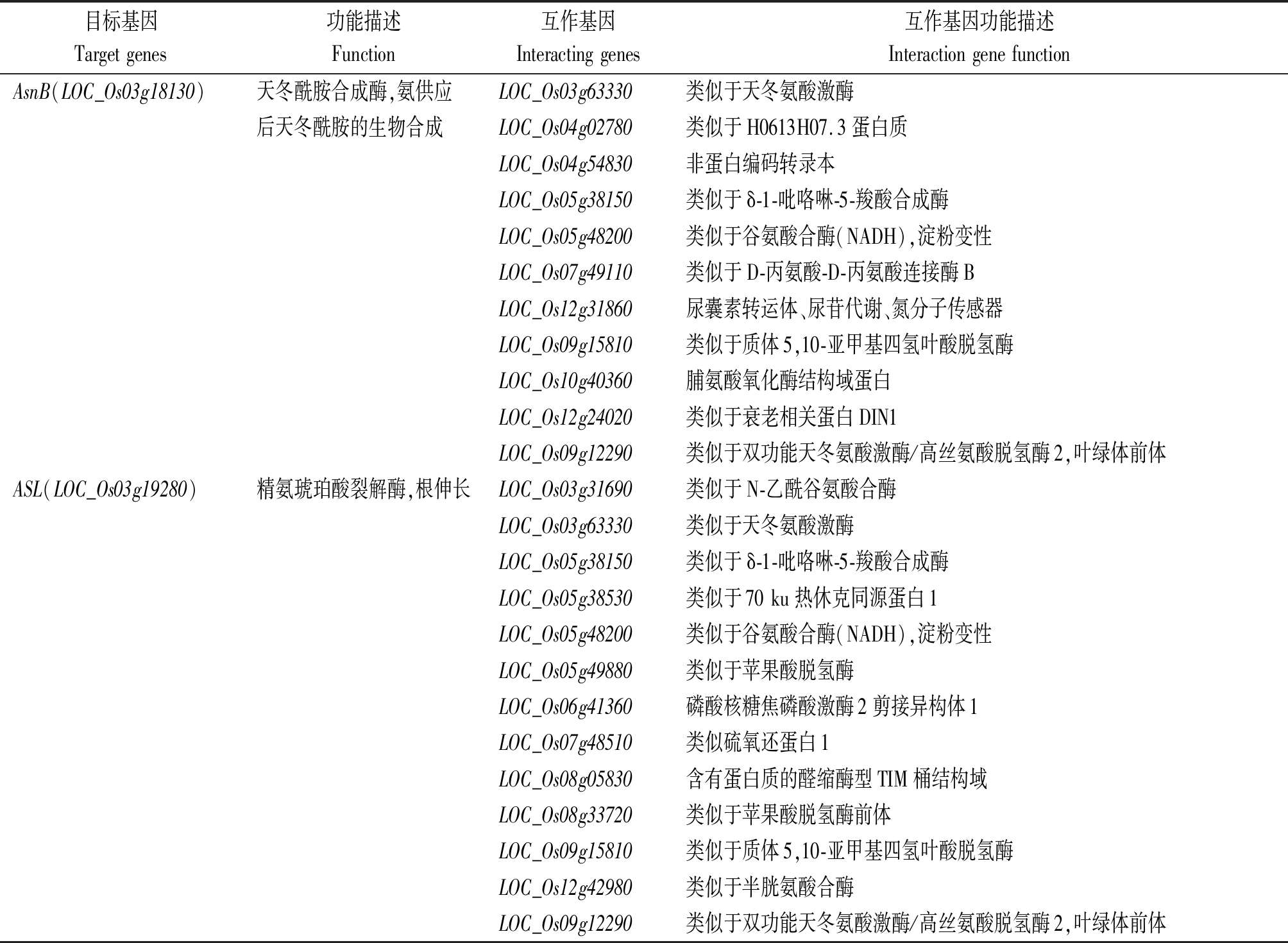
目标基因Targetgenes功能描述Function互作基因Interactinggenes互作基因功能描述InteractiongenefunctionAsnB(LOC_Os03g18130)天冬酰胺合成酶,氨供应LOC_Os03g63330类似于天冬氨酸激酶后天冬酰胺的生物合成LOC_Os04g02780类似于H0613H07.3蛋白质LOC_Os04g54830非蛋白编码转录本LOC_Os05g38150类似于δ-1-吡咯啉-5-羧酸合成酶LOC_Os05g48200类似于谷氨酸合酶(NADH),淀粉变性LOC_Os07g49110类似于D-丙氨酸-D-丙氨酸连接酶BLOC_Os12g31860尿囊素转运体、尿苷代谢、氮分子传感器LOC_Os09g15810类似于质体5,10-亚甲基四氢叶酸脱氢酶LOC_Os10g40360脯氨酸氧化酶结构域蛋白LOC_Os12g24020类似于衰老相关蛋白DIN1LOC_Os09g12290类似于双功能天冬氨酸激酶/高丝氨酸脱氢酶2,叶绿体前体ASL(LOC_Os03g19280)精氨琥珀酸裂解酶,根伸长LOC_Os03g31690类似于N-乙酰谷氨酸合酶LOC_Os03g63330类似于天冬氨酸激酶LOC_Os05g38150类似于δ-1-吡咯啉-5-羧酸合成酶LOC_Os05g38530类似于70ku热休克同源蛋白1LOC_Os05g48200类似于谷氨酸合酶(NADH),淀粉变性LOC_Os05g49880类似于苹果酸脱氢酶LOC_Os06g41360磷酸核糖焦磷酸激酶2剪接异构体1LOC_Os07g48510类似硫氧还蛋白1LOC_Os08g05830含有蛋白质的醛缩酶型TIM桶结构域LOC_Os08g33720类似于苹果酸脱氢酶前体LOC_Os09g15810类似于质体5,10-亚甲基四氢叶酸脱氢酶LOC_Os12g42980类似于半胱氨酸合酶LOC_Os09g12290类似于双功能天冬氨酸激酶/高丝氨酸脱氢酶2,叶绿体前体
碳氮循环也一直是研究的重点,糖类是碳氮循环的关键,Kim等[31]在水稻细胞悬浮液中提取得到galA(E3.2.1.22),galA可催化寡糖和多聚半乳甘露聚糖水解1,6-连接的a-半乳糖残基[32-33]。本研究发现,在氮处理前期分蘖期水稻叶片中galA的表达显著上调,同时调控半乳糖代谢的HK、malZ、galE(LOC_Os05g51670)的表达也显著上调,在施氮中期调控氨基糖和核苷酸糖的13个基因也差异表达,但是目前尚未有报道氮素调控这些基因的研究进展,这些基因可能是碳氮代谢的桥梁分子,为农业上施用氮肥精准调控水稻品质提供参考,但对于碳氮代谢的具体作用机制还有待研究。
氮素应答的差异表达基因有3 870个,主要与代谢过程、细胞过程、细胞组分、催化活性和结合等方面有关。在氮素处理后,前期应答的差异表达基因galA、HK、galE(LOC_Os05g51670)、malZ主要参与半乳糖代谢途径;中期应答的差异表达基因HEXA_B、GNPNAT1、GlmS、UAP1、RGP、XYL4、AXS、UXS1、UGDH、RHM、galE(LOC_Os09g15420)和GAUT主要参与氨基糖和核苷酸糖代谢途径;后期应答的差异表达基因AsnB、ASL、NadB、GOT2、GLT1和GlnA主要参与丙氨酸、天冬氨酸和谷氨酸的代谢途径;并且有35个蛋白与上述差异基因编码蛋白互作,说明氮素处理后不同时间点应答的代谢途径是有差异的,这些结果为水稻栽培过程中氮素调控提供了转录水平的参考依据。
[1] Khush G S. Green revolution:preparing for the 21st century[J]. Genome,1999,42(4):646-655. doi:10.1139/gen-42-4-646.
[2] Pingali P L. Green revolution:impacts,limits,and the path ahead[J]. Proceedings of the National Academy of Sciences of the United States of America,2012,109(31):12302-12308. doi:10.1073/pnas.0912953109.
[3] Evenson R E,Gollin D. Assessing the impact of the green revolution,1960 to 2000[J]. Science,2003,300(5620):758-762. doi:10.1126/science.1078710.
[4] Hedden P. The genes of the green revolution[J]. Trends in Genetics,2003,19(1):5-9. doi:10.1016/s0168-9525(02)00009-4.
[5] Li S,Tian Y H,Wu K,Ye Y F,Yu J P,Zhang J Q,Liu Q,Hu M Y,Li H,Tong Y P,Harberd N P,Fu X D. Modulating plant growth-metabolism coordination for sustainable agriculture[J]. Nature,2018,560(7720):595-600.doi:10.1038/s41586-018-0415-5.
[6] 黄东迈,朱培立,高家骅. 有机、无机态肥料氮在水田和旱地的残留效应[J]. 中国科学(B辑 化学 生物学 农学 医学 地学),1982,12(10):907-912.
Huang D M,Zhu P L,Gao J Y. Residual effect of organic and inorganic fertilizer nitrogen on paddy field and upland[J]. Scientia Sinica(Chimica),1982,12(10):907-912.
[7] 熊正琴,邢光熹,沈光裕,孙德玲. 太湖地区湖、河和井水中氮污染状况的研究[J]. 农村生态环境,2002,18(2):29-33. doi:10.3969/j.issn.1673-4831.2002.02.007.
Xiong Z Q,Xing G X,Shen G Y,Sun D L. Non-point source N pollution of lakes,rivers and wells in the Taihu Lake region[J]. Journal of Ecology and Rural Environment,2002,18(2):29-33.
[8] 周学志. 地下水开发利用的环境问题及防治措施研究[J]. 环境科学丛刊,1992,13(3):1-32.
Zhou X Z. Study on environmental problems and prevention measures of groundwater development and utilization[J]. Chinese Journal of Environmental Engineering,1992,13(3):1-32.
[9] 邢光熹,颜晓元.中国农田N2O排放的分析估算与减缓对策[J].农村生态环境,2000,16(4):1-6.doi:10.3969/j.issn.1673-4831.2000.04.001.
Xing G X,Yan X Y.Analysis and estimation of N2O emissions from croplands in China and its mitigation options[J]. Journal of Ecology and Rural Environment,2000,16(4):1-6.
[10] Baker A J M,Mcgrath S P,Sidoli C M D,Reeves R D. The possibility of in situ heavy metal decontamination of polluted soils using crops of metal-accumulating plants[J]. Resources Conservation and Recycling,1994,11(1-4):41-49. doi:10.1016/0921-3449(94)90077-9.
[11] Wu K,Wang S S,Song W Z,Zhang J Q,Wang Y,Liu Q,Yu J P,Ye Y F,Li S,Chen J F,Zhao Y,Wang J,Wu X K,Wang M Y,Zhang Y J,Liu B M,Wu Y J,Harberd N P,Fu X D. Enhanced sustainable green revolution yield via nitrogen-responsive chromatin modulation in rice[J]. Science,2020,367(6478):2046. doi:10.1126/science.aaz2046.
[12] Wei H Y,Zhu Y,Qiu S,Han C,Hu L,Xu D,Zhou N B,Xing Z P,Hu Y J,Cui P Y,Dai Q G,Zhang H C. Combined effect of shading time and nitrogen level on grain filling and grain quality in japonica super rice[J]. Journal of Integrative Agriculture,2018,17(11):2405-2417. doi:10.1016/S2095-3119(18)62025-8.
[13] 孙新,施卫明. 氮素形态对水稻蔗糖分配的影响[J]. 安徽农业科学,2008,36(13):5344-5346,5677. doi:10.3969/j.issn.0517-6611.2008.13.037.
Sun X,Shi W M. Influence of different nitrogen forms on the sucrose partitioning characteristics of rice(Oryza sativa L.)plants[J]. Journal of Anhui Agri Sci,2008,36(13):5344-5346,5677.
[14] 刘洪,李之林,徐振江,王维,任永浩,肖立中. 施氮对大穗型两系杂交水稻弱势粒碳氮代谢的影响[J]. 湖北农业科学,2013,52(8):1755-1759. doi:10.3969/j.issn.0439-8114.2013.08.005.
Liu H,Li Z L,Xu Z J,Wang W,Ren Y H,Xiao L Z. Effects of nitrogen application on metabolism of carbohydrate and nitrogen in the inferior grains of hybrid rice with large panicle[J]. Hubei Agricultural Sciences,2013,52(8):1755-1759.
[15] 王德仁,卢婉芳,温怀南,杨志根. 水稻高产、高效、高氨基酸含量及营养价的施氮量优化[J]. 中国农学通报,1995,11(2):24-27.
Wang D R,Lu W F,Wen H N,Yang Z G. Optimization of napplication for high yield,high beneift,high amino aeid content and nutrient value in rice[J]. Chinese Agrieultural Science Bulletin,1995,11(2):24-27.
[16] 常二华,张慎凤,王志琴,王学明,杨建昌. 结实期氮磷营养水平对水稻根系和籽粒氨基酸含量的影响[J]. 作物学报,2008,34(4):612-618. doi:10.3321/j.issn:0496-3490.2008.04.012.
Chang E H,Zhang S F,Wang Z Q,Wang X M,Yang J C. Effect of nitrogen and phosphorus on the amino acids in root exudates and grains of rice during grain filling[J]. Acta Agronomica Sinica,2008,34(4):612-618.
[17] Hu B, Wang W, Ou S J, Tang J Y, Li H, Che R H, Zhang Z H, Chai X Y, Wang H R, Wang Y Q, Liang C Z, Liu L C, Piao Z Z, Deng Q Y, Deng K, Xu C, Liang Y, Zhang L H, Li L G, Chu C C. Variation in NRT1.1B contributes to nitrate-use divergence between rice subspecies[J]. Nature Genetics,2015,47(7):834-838. doi:10.1038/ng.3337.
[18] Wang W,Hu B,Yuan D Y,Liu Y Q,Che R H,Hu Y C,Ou S J,Liu Y X,Zhang Z H,Wang H R,Li H,Jiang Z M,Zhang Z L,Gao X K,Qiu Y H,Meng X B,Liu Y X,Bai Y,Liang Y,Wang Y Q,Zhang L H,Li L G,Sodmergen,Jing H C,Li J Y,Chu C C. Expression of the nitrate transporter gene OsNRT1.1A/OsNPF6.3 confers high yield and early maturation in rice[J].The Plant Cell,2018,30(3):638-651. doi:10.1105/tpc.17.00809 .
[19] Chen J G,Zhang Y,Tan Y W,Zhang M,Zhu L L,Xu G H,Fan X R. Agronomic nitrogen-use efficiency of rice can be increased by driving OsNRT2.1 expression with the OsNAR2.1 promoter[J].Plant Biotechnology Journal,2016,14(8):1705-1715. doi:10.1111/pbi.12531.
[20] Fan X R,Tang Z,Tan Y W,Zhang Y,Luo B B,Yang M,Lian X M,Shen Q R,Miller A J,Xu G H.Overexpression of a pH-sensitive nitrate transporter in rice increases crop yields[J]. Proceedings of the National Academy of Sciences,2016,113(26):7118-7123. doi:10.1073/pnas.1525184113.
[21] Fang Z M,Xia K F,Yang X,Grotemeyer M S,Meier S,Rentsch D,Xu X L,Zhang M Y. Altered expression of the PTR/NRT1 homologue OsPTR9 affects nitrogen utilization efficiency,growth and grain yield in rice[J]. Plant Biotechnology Journal,2013,111(4):446-458. doi:10.1111/pbi.12031.
[22] Shu S H,Chen B,Zhou M C,Zhao X M,Xia H Y,Wang M. De novo sequencing and transcriptome analysis of Wolfiporia cocos to reveal genes related to biosynthesis of triterpenoids[J]. PLoS One,2013,8(8):e71350. doi:10.1371/journal.pone.0071350.
[23] 刘学英,李姗,吴昆,刘倩,高秀华,傅向东. 提高农作物氮肥利用效率的关键基因发掘与应用[J]. 科学通报,2019,64(25):2633-2640. doi:10.1360/TB-2019-0043.
Liu X Y,Li S,Wu K,Liu Q,Gao X H,Fu X D. Sustainable crop yields from the coordinated modulation of plant growth and nitrogen metabolism[J]. Chin Sci Bull,2019,64(25):2633-2640.
[24] Lam H M,Peng S S Y,Coruzzi G M. Metabolic regulation of the gene encoding glutamine-dependent asparagine synthetase in Arabidopsis thaliana[J]. Plant Physiol,1994,106(4):1347-1357. doi:10.1104/pp.106.4.1347.
[25] Lu Y E,Luo F,Yang M,Li X H,Lian X M. Suppression of glutamate synthase genes significantly affects carbon and nitrogen metabolism in rice(Oryza sativa L.)[J]. Science China Life Sciences,2011,54(7):651-663. doi:10.1007/s11427-011-4191-9.
[26] Lea P J,Miflin B J .16-transport and metabolism of asparagine and other nitrogen compounds within the plant[M].Amino Acids and Derivatives.Pittsburgh:Academic Press,1980:569-607.doi:10.1016/B978-0-12-675405-6.50022-x.
[27] Sieciechowicz K A,Joy K W,Ireland R J. The metabolism of asparagine in plants[J]. Phytochemistry,1988,27(3):663-671. doi:10.1016/0031-9422(88)84071-8.
[28] Lea P J,Robinson S A,Stewart G R . 4-The enzymology and metabolism of glutamine,glutamate,and asparagine[J]. Intermediary Nitrogen Metabolism,1990,16:121-159.doi:10.1016/B978-0-08-092616-2.50010-3.
[29] Lam H M,Coschigano K,Schultz C,Melo-Oliveira R,Tjaden G,Oliveira I,Ngai N,Hsieh M H,Coruzzi G. Use of Arabidopsis mut ants and genes to study amide amino acid biosynthesis[J]. The Plant Cell,1995,7(7):887-898. doi:10.1105/tpc.7.7.887.
[30] Lam H M,Wong P,Chan H K,Yam K M,Chen L,Chow C M,Coruzzi G M. Overexpression of the ASN1 gene enhances nitrogen status in seeds of Arabidopsis[J]. Plant Physiology,2003,132(2):926-935. doi:10.1104/pp.103.020123.
[31] Kim W D,Kobayashi O,Kaneko S,Sakakibara Y,Park G G,Kusakabe I, Tanaka H, Kobayashi H. α-Galactosidase from cultured rice(Oryza sativa L. var. Nipponbare)cells[J]. Phytochemistry,2002,61(6):621-630. doi:10.1016/S0031-9422(02)00368-0.
[32] Shibuya H,Kobayashi H,Sato T,Kim W S,Yoshida S,Kaneko S,Kasamo K,Kusakabe I. Purification,characterization,and cDNA cloning of a novel α-galactosidase from Mortierella vinacea[J]. Bioscience,Biotechnology and Biochemistry,1997,61(4):592-598. doi:10.1271/bbb.61.592.
[33] Shibuya H,Kobayashi H,Park G G,Komatusu Y,Sato T,Kaneko R,Nagasaki H,Yoshidsa S,Kasamo K,Kusakabe I . Purification and some properties of α-Galactosidase from Penicillium purpurogenum[J]. Bioscience,Biotechnology and Biochemistry,1995,59(12):2333-2335. doi:10.1271/bbb.59.2333.
