甘薯(Ipomoea batatas L.)是继小麦、水稻、玉米、马铃薯、大麦和木薯之后的世界第七大粮食作物,在世界100多个国家都被广泛种植[1]。特别在发展中国家,甘薯在粮食安全中扮演着重要的角色。近年来,甘薯被广泛用作动物饲料、工业原料,在许多发达国家作为保健食品也越来越受到消费者的青睐[2]。
甘薯最早是在美洲驯化后[3-4],再传播到世界其他地方的。目前甘薯属已经发现近700个物种,其中大部分来自美洲[3],甘薯是甘薯属中唯一被作为农作物种植的植物。据统计,全世界累计保存的甘薯属种质资源约26 000份,其中甘薯种质资源材料约8 000份[5-6]。甘薯近缘野生种是甘薯遗传改良的重要基因资源[7],越来越受到研究者的关注,但是目前甘薯与野生种的遗传关系仍未明确,因此深入研究甘薯及其相关野生种的遗传关系十分重要。
迄今为止,SSR(Simple sequence repeats)、RAPD(Randomly amplified polymorphic DNA)和AFLP(Amplified fragment length polymorphism)等分子标记在甘薯遗传多样性分析、遗传连锁构建和QTL定位研究等方面得到了广泛应用[8-11],发挥了重要作用。但与其他主要作物相比,适合甘薯遗传研究的分子标记非常有限。虽然近年来SLAF-seq(Specific-locus amplified fragment sequencing)、RAD-seq(Restriction-site associated DNA sequencing)、GBS-seq(Genotyping by sequencing)等基于简化基因组测序的分子标记技术开始在甘薯研究中应用[12-15],但是相比其他传统分子标记技术,昂贵的成本限制了其广泛应用。2009年,Collard和Mackill[16]根据植物基因ATG起始密码子翻译起始位点的侧翼保守序列设计单引物,用来扩增偏向候选功能基因区显性多态性标记,建立了启动密码子靶向(Start codon targeted polymorphism,SCoT)标记,该技术在PCR反应中使用单条引物,扩增产物为显性标记,使用琼脂糖凝胶电泳进行分离,具有操作简单、成本低,且基因关联度高等优点,已在许多植物研究中得到应用[17],但目前在甘薯研究应用的报道较少。本研究旨在利用SCoT分子标记技术对甘薯及其野生种进行扫描,揭示甘薯及其野生种之间的遗传多样性,分析甘薯及其野生种之间的遗传关系,为SCoT标记技术在甘薯研究中的应用提供参考。
1 材料和方法
1.1 试验材料
本研究共收集到22份甘薯及其野生种种质材料,其中包括8个甘薯种质材料,1份甘薯和三浅裂野牵牛(Ipomoea trifida L.)杂交后代材料,6份三浅裂野牵牛种质材料,7份三裂叶薯(Ipomoea triloba L.)种质材料,全部试验材料均由四川省农科院生物技术核技术研究所收集保存(表1)。
表1 甘薯及其野生种种质材料
Tab.1 The materials of sweet potato and its wild germplasm
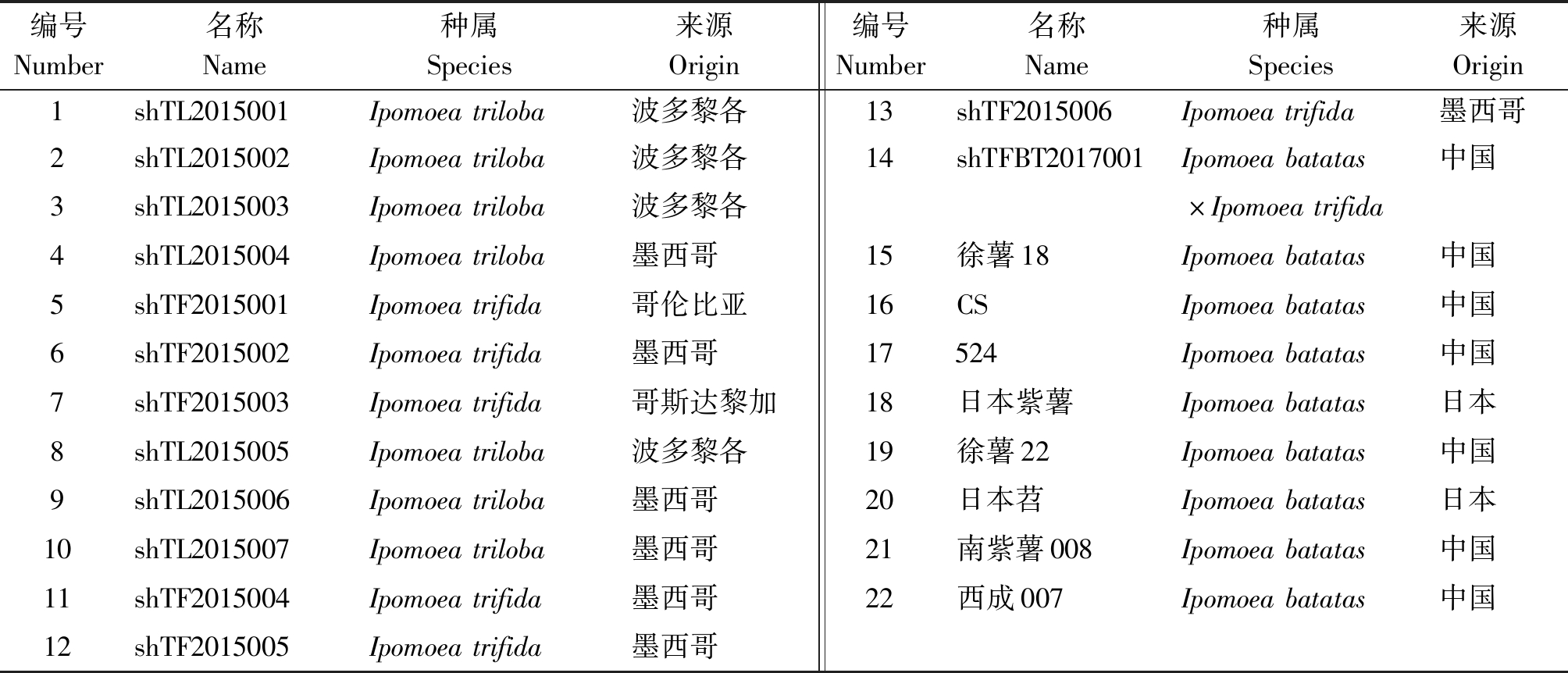
编号Number名称Name种属Species来源Origin编号Number名称Name种属Species来源Origin1shTL2015001Ipomoeatriloba波多黎各13shTF2015006Ipomoeatrifida墨西哥2shTL2015002Ipomoeatriloba波多黎各14shTFBT2017001Ipomoeabatatas中国3shTL2015003Ipomoeatriloba波多黎各×Ipomoeatrifida4shTL2015004Ipomoeatriloba墨西哥15徐薯18Ipomoeabatatas中国5shTF2015001Ipomoeatrifida哥伦比亚16CSIpomoeabatatas中国6shTF2015002Ipomoeatrifida墨西哥17524Ipomoeabatatas中国7shTF2015003Ipomoeatrifida哥斯达黎加18日本紫薯Ipomoeabatatas日本8shTL2015005Ipomoeatriloba波多黎各19徐薯22Ipomoeabatatas中国9shTL2015006Ipomoeatriloba墨西哥20日本苕Ipomoeabatatas日本10shTL2015007Ipomoeatriloba墨西哥21南紫薯008Ipomoeabatatas中国11shTF2015004Ipomoeatrifida墨西哥22西成007Ipomoeabatatas中国12shTF2015005Ipomoeatrifida墨西哥
1.2 试验方法
1.2.1 基因组DNA提取 2018年22份参试甘薯及其野生种材料种植于四川省农业科学院生物技术核技术研究所成都试验基地。2018年9月,取各材料幼嫩叶片0.3 g,按照CTAB(十六烷基三甲基溴化铵)法提取全基因组DNA[17-18]。用含有20 ng/μL RNA酶的1×TE缓冲液溶解DNA,除去DNA中的RNA。抽取2 μL加5 μL溴酚蓝,用1%琼脂糖检测DNA质量。使用Scandrop(Analyticgena公司)微量核酸测定仪检测DNA浓度,根据测定DNA浓度,将DNA原液稀释到50 ng/μL,-20 ℃冷冻待用。
1.2.2 PCR反应条件及引物合成 本研究所使用的43条SCoT引物信息均来自于Collard和Mackill发表文章[16],由生工生物工程(上海)股份有限公司合成,PCR反应程序及体系均根据Collard和Mackill、冯俊彦等的方法略有修改[16-17](表2)。
表2 本研究使用的SCoT引物
Tab.2 The information of SCoT primer sused in this study
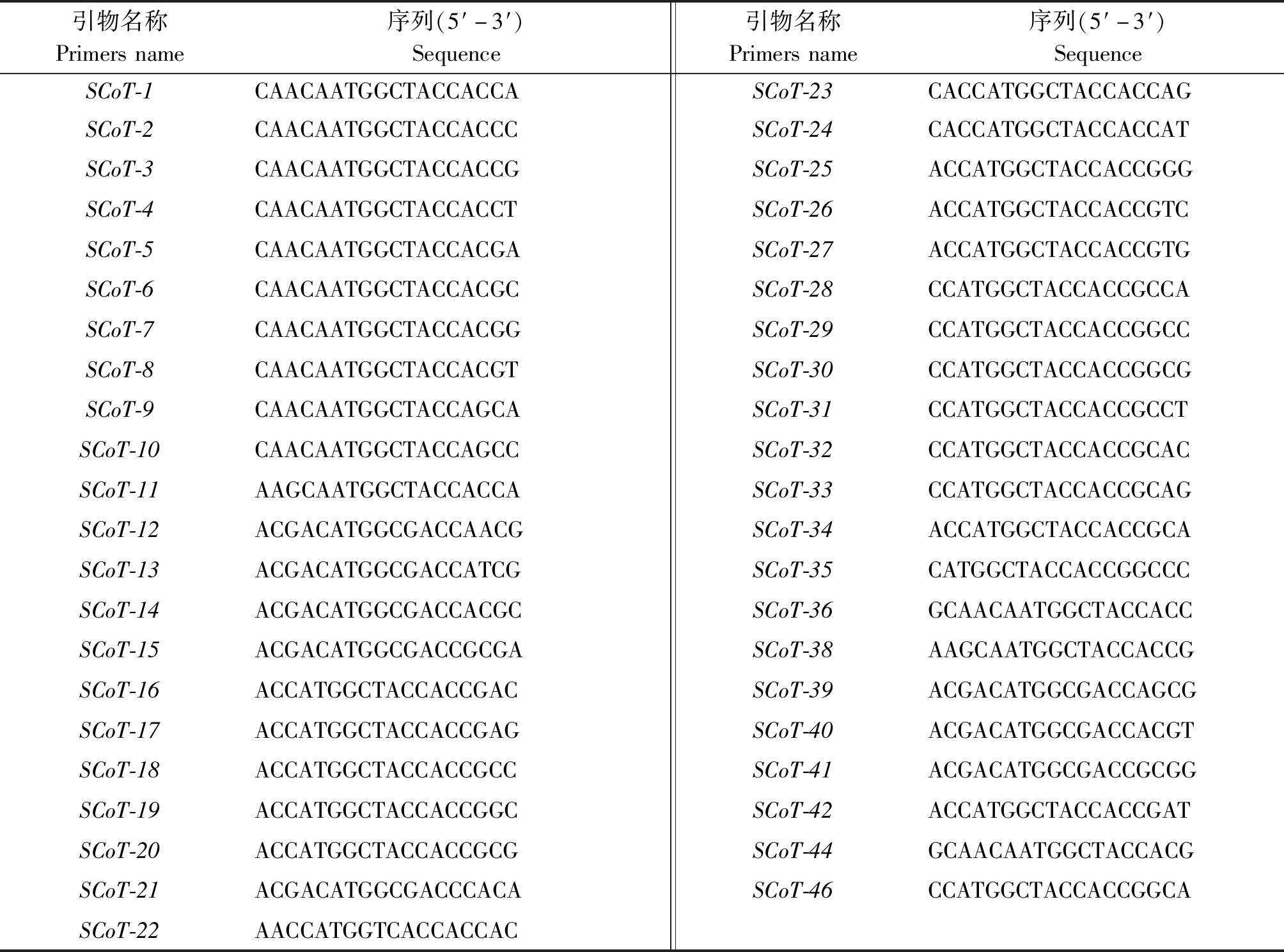
引物名称Primersname序列(5′-3′)Sequence引物名称Primersname序列(5′-3′)SequenceSCoT-1CAACAATGGCTACCACCASCoT-23CACCATGGCTACCACCAGSCoT-2CAACAATGGCTACCACCCSCoT-24CACCATGGCTACCACCATSCoT-3CAACAATGGCTACCACCGSCoT-25ACCATGGCTACCACCGGGSCoT-4CAACAATGGCTACCACCTSCoT-26ACCATGGCTACCACCGTCSCoT-5CAACAATGGCTACCACGASCoT-27ACCATGGCTACCACCGTGSCoT-6CAACAATGGCTACCACGCSCoT-28CCATGGCTACCACCGCCASCoT-7CAACAATGGCTACCACGGSCoT-29CCATGGCTACCACCGGCCSCoT-8CAACAATGGCTACCACGTSCoT-30CCATGGCTACCACCGGCGSCoT-9CAACAATGGCTACCAGCASCoT-31CCATGGCTACCACCGCCTSCoT-10CAACAATGGCTACCAGCCSCoT-32CCATGGCTACCACCGCACSCoT-11AAGCAATGGCTACCACCASCoT-33CCATGGCTACCACCGCAGSCoT-12ACGACATGGCGACCAACGSCoT-34ACCATGGCTACCACCGCASCoT-13ACGACATGGCGACCATCGSCoT-35CATGGCTACCACCGGCCCSCoT-14ACGACATGGCGACCACGCSCoT-36GCAACAATGGCTACCACCSCoT-15ACGACATGGCGACCGCGASCoT-38AAGCAATGGCTACCACCGSCoT-16ACCATGGCTACCACCGACSCoT-39ACGACATGGCGACCAGCGSCoT-17ACCATGGCTACCACCGAGSCoT-40ACGACATGGCGACCACGTSCoT-18ACCATGGCTACCACCGCCSCoT-41ACGACATGGCGACCGCGGSCoT-19ACCATGGCTACCACCGGCSCoT-42ACCATGGCTACCACCGATSCoT-20ACCATGGCTACCACCGCGSCoT-44GCAACAATGGCTACCACGSCoT-21ACGACATGGCGACCCACASCoT-46CCATGGCTACCACCGGCASCoT-22AACCATGGTCACCACCAC
具体PCR反应体系:总体系20 μL,包括100 ng参试植物基因组DNA,1×Buffer,1.5 mmol/L MgCl2,200 μmol/L dNTP,0.25 μmol/L引物,0.5 UTaq酶。所需试剂购自北京天根生物技术公司。
SCoT反应程序:94 ℃预变性3 min;94 ℃变性1 min,55~58 ℃退火1 min,72 ℃延伸2 min,35个循环;最后72 ℃终延伸5 min,慢慢冷却至12 ℃[16-17]。
1.2.3 PCR扩增产物检测 PCR产物中加入5 μL上样缓冲液(40%蔗糖,0.025%溴酚蓝),用2.0%(m/V)的琼脂糖凝胶进行电泳分离。再荧光成像仪上检测拍照,统计结果。
1.3 数据统计及处理
根据电泳结果中条带的有、无,采用二进位制进行记录。在相同迁移率处有带记为“1”,无带记为“0”。利用Excle 2007软件对数据进行初步处理,并进行数据相应格式转化。利用Ntsys-pc 2.1软件(http://www.exetersoftware.com/cat/ntsyspc/ntsyspc.html)进行遗传多样性分析,计算遗传距离。使用Jacard方法计算遗传距离,用非加权配对算术平均法UPGMA(Unweighted pair grouping method with arithmetic mean)进行聚类分析,使用Neighbor-joining 算法绘制遗传进化树。利用Ntsys-pc 2.1软件对遗传矩阵和相关矩阵进行相关性检验。
利用Structure 2.3.4软件进行基于贝叶斯(Bayesian)数学模型的遗传结构分析[19]。推断种群结构,先设置K值从2到7,将不作数迭代(Length of burn-in period)的MCMC(Markov chain monte carlo)值设为10 000次,将不作数迭代后的MCMC值设为100 000,每个K值运行3次,其余参数均为默认值,计算各参试材料的Q值(参试材料基于SCoT标记变异归于不同群的概率)。将计算结果上传到在http://taylor0.biology.ucla.edu/struct_harvest网站,计算ΔK值,确定最优K值[20]。利用CLUMPP(Windows.1.1.2b)软件和Distruct V1.1软件绘制遗传结构图。
利用Popgene V1.32软件[21]计算遗传多样性参数:等位基因位点数(Number of putative alleles,Na)、有效等位基因位点(Effective allele number,Ne)、Shannon′s信息指数(Shannon′s information index,I);Nei′s基因多样性指数(Nei′s gene diversity,H)[22]、多态性信息含量(Polymorphic information content,PIC)、群体遗传距离及遗传一致性等数据。
2 结果与分析
2.1 SCoT标记多态性分析
扩增结果显示,43条SCoT标记在22份甘薯及其近缘野生材料中总共获得278条稳定条带,多态性百分率99.65%。片段大小主要分布在100~3 000 bp,平均每条引物可扩增6.78条产物(图 1)。引物SCoT-26扩增产物最多,获得了12条带。引物SCoT-24扩增产物最少,仅有4条带。扩增多态性丰富的5条引物分别是:SCoT-8、SCoT-20、SCoT-26、SCoT-36、SCoT-35。
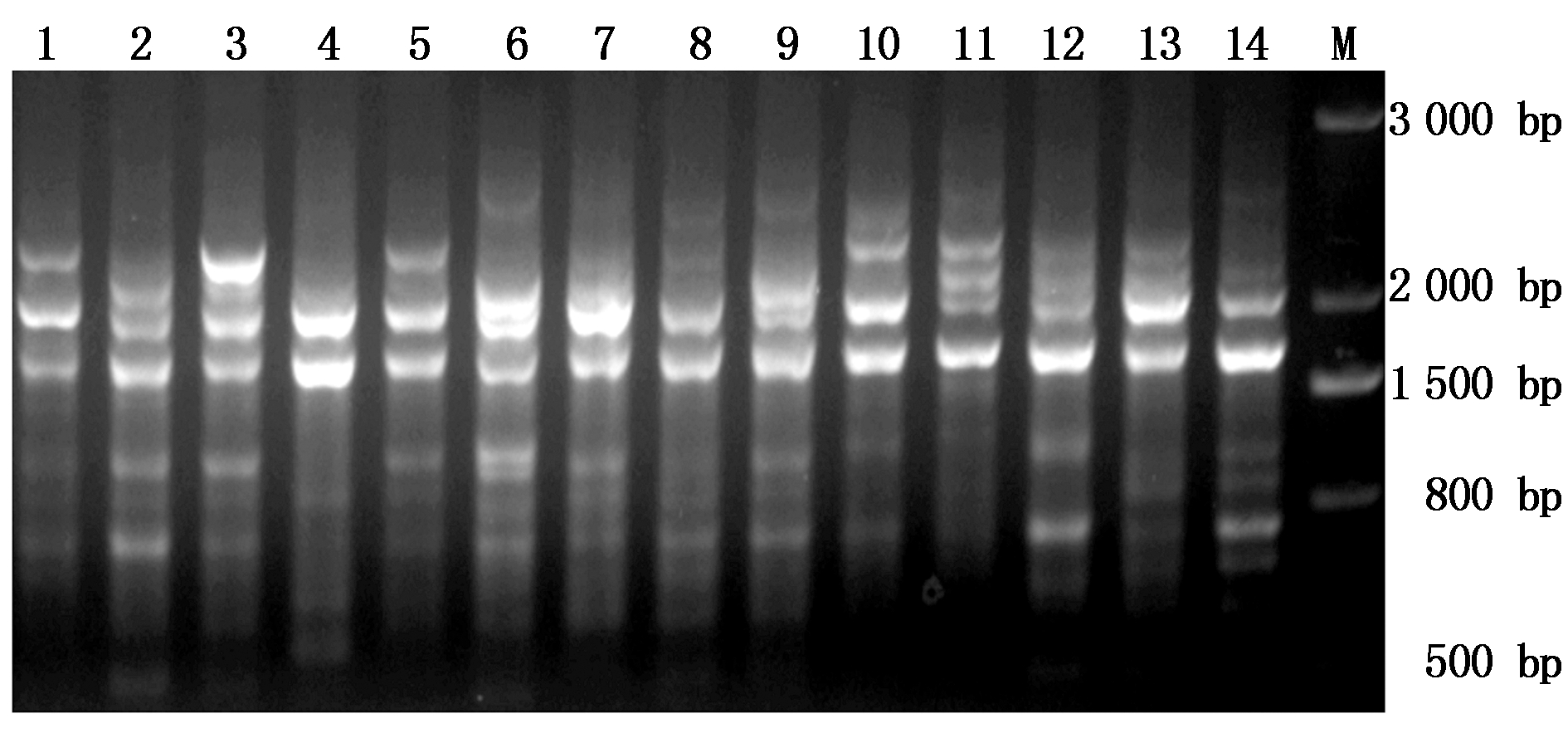
M.DNA Marker,标准条带分别为:500,800,1 500,2 000,3 000 bp;1-14.不同的参试材料。
M.DNA Marker,the size of bands is 500,800,1 500,2 000,3 000 bp,respectively;1-14.Different accessions.
图1 引物SCoT-46在参试材料中扩增产物电泳图
Fig.1 The electrophoretogram result amplified by primer SCoT-46 in different accessions
以SCoT标记扫描结果为基础,使用Popgene V1.32软件对甘薯、三浅裂野牵牛和三裂叶薯的遗传多样性分析表明,甘薯、三浅裂野牵牛和三裂叶薯平均等位基因数(Na)分别为1.84,1.92,1.52,均值为1.76。Nei′s有效等位变异数(Ne)分别为1.54,1.65,1.29,均值为1.50;基因多样性指数(H)分别为0.32,0.37和0.18,平均为0.29;Shannon′s多样性指数(I)分别为0.47,0.54,0.27,平均为0.43;多样性信息分别为84.38,92.36,52.43,均值为76.39(表3)。从结果可以看出,三浅裂野牵牛的遗传多样性最高,甘薯次之,三裂叶薯多样性最小。
表3 基于SCoT标记的3个物种间遗传多样性比较
Tab.3 Comparison of genetic diversity among three species based on SCoT markers
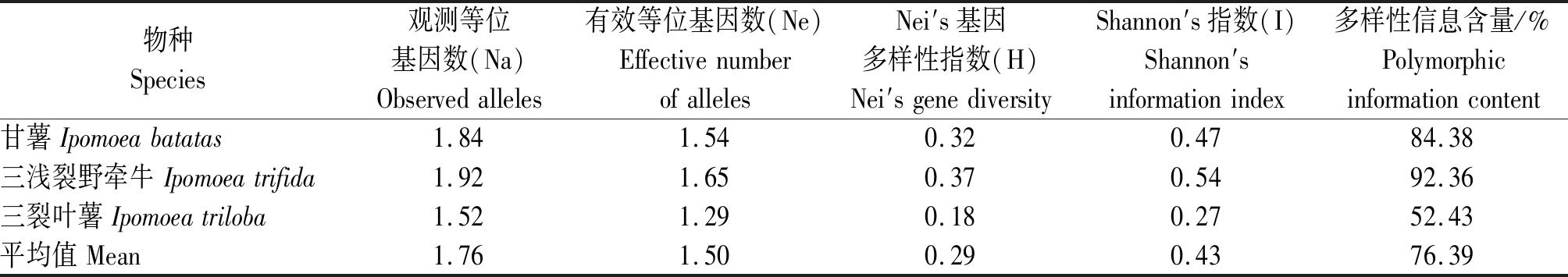
物种Species观测等位基因数(Na)Observedalleles有效等位基因数(Ne)EffectivenumberofallelesNei′s基因多样性指数(H)Nei′sgenediversityShannon′s指数(I)Shannon′sinformationindex多样性信息含量/%Polymorphicinformationcontent甘薯Ipomoeabatatas1.841.540.320.4784.38三浅裂野牵牛Ipomoeatrifida1.921.650.370.5492.36三裂叶薯Ipomoeatriloba1.521.290.180.2752.43平均值Mean1.761.500.290.4376.39
2.2 参试甘薯及其野生种聚类分析
基于22份参试材料的分子标记数据,利用Ntsys-pc 2.1软件计算不同材料间的Jaccard遗传距离,使用Neighbor-joining法进行聚类分析,聚类结果(图 2)表明,在遗传距离为0.003时,22份参试材料可分为两大类,其中第一大类群包含8份甘薯材料和1份甘薯与三浅裂野牵牛杂交的后代材料;另一大类群主要包括6份三裂叶薯材料和7份三浅裂野牵牛材料。在遗传距离0.008处,两大类群中的第一大类又被划分为两小类群。第一小类群中类包括8份甘薯种质材料,分别为西成007、徐薯18、CS、524、日本紫薯、徐薯22、日本苕、南紫薯008。其中来自日本的2份甘薯材料日本紫薯和日本苕被聚到一起,其遗传关系较近,可能与其来自国外同一生态区有关。种间杂交后代材料shTFBT2017001被单独聚到另一小类群中,可能与其既含有甘薯血缘又含有三浅裂野牵牛血缘有关。在遗传距离0.059处,2个大类中的第二大类又被划分为3个小类群,分别包含7,2,4份材料。其中2个小类群中分别包括7份三裂叶薯材料和4份三浅裂野牵牛材料。7份三裂叶薯材料间遗传关系较近,4份三浅裂野牵牛材料间的遗传关系较远。其余2份三浅裂野牵牛种质材料被单独聚到另一小类群中。参试6份三浅裂野牵牛种质材料被聚到2个小类群中,可能与其不同地理来源有关,还需进一步研究。
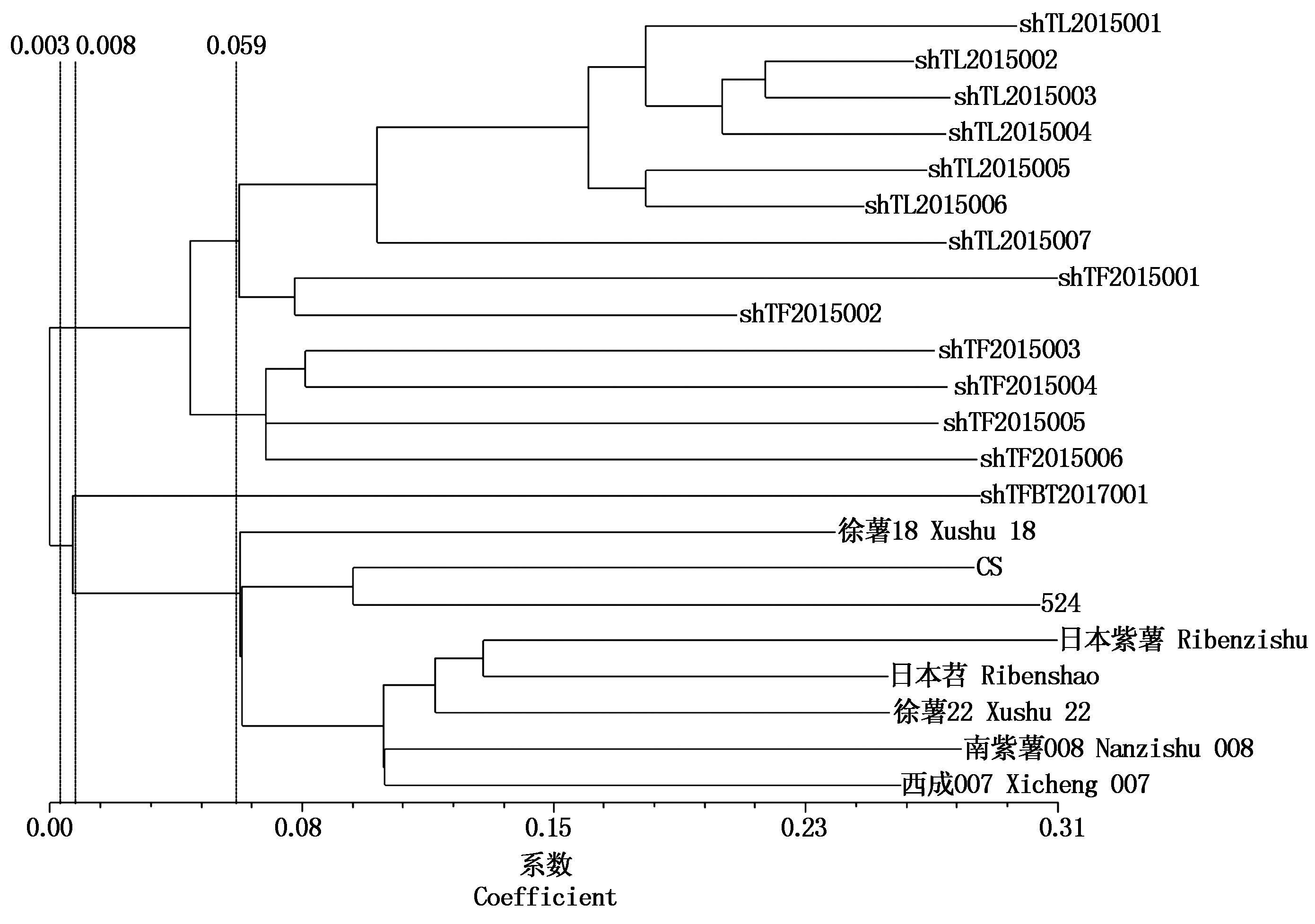
图2 基于SCoT分子标记的参试材料遗传聚类图
Fig.2 Phylogenetic tree of different accessions based on SCoT molecular markers
为了对遗传聚类结果的准确性进行评价,利用Ntsys-pc 2.1软件的协表(Cophenetic correlation)相关性检验功能,对遗传距离矩阵和相关矩阵进行分析,结果表明(图3),2个矩阵的相关系数为r=0.915 06,说明遗传聚类结果较好。
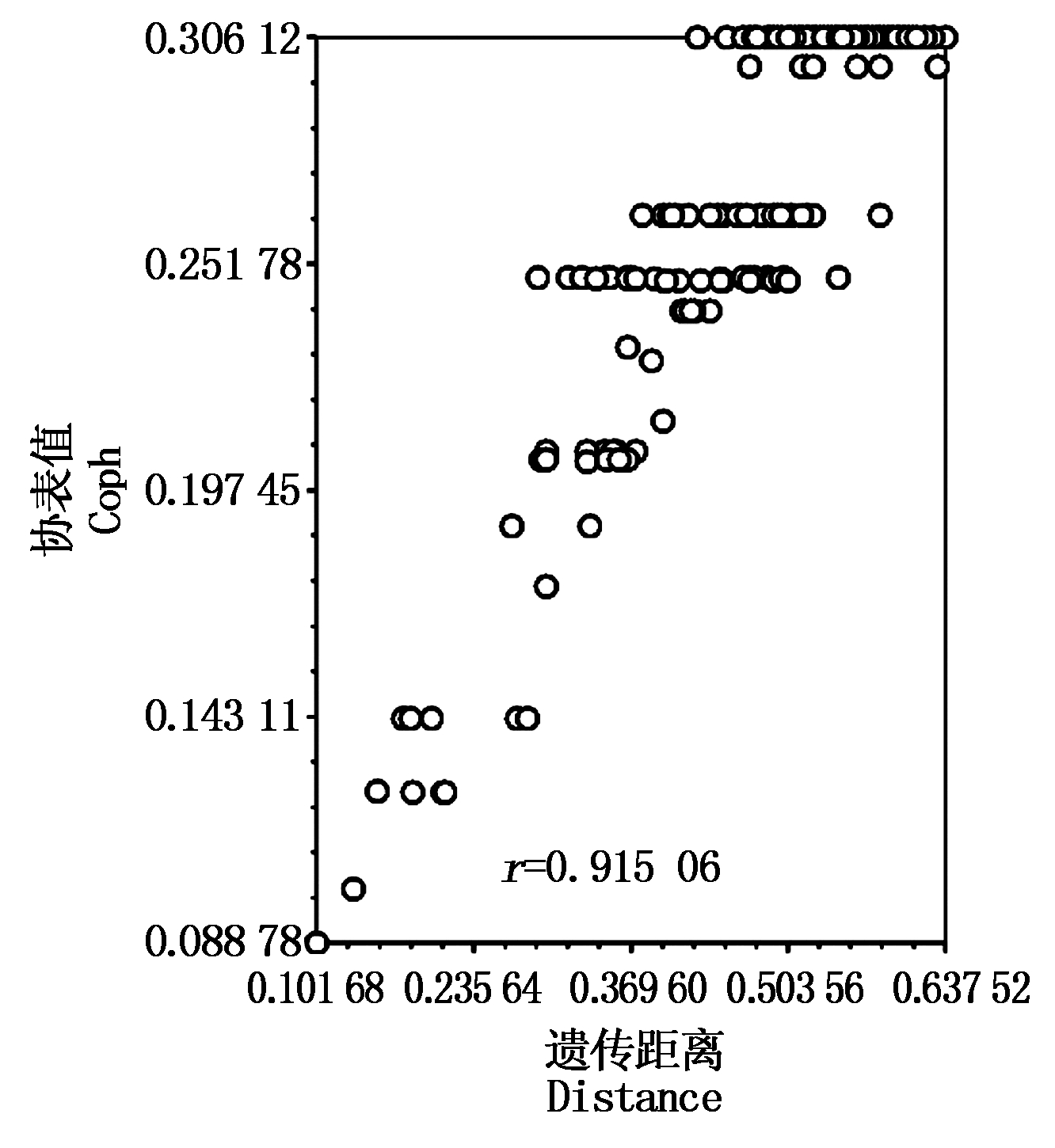
图3 遗传距离矩阵和相关矩阵的协表相关性检验结果
Fig.3 The cophenetic correlation test results of correlation matrix and genetic distance matrix
2.3 参试甘薯及其野生种遗传结构分析
基于SCoT分子标记数据,利用Structure v2.3.4软件对22份甘薯及其近缘属材料进行群体遗传结构分析,计算每份材料的Q值。设置K值从2到7,分析结果表明,LnP(D)值随着K值先增大后逐渐减小,当K=3时,LnP(D)最大为-6 323.9,随后急剧减小,出现拐点,后期变化趋小,初步确定最佳K值(图4-A)。为进一步验证该结果的正确性,参照Evanno等[23]的方法,通过计算ΔK值来确定最佳K值。通过对ΔK值变化的分析发现,当K=3时,ΔK值最大,当K=4时,ΔK值急剧减小,随后变化逐渐变小,故以K=3作为所有参试材料的最佳分群数(图 4-B)。分析LnP(D)值和ΔK值的变化趋势,获得的最佳K值一致,因此,初步确定参试22份材料被划分到3个亚群,不同颜色表示不同的类群,分别用S1(绿色)、S2(蓝色)和S3(红色)表示(图 5)。S1(绿色)亚群包括7份,分别为shTL2015001~shTL2015007,该亚群材料均属于三裂叶薯,占参试材料的31.82%。S2(蓝色)亚群包括6份,分别为shTF2015001~shTF2015006,该亚群材料均属于三浅裂野牵牛,占参试材料的27.27%。S3(红色)亚群包括9份,分别为西成007、徐薯18、CS、524、日本紫薯、徐薯22、日本苕、南紫薯008、shTFBT2017001,该亚群材料除shTFBT2017001为甘薯和三浅裂野牵牛杂交后代,其余材料均属于甘薯,占参试材料的40.91%。
以0.6作为区分亚群和混合群的阈值[24],分析每份参试材料的Q值分布,当Q≥0.6作为遗传背景单一,Q<0.6视为具有混合来源,遗传背景复杂。分析结果表明,22份材料中,21份参试材料(占95.45%)的Q值大于0.6。其中16份材料的Q值大于0.8,占供试材料总数的72.73%,说明参试材料间可能由于物种差异,相互之间血缘交流少,遗传组成比较单一。仅有1份材料(占4.55%)的Q值小于0.6,可能与其遗传组成中含有甘薯和三浅裂野牵牛血缘有关(图 5)。
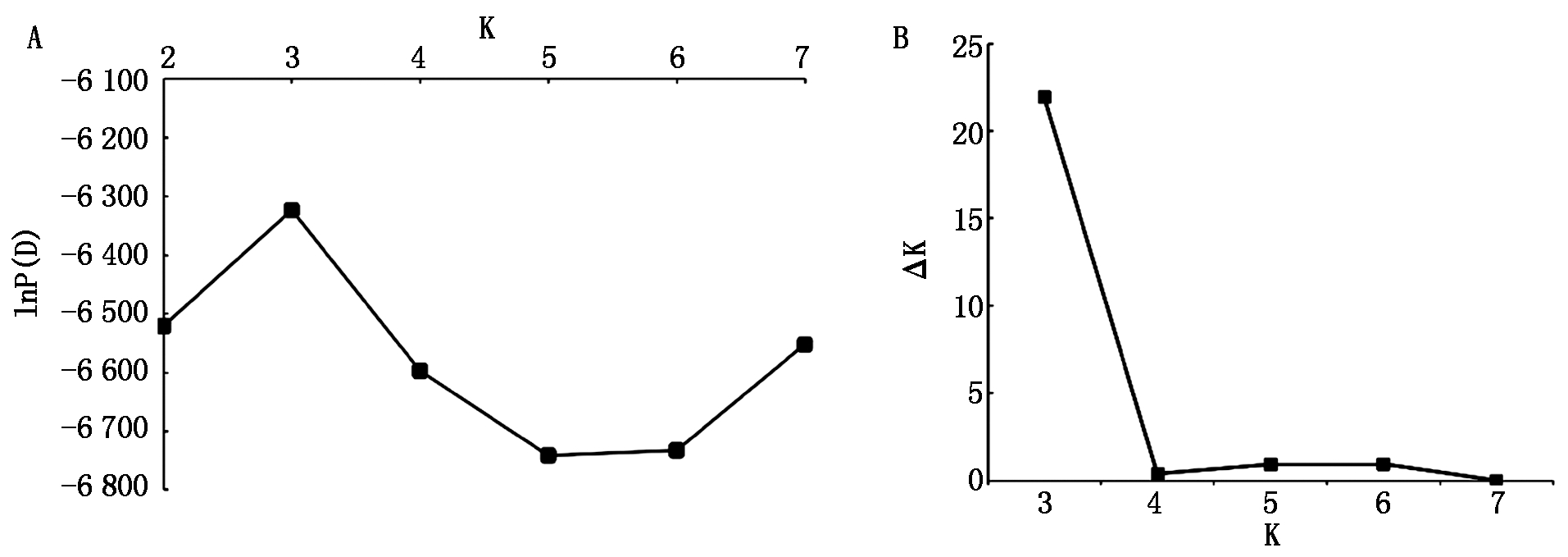
A.lnP(D)值随K值变化趋势图;B.ΔK值随K值变化趋势图。
A.The trends of lnP(D)with different K value;B.The trends of ΔK with different K value.
图 4 22份甘薯及其野生种材料群体结构分析的lnP(D)值、ΔK值随K值变化趋势图
Fig.4 The trends of lnP(D)and ΔK with different K value based on population structure analysis for 22 sweet potato and its wild species accessions
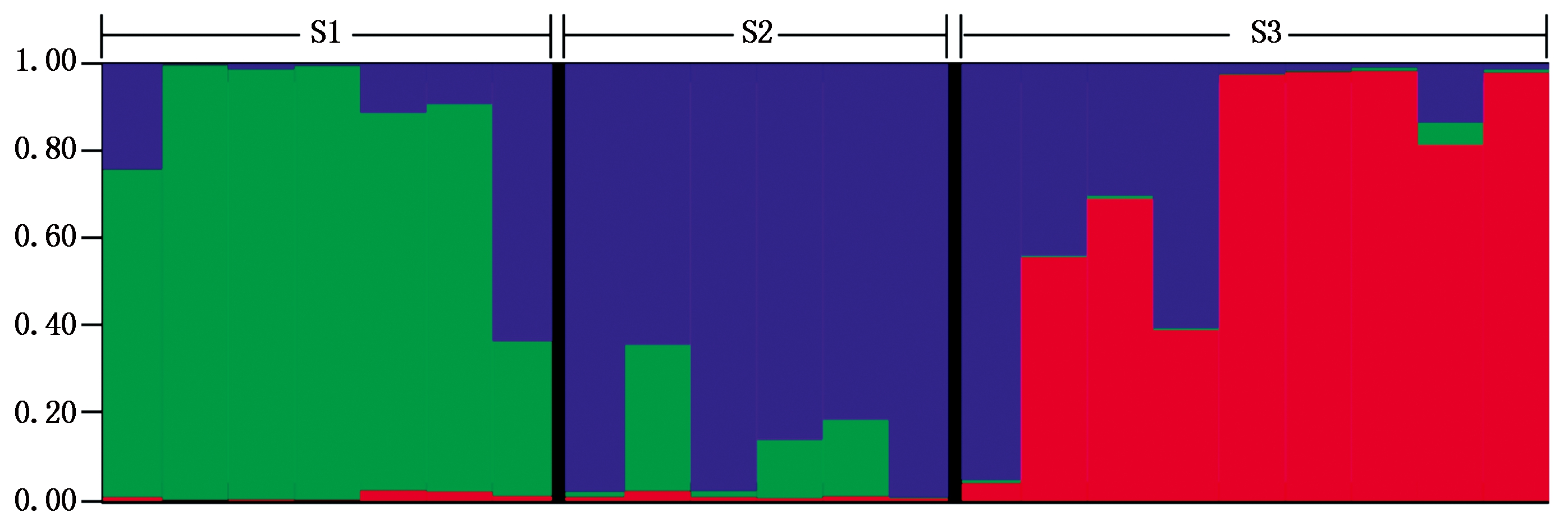
图 5 22份甘薯及其野生种材料的群体遗传结构
Fig.5 Population structure of 22 sweet potato and its wild species accessions
2.4 参试甘薯及其野生种遗传关系分析
利用Popgene V1.32软件对参试的22份甘薯及其野生种材料所属的4种类型进行遗传分析,其中三裂叶薯类型中包括7份材料,三浅裂野牵牛类型中包括6份材料,甘薯类型中包括8份材料,剩余类型仅包括1份甘薯和三浅裂野牵牛杂交材料shTFBT2017001,分析结果显示,shTFBT2017001与三裂叶薯、三浅裂野牵牛、甘薯之间的遗传距离较远,其中与三裂叶薯之间的遗传距离最远,达到0.506 7。可能与其仅有一份材料,而且其遗传组成含有三浅裂野牵牛、甘薯血缘,不含有三裂叶薯血缘有关。三裂叶薯和三浅裂野牵牛之间的遗传距离最近,可能与其野生种倍型有关,还需要进一步研究。甘薯与三浅裂野牵牛之间的遗传距离比其与三裂叶薯之间的遗传距离更近,这一结果与现有研究结果基本一致(表 4)。在遗传一致性方面,本研究中4种不同类型材料间的遗传一致性较低,可能与其来源于不同物种有关。其中,三裂叶薯和三浅裂野牵牛之间的遗传一致性最高,达到0.838 8。shTFBT2017001与甘薯和三浅裂野牵牛的遗传一致性均高于其与三裂叶薯的遗传一致性(表 4)。甘薯与三浅裂野牵牛的遗传一致性高于其与三裂叶薯间的遗传一致性。
表4 基于SCoT分子标记的不同类型甘薯及其野生种间遗传一致性及遗传距离分析
Tab.4 Genetic identity and genetic distance of four sweet potato and it′s wild species based on SCoT molecular markers

类型Type三裂叶薯I.triloba三浅裂野牵牛I.trifidashTFBT2017001甘薯I.batatas三裂叶薯I.triloba∗∗∗∗0.83880.60250.7122三浅裂野牵牛I.trifida0.1757∗∗∗∗0.62240.7814shTFBT20170010.50670.4741∗∗∗∗0.6498甘薯I.batatas0.33940.24660.4311∗∗∗∗
注:对角线上三角为遗传一致性;对角线下三角为遗传距离。
Note:The genetic uniformity above the diagonal;The genetic distance blow the diagonal.
3 讨论
3.1 分子标记在甘薯遗传研究中的应用
甘薯是六倍体(2n=6 x=90)作物,染色体多,基因组大,在基因定位、遗传图谱构建等研究上比其他作物困难。一直以来,甘薯遗传连锁图谱构建、分子标记研究主要使用AFLP(Amplified fragment length polymorphism)[10,25]、SSR(Simple sequence repeats)[26-27]、RAPD(Randomly amplified polymorphic DNA)[28]等分子标记技术。在甘薯重要农艺性状的分子标记研究方面,利用这些分子标记技术目前已定位了多个与抗病[10]、品质[25-27]等相关的遗传位点。虽然这些分子标记技术在甘薯遗传图谱构建和农艺性状标记方面发挥了重要作用,但是其各自的不足也显而易见。随着甘薯分子标记研究的不断深入,发掘成本低、多态性丰富、操作简便的分子标记,对甘薯遗传育种研究具有重要作用。
目标起始密码子多态性(Start codon targeted polymorphism,SCoT)标记是单引物、显性分子标记,扩增区域集中候选基因功能区[16],该标记技术操作简单、重复性好。而且它在不同物种间通用,可同时检测多个位点,大大节约了试验成本。目前,该标记技术已在生物多样性分析、种质资源评价、遗传图谱构建等方面得到了广泛应用。
本研究利用43条SCoT引物对22份甘薯及其野生种进行扫描,结果证明该标记在参试材料中扩增稳定、多态性丰富,能够较好的发掘甘薯及其野生种基因组遗传多态性信息。通过对22份甘薯及野生种进行分子标记数据分析发现,全部参试材料平均等位基因1.76个,平均基因多样性指数0.29,平均Shannon′s指数为0.43,平均多样性信息含量(PIC)为76.39。通过比较参试3个物种间遗传多样性指标发现,三浅裂野牵牛的多样性指标均最高,甘薯次之,三裂叶薯最小,原因可能是三浅裂野牵牛在自然条件下既可以自交也可以异交,遗传背景比较广,而甘薯虽然可以异交,但因长期使用少数亲本材料,遗传背景变窄。三裂叶薯在自然环境下主要以自交为主,其遗传多样性更低。目前,遗传多样性降低,遗传背景变窄已经成为甘薯育种中面临的主要问题,在今后甘薯育种中,开展远缘杂交,引入三浅裂野牵牛等野生种血缘,对提高甘薯遗传多样性无疑具有重要作用。
3.2 甘薯及其野生种遗传关系分析
早期形态学、细胞学方面的研究支持三浅裂野牵牛是甘薯的直接祖先之一[29-31]。近年来,分子标记技术在植物遗传研究方面的优势逐渐显现[32-34],该技术在甘薯及其野生种中遗传多样性研究中的应用也进一步证实了甘薯和三浅裂野牵牛之间的密切关系[35-37]。但是三裂叶薯是否是甘薯起源种,目前仍有争议[3,29]。本研究中的22份甘薯及野生种种质材料分别属于甘薯、三浅裂野牵牛和三裂叶薯,通过对全部材料的NJ聚类及遗传结构分析,可以将22份种质材料分为三大类群,分类结果与各材料所属物种基本一致,而且在分类结果显示甘薯和三浅裂野牵牛的杂交后代材料聚类位置处于甘薯类群和三浅裂野牵牛类群之间,初步说明SCoT分子标记可以区分甘薯及其野生种,证明了该标记在甘薯进化研究中具有潜在应用价值。
本研究对不同甘薯及其野生种群体的遗传距离和遗传一致性分析结果支持甘薯和三浅裂野牵牛之间的遗传关系比其与三裂叶薯更近。此外遗传结构分析结果也显示,在血缘交流方面,甘薯与三浅裂野牵牛的血缘交流较多,而其与三裂叶薯之间的血缘交流极少,也进一步说明甘薯与三浅裂野牵牛遗传关系较三裂叶薯更近,这一研究结果与前人研究结果[38-40]基本一致。但是由于本研究使用材料数较少,而且SCoT分子标记也主要揭示的是基因功能区的遗传多样性,因此,要完全弄清甘薯及野生种的遗传演化关系,还需应用测序等技术在全基因水平上开展深入研究。
[1] Woolfe J A. Sweet potato:an untapped food resource[M].Cambridge:Cambridge University Press,1992:15-40.
[2] 谢一芝,郭小丁,贾赵东,马佩勇,边小峰,禹阳. 中国食用甘薯育种现状及展望[J]. 江苏农业学报,2018,34(6):1419-1424. doi:10.3969/j.issn.1000-4440.2018.06.030.
Xie Y Z,Guo X D,Jia Z D,Ma P Y,Bian X F,Yu Y. Progresses and prospects on edible sweetpotato breeding in China[J].Jiangsu Journal of Agricultural Sciences,2018,34(6):1419-1424.
[3] Austin D F. The taxonomy,evolution and genetic diversity of sweet potatoes and related wild species[C]//CIP.Exploration,maintenance and utilization of sweetpotato genetic resources: Report 1st sweet potato planning conference. Lima:International Potato Center,1988:27-60.
[4] Zhang D P,Cervantes J,Huam n Z,Carey E,Ghislain M. Assessing genetic diversity of sweet potato(Ipomoea batatas (L.)Lam.)cultivars from tropical America using AFLP[J].Genetic Resources and Crop Evolution,2000,47:659-665. doi:10.1023/A:1026520507223.
n Z,Carey E,Ghislain M. Assessing genetic diversity of sweet potato(Ipomoea batatas (L.)Lam.)cultivars from tropical America using AFLP[J].Genetic Resources and Crop Evolution,2000,47:659-665. doi:10.1023/A:1026520507223.
[5] Takagi H. Sweetpotato collections in Papua New Guinea[C]//CIP.Exploration,maintenance and utilization of sweetpotato genetic resources: Report 1st sweet potato planning conference. Lima: International Potato Center, 1988: 233-246.
[6] Rao V R,Debouck T,Iwanaga M. The role of international organizations in root and tuber crop conservation[C]//International workshop on genetic resources-root and tubercrops. Tsukuba: 1st Ministry of Agriculture, Forestry and Fisheries, 1994:7-22.
[7] Komaki K. Breeding value of wild species closely related to sweetpotato[C].Korea:Proceedings of international workshop on production, utilization and development of sweetpotato, 2004:164-172.
[8] Kriegner A,Cervantes J C,Burg K,Mwanga R O M,Zhang D P. A genetic linkage map of sweetpotato(Ipomoea batatas (L.)Lam.)based on AFLP markers[J].Molecular Breeding,2003,11:169-185. doi:10.1023/A:1022870917230.
[9] Cervantes-Flores J C,Yencho G C,Kriegner A,Pecota K V,Faulk M A,Mwanga R O M,Sosinski B R. Development of a genetic linkage map and identification of homologous linkage groups in sweetpotato using multiple-dose AFLP markers[J].Molecular Breeding,2008,21:511-532. doi:10.1007/s11032-007-9150-6.
[10] Li H,Zhao N,Yu X X,Liu Y X,Zhai H,He S Z,Li Q,Ma D F,Liu Q C. Identification of QTLs for storage root yield in sweetpotato[J].Scientia Horticultural,2014,170:182-188. doi:10.1016/j.scienta.2014.03.017.
[11] Monden Y,Hara T,Okada Y,Jahana O,Kobaysahi A,Tabuchi H,Onaga S,Tahara M. Construction of a linkage map based on retrotransposon insertion polymorphisms in sweetpotato via high-throughput sequencing[J].Breeding Science,2015,65(2):145-153. doi:10.1270/jsbbs.65.145.
[12] Shirasawa K,Tanaka M,Takahata Y,Ma D F,Cao Q H,Liu Q C,Zhai H,Kwak S S,Jeong J C,Yoon U H,Lee H U,Hirakawa H,Isobe S. A high-density SNP genetic map consisting of a complete set of homologous groups in autohexaploid sweetpotato(Ipomoea batatas)[J].Scientific Reports,2018,7:44207. doi:10.1038/srep44207.
[13] Feng J Y,Li M,Zhao S,Zhang C,Yang S T,Qiao S,Tan W F,Qu H J,Wang D Y,Pu Z G. Analysis of evolution and genetic diversity of sweetpotato and its related different polyploidy wild species I.trifida using RAD-seq[J].BMC Plant Biology,2018,18:181. doi:10.1186/s12870-018-1399-x.
[14] Feng J Y,Zhao S,Li M,Zhang C,Qu H J,Li Q,Li J W,Lin Y,Pu Z G. Genome-wide genetic diversity detection and population structure analysis in sweetpotato(Ipomoea batatas)using RAD-seq[J].Genomics,2020,112(2):1978-1987. doi:10.1016/j.ygeno.2019.11.010.
[15] Yang X S,Su W J,Wang L J,Lei J,Chai S S,Liu Q C. Molecular diversity and genetic structure of 380 sweetpotato accessions as revealed by SSR markers[J].Journal of Integrative Agriculture,2015,14(4):633-641.doi:10.1016/S2095-3119(14)60794-2.
[16] Collard B C Y,Mackill D J. Start Codon Targeted(SCoT)polymorphism:a simple,novel DNA marker technique for generating gene-targeted markers in plants[J].Plant Molecular Biology Reporter,2009,27(1):86-93. doi:10.1007/s11105-008-0060-5.
[17] 冯俊彦,赵珊,李明,张聪,屈会娟,杨松涛,乔帅,谭文芳,王大一,蒲志刚.四种分子标记技术在甘薯及I.trifida遗传多样性研究中的应用[J]. 分子植物育种,2018,16(17):5667-5674. doi:10.13271/j.mpb.016.005667.
Feng J Y,Zhao S,Li M,Zhang C,Qu H J,Yang S Y,Qiao S,Tan W F,Wang D Y,Pu Z G. The application of four molecular marking techniques in the genetic diversity of sweetpotato and I.trifida[J].Molecular Plant Breeding,2018,16(17):5667-5674.
[18] Murray M G,Thompson W F. Rapid isolation of high molecular weight plant DNA[J]. Nnucleic Acids Research,1980,8(19):4321-4326. doi:10.1093/nar/8.19.4321.
[19] Hubisz M J,Falush D,Stephens M,Pritchard J K. Inferring weak population structure with the assistance of sample group information[J]. Molecular Ecology Resources,2009,9(5):1322-1332. doi:10.1111/j.1755-0998.2009.02591.x.
[20] Earl D A,Vonholdt B M. STRUCTURE HAVESTER:a website and program for visualizing STRUCTURE output and implementing the Evanno method[J]. Conservation Genetics Resources,2012,4(2):359-361. doi:10.1007/s12686-011-9548-7.
[21] Yeh F C,Boyle T B J. Population genetic analysis of co-dominant and dominant markers and quantitative traits[J].Belgian Journal of Botany,1997,129:157-163.
[22] Nei M,Li W H. Mathematical model for studying genetic variation in terms of restriction endonucleases[J]. Proceedings of the National Academy of Sciences of the United States of America, 1979,76(10):5269-5273. doi:10.1073/pnas.76.10.5269.
[23] Evanno G,Regnaunts S,Goudet J. Detecting the number of clusters of individuals using the software structure:A simulation study[J]. Molecular Ecology, 2005,14(8):2611-2620. doi:10.1111/j.1365-294X.2005.02553.x.
[24] 苏一钧,王娇,戴习彬,唐君,赵冬兰,张安,周志林,曹清河. 303份甘薯地方种SSR遗传多样性与群体结构分析[J]. 植物遗传资源学报,2018,19(2):243-251. doi:10.13430/j.cnki.jpgr.2018.02.007.
Su Y J,Wang J,Dai X B,Tang J,Zhao D L,Zhang A,Zhou Z L,Cao Q H. Genetic diversity and population Structure analysis of 303 sweetpotato landraces using SSR markers[J].Journal of Plant Genetic Resource,2018,19(2):243-251.
[25] Elameen A,Fjellheim S,Larsen A,Rognli O A,Sundheim L,Msolla S,Masumba E,Mtunda K,Klemsdal S S. Analysis of genetic diversity in a sweetpotato(Ipomoea batatas L.)germplasm collection from Tanzania as revealed by AFLP[J].Genetic Resources and Crop Evolution,2008,55(3):397-408.doi:10.1007/s10722-007-9247-0.
[26] Cervantes-Flores J C,Sosinski B,Pecota K V,Mwanga R O M,Catignani G L,Truong V D,Watkins R H,Yencho G C. Identification of quantitative trait loci for dry-matter,starch,and β-carotene content in sweetpotato[J].Molecular Breeding,2011,28:201-216. doi:10.1007/s11032-010-9474-5.
[27] Zhao N,Yu X X,Jie Q,Li H,Li H,Hu J,Zhai H,He S Z,Liu Q C. A genetic linkage map based on AFLP and SSR markers and mapping of QTL for dry-matter content in sweetpotato[J].Molecular Breeding, 2013,32(4):807-820. doi:10.1007/s11032-013-9908-y.
[28] Kim J H,Chung I K,Kim K M. Construction of a genetic map using EST-SSR markers and QTL analysis of major agronomic characters in hexaploid sweetpotato(Ipomoeabatatas(L.)Lam)[J].PLoS One,2017,12:e0185073. doi:10.1371/journal.pone.0185073.
[29] Roullier C,Duputié A,Wennekes P,Benoit L,Bringas V M F,Rossel G,Tay D,McKey D,Lebot V. Disentangling the origins of cultivated sweet potato(Ipomoeabatatas(L.)Lam.)[J].PLoS One, 2013,8:e62707. doi:10.1371/journal.pone.0062707.
[30] Gichuki S T,Berenyi M,Zhang D P,Hermann M,Schmidt J,Glössl J,Burg K. Genetic diversity in sweetpotato [Ipomoea batatas (L.)Lam.]in relationship to geographic sources as assessed with RAPD markers[J].Genetic Resources and Crop Evolution, 2003,50:429-437. doi:10.1023/A:1023998522845.
[31] Nishiyama I. The origin of the sweetpotato[M]. Honolulu:Tenth Pacific Sci Congr Univ Hawaii,1961:119-128.
[32] 李祥栋,石明,陆秀娟,潘虹,魏心元,陆平.利用叶绿体基因组SSR标记揭示薏苡属种质资源的遗传多样性[J].华北农学报,2019,34(S1):6-14. doi:10.7668/hbnxb.20190745.
Li X D,Shi M,Lu X J,Pan H,Wei X Y,Lu P. Genetic diversity of germplasm resources in job′s tears revealed by chloroplast genome SSR markers[J]. Acta Agriculturae Boreali-Sinica,2019,34(S1):6-14.
[33] 梁铁军,陈雄辉,崔玉梅,罗方雄,张泽民,彭海峰.影响温敏核不育水稻育性转换的低温敏感度QTL定位[J]. 华北农学报,2020,35(2):29-34. doi:10.7668/hbnxb.20190431.
Liang T J,Chen X H,Cui Y M,Luo F X,Zhang Z M,Peng H F. QTL mapping of low temperature sensitivity affecting the fertility reversibility in thermo-sensitive genic male sterile rice[J]. Acta Agriculturae Boreali-Sinica,2020,35(2):29-34.
[34] 白志元,张瑞军,武国平,连世超,杨玉花,卫保国.基于SSR标记的杂交大豆主要亲本遗传多样性分析[J].华北农学报,2018,33(4):120-125. doi:10.7668/hbnxb.2018.04.017.
Bai Z Y,Zhang R J,Wu G P,Lian S C,Yang Y H,Wei B G. Genetic diversity analysis of main parents for hybrid soybean based on SSR markers[J]. Acta Agriculturae Boreali-Sinica,2018,33(4):120-125.
[35] Nishiyama I,Fujise K,Teramura T,Miyazaki T. Studies of sweetpotato and its related species:I. Comparative investigationson the chromosome numbers and the main plant characters of Ipomoea species in section Batatas[J]. Japanese Journal of Breeding,1961,11(1):37-43. doi:10.1270/jsbbs1951.11.37.
[36] Kobayashi M,Miyazaki T.Sweetpotato breeding using wild related specie[M]//James C, Reginald M, Michael G. Tropical root and tuber crops:Fourth Triennial Symposium. Columbia: Centro Internacional de Agricultura Tropical, 1976: 53-57.
[37] Huang J C,Sun M. Genetic diversity and relationships of sweetpotato and its wild relatives in Ipomoea series Batatas(Convolvulaceae)as revealed by inter-simple sequence repeat(ISSR)and restriction analysis of chloroplast DNA[J].Theoretical and Applied Genetics, 2000,100:1050-1060. doi:10.1007/s001220051386.
[38] Srisuwan S,Sihachakr D,Siljak-Yakovlev S. The origin and evolution of sweetpotato(Ipomoea batatas Lam.)and its wild relatives through the cytogenetic approaches[J].Plant Science, 2006,171(3):424-433. doi:10.1016/j.plantsci.2006.05.007.
[39] Jarret R L,Austin D F. Genetic diversity and systematic relationships in sweetpotato(Ipomoea batatas (L.)Lam.)and related species as revealed by RAPD analysis[J].Genetic Resources and Crop Evolution, 1994,41(3):165-173. doi:10.1007/BF00051633.
[40] Kobayashi M. The Ipomoea trifida complex closely related to sweetpotato[C]//Peru:Centro Internacional de la Papua,Proc 6th Symp Int Soc Trop Root Crops,Lima,1984:561-568.
