玉米(Zea mays L.)在人类生产生活中发挥着巨大的作用,又是我国主要的经济作物和部分饲料的原材料。玉米籽粒中包括淀粉、蛋白质、脂肪等重要组分,而玉米胚乳约占玉米籽粒重的80%~85%,而淀粉又是胚乳细胞中的主要成分,是玉米籽粒的重要组成成分[1]。籽粒淀粉是决定玉米产量和品质的关键因素,提高玉米淀粉的含量和淀粉的品质是玉米育种工作者最重要的任务[2]。淀粉是在一系列淀粉合成相关酶的催化作用下完成的,在淀粉的合成过程中,有4种酶起主要催化作用,包括淀粉去分支酶(Starch debranching enzyme,SDBE)、ADP葡萄糖焦磷酸化酶(ADP-glucose pyrophosphorylase,AGPase)、淀粉分支酶(Starch branching enzyme,SBE)以及淀粉合成酶(Starch synthase,SS) [3-4]。
玉米ZmAGP2 基因(ADP glucose pyrophosphorylase 2)编码ADP葡萄糖焦磷酸化酶大亚基。ADP葡萄糖焦磷酸化酶是一个异源四聚体[5],由2个相同的大亚基(Sh2)和2个相同的小亚基(Bt2)组成[6]。ADP葡萄糖焦磷酸化酶催化ATP和Glc-1-p[7],合成ADP-葡萄糖和二磷酸,在α-1,4-葡聚糖合成的过程中,ADP-葡萄糖作为活化的葡萄糖供体[8-9]。该反应是玉米胚乳淀粉合成的关键限速步骤。Shrunken2(Sh2)基因编码玉米ADP葡萄糖焦磷酸化酶大亚基,Brittle2(Bt2)基因编码玉米ADP葡萄糖焦磷酸化酶小亚基。Deng等[4]分离了一个编码小麦(Triticum aestivum L.)中AGP酶质体小亚基(TaAGPS1b)的cDNA序列,TaAGPS1b与水稻原生质体细胞中的绿色荧光蛋白(GFP)融合,共聚焦荧光显微镜观察表明,TaAGPS1b-GFP定位于叶绿体中。Smidansky等[10]将玉米中编码ADP葡萄糖焦磷酸化酶大亚基的Shrunken2基因改造后(Sh2r6hs)转入小麦中,发现转基因小麦株系平均每株比对照多出38%的粒质量。Zhang等[3]将小麦中的TaAGP突变,结果发现,导致其他4个淀粉合成基因的表达模式发生变化,导致淀粉和支链淀粉含量降低。Rose等[11]发现在小麦自然群体中TaAGP的SNPs引起千粒质量和种子数量的变化,但总淀粉含量没有显著变化。灌浆时间和速率对最终的晶粒质量至关重要。谷物发育初期的干旱胁迫会干扰器官建立和淀粉生物合成所需的细胞分裂和分化过程。在籽粒灌浆过程中,在水分亏缺条件下,AGPase在蛋白质和转录水平上的活性下调是淀粉积累显著下降的原因[12-13]。ZmAGP2基因在优质玉米自交系的核苷酸多态性尚不清楚,本研究的目的就是对103份玉米自交系的ZmAGP2基因的序列进行定点捕获,分析该基因的核苷酸多态性和单倍型密度。
1 材料和方法
1.1 试验材料
本研究选用了103份优良玉米自交系,其中包括来自于国外的种质材料、热带、亚热带种质以及我国温带玉米的5个杂种优势群的代表性种质(表1)。
表1 本研究所用玉米自交系列表
Tab.1 List of maize inbred lines of the study
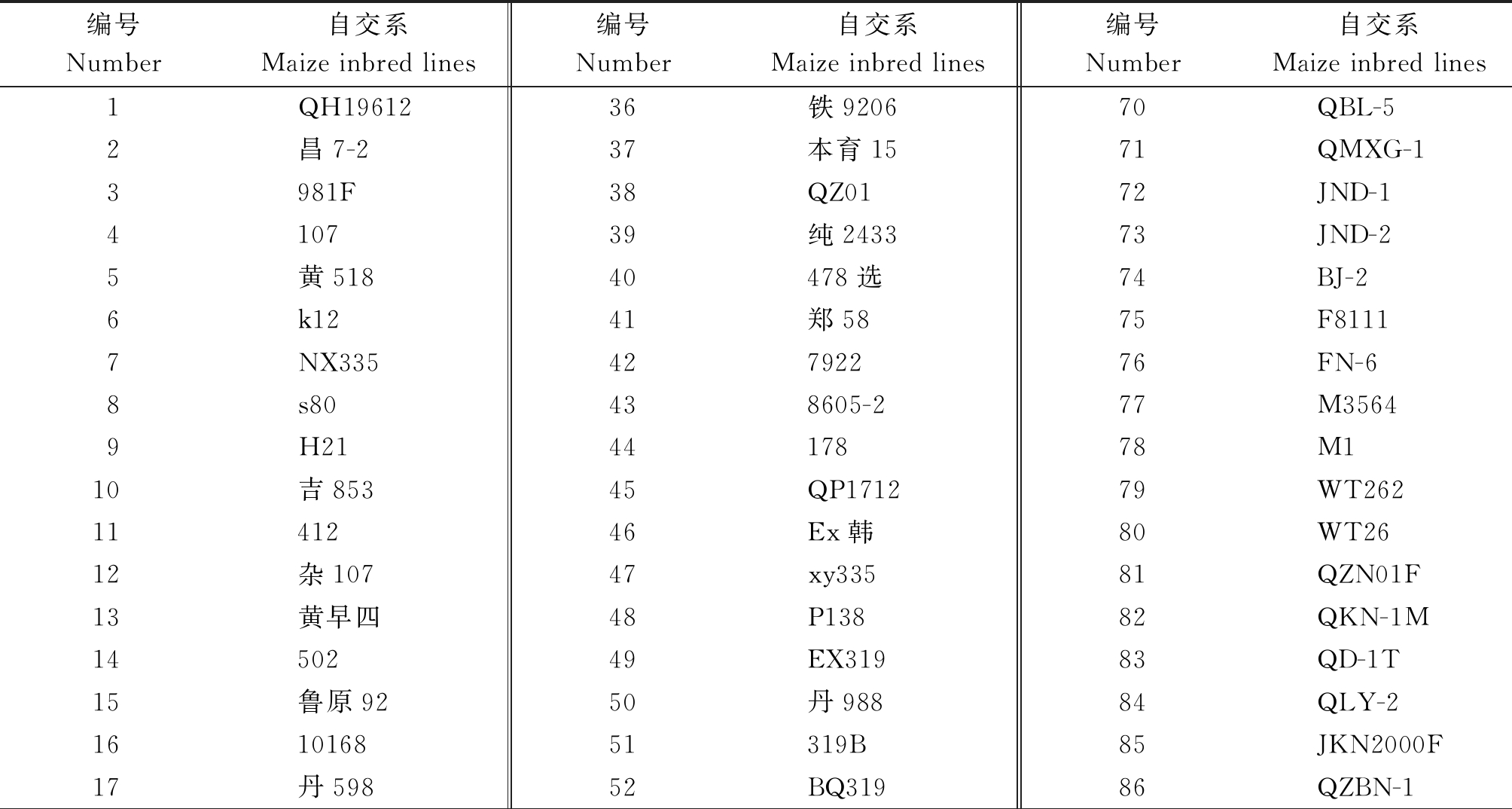
编号Number自交系Maize inbred lines编号Number自交系Maize inbred lines编号Number自交系Maize inbred lines1QH1961236铁920670QBL-52昌7-237本育1571QMXG-13981F38QZ0172JND-1410739纯243373JND-25黄51840478选74BJ-26k1241郑5875F81117NX33542792276FN-68s80438605-277M35649H214417878M110吉85345QP171279WT2621141246Ex韩80WT2612杂10747xy33581QZN01F13黄早四48P13882QKN-1M1450249EX31983QD-1T15鲁原9250丹98884QLY-2161016851319B85JKN2000F17丹59852BQ31986QZBN-1
表1(续)
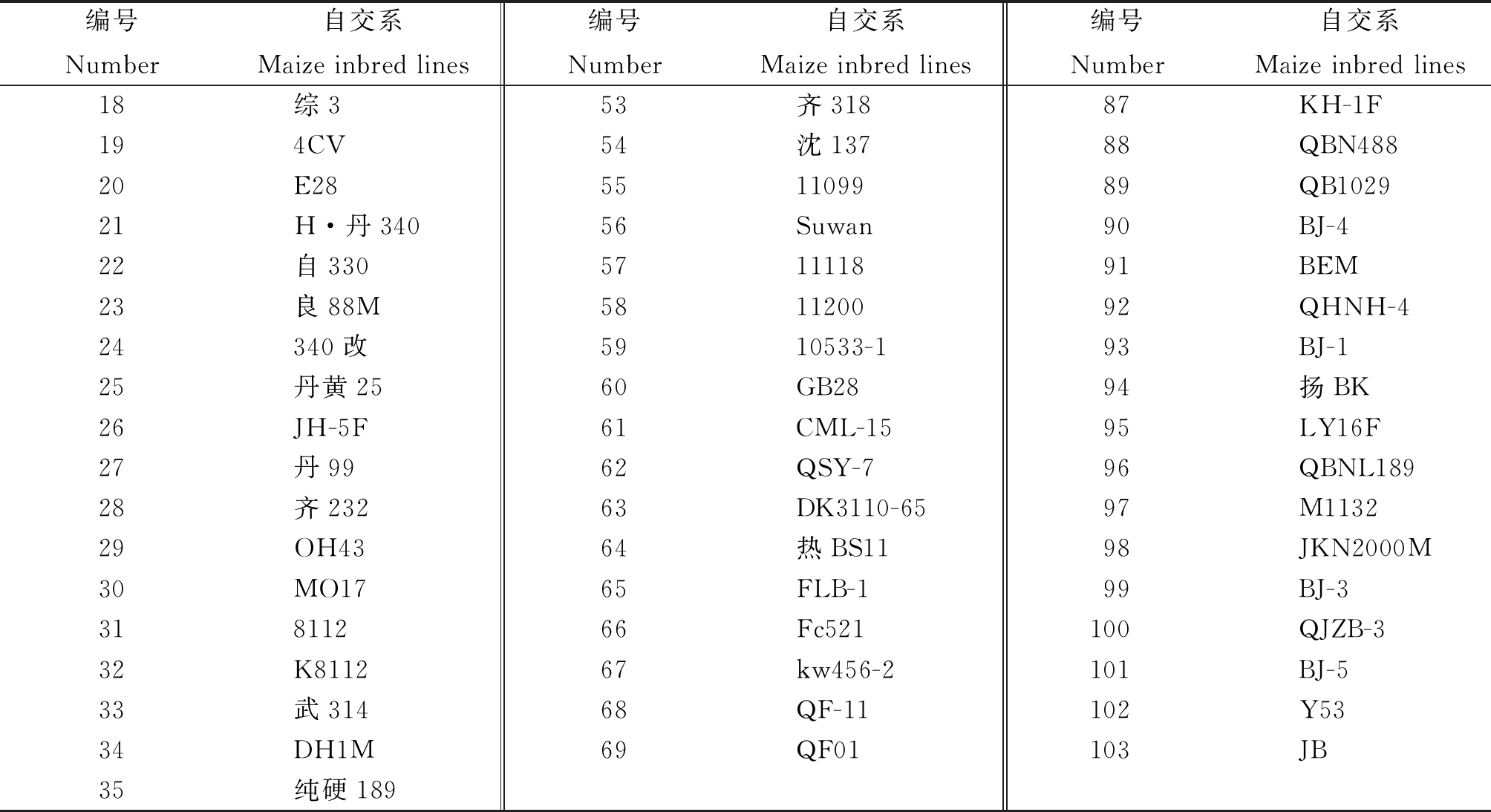
编号Number自交系Maize inbred lines编号Number自交系Maize inbred lines编号Number自交系Maize inbred lines18综353齐31887KH-1F194CV54沈13788QBN48820E28551109989QB102921H·丹34056Suwan90BJ-422自330571111891BEM23良88M581120092QHNH-424340改5910533-193BJ-125丹黄2560GB2894扬BK26JH-5F61CML-1595LY16F27丹9962QSY-796QBNL18928齐23263DK3110-6597M113229OH4364热BS1198JKN2000M30MO1765FLB-199BJ-331811266Fc521100QJZB-332K811267kw456-2101BJ-533武31468QF-11102Y5334DH1M69QF01103JB35纯硬189
1.2 DNA提取和ZmAGP2基因测序
通过水培的方式,将所有的自交系种子种植在28 °C/25 °C,16 h光照、8 h黑暗的昼夜交替的温室里。各样本的DNA的提取是利用改良过的CTAB法,利用天根®超微量分光光度计来测定DNA的浓度,各样本DNA的质量的检测是用1%的琼脂糖胶。以B73的ZmAGP2基因序列为模板,103份自交系材料的ZmAGP2基因的定点捕获和测序是通过NimbleGen平台进行的,华大公司完成该序列的定点捕获和测序。
1.3 序列数据分析
本研究用到的软件有ClustalX 2.0[14]和DnaSP 6.0[15]等。分别对获得的ZmAGP2的基因序列进行比对和进一步人工校对,估算各区段以及全长的核苷酸多态性。基因的中性进化检测利用Tajima[16]、Fu与Li[17]的D*和F*的参数来衡量。
2 结果与分析
2.1 玉米ZmAGP2基因的序列变异
ZmAGP2基因序列的全长是以玉米自交系B73的ZmAGP2基因为模板在103个玉米供试自交系中获得的。经过多序列比对后,获得了ZmAGP2基因的全长序列为4 111 bp。包括启动子序列246 bp,5′UTR区段序列1 334 bp,3′UTR区段序列370 bp,以及内含子1 106 bp和外显子1 055 bp。以100 bp为滑动框,25 bp为步长,分析得到ZmAGP2基因的核苷酸多态性图(图1)。结果显示,5′UTR 区段与其他区段相比,核苷酸多态性较高。
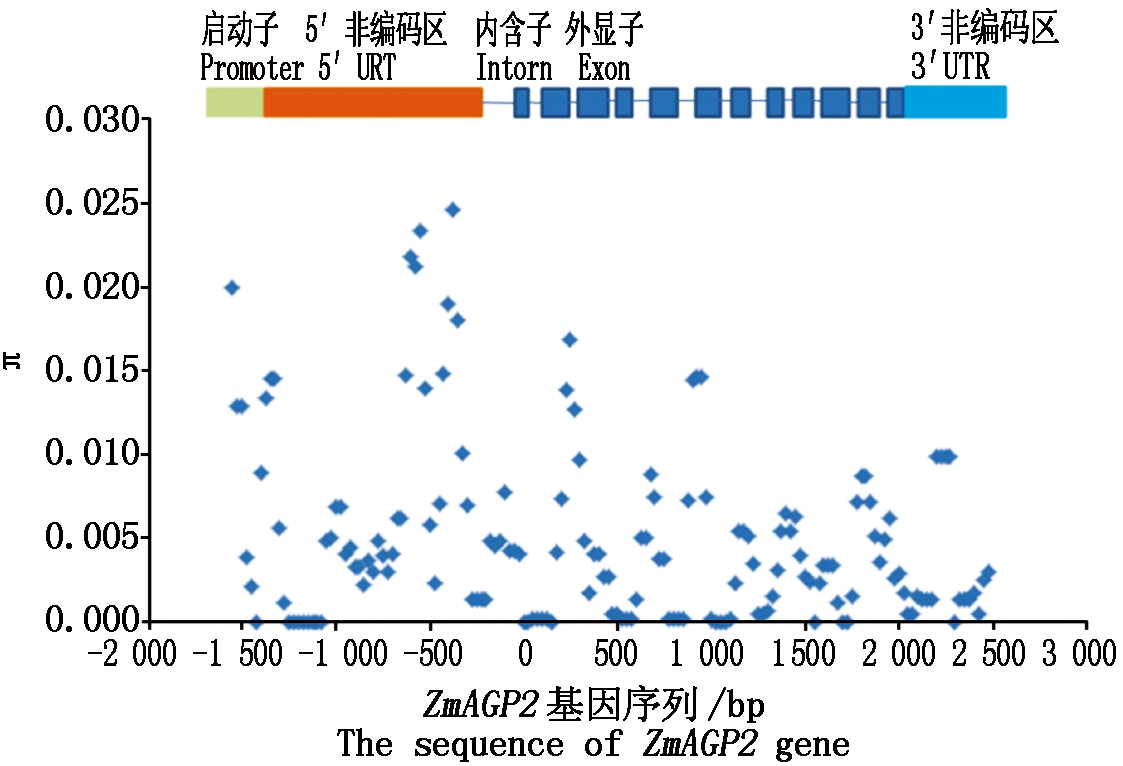
图1 ZmAGP2基因序列核苷酸多样性
Fig.1 The nucleotide diversity estimated along the ZmAGP2 gene sequence
对ZmAGP2基因全长序列进一步分析发现,共检测到112个变异位点,没有检测到Indel变异,均为SNP位点(表2)。非编码区含有103个SNP,其中内含子区段有42个SNP,5′UTR区段有40个SNP。编码区含有9个SNP。说明内含子区段和5′UTR区段是主要的遗传变异来源。变异频率最高的是第六内含子区段,为0.077。在6个区段没有发现变异位点。
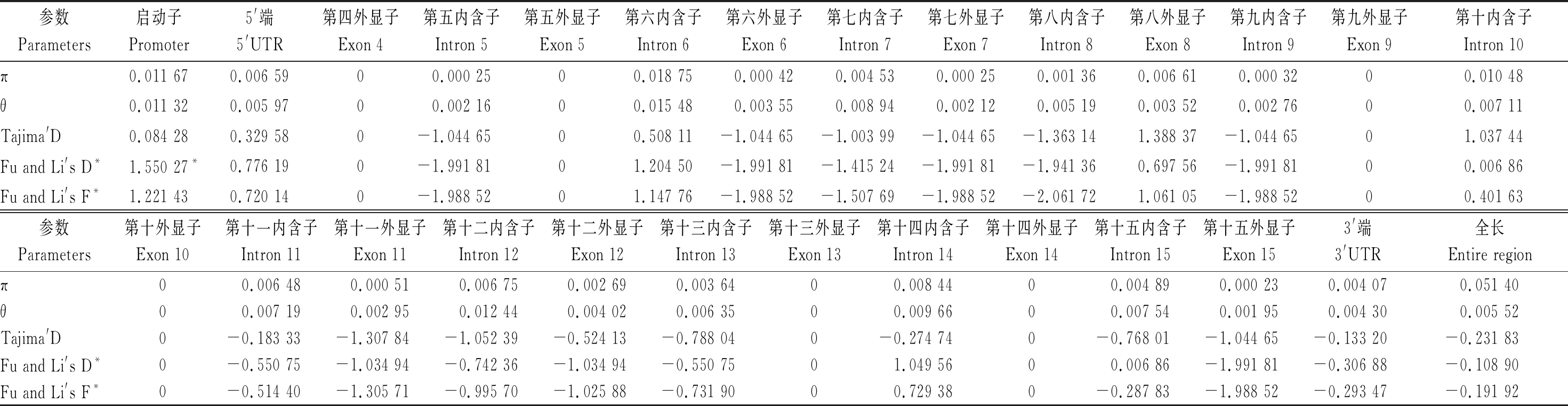
常用参数θ和π分析核苷酸多态性。在本研究的103份玉米自交系中,ZmAGP2基因全长序列的π值和θ值分别为0.051 40和0.005 52(表3)。Tajima′s D测验ZmAGP2基因序列中的多态性位点。结果显示,Tajima′s D没有达到显著水平,说明整段序列没有经过明显的选择作用。而只有启动子区段的Fu与Li的D*值达到了显著水平。
表2 ZmAGP2基因序列变异参数
Tab.2 Parameters for the sequences variants of the ZmAGP2 gene

参数Parameters启动子Promoter5'端5'UTR第四外显子Exon 4第五内含子Intron 5第五外显子Exon 5第六内含子Intron 6第六外显子Exon 6第七内含子Intron 7第七外显子Exon 7第八内含子Intron 8第八外显子Exon 8第九内含子Intron 9第九外显子Exon 9第十内含子Intron 10第十外显子Exon 10序列长度/bp Total length of amplicons2461 33447928790568994115113727314081变异位点(SNPs+ Indels) Number of all sequence variants(SNPs and Indels)13400107141221050变异频率 Frequency of all sequence variants0.0530.0300.01100.0770.0170.0440.0100.0170.0180.01300.0350SNP数目 Number of nucleotide substitutions13400107141221050SNP频率 Frequency of SNP sites0.0530.0300.01100.0770.0170.0440.0100.0170.0180.01300.0350Indels数目 Number of Indels000000000000000参数Parameters第十一内含子Intron 11第十一外显子Exon 11第十二内含子Intron 12第十二外显子Exon 12第十三内含子Intron 13第十三外显子Exon 13第十四内含子Intron 14第十四外显子Exon 14第十五内含子Intron 15第十五外显子Exon 153'端3'UTR全长Entire region序列长度/bp Total length of amplicons83135969994107103611321023704 111变异位点(SNPs+ Indels) Number of all sequence variants(SNPs and Indels)32623050518112变异频率 Frequency of all sequence variants0.0360.0140.0620.020.03100.04800.0370.0090.0210.027SNP数目 Number of nucleotide substitutions32623050518112SNP频率 Frequency of SNP sites0.0360.0140.0620.020.03100.04800.0370.0090.0210.027Indels数目 Number of Indels000000000000
表3 中性检验相关参数
Tab.3 Neutral test related parameters
注:*.P<0.05;**.P<0.01
Note: *.The significance at P<0.05 level;**.P<0.01.
参数Parameters启动子Promoter5'端5'UTR第四外显子Exon 4第五内含子Intron 5第五外显子Exon 5第六内含子Intron 6第六外显子Exon 6第七内含子Intron 7第七外显子Exon 7第八内含子Intron 8第八外显子Exon 8第九内含子Intron 9第九外显子Exon 9第十内含子Intron 10π0.011 670.006 5900.000 2500.018 750.000 420.004 530.000 250.001 360.006 610.000 3200.010 48θ0.011 320.005 9700.002 1600.015 480.003 550.008 940.002 120.005 190.003 520.002 7600.007 11Tajima'D0.084 280.329 580-1.044 6500.508 11-1.044 65-1.003 99-1.044 65-1.363 141.388 37-1.044 6501.037 44Fu and Li's D*1.550 27*0.776 190-1.991 8101.204 50-1.991 81-1.415 24-1.991 81-1.941 360.697 56-1.991 8100.006 86Fu and Li's F*1.221 430.720 140-1.988 5201.147 76-1.988 52-1.507 69-1.988 52-2.061 721.061 05-1.988 5200.401 63参数Parameters第十外显子Exon 10第十一内含子Intron 11第十一外显子Exon 11第十二内含子Intron 12第十二外显子Exon 12第十三内含子Intron 13第十三外显子Exon 13第十四内含子Intron 14第十四外显子Exon 14第十五内含子Intron 15第十五外显子Exon 153'端3'UTR全长Entire regionπ00.006 480.000 510.006 750.002 690.003 6400.008 4400.004 890.000 230.004 070.051 40θ00.007 190.002 950.012 440.004 020.006 3500.009 6600.007 540.001 950.004 300.005 52Tajima'D0-0.183 33-1.307 84-1.052 39-0.524 13-0.788 040-0.274 740-0.768 01-1.044 65-0.133 20-0.231 83Fu and Li's D*0-0.550 75-1.034 94-0.742 36-1.034 94-0.550 7501.049 5600.006 86-1.991 81-0.306 88-0.108 90Fu and Li's F*0-0.514 40-1.305 71-0.995 70-1.025 88-0.731 9000.729 380-0.287 83-1.988 52-0.293 47-0.191 92
2.2 ZmAGP2基因的单倍型分析
单倍型分析可以为育种工作者提供丰富的遗传信息。本研究通过对103份自交系的ZmAGP2基因的全长进行单倍型分析,共检测到38个单倍型(图2),单倍型Hap_1的供试材料数目最多,共38个自交系,单倍型多态性达到0.80。同时,单倍型Hap_2-Hap_38均与Hap_1表现显著性差异。Hap_2与Hap_24差异不显著,Hap_4、Hap_11、Hap_15、Hap_25和Hap_33没有显著性差异。表明自交系在每个单倍型中的分布并不均匀,单倍型Hap_1中含有的单倍型最多。
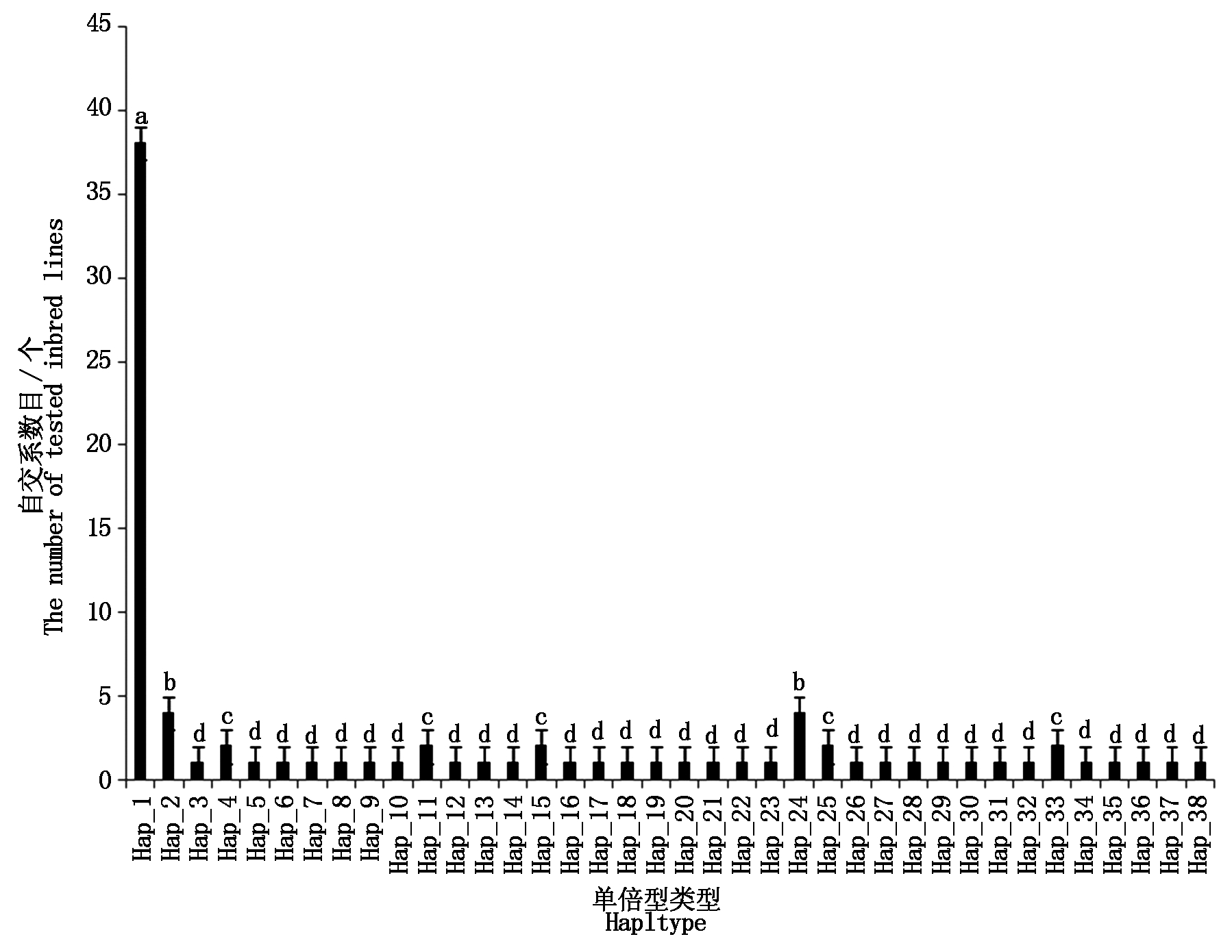
显著水平为P<0.05。
Significance at P<0.05 level.
图2 供试玉米自交系中ZmAGP2基因全长单倍型分布
Fig.2 Distribution of major haplotypes of the ZmAGP2 gene total sequences among tested inbred lines sequence
为进一步深入研究,针对 ZmAGP2基因的编码区对单倍型进行分析(图3)。共检测到8个单倍型,CDS Hap_1和CDS Hap_2包括75个自交系,占供试材料的87.2%。其中CDS Hap_1包含63个自交系,数目最多;CDS Hap_3、CDS Hap_5、CDS Hap_6、CDS Hap_7和CDS Hap_8包含自交系数目最少为1个;CDS Hap_2包含12个自交系;CDS Hap_4包含6个自交系。单倍型CDS Hap_2-8与CDS Hap_1具有显著差异,CDS Hap_2与CDS Hap_3-8也具有显著差异性,单倍型CDS Hap_3、CDS Hap_5、CDS Hap_6、CDS Hap_7和CDS Hap_8没有显著差异,说明自交系在编码区的单倍型分布也不均匀,CDS Hap_1含有的玉米自交系个数最多。
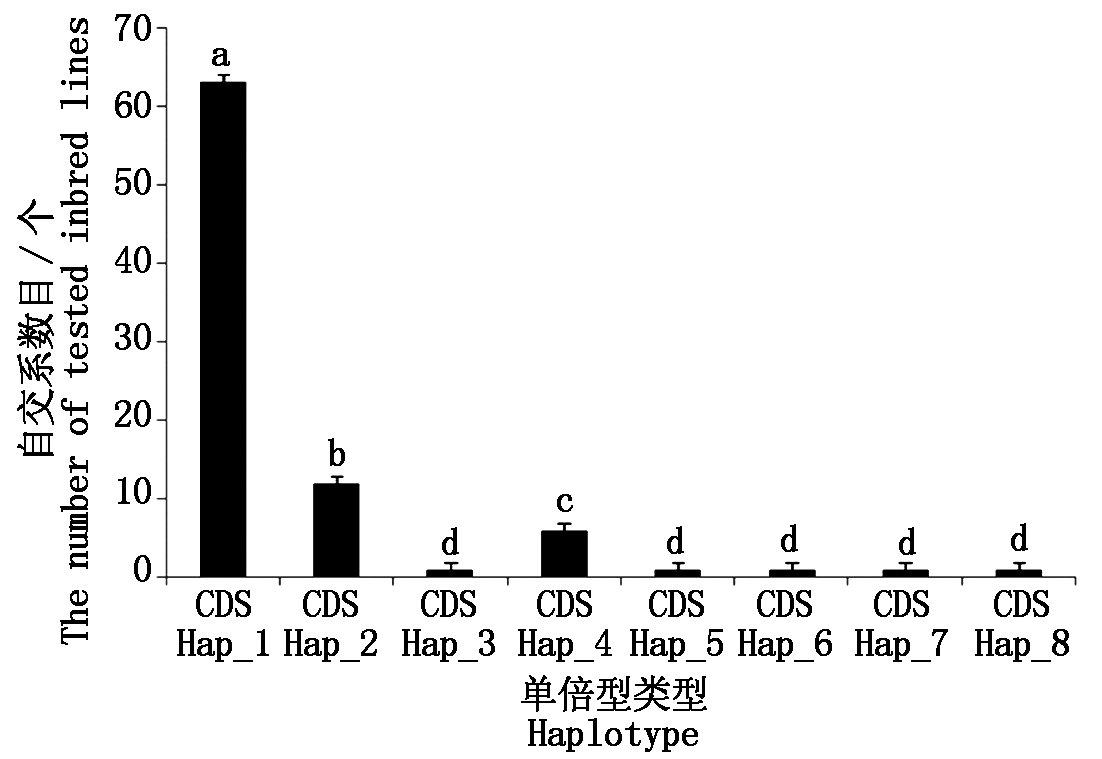
图3 供试玉米自交系中ZmAGP2基因编码区单倍型分布
Fig.3 Distribution of major haplotypes of the ZmAGP2 gene coding sequences among tested inbred lines
ZmAGP2基因共编码了5种蛋白质序列,氨基酸全长为334 aa(图4)。ZmAGP2基因编码区有9个SNP,包括3个同义突变,6个为非同义突变,后者导致氨基酸序列的突变,产生5种不同类型的ZmAGP2蛋白。
3 讨论
丰富的遗传变异使植物育种家能够创造出新的植物基因组合,并可以选择更适合不同农业系统需要的作物品种。作物功能基因遗传多样性的分析对于理解表型变异的遗传背景至关重要,进而为作物改良提供了极大的帮助[18-19]。本研究在玉米ZmAGP2基因全长序列中检测到112个变异位点,变异类型均为SNP。在这些SNP中,有9个位于编码区,其中3个为同义突变,未改变氨基酸序列,6个为非同义突变,是改变蛋白序列的最主要的原因。编码区的9个SNP将被检测的自交系划分为8个单倍型,编码5种ZmAGP2蛋白。然而发挥主要作用的可能是非编码区段的位点变异,尤其是5′UTR和内含子区段,分别检测到40,42个SNP,其中的原因可能是非编码区段的序列长度要远远大于编码区。经分析发现B73中该基因含有15个内含子和15个外显子,但对103份自交系ZmAGP2基因的全长序列进行重测序后,发现前4个内含子和前3个外显子丢失,其原因可能因为在转录过程中基因发生可变剪切或者测序过程中出现的错误导致,其碱基的大量缺失产生的性状和表型以及是否对淀粉的合成产生影响尚不清楚,需进一步的研究。Ching等[20]对36个玉米自交系进行序列变异分析,发现在非编码区每31 bp就发生一个变异位点,而在编码区每124 bp会产生一个变异位点。Zhang等[21]对73个中国优良自交系的RTCS基因的核苷酸多态性进行分析,编码区每45.9 bp有一个SNP,在非编码区每10.9 bp就有一个SNP。本研究中在非编码区每30 bp就会有一个SNP;在编码区每117 bp就有一个SNP。

图4 不同CDS单倍型编码的ZmAGP2蛋白序列比对
Fig.4 Sequence alignment of the ZmAGP2 proteins encoded by different CDS haplotypes
本研究对玉米103份自交系的ZmAGP2基因各区段以及全长进行了Tajima[16]以及Fu与Li[17]的D*和F*测验,没有检测到明显的人工选择的信号。因此,ZmAGP2基因虽然参与淀粉的合成,但是可能不是影响淀粉合成过程的关键基因。同时,若要研究该基因对产量和品质的影响时,应该考虑更大的群体。
[1] Qu J Z,Xu S T,Zhang Z Q,Chen G Z,Zhong Y Y,Liu L S,Zhang R H,Xue J Q,Guo D W.Evolutionary,structural and expression analysis of core genes involved in starch synthesis[J]. Sci Rep,2018,8(1):12736.doi:10.1038/s41598-018-30411-y.
[2] Yu G W,Lü Y N,Shen L Y,Wang Y B,Qing Y,Wu N,Li Y P,Huang H H,Zhang N,Liu Y H,Hu Y F,Liu H M,Zhang J J,Huang Y B.The proteomic analysis of maize endosperm protein enriched by Phos-tag(tm)reveals the phosphorylation of Brittle-2 subunit of ADP-Glc pyrophosphorylase in starch biosynthesis process[J]. Int J Mol Sci,2019,20(4):986.doi:10.3390/ijms20040986.
[3] Zhang S L,Guo H J,Irshad A,Xie Y D,Zhao L S,Xiong H C,Gu J Y,Zhao S R,Ding Y P,Liu L X.The synergistic effects of TaAGP.L-B1 and TaSSIVb-D mutations in wheat lead to alterations of gene expression patterns and starch content in grain development[J].PLoS One,2019,14(10):e0223783. doi:10.1371/journal.pone.0223783.
[4] Deng X,Liu Y,Xu X X,Liu D M,Zhu G R,Yan X,Wang Z M,Yan Y M. Comparative analysis of AGPase genes and encoded proteins in eight monocots and three dicots with emphasis on wheat[J].Front Plant Sci,2017,8:19. doi:10.3389/fpls.2018.00425.
[5] Slattery C J,Kavakli I H and Okita T W,Engineering starch for increased quantity and quality[J].Trends Plant Sci, 2000,5(7):291-298.doi:10.1016/s1360-1385(00)01657-5.c.
[6] Hou J,Li T,Wang Y M,Hao C Y,Liu H X,Zhang X Y. ADP-glucose pyrophosphorylase genes,associated with kernel weight,underwent selection during wheat domestication and breeding[J].Plant Biotechnol J, 2017,15(12):1533-1543.doi:10.1111/pbi.12735.
[7] Miao H X,Sun P G,Liu Q,Liu J H,Xu B Y,Jin Z Q. The AGPase family proteins in banana:Genome-wide identification,phylogeny,and expression analyses reveal their involvement in the development,ripening,and abiotic/biotic stress responses[J]. Int J Mol Sci, 2017,18(8):1581.doi:10.3390/ijms18081581.
[8] Zeeman S C,Kossmann J S,Alison M K,Smith A M.Starch:its metabolism,evolution,and biotechnological modification in plants[J].Annu Rev Plant Biol,2010,61:209-234.doi:10.1146/annurev-arplant-042809-112301.
[9] Tiessen A,Nerlich A,Faix B,Hummer C,Fox S,Trafford K,Weber H,Weschke W,Geigenberger P.Sub cellular analysis of starch metabolism in developing barley seeds using a non-aqueous fractionation method[J].J Exp Bot,2012,63(5):2071-2087.doi:10.1093/jxb/err408.
[10] Smidansky E D,Clancy M,Meyer F D,Lanning S P,Blake N K,Talbert L E,Giroux M J.Enhanced ADP-glucose pyrophosphorylase activity in wheat endosperm increases seed yield[J]. Proc Natl Acad Sci USA,2002,99(3):1724-1729. doi:10.1073/pnas.022635299.
[11] Rose M K,Huang X Q,BruleB A. Molecular characterization and sequence diversity of genes encoding the large subunit of the ADP-glucose pyrophosphorylase in wheat(Triticum aestivum L.)[J]. J Appl Genet, 2016,57(1):15-25.doi:10.1007/s13353-015-0298-1.
[12] Deng X,Liu Y,Xu X X,Liu D M,Zhu G R,Yan X,Wang Z M,Yan Y M.Comparative proteome analysis of wheat flag leaves and developing grains under water deficit[J]. Front Plant Sci,2018,9:425.doi:10.3389/fpls.2018.00425.
[13] Chen G X,Zhen S M,Liu Y L,Yan X,Zhang M,Yan Y M. In vivo phosphoproteome characterization reveals key starch granule-binding phosphoproteins involved in wheat water-deficit response[J]. BMC Plant Biol,2017,17(1):168.doi:10.1186/s12870-017-1118-z.
[14] Murray M G,Thompson W F.Rapid isolation of high molecular weight plant DNA[J].Nucleic Acids Res,1980,8(19):4321-4325.doi:10.1093/nar/8.19.4321.
[15] Rozas J, Ferrer-Mata A, Sánchez-DelBarrio J C, Guirao-Rico S, Librado P, Ramos-Onsins S E , Sánchez-Gracia A . DnaSP 6:DNA sequence polymorphism analysis of large data pets[J]. Mol Biol Evol,2017,34(12):3299-3302.doi:10.1093/molbev/msx248.
[16] Tajima F.Statistical method for testing the neutral mutation hypothesis by DNA polymorphism[J].Genetics,1989,123(3):585-595.
[17] Fu Y X,Li W H.Statistical tests of neutrality of mutations[J]. Genetics,1993,133(3):693-709.
[18] Xiao Y J,Liu H J,Wu L J,Warburton M,Yan J B. Genome-wide association studies in maize:praise and stargaze[J].Mol Plant, 2017,10(3):359-374.doi:10.1016/j.molp.2016.12.008.
[19] Mafra G S,Amaral J,Antonio T D,Almeida F,Janeo E D,Vivas M,Santos P H A D,Santos J S,Pena G F,Lima V J D,Kamphorst S H,Oliveira FT D,Souza Y P D,Schwantes I A,Santos T D,Oliveira B,Rosimeire B,Maldonado C,Mora F. SNP-based mixed model association of growth-and yield-related traits in popcorn[J]. PLoS One,2019,14(6):e0218552. doi:10.1371/journal.pone.0218552.
[20] Ching A,Caldwell K S,Jung M,Dolan M,Smith O S,Tingey S,Morgante M,Rafalski A J.SNP frequency,haplotype structure and linkage disequilibrium in elite maize inbred lines[J]. BMC Genet,2002,3:19.doi:10.1186/1471-2156-3-19.
[21] Zhang E Y,Yang Z F,Wang Y F,Hu Y Y,Song X Y,Xu C W. Nucleotide polymorphisms and haplotype diversity of RTCS gene in China elite maize inbred lines[J].PLoS One,2013,8(2):e56495.doi:10.1371/journal.pone.0056495.