C1q(Complement 1)分子由6个亚单位共18条多肽链组成,C1QA、C1QB、C1QC基因分别编码补体C1q一个亚单位的A、B、C 3条多肽链[1]。C1q对体内病原体的清除、氧化应激、细胞凋亡、维护机体内环境稳定等过程发挥着重要作用[2]。
C1QA、C1QB、C1QC基因的相关研究在人[3]、鼠[4]和农业经济动物中均有报道。在人和鼠中,以上基因的研究主要集中在缺氧心肌血管的形成[5-7]、系统性红斑狼疮和免疫性肾病等方面[8]。在2000年Tohgi 等[9]研究中发现缺氧后的小鼠PC12细胞会立即表达补体C1q mRNA,细胞通过复氧翻译出C1q蛋白的A、B、C链,以致缺氧细胞受损。2003年Luo等[10]研究证实大脑缺氧后C1q沉积是诱导阿尔茨海默病中神经元死亡和氧化应激的关键因素,即C1q是导致缺氧细胞受损的重要原因。Theerut等[11]在2018年的研究中发现,缺氧组织中补体C1q沉积会诱导巨噬细胞刺激中性粒细胞产生活性氧ROS。尽管ROS过量会干扰细胞的正常功能导致细胞抗氧化防御能力的减弱和病变[12],但同时ROS又可激活相关信号分子、细胞因子,作为调节氧化应激的重要转录因子,调节肝脏中抗氧化防御相关基因的表达[13]。综上所述,体内C1q蛋白的沉积可导致机体产生ROS来激活氧化应激相关转录因子,使得机体的内源性抗氧化系统增强,从而缓解机体的氧化应激反应。
在农业经济动物中,C1QA、C1QB、C1QC基因在猪[14]、普通牛[15]等哺乳动物中均已被克隆和测序,研究显示C1QA、C1QB、C1QC基因在绵羊各个组织中也均有表达[16]。在笔者先前的研究中已对牦牛C1QA、C1QB、C1QC基因进行了克隆、组织表达和生物信息学分析[17],但以上基因是否与牦牛高原适应性相关也成为本研究探讨的主要方面。牦牛因长期生活在高寒、低氧、强紫外线照射等特殊环境中[18-19],这些因素均使牦牛体内产生氧化应激,因此,C1QA、C1QB、C1QC基因在牦牛体内的时空表达状况可以反映出牦牛对高原环境的适应性。
本研究以麦洼牦牛为研究对象,通过RT-qPCR检测牦牛C1QA、C1QB、C1QC基因在牦牛0.5,1.5,2.5岁的心脏、脾脏、肺脏、大脑、小脑组织中的表达来分析其在牦牛高原适应性中的作用。
1 材料和方法
1.1 试验材料
样本采自四川省阿坝藏族羌族自治州红原县,选取0.5,1.5,2.5岁麦洼牦牛各3头,屠宰后采集9头牦牛的心脏、肺脏、大脑、脾脏、小脑组织,用新配置的DEPC水预处理后立即保存于液氮中。
1.2 试验方法
1.2.1 引物设计与合成 根据NCBI-GenBank中公布的牦牛C1QB(XM_005905180.2)、C1QA(XM_005905182.20、C1QC(XM_005905183)基因mRNA 的预测序列,利用Primer Premier 5.0软件设计荧光定量引物,由擎科生物工程(上海)股份有限公司合成(表1)。
表1 RT-qPCR引物序列
Tab.1 RT-qPCR primer sequence

基因Gene引物序列(5'-3')Primer sequence(5'-3')Tm/℃产物长度/bpProduct size用途ApplicationC1QAF:GTGGTCGTCTTCGGAAAGGT59133RT-qPCRR:ACAGGCAGATGTCCCAGTTGC1QBF:AGCCACCTCAACGAGAATGG57141RT-qPCRR:GTCTCCCGATTCACCCTTGGC1QCF:CTAAACCTGCTGCTGCTCCT58118RT-qPCRR:CGTCATACCCATCCTTCCCGβ-actinF:TGATGATATTGCTGCGCTCG57153RT-qPCRR:TACGAGTCCTTCTGGCCCAT
1.2.2 组织总RNA提取和质量检测 利用液氮将心脏、脾脏、肺脏、大脑、小脑组织样本于研钵中迅速研磨至粉末状后转移至无RNase 的1.5 mL离心管中,加入1 mL TRIzol(Invitrogen)试剂,使组织粉末完全溶解。根据TRIzol法提取所需组织的总RNA。用NanoDropTM One 2000分光光度计检测RNA样品的浓度、纯度,总RNA的完整性用1.0%琼脂糖凝胶电泳检测。
1.2.3 RT-PCR cDNA 的制备根据PrimeScriptTM RT reagent Kit with gDNA Eraser(Perfect Real Time)(TaKaRa)反转录试剂盒的操作手册完成,并保存于-20 ℃备用。
1.2.4 实时荧光定量PCR 为实时定量PCR检测C1QA、C1QB、C1QC基因在心脏、脾脏、肺脏、大脑和小脑组织中的相对表达量,本试验选用TB GreenTM Premix Ex TaqTMⅡ试剂(TaKaRa),具体反应体系和程序详见文献[20]。每个样品设置3个生物学重复,3个技术重复,选择牦牛β-actin作为内参基因,并利用熔解曲线来鉴定引物的特异性,记录样品Ct值,利用 2-ΔΔCt 法计算样本的相对表达量。
基因在组织中的相对表达柱状图用GraphPad Prism 5绘制,使用SPSS 22.0软件分析各组织间的差异显著性,P<0.01表示差异极显著,P<0.05表示差异显著,P>0.05表示差异不显著。
2 结果与分析
RT-qPCR检测C1QA、C1QB、C1QC 基因在0.5,1.5,2.5岁牦牛心脏、脾脏、肺脏、大脑和小脑组织中的表达水平,荧光定量结果显示C1QA、C1QB、C1QC基因在牦牛3个年龄段各组织中均有表达,具体结果如下。
2.1 C1QA、C1QB、C1QC基因在麦洼牦牛不同年龄段的表达差异
2.1.1 C1QA基因在麦洼牦牛同组织3个年龄中的差异表达分析 时序表达结果显示(图 1), C1QA基因随着牦牛年龄的增加,在肺脏中的表达量逐渐下降,大、小脑中是先下降后增加,在心脏中则是先上升后下降,其在牦牛1.5岁的心脏和0.5岁的脾脏、肺脏、大脑、小脑组织中表达量最高。该基因在牦牛1.5岁心脏中的表达量显著高于0.5岁中的表达量(P<0.05),极显著高于2.5岁中的表达量(P<0.01),在2.5与0.5岁间差异不显著(P > 0.05);C1QA基因在2.5岁脾脏中的表达量显著高于1.5岁(P<0.05),在1.5,2.5岁脾脏中的表达量极显著低于0.5岁脾脏(P<0.01);该基因在牦牛0.5岁肺脏中的表达量极显著高于0.5,2.5岁中肺脏的表达量(P<0.01),在2.5,1.5岁间则差异不显著(P>0.05);C1QA在牦牛2.5岁大脑中的表达量显著高于1.5岁大脑中的表达量(P<0.05),1.5岁大脑的表达量极显著低于0.5岁中的表达量(P<0.01),2.5与0.5岁大脑间差异不显著(P>0.05);C1QA基因在牦牛0.5岁小脑中的表达量极显著高于1.5,2.5岁(P<0.01),2.5岁小脑中的表达量显著高于1.5岁(P<0.05)。
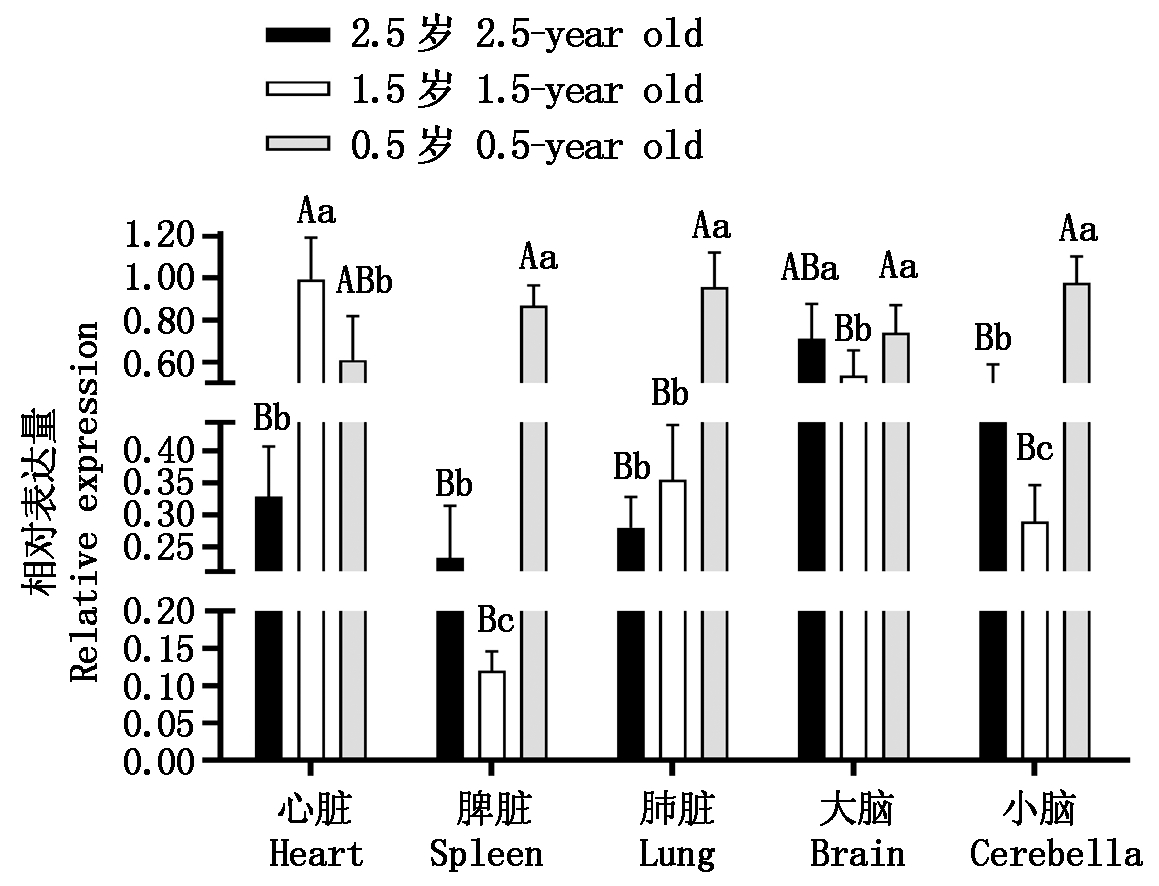
同组织间不同大写字母表示差异极显著( P<0.01);不同小写字母表示差异显著( P<0.05);相同字母表示差异不显著( P>0.05);不同组织间不做显著性分析。图2-3同。
In the same tissue different capital letters mean extremely significant difference(P<0.01);Different lowercase letters mean significant difference(P<0.05);The same letters mean no significant difference(P>0.05);No significance analysis was done between the different tissues.The same as Fig.2-3.
图1 C1QA在牦牛各组织发育阶段的表达量
Fig.1 Expression of C1QA in yak tissues at different stages of development
2.1.2 C1QB基因在麦洼牦牛同组织3个年龄中的差异表达分析 RT-qPCR结果显示(图 2), C1QB在组织中的表达量随牦牛年龄的增加呈先上升后下降的趋势,在麦洼牦牛1.5岁各组织中的表达量最高。该基因在牦牛1.5岁心脏中的表达量极显著高于2.5,0.5岁中的表达量(P<0.01),在0.5岁中的表达量极显著高于2.5岁中的表达量(P<0.01);C1QB基因在牦牛1.5岁脾脏中的表达量极显著高于0.5,2.5岁(P<0.01),在2.5与0.5岁间差异不显著(P>0.05);它在牦牛1.5岁肺脏中的表达量极显著高于0.5,2.5岁中的表达量(P<0.01),在0.5岁中的表达量显著高于2.5岁中的表达量(P<0.05);该基因在牦牛1.5岁大脑中的表达量显著高于2.5岁中的表达量(P<0.05),极显著高于0.5岁中的表达量(P<0.01),2.5岁中表达量也极显著高于0.5岁中的表达量(P<0.01);C1QB基因在牦牛1.5岁小脑中的表达量极显著高于2.5,0.5岁(P<0.01),在0.5岁中的表达量显著高于2.5岁(P<0.05)。
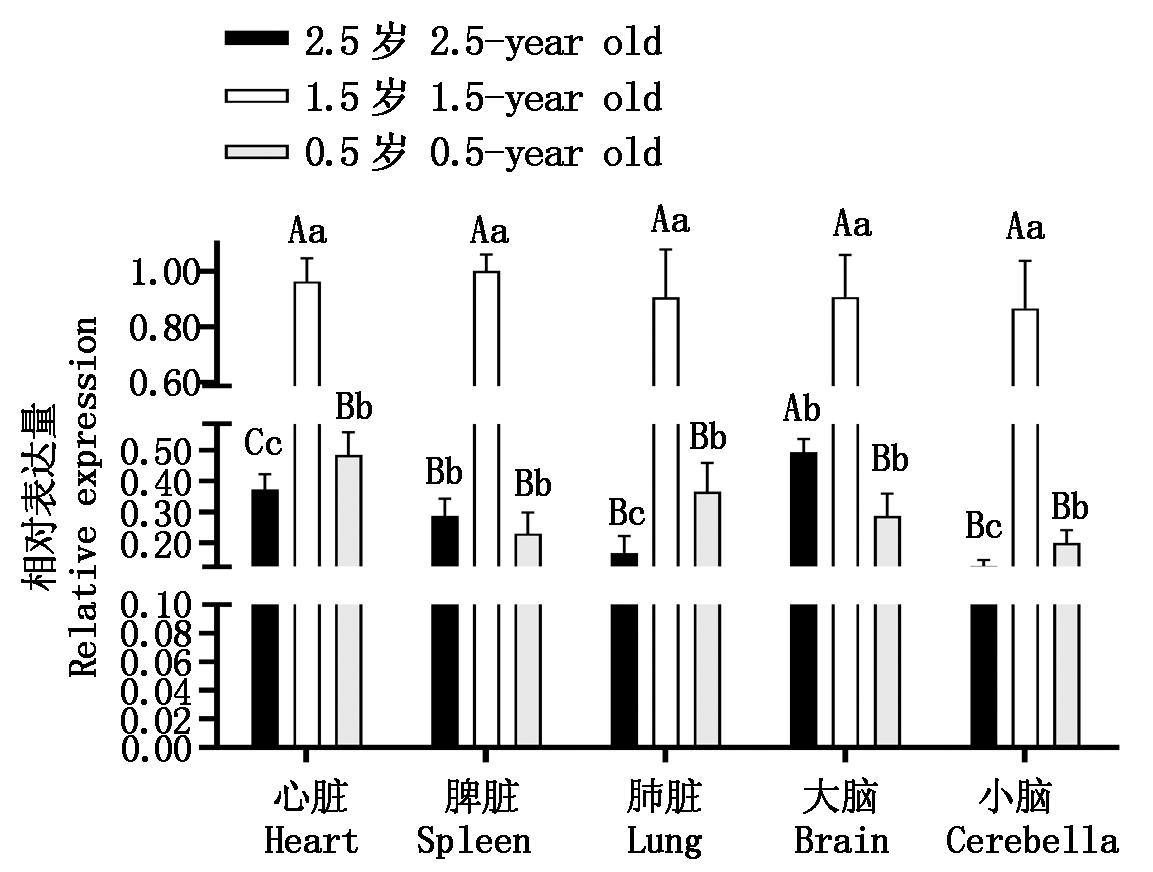
图2 C1QB在牦牛各组织发育阶段的表达量
Fig.2 Expression of C1QB in yak tissues at different stages of development
2.1.3 C1QC基因在麦洼牦牛同组织3个年龄中的差异表达分析 组织表达结果显示(图 3),随着牦牛年龄的增加C1QC基因在心脏、大脑、小脑组织中的表达量呈逐渐上升的趋势,在脾脏、肺脏中则是先上升后下降。在牦牛2.5岁心脏、大脑、小脑组织和1.5岁脾脏、肺脏组织中C1QC基因的表达量最高。C1QC基因在牦牛0.5,1.5,2.5岁心脏中的表达量差异不显著(P>0.05);C1QC基因在牦牛2.5岁脾脏中的表达量显著高于0.5岁(P<0.05),1.5岁脾脏中的表达量极显著高于0.5岁(P<0.01),在2.5,1.5岁间差异不显著(P>0.05);该基因在牦牛1.5岁肺脏中的表达量极显著高于0.5,2.5岁中的表达量(P<0.01),在2.5,1.5岁间差异不显著(P>0.05);该基因在牦牛2.5岁大脑中的表达量极显著高于1.5,0.5岁中的表达量(P<0.01),在1.5,0.5岁间差异不显著(P>0.05);C1QC基因在牦牛2.5岁小脑中的表达量极显著高于1.5,2.5岁(P<0.01),在1.5岁中的表达量显著高于0.5岁(P<0.05)。
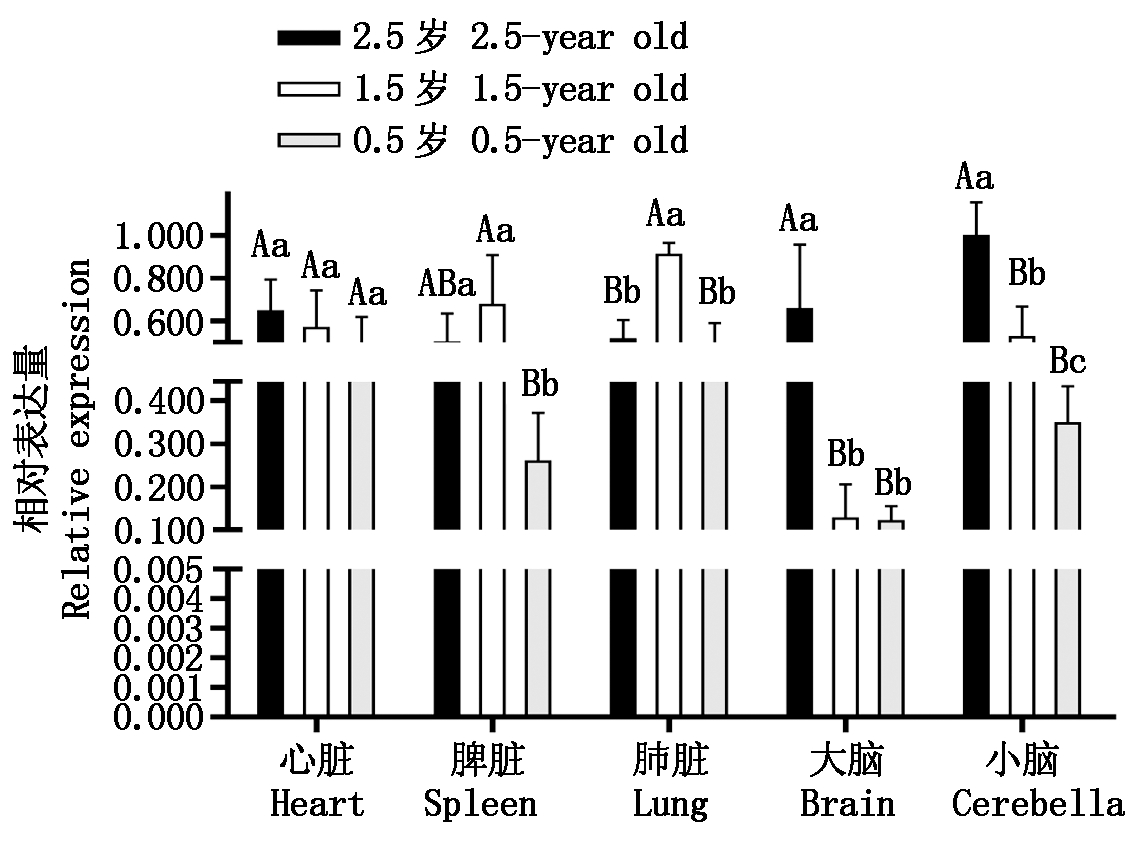
图3 C1QC在牦牛各组织发育阶段的表达量
Fig.3 Expression of C1QC in yak tissuesat different stages of development
2.2 C1QA、C1QB、C1QC基因在麦洼牦牛不同组织中的表达谱
2.2.1 C1QA基因在麦洼牦牛0.5,1.5,2.5岁中的组织表达谱 定量结果表明(图4),C1QA基因在0.5,1.5,2.5岁牦牛肺脏中的表达量最高,脾脏次之,然后是心脏,最后是大脑、小脑。其中C1QA基因在0.5岁牦牛肺脏中的表达量显著高于脾脏(P<0.05),而在1.5,2.5岁牦牛肺脏中的表达量是极显著高于脾脏(P<0.01);C1QA基因在0.5,2.5岁牦牛脾脏中的表达量极显著高于心脏(P<0.01),1.5岁时期两者差异不显著(P>0.05);C1QA基因在1.5,2.5岁牦牛心脏中的表达量极显著高于大脑和小脑(P<0.01),在0.5岁时期心脏中的表达量显著高于大脑和小脑(P<0.05);而0.5,1.5,2.5岁牦牛的大脑与小脑中的表达量差异不显著(P>0.05)。
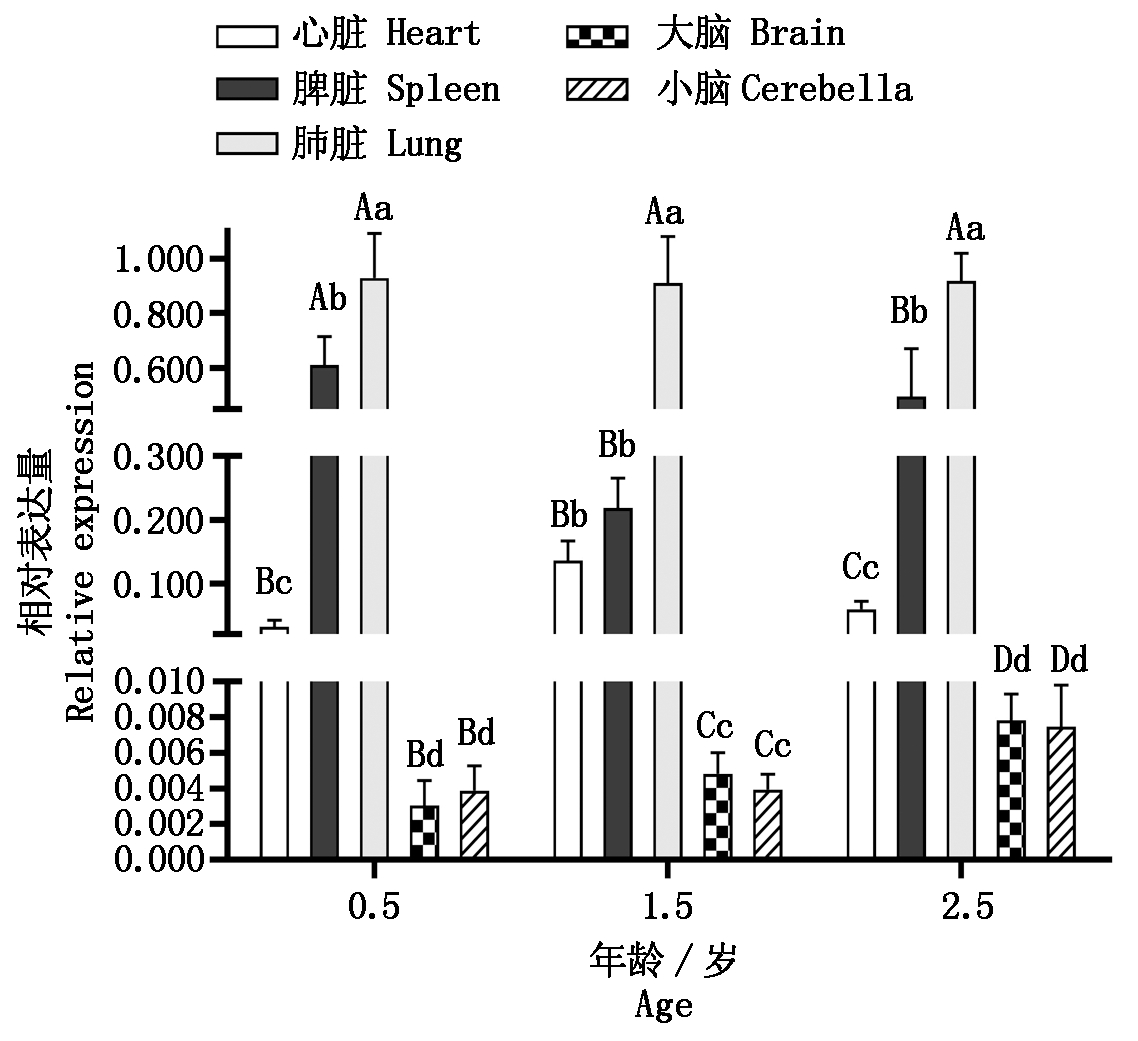
同年龄间不同大写字母表示差异极显著( P<0.01);不同小写字母表示差异显著(P<0.05);相同字母表示差异不显著(P>0.05);不同年龄间不做比较。图5-6同。
In the same ages different capital letters mean extremely significant difference(P<0.01);Different lowercase letters mean significant difference(P<0.05);The same letters mean no significant difference(P>0.05);No comparison was made between different ages.The same as Fig.5-6.
图4 C1QA在不同年龄牦牛组织中的表达
Fig.4 Expression of C1QA in yak tissues of different ages
2.2.2 C1QB基因在麦洼牦牛0.5,1.5,2.5岁中的组织表达谱 结果显示(图5),C1QB基因在0.5,1.5,2.5岁牦牛肺脏中的表达量最高,脾脏次之,然后是心脏,最后是大脑、小脑。其中C1QB基因在0.5岁牦牛肺脏中的表达量极显著高于脾脏、心脏、大脑、小脑(P<0.01),在1.5岁肺脏中的表达量极显著高于脾脏、心脏、大脑、小脑(P<0.01),而在2.5岁肺脏与脾脏间表达量差异不显著(P>0.05);C1QB基因在3个年龄牦牛脾脏中的表达量均极显著高于心脏、大脑和小脑(P<0.01),0.5岁的表达量显著高于心脏(P<0.05);C1QB基因在1.5,2.5岁牦牛心脏中的表达量极显著高于大脑和小脑(P<0.01);而0.5,1.5,2.5岁牦牛的大脑与小脑中的表达量差异不显著(P>0.05)。
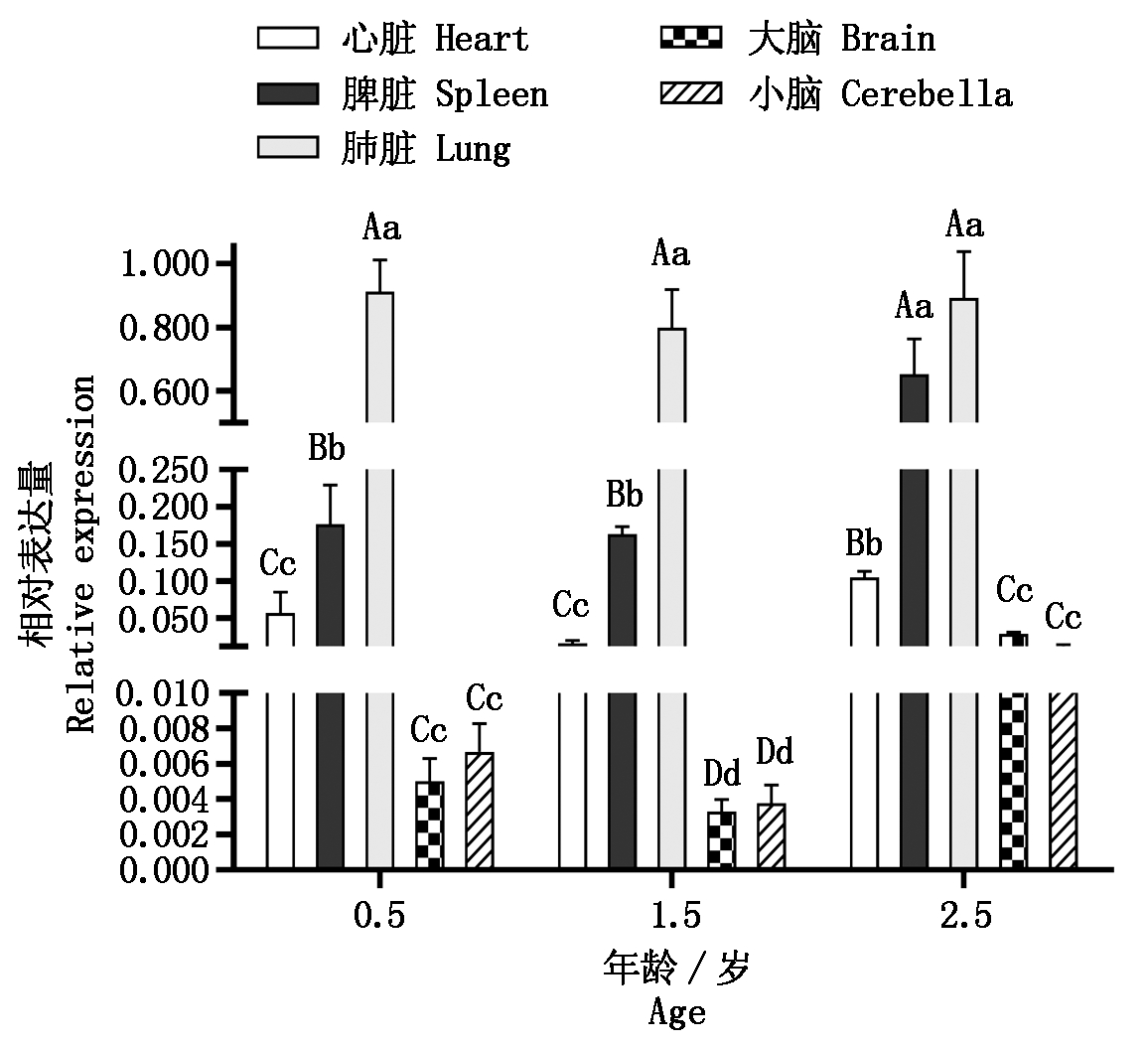
图5 C1QB在不同年龄牦牛组织中的表达
Fig.5 Expression of C1QB in yak tissues of different ages
2.2.3 C1QC基因在麦洼牦牛0.5,1.5,2.5岁中的组织表达谱 结果表明(图6),C1QC基因在0.5,1.5,2.5岁牦牛肺脏中的表达量最高,脾脏次之,然后是心脏,最后是大脑、小脑。其中C1QC基因在0.5岁牦牛肺脏中的表达量显著高于脾脏(P<0.05),而在1.5岁牦牛肺脏中的表达量是极显著高于脾脏(P<0.01),而在2.5岁肺脏与脾脏间表达量不显著(P>0.05);C1QC基因在1.5,2.5岁牦牛脾脏中的表达量极显著高于心脏(P<0.01),0.5岁时期两者差异显著(P<0.05);C1QC基因在1.5,2.5岁牦牛心脏中的表达量极显著高于大脑和小脑(P<0.01),在0.5岁时心脏中的表达量显著高于大脑和小脑(P < 0.05);而0.5,1.5,2.5岁牦牛的大脑与小脑中的表达量差异不显著(P>0.05)。
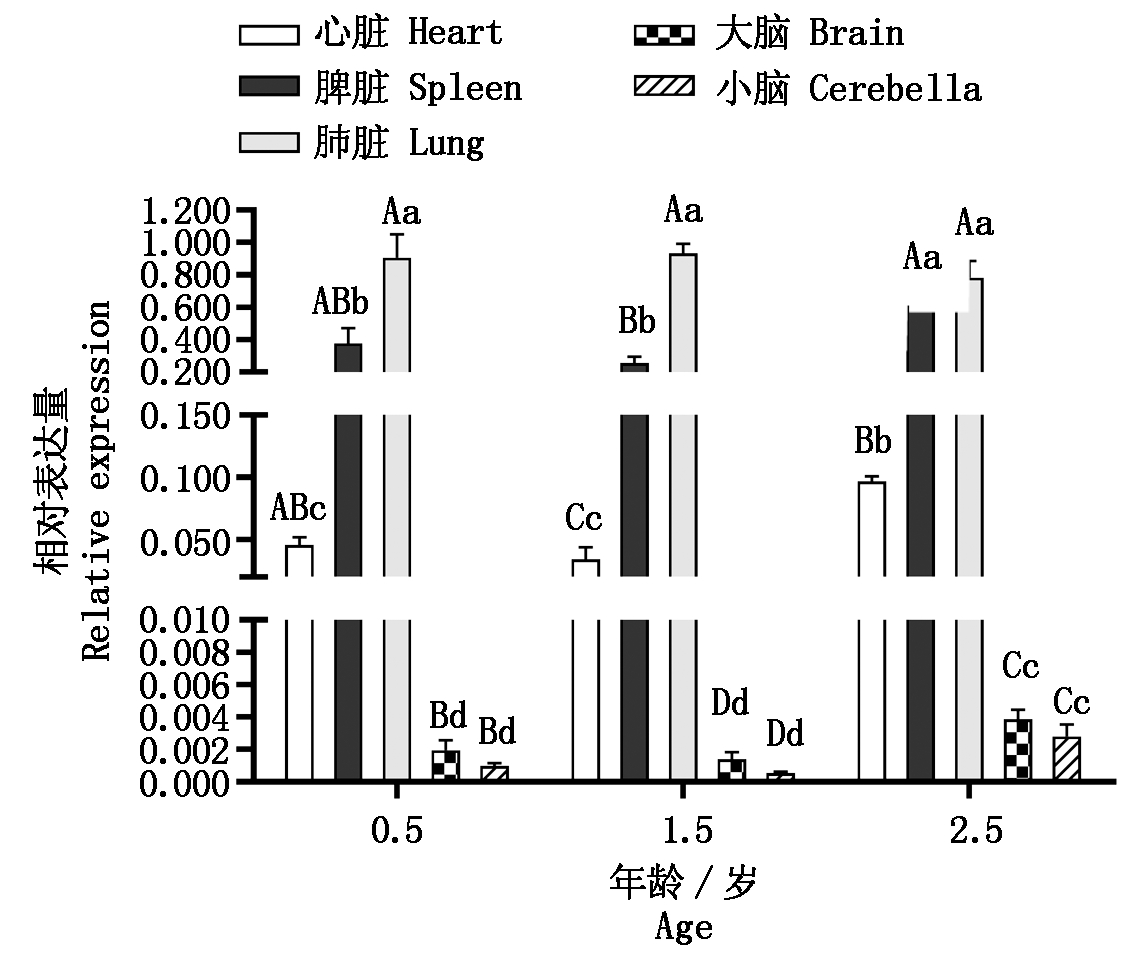
图6 C1QC在不同年龄牦牛组织中的表达
Fig.6 Expression of C1QC in yak tissues of different ages
3 讨论与结论
研究证明,C1q在人和小鼠缺氧心肌中血管的生成[6-7]、机体内源性抗氧化系统增强[13]等方面发挥作用,C1QA、C1QB、C1QC基因作为构成补体C1q的亚单位,在牦牛体内的表达量对C1q形成及C1q功能的发挥至关重要。本试验通过研究C1QA、C1QB、C1QC基因在麦洼牦牛0.5,1.5,2.5岁的心脏、脾脏、肺脏、大脑、小脑组织中的组织表达和时序表达,为C1QA、C1QB、C1QC基因在牦牛高原适应性中具体作用机制提供基础数据。
时序表达结果显示,随着牦牛年龄的增加C1QA基因在多数组织中的表达量呈显著下降趋势,C1QB基因的表达量则是先显著上升再显著下降,而C1QC基因在多数组织中的表达量则呈显著上升趋势,少数组织中趋势不一致,这与梁栋等[21]研究显示,C1q蛋白在人大脑海马组织中随年龄增加表达明显增加不完全一致;与陈文举等[22]在健康成人血清中发现补体C1q的含量存在性别差异,与年龄呈正相关也不完全一致,可能因为C1QA、C1QB、C1QC基因在不同组织中的表达量与研究物种遗传结构的性别[22]及所处的环境均存在一定关系[23]。表明C1QA、C1QB、C1QC基因在牦牛不同生长发育阶段发挥作用不同,但具体分子机制有待进一步研究。
C1QA、C1QB、C1QC基因同年龄不同组织定量结果显示,各年龄段组织中的表达趋势基本一致,均为肺脏显著高于脾脏、心脏、大脑、小脑;脾脏显著高于心脏、大脑、小脑;而心脏显著高于大脑、小脑,大脑和小脑间差异不显著。其中牦牛0.5岁时期C1QA、C1QB、C1QC基因在肺脏中的表达量显著高于脾脏(P<0.05),到了1.5岁时期则极显著高于脾脏(P<0.01),2.5岁时期则只有C1QA极显著高于脾脏(P<0.01),C1QB显著高于脾脏(P<0.05),C1QC基因则差异不显著(P>0.05)。与Feng等[24]发现小家鼠脾脏中表达量最高,在大脑和小脑中的表达量低于脾脏、肺脏的表达趋势一致,但与3个基因在成年小家鼠脾脏中的表达量显著高于成年小家鼠肺脏和Jiang等[16]在绵羊脾脏中发现其表达量最高,其次是肺脏的结果不同,说明C1QA、C1QB、C1QC基因的组织表达具有物种特异性,也可能与牦牛高原适应性和氧化应激调节造成,因为补体C1q可以促进机体内源性抗氧化系统增强[14],使牦牛更适应高原低氧环境。C1QA、C1QB、C1QC基因在各物种脾脏中表达量都较高是因为补体C1q分子是免疫蛋白,而脾脏是重要的免疫器官,同时也是机体的“血库”,当机体在失血、缺氧等应激状态时将血液排送到血循环中增加血容量,保证机体的能量供应[25-27]。与本研究发现3个基因在牦牛各发育时期的脾脏中表达量都较高一致;而在牦牛肺脏组织中表达量与其他物种存在差异的原因可能是C1QA、C1QB、C1QC基因参与调控牦牛肺部低氧适应机制。
本研究定量检测C1QA、C1QB、C1QC基因在2.5,1.5,0.5岁3个年龄段麦洼牦牛的心脏、脾脏、肺脏、大脑和小脑5个组织中的相对表达量以及探索3个基因的时空表达特性,为进一步研究C1QA、C1QB、C1QC基因在牦牛高原适应性中的分子机制提供基础数据。
[1] Lu J H,Teh B K,Wang L,Wang Y N,Tan Y S,Lai M C,Reid K B M.The classical and regulatory functions of clq in immunity and autoimmunity[J].Cellular & Molecular Immunology,2008(1):9-21.doi:10.1038/cmi.2008.2.
[2] Kishore U,Gaboriaud C,Waters P,Shrive A K,Greenhough T J,Reid K B M,Sim R B,Arlaud G J.C1q and tumor necrosis factor superfamily:modularity and versatility[J].Trends in Immunology,2004,25(10):551-561.doi:10.1016/j.it.2004.08.006.
[3] Kishore U,Reid K.C1q:Structure,function,and receptors[J].Immunopharmacology,2000,49(1-2):159-170.doi:10.1016/S0162-3109(00)80301-X.
[4] Sellar G C,Blake D J,Reid K B.Characterization and organization of the genes encoding the A-,B-and C-chains of human complement subcomponent C1q.The complete derived amino acid sequence of human C1q[J].Biochemical Journal,1991,274(2):481-490.doi:10.1042/bj2740481.
[5] Petry F,Reid K B,Loos M.Gene expression of the A-and B-chain of mouse C1q in different tissues and the characterization of the recombinant A-chain[J].Journal of Immunolog,1991,147(11):3988-3993.
[6] Atlante A,Calissano P,Bobba A,Giannattasio S,Marra E,Passarella S.Glutamate neurotoxicity,oxidative stress and mitochondria[J].FEBS Letters,2001,497(1):1-5.doi:10.1016/S0014-5793(01)02437-1.
[7] Ten V S,Sosunov S A,Mazer S P,Stark R I,Pinsky D J.C1q-deficiency is neuroprotective against hypoxic-ischemic brain injury in neonatal mice[J].Stroke,2005,36(10):2244-2250.doi:10.1161/01.STR.0000182237.20807.d0.
[8] 于歌,谢风.补体C1q(complement C1q)在临床研究中的最新进[J].中国实验诊断学,2018,22(5):920-922.doi:10.3969/j.issn.1007-4287.2018.05.055.
Yu G,Xie F.The latest progress of complement C1q in clinical research[J].Chinese Journal of Laboratory Diagnosis,2018,22(5):920-922.
[9] Tohgi H,Utsugisawa K,Nagane Y.Hypoxia-induced expression of C1q,a subcomponent of the complement system,in cultured rat PC12 cells[J].Neuroscience Letters,2000,291(3):151-154.doi:10.1016/S0304-3940(00)01399-9.
[10] Luo X,Weber G.A,Zheng J,Gendelman H E,Ikezu T.C1q-calreticulin induced oxidative neurotoxicity:relevance for the neuropathogenesis of Alzheimer′s disease[J].Journal of Neuroimmunology,2003,135(1):62-71.doi:10.1016/S0165-5728(02)00444-7.
[11] Theerut L,Su S,Henricus A M,Geny M M,Peter O,Miriam B.Targeting oxidative stress for the treatment of liver fibrosis[J].Rev Physiol Biochem Pharmacol,2018,175:71-102.doi:10.1007/112_2018_10.
[12] 赵杰,齐永芬,鱼艳荣.氧化应激在肝纤维化发生发展中的作用[J].临床肝胆病杂志,2019,35(9):2067-2071.
Zhao J,Qi Y F,Yu Y R.Research advances in the role of oxidative stress in the development and progression of liver fibrosis[J].Journal of Hepatology,2019,35(9):2067-2071.
[13] Mi S S,Hye Y J,Hwan K S.Role of the Nrf2-ARE pathway in liver diseases[J].Oxid Med Cell Longev,2013,2013:763257.doi:10.1155/2013/763257.
[14] Fahrenkrug S C,Smith T P L,Freking B A,Cho J,White J,Vallet J,Wise T,Rohrer G,Pertea G,Sultana R,Quackenbush,Keele J W.Porcine gene discovery by normalized cDNA-library sequencing and EST cluster assembly[J].Mammalian Genome,2002,13(8):475-478.doi:10.1007/s00335-001-2072-4.
[15] Harhay G P,Sonstegard T S,Keele J W,Heaton M P,Clawson M L,Snelling W M,Wiedmann R T,Tassell C P V,Smith T P L S.Characterization of 954 bovine full-CDS cDNA sequences[J].Bmc Genomics,2005,6(1):166-170.doi:10.1186/1471-2164-6-166.
[16] Jiang Y,Xie M,Chen W,Talbot R,Maddox J F,Faraut T,Wu C,Muzny D M,Li Y,Zhang W,Stanton J A,Brauning R,Barris W C,Hourlier T,Aken B L,Searle S M J,Adelson D L,Bian C,Cam G R,Chen Y,Cheng S,DeSilva U,Dixen K,Dong Y,Fan G,Franklin I R,Fu S,Guan R,Highland M A,Holder M E,Huang G,Ingham A B,Jhangiani S N,Kalra D,Kovar C L,Lee S L,Liu W,Liu X,Lu C,Lü T,Mathew T,McWilliam S,Menzies M,Pan S,Robelin D,Servin B,Townley D,Wang W,Wei B,White S N,Yang X,Ye C,Yue Y,Zeng P,Zhou Q,Hansen J B,Kristensen K,Gibbs R A,Flicek P,Warkup C C,Jones H E,Oddy V H,Nicholas F W,McEwan J C,Kijas J,Wang J,Worley K C,Archibald A L,Cockett N,Xu X,Wang W,Dalrymple B P.The sheep genome illuminates biology of the rumen and lipid metabolism[J].Science,2014,344(6188):1168-1173.doi:10.1126/science.1252806.
[17] 钟美,王吉坤,柴志欣,王会,王嘉博,武志娟,信金伟,张成福,姬秋梅,钟金城.类乌齐牦牛C1QA、C1QB、C1QC基因克隆与组织表达分析[J].华北农学报,2020,35(4):211-221.doi:10.7668/hbnxb.20190863.
Zhong M,Wang J K,Chai Z X,Wang H,Wang J B,Wu Z J,Xin J W,Zhang C F,Ji Q M,Zhong J C.Cloning and tissue expression of C1QA,C1QB and C1QC genes in yak[J].Acta Agriculturae Boreali-Sinica,2020,35(4):211-221.
[18] 范国琴,李振宇,徐开林.DNA氧化损伤及其修复酶与肿瘤关系的研究进展[J].国际输血及血液学杂志,2015,38(2):174-177.doi:10.3760/cma.j.issn.1673-419X.2015.02.020.
Fan G Q,Li Z N,Xu K L.Research progress of relationship among DNA oxidative damage,its repair enzymes and neoplasms[J].International Journal of Blood Transfusion and Hematology,2015,38(2):174-177.
[19] Cadet J,Vigny P.The photochemistry of nucleic acids[M]//Bioorganic photochemistry.New York:John Wiley & Sons,1990:1-272.
[20] 侯孟典,王会,柴志欣,王吉坤,信金伟,姬秋梅,钟金城.牦牛RETN基因克隆及组织表达分析[J].黑龙江畜牧兽医,2019(7):123-128,171.doi:10.13881/j.cnki.hljxmsy.2018.09.0091.
Hou M D,Wang H,Chai Z X,Wang J K,Xin J W,Ji Q M,Zhong J C,Cloning and tissue expression of yak RETN gene[J].Heilongjiang Animal Science and Veterinary Medicine,2019(7):123-128,171.
[21] 梁栋,韩琪,陈飞,曹楷,郑茂金,王超群.补体C1q蛋白在人体海马中表达的増龄性改变及其突触清除作用的研究[J].医药卫生,2016(9):46-48.
Liang D,Han Q,Chen F,Cao K,Zheng M J,Wang C Q.Aging changes of complement C1q protein expression in human hippocampus and its synaptic scavenging effect[J].Medicine and Health,2016(9):46-48.
[22] 陈文举,徐佳佳.免疫透射比浊法检测健康成人血清补体C1q参考区间的初步建立[C]∥2016年浙江省检验医学学术年会论文汇编.湖州,2016.
Chen W J,Xu J J.Preliminary establishment of serum complement C1q reference interval for healthy adults by immunoturbidimetry[C]∥ Papers of zhejiang Provincial Annual Conference of Laboratory Medicine in 2016.Huzhou,2016.
[23] Kondrashov N,Pusic A,Stumpf C R,Shimizu K,Hsieh A C,Xue S F,Ishijima J,Shiroishi T,Ribosomemediated M B.Specificity in Hox mRNA translation and vertebrate tissue patterning[J].Cell,2011,145:383-397.doi:10.1016/j.cell.2011.03.028.
[24] Feng Y,Cheng Y,Breschi A,Vierstra J,Wu W,Ryba T,Sandstrom R,Ma Z,Davis C,Pope B D,Shen Y,Pervouchine D D,Djebali S,Thurman R E,Kaul R,Rynes E,Kirilusha A,Marinov G K,Williams B A,Trout D,Amrhein H,Fisher-Aylor K,Antoshechkin I,DeSalvo G,See L H,Fastuca M,Drenkow J,Zaleski C,Dobin A,Prieto P,Lagarde J,Bussotti G,Tanzer A,Denas O,Li K,Bender M A,Zhang M,Byron R,Groudine M T,McCleary D,Pham L,Ye Z,Kuan S,Edsall L,Wu Y C,Rasmussen M D,Bansal M S,Kellis M,Keller C A,Morrissey C S,Mishra T,Jain D,Dogan N,Harris R S,Cayting P,Kawli T,Boyle A P,Euskirchen G,Kundaje A,Lin S,Lin Y,Jansen C,Malladi V S,Cline M S,Erickson D T,Kirkup V M,Learned K,Sloan C A,Rosenbloom K R,Lacerda de Sousa B,Beal K,Pignatelli M,Flicek P,Lian J,Kahveci T,Lee D,Kent W J,Ramalho Santos M,Herrero J,Notredame C,Johnson A,Vong S,Lee K,Bates D,Neri F,Diegel M,Canfield T,Sabo P J,Wilken M S,Reh T A,Giste E,Shafer A,Kutyavin T,Haugen E,Dunn D,Reynolds A P,Neph S,Humbert R,Hansen R S,De Bruijn M,Selleri L,Rudensky A,Josefowicz S,Samstein R,Eichler E E,Orkin S H,Levasseur D,Papayannopoulou T,Chang K H,Skoultchi A,Gosh S,Disteche C,Treuting P,Wang Y,Weiss M J,Blobel G A,Cao X,Zhong S,Wang T,Good P J,Lowdon R F,Adams L B,Zhou X Q,Pazin M J,Feingold E A,Wold B,Taylor J,Mortazavi A,Weissman S M,Stamatoyannopoulos J A,Snyder M P,Guigo R,Gingeras T R,Gilbert D M,Hardison R C,Beer M A,Ren B.A comparative encyclopedia of DNA elements in the mouse genome[J].Nature,2014,515(7527):355-364.doi:10.1038/nature13992.
[25] Tassopoulos A,Wein M,Segura A.Traumatic rupture of a giant congenital splenic cyst presenting as peritonitis[J].Radiology Case Reports,2017,12(2):401-404.doi:10.1016/j.radcr.2017.01.001.
[26] Anyanwu C T,Reynal S D.Delayed splenic rupture resulting in massive intraperitoneal hemorrhage post ambulatory-related injury[J].Cureus,2018,10(2):e2160 doi:10.7759/cureus.2160.
[27] Oh J S,Mentzer C J,Abuzeid A M,Holsten S B.Delayed splenic rupture with normal admission CT scan after blunt trauma[J].American Surgeon,2016,82(8):223-224 doi:10.1177/000313481608200820.