黄萎病是棉花生产上重要的世界性病害,自1935年传入我国以来[1],迅速蔓延扩散,危害猖獗,成为制约我国棉花发展的突出问题之一。目前,棉花黄萎病缺乏有效的防治药剂,国际上认可的最有效措施就是种植抗病棉花品种[2]。因此,利用分子育种技术挖掘抗病基因,揭示抗病机制,对抗病棉花新品种的培育具有重要意义。
棉花在与黄萎病菌等病原微生物长期共同进化过程中形成了1套自身的防御系统,包括组织结构抗性(固有组织结构抗性和诱导组织结构抗性)和生理生化抗性[3]。当受到黄萎病菌侵染后,棉花组织结构抗性被诱导,导致细胞壁木质化、薄壁组织增生,侵填体、胼胝体等结构生成,这些均能够阻碍黄萎病菌的入侵和扩展;同时,棉花组织里的棉酚、植保素、单宁等[4]和各种酶类(氧化还原酶类、水解酶类等)不断积累[5-7]、某些可溶性糖含量也发生变化[8],都为棉花提高黄萎病抗性提供了生理基础。另外,植物内源激素在棉花的抗病反应中也起着非常重要的作用。比如,王天义[9]研究发现,在抑制棉花乙烯(ET)信号路径后可显著降低接种强致病力落叶型黄萎病菌导致的棉花落叶,显著提高棉花对黄萎病的耐性。Gao等[10]发现茉莉酸(JA)和油菜素内酯(BR)对棉花抵抗黄萎病起正向调控的作用。Zhang等[11]发现水杨酸(SA)通过诱导胞间连丝中胼胝质的积累,有助于赋予海岛棉(Gossypium barbadense)抗性。
近年来,随着多组学的发展,棉花抵御黄萎病菌侵染的机理研究不断深入,为棉花抗黄萎病调控机制中关键因子的解析、候选基因的挖掘搭建了平台。Zhang等[1,12]利用接种大丽轮枝菌后的海岛棉转录组,首次清晰说明了“SA→NPR1→TGA→PR-1→抗病”信号途径中的相关基因,获得了大量的棉花抗黄萎病的防卫基因序列。Shao等[13]通过转录组分析发现了16个候选基因与棉花抗黄萎病相关,其中TGF-β在棉花抗病过程中可能起重要作用。Fang等[14]通过全基因组关联分析(Genome-wide association study,GWAS),筛选到3个与棉花抗黄萎病相关的位点,其中Gh_D06G0687编码1个NB-ARC蛋白结构域,与棉花抗黄萎病性显著相关。Wang等[15]以海岛棉根部为材料研究了病菌侵染后棉花蛋白质组的变化,发现差异蛋白主要参与防御和应激反应、初级和次级代谢、脂质转运和细胞骨架形成,并指出Bet v家族蛋白可能在黄萎病的防御反应中发挥重要作用。Fang等[16]对侵染了黄萎病菌的瑟伯氏棉(Gossypium thurberi)根部进行蛋白质组分析,在这些已鉴定的蛋白质中,主要功能涉及细胞壁形成和强化、次级代谢、激素信号传导、致病相关蛋白和抗病蛋白。与此同时,在大量组学数据基础上,越来越多的抗病相关基因被挖掘,如GbERF1-like[10]、NBS-LRR基因家族[17-19],TGF-β[13]、GbNRX1[20]、GaRPL18[21]等基因被证明在棉花抗黄萎病过程中发挥积极作用。
黄萎病属于土传病害,棉花根部作为抵御病菌侵入的第一道防线,对于阻碍黄萎病菌的传播具有重要作用。目前,研究已经证实mRNA与蛋白质变化水平不一致[22],蛋白质作为生命活动的直接执行者,更能够直观反映出基因的表达水平。因此,研究黄萎病菌入侵后棉花根部蛋白质组的变化对于挖掘抗病基因,揭示根部抵抗黄萎病菌侵入机理,培育转基因抗病品种具有重要意义。为了更全面地了解陆地棉应对黄萎病菌侵染初期的机制,本研究以大丽轮枝杆菌侵染1 h的抗病棉花品种冀2658的根系为材料,利用iTRAQ(Isobaric tag for relative and absolute quantitation,iTRAQ)技术对黄萎病菌胁迫下棉花根系的蛋白质组进行了研究,这将进一步丰富棉花防御反应的分子机制。
1 材料和方法
1.1 试验材料
试验材料为河北省农林科学院棉花研究所培育的抗黄萎病品种冀2658。冀2658在河北省区域试验中鉴定黄萎病相对病指为12.8,属于抗黄萎病品种。
黄萎病菌为大丽轮枝菌(Verticillium dahliae)强致病菌系Vd991,由中国农业科学院植物保护研究所简桂良研究员提供。
1.2 材料处理
采用培养室内纸质营养钵育苗,基质蛭石高温灭菌,所用的营养液为Hoagland营养液。在棉花长至两叶一心时,先用清水将棉花根清洗干净,而后用强致病菌系Vd991孢子悬浮液浸根接种1 h,对照用清水浸根1 h。处理完成后用吸水纸将所有棉花根处理干净,液氮冷冻后储藏在-80 ℃冰箱中备用。用于基因验证的棉花则取接种后15 min,30 min,1 h,3 h,6 h,12 h,24 h的棉花。
1.3 RNA提取和定量实时PCR
用RNAprep Pure Plant Kit(Polysaccharides & Polyphenolics-rich)(天根)试剂盒按照说明书制备总RNA。总RNA经DNase Ⅰ处理去除基因组DNA污染。第一链cDNA合成和qRT-PCR分别采用FastKing-RT试剂盒(含gDNase)(天根)和SuperReal PreMix Plus(SYBR-Green)(天根)进行。在BIO-RAD CFX96实时系统中进行qRT-PCR,以1 μL合成的cDNA为模板。利用引物Premier 5设计基因特异性引物,以棉花Actin 1作为参考基因,进行qRT-PCR试验。所用的引物序列为:
Forward primer:5′-ATTTACGGGTATTACAGCAGTCCA-3′;
Reverse primer:5′-TGAACATTTCCAATCAACTCCG-3′。
1.4 蛋白质提取、消化及标记
预冷研磨器具及其他需要接触到样品的工具,样品加入液氮研磨。加入TCA-冰丙酮(TCA-丙酮的配制:500 mL丙酮中加入50 g TCA,摇匀使其充分溶解)(-20 ℃预冷),-20 ℃沉淀2 h,后用低温离心机13 000 r/min,4 ℃,离心30 min。弃上清,取沉淀。沉淀中再加入预冷的纯丙酮,清洗沉淀(将沉淀捣碎或是悬起),-20 ℃沉淀30 min,4 ℃,离心机13 000 r/min,离心30 min,此操作可进行3~5次,洗至沉淀基本为白色。在沉淀中加入裂解液(8 mol/L尿素,30 mmol/L HEPES,1 mmol/L PMSF,2 mmol/L EDTA,10 mmol/L DTT),超声5 min助溶(超声条件:脉冲打开2 s,脉冲关闭3 s,功率180 W)。超声后,离心机13 000 r/min,离心30 min,取上清。加入固体DTT至终浓度为10 mmol/L,56 ℃水浴 1 h。取出后,迅速加入IAM至终浓度55 mmol/L,暗室静置1 h。加入4倍于样品溶液体积的预冷丙酮,-20 ℃沉淀3 h以上。离心机13 000 r/min,4 ℃,离心30 min,取沉淀。加入300 μL的复溶Buffer,终浓度为50%TEAB,0.1%SDS。超声3 min助溶,超声条件同上。离心机13 000 r/min,4 ℃,离心30 min,取上清。使用Bradford法[23]对蛋白进行定量。十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(SDS-PAGE)(12%凝胶)用于验证蛋白质的质量。每个样品取100 μg蛋白体积,用含0.1%SDS的TEAB补齐所有样品体积。加入1 μg/μL的Trypsin,每100 μg蛋白质底物加入3.3 μg的酶,37 ℃水浴24 h,然后补加Trypsin 1 μg,37 ℃水浴12 h。冻干消化液,然后使用TEAB(水∶TEAB=1∶1)每管30 μL复溶肽段。标记试剂平衡至室温,每管标记试剂中加入70 μL异丙醇,涡旋1 min,离心甩至管底。将混好的标记试剂加入到肽段中,不同样品用不同大小的同位素标记。混匀后,甩至管底,室温静置2 h。真空抽干标记后样品。
1.5 强阳离子交换(SCX)分馏和液相色谱-串联质谱法(LC-MS/MS)
SCX和LC-MS/MS在以前试验的基础上进行了轻微改动[24]。SCX改动如下:预分离从40 min开始混入B液,在40 min时A液浓度为100%;40~41 min,B液比例增加到5%;41~61 min,B液比例增加到30%;61~66 min,B液比例增加到50%并保持5 min;71~76 min,B液比例增加到100%并保持10 min,第86.01 min停止(A 液的成分:25% ACN,10 mmol/L KH2PO4,用磷酸调pH 值至 3.0。B 液的成分:25%ACN,2 mol/L KCL,10 mmol/L KH2PO4,用磷酸调pH 值至3.0)。LC-MS/MS 参数如下:使用C18分析反相柱以400 nL/min的溶剂流速分离肽(溶液A:水/0.1%甲酸;溶液B,100%乙腈/0.1%甲酸)。进样后,用5% 溶液B平衡10 min,然后启动以下梯度程序:在40 min溶液B比例达到30%;在45 min溶液B比例达到60%;在48 min溶液B比例达到80%,保持7 min;在58 min溶液B比例降到5%并保持7 min,然后逐渐回到初始溶剂条件。
1.6 蛋白质定性与定量分析
质谱扫描完毕,得到质谱信号总图。将质谱原始数据输入到 PD(Proteome Discoverer 1.3,thermo)软件后,软件先对质谱谱图进行筛选。质谱图筛选参数如下:母离子质量为350~6 000 ku,二级质谱图中最小峰数为10,信噪比 S/N 域值为1.5。PD提取后的谱图用Mascot进行搜索,搜索结束后,PD软件根据Mascot搜索结果和第一步筛选后的谱图进行定量分析。鉴定检索参数如下:Mascot版本号为2.3.0,固定修饰为Carbamidomethyl(C),可变修饰为Oxidation(M)、Gln→Pyro-Glu(N-term Q)、iTRAQ 8 plex(K)、iTRAQ 8 plex(Y)、iTRAQ 8 plex(N-term),一级质量偏差为15 ppm,二级质量偏差为20 mmu,最大允许漏切数为1,酶的类型为Trypsin,数据库为Gossypium_ncbi_3633。定量分析参数如下:蛋白定量取值类型为median,最少unique peptide数为1,归一化方法为median,蛋白定量的显著性差异(P-value)<0.05,差异倍数(Ratio)>1.2倍。
1.7 生物信息学分析
利用Gene Ontology 分析(http://www.gene ontology.org)对不同表达的蛋白质物种进行功能注释。Kyoto Encyclopedia of Genes and Genomes(KEGG)(http://www.genome.jp/kegg/或http://www.kegg.jp/)用于预测主要代谢途径和生化信号转导途径。
2 结果与分析
2.1 数据初步分析
融合了处理和对照的数据之后,共得到总谱图数(All spectra)322 079个,匹配谱图数(Matched spectra)39 713个,匹配肽段种类数(Peptide)10 427个,匹配蛋白组数(Protein group)2 414个(图1)。从肽数量(Peptide number)(图2-A)和长度(Length of peptide)(图2-B)以及蛋白质质量(Protein mass)(图2-C)的分布可以看出,大部分的蛋白质至少包含2条多肽链,并且大部分肽段集中在30个氨基酸以内。对鉴定到的蛋白质数据进行整理,通过控制差异比值1.2倍以上,P<0.05[25],以水处理作为对照,黄萎病菌处理筛选得到差异表达蛋白质(Differentially expressed proteins,DEPs)总数为46个,其中9个下调表达,37个上调表达(图3)。
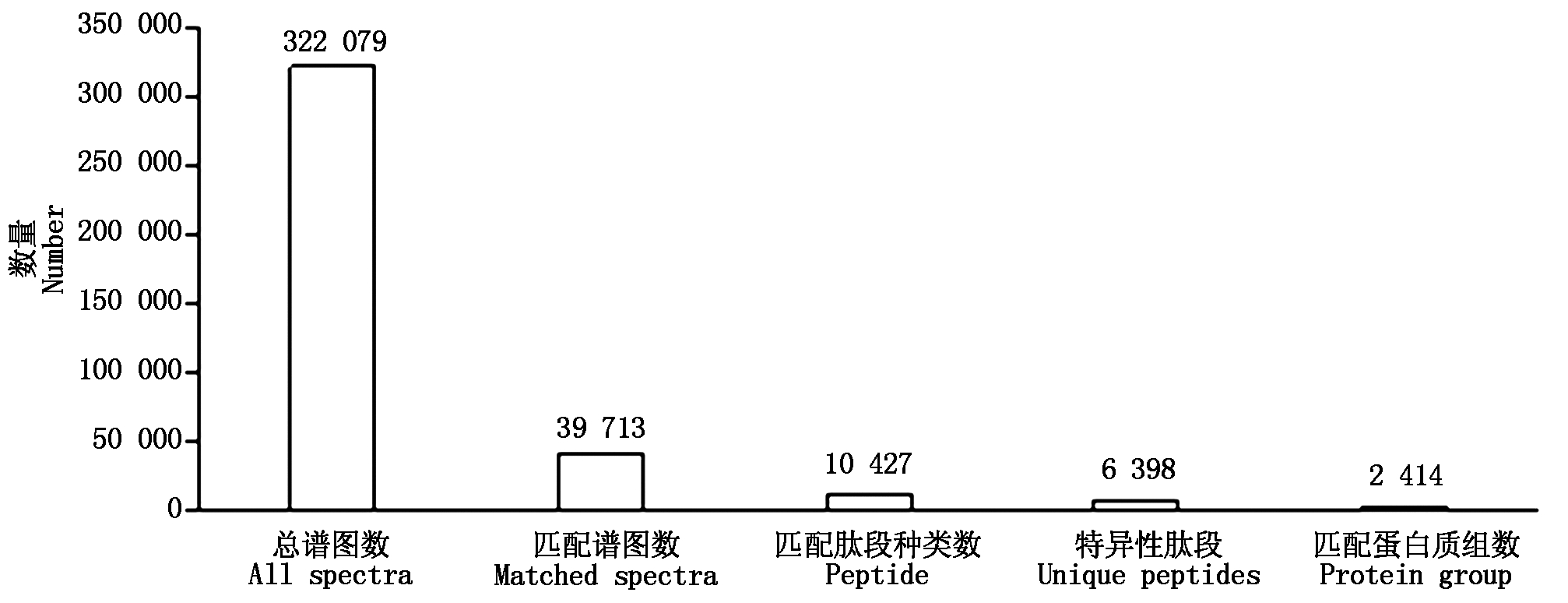
图1 iTRAQ蛋白质组学的基本信息
Fig.1 The basic information of iTRAQ proteomics
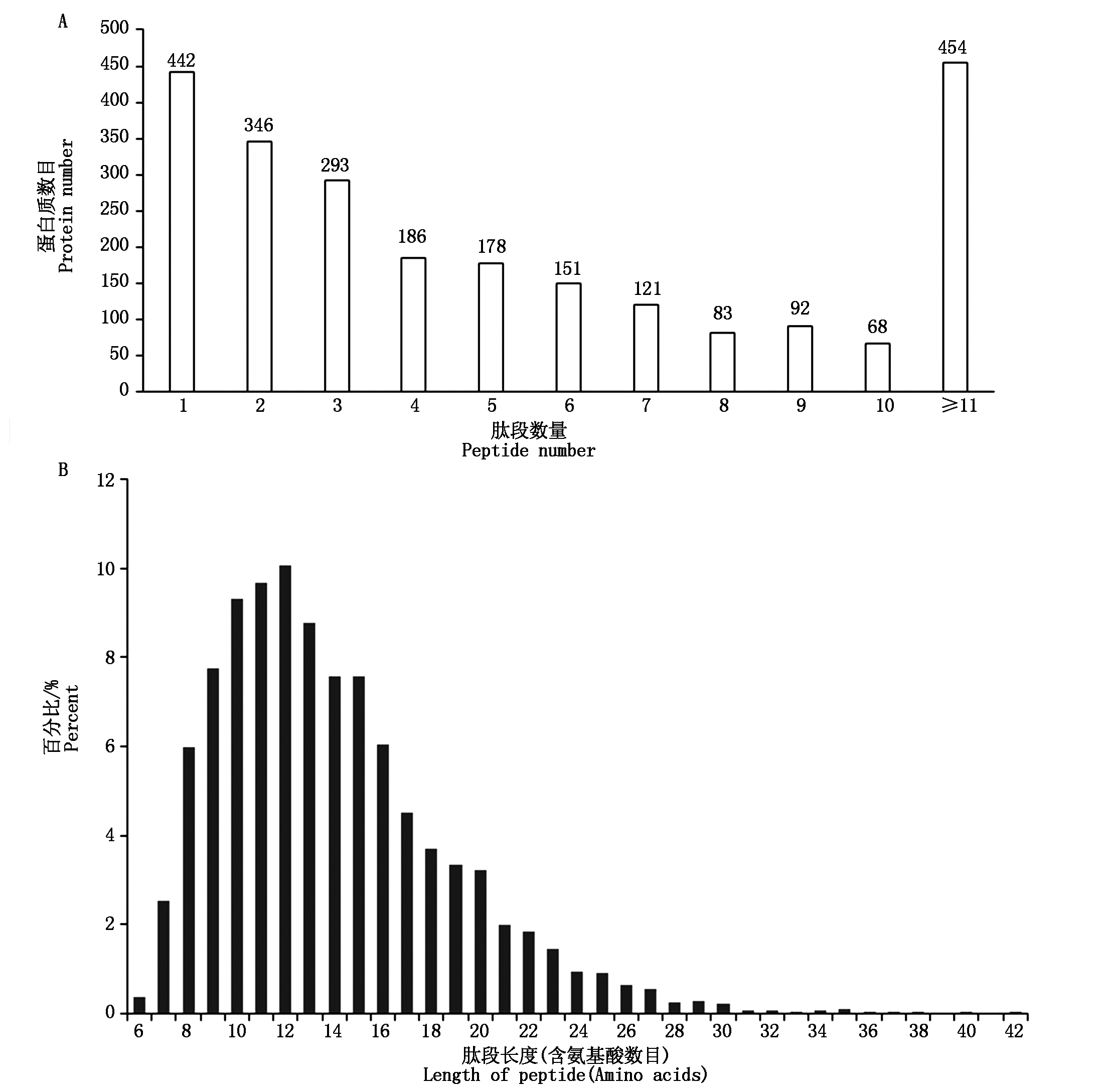
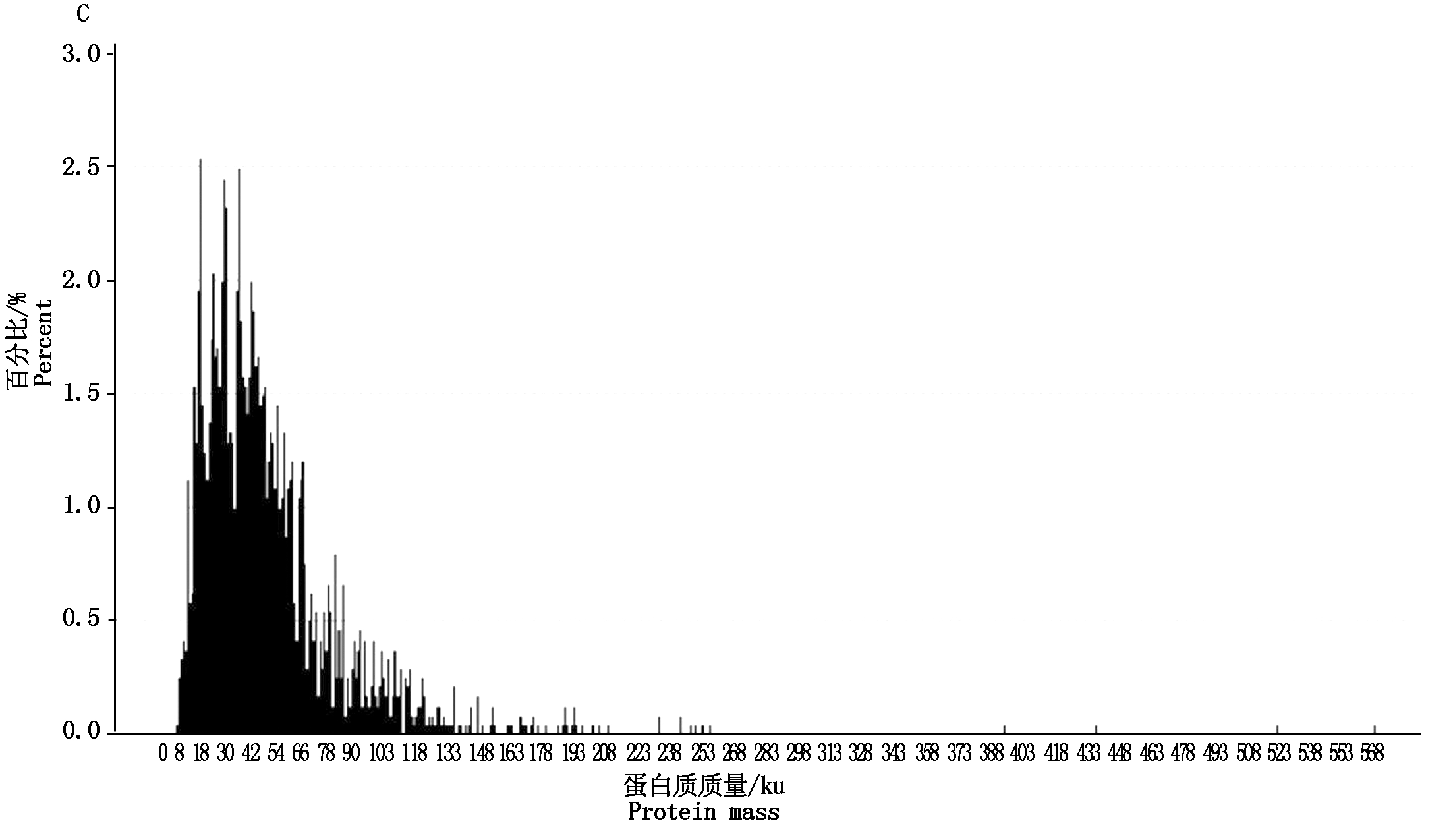
A.肽段数量分布;B.肽长分布;C.蛋白质质量分布图。
A.The distribution of length of peptides;B.The distribution of length of peptides;C.The distribution of protein mass.
图2 iTRAQ蛋白质组学鉴定的多肽长度、数量和蛋白质质量分布
Fig.2 The distribution of peptide length,peptide number and protein mass identified by iTRAQ proteomics
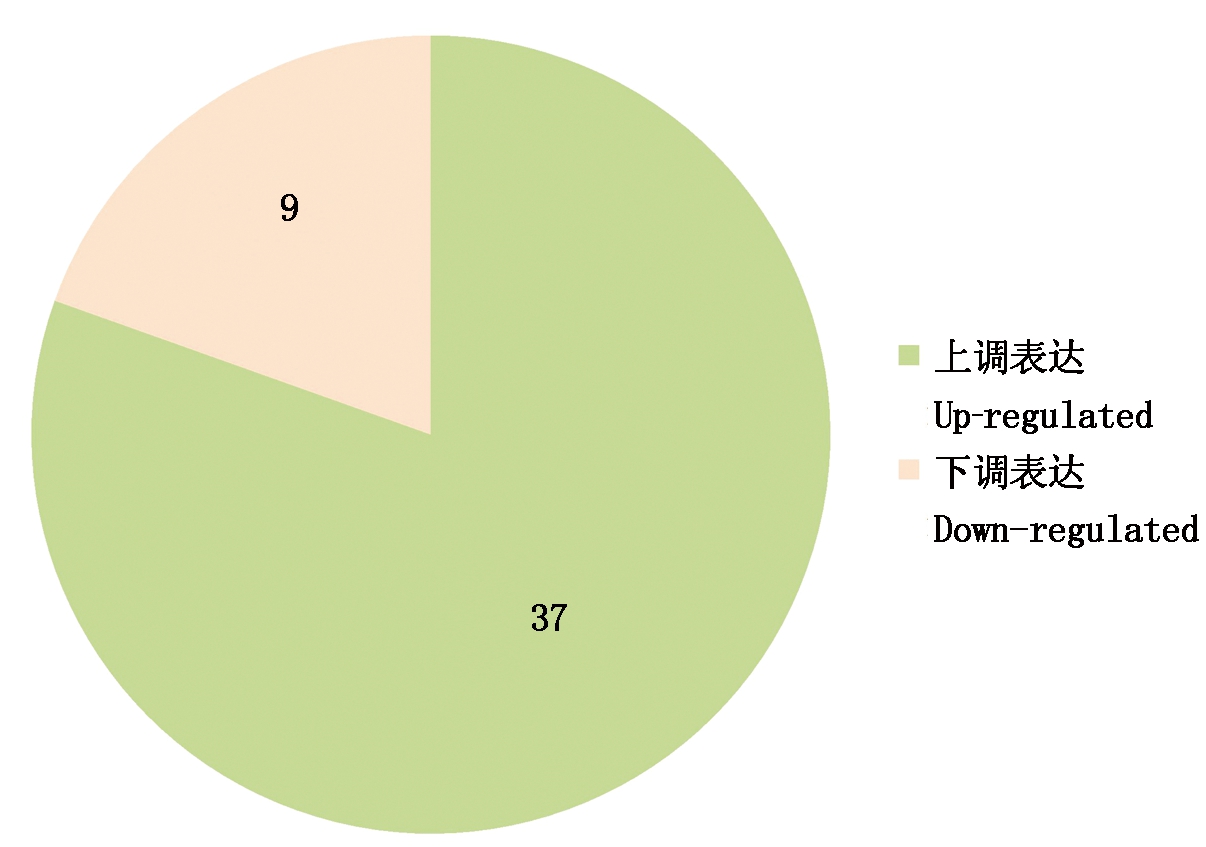
图3 黄萎病菌胁迫下冀2658中差异表达蛋白的数目
Fig.3 The number of differentially expressed proteins(DEPs)in Ji 2658 under the stress of Verticillium dahliae
2.2 差异蛋白GO注释分析
GO(Gene Ontology)数据库是全面描述基因以及基因产物属性的数据库,从细胞组成(Cellular component)、生物过程(Biological process)和分子功能(Molecular function)3个方面对差异蛋白质在功能方面进行富集。本研究对差异蛋白进行GO注释分析(图4),发现差异蛋白的功能分为24类,生物过程占8类,代谢过程(Metabolic process)和细胞过程(Cellular process)中所含差异蛋白数目最多;细胞组分占9类,大部分差异蛋白分布在细胞组分(Cell part)、细胞(Cell)和细胞器(Organelle)中;分子功能占7类,具有蛋白结合(Binding)功能和催化活性(Catalytic activity)的差异蛋白数目最多。
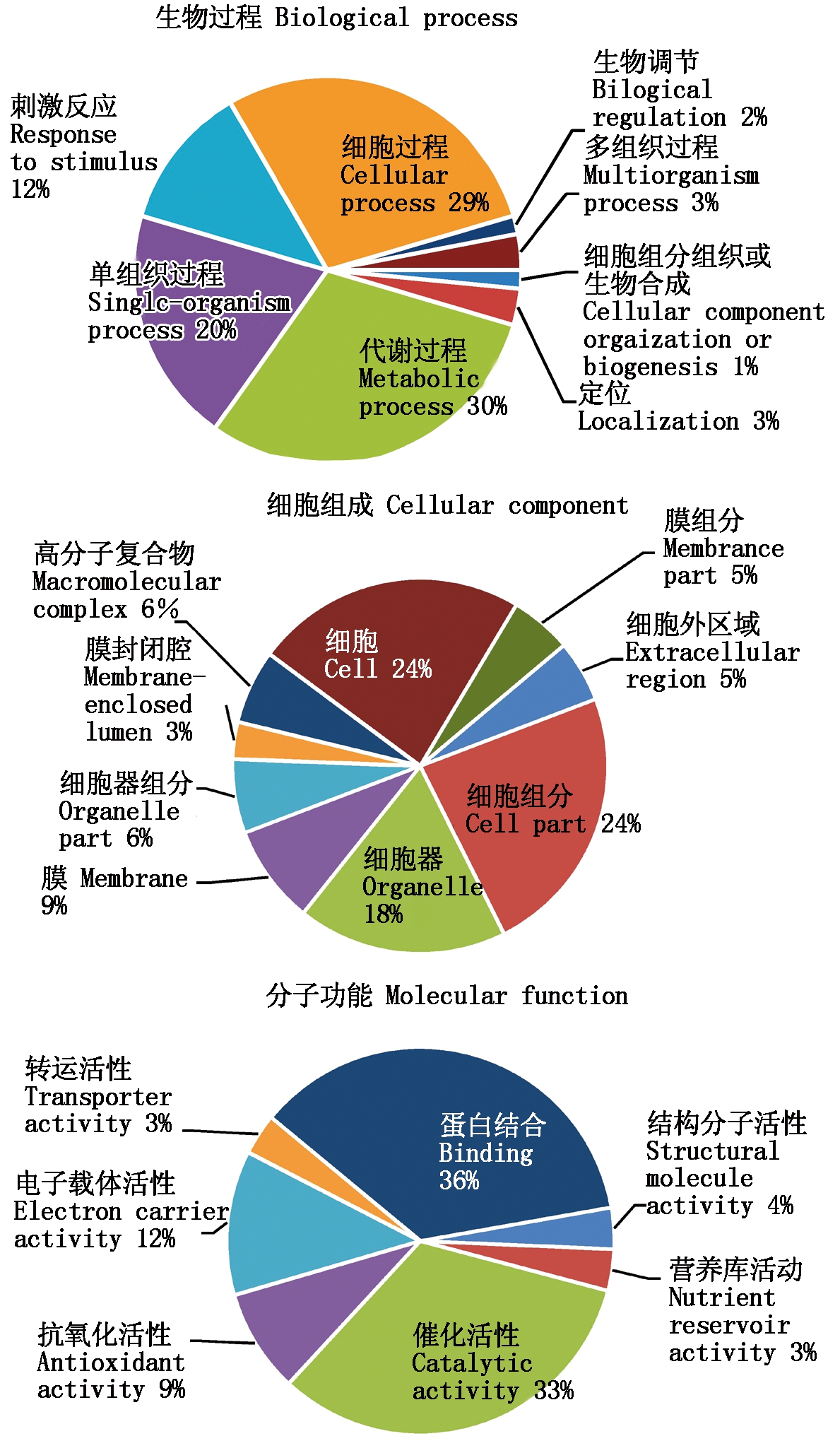
图4 黄萎病菌胁迫下差异表达蛋白GO注释分析
Fig.4 GO annotation analysis of differentially expressed proteins under Verticillium dahliae
2.3 差异蛋白KEGG(Kyoto Encyclopedia of Genes and Genomes)Pathway分析
对差异蛋白进行Pathway分析,黄萎病菌胁迫下差异蛋白参与32个代谢通路。其中苯丙氨酸代谢(Phenylalanine metabolism)P值(0.025 308 8)最小,达到显著水平;并且有4个DEPs(3个上调表达蛋白,1个下调表达蛋白)参与其中。苯丙氨酸代谢途径是植物次生代谢产物合成的重要途径,在植物的抗病反应中起重要作用,通过该途径能合成异黄酮类植物防御素和木质素等多种抗病物质。在3个上调表达的蛋白中,巨噬细胞迁移抑制因子同源物(Macrophage migration inhibitory factor homolog)差异表达倍数最高。巨噬细胞迁移抑制因子(Macrophage migration inhibitory factor,MIF)在动物中的研究比较多,是一类具有独特结构的多效免疫调节细胞因子,在固有免疫、免疫细胞招募和炎症反应等方面有了很多研究。MIF还具有促炎症反应,促进细胞定向迁移,拮抗糖皮质激素,抑制细胞凋亡,促进其他促炎因子释放等功能[26]。在植物中,有关此基因的研究较少。目前,Panstruga等[27]在拟南芥中发现3个MIF-like基因,其中一个定位在过氧化物酶体中,可能和植物病害防御相关。
3 结论与讨论
3.1 在黄萎病菌侵染初期,棉花为应对病菌侵入需合成更多能量
能量代谢维持着植物体正常的生命活动,许多研究表明,植物体在胁迫环境下,能量代谢相关蛋白会积累更多[25,28-29]。沈凡瑞[29]的研究发现,在棉花接种黄萎病菌3 d之后,能量代谢相关蛋白RuBisCO活化酶、ATP合成酶β亚基以及控制光合速率的果糖-1,6-二磷酸醛缩酶的表达量升高,减轻了病原菌的伤害对能量代谢的负面影响。本研究中,有3个能量代谢相关蛋白上调表达,它们分别是参与三羧酸循环的柠檬酸合酶和苹果酸脱氢酶,还有一个ATP合成酶δ亚基,这3个蛋白的上调表达为植物抵御黄萎病菌侵染提供更多能量。此外,王祎[30]研究发现,超量表达三羧酸循环中的关键酶-柠檬酸合酶可以提高油菜对土壤中磷铝胁迫的抗性,这为进一步研究能量代谢相关蛋白抵抗黄萎病菌侵染提供了依据。
3.2 在黄萎病菌侵染初期,蛋白质合成相关蛋白表达平衡
蛋白质的合成和降解平衡有利于维持植物体细胞正常的代谢活动,翻译机制中不同的成分在胁迫下表达差异有助于棉花适应生物逆境和非生物逆境胁迫[31]。核糖体是一种高度保守的蛋白质,对细胞活动至关重要,主要功能是合成蛋白质。本研究中,有2个核糖体蛋白-泛素40S核糖体蛋白和60S核糖体蛋白L17同工型X3下调表达,2个60S酸性核糖体蛋白P2A-like上调表达。此结果与李武[31]的研究结果类似,他发现在盐胁迫下棉花根部2个核糖体蛋白丰度降低,还有2个核糖体蛋白表达增强。另一项研究表明,核糖体蛋白L12和核糖体蛋白L19对拟南芥的非宿主抗病性具有重要作用[32]。此外,Gong等[21]将棉花中的核糖体蛋白基因GaRPL18在拟南芥中超量表达,发现转基因拟南芥提高了黄萎病抗性。分子伴侣对多肽链的正确折叠和组装有重要作用,有助于蛋白质的结构稳定和亚细胞定位,本研究中一个分子伴侣上调表达,这有利于保持胁迫环境下蛋白质正常的构象和功能。这些结果表明,陆地棉在受病菌侵染1 h时就开始为对抗病菌侵入做“原料”准备,而许菲菲[33]研究对象为盐胁迫处理4 h的棉花苗期叶片,发现此时棉花为对抗盐胁迫开始准备“原料”。
3.3 在黄萎病菌侵染初期,棉花为应对病菌在体内扩展启动细胞凋亡程序
病原菌诱导的防卫系统之一是局部抗性,主要指超敏反应(Hypersensitive reaction,HR),即在植物与病原物的非亲和互作系统中,受病原物侵染的植物细胞迅速死亡进而形成枯斑的反应[34]。其结果是病原物被限制在侵染点的坏死组织中,不能向周围扩展获得充足的营养物质,以致死亡[35]。细胞色素C(Cytochrome c,Cyt-c)是生物氧化过程中非常重要的电子传递体,在能量代谢和细胞氧化还原中发挥作用,同时也是线粒体启动凋亡程序的关键物质。当Cyt-c从植物线粒体内向胞浆释放,线粒体内电子传递链受阻,导致ATP合成减少,活性氧(Reactive oxygen species,ROS)过度生成,而过多的ROS可对细胞造成一系列损害,从而促进程序性细胞死亡(Programmed cell death,PCD)或者坏死[36]。奚经龙[35]对接种黄萎病菌的抗病品种进行差异显示分析,分离到一个与Cyt-c高度相似的蛋白,命名为Ghcytc,此基因受黄萎病菌诱导表达。在本研究中,一个Cyt-c上调表达,并且差异表达倍数最高。这说明棉花在受到黄萎病菌侵染初期就开始启动PCD阻止黄萎病菌在植物体内的传播。
3.4 在黄萎病菌侵染初期,组织结构代谢增强
黄萎病菌入侵根部,通过层层阻碍进入维管束,木质部结构在一定程度上决定着病菌是否顺利进入维管束系统[37]。与感病品种相比,抗黄萎病棉花品种木质部间隙小,单位面积薄壁细胞数量多,导管直径小,根、茎导管细胞壁厚,棉花的机械抗性强,所以能够把病原菌限制在进入维管束之前或者侧根中[38]。万琪[39]研究发现,将含GFP(Green fluorescent protein,GFP)基因的大丽轮枝菌分别接种耐黄萎病品种和感病品种,发现耐病品种体内各部分的GFP荧光强度远低于感病品种,且仅在耐病品种根中检测到GFP荧光,说明耐病品种具有一定的抗入侵和较强的抗扩展能力。本研究中,组织结构代谢相关的基因Dirigent-like(DIR)、类成束阿拉伯半乳聚糖蛋白FLA9的前体等上调表达,这对于构建致密的组织结构,阻碍黄萎病菌的扩展具有重要作用。
Dirigent-like蛋白是一类植物病原应答诱导蛋白,该家族内很多成员受病菌诱导,能参与木质素的形成,增强病菌防御能力[40]。郑娜等[41]利用VIGS(Virus induced gene silencing,VIGS)技术沉默植物病原应答诱导蛋白基因DIR,发现棉花植株对黄萎病菌的抗性明显减弱。郭宝生等[40]利用RNA-Seq技术获得12个陆地棉Dirigent-like蛋白基因家族成员,发现Dirigent-like蛋白与黄萎病菌侵入后棉花植株的抗性反应相关,其中,GhDIR4差异表达较高,在抗黄萎病反应中比较重要。在本研究中,有2个Dirigent-like 4蛋白上调表达。为了验证Dirigent-like 4在抗黄萎病中是否有重要作用,选取其中差异表达倍数较高的Dirigent-like 4进行qRT-PCR验证,在病菌处理6 h之后,基因表达量迅速升高,处理24 h时差异倍数达到23倍,这说明Dirigent-like 4对于棉花抵抗黄萎病具有重要作用。
阿拉伯半乳聚糖蛋白(AGPs)是许多植物细胞壁的重要结构蛋白,研究表明AGPs能够促进细胞膨大、根表皮细胞扩展、花粉管伸长和信号传导等[42]。类成束阿拉伯半乳聚糖蛋白(FLAs)是阿拉伯半乳聚糖蛋白中的一个亚类,已有的证据表明,FLA蛋白可能在细胞伸长、细胞黏连和次生壁成熟的过程中起作用[43]。李家宝[43]从棉花纤维发育早期cDNA文库中随机克隆了一个GhFLA cDNA,该基因在棉花的根、胚珠及快速伸长期纤维(5~15DPA)中优势表达,而在棉花的茎、叶和花中未检测到信号,超量表达GhFLA基因可以促进烟草种子提早萌发。Johnson等[44]发现,拟南芥中的FLA1可以促进侧根发育。Shi等[45]也发现fla4突变体拟南芥根系细胞壁较薄。本研究中,类成束阿拉伯半乳聚糖蛋白FLA9的前体表达量增多,基于以上研究,笔者认为该基因可能促进陆地棉根部细胞发育和细胞壁增厚,从而有利于阻碍黄萎病菌的扩展。另外,通过Pathway分析也发现,有2个上调表达的过氧化物酶(POD)参与苯丙烷类生物合成(Phenylpropanoid biosynthesis),这也可以促进木质素的积累。
3.5 在黄萎病菌侵染初期,核苷二磷酸激酶参与感病应激
核苷二磷酸激酶(NDPK)是一类高度保守的基因,在生物体内普遍存在,具有催化底物磷酸化的作用,与生物体的能量代谢、生物合成代谢息息相关。植物中发现的NDPK主要有NDPK1、NDPK2和NDPK3 3种类型[46]。本研究中,一个类核苷二磷酸激酶B上调表达。通过NCBI比对发现,该基因与多个物种的NDPK1序列高度相似。在植物体内,NDPK1在植物生长发育、非生物胁迫、感病应激、激素响应、DNA复制、淀粉纤维素合成等方面起到重要作用[47]。有研究发现,当油菜感染根癌农杆菌和黑斑菌时,NDPK1上调表达[48-49]。转OsNDPK1的拟南芥植株对丁香假单胞杆菌、灰葡萄孢真菌、番茄致病变种细菌的抗性增强,并且发现,OsNDPK1通过正调控BR信号转导来增强植物抗病防御机制[50]。基于上述研究,推测本研究中上调表达的类核苷二磷酸激酶B对棉花抵抗黄萎病具有一定作用,这为后续的研究提供了现实基础。
3.6 在黄萎病菌侵染初期,抗氧化物酶表达丰度提高
生物胁迫诱导植物根部产生大量的ROS,从而引起膜质的氧化伤害,扰乱植物的正常代谢。植物体内存在多个ROS清除系统,超氧化物歧化酶(SOD)是第一道ROS清除系统[31];POD是另一个关键途径,可以利用各种电子供体催化还原H2O2[51]。本研究中,2个SODs和2个PODs上调表达,这与其他作物在环境胁迫下的反应一致[52-53]。王亮[54]将过氧化物酶基因转入小麦中,与对照相比,转基因小麦明显提高了对干旱和盐碱的抗性。胡根海等[55]在棉花中过表达超氧化物歧化酶基因提高了植株的耐盐性。这些研究说明,转抗氧化酶可以提高植物对非生物胁迫的抗性,这对接下来研究抗氧化物酶对棉花黄萎病的影响提供了基础。另外,POD在脱氢酶的参与下,可以催化许多重要酚类物质的氧化反应,参与木质素的聚合过程[56],促进受侵组织的木质化作用。比如,邓接楼等[57]在拟南芥体内过表达红麻POD基因能显著提高拟南芥的木质素含量水平。
棉花抵抗黄萎病菌侵染的过程复杂,涉及许多代谢通路,是一系列级联反应的结果。通过本研究,初步了解了陆地棉在受黄萎病菌侵染初期(1 h)抵抗黄萎病菌的机制,进一步丰富了棉花防御反应的分子机制。
[1] 朱荷琴,李志芳,冯自力,冯鸿杰,魏锋,赵丽红,师勇强,刘世超,周京龙.我国棉花黄萎病研究十年回顾及展望[J].棉花学报,2017,29:37-50.doi:10.11963/1002-7807.zhqzhq.20170825.
Zhu H Q,Li Z F,Feng Z L,Feng H J,Wei F,Zhao L H,Shi Y Q,Liu S C,Zhou J L.Overview of cotton Verticillium wilt research over the past decade in China and its prospect in future[J].Cotton Science,2017,29:37-50.
[2] 许琦,武林琳,王咪,李晓萍,郭文治,裴蕾.抗黄萎病海岛棉叶片在大丽轮枝菌胁迫下的蛋白组学分析[J].棉花学报,2017,29(6):533-540.doi:10.11963/1002-7807.xqxq.20171012.
Xu Q,Wu L L,Wang M,Li X P,Guo W Z,Pei L.Proteomics analysis of Gossypium barbadense leaves with resistance to Verticillium dahliae under infection stress[J].Cotton Science,2017,29(6):533-540.
[3] 宋学贞,杨国正.棉花抗黄萎病育种研究进展[J].中国农学通报,2013,29(21):16-22.
Song G Z,Yang G Z.The progress on the breeding of resistance to Verticillium wilt in cotton[J].Chinese Agricultural Science Bulletin,2013,29(21):16-22.
[4] 李成伟,丁锦平,刘冬梅,周瑞阳, 李付广.棉花黄萎病及抗病育种研究进展[J].棉花学报,2008,20(5):385-390.doi:10.3969/j.issn.1002-7807.2008.05.012.
Li C W,Ding J P,Liu D M,Zhou R Y,Li F G.Research progress on cotton Verticillium wilt and resistance breeding[J].Cotton Science,2008,20(5):385-390.
[5] Plekhanova L S,Gevork′ian A G,Ashirova Z K,Ibragimov AI. Biosynthesis of RNA in the nuclear structure of wilted cotton plant leaves[J].Biokhimiia,1978,43(3):407-412.
[6] Dubery I A,Smit F.Phenylalanine ammonia-lyase from cotton(Gossypium hirsutum)hypocotyls:properties of the enzyme induced by a Verticillium dahliae phytotoxin[J].Biochim Biophys Acta-Protein Structure and Molecular Enzymology,1994,1207(1):24-30.doi:10.1016/0167-4838(94)90047-7.
[7] Zhen X H,Li Y Z.Ultrastructural changes and location of β-1,3-glucanase in resistant and susceptible cotton callus cells in response to treatment with toxin of Verticillium dahliae and salicylic acid[J].Journal of Plant Physiology,2004,161(12):1367-1377.doi:10.1016/j.jplph.2004.04.007.
[8] Davis D A,Low P S,Heinstein P.Purification of a glycoprotein elicitor phytoalexin formation from Verticillium dahliae[J].Physiological and Molecular Plant Pathology,1998,52(4):259-273.doi:10.1006/pmpp.1998.0150.
[9] 王天义.乙烯在棉花与黄萎病菌互作中的作用研究[D].武汉:华中农业大学,2018.
Wang T Y.Function analysis of ethylene in the interaction between cotton and Verticillium dahliae[D].Wuhan:Huazhong Agricultural University,2018.
[10] Gao W, Long L, Zhu L F, Xu L,Gao W H,Sun L Q,Liu L L,Zhang X L.Proteomic and virus-induced gene silencing(VIGS)analyses reveal that gossypol,brassinosteroids,and jasmonic acid contribute to the resistance of cotton to Verticillium dahliae[J].Molecular and Cellular Proteomics,2013,12(12):3690-3703.doi:10.1074/mcp.M113.031013.
[11] Zhang Y,Wang X F,Rong W,Yang J,Li Z K,Wu L Q,Zhang G Y,Ma Z Y.Histochemical analyses reveal that stronger intrinsic defenses in Gossypium barbadense than in G.hirsutum are associated with resistance to Verticillium dahliae[J].Molecular Plant Microbe Interactions,2017,30(12):984-996.doi:10.1094/MPMI-03-17-0067-R.
[12] Zhang Y,Wang X F,Ding Z G,Ma Q,Zhang G R,Zhang S L,Li Z K,Wu L Q,Zhang G Y,Ma Z Y.Transcriptome profiling of Gossypium barbadense inoculated with Verticillium dahliae provides a resource for cotton improvement[J].BMC Genomics,2013,14(1):637.doi:10.1186/1471-2164-14-637.
[13] Shao B X,Zhao Y L,Chen W,Wang H M,Guo Z J,Gong H Y,Sang X H,Cui Y L,Wang C H.Analysis of upland cotton(Gossypium hirsutum)response to Verticillium dahliae inoculation by transcriptome sequencing[J].Genetics and Molecular Research,2015,14(4):13120-13130.doi:10.4238/2015.October.26.8.
[14] Fang L,Wang Q,Hu Y,Jia Y H,Chen J D,Liu B L,Zhang Z Y,Guan X Y,Chen S Q,Zhou B L,Mei G F,Sun J L,Pan Z E,He S P,Xiao S H,Shi W J,Gong W F,Liu J G,Ma J,Cai C P,Zhu X F,Guo W Z,Du X M,Zhang T Z.Genomic analyses in cotton identify signatures of selection and loci associated with fiber quality and yield traits[J].Nature Genetics,2017,49(7):1089-1098.doi:10.1038/ng.3887.
[15] Wang F X, Ma Y P, Yang C L, Zhao P M,Yao Y,Jian G L,Luo Y M,Xia G X. Proteomic analysis of the sea-island cotton roots infected by wilt pathogen Verticillium dahliae[J].Proteomics,2011,11(22):4296-4309.doi:10.1002/pmic.201100062.
[16] Fang W F,Xie D Y,Zhu H Q,Li W,Xu Z Z,Yang L R,Li Z F,Sun L,Wang J X,Nie L H,Tang Z G,Lv S P,Zhao F A,Sun Y,Zhao Y M,Hou J N,Yang X J.Comparative proteomic analysis of Gossypium thurberi in response to Verticillium dahliae inoculation[J].International Journal of Molecular Sciences,2015,16(10):25121-25140.doi:10.3390/ijms161025121.
[17] Wang K B, Wang Z W, Li F G, Ye W W,Wang J Y,Song G L,et al.The draft genome of a diploid cotton Gossypium raimondii[J].Nature Genetics,2012,44(10):1098-1103.doi:10.1038/ng.2371.
[18] Li F G,Fan G Y,Wang K B,Sun F M,Yuan Y L,Song G L,et al.Genome sequence of the cultivated cotton Gossypium arboreum[J].Nature Genetics,2014,46(6):567-574.doi:10.1038/ng.2987.
[19] Li F G,Fan G Y,Lu C R,Xiao G H,Zou C S,Kohel R J,et al.Genome sequence of cultivated upland cotton(Gossypium hirsutum TM-1)provides insights into genome evolution[J].Nature Biotechnology,2015,33(5):524-530.doi:10.1038/nbt.3208.
[20] Li Y B,Han L B,Wang H Y,Zhang J,Sun S T,Feng D Q,Yang C L,Sun Y D,Zhong N Q,Xia G X.The thioredoxin GbNRX1 plays a crucial role in homeostasis of apoplastic reactive oxygen species in response to Verticillium dahliae infection in cotton[J].Plant Physiology,2016,170(4):2392-2406.doi:10.1104/pp.15.01930.
[21] Gong Q,Yang Z E,Wang X Q,Butt H I,Chen E R,He S P,Zhang C J,Zhang X Y,Li F G.Salicylic acid-related cotton(Gossypium arboreum)ribosomal protein GaRPL18 contributes to resistance to Verticillium dahliae[J].BMC Plant Biology,2017,17(1):59.doi:10.1186/s12870-017-1007-5.
[22] Jiang L G,Li B,Liu S X,Wang H W,Li C P,Song S H,Beatty M,Zastrow-Hayes G,Yang X H,Qin F,He Y.Characterization of proteome variation during modern maize breeding[J].Molecular & Cellular Proteomics,2019,18:263-276.doi:10.1074/mcp.ra118.001021.
[23] Kruger N J.The Bradford method for protein quantitation[J].Methods in Molecular Biology,1994,32:9-15.doi:10.1385/0-89603-126-8:25.
[24] Qin J,Gu F,Liu D,Yin C C,Zhao S J,Chen H,Zhang J N,Yang C Y,Zhan X,Zhang M C.Proteomic analysis of elite soybean Jidou17 and its parents using iTRAQ-based quantitative approaches[J].Proteome Science,2013,11(1):12.doi:10.1186/1477-5956-11-12.
[25] Gong W F,Xu F F,Sun J L,Peng Z,He S P,Pan Z E,Du X M.iTRAQ-Based comparative proteomic analysis of seedling leaves of two upland cotton genotypes differing in salt tolerance frontiers in plant science[J].Frontiers in Plant Science,2017,8:2113.doi:10.3389/fpls.2017.02113.
[26] 赵然,姚伟娟.巨噬细胞迁移抑制因子的功能及临床研究进展[J].生理科学进展,2014,45(2):93-99.
Zhao R,Yao W J.The functions and clinical studies of macrophage migration inhibitory factor[J].Progress in Physiological Sciences,2014,45(2):93-99.
[27] Panstruga R,Baumgarten K,Bernhagen J.Phylogeny and evolution of plant macrophage migration inhibitory factor/D-dopachrome tautomerase-like proteins[J].BMC Evolutionary Biology,2015,15(1):64.doi:10.1186/s12862-015-0337-x.
[28] Wang X,Shan X,Wu Y,Su S Z,Li S P,Liu H K,Han J Y,Xue C M,Yuan Y P.iTRAQ-based quantitative proteomic analysis reveals new metabolic pathways responding to chilling stress in maize seedlings[J].Journal of Proteomics,2016,146:14-24.doi:10.1016/j.jprot.2016.06.007.
[29] 沈凡瑞.黄萎病菌侵染对感病棉花品种叶片蛋白质组的影响[D].安阳:中国农业科学院,2014.
Shen F R.Proteomic analysis of susceptible cotton leaves after infection of Verticillium dahliae[D].Anyang:Chinese Academy of Agricultural Sciences Dissertation,2014.
[30] 王祎.油菜超量表达柠檬酸合成酶和植酸酶基因提高抗土壤磷铝胁迫的研究[D].武汉:华中农业大学,2012.
Wang Y.Study on overexpression of citrate synthase and phytase genes in rapeseed to improve resistance to soil phosphorus and aluminum stress[D].Wuhan:Huazhong Agricultural University,2012.
[31] 李武.盐胁迫下陆地棉根蛋白差异表达分析及耐盐相关基因的功能鉴定[D].开封:河南大学,2016.
Li W.Differential expression analysis of the proteins in upland cotton roots under salt stress and function identification of the salt-tolerance related genes[D].Kaifeng:Henan University,2016.
[32] Nagaraj S,Senthil-Kumar M,Ramu V S,Wang K,Mysore K S.Plant ribosomal proteins,RPL12 and RPL19,play a role in nonhost disease resistance against bacterial pathogens[J].Frontiers in Plant Science,2016,6:1192.doi:10.3389/fpls.2015.01192.
[33] 许菲菲.棉花苗期叶片盐胁迫差异蛋白质分析[D].安阳:中国农业科学院,2014.
Xu F F.Differentially expressed proteins analysis of seedling leaf of cotton under salt stress[D].Anyang:Chinese Academy of Agricultural Sciences,2014.
[34] Baker B,Zambryski P,Staskawicz B,Dinesh-Kumar S P.Signaling in plant-microbe interactions[J].Science,1997,276(5313):726-733.doi:10.1126/science.276.5313.726.
[35] 奚经龙.两个与棉花黄萎病抗性相关基因的克隆与特征分析[D].南京:南京农业大学,2009.
Xi J L.Isolation and characterization of two genes related with resistance to Verticillium wilt in cotton(Gossypium)[D].Nanjing:Nanjing Agriculture University,2009.
[36] Barros M H,Netto L E S,Kowaltowski A J.H2O2 generation in Saccharomyces cerevisiae respiratory pet mutants:effect of cytochrome c[J].Free Radical Biology and Medicine,2003,35(2):179-188.doi:10.1016/S0891-5849(03)00307-1.
[37] Báidez A G, Gómez P, Río J A D, ![]() A. Dysfunctionality of the xylem in Olea europaea L.plants associated with the infection process by Verticillium dahliae Kleb.role of phenolic compounds in plant defense mechanism[J].Journal of Agricultural & Food Chemistry,2007,55(9):3373-3377.doi:10.1021/jf063166d.
A. Dysfunctionality of the xylem in Olea europaea L.plants associated with the infection process by Verticillium dahliae Kleb.role of phenolic compounds in plant defense mechanism[J].Journal of Agricultural & Food Chemistry,2007,55(9):3373-3377.doi:10.1021/jf063166d.
[38] Vallad G E,Subbarao K V.Colonization of resistant and susceptible lettuce cultivars by a green fluorescent protein-tagged isolate of Verticillium dahliae[J].Phytopathology,2008,98(8):871-885.doi:10.1094/PHYTO-98-8-0871.
[39] 万琪.大丽轮枝菌对棉花的侵染及其转录组和磷酸蛋白质组学分析[D].南京:南京农业大学,2016.
Wan Q.Verticillium dahliae infection inspection and transcriptomic and phosphoproteomic analysis in cotton[D].Nanjing:Nanjing Agriculture University,2016.
[40] 郭宝生,师恭曜,王凯辉,刘素思,赵存鹏,王兆晓,耿军义,华金平.黄萎病菌侵染下陆地棉Dirigent-like蛋白基因表达差异分析[J].中国农业科学,2014,47(22):4349-4359.doi:10.3864/j.issn.0578-1752.2014.22.001.
Guo B S,Shi G Y,Wang K H,Liu S E,Zhao C P,Wang Z X,Geng J Y,Hua J P.Expression differences of dirigent-like protein genes in upland cotton responsed to infection by Verticillium dahliae[J].Scientia Agricultura Sinica,2014,47(22):4349-4359.
[41] 郑娜,郭安慧,翟伟卜,郭慧敏,张珊珊,张文蔚,简桂良,韩榕,齐放军.VIGS 技术高通量筛选棉花抗黄萎病相关基因[J].中国科学(生命科学),2016,46(5):646-656.doi:10.1360/N052016-00035.
Zheng N,Guo A H,Zhai W B,Guo H M,Zhang S S,Zhang W W,Jian G L,Han R,Qi F J.High throughput screening of Verticillium dahliae resistance genes by VIGS technology in cotton[J].Scientia SinicaVitae,2016,46(5):646-656.
[42] 王雅琴.两个棉花纤维优势表达AGP基因的克隆及功能初步分析[D].石河子:石河子大学,2013.
Wang Y Q.Cloning and expression analysis of two cotton fiber advantage express gene AGP[D].Shihezi:Shihezi University,2013.
[43] 李家宝.棉花类成束蛋白阿拉伯半乳聚糖蛋白基因(GhFLA)的克隆与功能分析[D].重庆:西南大学,2007.
Li J B.Cloning and function analysis of a fasciclin-like arabinogalactan protein gene(GhFLA)from cotton(Gossypium hirsutum L.)[D].Chongqing:Xinan University,2007.
[44] Johnson K L,Kibble N A J,Bacic A,Schultz C J.A Fasciclin-Like Arabinogalactan-Protein(FLA)mutant of Arabidopsis thaliana,fla1,shows defects in shoot regeneration[J].PLoS One,2011,6(9):e25154.doi:10.1371/journal.pone.0025154.
[45] Shi H,Kim Y,Guo Y,Stevenson B,Zhu J K.The Arabidopsis SOS5 locus encodes a putative cell surface adhesion protein and is required for normal cell expansion[J].The Plant Cell Online,2003,15(1):19-32.doi:10.1105/tpc.007872.
[46] 王文斌,王金胜,邓西平,刘建东.植物核苷二磷酸激酶(NDPKs)研究进展[J].农学学报,2011,1(4):1-5.doi:10.3969/j.issn.1007-7774.2011.04.001.
Wang W B,Wang J S,Deng X P,Liu J D.Research advances on nucleoside diphosphate kinases(NDPKs)of plants[J].Journal of Agriculture,2011,1(4):1-5.
[47] 朱馨妮,汪珊珊,周佳琴,朱世华.植物核苷二磷酸激酶研究进展[J].生物技术通报,2017,33(11):19-28.doi:10.13560/j.cnki.biotech.bull.1985.2017-0261.
Zhu X N,Wang S S,Zhou J Q,Zhu S H.Research advances of NDPKs in plants[J].Biotechnology Bulletin,2017,33(11):19-28.
[48] Cao T,Srivastava S,Rahman M H,Kav N N V,Hotte N,Deyholos M K,Strelkov E.Proteome-level changes in the roots of Brassica napus as a result of Plasmodiophora brassicae infection[J].Plant Science,2008,174(1):97-115.doi:10.1016/j.plantsci.2007.10.002.
[49] Sharma N,Rahman M H,Strelkov S,Thiagarajah M,Bansal V K,Kav N N V.Proteome-level changes in two Brassica napus lines exhibiting differential responses to the fungal pathogen Alternaria brassicae[J].Plant Science(Oxford),2007,172(1):95-110.doi:10.1016/j.plantsci.2006.07.016.
[50] 李生辉.OsNDPK1提高植物抗病性及促进细胞分裂的机理研究[D].保定:河北农业大学,2012.
Li S H.Mechanisms of OsNDPK1 improving disease-resistance and promoting cell division of plant[D].Baoding:Agricultural University of Hebei Province,2012.
[51] Boerjan W,Ralph J,Baucher M.Lignin biosynthesis[J].Annual Review of Plant Biology,2003,54(1):519-546.doi:10.1146/annurev.arplant.54.031902.134938.
[52] Du C X,Fan H F,Guo S R,Tezuka T,Li J.Proteomic analysis of cucumber seedling roots subjected to salt stress[J].Phytochemistry,2010,71(13):1450-1459.doi:10.1016/j.phytochem.2010.05.020.
[53] Jiang Y,Yang B,Harris N S,Deyholos M K.Comparative proteomic analysis of NaCl stress-responsive proteins in Arabidopsis roots[J].Journal of Experimental Botany,2007,58(13):3591-3607.doi:10.1093/jxb/erm207.
[54] 王亮.小麦过氧化物酶基因TaPrx的遗传转化及耐旱耐盐功能鉴定[D].泰安:山东农业大学,2015.
Wang L.Genetic transformation of TaPrx gene in wheat and identification of its drought and salt tolerance[D].Taian:Shandong Agricultural University,2015.
[55] 胡根海,张晓红,付远志,董娜,王清连.棉花过表达超氧化物歧化酶基因对植株耐盐性的影响[J].华北农学报,2017,32(6):54-59.doi:10.7668/hbnxb.2017.06.008.
Hu G H,Zhang X H,Fu Y Z,Dong N,Wang Q L.Effects of overexpression of cotton superoxide dismutase genes on salt tolerant capability in upland cotton[J].Acta Agriculture Boreali-Sinica,2017,32(6):54-59.
[56] Baucher M,Halpin C,Petit-Conil M,Boerjan W.Lignin:genetic engineering and impact on pulping[J].Critical Reviews in Biochemistry and Molecular Biology,2003,38(4):305-350.doi:10.1080/10409230391036757.
[57] 邓接楼,张高阳,黄思齐,李德芳.红麻过氧化物酶基因分离、过表达载体构建及转化拟南芥分析[J].基因组学与应用生物学,2017,36(4):1570-1574.doi:10.13417/j.gab.036.001570.
Deng J L,Zhang G Y,Huang S Q,Li D F.The Analysis of peroxidase gene isolation,over-expression vector construction in Kenaf and its transformation in Arabidopsis thaliana[J].Genomics and Applied Biology,2017,36(4):1570-1574.