小麦(Triticum aestivum L.)是世界重要的粮食作物,为人类提供约20%的食物热量和蛋白质[1]。黄淮麦区作为我国小麦第一大主产区,占小麦总种植面积和总产量的60%~70%[2]。穗粒数作为小麦产量构成三要素之一,是黄淮麦区小麦高产育种的驱动力量[3]。穗部作为小麦穗粒数形成的器官,合理的穗部结构对提高小麦产量具有重要作用[4]。近年来,穗部性状成为小麦遗传改良研究热点之一[5]。
关联分析又称连锁不平衡作图(Linkage disequilibrium mapping)或关联作图(Association mapping),被广泛用于农作物复杂性状的遗传研究[6]。相较于QTL分析,全基因组关联分析(Genome-wide association studies,GWAS)可更好的解析复杂性状的遗传结构[7],挖掘重要农艺性状关键遗传位点及相关位点的优异等位基因。武玉国等[8]利用黄淮麦区175个小麦品种进行关联分析,检测到wmc128-1B和wmc236-3B与小穗数极显著关联,分别解释10.5%,8.0%的表型变异。Zhang等[9]利用中国小麦微核心种质(Mini core collection,MCC)群体,通过全基因组关联分析发现4个穗粒数关联位点,其中Xgwm131-3B与Xcfe273-6A位点与穗粒数极显著相关,Xgwm131110与Xcfe273306分别为两者的优良等位变异。近年来,随着9K[10]、90K[11]、820K[12]、660K[13]和55K[14]SNP芯片的开发,利用GWAS发掘小麦重要农艺性状基因已成为研究复杂农艺性状的重要手段[15]。Mora等[16]基于SNP标记对382份小麦栽培品种进行关联分析,检测到15个与穗粒数显著关联的位点。Sun等[17]利用90K SNP芯片对黄淮麦区163个小麦品种的13个产量相关性状进行全基因组关联分析,在过半环境中检测到多个与穗长、穗粒数显著关联的SNP位点,分布于1BL、2B、3B、6B、6DL染色体。Muqaddasi等[18]对518份欧洲小麦品种的穗部性状进行GWAS分析,发现了一个位于7A染色体上的与穗粒数显著关联的位点,并确定了该位点的候选基因及遗传效应。
本研究利用Affymetrix Wheat 55K SNP芯片扫描132份黄淮麦区北片小麦主栽品种及其衍生品系,结合2 a 8个环境的穗部性状表型鉴定结果开展标记-性状关联分析,探索控制小麦穗部性状的显著关联位点,挖掘其优异等位基因,解析其聚合效应,为黄淮麦区小麦遗传改良提供参考。
1 材料和方法
1.1 试验材料
供试小麦材料共132份,包括河北省63个、河南省26个、山东省15个、山西省14个、陕西省7个和北京市7个,为黄淮麦区北片近30年培育的小麦主栽品种及其衍生品系组成的自然群体(表1)。
表1 供试132个小麦品种/系的编号及其属地来源
Tab.1 The names and district origins of the 132 wheat accessions investigated in this study
编号Code品种/系Varieties/lines来源Origins编号Code品种/系Varieties/lines来源Origins编号Code品种/系Varieties/lines来源Origins1冀麦38河北45科农2011河北89长6878山西2石4185河北46科农8024河北90运旱22-33山西3石家庄8号河北47科农8162河北91运旱618山西4石麦15河北48科农1002河北92晋太170山西5石麦19河北49邯麦4号河北93郯麦98山西6石优20河北50邯麦7号河北94太10604山西7石麦22河北51邯麦13号河北95晋麦47 山西8石麦18河北52邯6172河北96晋麦79山西
表1(续)

编号Code品种/系Varieties/lines来源Origins编号Code品种/系Varieties/lines来源Origins编号Code品种/系Varieties/lines来源Origins9石优4366河北53邯7086河北97临旱6号山西10石麦26河北54邯4564河北98临远8号山西11石4195河北55邯6012河北99临优7287山西12石CG15-009河北56邢麦6号河北100舜麦1718陕西13石5139河北57沧麦028河北101尧麦16陕西14石麦28河北58沧麦6005河北102小偃22陕西15石6650河北59师栾02-1河北103西农928陕西16冀5265河北60金禾912河北104西农556陕西17冀麦585河北61河农825河北105西农538陕西18冀麦325河北62河农6049河北106西农558陕西19冀麦418河北63保麦10号河北107周麦16河南20冀麦867河北64京冬18北京108周麦18河南21冀麦120河北65京花9号北京109周麦22河南22冀麦738河北66中麦175北京110周麦24河南23冀麦518河北67中麦415北京111周麦26河南24冀麦181河北68轮选987北京112周麦27河南25冀麦161河北69轮选518北京113洛旱2号河南26冀麦631河北70轮选988北京114洛旱6号河南27冀麦403河北71烟农836山东115洛旱7号河南28衡观35河北72烟农0428山东116洛旱9号河南29衡4399河北73烟农21山东117洛旱11河南30衡136河北74汶农14号山东118洛旱13河南31衡5299河北75鲁原502山东119洛麦21河南32衡0628河北76济麦19山东120丰德存麦1号河南33衡6632河北77济麦20山东121矮抗58河南34衡5366河北78济麦21山东122豫麦66号河南35衡5835河北79济麦22山东123兰考矮早8河南36衡1589河北80泰山21山东124郑麦9023河南37衡9966河北81泰山22山东125郑麦004河南38衡科6021河北82泰山5366山东126郑麦366河南39衡11-6021河北83良星99山东127郑麦7698河南40衡S13-5005河北84良星66山东128新麦11号河南41衡S13-5022河北85鲁星麦9号山东129新麦18号河南42科农9204河北86长6359山西130新麦19号河南43科农199河北87长4738山西131新麦20号河南44科农2009河北88长4640山西132新麦26号河南
1.2 表型鉴定
供试材料在2016-2017、2017-2018分别种植于中国科学院农业资源研究中心(栾城聂家庄)、河北省粮油作物研究所(藁城堤上)、石家庄市农林科学研究院(辛集马兰)、河北省旱作农业研究所(衡水护驾迟)所属农业试验站。采用常规田间管理,每个品种播2行,行距25 cm,株距10 cm,行长2~3 m,随机排列,重复2次。每个材料取典型植株5株,测定主茎穗穗长(Spike length,SL,cm)、总小穗数(Total spikelet number per spike,TSNS)、穗密度(Spike compactness,SCN)和穗粒数(Kernel number per spike,KNS)等穗部性状。对表型值进行最优线性无偏估计(The best linear unbiased prediction,BLUP),运行基于Merk等[19]描述的线性模式的R程序脚本(https://articles.extension.org/pages/61006/),获得各个性状的BLUP值。
1.3 性状-标记的关联分析
按照Zhang等[20]的方法,使用Affymetrix Wheat 55K SNP芯片对自然群体进行基因分型,去除MAF<5%和缺失数据>30%的标记,过滤后得到7 948个SNP标记用于分析供试材料的群体结构,结合表型数据、Q矩阵、亲缘关系Kinship值,使用 TASSEL 5.0软件中的 MLM(Mixed linear model)模型进行性状和标记之间的关联分析。当SNP标记P ≤ 10-4时认为标记与性状存在显著关联。
2 结果与分析
2.1 表型统计结果分析
穗部相关性状统计分析结果(表2)表明,8个环境中各个性状表现出较大差异。其中穗长、穗密度变异系数较小,总小穗数变异系数较大,推测是温度、降水等环境因子的年度间变化对小麦生长发育造成的影响。
表2 供试群体穗部相关性状的描述性统计
Tab.2 Descriptive statistics for spike-related traits of the mapping panel
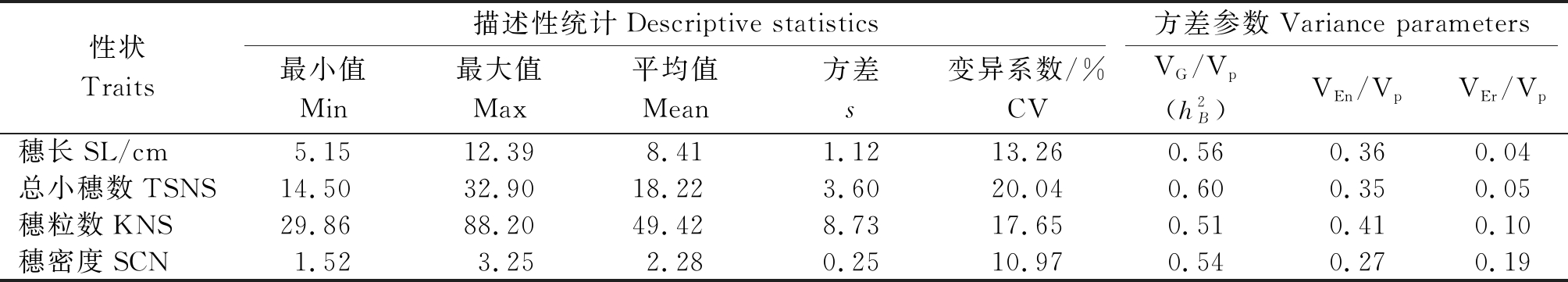
性状Traits描述性统计Descriptive statistics方差参数Variance parameters最小值Min最大值Max平均值Mean方差s变异系数/%CVVG/Vp(h2B)VEn/VpVEr/Vp穗长SL/cm5.1512.398.411.1213.260.560.360.04总小穗数TSNS14.5032.9018.223.6020.040.600.350.05穗粒数KNS29.8688.2049.428.7317.650.510.410.10穗密度SCN1.523.252.280.2510.970.540.270.19
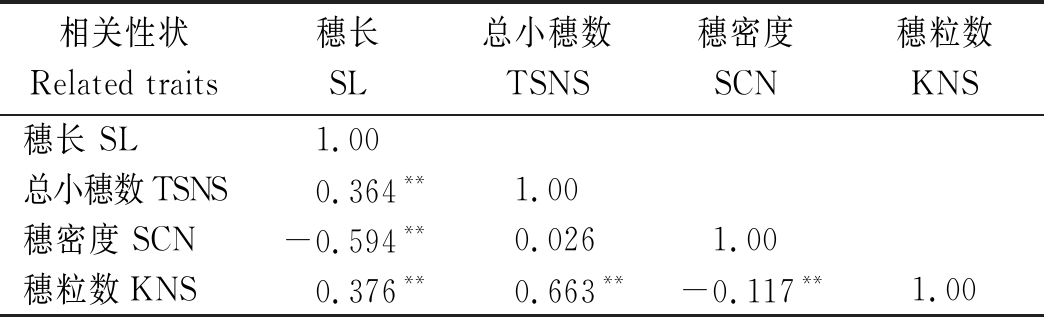
相关性分析结果表明(表3),除总小穗数与穗密度相关性不显著外,其他性状之间都密切相关,暗示控制穗部性状的基因可能存在一因多效的现象,小麦穗部性状遗传调控网络高度复杂。
表 3 各穗部性状之间的相关性分析
Tab.3 The phenotypic correlations among the spike-related traits investigated
注:**.在0.01水平上显著相关。
Note:**.Significant correlation at the 0.01 level.
相关性状Related traits穗长SL总小穗数TSNS穗密度SCN穗粒数KNS穗长 SL1.00总小穗数TSNS0.364**1.00穗密度 SCN-0.594**0.0261.00穗粒数KNS0.376**0.663**-0.117**1.00
2.2 全基因组关联分析
2 a 8个环境下共检测到121个穗部性状-标记关联位点(Marker-trait association,MTA)。其中,与穗长显著关联的位点38个,与总小穗数显著关联的位点15个,与穗密度显著相关的位点47个,与穗粒数显著关联的位点21个,8个显著关联位点在3个及3个以上环境中被重复检测到(表4)。其中,与穗长显著关联的3个位点分别位于1D、2A和6D染色体上,分别可解释9.00%~15.70%,10.50%~15.80%,9.40%~11.70%的表型变异。与总小穗数显著关联的2个位点位于2A染色体上,可分别解释11.10%~12.80%,10.40%~14.80%的表型变异。与穗密度显著关联的2个位点位于1D染色体上相近位置,可解释11.00%~24.53%的表型变异。与穗粒数显著关联的位点AX-110378404位于5A染色体580 Mb位置,可解释9.70%~14.10%的表型变异。
表 4 可重复检测到的穗部性状显著关联位点
Tab.4 The repetitively detected significant SNP loci associated with spike-related traits

注:E1、E5.低氮环境;E2、E4、E7和E8.高氮环境;BLUP.E1~E8的最佳线性无偏预测。
Note:E1,E5.Low nitrogen environments;E2,E4,E7,E8.High nitrogen environments; BLUP.The best linear unbiased prediction of E1 to E8.
性状Traits标记Markers染色体Chromosome物理位置Physical positionE1E2E4PPVE/%PPVE/%PPVE/%SLAX-1087305441D195274311.30E-0515.70%SLAX-947555702A152367379.00E-0513.203.70E-0411.10SLAX-1117038516D38497968.50E-0410.20TSNSAX-1112355322A1254534878.00E-0410.502.60E-0412.105.20E-0411.10TSNSAX-1109261422A7541637033.20E-0514.802.80E-0412.00SCNAX-1087305441D195274319.62E-0411.12SCNAX-1109632991D193685285.31E-0412.21KNSAX-1103784045A5756028674.10E-0411.201.30E-049.70性状TraitsE5E7E8BLUPPPVE/%PPVE/%PPVE/%PPVE/%SL1.00E-049.004.80E-0412.60SL2.00E-0412.106.70E-0410.508.00E-0515.80SL1.40E-049.402.60E-0411.70TSNS4.90E-0411.207.40E-0412.80TSNS8.80E-0410.40SCN9.71E-0411.006.21E-0411.77SCN5.61E-0724.532.30E-0413.57KNS1.40E-0414.10
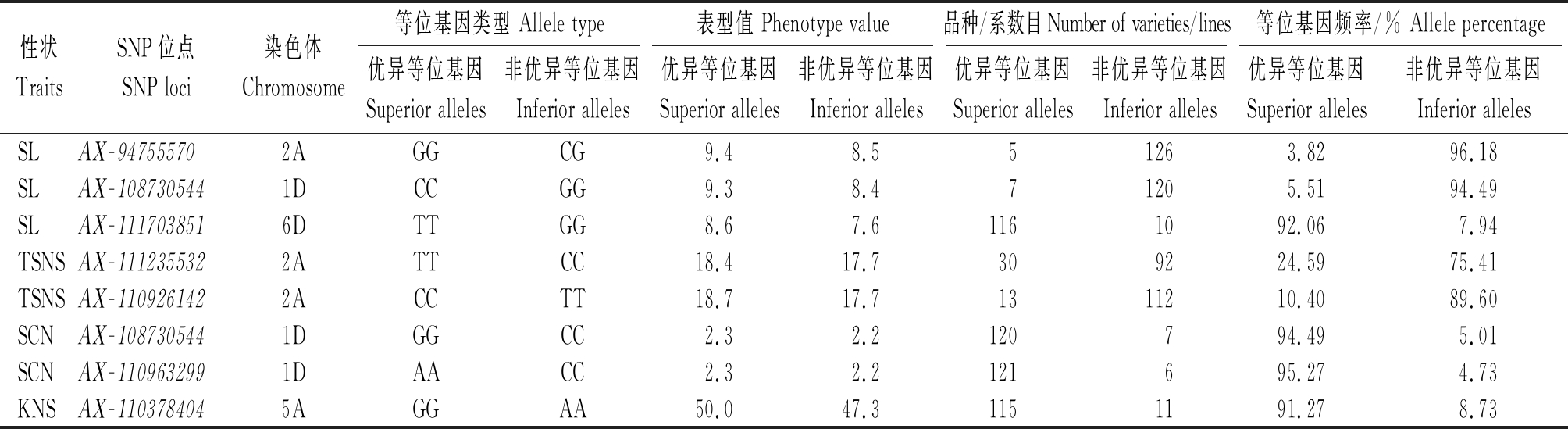
2.3 穗部性状显著关联位点优异等位基因的频率
对这些可重复检测到的显著关联位点的优异等位基因在本区域小麦中的频率进行分析(表5),发现穗长显著关联位点AX-108730544-1D的优良等位变异为CC,平均穗长9.3 cm,优异等位基因存在于衡S13-5005、济麦21、泰山5366、临旱6号、临远8号和西农928等6个品种中,在供试材料中的频率为5.51%。AX-94755570-2A位点的优良等位变异为GG,平均穗长9.4 cm,优异等位基因仅存在于晋麦47、晋麦79、临旱6号、周麦24、丰德存麦1号等5个品种中,频率仅为3.82%。AX-111703851-6D位点的优良等位变异为TT,平均穗长8.6 cm,优异等位基因频率为92.06%。仅临旱6号在3个位点都具有优异等位基因,7.58%的品种/系2个位点具有优异等位基因,75.76%的品种/系仅1个位点具有优异等位基因,15.91%的品种/系3个位点均不具备优异等位基因。
与总小穗数显著关联的AX-111235532-2A位点的优良等位变异为TT,平均总小穗数18.4个,24.59%的供试品种/系含有此优异等位基因。AX-110926142-2A位点的优良等位变异为CC,平均总小穗数18.7个,石10-4393、石CG15-009、石11-5139、石麦28、河农825、轮选518、小偃22、周麦18、洛旱13、洛麦21、豫麦66号、兰考矮早8和郑麦366等13个品种具有此优良等位变异,该位点的优异等位基因在本区域小麦中的频率为10.40%。
穗密度显著关联位点AX-108730544-1D的优良等位变异为GG,平均穗密度为2.26个/cm,94.49%的供试材料含有此优异等位基因。AX-110963299-1D位点的优良等位变异为AA,平均穗密度为2.25个/cm,95.27%的品种含有此优异等位基因。
穗粒数显著关联位点AX-110378404-5A的优良等位变异为GG,平均穗粒数50.0个,优异等位基因频率为91.27%,大多数品种都含有此优良等位变异。
表5 显著关联SNP标记位点优异等位基因在自然群体中的频率
Tab.5 Frequency of the superior alleles at the significant association SNP loci in the mapping panel
性状TraitsSNP位点SNP loci染色体Chromosome等位基因类型 Allele type表型值 Phenotype value品种/系数目Number of varieties/lines等位基因频率/% Allele percentage优异等位基因Superior alleles非优异等位基因Inferior alleles优异等位基因Superior alleles非优异等位基因Inferior alleles优异等位基因Superior alleles非优异等位基因 Inferior alleles优异等位基因Superior alleles非优异等位基因Inferior allelesSLAX-947555702AGGCG9.48.551263.8296.18SLAX-1087305441DCCGG9.38.471205.5194.49SLAX-1117038516DTTGG8.67.61161092.067.94TSNSAX-1112355322ATTCC18.417.7309224.5975.41TSNSAX-1109261422ACCTT18.717.71311210.4089.60SCNAX-1087305441DGGCC2.32.2120794.495.01SCNAX-1109632991DAACC2.32.2121695.274.73KNSAX-1103784045AGGAA50.047.31151191.278.73
2.4 穗部性状显著关联位点优异等位基因的地域分布
对可重复检测到的显著关联位点的优异等位基因的地域分布进行分析,结果(图1)显示,3个穗长显著关联位点在山西、陕西、山东等地品种中优异等位基因频率较高,总小穗数和穗密度显著关联位点在河南、河北两省品种中优异等位基因频率较高,表明这2个性状紧密相关。山西、陕西两地品种中穗粒数位点优异等位基因频率较高,可能与两地品种穗长较长有关。

A、B、C和D.穗长、总小穗数、穗密度和穗粒数的优异/非优异等位基因在黄淮麦区的分布及其频率;深色.优异等位基因;浅色.非优异等位基因。
A,B,C and D.The distribution of the superior/inferior alleles of SL,TSNS,SCN and KNS in the Huanghuai River Valley Wheat Region,respectively;Deep/light colour.Frequency of the superior/inferioralleles of corresponding traits,respectively.
图1 穗部性状显著关联SNP位点优异等位基因在不同省市品种/系中的分布
Fig.1 Geographic distribution of the superior alleles at the significant association SNP loci of spike-related traits in the tested varieties/lines from different provinces
2.5 穗部性状显著关联位点优异等位基因聚合效应
针对多环境下重复检测到的显著关联位点,通过线性回归估计优异等位基因的聚合效应,发现3个穗长显著关联位点(AX-108730544-1D、AX-94755570-2A和AX-111703851-6D)的优异等位基因聚合时穗长增加,两者存在显著的正相关关系,相关系数为0.908(图2)。小穗数和穗密度表型值随着优异等位基因聚合数目的增加也呈现出提高的趋势(表6)。高低氮2个环境中,穗部性状显著关联位点优异等位基因的聚合效应表现相似,较多优异等位基因聚合时相应性状的表型值也达到极端值。
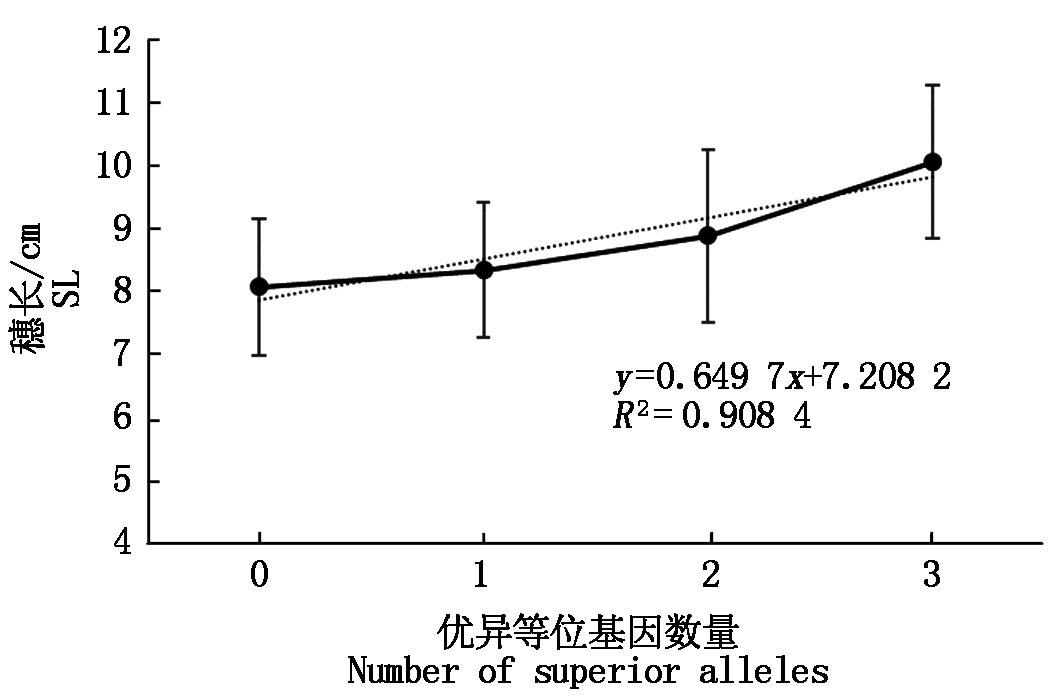
图2 穗长显著关联位点优异等位基因的聚合效应
Fig.2 The pyramiding effect of superior alleles at the significant association SNP loci
表6 与总小穗数和穗密度显著关联位点优异等位基因的聚合效应
Tab.6 The pyramiding effect of the superior alleles related to TSNS and SCN
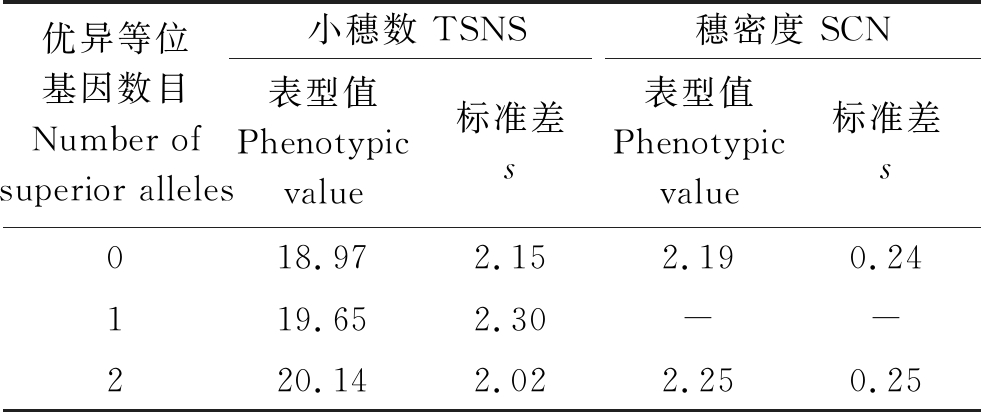
注:-.无相应数据。
Note:-.No matching value.
优异等位基因数目Number of superior alleles小穗数 TSNS穗密度 SCN表型值Phenotypic value标准差s表型值Phenotypic value标准差s018.972.152.190.24119.652.30--220.142.022.250.25
2.6 穗部性状显著关联位点优异等位基因在科农系列小麦中的传递
基于至少4个环境中重复检测到的穗部性状显著关联位点优异等位基因频率,统计科农系列小麦穗长、总小穗数和穗粒数关联位点的优异等位基因频率(表7),科农9204和科农199在穗长关联位点的优异等位基因频率为33.3%,低于其亲本冀麦38;总小穗数和穗粒数关联位点的优异位基因频率分别为50.0%和100%,这些优异等位基因均传递给后代。与黄淮麦区北片国家区域试验对照品种济麦22和良星99相比,科农8024和科农1002 2个新品系穗长关联位点优异等位基因频率显著提高。
表7 穗部性状显著关联位点优异等位基因在科农系列小麦的分布频率
Tab.7 The frequency of the superior alleles at the significant association SNP loci in wheat Kenong 9204 and its derivatives
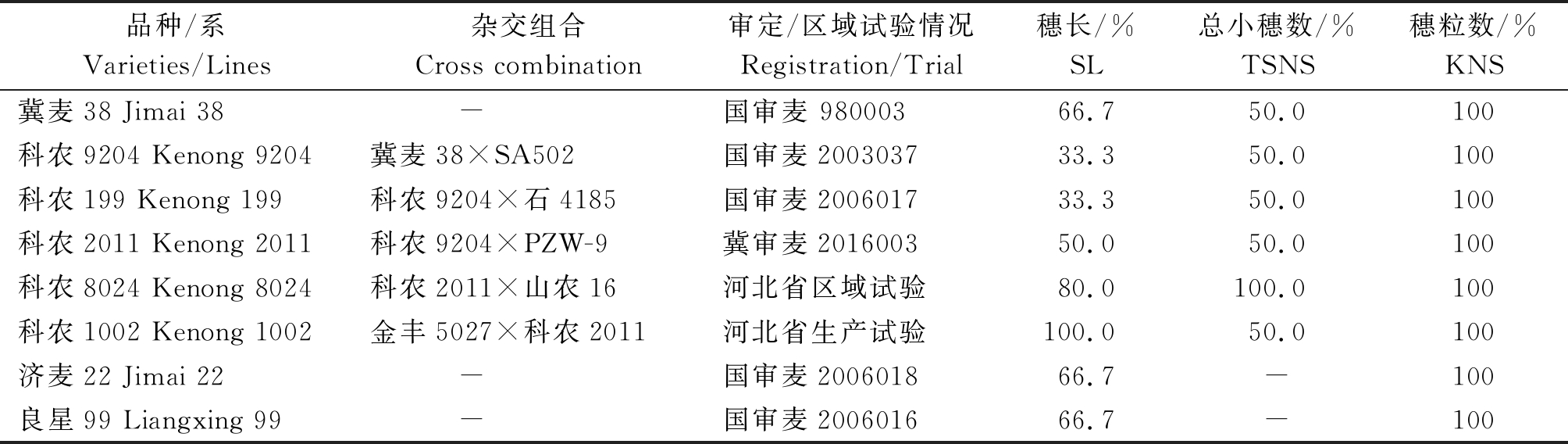
注:冀麦38.科农9204的亲本;济麦22、良星99.黄淮麦区北片冬小麦区域试验的对照品种;-.未检测到优异等位基因。
Note:Jimai 38.The maternal parent of Kenong 9204;Jimai 22,Liangxing 99.Two control varieties in the winter wheat performance trial of the North Huanghuai Regional Nursery of China;-.None superior allele were detected.
品种/系Varieties/Lines杂交组合Cross combination审定/区域试验情况Registration/Trial 穗长/%SL总小穗数/%TSNS穗粒数/%KNS冀麦38 Jimai 38 -国审麦 98000366.750.0100科农9204 Kenong 9204冀麦38×SA502国审麦200303733.350.0100科农199 Kenong 199科农9204×石4185国审麦200601733.350.0100科农2011 Kenong 2011科农9204×PZW-9冀审麦201600350.050.0100科农8024 Kenong 8024科农2011×山农16河北省区域试验80.0100.0100科农1002 Kenong 1002金丰5027×科农2011河北省生产试验100.050.0100济麦22 Jimai 22 -国审麦200601866.7-100良星99 Liangxing 99 -国审麦200601666.7-100
3 讨论
GWAS是发掘农作物复杂农艺性状基因的有效手段。在全基因组关联分析中,群体结构对分析结果的准确性有着重要影响。Zhang等[20]利用Affymetrix Wheat 55K SNP芯片对本自然群体进行遗传多样性分析,发现本群体平均遗传多样性指数(Genetic diversity index)为0.377,平均多态性信息含量(Polymorphism information content,PIC)为0.341。与已报道的本地区小麦关联分析研究群体相比[21],遗传多样性较高,能够充分反映本区域小麦品种材料的遗传信息。群体结构分析结果表明,本自然群体可分为2个亚群,即主要由河南、陕西品种组成的第一亚群,以及主要由河北、山西、山东和北京品种/系组成的第二亚群,适合开展全基因组关联分析。
穗长是小麦重要农艺性状之一,在穗型和产量形成中起着重要作用。已有研究结果表明,除1D和4D外的染色体上都有关于穗长QTL的报道。本研究中,穗长关联标记AX-94755570-2A(15.2 Mb)与前人[22-23]在2A染色体上定位到的穗长相关标记gwm359(24Mb)和RAC875_c74_204(24 Mb)具有相近的物理位置。标记AX-111703851-6D(3.8 Mb)与Sun等[17]在6D染色体上检测到的与穗长显著关联的标记BS00046263_51具有相近的物理位置。暂未见AX-10873054-1D位点附近穗长相关QTL的报道,该SNP位点可能为新发现的位于1D染色体的穗长相关位点。AX-108730544-1D和AX-94755570-2A位点的优异等位基因频率分别仅为5.51%和3.82%,说明这些位点在本区域小麦育种实践中尚未被充分选择利用。临旱6 号在AX-108730544-1D和AX-94755570-2A 2个位点均具备优异等位基因,具有较大的育种利用价值,可作为穗长性状遗传改良的优良供体。而AX-111703851-6D位点优异等位基因频率高达92.06%,说明该位点在育种过程中已被强烈选择。
本研究中与总小穗数显著关联的位点AX-111235532-2A(125.4 Mb)与Cui等[24]在2A染色体上定位到的与总小穗数相关的标记Xpsp3088(114 Mb)具有相近的物理位置,说明在2A染色体上存在着与总小穗数相关的位点。与穗粒数显著关联的位点AX-110378404-5A(576 Mb)与Shi等[25]检测到的穗粒数显著关联标记wsnp_Ex_c31799_40545376(585 Mb)和wsnp_BF293620A_Ta_2_3(588 Mb)物理位置接近,Sukumaran等[26]也在此物理位置附近检测到一个与穗粒数显著关联的标记BobWhite_c14689_172,暗示该位点可能在春化特性不同的小麦品种中都发挥功能。与他人利用其他自然群体开展关联分析所得结果相比[17],本研究所检测到的总小穗数、穗粒数显著关联位点较少,原因一方面可能是受试验材料选择或试验环境设置的局限,尤其是年度间温度变化影响小麦穗分化、小穗小花生长发育,造成总小穗数和穗粒数变异系数较大,影响关联分析精度;另一方面更可能是本区域近30 a的小麦育种工作使得穗部性状相关位点的遗传改良已取得显著功效,构成本自然群体的绝大多数品种总小穗数和穗粒数相关位点的遗传构成达到同质化的优化状态,使得本关联群体穗部性状相关位点的遗传多样性降低,多态性位点较少。
优良基因聚合已被证明是提高小麦赤霉病抗性的有效途径[27],也是增加小麦籽粒产量的正确选择[28]。Qin等[29]利用自然群体开展粒质量相关基因TaGW2关联分析,发现TaGW2-6A/TaGW2-6B单倍型互作产生显著的加性效应,Hap-6A-A/Hap-6B-1是提高千粒质量的最佳优异单倍型组合。利用本自然群体研究粒质量关联位点优异等位基因的聚合效应,发现粒长、粒宽和千粒质量表型值随着显著关联位点优异等位基因聚合数目的增加而提高,随着非优异等位基因聚合数目的增加而降低,表型性状与优异/非优异等位基因的聚合数目呈线性相关关系[20]。本研究仅检测到一个与穗粒数显著关联位点AX-110378404-5A,且该位点在黄淮麦区小麦育种过程中已经历强烈选择,优异等位基因频率为91.3%。小麦穗长、总小穗数直接影响穗粒数多少,本研究中穗粒数与穗长和总小穗数极显著正相关,相关系数分别为0.376和0.633。穗长在本研究中变异系数较小,并被重复检测到3个显著关联位点,这些位点的优异等位基因聚合时呈现加性效应,3个位点均具备优异等位基因的品种/系穗长平均增加17.18%。总小穗数随着优异等位基因的聚合数目增加也呈现出增加的趋势,说明总小穗数关联位点优异等位基因聚合时也具有加性效应。那么,可否通过强化对穗长和总小穗数关联位点的选择来增加穗粒数,从而达到提高籽粒产量的育种目标呢?实践中长穗型小麦材料往往伴随着顶部小穗不育的现象,对穗长这一性状的选择过程中需要注意与其他产量相关性状基因的平衡,否则不一定收获好的效果。考虑到穗粒数与粒质量间的负相关关系[30],黄淮麦区小麦高产育种过程中对穗部性状的遗传改良必须考虑与籽粒性状的平衡。
比较科农9204小麦与其母本冀麦38在穗部性状显著关联位点的优异等位基因频率,可以看到科农9204小麦穗长关联位点的优异等位基因频率较低,但总小穗数关联位点的优异等位基因频率与其亲本冀麦38相当。分析科农系列小麦穗部性状显著关联位点的遗传构成,可以看到,穗长和总小穗数关联位点的优异等位基因在子一代品种科农199和科农2011中得以传递,子二代品系科农8024(河北省冬小麦区域试验)和科农1002(河北省冬小麦生产试验)在更多的位点上聚合了优异等位基因,其穗长关联位点的优异等位基因频率高出黄淮麦区北片国家区域试验对照品种济麦22和良星99。随着越来越多的重要农艺性状关键基因位点的挖掘,利用分子标记选择技术聚合优异等位基因,有助于产量、品质等复杂性状遗传改良取得突破。本研究利用7948个SNP标记,结合8个环境下的穗部性状表型数据进行关联分析,共检测到74个与穗长、总小穗数、穗粒数和穗密度相关联的标记位点,其中8个位点为多环境稳定SNP位点。分析该8个位点的优异等位基因在黄淮麦区小麦主栽品种/系的分布及其聚合效应,明确了本区域小麦亟须改良的穗部性状遗传位点,为小麦产量性状分子改良提供理论依据。
[1] 何中虎,庄巧生,程顺和,于振文,赵振东,刘旭.中国小麦产业发展与科技进步[J]. 农学学报,2018,8(1):99-106.
He Z H,Zhuang Q S,Cheng S H,Yu Z W,Zhao Z D,Liu X. Wheat production and technology improvement in China[J]. Journal of Agriculture,2018,8(1):99-106.
[2] Chen F,Gao M X,Zhang J H,Zuo A H,Shang X L,Cui D Q. Molecular characterization of vernalization and response genes in bread wheat from the Yellow and Huai Valley of China[J]. BMC Plant Biology,2013,13:199. doi:10.1186/1471-2229-13-199.
[3] 茹振钢,冯素伟,李淦.黄淮麦区小麦品种的高产潜力与实现途径[J].中国农业科学,2015,48(17):3388-3393. doi:10.3864/j.issn.0578-1752.2015.17.006.
Ru Z G,Feng S W,Li G. High-yield potential and effective ways of wheat in Yellow and Huai River Valley facultative winter wheat region[J]. Scientia Agricultura Sinica,2015,48(17):3388-3393.
[4] Guo Z F, Zhao Y S, Röder M S, Reif J C, Ganal M W, Chen D J, Schnurbusch T. Manipulation and prediction of spike morphology traits for the improvement of grain yield in wheat[J].Scientific Reports,2018,8:14435. doi:10.1038/s41598-018-31977-3.
[5] Zhou Y P,Conway B,Miller D,Marshall D,Cooper A,Murphy P,Chao S M,Brown-Guedira G,Costa J. Quantitative trait loci mapping for spike characteristics in hexaploid wheat[J]. The Plant Genome,2017,10(2):1-15.doi:10.3835/plantgenome2016.10.0101.
[6] Yao J,Wang L X,Liu L H,Zhao C P,Zheng Y L. Association mapping of agronomic traits on chromosome 2A of wheat[J]. Genetica,2009,137(1):67-75.doi:10.1007/s10709-009-9351-5.
[7] Sukumaran S,Dreisigacker S,Lopes M,Chavez P,Reynolds M P. Genome-wide association study for grain yield and related traits in an elite spring wheat population grown in temperate irrigated environments[J].Theoretical and Applied Genetics,2015,128(2):353-363. doi:10.1007/s00122-014-2435-3.
[8] 武玉国,吴承来,秦保平,王振林,黄玮,杨敏,尹燕枰.黄淮冬麦区175个小麦品种的遗传多样性及SSR标记与株高和产量相关性状的关联分析[J].作物学报,2012,38(6):1018-1028.
Wu Y G,Wu C L,Qin B P,Wang Z L,Huang W,Yang M,Yi Y P. Diversity of 175 wheat varieties from yellow and Huai River Valleys facultative wheat zone and association of SSR markers with plant height and yield related traits[J]. Acta Agronomica Sinica,2012,38(6):1018-1028.doi:10.3724/SP.J.1006.2012.01018.
[9] Zhang D L,Hao C Y,Wang L F,Zhang X Y. Identifying loci influencing grain number by microsatellite screening in bread wheat (Triticum aestivum L.)[J]. Planta,2012,236:1507-1517. doi:10.1007/s00425-012-1708-9.
[10] Cavanagh C R,Chao S M,Wang S C,Huang B E,Stephen S,Kiani S,Forrest K,Saintenac C,Brown-Guedira G L,Akhunova A,See D,Bai G H,Pumphrey M,Tomar L,Wong D,Kong S,Reynolds M,da Silva M L,Bockelman H,Talbert L,Anderson J A,Dreisigacker S,Baenziger S,Carter A,Korzun V,Morrell P L,Dubcovsky J,Morell M K,Sorrells M E,Hayden M J,Akhunov E. Genome-wide comparative diversity uncovers multiple targets of selection for improvement in hexaploid wheat landraces and cultivars[J]. Proceedings of the National Academy of Sciences,2013,14(110):8057-8062. doi:10.1073/pnas.1217133110.
[11] Wang S H,Wong D,Forrest K,Allen A,Chao S M,Huang B E,Maccaferri M,Salvi S,Milner S G,Cattivelli L,Mastrangelo A M,Whan A,Stephen S,Barker G,Wieseke R,Plieske J,Lillemo M,Mather D,Appels R,Dolferus R,Brown-Guedira G,Korol A,Akhunova A R,Feuillet C,Salse J,Morgante M,Pozniak C,Luo M C,Dvorak J,Morell M,Dubcovsky J,Ganal M,Tuberosa R,Lawley C,Mikoulitch I,Cavanagh C,Edwards K J,Hayden M,Akhunov E. Characterization of polyploid wheat genomic diversity using a high-density 90 000 single nucleotide polymorphism array[J]. Plant Biotechnology Journal,2014,12(6):787-796. doi:10.1111/pbi.12183.
[12] Winfield M O,Allen A M,Burridge A J,Barker G L,Benbow H R,Wilkinson P A,Coghill J,Waterfall C,Davassi A,Scopes G,Pirani A,Webster T,Brew F,Bloor C,King J,West C,Griffiths S,King I,Bentley A R,Edwards K J. High-density SNP genotyping array for hexaploid wheat and its secondary and tertiary gene pool[J]. Plant Biotechnology Journal,2016,14(5):1195-1206. doi:10.1111/pbi.12485.
[13] Cui F,Zhang N,Fan X L,Zhang W,Zhao C H,Yang L J,Pan R Q,Chen M,Han J,Zhao X Q,Ji J,Tong Y P,Zhang H X,Jia J Z,Zhao G Y,Li J M. Utilization of a Wheat660K SNP array-derived high-density genetic map for high-resolution mapping of a major QTL for kernel number[J]. Scientific Reports,2017,7:3788. doi:10.1038/s41598-017-04028-6.
[14] Ren T H,Hu Y S,Tang Y Z,Li C S,Yan B Z,Ren Z L,Tan F Q,Tang Z X,Fu S L,Li Z. Utilization of a Wheat55K SNP array for mapping of major QTL for temporal expression of the tiller number[J]. Frontiers in Plant Science,2018,9:333. doi:10.3389/fpls.2018.00333.
[15] Juliana P,Poland J,Huerta-Espino J,Shrestha S,Crossa J,Crespo-Herrera L,Toledo F H,Govindan V,Mondal S,Kumar U,Bhavani S,Singh P K,Randhawa M S,He X Y,Guzman C,Dreisigacker S,Rouse M N,Jin Y,Pérez-Rodríguez P,Montesinos-López O A,Singh D,Rahman M M,Marza F,Singh R P. Improving grain yield,stress resilience and quality of bread wheat using large-scale genomics[J]. Nature Genetics,2019,51:1530-1539. doi:10.1038/s41588-019-0496-6.
[16] Mora F,Castillo D,Lado B,Matus I,Poland J,Belzile F,Von Zitzewitz J,del Pozo A. Genome-wide association mapping of agronomic traits and carbon isotope discrimination in a worldwide germplasm collection of spring wheat using SNP markers[J]. Molecular Breeding,2015,35:69.doi:10.1007/s11032-015-0264-y.
[17] Sun C W,Zhang F Y,Yan X F,Zhang X F,Dong Z D,Cui D Q,Chen F. Genome-wide association study for 13 agronomic traits reveals distribution of superior alleles in bread wheat from the Yellow and Huai Valley of China[J]. Plant Biotechnology Journal,2017,15 (8):953-969. doi:10.1111/pbi.12690.
[18] Muqaddasi Q H,Brassac J,Koppolu R,Plieske J,Ganal M W,Röder M S. TaAPO-A1,an ortholog of rice ABERRANT PANICLE ORGANIZATION 1,is associated with total spikelet number per spike in elite European hexaploid winter wheat (Triticum aestivum L.) varieties[J]. Scientific Reports,2019,9:13853. doi:10.1038/s41598-019-50331-9.
[19] Merk H L,Yarnes S C,Van Deynze A,Tong N K,Menda N,Mueller L A,Mutschler M A,Loewen S A,Myers J R,Francis D M. Trait diversity and potential for selection indices based on variation among regionally adapted processing tomato germplasm[J]. Journal of the American Society for Horticultural Science,2012,137(6):427-437.doi:10.21273/Jashs.137.6.427.
[20] Zhang N,Zhang X L,Song L Q,Su Q N,Zhang S,Liu J J,Zhang W,Fu X Y,Zhao M H,Sun L J,Ji J,Qiao W C,Li H,He M Q,Li J M. Identification and validation of the favorable alleles for wheat kernel traits detected by genome-wide association study under different nitrogen environments[J].Euphytica,2020,216:52.doi:10.1007/s10681-020-2572-5.
[21] Chen G F,Zhang H,Deng Z Y,Wu R G,Li D M,Wang M Y,Tian J C. Genome-wide association study for kernel weight-related traits using SNPs in a Chinese winter wheat population[J]. Euphytica,2016,212(2):1-13.doi:10.1007/s10681-016-1750-y.
[22] Zhang H X,Zhang F N,Li G D,Zhang S N,Zhang Z G,Ma L J. Genetic diversity and association mapping of agronomic yield traits in eighty six synthetic hexaploid wheat[J].Euphytica,2017,213:111.doi:10.1007/s10681-017-1887-3.
[23] Ma F F,Xu Y F,Ma Z Q,Li L H,An D G. Genome-wide association and validation of key loci for yield-related traits in wheat founder parent Xiaoyan 6[J]. Molecular Breeding,2018,38(7):91.doi:10.1007/s11032-018-0837-7.
[24] Cui F,Ding A M,Li J,Zhao C H,Wang L,Wang X Q,Qi X L,Li X F,Li G Y,Gao J R,Wang H G. QTL detection of seven spike-related traits and their genetic correlations in wheat using two related RIL populations[J]. Euphytica,2012,186:177-192. doi:10.1007/s10681-011-0550-7.
[25] Shi W P,Yue L Q,Cheng J Y,Guo J H,Li L L,Xie K D,Dong J R,Xu Y H,Guo J,Zhou M X. A genome-wide associate study reveals favorable alleles conferring apical and basal spikelet fertility in wheat (Triticum aestivum L.)[J]. Molecular Breeding,2018,38:146. doi:10.1007/s11032-018-0906-y.
[26] Sukumaran S,Lopes M,Dreisigacker S,Reynolds M. Genetic analysis of multi-environmental spring wheat trials identifies genomic regions for locus-specific trade-offs for grain weight and grain number[J].Theoretical and Applied Genetics,2018,131:985-998. doi:10.1007/s00122-017-3037-7.
[27] Zheng Z,Gao S,Zhou M X,Yan G J,Liu C J. Enhancing Fusarium crown rot resistance by pyramiding large-effect QTL in common wheat (Triticum aestivum L.)[J]. Molecular Breeding,2017,37:107. doi:10.1007/s11032-017-0708-7.
[28] Li F J,Wen W E,Liu J D,Zhang Y,Cao S H,He Z H,Rasheed A,Jin H,Zhang C,Yan J,Zhang P Z,Wan Y X,Xia X C. Genetic architecture of grain yield in bread wheat based on genome-wide association studies[J]. BMC Plant Biology,2019,19:168. doi:10.1186/s12870-019-1781-3.
[29] Qin L,Hao C Y,Hou J,Wang Y Q,Li T,Wang L F,Ma Z Q,Zhang X Y. Homologous haplotypes,expression,genetic effects and geographic distribution of the wheat yield gene TaGW2[J]. BMC Plant Biology,2014,14:107. doi:10.1186/1471-2229-14-107.
[30] Miralles D J,Slafer G A. Sink limitations to yield in wheat :how could it be reduced?[J].The Journal of Agricultural Science,2007,145 (2):139-149. doi:10.1017/S0021859607006752.