大豆是人类食用油和植物蛋白的主要来源[1]。随着经济的快速发展和生活水平的提高,人们对大豆需求逐渐增加。2019年我国的大豆总产量达到1 810万t,但是还不能满足国内需求,需要大量进口[2]。提高产量成为大豆科研工作者关注的重点[3]。株高和大豆单产密切相关。株高过高容易产生倒伏,株高过矮达不到大豆高产的生物量要求。因此,鉴定大豆株高相关基因,进而利用分子辅助育种手段对大豆株型进行调控,对增加大豆产量和大豆株型改良都具有重要意义[4]。
目前,国内外学者利用不同方法对大豆株高进行系统研究并取得显著进展。Lee等[5]、吴晓雷等[6]、Kabelka等[7]、Wang等[8]、陈庆山等[9]利用RIL群体检测到株高相关QTL。孙亚男等[10]利用RHL材料定位到与株高相关15个位点。随后,张峰阁[11]以黑农35突变体ZK1917和黑河43杂交构建F2群体为材料通过BSA分析方法得到控制大豆株高的位点。雷硕[12]也利用BSA方法对大豆株高相关位点进行了定位。尹振功等[13]利用Overview分析方法得到与大豆株高相关的13个基因。从前人研究看出,虽然已经有株高相关QTL和基因被鉴定到,但是不同研究共同鉴定到的株高主效基因较少,说明大豆株高遗传为典型的数量性状,控制大豆株高基因较多,受遗传背景和环境条件影响均较大,需要深入研究大豆株高的遗传调控机制;而且上述研究多以单一群体单一方法进行分析,研究结果稳定性较低。利用多群体多种方法对大豆株高基因进行鉴定能够有效提高候选基因染色体区间的准确性,结合拟南芥等作物同源基因功能可以有效鉴定大豆株高调控基因,但是相关研究报道较少。本研究利用BSA关联分析的SNP-index方法和ED方法对F2和RIL 2种群体的株高基因进行检测,试图提高大豆株高相关基因检测的可靠性,为大豆株型改良和高产抗倒伏大豆新品种培育提供理论和材料基础。
本研究利用株高差异显著的东农594为父本(高秆),Charleston为母本(矮秆)杂交构建F2群体和RIL群体,然后选择亲本及F2群体极高、极矮单株各40株建立高矮秆池,同时建立RIL群体各20株的高矮秆池;进而分析亲本及2个高矮秆池DNA二代测序结果,筛选高可靠SNP,再利用亲本及高矮秆池间多态性SNP标记对大豆F2群体、RIL群体的染色体进行基于ΔSNP-index和ED方法的株高QTL关联分析,取2个群体2种方法的交集进行株高QTL关联分析,鉴定调控大豆株高的染色体区间。最后分析这些区间内候选基因,并根据拟南芥同源基因注释信息初步鉴定候选基因与株高的相关性,联合大豆旺盛生长期(R1期)高矮秆池顶芽的转录组数据进一步确认株高调控基因功能,研究结果能够为大豆株型改良和提高大豆产量提供理论依据。
1 材料和方法
1.1 试验材料
本试验的群体材料均种植于东北农业大学向阳基地。2013年再次选用株高较高的东农594(父本)和Charleston(母本)杂交获得F0种子,2014年收获F1种子,2015年获得F2群体。每年田间种植的行长为3.00 m,株距为0.05 m,垄距为0.65 m。
2016年利用Illumina Hiseq2500平台对亲本进行50倍深度测序,对RIL群体中147个株系的极高和极矮株系各20份等量合并进行20倍高通量测序;同时,对1 500株F2∶3群体中株高极高和极矮单株各40份,等量合并进行20倍高通量测序。完成测序数据质量评估及整理基础上,2018年进行调控大豆株高全基因组QTL分析。
1.2 数据分析方法
1.2.1 SNP位点的开发与关联分析 过滤接头;过滤低质量数据;去N:删除reads中的N含量大于2%的整条read。然后利用BWA软件,将测序数据比对参考基因组。将得到的SNP利用 SNP-index方法和ED方法进行关联分析,获得与性状相关的关联位点。
1.2.2 BSA分析 ΔSNP-index原理:首先对read读数大于4的SNP位点进行筛选得到高质量可信的SNP位点。然后联合亲本SNP数据计算2个混池的SNP-index,并通过ΔSNP-index计算与性状相关的SNP区间。分别对每条染色体ΔSNP-index的中值作为拟合后的阈值,当拟合标记值大于90%置信系数阈值时,与性状相关的基因组区间被识别。随后对筛选出的SNP位点进行分析[14]。计算公式如下:
SNP-index(Mut)=ρx/(ρX+ρx)
SNP-index(WT)=ρx/(ρX+ρx)
ΔSNP-index=SNP-index(Mut)-SNP-index(WT)
在这里Mut和WT分别为子代的高秆池与矮秆池,ρX和ρx分别为高秆和矮秆亲本的等位基因型。
欧氏距离计算方法:本研究还利用欧氏距离算法计算了两群体和株高相关的SNP位点。设置A高、T高、C高、G高代表 SNP位点上A、T、C、G碱基在高秆混池测序reads中出现的频率;A低、T低、C低、G低代表SNP位点上A、T、C、G碱基在矮秆混池测序reads中出现的频率。通过公式
![]()
计算每一个位点的ED值,对ED值的原始数值进行乘方处理以消除背景杂音,并对同一染色体上的ED值进行拟合并计算阈值来消除假阳性。然后选择高于阈值的区间作为株高调控基因的候选区域。本研究的关联阈值计算方法是median+3SD,将计算结果作为所有位点拟合值进行区间定位[14]。
1.2.3 转录组测序分析 为验证关联分析结果准确性,本研究对RIL群体和F2群体后代在株高形成关键时期(R1期)的高矮秆池的顶芽转录组数据进行分析,明确LOD值大于2的株高候选基因表达规律,对定位到的株高候选基因进行验证。
1.2.4 候选基因功能查找 利用拟南芥数据库(www.arabidopsis.org)和大豆数据库(www.soybase.org)信息对候选基因进行功能注释。
1.2.5 数据分析和作图 利用Excel和Photoshop 9.0对SNP位点计算结果进行整理和作图。
2 结果与分析
2.1 全基因组关联分析大豆株高QTL
首先对RIL群体的株高QTL进行BSA关联分析,共得到read读数大于4的高质量SNP位点20 285个。经计算,当置信度为0.90时,RIL群体ΔSNP-index方法的阈值为0.17,ED方法的阈值为1(图1)。计算结果表明,在18个染色体有高于阈值的区间。其中,16号染色体有1个;1,4,12,14,15,17号染色体有2个;3,8,13号染色体有3个;5,7,9,10,11,19号染色体有4个;6号染色体有5个;2号染色体有8个。
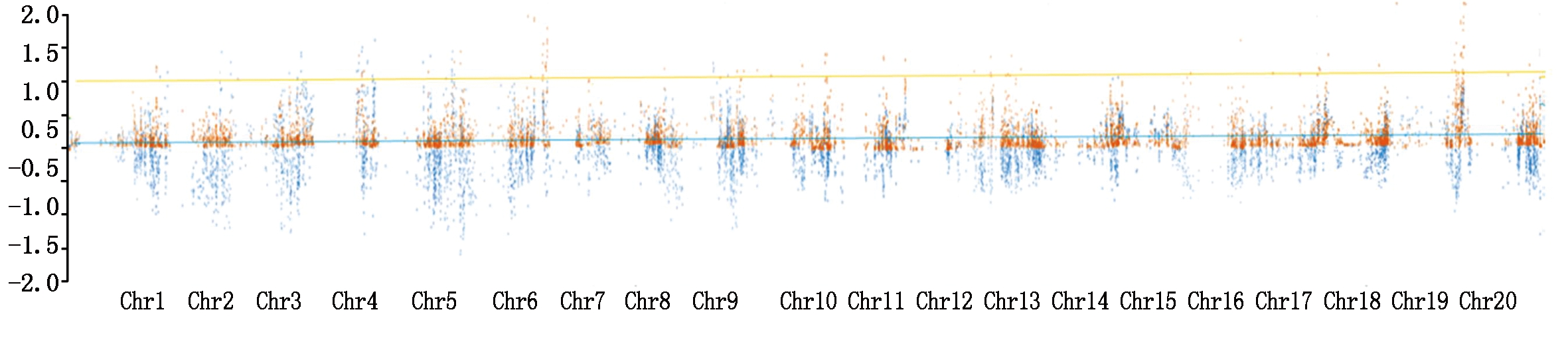
蓝色.ΔSNP-index关联分析结果;橙色线条.ED关联分析结果。图2-4同。
Blue color.The results of ΔSNP-index;Orange color.The results of ED.The same as Fig.2-4.
图1 RIL群体全基因组阈值
Fig.1 Whole genome threshold of RIL population
接下来对F2群体株高QTL进行BSA关联分析,得到read读数大于4的高质量SNP位点20 187个。当置信度为0.90时,F2群体ΔSNP-index的阈值为0.5,ED关联分析阈值为1.2(图2)。计算结果表明,19个染色体有高于阈值的区间。其中,1号染色体有1个;9,12,20号染色体有2个;10,13,16号染色体有3个;2,4,5,6,7,8,11,14,17号染色体有4个;3,15,19号染色体有5个。
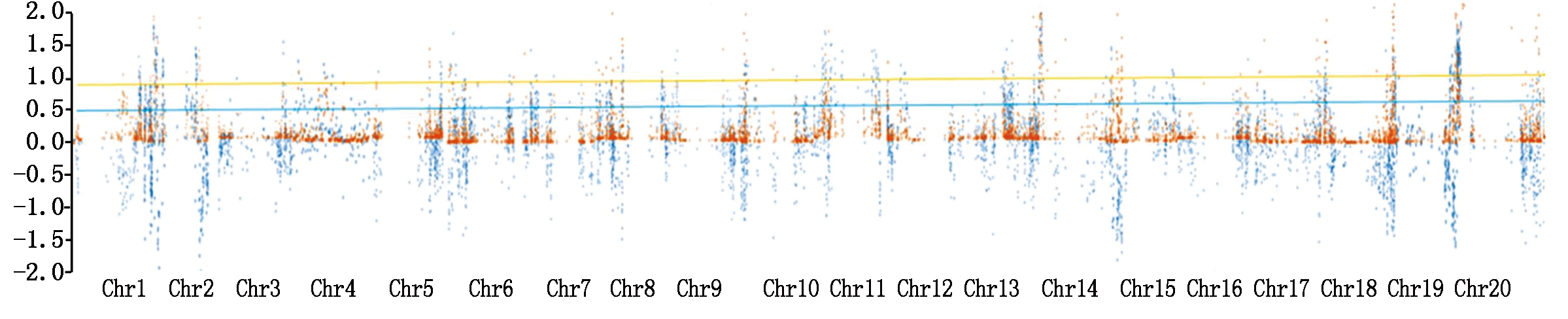
图2 F2群体全基因组阈值
Fig.2 Whole genome threshold of F2 population
由以上分析可知,2个群体2种方法分别在18和19条染色体上定位到控制株高的QTL,数量较多,为了提高定位结果的准确性,借鉴玉米果皮纤维素QTL定位方法,对存在关联区间的染色体重新计算阈值并检测QTL,最终将调控大豆株高QTL定位到1,2,3,4,5,9,11,12,15和19号染色体。
2.2 按染色体计算阈值重新分析株高关联QTL区间
2.2.1 BSA关联分析大豆株高QTL 重新分析了RIL群体各染色体高质量SNP分布情况(图3)。结果在Chr1有607个SNP位点;Chr2有1 067个;Chr3有995个;Chr4有946个;Chr5有1 023个;Chr9有1 058个;Chr11有820个;Chr12有820个;Chr15有944个。然后分别计算置信度为0.90时各染色体ΔSNP-index关联分析阈值:Chr1为0.21;Chr2、Chr5为0.19;Chr3 、Chr9为0.13;Chr4为0.70;Chr11为0.10;Chr12为0.08;Chr15为0.28;ED关联分析取各染色体所有位点拟合值的median+3SD作为关联阈值:Chr1为0.25;Chr2为0.67;Chr3和Chr4为0.38;Chr5为0.37;Chr9为0.36;Chr11为0.80;Chr12为0.64;Chr15为0.60(表1)。
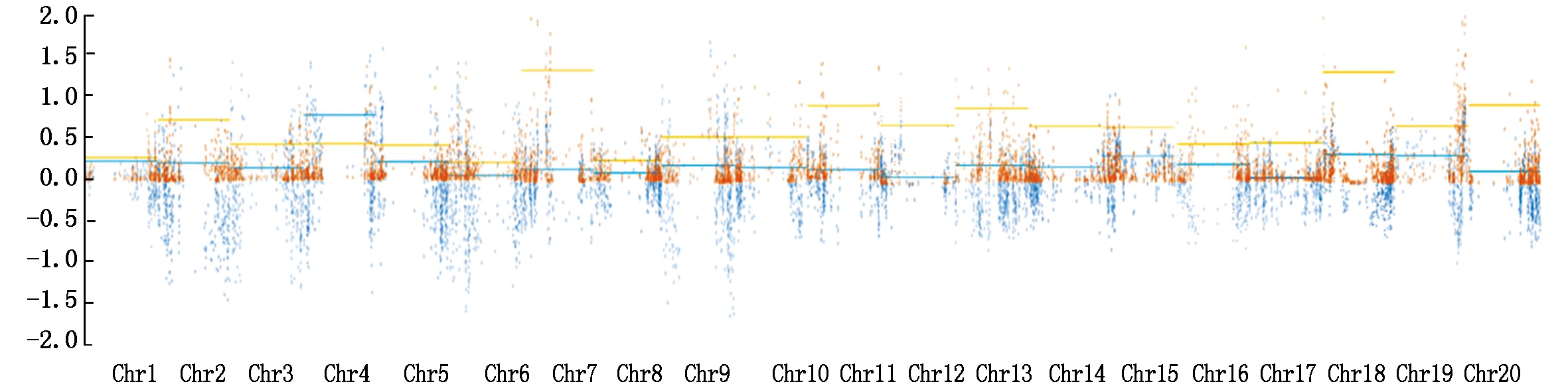
图3 RIL群体各染色体单独计算阈值
Fig.3 Thresholds of each chromosome in the RIL population are calculated separately
重新分析了RIL群体各染色体高质量SNP分布情况(图3)。结果在Chr1有607个SNP位点;Chr2有1 067个;Chr3有995个;Chr4有946个;Chr5有1 023个;Chr9有1 058个;Chr11有820个;Chr12有820个;Chr15有944个。然后分别计算置信度为0.90时各染色体ΔSNP-index关联分析阈值:Chr1为0.21;Chr2、Chr5为0.19;Chr3 、Chr9为0.13;Chr4为0.70;Chr11为0.10;Chr12为0.08;Chr15为0.28;ED关联分析取各染色体所有位点拟合值的median+3SD作为关联阈值:Chr1为0.25;Chr2为0.67;Chr3和Chr4为0.38;Chr5为0.37;Chr9为0.36;Chr11为0.80;Chr12为0.64;Chr15为0.60(表1)。
对F2群体各染色体的SNP位点分别分析结果表明Chr1有609个SNP位点;Chr2有1 075个;Chr3有995个;Chr4有937个;Chr5有1 012个;Chr9有1 058个;Chr11有820;Chr12有845个;Chr15有967个(图4)。然后计算了置信度为0.90时各染色体ΔSNP-index关联分析阈值:Chr1为0.44;Chr2为0.80;Chr3、Chr4为0.40;Chr5为0.08;Chr9、Chr11、Chr12为0.24;Chr15为0.06。通过ED关联分析取每条染色体所有位点拟合值的median+3SD作为分析的关联阈值:Chr1为0.46;Chr2为1.00;Chr3为0.58;Chr4为0.40;Chr5、Chr11、Chr12为0.45;Chr9为0.22;Chr15为0.29(表1)。
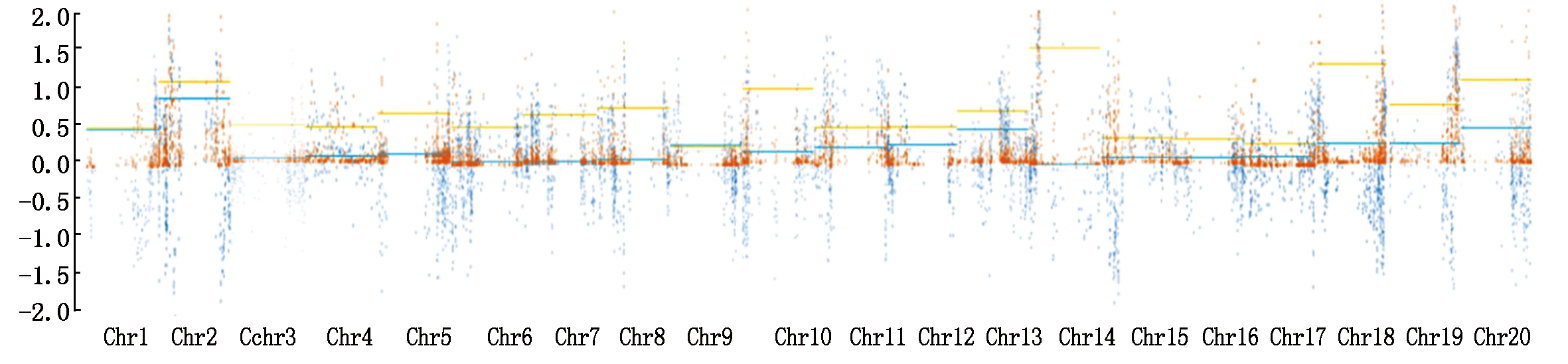
图4 F2群体各染色体单独计算阈值
Fig.4 Thresholds of each chromosome in the F2 population are calculated separately
表1 各染色体BSA关联分析阈值
Tab.1 Threshold of BSA association analysis for each chromosome
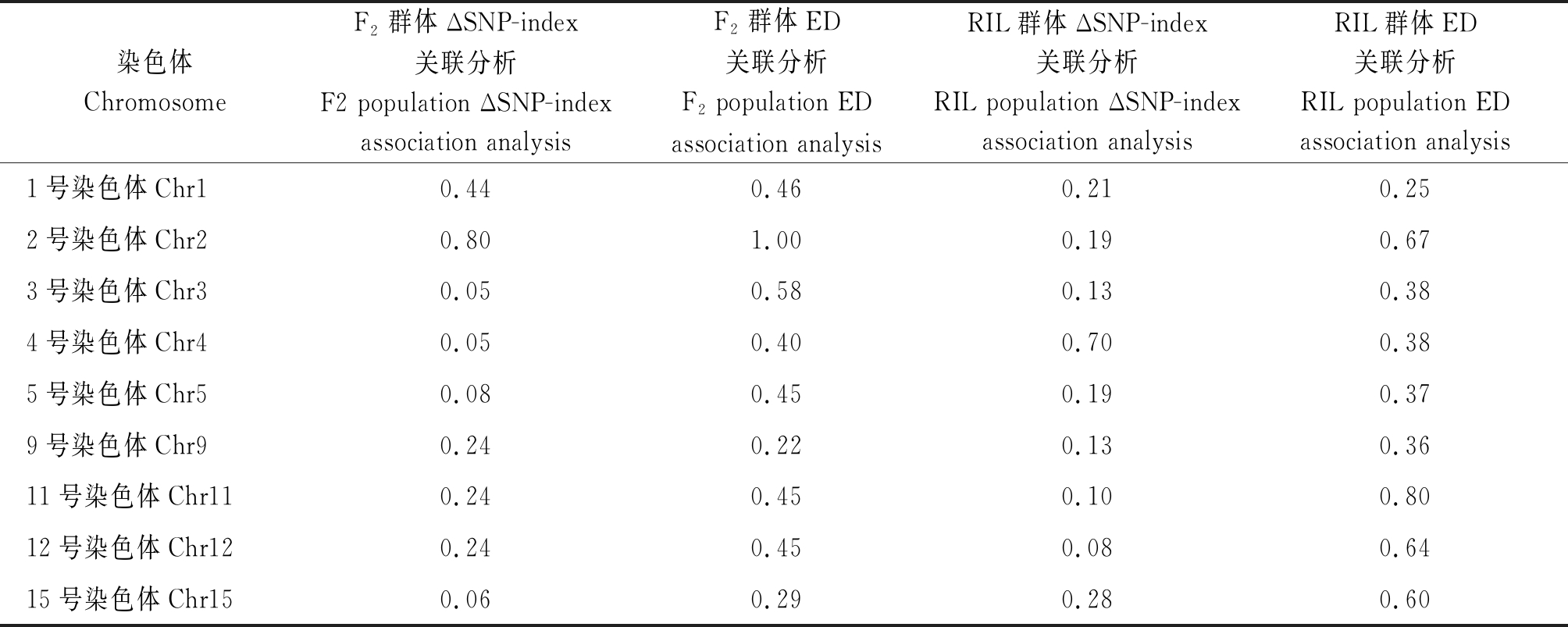
染色体ChromosomeF2群体ΔSNP-index关联分析F2 population ΔSNP-indexassociation analysisF2群体ED关联分析F2 population EDassociation analysisRIL群体ΔSNP-index关联分析RIL population ΔSNP-indexassociation analysisRIL群体ED关联分析RIL population EDassociation analysis1号染色体Chr10.440.460.210.252号染色体Chr20.801.000.190.673号染色体Chr30.050.580.130.384号染色体Chr40.050.400.700.385号染色体Chr50.080.450.190.379号染色体Chr90.240.220.130.3611号染色体Chr110.240.450.100.8012号染色体Chr120.240.450.080.6415号染色体Chr150.060.290.280.60
2.2.2 各染色体重合区间分析 合并RIL群体和F2群体定位结果,2个群体共同关联区域各染色体阈值以上的重合区间分别为:Chr1、Chr2、Chr5、Chr9、Chr11、Chr12、Chr15有1个,Ch3有2个,Ch4有4个(表2)。
表2 RIL群体和F2群体重合区间
Tab.2 RIL population and F2 population coincidence interval
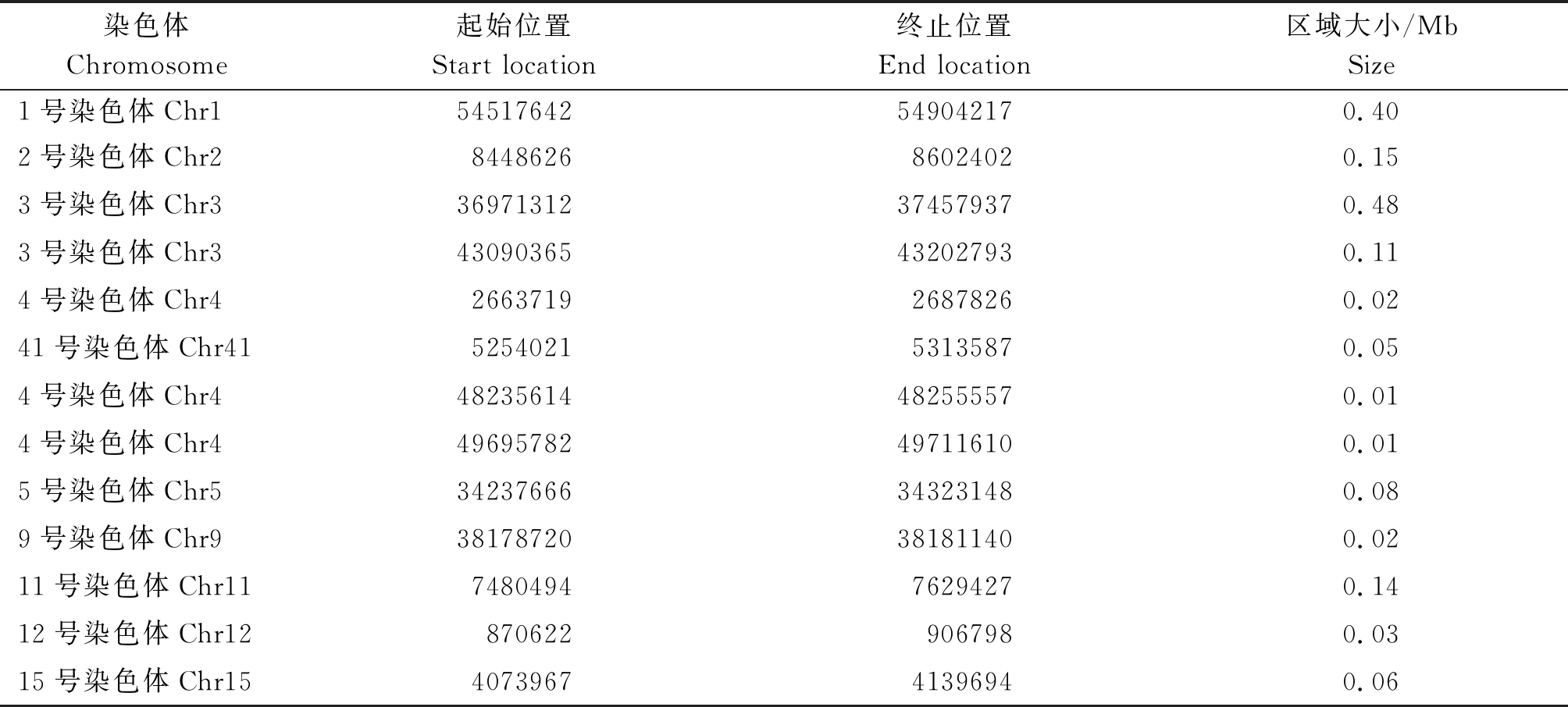
染色体Chromosome起始位置Start location终止位置End location区域大小/MbSize1号染色体Chr154517642549042170.402号染色体Chr2844862686024020.153号染色体Chr336971312374579370.483号染色体Chr343090365432027930.114号染色体Chr4266371926878260.0241号染色体Chr41525402153135870.054号染色体Chr448235614482555570.014号染色体Chr449695782497116100.015号染色体Chr534237666343231480.089号染色体Chr938178720381811400.0211号染色体Chr11748049476294270.1412号染色体Chr128706229067980.0315号染色体Chr15407396741396940.06
2.2.3 各染色体差异SNP分析 在2个群体共同关联区域内检索亲本间差异SNP位点,Chr2、Chr5、Chr9、Chr11、Chr12、Chr15有1个、Chr1有2个,Chr3有2个、Chr4有8个(表2)。然后用拟南芥基因组信息为参考,对18个候选基因功能进行注释(表3)。
2.2.4 候选基因表达分析 为验证上述基因是否参与大豆株高形成,分析了大豆旺盛生长期(R1期)顶芽的转录组数据,Glyma.03g230000、Glyma.04g065600和Glyma.04g227700在RIL群体的高矮秆池的顶芽差异表达(表4)。其中,Glyma.03g230000的拟南芥同源基因AT1G09340.1[19]编码叶绿体RNA结合(CRB)蛋白影响系统昼夜节律,突变体矮化;Glyma.04g065600的拟南芥同源基因AT2G45180.1具有脂转运蛋白活性及蛋白酶抑制剂活性,突变体株高矮化;而Glyma.04g227700的拟南芥同源基因AT5G54160.1是甲基转移酶,和细胞壁发育相关,参与油菜素内酯信号途径调控。
表3 关联区间的候选基因分析
Tab.3 Candidate gene analysis of related segments

基因Genes区间Interval拟南芥基因Gene loci in Arabidopsis拟南芥基因注释Gene annotationin Arabidopsis支持文献ReferencesGlyma.01G21430054534491-54538724AT4G22890.5类PGR5 A[15]Glyma.01G22070054977695-54985676AT1G63700.1激酶蛋白家族蛋白[16]Glyma.02G0956008598036-8600822AT5G24330.1拟南芥TRITHORAX相关蛋白6[17]Glyma.03G15810037349906-37351578AT3G54560.1组蛋白 H2A 11[18]Glyma.03g23000043190797-43194898AT1G09340.1叶绿体RNA结合[19]Glyma.04G0338002687701-2689839AT1G75080.1油菜素内脂信号正调控家族蛋白(BZR1) [20]Glyma.04G0638005293426-5299847AT4G18780.1纤维素合成蛋白[21]Glyma.04g0656005457842-5458781AT2G45180.1双功能抑制剂/脂转运/种子2S 清蛋白超级家族蛋白[22]Glyma.04G19080046143080-46147709AT1G09340.1蛋氨酸硫氧化物还原酶2[23]Glyma.04G21000048203384-48210621AT5G09230.7Sirtuin蛋白 2[24]Glyma.04G21070048245635-48265039AT5G63960.1DNA 结合蛋白[25]Glyma.04G21290048454974-48460147AT4G33460.1ABC转运家族蛋白[26]Glyma.04g22770049704598-49706834AT5G54160.1甲基转移酶1[27]Glyma.05G14870034274745-34292164AT5G65060.1MADS转录因子[28]Glyma.09G15840038175841-38181729AT2G38880.1核因子Y亚基B1[29]Glyma.11G1004007605906-7620986AT5G23150.1Tudor/PWWP/MBT 结构域蛋白[30]Glyma.12G012400904135-909947AT3G47450.2含P环的三磷酸核苷水解酶[31]Glyma.15g0524004132856-4135122AT1G15820.1光系统Ⅱ光捕获复合体 [32]
表4 大豆旺盛生长期转录组数据
Tab.4 Transcriptome data in vigorous growth stage of soybean

染色体Chromosome群体Group基因GeneLOD绝对值log absolute value3号染色体Chr3RILGlyma.03g2300002.114号染色体Chr4RILGlyma.04g0656003.014号染色体Chr4F2Glyma.04g0656002.564号染色体Chr4F2Glyma.04g2277002.01
3 讨论与结论
BSA方法原理简单,操作方便。与传统育种相比,BSA方法更加省时省力,可以缩短育种年限,目前,被广泛应用到各种作物重要QTL和主效基因检测。王瑞等[14]利用高粱K35-Y5和1383杂交构建F2群体,利用BSA法找到4个与株高关联的SNP位点。杜龙岗等[33]用重组自交系群体(RILs),定位到9个控制果皮纤维素候选基因。前人的研究证明此方法效率高、可靠性好。
利用BSA法定位大豆剩余杂合系重要性状QTL的报道已有很多[11-13,34-35]。但是前人的研究均利用剩余杂合系F2群体对质量性状进行BSA关联分析挖掘候选基因,群体的选择比较单一,而且主要研究质量性状。株高等数量性状由多个主效基因调控,受环境影响较大,单一群体和单一定位方法的定位结果不理想;利用多群体与多个定位方法相结合对株高等数量性状QTL及主效调控基因进行检测,能够提高数量性状主效基因定位结果的准确度。因此,本研究利用株高差异显著的亲本构建F2及RIL群体,运用BSA关联分析方法对株高性状进行分析。RIL群体和F2群体的全基因组关联分析结果定位到的染色体相对较多,为了提高染色体关联区间鉴定结果准确性,借鉴玉米果皮纤维素主效基因鉴定方法[33],利用2个群体2种分析方法对入选染色体用重新计算分析,最后发现10条染色体存在共同区间。本研究结果表明多群体多方法鉴定重要性状候选基因结果比单一群体更加可靠,也证明利用BSA方法进行多群体和多方法联合鉴定复杂调控的数量性状是可行性。同时候选基因鉴定结果与转录组数据结合能更好地验证候选基因功能,为筛选主效基因提供一种新思路。
大豆各染色体控制大豆株高相关QTL共有440个,分布于20条染色体(www.soybase.org)。其中,Hu等[34]得到控制大豆株高的QTL区间Satt531(Gm01:3779070)-Satt129(Gm01:55166202)。Reinprecht等[35]鉴定到大豆株高的QTL区间BARC-037223-06748(Gm02:7055032)-Satt173(Gm02:9482333)。Kabelka等[36]确认大豆株高QTL区间Satt549(Gm03:37342681)-Satt304(Gm03:39088673)。Sun等[37]鉴定大豆株高的QTL区间在Satt549(Gm03:37342681)-Satt304(Gm03:39088673)。Kim等[38]鉴定到大豆株高QTL区间BARC-021961-04241(Gm04:1601042)-Satt367(Gm04:3300829)。Yao等[39]筛选到大豆株高的QTL区间Satt171(Gm05:34434531)-Satt217(Gm05:41679029)。Chen等[40]鉴定到大豆株高QTL区间Satt509(Gm11:6216988)-Satt197(Gm11:8898878)。随着研究深入,单倍型数据也被引入到大豆株高遗传分析。Contreras等[41]基于SNP单倍型数据定位到调控大豆株高单倍型,包括Gm9_Hap24a、Gm9_Hap24b、Gm12_Hap12a、Gm12_Hap12b等。Fang等[42]通过多位点全基因组关联分析挖掘到Glyma.04G030000、Glyma.13G33430等调控大豆株高基因。从前人研究结果可以看出,SSR其定位区间相对于SNP来说定位区间相对较大,候选基因数量很多,不能对所在区间基因进行筛选,鉴定不到主效基因。而基于SNP标记数据大大降低了QTL定位的区间,提高了主效基因的鉴定能力。本研究利用2个群体2种方法的交集进行株高候选基因挖掘,共筛选到18个候选基因,其中Glyma.09G158400与Glyma.12G012400存在于Contreras等 [41]所定位的SNP区间;Glyma.03G158100与Fang等 [42]挖掘的Glyma.13G334300同属于组蛋白H2A家族来调控株高;Glyma.04G033800与Fang等 [42]挖掘的Glyma.04G030000所在的QTL区间一致,且物理图谱距离相对较近,基因功能还需下一步验证。经比对分析,本研究所定位候选基因均位于前人株高定位QTL区间,证明本研究结果是可靠的,鉴定到的基因可能为该QTL的主效基因,表明本研究能够为大豆株高分子调控及大豆优异株型育种提供目标基因。
为分析大豆株高候选基因功能及调控通路,对18个株高候选基因在拟南芥中的同源基因注释信息进行了分析,结果表明大豆株高受光质、昼夜节律、Br信号通路、纤维素含量、生长素信号通路、染色体表观修饰等调控。其中,Glyma.01G214300同源基因AT4G22890.5[15]、Glyma.01G220700同源基因AT1G63700.1[16]分别编码PGRL1A和MEKK亚家族成员,前者调控蓝光和远红光反应,和光合作用过程电子流有关,后者参与丝氨酸和苏氨酸蛋白激酶的调控,是气孔发育通路的组成部分,突变体均矮化,被确定为1号染色体的候选基因;Glyma.02G095600的拟南芥同源基因AT4G22890.5[17]编码结构域蛋白SET,调控染色质结构所需的H3K27甲基转移酶与开花时间相关,突变体矮化,被确定为2号染色体的候选基因;Glyma.03G158100的拟南芥同源基因AT3G54560.1[18]、Glyma.03g230000的拟南芥同源基因AT1G09340.1[19],分别编码HTA11蛋白和叶绿体RNA结合(CRB)蛋白,前者调控DNA甲基化而后者调控叶绿体表达量,影响系统昼夜节律,突变体均矮化,被确定为3号染色体的候选基因;Glyma.04G033800的拟南芥同源基因AT1G75080.1[20]、Glyma.04G063800的拟南芥同源基因AT4G18780.1[21]、Glyma.04g065600的拟南芥同源基因AT2G45180.1[22]、Glyma.04G190800的拟南芥同源基因AT1G09340.1[23]、Glyma.04G210000的拟南芥同源基因AT5G09230.7[24]、Glyma.04G210700的拟南芥同源基因AT5G63960.1[25]、Glyma.04G212900的拟南芥同源基因AT4G33460.1[26]、Glyma.04g227700的拟南芥同源基因AT5G54160.1[27],分别编码BR信号通路、纤维素合成酶、双功能抑制剂超家族蛋白、甲硫氨酸亚砜的还原酶活性,调控BR信号通路下游反应及次生壁合成。突变体株型均矮化,被确定为4号染色体的候选基因;Glyma.05G148700的拟南芥同源基因AT5G65060.1[28]编码K-box区域的MADS-box转录因子家族蛋白,突变体矮化,定为5号染色体的候选基因;Glyma.09G158400的拟南芥同源基因AT2G38880.1[29]编码核因子Y(NF-Y)家族的转录因子AtNF-YB1,影响植株开花时间,突变体矮化,被定为9号染色体的候选基因;Glyma.11G100400的拟南芥同源基因AT5G23150.1[30]编码含Tudor/PWWP/MBT域的蛋白,使植株早开花,突变体矮化,被定为11号染色体的候选基因;Glyma.12G012400拟南芥同源基因AT3G47450.2[31]编码含P环的核苷三磷酸水解酶超家族蛋白,调控生长激素信号传导,下调氧化应激酶和活性氧(ROS)反应,使叶片发育迟缓,突变体矮化,被确定为12号染色体候选基因;Glyma.15g052400的拟南芥同源基因AT1G15820.1[32]编码光系统II亚基6蛋白,突变体矮化,被定为15号染色体的候选基因。同时我们分析了候选基因在水稻中同源基因功能。其中,Glyma.02G095600[43]和Glyma.04G210000[44]在水稻中的同源基因通过组蛋白修饰和DNA甲基化导致植株矮化;Glyma.01G214300[45]在水稻中的同源基因通过影响pgr5在PSI循环中电子传递导致株型矮化。另外,Glyma.03G158100[46]在水稻中的同源基因为染色质重塑因子CHR729,Glyma.04g227700[47]在水稻中的同源基因和温敏不育相关,Glyma.03g230000[48]在水稻中的同源基因OsSRO1a与RNA结合域蛋白(OsRBD1)相互作用,都和植株矮化密切相关;Glyma.04G063800[49]在水稻中的同源基因突变体也导致株型矮化。在大豆株高形成的关键时期,Glyma.01G214300、Glyma.03g230000、Glyma.04g065600、Glyma.04G210000、Glyma.04g227700、Glyma.15G052400在本研究的转录组数据被检测到,表明本研究定位的候选基因都和大豆株高形成密切相关,通过不同分子途径参与大豆株高形成,而且鉴定到新的调控大豆株高基因,为从不同分子途径调控大豆株高提供了候选基因。
通过BSA分析合并RIL群体和F2群体定位结果对候选基因进行定位,共检测到18个候选基因与株高显著相关。其中1号染色体有2个;2号染色体有1个;3号染色体有2个;4号染色体有8个;5号染色体有1个;9号染色体有1个;11号染色体有1个;12号染色体有1个;15号染色体有1个。
同转录组数据相比较,当LOD值大于2时只有Glyma.03g230000、Glyma.04g065600、Glyma.04g227700 3个基因被检测到,说明这3个基因在生长旺盛期(R1期)表达。
[1] 毕影东,李炜,肖佳雷,李琬,刘明,刘淼,张必弦,林红,来永才. 大豆分子育种现状,挑战与展望[J]. 中国农学通报,2014,30(6):33-39.
Bi Y D,Li W,Xiao J L,Li W,Liu M,Liu M,Zhang B X,Lin H,Lai Y C. Soybean molecular breeding:current status,challenges and perspectives[J]. Chinese Agricultural Science Bulletin,2014,30(6):33-39.
[2] Westcott P. USDA agricultural baseline projections to 2012[J].Situation Outlook Report Rice,2010.
[3] 王育民,卜实,刘忠臣. 国内外大豆生产和贸易现状分析及前景展望[J]. 大豆科技,2001(6):21-23. doi:10.3969/j.issn.1674-3547.2001.06.016.
Wang Y M,Bu S,Liu Z C. Analysis and prospect of soybean production and trade at home and abroad[J]. Soybean Technology,2001(6):21-23.
[4] 王曙明,范旭红. 大豆倒伏问题应引起高度重视[J]. 中国农业信息,2008(11):29. doi:10.3969/j.issn.1674-3547.2009.01.003.
Wang S M,fan X H. Soybean lodging should be paid more attention[J]. China Agricultural Information,2008(11):29.
[5] Lee S H,Bailey M A,Mian M A R,Carter T E,Ashley D A,Hussey R S. Molecular markers associated with soybean plant height,lodging,and maturity across locations[J]. Crop Sci,1996,36(3):728-735.
[6] 吴晓雷,王永军,贺超英,陈受宜,盖钧镒,王学臣. 大豆重要农艺性状的QTL分析[J].遗传学报,2001,28(10):947-955.
Wu X L,Wang Y J,He C Y,Chen S Y,Gai J Y,Wang X C. Mapping of some agronomic traits of soybean[J]. J Genet Genomics,2001,28(10):947-955.
[7] Kabelka E A,Diers B W,Fehr W R,LeRoy A R,Baianu I C,You T,Neece D J,Nelson R L. Putative alleles for increased yield from soybean plant introductions[J]. Crop Sci,2004,44(3):784-791. doi:10.2135/cropsci2004.0784.
[8] Wang D,Graef G L,Procopiuk A M,Diers B W. Identification of putative QTL that underlie yield in interspecific soybean backcross populations[J]. Theor Appl Genet,2004,108(3):458-467. doi:10.1007/s00122-003-1449-z.
[9] 陈庆山,张忠臣,刘春燕,辛大伟,单大鹏,邱红梅. 大豆主要农艺性状的QTL分析[J]. 中国农业科学,2007,40(1):41-47. doi:10.3321/j.issn:0578-1752.2007.01.006.
Chen Q S,Zhang Z C,Liu C Y,Xin D W,Shan D P,Qiu H M. QTL analysis of main agronomic characters of soybean[J]. China Agricultural Science,2007,40(1):41-47.
[10] 孙亚男,齐照明,单大鹏,刘春燕,胡国华,陈庆山. 大豆株高QTL的定位与整合分析[J]. 分子植物育种,2010(4):69-75.
Sun Y N,Qi Z M,Shan D P,Liu C Y,Hu G H,Chen Q S. Location and integration analysis of QTL for plant height of soybean[J]. Molecular Plant Breeding,2010(4):69-75.
[11] 张峰阁. 大豆结荚习性控制基因的遗传定位[D]. 北京:中国科学院,2018.
Zhang F G. Genetic mapping of controlling genes of soybean podding habit[D]. Beijing:Chinese Academy of Sciences,2018.
[12] 雷硕. 大豆顶生花序长度性状的种质资源筛选及基因定位[D]. 长春:吉林大学,2018.
Lei S. Selection of germplasm resources and gene mapping of soybean terminal inflorescence length[D]. Changchun:Jilin University,2018.
[13] 尹振功,王强,孟宪欣,刘广阳,郭怡璠,王秀君. 基于Overview和物理图谱的大豆株高性状候选基因挖掘[J]. 大豆科学,2019(6):914-920. doi:10.11861/j.issn.1000-9841.2019.06.0914.
Yin Z G,Wang Q,Meng X X,Liu G Y,Guo Y L,Wang X J. Candidate gene mining for plant height traits based on overview and physical map[J]. Soybean Science,2019(6):914-920.
[14] 王瑞,凌亮,詹鹏杰,于纪珍,楚建强,平俊爱,张福耀. 控制高粱分蘖与主茎株高一致性的基因定位[J]. 作物学报,2019,45(6):829-838. doi:10.3724/SP.J.1006.2019.84111.
Wang R,Ling L,Zhan P J,Yu J Z,Chu J Q,Ping J A,Zhang F Y. Gene mapping to control the consistency between tillering and main stem height of sorghum[J]. Acta Agronomica Sinica,2019,45(6):829-838.
[15] DalCorso G,Pesaresi P,Masiero S,Aseeva E,Sch nemann D,Finazzi G,Joliot P,Barbato R,Leister D. A complex containing PGRL1 and PGR5 is involved in the switch between linear and cyclic electron flow in arabidopsis[J]. Cell,2008,132(2):273-285. doi:10.1016/j.cell.2007.12.028.
[16] Wang H,Ngwenyama N,Liu Y D,Walker J C,Zhang S Q. Stomatal development and patterning are regulated by environmentally responsive mitogen-activated protein kinase in Arabidopsis[J]. Plant Cell,2007,19(1):63-73. doi:10.1105/tpc.106.048298.
[17] Zhao H W,Xing D H,Li Q Q. Unique features of plant cleavage and polyadenylation specificity factor revealed by proteomic studies[J]. Plant Physiol,2009,151(3):1546-1556. doi:10.2307/40537975.
[18] Cao D N,Cheng H,Wu W,Soo H M,Peng J R. Gibberellin mobilizes distinct DELLA-Dependent transcriptomes to regulate seins are related to Arabidopsis seedling development[J]. Proteomics,2006,142(2):509-525. doi:10.2307/20205944.
[19] Hassidim M,Yakir E,Fradkin D,Hilman D,Kron I,Keren N,Harir Y,Yerushalmi S,Green R M. Mutations in chloroplast RNA binding provide evidence for the involvement of the chloroplast in the regulation of the circadian clock in Arabidopsis[J]. Plant J,2007,51(4):551-562. doi:10.1111/j.1365-313x.2007.03160.x.
[20] Kanno Y,Oikawa T,Chiba Y,Ishimaru Y,Shimizu T,Sano N,Koshiba T,Kamiya Y,Ueda M,Seo M. AtSWEET13 and AtSWEET14 regulate gibberellin-mediated physiological processes[J]. Nature Commun,2016,7:13245. doi:10.1038/ncomms13245.
[21] Somerville T C R. Collapsed xylem phenotype of Arabidopsis identifies mutants deficient in cellulose deposition in the secondary cell wall[J]. Plant Cell,1997,9(5):689-701. doi:org/10.1105/tpc.9.5.689.
[22] Li F L,Asami T,Wu X Z,Tsang E W T,Cutler A J. A putative hydroxysteroid dehydrogenase involved in regulating plant growth and development[J]. Plant Physiol,2007,45(1):87-97. doi:10.2307/40065611.
[23] Bechtold U,Murphy D J,Mullineaux P M. Arabidopsis peptide methionine sulfoxide reductase2 prevents cellular oxidative damage in long nights[J].Plant Cell,2004,16(4):908-919. doi:10.1105/tpc.015818.
[24] Zhang F,Wang L K,Esther K E,Kevin S,Hong Q. Histone deacetylases SRT1 and SRT2 interact with eNAP1 to mediate ethylene-induced transcriptional repression[J]. Plant Cell,2018,30(1):153-166. doi:10.1105/tpc.17.00671.
[25] Eduardo M B,David E B,Lucía J V,Riad N,Hector C,María L F. In CURVATA11 and CUPULIFORMIS2 are redundant genes that encode epigenetic machinery components in Arabidopsis[J]. Plant Cell,2018,30(7):1596-1616. doi:10.1105/tpc.18.00300.
[26] Lena V,Jiyoung P,Roland S,Christopher L. A novel prokaryote-type ECF/ABC transporter module in chloroplast metal homeostasis[J]. Front Plant Sci,2019,1:264. doi:10.3389/fpls.2019.01264.
[27] Shi H T,Wei Y X,He C Z. Melatonin-induced CBF/DREB1s are essential for diurnal change of disease resistance and CCA1 expression in Arabidopsis[J]. Plant Physiol Bioch,2016,100:150-155. doi:10.1016/j.plaphy.2016.01.018.
[28] Schonrock N. Polycomb-group proteins repressthe floral activator AGL19 in the FLC-independent vernalization pathway[J]. Genes Dev,2006,20(12):1667-1678. doi:10.1101/gad.377206.
[29] Wenkel S,Turck F,Singer K,Gissot L,Gourrierec J L,Samach A,Coupland G. CONSTANS and the CCAAT box binding complex share a functionally important domain and interact to regulate flowering of Arabidopsis[J]. Plant Cell,2006,18(11):2971-2984. doi:10.1105/tpc.106.043299.
[30] Havaux M,Eymery F,Porfirova S,Rey P,Dörmann P. Vitamin E protects against photoinhibition and photooxidative stress in Arabidopsis thaliana[J]. Plant Cell,2005,17(12):3451-3469. doi:10.1105/tpc.105.037036.
[31] Flores-Pérez,Sauret-Güeto S,Gas E,Jarvis P,Rodrí guez-Concepción M. A mutant impaired in the production of plastome-encoded proteins uncovers a mechanism for the homeostasis of isoprenoid biosynthetic enzymes in Arabidopsis plastids[J].Plant Cell,2008,20(5):1303-1315. doi:10.1105/tpc.108.058768.
[32] Silvia de B,Dall′Osto L,Giuseppe T,Tomas M,Roberto B. Minor antenna proteins CP24 and CP26 affect the interactions between photosystem Ⅱ subunits and the electron transport rate in grana membranes of Arabidopsis[J]. The Plant Cell,2008,20(4):1012-1028. doi:10.1105/tpc.107.055749.
[33] 杜龙岗,王美兴. 玉米SLAF标记的开发及其在玉米果皮纤维素含量BSA分析中的应用[J]. 中国农业科学,2018,51(8):1421-1430. doi:10.3864/j.issn.0578-1752.2018.08.001.
Du L G,Wang M X. SLAF-Marker development and its application in BSA analysis of cellulose content in pericarp of maize[J]. Sci Agric Sin,2018,51(8):1421-1430.
[34] Hu Z B,Zhang H R,Kan G Z,Ma D Y,Zhang D,Shi G X,Hong D L,Zhang G Z,Yu D Y. Determination of the genetic architecture of seed size and shape via linkage and association analysis in soybean(Glycine max L. Merr.)[J]. Genetica,2013,141(4-6):247-254. doi:10.1007/s10709-013-9723-8.
[35] Reinprecht Y,Poysa V,Yu K,Rajcan I,Ablett G,Pauls K. Seed and agronomic QTL in low linolenic acid,lipoxygenase-free soybean(Glycine max (L.)Merrill)germplasm[J]. Genome,2006,49(12):1510-1527. doi:10.1139/g06-112.
[36] Kabelka E A,Diers B W,Fehr W R,LeRoy A R,Baianu I C,You T,Neece D J,Nelson R L. Putative alleles for increased yield from soybean plant introductions[J]. Crop Sci,2004,44(3):784-791. doi:10.2135/cropsci2004.0784.
[37] Sun D S,Li W B,Zhang Z C,Chen Q S,Ning H L,Qiu L J,Sun G L. Quantitative trait loci analysis for the developmental behavior of soybean[J]. Springer Nature Journal,2006,112(4):665-673. doi:10.1007/s001220050895.
[38] Kim K S,Diers B W,Hyten D L,Mian M A,Shannon J G,Nelson R L. Identification of positive yield QTL alleles from exotic soybean germplasm in two backcross populations[J]. Theor Appl Genet,2012,125(6):1353-1369. doi:10.1007/s00122-012-1944-1.
[39] Yao D,Liu Z Z,Zhang J,Liu S Y,Wang P W. Analysis of quantitative trait loci for main plant traits in soybean[J]. Genetics Molecular Research,2015,14(2):6101-6109. doi:10.4238/2015.June.8.8.
[40] Chen Q S,Zhang Z C,Liu C Y,Xin D W,Shan D P,Qiu H M. QTL analysis of major agronomic traits in soybean[J]. Entia Agricultura Sinica,2007,6(4):399-405. doi:10.1016/S1671-2927(07)60062-5.
[41] Contreras R,Mora F,Mar O,Higashi W,Schuster I. A Genome-wide association study for agronomic traits in soybean using SNP Markers and SNP-based haplotype analysis[J]. PLoS One,2017,12(2):e0171105. doi:10.1371/journal.pone.0171105.
[42] Fang Y L,Liu S L,Dong Q Z,Zhang K X,Tian Z X,Li X Y,Li W B,Qi Z Y,Wang Y,Tian X C,Song J,Wang J J,Yang C,Jiang S T,Li W X,Ning H L. Linkage analysis and multi-locus genome-wide association studies identify QTNs controlling soybean plant height[J]. Front Plant Sci,2020,11:9. doi:10.3389/fpls.2020.00009.
[43] Choi S C,Lee S,Kim S R,Lee Y S,Liu C,Cao X. Trithorax group protein oryza sativa trithorax1 controls flowering time in rice via interaction with early heading date3[J]. Plant Physiol,2014,164(3):1326-1337. doi:10.1104/pp.113.228049.
[44] 周少立. 组蛋白修饰和DNA甲基化调控水稻发育的表观遗传机制研究[D]. 武汉:华中农业大学,2017.
Zhou S L. Epigenetic mechanism of histone modification and DNA methylation regulating rice development[D]. Wuhan:Huazhong Agricultural University,2017.
[45] Yuri N,Hiroshi Y,Yuki O,Shinya W,Nozomi S,Yoshichika T,Kazuhiko S,Amane M,Toshiharu S. PGR5-Dependent cyclic electron transport around PSI contributes to the redox homeostasis in chloroplasts rather than CO2 fixation and biomass croduction in rice[J]. Plant Physiol,2012,53(12):2117-2126. doi:10.1093/pcp/pcs153.
[46] 李健健. 水稻染色质重塑因子CHR729对激素代谢的影响[J]. 湖北农业科学,2015,54(2):468-473. doi:10.14088/j.cnki.issn0439-8114.2015.02.055.
Li J J. Effect of rice chromatin remodeling factor CHR729 on hormone metabolism[J]. Hubei Agricultural Science,2015,54(2):468-473.
[47] 张莹雪. 水稻温敏不育系育性转换的蛋白质组学分析[D].开封:河南大学,2015.
Zhang Y X. Proteomic analysis of fertility transformation of thermo sensitive male sterile lines in rice[D]. Kaifeng:Henan University,2015.
[48] Sharma S,Kaur C,Singla-Pareek S L,Sopory S K. OsSRO1a Interacts with RNA binding domain-containing protein(OsRBD1)and functions in abiotic stress tolerance in yeast[J]. Front Plant Sci,2016,7:62. doi:10.3389/fpls.2016.00062.
[49] 娄腊梅,解志伟,尹亮,赵金凤,袁守江,张文会,赵宝华,李学勇. 两个水稻细卷叶等位突变体的基因定位[J]. 核农学报,2014,28(1):7-13. doi:10.11869/j.issn.100-8551.2014.01.0007.
Lou L M,Xie Z W,Yin L,Zhao J F,Yuan S J,Zhang W H,Zhao B H,Li X Y. Gene mapping of two rice fine roll leaf allelic mutants[J]. Journal of Nuclear Agriculture Science,2014,28(1):7-13.