水稻抽穗期(Heading stage)作为水稻生命活动的一个重要生理时期,它决定了水稻品种的种植区域和环境适应性。水稻的抽穗期(开花时间)主要是由感光性(Photoperiod sensitivity,PS)、感温性(Temperature sensitivity,TS)和基本营养生长状况(Basic vegetative growth,BVG)3个因素决定的[1]。水稻属于短日照植物,一般来说在长日照条件下抽穗期延长,短日照条件下抽穗期缩短,高温环境下抽穗期缩短,低温环境下抽穗期延长。水稻营养物质的积累时间和光合作用的长短,及籽粒的灌浆时间和生长进程都受到抽穗期的影响,因此,要使种植品种达到高产稳产的效果,必需关注抽穗期这一性状。研究学者从粳稻品种日本晴和籼稻品种(Kasalath)杂交后代的分离群体中鉴定出了一定数量的与开花调控有关的QTLs,包括Hd1、Hd2/Ghd7.1/OsPRR37、Hd3a、Ghd7和DTH8/Ghd8[1-5]。一些开花基因如Ghd7、DTH8等存在一因多效现象,这些基因不仅能作为主效基因调控水稻的生育期,同时可以通过调控生长发育过程和植株的形态结构来调控水稻产量构成因素。DTH8(Ghd8/Hd5)是株高和抽穗期多效性控制基因,全长1 156 bp,含有1个外显子,编码一个由297个氨基酸组成的多肽[2,5-6]。DTH8在许多组织中表达,长日照条件下能下调Ehd1和Hd3a的转录,且独立于Ghd7和Hd1;DTH8通过调节Ehd1、RFT1和Hd3a使水稻开花期延长,但短日照条件下促进水稻提前开花[5]。长日照条件下,导入Asominori中有功能的DTH8等位基因能显著增加CSSL61的抽穗期和株高[6]。综上所述,DTH8不仅能同时调控水稻的生育期、株高和产量性状,还能调控分蘖数和穗部分枝,当DTH8突变后,能有效减弱水稻的光周期敏感性。
CRISPR/Cas9(Clustered Regularly Inter-spaced Short Palindromic Repeat,CRISPR)技术是近几年来发展较快的一项基因编辑技术,它具有精准、高效、快捷等优点[7],通过核酸酶引起靶位点处的DNA双链断裂,激活细胞内非同源末端连接(Nor homologous End Joining,NHEJ)和同源重组(Homologous Recombination,HR)2种修复机制进行修复。目前,CRISPR/Cas9技术不但在酵母、人、鼠、果蝇中广泛应用[8-11],在拟南芥、小麦、甜橙、烟草、水稻、玉米等植物中也实现了基因的定点编辑[12-16]。该技术在多种生物中已逐渐成为一种成熟的基因组编辑工具,在研究基因功能方面具有一定的促进作用[17]。CRISPR/Cas9系统即成簇的、有规律的、间隔短回文重复序列在古细菌和细菌中普遍存在,能够为机体提供一种特异性免疫保护机制[18]。CRISPR/Cas系统能够特异地识别外源DNA,切割外源DNA造成基因的沉默。由于Ⅰ型和Ⅲ型结构复杂,在切割DNA链时,需要多个Cas蛋白形成复合体发挥作用,而Ⅱ型系统在对靶DNA进行切割时,只需一个Cas9蛋白即可,该系统中只含有一个Cas9蛋白和一小段sgRNA序列,约20 bp,主要用来识别目的序列,Cas9指导前体crRNA的成熟和对外源DNA的切割。DNA双链断裂能够引起细胞内的自我修复机制,非同源末端连接保真度较低,在切割位点能够引起碱基的缺失或插入,造成基因沉默或移码突变等,都可能导致基因失去功能;在修复过程中,若存在DNA模板时,则会启动高保真的同源重组修复机制,通过插入一段同源序列与目的基因进行同源交换,从而完成基因组的定向精准编辑[19]。
香糯99-25稻米品质优良,全生育期168 d,抗倒伏,抽穗期较晚,抗病能力一般,产量约9 000~9 750 kg/hm2。生产上对照品种临稻10号于8月25日抽穗,香糯99-25于9月6日抽穗,大约比生产上对照品种临稻10号晚11 d左右,极大地制约了其在生产上的应用。由于我国北方地区秋冬两季长日低温,不适宜种植熟期较长的水稻品种,在一定程度上限制了一些高产、稳产水稻品种的种植。不少研究表明,水稻的生物学和经济学产量会随着抽穗期的缩短而降低[20],如果能利用基因编辑技术对控制抽穗期的基因进行编辑,将有利于培育早熟、丰产、优质的种质资源,对于水稻研究进展的各个方面及指导今后的育种工作、品种改良都具有极其重要的意义。本研究利用CRISPR/Cas9基因编辑技术对水稻抽穗期DTH8基因进行了编辑,获得了抽穗期较野生型香糯99-25明显提前的突变体水稻材料,结合田间抽穗期调查,结果符合预期,为后续DTH8基因功能的研究提供了重要的理论基础。
1 材料和方法
1.1 试验材料
本试验使用的水稻野生型粳稻品种香糯99-25,该品种农艺性状优良,在黄淮区抽穗期比生产上主栽品种平均晚抽穗10 d左右,限制了在生产上的推广应用。该品种夏季种植在山东省农业科学院饮马泉试验基地,冬季种植在三亚南繁站试验田。
1.2 菌株、质粒和载体
大肠杆菌DH5α感受态细胞来自上海生工生物工程有限公司,EHA105农杆菌感受态细胞来自济南博门特生物科技有限公司,本试验所采用的CRISPR/Cas9载体由华南农业大学生命科学学院刘耀光老师提供。系统包括2个载体,分别为中间载体和表达载体。中间载体AtU6-26-sgRNA-SK构建Single Guide RNA(sgRNA)cassette,表达载体pCAMBIA1300-YAO∶Cas9转化植物。
1.3 主要试剂
限制性内切酶Bsa Ⅰ、Spe Ⅰ和Nhe Ⅰ(New England Biolabs),DNA连接酶T4 DNA ligase(TaKaRa);质粒提取试剂盒(上海生工生物工程有限公司);琼脂糖凝胶回收试剂盒(上海生工生物工程有限公司);卡那霉素(Kanamycin)、氨苄青霉素(Ampicillin)(上海先锋药业公司);DNA测序服务(上海生工生物工程有限公司);本研究所有引物均由上海生工生物工程有限公司合成(表1)。
表1 本研究所用的引物
Tab.1 Primers used in this research

引物名称Primername引物序列(5′-3′)Primersequence(5′-3′)用途ApplicationDTH8-FCAGGAGTGCGTGTCGGAGTTqRT-PCR正向引物DTH8-RGGTCGTCGCCGTTGATGGTqRT-PCR反向引物DTH8-M1FGCCGGTCCGGCGAGGCGGCGGCGG正向靶点引物DTH8-M1RAAACCCGCCGCCGCCTCGCCGGAC反向靶点引物DTH8-M2FGCCGGGTCATCAGCAGCAGGACGG正向靶点引物DTH8-M2RAAACCCGTCCTGCTGCTGATGACC反向靶点引物1300-FCCAGTCACGACGTTGTAAAAC菌液PCR正向引物1300-RCAATGAATTTCCCATCGTCGAG菌液PCR反向引物U6-sgRNA-FCTCACTATAGGGCGAATTGG菌液PCR正向引物Hpt-FACACTACATGGCGTGATTTCAT检测正向引物Hpt-RTCCACTATCGGCGAGTACTTCT检测反向引物DTH8-1-FTCGGGTAGGGACGAGAATGT克隆DTH8-1-RGCGGTAGCGGTTGAGGTAGG克隆DTH8-2-FCTCAAGTCCTACCTCAACCG克隆DTH8-2-RAAACAGCATCAGCATCAACA克隆
注:下划线碱基代表水稻固定接头。
Note:Underlined bases represent rice fixed linker.
1.4 靶点的选择及sgRNA设计
在CRISPR-P网站(http://cbi.hzau.edu.cn/crispr/)输入DTH8基因序列号进行靶位点设计。根据CRISPR/Cas9系统识别PAM上游大约20个碱基序列的特点来筛选合适的靶位点[21],通过Blast分析进行靶位点特异性的比对验证。确定靶位点后,在靶位点序列前面加上Bsa Ⅰ限制性内切酶的黏性末端的接头,具体引物序列见表1。
1.5 CRISPR-Cas9表达载体的构建
用双蒸水将靶点引物(DTH8-M1F/R、DTH8-M2F/R)稀释到10 μmol/L,通过退火形成引物二聚体,F引物和R引物各取1 μL加8 μL ddH2O稀释,沸水浴30 s立刻取出,自然冷却。通过限制性内切酶Bsa Ⅰ酶切将AtU6-26-sgRNA-SK载体线性化,经1%琼脂糖凝胶电泳检测,切胶回收酶切产物,利用T4连接酶将目的片段(Target)连接到线性化AtU6-26-sgRNA-SK载体上,用热激法转化大肠杆菌感受态细胞DH5α,次日挑取单克隆菌落用U6-sgRNA-F和DTH8-M1R进行菌落PCR鉴定,并测序验证。获得的中间载体AtU6-26-sgRNA-SK-DTH8用Spe Ⅰ和Nhe Ⅰ双酶切后,切胶回收约642 bp的片段,与Spe Ⅰ酶切后的pCAMBIA1300∶Cas9载体通过T4 DNA连接酶进行连接,热激法转化大肠杆菌感受态细胞DH5α,次日挑取单克隆菌落用1300-F和1300-R进行菌落PCR鉴定,并测序验证,获得双元载体CRISPR-DTH8。
1.6 T0转基因植株的获得与检测
利用农杆菌介导法将CRISPR-DTH8载体转入香糯水稻愈伤组织,利用潮霉素进行筛选。在分蘖盛期,采用CTAB法[22]提取水稻T0基因编辑植株叶片DNA,利用潮霉素引物Hpt-F/R对载体中的潮霉素抗性基因进行PCR检测,得到基因编辑阳性植株。
1.7 转基因植株的突变检测及qRT-PCR分析
分蘖盛期取基因编辑植株叶片,采用CTAB法[22]提取水稻基因组DNA。根据不同靶位点所在位置的附近序列,利用Primer 5.0设计引物对不同靶位点的突变类型进行检测。对试验中的靶位点的转基因阳性植株设计2对引物DTH8-1-F/R、DTH8-2-F/R(表1)分别进行目的片段的扩增。PCR体系:DNA模板2 μL,2×Power Taq PCR Master Mix 10 μL,正向引物1 μL,反向引物 1 μL,ddH2O 6 μL混合,共20 μL。PCR反应程序:98 ℃预变性2 min;98 ℃变性30 s,60 ℃退火30 s,72 ℃延伸1 min,30个循环;72 ℃延伸7 min。经120 V 1%琼脂糖凝胶电泳25 min,将产物送至上海生工生物有限公司进行测序,并利用DNAMAN对测序结果进行分析。
采用TRIzol法[23]提取突变体及野生型叶片总RNA。首先利用DNase Ⅰ处理总RNA,以消化处理后的RNA为模板,采用cDNA合成试剂盒反转录合成第一链cDNA,然后利用实时荧光定量PCR(qRT-PCR)方法分析DTH8在野生型和突变体中的表达量,其中Ubiquitin基因作为内参基因。qRT-PCR体系:cDNA模板2 μL,正向引物 1 μL,反向引物 1 μL,2×SYBR qPCR Mix 12.5 μL,ddH2O 8.5 μL,共计 25 μL。PCR扩增程序:预变性95 ℃ 5 min;变性95 ℃ 15 s,退火60 ℃ 20 s,延伸72 ℃ 30 s,42个循环。利用公式2-ΔΔCT方法计算基因相对表达量。
1.8 突变体表型分析
1.8.1 抽穗期及主要农艺性状调查 T1种植筛选得到的基因型纯合的株系,野生型和每个突变体各种植40株。野生型材料和每个突变体材料各调查20株的抽穗期,求其平均抽穗期;同时考察株高、每穗实粒数、千粒质量、穗长等产量性状,获得数据通过SPSS 16.0进行统计分析。
1.8.2 通过t检验验证基因编辑的抽穗期 利用t检验来比较野生型水稻材料和不同突变体之间的差异显著性。以概率α=10-6为阈值,即当突变体的抽穗天数与对照抽穗天数之间的t检验的P≤10-6时,证明该突变体上基因编辑引起了抽穗期的变化。
2 结果与分析
2.1 构建DTH8的CRISPR-Cas9表达载体
分析DTH8基因序列,设计引物DTH8-M1F和DTH8-M1R、DTH8-M2F和DTH8-M2R,将退火后的靶点DTH8-M1、DTH8-M2分别与Bsa Ⅰ 酶切后的AtU6-26-sgRNA-SK(图1-A)连接,连接后转化涂板过夜培养后挑单克隆菌,通过菌落PCR检测,琼脂糖凝胶电泳后片段大小为499 bp(图1-C),连接正确。菌液经过摇菌提质粒(PCR产物)分别测序,测序结果含有靶点DTH8-M1、DTH8-M2证明连接正确。
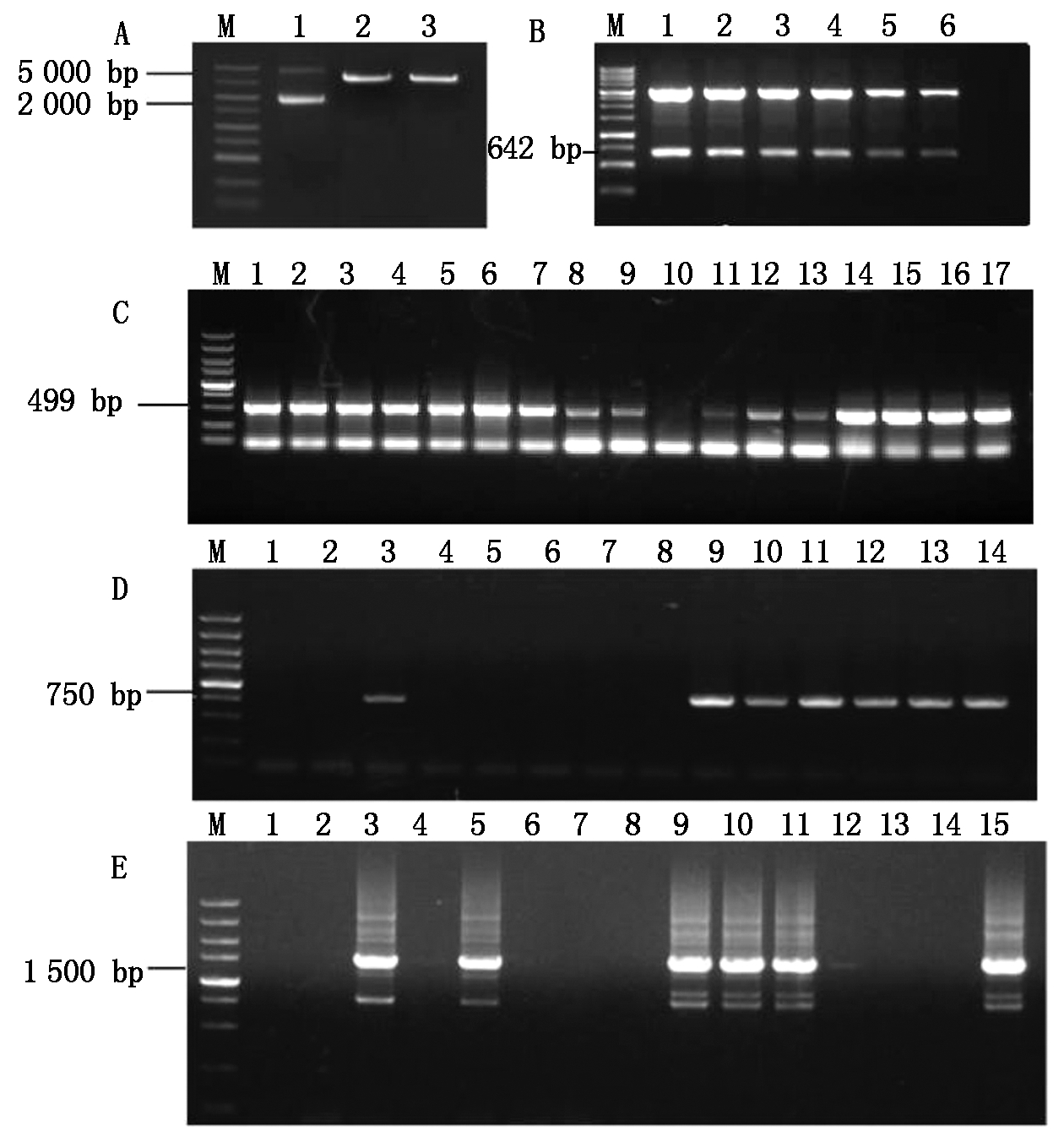
M.DL5000 DNA 标记(由大到小分别为5 000,3 000,1 500,1 000,750,500,250,100 bp);A.AtU6-26-sgRNA-SK 酶切琼脂糖凝胶电泳图;1.AtU6-26-sgRNA-SK质粒;2-3.AtU6-26-sgRNA-SK质粒酶切产物;B.AtU6-26-sgRNA-SK-靶点质粒双酶切琼脂糖凝胶电泳图;1-6.AtU6-26-sgRNA-SK-靶点质粒双酶切产物;C.AtU6-26-sgRNA-SK-靶点菌液PCR琼脂糖凝胶电泳图;1-17.AtU6-26-sgRNA-SK-靶点菌落1-17;D.pCAMBIA1300-DTH8-M1菌液PCR琼脂糖凝胶电泳图;1-14.pCAMBIA1300-DTH8-M1菌落1-14;E.pCAMBIA1300-DTH8-M1-DTH8-M2菌液PCR琼脂糖凝胶电泳图;1-15.pCAMBIA1300-DTH8-M1-DTH8-M2菌落1-15。
M.The DNA Marker of DL5000(from large to small, 5 000,3 000,1 500, 1 000, 750, 500, 250 and 100 bp);A.The agarose electrophoresis of digestion of AtU6-26-sgRNA-SK plasmid; 1.AtU6-26-sgRNA-SK plasmid; 2-3.AtU6-26-sgRNA-SK plasmid digestion products;B.The agarose electrophoresis of double-digestion of AtU6-26-sgRNA-SK-target spot plasmid; 1-6.AtU6-26-sgRNA-SK-target spot plasmid double enzyme digestion product;C.The AtU6-26-sgRNA-SK-target spot colony PCR agarose electrophoresis; 1-17.AtU6-26-sgRNA-SK-target colony 1-17;D.The pCAMBIA1300-DTH8-M1 colony PCR agarose electrophoresis; 1-14.pCAMBIA1300-DTH8-M1 colony 1-14;E.The pCAMBIA1300-DTH8-M1-DTH8-M2 colony PCR agarose electrophoresis; 1-15.pCAMBIA1300-DTH8-M1-DTH8-M2 colony 1-15.
图1 DTH8的CRISPR-Cas9表达载体构建
Fig.1 Construction of CRISPR-Cas9 expression vector of DTH8
挑菌后提质粒分别进行Nhe Ⅰ和Spe Ⅰ双酶切,回收大约为642 bp的片段(图1-B),得到DTH8-M1-sgRNA cassette、DTH8-M2-sgRNA cassette,然后分别与Spe Ⅰ单酶切后的pCAMBIA1300∶Cas9质粒连接,转化大肠杆菌后,菌落PCR检测电泳片段约为750 bp(图1-D),测序结果包含靶点DTH8-M1、DTH8-M2。选取正确的菌落过夜培养摇菌提取pCAMBIA1300-DTH8-M1、pCAMBIA1300-DTH8-M2质粒。基因编辑单靶点载体pCAMBIA1300-DTH8-M1、pCAMBIA1300-DTH8-M2 构建完成。命名为CRISPR-DTH8-1、CRISPR-DTH8-2。将DTH8-M2-sgRNA cassette与Spe Ⅰ单酶切后的pCAMBIA1300-DTH8-M1质粒连接,转化涂板过夜培养后挑取单克隆菌,PCR检测电泳片段约为1 500 bp(图1-E),测序结果包含靶点DTH8-M1和DTH8-M2。选取正确的菌落过夜培养摇菌提取pCAMBIA1300-DTH8-M1-DTH8-M2质粒。基因编辑双靶点载体构建完成,命名为CRISPR-DTH8-M1-DTH8-M2。
2.2 农杆菌介导的遗传转化及T0转基因植株检测
使用农杆菌介导的水稻遗传转化方法共获得30株再生植株,利用CTAB法提取水稻叶片DNA,用潮霉素鉴定Cas9载体元件是否整合到再生植株基因组上,经潮霉素筛选标记,特异性扩增的片段大小为557 bp,结果表明,在获得的30株T0再生苗中有29株能扩增出目的条带,说明这些植株均为基因编辑阳性植株,阳性转化率约为96.7%(图2)。
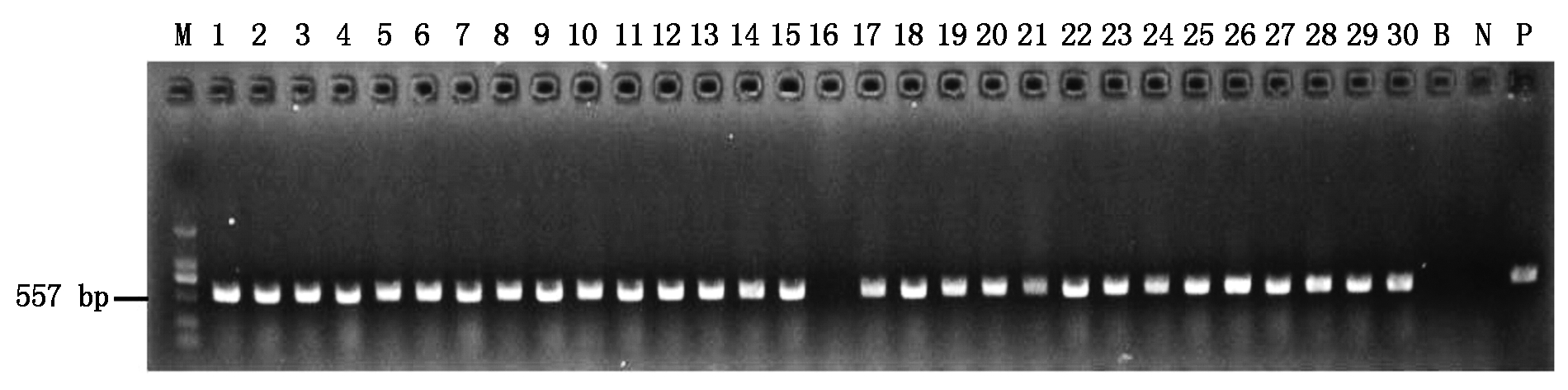
M.DL2000 DNA标记(由大到小分别为2 000,1 000,750,500,250,100 bp);Hpt引物对CRISPR-DTH8-1-DTH8-2转化植株进行PCR检测;B.空白对照;N.阴性对照;P.阳性对照。
M.DL2000DNA marker(from large to small, 2 000, 1 000, 750, 500, 250,100 bp, respectively);Hpt primer was used for PCR detection of CRISPR-DTH8-1-DTH8-2 transformed plants;B.Blank control; N.Negative control; P.Positive control.
图2 转化再生植株PCR检测
Fig.2 PCR detection of transformed and regenerated plants
2.3 靶位点测序结果及突变类型分析
利用设计好的特异性引物对阳性植株进行扩增,通过对PCR扩增产物的测序发现靶位点1附近未发生突变;靶位点2附近发生了2种类型的碱基置换和插入突变。通过刘耀光课题组的DSDecode解码方法(http:/cbi.hzau.edu.cn/crispr/),分析该突变体的具体碱基突变类型,编号为DTH8-5、DTH8-10、DTH8-12、DTH8-21结果显示在外显子第602号碱基G被C替换、603号碱基T被C替换、609号碱基G被T替换、610号碱基C被G替换、614号碱基A被C替换、615号碱基G被C替换、620号碱基G被T替换的纯合突变类型(图3);该类型突变导致靶位点编码蛋白的第201位氨基酸处形成甘氨酸→丙氨酸的置换;第203位氨基酸处形成谷氨酰胺→组氨酸的置换;第204位氨基酸处形成谷氨酰胺→谷氨酸的置换;第205位氨基酸处形成谷氨酰胺→脯氨酸的置换;第207位氨基酸处形成甘氨酸→缬氨酸的置换;DTH8-9、DTH8-17结果显示在外显子872号碱基G被A替换,导致第291位氨基酸由精氨酸→赖氨酸的置换。DTH8-2测序结果显示在外显子第671号碱基后面插入碱基AA,结果导致第234位氨基酸处发生提前终止(图3)。
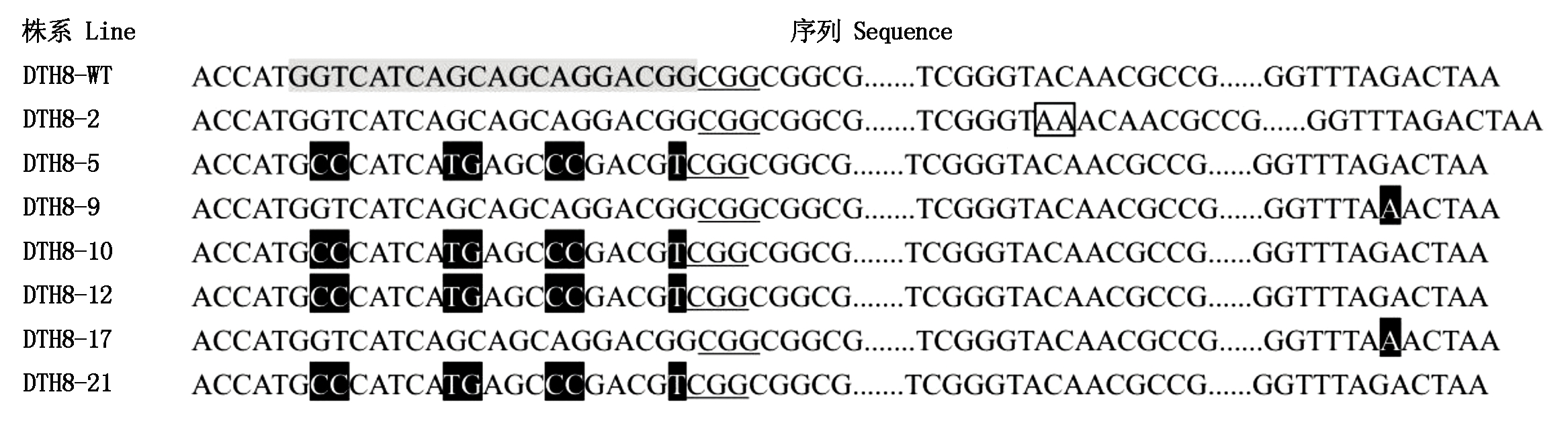
灰色字体.靶位点序列;下划线.候选识别位点的毗邻基序(PAM)序列;方框.插入碱基;黑色阴影.替换碱基;省略号.中间省略的碱基序列。
The grey letters.The target sequence;The underline.Protospacer adjacent motif(PAM)sequence; The box.The insert base;The black shadows.Replacement base;Ellipsis.Base sequence omitted in the middle.
图3 DTH8基因2种突变类型序列比对
Fig.3 Sequencing alignment of two mutation types of DTH8 gene
2.4 转基因T0表型及RNA表达水平分析
在T0获得转基因苗30株,其中阳性苗29株,编号分别为DTH8-1~15、DTH8-17~30。其中,有7份材料较野生型相比抽穗期提前,分别为DTH8-2、DTH8-5、DTH8-9、DTH8-10、DTH8-12、DTH8-17、DTH8-21(图4-A),其余材料均表现为正常表型。进一步研究了DTH8基因在T0的表达情况,对香糯99-25和7个突变体材料的抽穗期基因进行了RNA表达水平的检测。提取了8组材料抽穗期叶片总RNA,通过实时荧光定量PCR技术,定量分析了DTH8基因的表达情况,经统计分析发现,突变体材料中DTH8基因在RNA水平下表达量呈现出下降趋势(图4-B)。该结果有力地证明了DTH8基因外显子的碱基置换或插入影响了该基因的转录水平,导致该基因在突变体材料DTH8-2、DTH8-9和DTH8-17中表达显著下调。
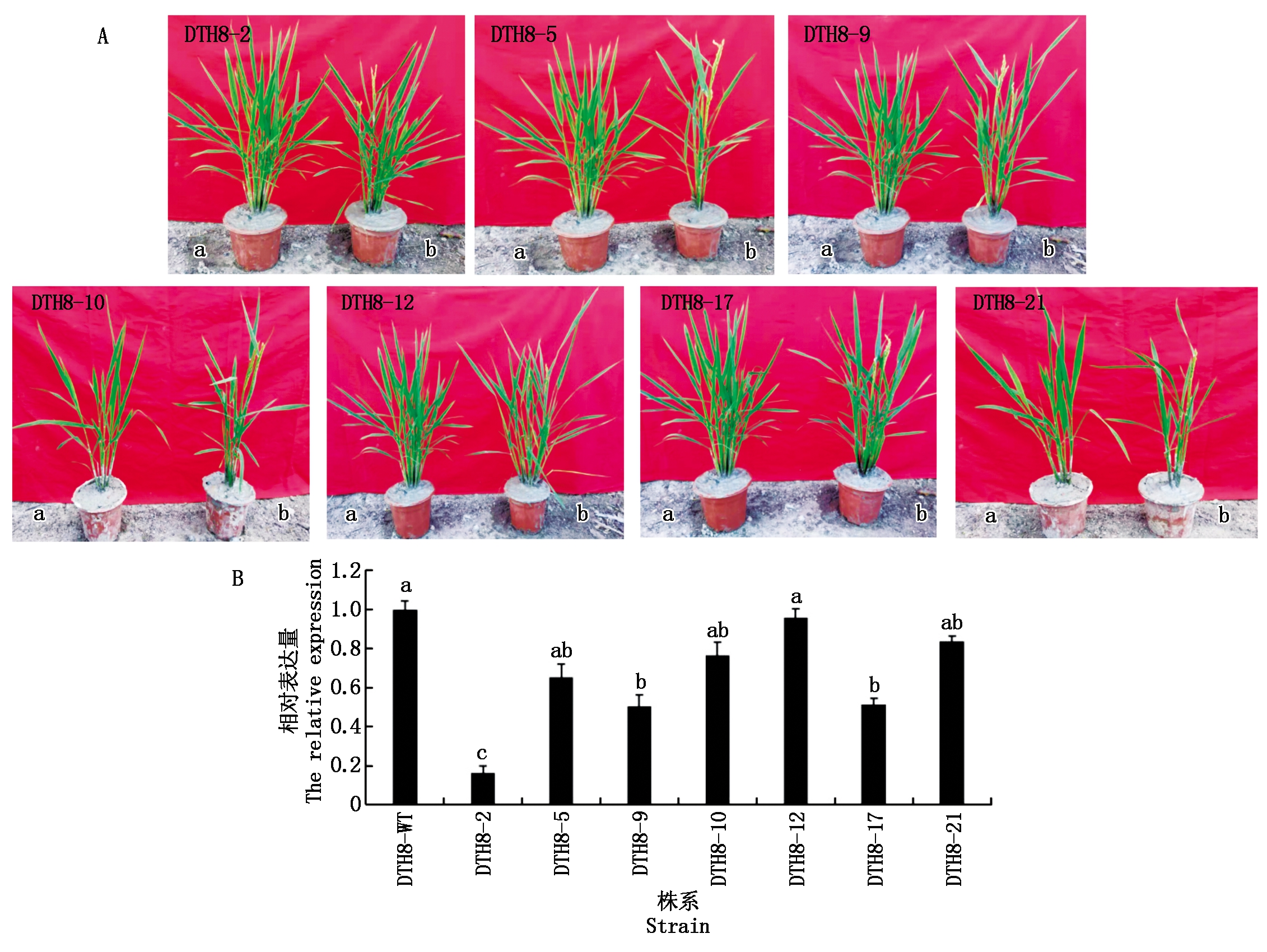
A.香糯99-25及DTH8突变体植株表现;a.WT;b.突变体;B.香糯99-25及DTH8突变体中DTH8 RNA水平。不同小写字母表示在0.05水平上差异显著(P<0.05)。图5同。
A.Plant expression of Xiangnuo 99-25 and DTH8 mutants;a.WT;b.Mutants;B.The levels of DTH8 RNA in Xiangnuo 99-25 and DTH8 mutant.Different lowercase letters indicate significant differences at the 0.05 level(P<0.05).The same as Fig.5.
图4 T0转基因部分株系的表型及RNA表达水平检测
Fig.4 Phenotype and RNA expression level of partial transgenic lines of T0 generation were detected
2.5 T1植株性状分析
在T1随机挑选了野生型材料和纯合的7个突变株系各20株,进一步对其抽穗期进行考察。经统计结果分析表明,DTH8基因成功编辑后代DTH8-2、DTH8-9、DTH8-17的抽穗期与野生型香糯99-25有显著性差异。以概率α=10-6为阈值,根据P≤10-6,结果表明,所调查的突变体植株抽穗期与对照存在显著差异;值得注意的是,在考察的野生型和7个突变株系的株高、每穗实粒数、千粒质量和穗长等性状中,7个突变株系的农艺性状与野生型基本一致,并经统计学分析发现,除抽穗期外,其他农艺性状与野生型相比均未达到显著水平(图5)。进一步证明了突变体抽穗期的变化是由于编辑抽穗期基因DTH8引起的。

图5 野生型与T1 DTH8突变体的农艺性状
Fig.5 Agronomic traits of wild type and T1-generation DTH8 mutants
3 结论与讨论
抽穗期作为决定水稻品种的地域种植和季节适应性的重要生理时期[24],近年来,受到育种工作者的关注。我国北方地区地处秦岭-淮河以北,内蒙古高原以南,大兴安岭、青藏高原以东,主要包括东北地区和华北地区,约占全国面积的20%,由于秋冬两季长日低温,在水稻育种过程中,不适宜种植抽穗期较长的水稻品种,同时早熟和高产一直是无法兼得的性状,早熟可能就要面对光合作用和物质积累时间长度不够的矛盾,因此,培育适宜在北方地区种植的提前抽穗的水稻品种具有重要意义。DTH8是一个控制水稻抽穗期的关键基因,据报道显示,该基因有4个等位基因,即Ghd8、LHD1、EF8和DTH8。本研究对抽穗期较晚的粳稻品种香糯99-25中的DTH8基因进行了定向编辑,获得了7个DTH8功能缺失的早抽穗突变体,与野生型香糯99-25相比,DTH8编码区的第602号碱基G被C替换、603号碱基T被C替换、609号碱基G被T替换、610号碱基C被G替换、614号碱基A被C替换、615号碱基G被C替换、620号碱基G被T替换。Zhang等[25]研究表明,利用CRISPR/Cas9技术对水稻进行编辑,在T0即可获得纯合的基因突变植株,并且这些突变能够稳定遗传给下一代。
CRISPR/Cas9系统作为一种DNA分子剪刀,是一种较为成熟的基因编辑技术,它能够精准地对基因组靶标部位进行定点编辑,彻底改变基因组序列,进而实现对目的基因的敲除和编辑。与传统的基因编辑技术TALEN和RNAi相比,该技术更加精准、操作简便快捷,同时具有多靶点突变且敲除效率高等优点。自2013年实现第一次应用后,目前已广泛应用于动植物、细菌、真菌基因功能研究中[7]。CRISPR/Cas9技术已经成功地应用到水稻[26-27]、玉米[28-29]、小麦[30]、大麦[31]、大豆[32]等多种作物中,并获得了基因功能缺失的突变体。陈炜等[33]利用此技术定点敲除水稻叶型相关基因NRL2,获得突变体耐旱基因表达水平显著低于野生型,进一步验证了NRL2影响植物耐旱性。本研究的抽穗期调查结果表明,CRISPR/Cas9技术编辑后获得的突变体均可促进抽穗。由此证明了CRISPR/Cas9系统能高效介导水稻基因组编辑修饰。目前,笔者正在将改良后的纯合突变体材料进行自交,进一步纯化其后代的遗传背景。随着CRISPR/Cas9基因编辑技术的不断完善,为定向改良某些水稻品种的主要农艺性状提供了一个高效、快速、便捷的分子育种方法,并在基因功能的研究、创建基因功能缺失突变体等方面发挥了重要作用。
[1] Yano M, Katayose Y, Ashikari M, Yamanouchi U, Monna L, Fuse T, Baba T, Yamamoto K, Umehara Y, Nagamura Y, Sasaki T.Hd1, a major photoperiod sensitivity quantitative trait locus in rice, is closely related to the Arabidopsis flowering time gene constans[J].The Plant Cell, 2001, 12(12):2473-2483.doi:10.1105/tpc.12.12.2473.
[2] Dai X D, Ding Y N, Tan L B, Fu Y C, Liu F X, Zhu Z F, Sun X Y, Sun X W, Gu P, Cai H W, Sun C Q.LHD1,an allele of DTH8/Ghd8, controls late heading date in common wild rice(Oryza rufipogon)[J].Journal of Integrative Plant Biology, 2012,54(10):790-799.doi:10.1111/j.1744-7909.2012.01166.x.
[3] Gao H, Jin M N, Zheng X M, Chen J, Yuan D Y, Xin Y Y, Wang M Q, Huang D Y, Zhang Z, Zhou K N, Sheng P K, Ma J, Ma W W, Deng H F, Jiang L, Liu S J, Wang H Y, Wu C Y, Yuan L P, Wan J M.Days to heading 7, a major quantitative locus determining photoperiod sensitivity and adaptation in rice[J].PNAS,2014,111(46):16377-16342.doi:10.1073/pnas.1418204111.
[4] Yan W H, Liu H Y, Zhou X C, Li Q P, Zhang J, Lu L, Liu T M, Liu H J, Zhang C J,Zhang Z Y, Shen G J, Yao W, Chen H X, Yu S B, Xie W B, Xing Y Z.Natural variation in Ghd7.1 plays an important role in grain yield and adaptation in rice[J].Cell Research,2013,23(7):969-971.doi:10.1038/cr.2013.43.
[5] Yan W H, Wang P, Chen H X, Zhou H J, Li Q P, Wang C R, Ding Z H, Zhang Y S, Yu S B, Xing Y Z, Zhang Q F.A major QTL, Ghd8, plays pleiotropic roles in regulating grain productivity, plant height, and heading date in rice[J].Molecular Plant,2011,4(2):319-330.doi:10.1093/mp/ssq070.
[6] Wei X J, Xu J F, Guo H N, Jiang L, Chen S H, Yu C Y, Zhou Z L, Hu P S, Zhai H Q, Wan J M.DTH8 suppresses flowering in rice, influencing plant height and yield potential simultaneously[J].Plant Physiology,2010,153(4):1747-1758.doi:10.1104/pp.110.156943.
[7] 何先畅,黄国强,王道洋,常淑伟,张大兵.利用CRISPR-Cas9基因编辑技术获得水稻OsYUCCA1基因突变体[J].基因组学与应用生物学,2017,36(11):4778-4784.doi:10.13417/j.gab.036.004778.
He X Y, Huang G Q, Wang D Y, Chang S W, Zhang D B.Generation of the mutations for OsYUCCA1 in rice using CRISPR/Cas9 approach[J].Genomic and Applied Biology, 2017,36(11):4778-4784.
[8] Cong L, Ran F A, Cox D, Lin S L, Barretto R, Habib N,Hsu P D, Wu X B, Jiang W Y,Marraffini L A, Zhang F.Multiplex genome engineering using CRISPR/Cas systems[J].Science,2013,339(6121):819-823.doi:10.1126/science.1231143.
[9] DiCarlo J E, Norville J E, Mail P, Rios X, Aach J, Church G M.Genome engineering in Saccharomyces cere visiae using CRISPR-Cas systems[J].Nucleic Acids Research, 2013, 41(7):4336-4343.doi:10.1093/nar/gkt135.
[10] Mali P, Yang L H, Esvelt K M, Aach J, Guell M, DiCarlo J E, Norville J E, Church G M.RNA-Guided human genome engineering via Cas9[J].Science,2013,339(6121):823-826.doi:10.1126/science.1232033.
[11] Wang H Y, Yang H, Shivalila C S, Dawlaty M M, Cheng A W, Zhang F, Jaenisch R.One-Step generation of mice carrying mutations in multiple genes by CRISPR/Cas-Mediated genome engineering[J].Cell, 2013,153(4):910-918.doi:10.1016/j.cell.2013.04.025.
[12] Feng Z Y, Zhang B T, Ding W N, Liu X D, Yang D L, Wei P L, Cao F Q, Zhu S H, Zhang F, Mao Y F, Zhu J K.Efficient genome editing in plants using a CRISPR/Cas system[J].Cell Research,2013,23(10):1229-1232.doi:10.1038/cr.2013.114.
[13] Jiang W Y, Bikard D, Cox D, Zhang F, Marraffini L A.RNA-guided editing of bacterial genomes using CRISPR-Cas systems[J].Nature Biotechnology,2013,31(3):233-239.doi:10.1038/nbt.2508.
[14] Bortesi L, Fischer R.The CRISPR/Cas9 system for plant genome editing and beyond[J].Biotechnology Advances,2015,33(1):41-52.doi:10.1016/j.biotechadv.2014.12.006.
[15] Ma X L, Zhang Q Y, Zhu Q L, Liu W, Chen Y, Qiu R, Wang B, Yang Z F, Li H Y, Lin Y R, Xie Y Y, Shen R X,Chen S F, Wang Z, Chen Y L, Guo J X, Chen L T, Zhao X C, Dong Z C, Liu Y G.A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants[J].Molecular Plant, 2015, 8(8):1274-1284.doi:10.1016/j.molp.2015.04.007.
[16] Xu R F, Li H, Qin R Y, Li J, Qiu C H, Yang Y C, Ma H, Li L, Wei P C, Yang J B.Generation of inheritable and ′transgene clean′ targeted genome-modified rice in later generations using the CRISPR/Cas9 system[J].Scientific Reports,2015,5:11491.doi:10.1038/srep11491.
[17] 陈勇龙,黄华荣,唐平平,羊雪芹,李斐雪,许杰,张遵义.基因组编辑技术——CRISPR/Cas系统[J].杭州师范大学学报(自然科学版),2015,14(1):60-65.doi:10.3969/j.issn.1674-232X.2015.01.011.
Chen Y L, Huang H R, Tang P P, Yang X Q, Li F X, Xu J, Zhang Z Y.Genome editing techniques-CRISPR/Cas system[J].Journal of Hangzhou Normal University(Natural Science Edition),2015,14(1):60-65.
[18] Grissa I,Vergnaud G,Pourcel C.The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats[J].BMC Bioinformatics, 2007, 8(1):172.doi:10.1186/1471-2105-8-172.
[19] Jinek M, Jiang F G, Taylor D W, Sternberg S H, Kaya E, Ma E, Anders C, Hauer M, Zhou K H, Lin S, Kaplan M, Iavarone A T, Charpentier E, Nogales E, Doudna J A.Structures of Cas9 endonucleases reveal RNA-Mediated conformational activation[J].Science,2014,343(6176):1247997.doi:10.1126/science.1247997.
[20] 蔡春苗.光(温)敏雄性核不育水稻MS8S不育基因和抽穗期基因的遗传分析与分子定位[D].福州:福建师范大学,2008.doi:10.7666/d.y1332640.
Cai C M.Genetic analysis and molecular mapping of photoperiod(themo)-sensitive genic male sterile and heading-time gene in rice MS8S[D].Fuzhou: Fujian Normal University,2008.
[21] 叶世伟,方芳,梁婉琪.利用CRISPR-Cas9基因编辑技术获得水稻OsWOX9C基因突变体[J].分子植物育种,2018,16(15):4921-4928.doi:10.13271/j.mpb.016.004921.
Ye S W, Fang F, Liang W Q.Obtain of OsWOX9C gene mutant in rice using CRISPR-Cas9 gene editing technology[J].Molecular Plant Breeding,2018,16(15):4921-4928.
[22] 李荣华,夏岩石,刘顺枝,孙莉丽,郭培国,缪绅裕,陈健辉.改进的CTAB提取植物DNA方法[J].实验室研究与探索,2009,28(9):14-16.doi:10.3969/j.issn.1006-7167.2009.09.005.
Li R H, Xia Y S, Liu S Z, Sun L L, Guo P G, Liao S Y, Chen J H.CTAB-improved method of DNA extraction in plant[J].Research and Exploration in Laboratory,2009,28(9):14-16.
[23] 孙德权,郭启高,胡玉林,谢江辉.改良TRIzol法提取香蕉叶片总RNA[J].广东农业科学,2009(5):162-164.
Sun D Q, Guo Q G, Hu Y L, Xie J H.Extraction of total RNA from banana with improved TRIzol method[J].Guangdong Agricultural Sciences,2009(5):162-164.
[24] 李允振,黄永禄,谢旭阳,李志华,艾海粤,刘芳,邱永福,罗继景,李容柏,覃宝祥.水稻抽穗期基因EHD8的遗传分析及精细定位[J].中国科技论文,2017,12(12):1336-1340.doi:10.3969/j.issn.2095-2783.2017.12.003.
Li Y Z, Huang Y L, Xie X Y, Li Z H, Ai H Y, Liu F, Qiu Y F, Luo J J, Li R B, Qin B X.Genetic analysis and fine-mapping of a rice heading date gene EHD8[J].China Sciencepaper,2017,12(12):1336-1340.
[25] Zhang H, Zhang J S, Wei P L, Zhang B T, Gou F, Feng Z Y, Mao Y F, Yang L, Zhang H, Xu N F, Zhu J K.The CRISPR/Cas9 system produces specific and homozygous targeted gene editing in rice in one generation[J].Plant Biotechnology Journal, 2014, 12(6):797-807.doi:10.1111/pbi.12200.
[26] Shen L, Wang C, Fu Y P, Wang J J, Liu Q, Zhang X M, Yan C J, Qian Q, Wang K J.QTL editing confers opposing yield performance in different rice varieties[J].Journal of Integrative Plant Biology,2018,60(2):89-93.doi:10.1111/jipb.12501.
[27] Li M R, Li X X, Zhou Z J, Wu P Z, Fang M C, Pan X P, Lin Q P, Luo W B, Wu G J, Li H Q.Reassessment of the four yield-related genes Gn1a, DEP1, GS3, and IPA1 in rice using a CRISPR/Cas9 system[J].Frontiers in Plant Science,2016,7:377.doi:10.3389/fpls.2016.00377.
[28] Feng C, Yuan J, Wang R, Liu Y, Birchler J A, Han F P.Efficient targeted genome modification in maize using CRISPR/Cas9 system[J].Journal of Genetics and Genomics,2016,43(1):37-43.doi:10.1016/j.jgg.2015.10.002.
[29] Svitashev S, Young J K, Schwartz C, Gao H R, Falco S C, Cigan A M.Targeted mutagenesis, precise gene editing, and site-specific gene insertion in maize using Cas9 and guide RNA[J].Plant Physiology,2015,169(2):931-945.doi:10.1104/pp.15.00793.
[30] Shan Q W, Wang Y P, Li J, Gao C X.Genome editing in rice and wheat using the CRISPR/Cas system[J].Nature Protocols,2014,9(10):2395-2410.doi:10.1038/nprot.2014.157.
[31] Lawrenson T, Shorinola O, Stacey N, Li C D, Østergaard L, Patron N, Uauy C, Harwood W.Induction of targeted, heritable mutations in barley and Brassica oleracea using RNA-guided Cas9 nuclease[J].Genome Biology,2015,16(1):258.doi:10.1186/s13059-015-0826-7.
[32] Jacobs T B, LaFayette P R, Schmitz R J, Parrott W A.Targeted genome modifications in soybean with CRISPR/Cas9[J].BMC Biotechnology,2015,15(1):16.doi:10.1186/S12896-015-0131-2.
[33] 陈炜,邱牡丹,李潜龙,王建龙.基于CRISPR/Cas9研究叶形相关基因NRL2对水稻抗旱性的影响[J].分子植物育种,2018,16(22):7298-7303.doi:10.13271/j.mpb.016.007298.
Chen W, Qiu M D, Li Q L, Wang J L.Effect of leaf-shape related gene NRL2 on drought resistance of rice based on CRISPR/Cas9[J].Molecular Plant Breeding,2018,16(22):7298-7303.