实时荧光定量PCR(qRT-PCR)是研究基因表达的一项重要技术,通过对内参基因的检测从而达到对目的基因表达情况的实时监测[1]。qRT-PCR技术具有灵敏度高、特异性强和重复性好等优点,得到了广泛应用。但该技术对样品RNA的质量、反转录的效率、内参基因选择及引物特异性等都有着非常严格的要求[2]。其中,qRT-PCR分析结果准确可靠的前提是合适内参基因的选择。常用的内参基因包括EF1α(Elongation factor 1 alpha)、Actin(Actin-7)、GAPDH(Glyceraldehyde-3-phosphate dehydrogenase)、PP2A(Protein phosphatase 2A)、TIP41(TIP41-like family protein)、UPL7(E3 ubiquitin-protein ligase UPL7)、PEPKR1(Phosphoenolpyruvate carboxylase-related Kinase 1)、SAND(SAND family protein)和TUB(Tubulin)等[3-4]。理想的内参基因在植物不同生长发育阶段、不同组织中均能稳定表达,不受环境影响[5]。但研究表明,在不同植物中、不同时间及处理条件下,内参基因表达稳定性存在较大差异,如Actin在侧柏各组织中有较好的稳定性表达[6],但其表达稳定性在柚木各组织中却较差[7];18S rRNA在黄山栾树不同组织中表达稳定性较好[8],但在珙桐各器官中表达稳定性较差[9]。如直接使用未经筛选的内参基因,会导致试验数据出现偏差,影响目的基因表达水平的分析[10]。因此,为了确保qRT-PCR分析结果可靠准确,针对不同植物、组织、生长发育阶段及试验条件等因素,应对内参基因表达稳定性进行评价从而筛选出适宜的内参基因。
茉莉花(Jasminum sambac(L.)Aiton)又名茉莉、茶叶花,为木樨科(Oleaceae)素馨属常绿灌木。茉莉花原产于印度、巴基斯坦等地,在1 700多年前的汉代传入我国,最早在东南沿海地区栽培,然后渐次北上直到长江流域,现主要分布在广西、福建、四川、广东等地,是“一路一带”经济植物开发与应用的重要成员之一[11]。茉莉花具有较高的观赏、茶用和药用价值,历来受到我国人民的喜爱,被誉为“天下第一香”、“花树中的珍品”[12]。茉莉花因馥郁及优雅的香气而被称为“精油之王”,广泛用于花茶和香精加工产业。目前,对茉莉花分子生物学方面的研究日益增多,基因表达分析已被应用于茉莉花香气基因调控网络的研究中[13],如JsDXR和JsTPS在花发育不同阶段及花不同部位的表达水平[14]、α-法呢烯合成相关的HMGS、HMGR、FPPS、TPS基因在茉莉花不同花期的表达情况的研究[15]等。研究茉莉花香气合成代谢通路中的关键基因,明确其调控分子机制将有助于对香气成分进行调控。茉莉花基因表达的研究必须要有合适的内参基因作为参照,但目前关于茉莉花内参基因的研究尚未见报道。因此,本研究选取8个常用候选内参基因(EF1α、GAPDH、Actin、PP2A、TIP41、UPL7、PEPKR1和SAND),研究其在茉莉花不同组织和花发育不同时期的表达水平,并借助geNorm、NormFinder、BestKeeper和RefFinder软件对这些内参基因表达的稳定性进行评价,进一步利用目的基因对筛选获得的内参基因的稳定性和可靠性进行验证。本研究结果为后续茉莉花基因表达、功能及调控等相关研究奠定基础。
1 材料和方法
1.1 试验材料
供试材料为多年生双瓣茉莉花,种植于江苏省农业科学院茉莉花种质资源圃。不同组织样品分别取自根、茎、嫩叶、成熟叶和花;花发育不同阶段样品取样参照Deng等[16]分别取阶段Ⅰ(幼蕾期)、阶段Ⅲ(膨大花蕾期)、阶段Ⅴ(最长花蕾期)和阶段Ⅵ(初绽期)4个时期。每个样品设置3个生物学重复,液氮速冻后置-80 ℃冰箱保存。
1.2 试验方法
1.2.1 RNA提取及cDNA合成 用RNA提取试剂盒(TaKaRa, 日本)提取样品总RNA。总RNA样品用DNaseⅠ(TaKaRa, 日本)消化去除基因组DNA污染。利用ND-1000 spectrophotometer(NanoDrop, Wilmington,DE)和1.0%的琼脂糖凝胶电泳检测总RNA浓度、纯度和完整性。取各样品1 000 ng的RNA为模板,按照PrimeScript 1st strand cDNA synthesis kit(TaKaRa, 日本)的操作方法反转录合成cDNA,cDNA置于-20 ℃保存。
1.2.2 候选内参基因选择及引物设计 根据绣球与宿根花卉研究室前期茉莉花转录组数据,选择较常见的8个候选内参基因:EF1α、GAPDH、Actin、PP2A、TIP41、UPL7、PEPKR1和SAND。根据候选内参基因序列及引物设计原则,运用软件Primer Premier 5.0设计内参基因的引物(表1),并由南京思普金生物科技有限公司合成。
表1 茉莉花候选内参基因引物序列
Tab.1 Sequence of the primers for candidate reference genes in J.sambac
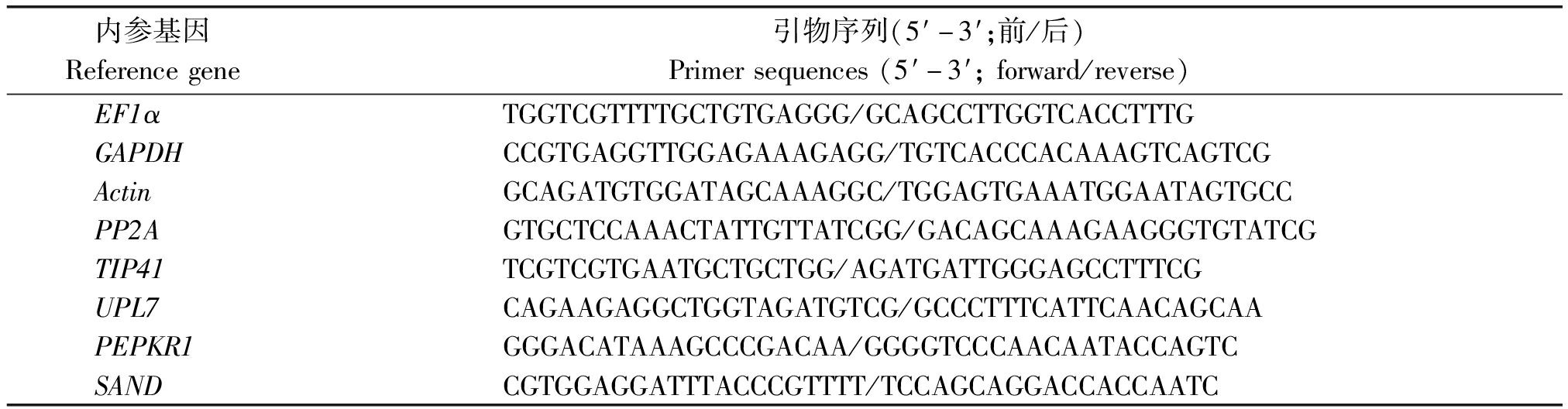
内参基因Referencegene引物序列(5′-3′;前/后)Primersequences(5′-3′;forward/reverse)EF1αTGGTCGTTTTGCTGTGAGGG/GCAGCCTTGGTCACCTTTGGAPDHCCGTGAGGTTGGAGAAAGAGG/TGTCACCCACAAAGTCAGTCGActinGCAGATGTGGATAGCAAAGGC/TGGAGTGAAATGGAATAGTGCCPP2AGTGCTCCAAACTATTGTTATCGG/GACAGCAAAGAAGGGTGTATCGTIP41TCGTCGTGAATGCTGCTGG/AGATGATTGGGAGCCTTTCGUPL7CAGAAGAGGCTGGTAGATGTCG/GCCCTTTCATTCAACAGCAAPEPKR1GGGACATAAAGCCCGACAA/GGGGTCCCAACAATACCAGTCSANDCGTGGAGGATTTACCCGTTTT/TCCAGCAGGACCACCAATC
1.2.3 候选内参基因引物特异性分析 用内参基因qRT-PCR引物,以茉莉花各样品cDNA等量混合为模板进行PCR扩增。PCR反应的体系为25.0 μL:cDNA模板1.0 μg、上下游引物(10 μmol/L)各1.0 μL、TaKaRa Taq(5 U/μL)0.2 μL、10× PCR Buffer 2.5 μL、dNTP(2.5 mmol/L each)2.0 μL,加水至25.0 μL。PCR反应条件为:94 ℃预变性5 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸30 s,35个循环;72 ℃延伸7 min。通过2%的琼脂糖凝胶电泳对引物的特异性进行检测。
1.2.4 内参基因的qRT-PCR分析 qRT-PCR扩增采用TaKaRa公司的TB Green Premix Ex Taq(Tli RNaseH Plus)试剂盒,具体操作按说明书进行。cDNA稀释10倍作为模板,反应体系20 μL:cDNA模板2.0 μL、TB Green Premix Ex Taq(Tli RNaseH Plus)(2×)10 μL、上下游引物(10 μmol/L)各0.4 μL、ROX Reference Dye Ⅱ(50×)0.4 μL、ddH2O 6.8 μL。反应在实时荧光定量PCR仪ABI 7500上进行,反应程序:95 ℃预变性30 s;95 ℃变性5 s,60 ℃退火34 s,40个循环;95 ℃变性15 s,60 ℃退火1 min,95 ℃变性15 s。每个样品设3个生物学重复和3次技术重复。
1.2.5 数据处理和分析 qRT-PCR后得出Ct值,分别利用geNorm[17-18]、NormFinder[19]和BestKeeper[20]软件对Ct值进行分析,对8个内参基因表达的稳定性进行比较,并从中选择最适内参基因。使用RefFinder程序对以上3种软件的分析结果进行综合分析,选出最适于茉莉花不同组织和花发育不同阶段的内参基因[21]。
1.2.6 内参基因稳定性验证 通过对茉莉花花香气形成过程中的5-磷酸脱氧木酮糖合成酶(DXS,Deoxyoxylulose-5-phosphate synthase)基因JsDXS和苯丙氨酸解氨酶(PAL,phenylalanine ammonia lyase)基因JsPAL2在花发育不同阶段的表达模式进行分析,从而对筛选的内参基因稳定性和可靠性进行验证。基因JsDXS和JsPAL2 qRT-PCR分析的引物序列分别参考孙君等[22]、熊青等[23]文献报道中的引物序列。
2 结果与分析
2.1 总RNA质量检测及引物特异性分析
NanoDrop检测各样品总RNA的OD260/280均在1.95~2.13,OD260/230均大于2.0,说明总RNA的纯度较好。通过1.0%的琼脂糖凝胶电泳检测样品总RNA完整性,所有样品28S rRNA和18S rRNA条带清晰完整,说明总RNA的完整性较好,达到后续试验的需求。
以各样品cDNA等量混合为模板,对8个内参基因进行PCR扩增。凝胶电泳结果显示,8个候选内参基因均扩增出单一条带,无非特异扩增及引物二聚体,且扩增条带大小符合预期(图1),说明设计的引物特异性好,可用于qRT-PCR试验。
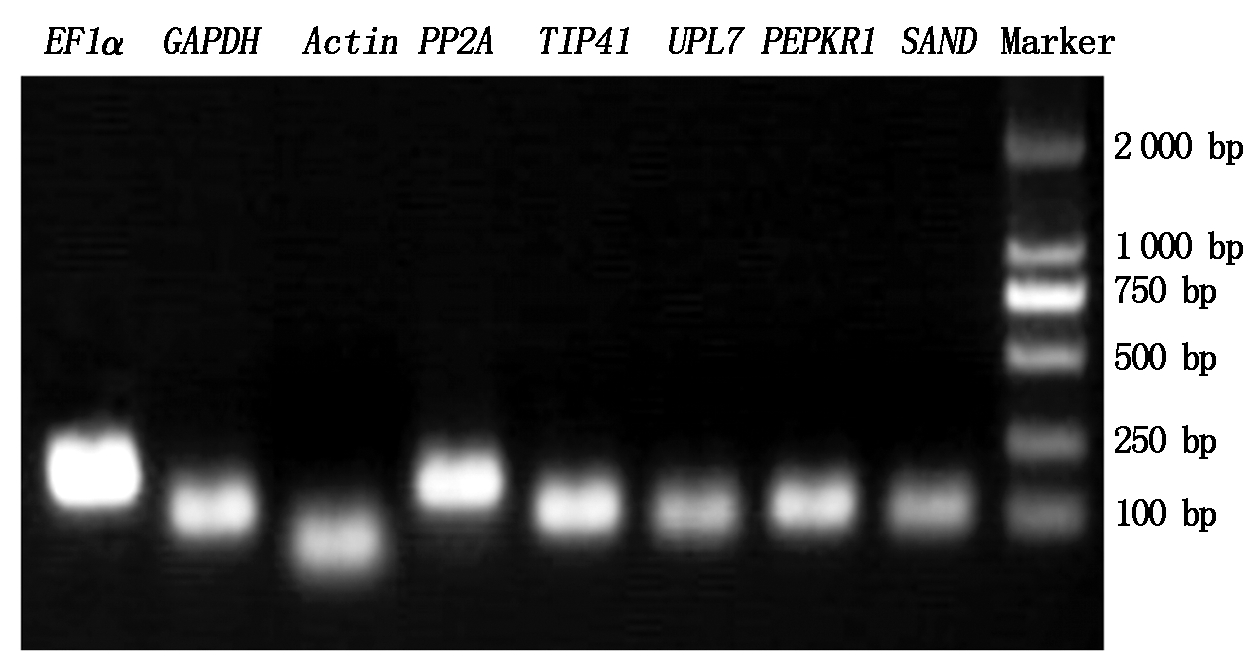
图1 茉莉花8个候选内参基因PCR产物
Fig.1 PCR products of the eight candidate reference genes in J.sambac
将各样品cDNA等量混合后,稀释10倍作为模板,分别用8个候选内参基因引物进行qRT-PCR扩增,结果如图2所示。各候选内参基因溶解曲线均只有明显的单一峰,说明8个候选内参基因引物扩增特异性强,符合qRT-PCR试验要求,可用于后续候选内参基因稳定性的分析。
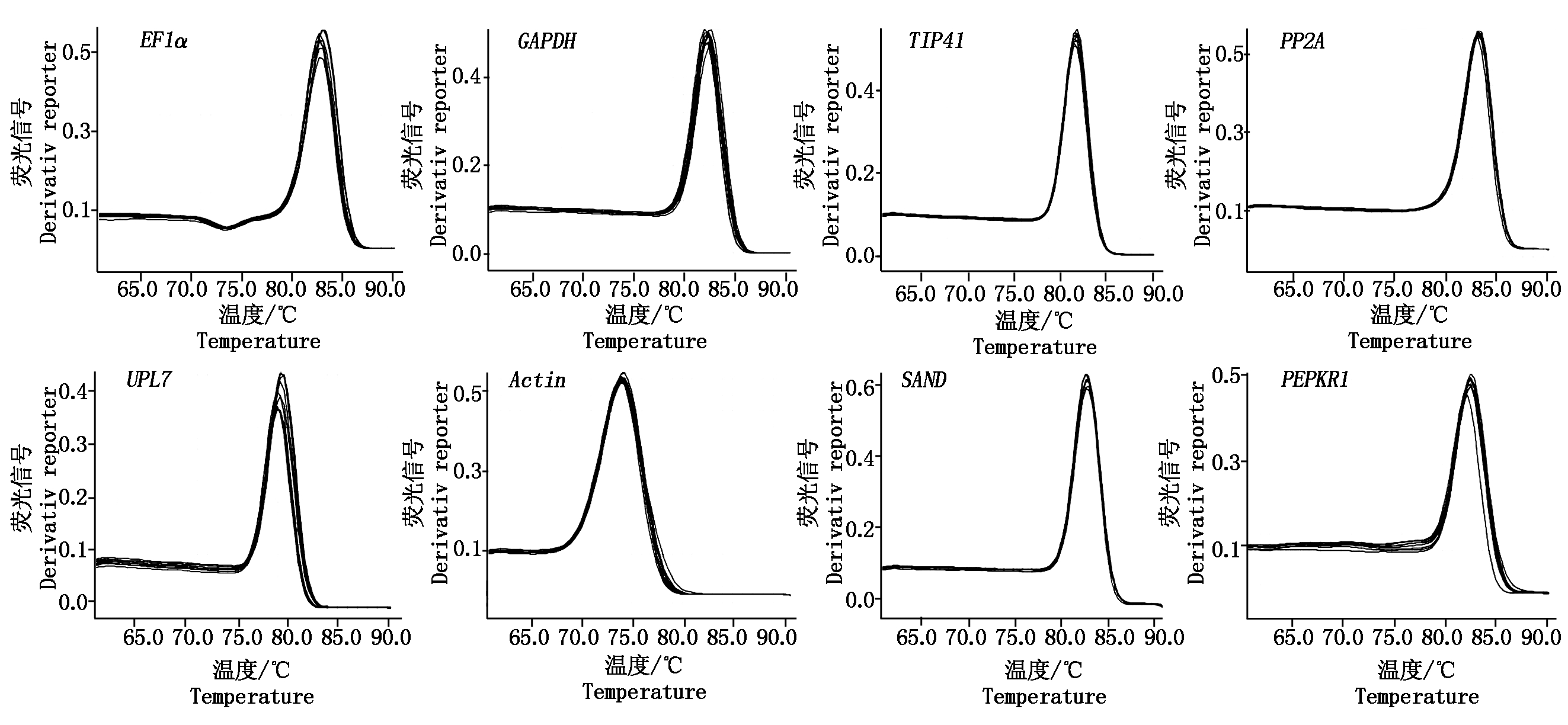
图2 茉莉花8个候选内参基因qRT-PCR溶解曲线
Fig.2 qRT-PCR melting curves of the eight candidate reference genes in J.sambac
2.2 候选内参基因表达分析
分别以茉莉花不同组织和花发育不同阶段cDNA为模板,进行qRT-PCR分析。结果如图3所示,8个候选内参基因在茉莉花不同样品中的Ct值均有一定的变化,变化的趋势各有不同。EF1α在不同样品的Ct值在19.09~23.27,GAPDH的Ct值在17.89~31.26,Actin的Ct值27.88~29.20,PP2A的Ct值在22.16~34.03,TIP41的Ct值在22.31~33.51,UPL7的Ct值在23.47~31.39,PEPKR1的Ct值在26.96~29.98,SAND的Ct值在25.54~31.20。除Actin外,其他所有候选内参基因在花中的Ct值均高于其他组织,即在花中的表达量均最低。因各候选内参基因的Ct值(除Actin)在花与其他组织中的差异较大,对后期结果影响较大,故在候选内参基因稳定性评价中,将不同组织分为“有花组”(包含根、茎、嫩叶、成熟叶、花)和“无花组”(包含根、茎、嫩叶、成熟叶)进行分析。8个候选内参基因平均Ct值为21.53~28.68,其中EF1α的Ct值最小,说明其表达丰度较高(图4)。
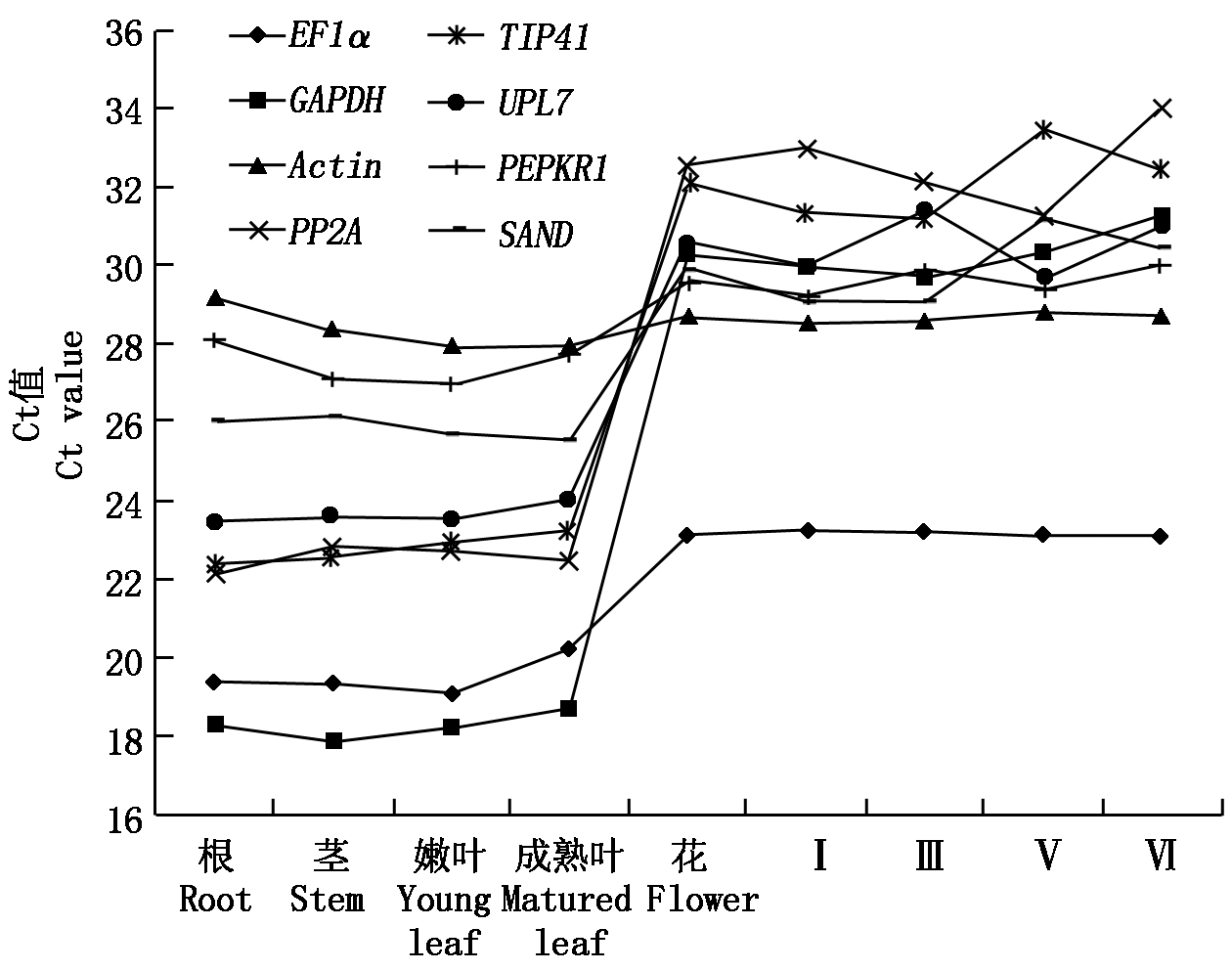
Ⅰ.幼蕾期;Ⅲ.膨大花蕾期;Ⅴ.最长花蕾期;Ⅵ.初绽期。图7同。
Ⅰ.Young floral bud; Ⅲ.Swollen floral bud; Ⅴ.Longest floral bud; Ⅵ.Initial opening flower.The same as Fig.7.
图3 茉莉花不同样品中候选内参基因的平均Ct值
Fig.3 Average Ct values of candidate reference genes in various samples in J.sambac
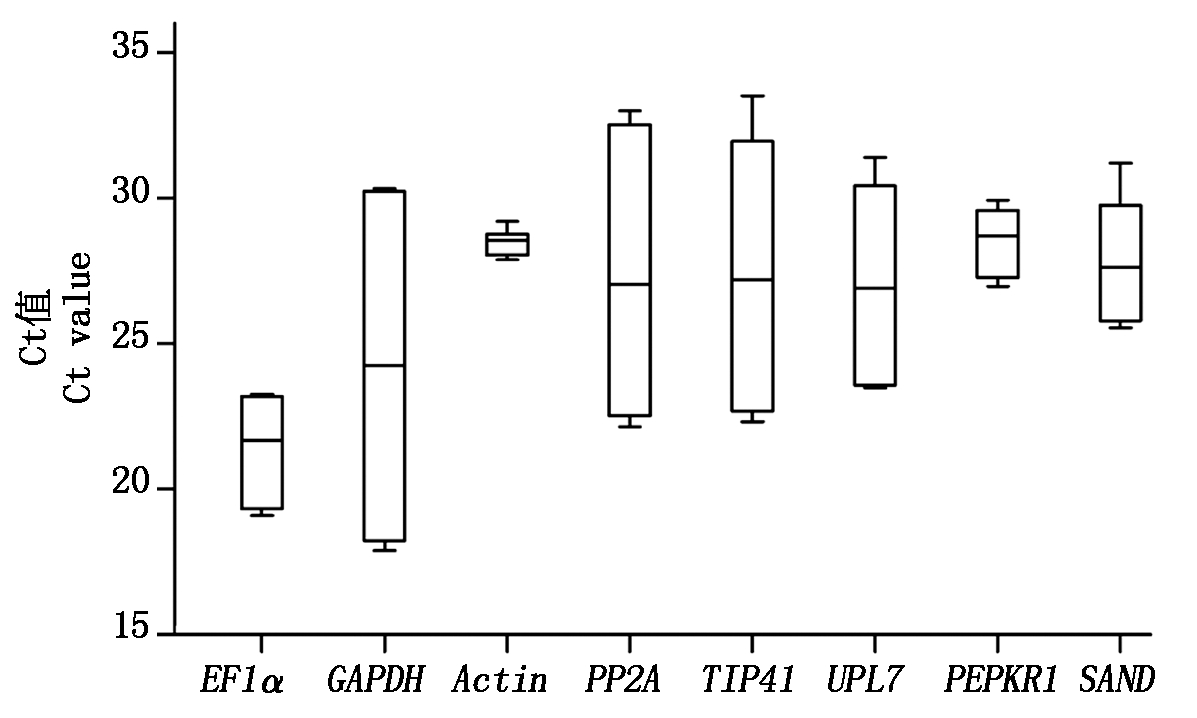
箱体内的线表示中位数;箱体下、上边分别表示25%,75%的百分位数;箱体上、下竖线分别表示最大值与最小值。
Lines in boxes represent median values;Lower and upper boxes show the 25th percentile to the 75th percentile; Whisker caps represent the maximum and minimum values.
图4 茉莉花不同样品中候选内参基因的Ct值
Fig.4 Ct values of candidate reference genes in various samples in J.sambac
2.3 候选内参基因表达的稳定性分析
2.3.1 geNorm分析 geNorm软件依据计算的每个基因表达稳定性M值来进行排序,M值越小稳定性越好,反之则稳定性越差,M<1.5时认为是理想内参。geNorm软件分析结果显示,在有花组中,EF1α、SAND、PEPKR1、UPL7的M值小于1.5,说明这些内参基因表达稳定性较好,表达的稳定性由高到低排序依次为:EF1α=SAND>PEPKR1>UPL7>TIP41>PP2A>Actin>GAPDH(图5-A);在无花组中,各候选内参基因M值均小于1.5,说明8个内参基因均具有良好的稳定性,表达稳定性由高到低排序依次为:GAPDH=UPL7>TIP41>EF1α>PP2A>SAND>PEPKR1>Actin(图5-B);在花发育不同阶段,各基因M值均小于1.5,说明8个内参基因均具有良好的稳定性,表达稳定性由高到低排序依次为:EF1α=Actin>PEPKR1>GAPDH>SAND>TIP41>UPL7>PP2A(图5-C)。
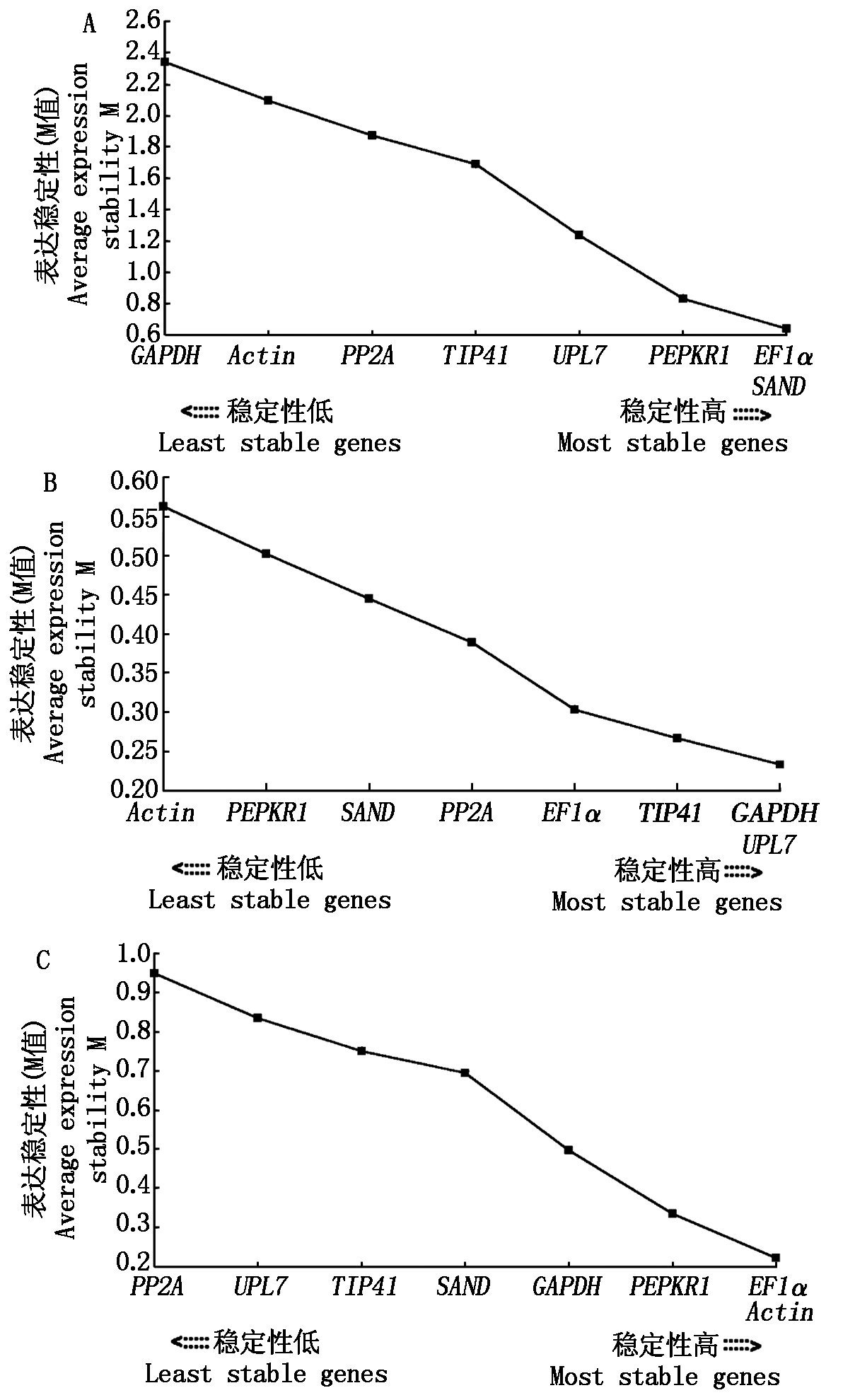
A.不同组织(有花组);B.不同组织(无花组);C.花发育不同阶段。图6同。
A.Different tissues(with flowers); B.Different tissues(without flowers); C.Flower samples of different developmental stages.The same as Fig.6.
图5 geNorm分析候选内参基因表达的稳定性
Fig.5 Analysis of the expression stability of candidate reference genes by geNorm
geNorm通过对配对差异分析(Vn/Vn+1)得出最佳内参基因个数,软件默认的Vn/Vn+1阈值为0.15,Vn/Vn+1比值小于0.15时,最适内参基因为n个;大于0.15时,最适内参基因为n+1个。对候选内参基因的配对差异值分析发现,在有花组中,Vn/Vn+1值均大于0.15,此时不能依靠阈值(0.15)来决定最适内参基因的个数,评估目的基因在茉莉花不同组织中的表达水平时,选取2~3个稳定性强的内参基因即可(图6-A);在无花组中,V2/V3=0.087<0.15,说明最适内参基因个数为2(图6-B);在花发育不同阶段,V2/V3=0.126<0.15,说明采用2个内参基因即可(图6-C)。
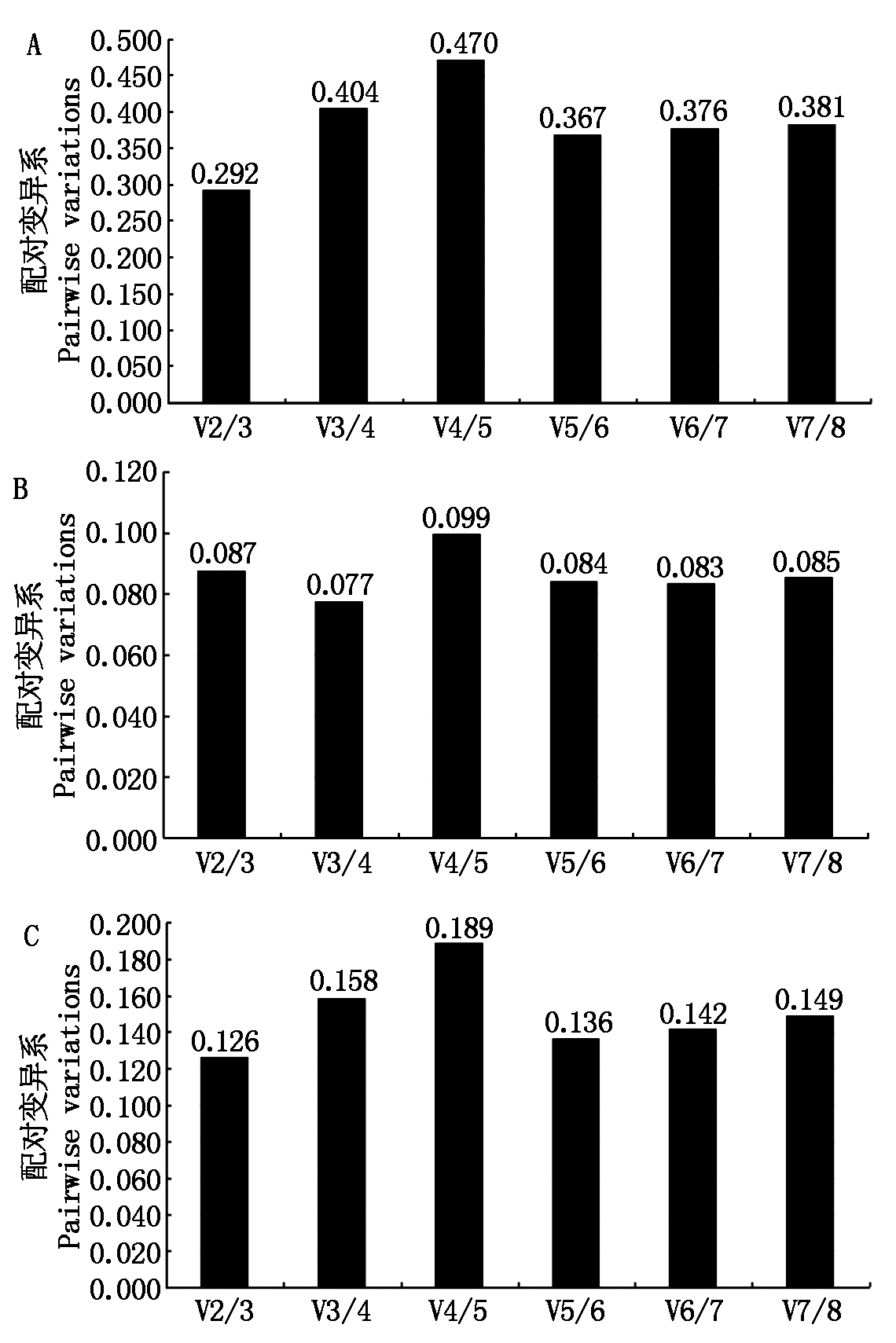
图6 geNorm分析确定候选内参基因数目
Fig.6 Determined the optimal number of candidate reference genes by geNorm
2.3.2 NormFinder分析 NormFinder软件通过方差分析对候选内参基因进行筛选,利用ΔCt法计算候选内参基因表达稳定值(S),从而对内参基因表达的稳定性进行评价,S值越小,基因越稳定。NormFinder分析结果显示,在有花组中,UPL7(S=0.172)和SAND(S=0.221)表达最稳定,GAPDH(S=0.898)表达最不稳定;在无花组中,UPL7(S=0.037)和GAPDH(S=0.072)表达最稳定,Actin(S=0.201)表达最不稳定;在花发育不同阶段,Actin(S=0.036)、PEPKR1(S=0.077)和GAPDH(S=0.077)表达最稳定,PP2A(S=0.350)表达最不稳定;是否有花,对GAPDH的稳定性排名有重要影响(表2)。
表2 NormFinder分析候选内参基因表达稳定性排名
Tab.2 Ranking of candidate reference genes by expression stability levels determined via NormFinder
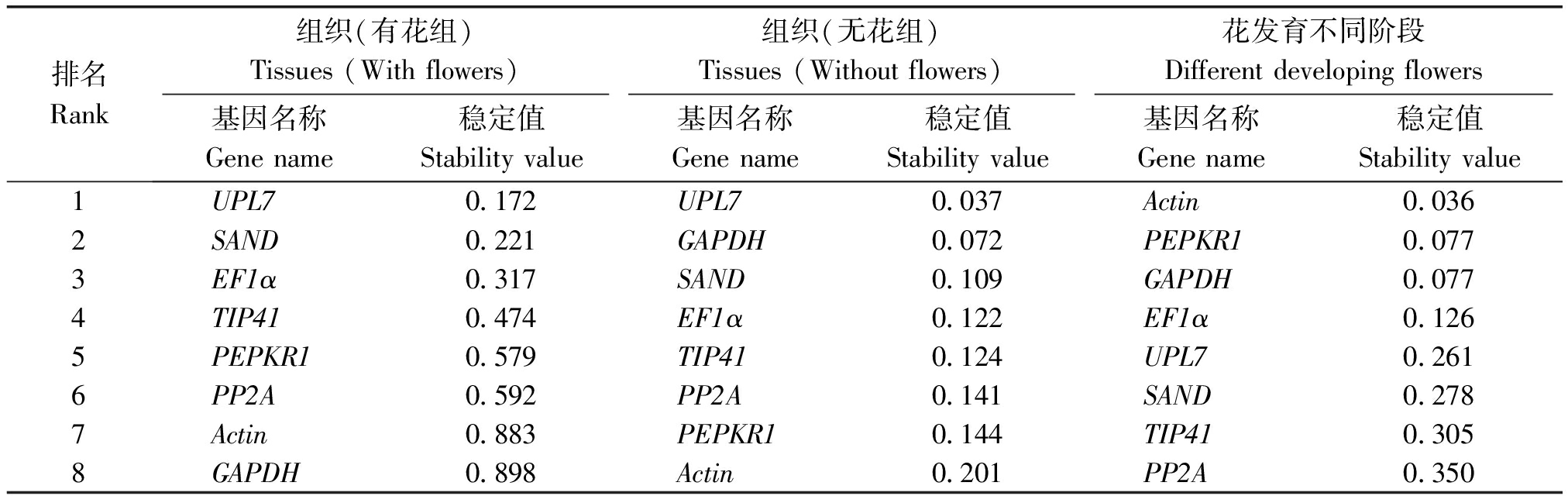
排名Rank组织(有花组)Tissues(Withflowers)组织(无花组)Tissues(Withoutflowers)花发育不同阶段Differentdevelopingflowers基因名称Genename稳定值Stabilityvalue基因名称Genename稳定值Stabilityvalue基因名称Genename稳定值Stabilityvalue1UPL70.172UPL70.037Actin0.0362SAND0.221GAPDH0.072PEPKR10.0773EF1α0.317SAND0.109GAPDH0.0774TIP410.474EF1α0.122EF1α0.1265PEPKR10.579TIP410.124UPL70.2616PP2A0.592PP2A0.141SAND0.2787Actin0.883PEPKR10.144TIP410.3058GAPDH0.898Actin0.201PP2A0.350
2.3.3 BestKeeper分析 BestKeeper通过计算不同样品间候选内参基因Ct值产生配对的变异系数(CV)、标准差(s)的大小来对各内参基因表达的稳定性进行评价。CV和s越小,说明该内参基因的稳定性越好;若s>1,则说明该内参基因表达不稳定。BestKeeper软件分析结果显示,在有花组中,Actin(s=0.42)和PEPKR1(s=0.78)s值小于1,说明这2个候选内参基因的表达稳定性较好;在无花组中,8个候选内参基因的s值均小于1,说明各内参基因的表达均为稳定,稳定性由高到低进行排序为:UPL7>SAND>GAPDH>PP2A>TIP41>EF1α>Actin>PEPKR1;在花发育不同阶段中,8个内参基因的s值均小于1,说明各基因的表达均为稳定,稳定性由高到低进行排序为:EF1α>Actin>PEPKR1>GAPDH>UPL7>TIP41>SAND>PP2A(表3)。
表3 BestKeeper分析候选内参基因表达稳定性排名
Tab.3 Expression stability values of candidate reference genes determined through BestKeeper
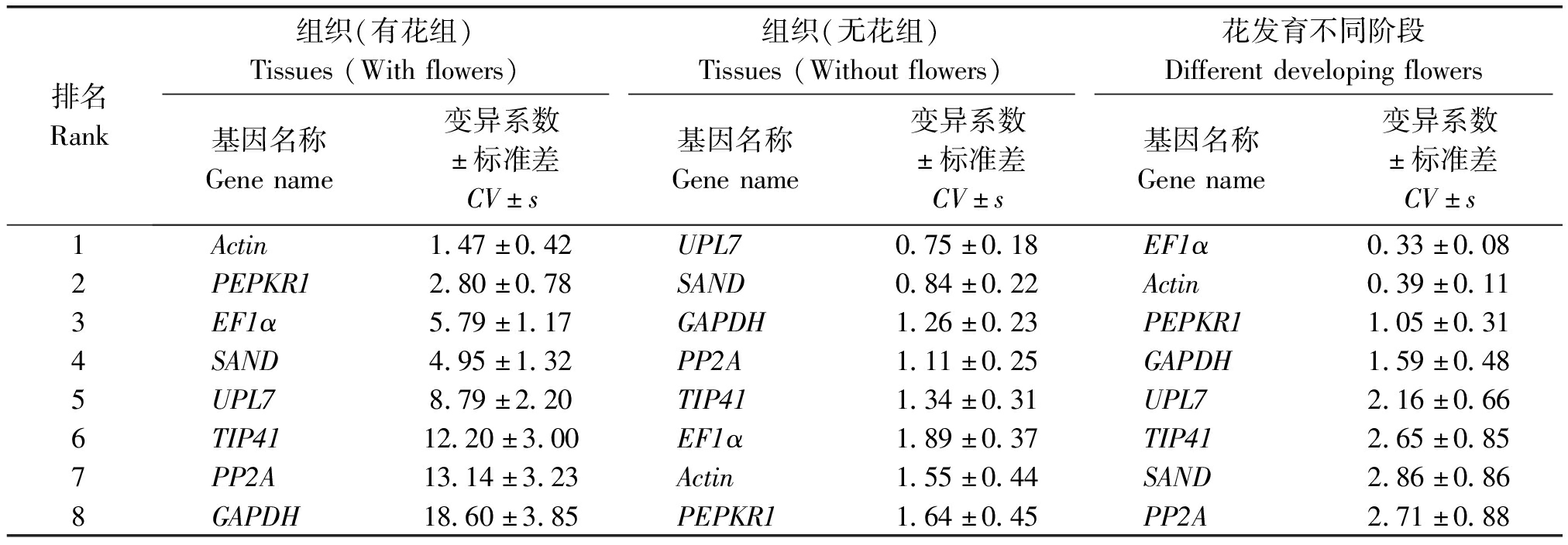
排名Rank组织(有花组)Tissues(Withflowers)组织(无花组)Tissues(Withoutflowers)花发育不同阶段Differentdevelopingflowers基因名称Genename变异系数±标准差CV±s基因名称Genename变异系数±标准差CV±s基因名称Genename变异系数±标准差CV±s1Actin1.47±0.42UPL70.75±0.18EF1α0.33±0.082PEPKR12.80±0.78SAND0.84±0.22Actin0.39±0.113EF1α5.79±1.17GAPDH1.26±0.23PEPKR11.05±0.314SAND4.95±1.32PP2A1.11±0.25GAPDH1.59±0.485UPL78.79±2.20TIP411.34±0.31UPL72.16±0.666TIP4112.20±3.00EF1α1.89±0.37TIP412.65±0.857PP2A13.14±3.23Actin1.55±0.44SAND2.86±0.868GAPDH18.60±3.85PEPKR11.64±0.45PP2A2.71±0.88
注:CV.变异系数;s.标准差。
Note:CV.Coefficient of variation; s.Standard deviation.
2.3.4 RefFinder综合排名分析 由于各个软件算法原理不同,3个软件获得的最稳定的内参基因排名有差异,最后使用RefFinder对8个候选内参基因稳定性进行综合分析,几何平均值越小,其表达稳定性越好。结果显示,在有花组中,8个候选内参基因中表达的稳定性由高到低依次为:SAND>UPL7>EF1α>PEPKR1>Actin>TIP41>PP2A>GAPDH;在无花组中,内参基因的稳定性由高到低依次为:UPL7>GAPDH>SAND>TIP41>EF1α>PP2A>PEPKR1>Actin;花发育不同阶段中内参基因的稳定性由高到低依次为:Actin>EF1α>PEPKR1>GAPDH>SAND>UPL7>TIP41>PP2A(表4)。
表4 候选内参基因表达稳定性综合排名
Tab.4 The overall ranking of expression stability values of candidate reference genes determined through RefFinder
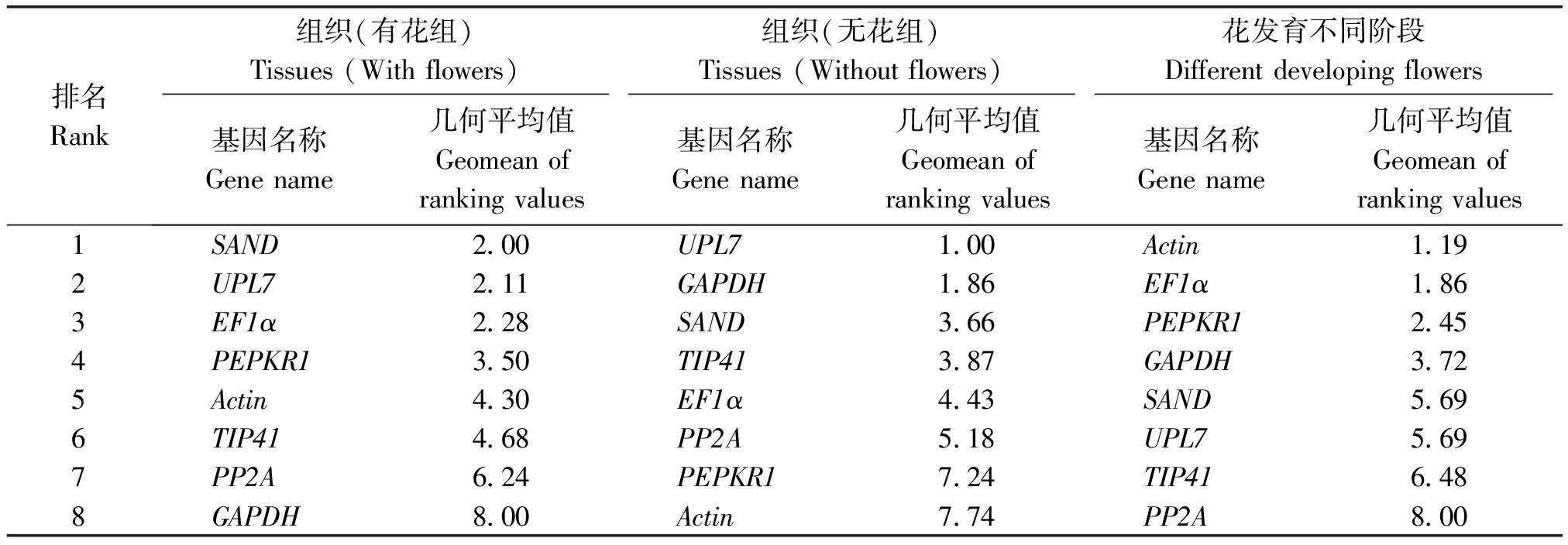
排名Rank组织(有花组)Tissues(Withflowers)组织(无花组)Tissues(Withoutflowers)花发育不同阶段Differentdevelopingflowers基因名称Genename几何平均值Geomeanofrankingvalues基因名称Genename几何平均值Geomeanofrankingvalues基因名称Genename几何平均值Geomeanofrankingvalues1SAND2.00UPL71.00Actin1.192UPL72.11GAPDH1.86EF1α1.863EF1α2.28SAND3.66PEPKR12.454PEPKR13.50TIP413.87GAPDH3.725Actin4.30EF1α4.43SAND5.696TIP414.68PP2A5.18UPL75.697PP2A6.24PEPKR17.24TIP416.488GAPDH8.00Actin7.74PP2A8.00
2.4 内参基因表达稳定性验证
为验证筛选的内参基因表达稳定性,分别用稳定性排名最高的2个内参基因Actin、EF1α及Actin+EF1α组合,以最不稳定的内参基因PP2A为对照,对花发育不同阶段的JsDXS和JsPAL2的表达水平进行分析。结果显示,以稳定性好的内参基因Actin、EF1α及Actin+EF1α组合为参照时,JsDXS基因在花发育不同阶段的表达水平的变化趋势基本一致,而用最不稳定的内参基因PP2A为参照时,花发育不同阶段的表达水平的变化趋势不一致(图7-A);同样,以Actin、EF1α及Actin+EF1α组合为参照时,JsDXS在花发育不同阶段的表达水平的变化趋势基本一致,而用PP2A为参照时,花发育不同阶段的表达水平的变化趋势不一致(图7-B)。
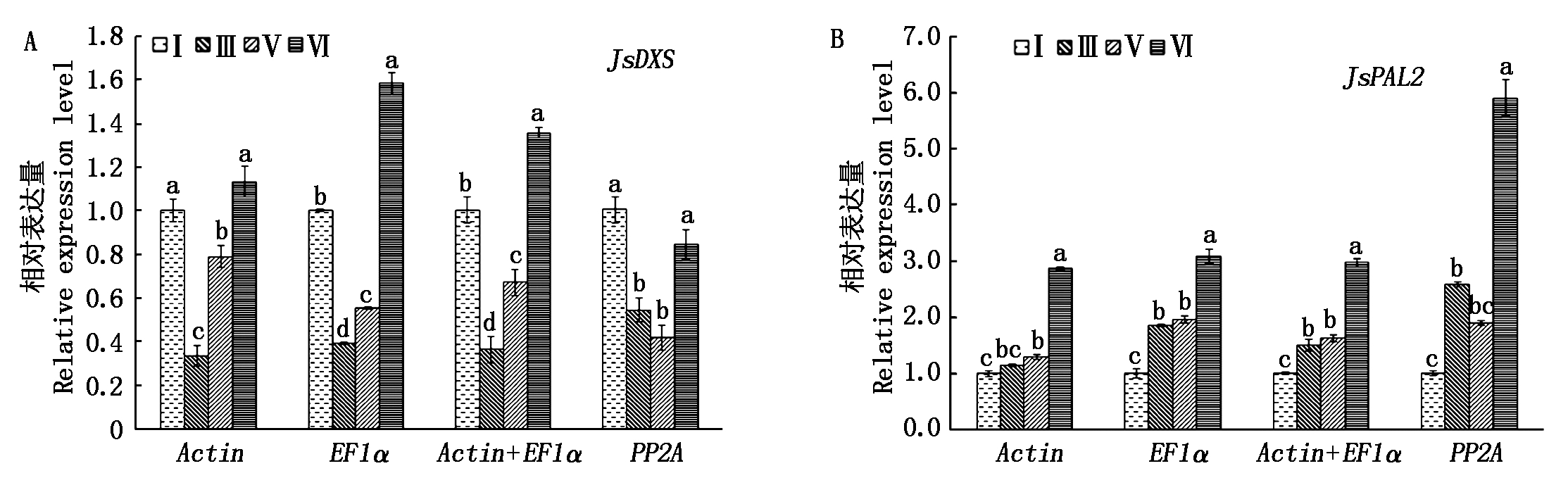
图7 不同内参基因或组合分析茉莉花花发育不同阶段JsDXS和JsPAL2表达水平
Fig.7 Expression level of JsDXS and JsPAL2 in flower samples of different developmental stages using Actin, EF1α, Actin+EF1α and PP2A
3 讨论与结论
qRT-PCR是目前在转录水平上研究基因表达最为普遍的技术,具有灵敏度高、特异性强、快速高效和重复性好等优点,常用于相对定量分析生物体不同组织间、不同生长发育时期、不同试验条件下基因表达的变化,而qRT-PCR对目的基因进行表达水平分析的前提是选择表达稳定的内参基因[4, 24]。试验中常选择管家基因作为内参基因,因为这些基因在生物体各组织和不同生长阶段表达水平较为稳定。近年来研究发现,这些管家基因在不同植物、组织、生长发育时期及环境下表达的水平并非稳定[25]。如非生物胁迫处理的海滨雀稗草中,内参基因U2AF和GAPDH在镉处理的叶和冷处理的根中稳定性最好,U2AF和FBOX在镉处理的根和冷处理的叶中稳定性最好,SAND和CACS在盐处理的叶中稳定性最好[4];火龙果果实不同发育期样本中,内参基因YLS8和TBP2稳定性最好[26];在水稻稻瘟病菌的发病机理研究中,Edf和TI是最稳定的内参基因,而常见的管家基因18S rRNA和UBQ5是最不稳定的内参基因[27]。本研究也有类似发现,同一基因在不同组织中稳定性存在较大差异,如GAPDH在不同组织(有花组)中是最不稳定的内参基因,而在不同组织(无花组)中是稳定的内参基因。由此可见,不只在不同物种间内参基因的表达存在差异,即便同一物种,若研究的组织、生长阶段和环境等不同,内参基因表达的稳定性也存在差异。因此,做目的基因表达分析之前,必须对具体条件下内参基因表达的稳定性进行筛选及验证。
geNorm、NormFinder和BestKeeper是目前对内参基因稳定性进行评价中最常用的3种软件。本研究中,发现3种软件的内参基因稳定性排名有所不同,其中,茉莉花不同组织(有花组)中,Actin在BestKeeper中的稳定性最好,但是其在geNorm和NormFinder中的稳定性都较差;SAND和UPL7在BestKeeper中的稳定性不好,但其在geNorm和NormFinder中的稳定性都较好。该现象在柑橘(Citrus reticulata)[28]、凤丹[29]的内参基因的筛选中也有报道,推测可能与计算机内置统计学算法不同有关,geNorm和NormFinder都是利用各内参基因Ct值换算成Q值进行分析,而BestKeeper直接用Ct值进行分析[30]。在实际选用内参基因时,为避免因软件分析差异带来的误差,通常利用几何平均法对不同软件得到的结果进行分析得出综合排名,该排名在一定程度上可以说明各内参基因的稳定性趋势[9]。本研究利用RefFinder对8个候选内参基因稳定性进行综合分析,从而筛选出了在不同样品中表达最为稳定的内参基因。geNorm分析发现,在茉莉花不同组织(有花组)中,Vn/Vn+1值均大于0.15,在榛[31]、梨[32]内参基因的筛选中也发现了类似现象,这可能是由于试验材料来自不同组织,跨度较大而导致变异系数均大于0.15。
在对目的基因表达水平进行分析时,不仅要求所用内参基因表达稳定,而且要求其表达量与目的基因的表达量相近[33]。本研究中,茉莉花不同组织(无花组)、花发育不同阶段最优内参基因中,GAPDH、EF1α表达丰度高,适合作为分析较高表达丰度目的基因时的内参;UPL7、Actin表达丰度低,适合作为分析较低表达丰度目的基因时的内参。本研究发现茉莉花发育不同时期最适内参基因为Actin和EF1α,这与已有研究中常用Actin作为内参基因一致[15, 22, 34],说明Actin基因适合做花的内参基因。利用最适内参基因Actin和EF1α对目的基因JsDXS和JsPAL2的表达水平进行分析,结果进一步说明筛选出来的Actin和EF1α稳定性较好。
总之,本研究对茉莉花的内参基因进行了筛选,不同组织(有花组)的最优内参基因为SAND和UPL7;不同组织(无花组)的最优内参基因为UPL7和GAPDH;花发育不同阶段的最优内参基因为Actin和EF1α。本研究结果为茉莉花基因表达分析提供了可选择的内参基因,为后续基因表达、功能及调控等相关研究奠定了基础。
[1] Giulietti A, Overbergh L, Valckx D, Decallonne B, Bouillon R, Mathieu C.An overview of Real-time quantitative PCR: applications to quantify cytokine gene expression[J].Methods, 2001, 25(4): 386-401.doi:10.1006/meth.2001.1261.
[2] VanGuilder H D, Vrana K E, Freeman W M.Twenty-five years of quantitative PCR for gene expression analysis[J].BioTechniques, 2008, 44(5): 619-626.doi:10.2144/000112776.
[3] Yang Z M, Chen Y, Hu B Y, Tan Z Q, Huang B R.Identification and validation of reference genes for quantification of target gene expression with quantitative Real-time PCR for tall fescue under four abiotic stresses[J].PLoS One, 2015, 10(3): e0119569.doi:10.1371/journal.pone.0119569.
[4] Liu Y, Liu J, Xu L, Lai H, Chen Y, Yang Z M, Huang B R.Identification and validation of reference genes for Seashore paspalum response to abiotic stresses[J].International Journal of Molecular Sciences, 2017, 18(6): 1322.doi:10.3390/ijms18061322.
[5] Czechowski T, Stitt M, Altmann T, Udvardi M K, Scheible W R.Genome-wide identification and testing of superior reference genes for transcript normalization in Arabidopsis[J].Plant Physiology, 2005, 139(1): 5-17.doi:10.1104/pp.105.063743.
[6] Chang E M, Shi S Q, Liu J F, Cheng T L, Xue L, Yang X Y, Yang W J, Lan Q, Jiang Z P.Selection of reference genes for quantitative gene expression studies in Platycladus orientalis(Cupressaceae)using Real-time PCR[J].PLoS One, 2012, 7(3): e33278.doi:10.1371/journal.pone.0033278.
[7] Galeano E, Vasconcelos T S, Ramiro D A, De Martin V D F, Carrer H.Identification and validation of quantitative real-time reverse transcription PCR reference genes for gene expression analysis in teak(Tectona grandis L.f.)[J].BMC Research Notes, 2014, 7: 464.doi:10.1186/1756-0500-7-464.
[8] 吕运舟, 董筱昀, 黄利斌.黄山栾树实时荧光定量PCR内参基因的筛选[J].分子植物育种, 2019, 17(2): 553-560.doi:10.13271/j.mpb.017.000553.
Lü Y Z, Dong X Y, Huang L B.The screening of reference genes for Real-time fluorescent quantitative PCR of Koelreuteria bipinnata[J].Molecular Plant Breeding, 2019, 17(2): 553-560.
[9] 任锐, 戴鹏辉, 李萌, 刘志明, 曹福祥.珙桐实时定量PCR内参基因的筛选及稳定性评价[J].植物生理学报, 2016, 52(10): 1565-1575.doi:10.13592/j.cnki.ppj.2016.0325.
Ren R, Dai P H, Li M, Liu Z M, Cao F X.Selection and stability evaluation of reference genes for Real-time quantitative PCR in dove tree(Davidia involucrata)[J].Plant Physiology Journal, 2016, 52(10): 1565-1575.
[10] Postollec F, Falentin H, Pavan S, Combrisson J, Sohier D.Recent advances in quantitative PCR(qPCR)applications in food microbiology[J].Food Microbiology, 2011, 28(5): 848-861.doi:10.1016/j.fm.2011.02.008.
[11] 袁昌齐, 束成杰, 贾晓东, 肖正春, 张卫明.“一带一路”经济植物应用与开发(南亚篇)[J].中国野生植物资源, 2016, 35(2): 1-2.doi:10.3969/j.issn.1006-9690.2016.02.001.
Yuan C Q, Shu C J, Jia X D, Xiao Z C, Zhang W M.Application and development of economic plantsin“One Belt and One Road”(Chapter South Asia)[J].Chinese Wild Plant Resources, 2016, 35(2): 1-2.
[12] 邓衍明, 孙晓波, 贾新平, 梁丽建, 苏家乐.秋水仙素浓度和处理时间对茉莉腋芽生长发育的影响[J].植物资源与环境学报, 2016, 25(3): 62-71.doi:10.3969/j.issn.1674-7895.2016.03.08.
Deng Y M, Sun X B, Jia X P, Liang L J, Su J L.Effects of concentration and treatment time of colchicine on growth and development of axillary buds of Jasminum sambac[J].Journal of Plant Resources and Environment, 2016, 25(3): 62-71.
[13] 邓衍明, 齐香玉, 苏家乐.茉莉生殖发育与分子生物学特性研究分析[J].核农学报, 2019, 33(2): 270-277.doi:10.11869/j.issn.100-8551.2019.02.0270.
Deng Y M, Qi X Y, Su J L.Studies on reproductive development and molecular biology characteristics of Jasmine sambac(L.)Aition[J].Journal of Nuclear Agricultural Sciences, 2019, 33(2): 270-277.
[14] Bera P, Mukherjee C, Mitra A.Enzymatic production and emission of floral scent volatiles in Jasminum sambac[J].Plant Science, 2017, 256: 25-38.doi:10.1016/j.plantsci.2016.11.013.
[15] Yu Y, Lyu S H, Chen D, Lin Y, Chen J J, Chen G X, Ye N X.Volatiles emitted at different flowering stages of Jasminum sambac and expression of genes related to α-farnesene biosynthesis[J].Molecules, 2017, 22(4): 546.doi:10.3390/molecules22040546.
[16] Deng Y M, Jia X P, Liang L J, Gu C S, Sun X B.Morphological anatomy, sporogenesis and gametogenesis in flowering process of jasmine(Jasminum sambac Aiton)[J].Scientia Horticulturae, 2016, 198: 257-266.doi:10.1016/j.scienta.2015.11.036.
[17] Wei L B, Miao H M, Zhao R H, Han X H, Zhang T D, Zhang H Y.Identification and testing of reference genes for sesame gene expression analysis by quantitative real-time PCR[J].Planta, 2013, 237(3): 873-889.doi:10.1007/s00425-012-1805-9.
[18] 付建新, 张超, 王艺光, 赵宏波.桂花组织基因表达中荧光定量PCR内参基因的筛选[J].浙江农林大学学报, 2016, 33(5): 727-733.doi:10.11833/j.issn.2095-0756.2016.05.001.
Fu J X, Zhang C, Wang Y G, Zhao H B.Reference gene selection for quantitative real-time polymerase chain reaction(qRT-PCR)normalization in the gene expression of sweet osmanthus tissues[J].Journal of Zhejiang A & F University, 2016, 33(5): 727-733.
[19] Andersen C L, Jensen J L, Ørntoft T F.Normalization of Real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets[J].Cancer Research, 2004, 64(15): 5245-5250.doi:10.1158/0008-5472.CAN-04-0496.
[20] Pfaffl M W, Tichopad A, Prgomet C, Neuvians T P.Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper-Excel-based tool using pair-wise correlations[J].Biotechnology Letters, 2004, 26(6): 509-515.doi:10.1023/B:BILE.0000019559.84305.47.
[21] Xie F L, Xiao P, Chen D L, Xu L, Zhang B H.miRDeepFinder: a miRNA analysis tool for deep sequencing of plant small RNAs[J].Plant Molecular Biology, 2012, 80(1): 75-84.doi:10.1007/s11103-012-9885-2.
[22] 孙君, 陈桂信, 叶乃兴, 吕恃衡, 刘志钦, 黄玮, 林志达.茉莉花香气相关基因JsDXS及其启动子的克隆与表达分析[J].园艺学报, 2014, 41(6): 1236-1244.doi:10.16420/j.issn.0513-353x.2014.06.011.
Sun J, Chen G X, Ye N X, Lü S H, Liu Z Q, Huang W, Lin Z D.Cloning and expression analysis of deoxyoxylulose-5-phosphate synthase gene related to aroma from Jasminum sambac and isolation of its promoter[J].Acta Horticulturae Sinica, 2014, 41(6): 1236-1244.
[23] 熊青, 宋姣敏, 崔萌, 许颖妍, 俞滢, 叶乃兴, 陈桂信.茉莉花JsPAL2基因的克隆与表达分析[J].热带作物学报, 2018, 39(7): 1359-1366.doi:10.3969/j.issn.1000-2561.2018.07.015.
Xiong Q, Song J M, Cui M, Xu Y Y, Yu Y, Ye N X, Chen G X.Cloning and expression analysis of JsPAL2 gene from Jasminum sambac[J].Chinese Journal of Tropical Crops, 2018, 39(7): 1359-1366.
[24] Xiao Z, Sun X B, Liu X Q, Li C, He L S, Chen S P, Su J L.Selection of reliable reference genes for gene expression studies on Rhododendron molle G.Don[J].Frontiers in Plant Science, 2016, 7: 1547.doi:10.3389/fpls.2016.01547.
[25] Die J V, Román B, Nadal S, González-Verdejo C I.Evaluation of candidate reference genes for expression studies in Pisum sativum under different experimental conditions[J].Planta, 2010, 232: 145-153.doi:10.1007/s00425-010-1158-1.
[26] 崇慧影, 文晓鹏.发育期火龙果内参基因的筛选和验证[J].生物技术, 2019, 29(1): 39-45,56.doi:10.16519/j.cnki.1004-311x.2019.01.0008.
Chong H Y, Wen X P.Screening and validation of reference genes during fruit development in potaya[J].Biotechnology, 2019, 29(1): 39-45,56.
[27] Sagun C M, Grandmottet F, Suachaowna N, Sujipuli K, Ratanasut K.Validation of suitable reference genes for normalization of quantitative reverse transcriptase-polymerase chain reaction in rice infected by Xanthomonas oryzae pv.oryzae[J].Plant Gene, 2020, 21: 100217.doi:10.1016/j.plgene.2019.100217.
[28] Mafra V, Kubo K S, Alves-Ferreira M, Ribeiro-Alves M, Stuart R M, Boava L P, Rodrigues C M, Machado M A.Reference genes for accurate transcript normalization in Citrus genotypes under different experimental conditions[J].PLoS One, 2012, 7(2): e31263.doi:10.1371/journal.pone.0031263.
[29] Li C Q, Hu L Z, Wang X Q, Liu H Z, Tian H H, Wang J S.Selection of reliable reference genes for gene expression analysis in seeds at diferent developmental stages and across various tissues in Paeonia ostia[J].Molecular Biology Reports, 2019, 46: 6003-6011.doi:10.1007/s11033-019-05036-7.
[30] De Spiegelaere W, Dern-Wieloch J, Weigel R, Schumacher V, Schorle H, Nettersheim D, Bergmann M, Brehm R, Kliesch S, Vandekerckhove L, Fink C.Reference gene validation for RT-qPCR, a note on different available software packages[J].PLoS One, 2015, 10(3): e0122515.doi:10.1371/journal.pone.0122515.
[31] 杨丹, 李清, 王贵禧, 马庆华, 朱利泉.平欧杂种榛实时荧光定量PCR内参基因的筛选与体系建立[J].中国农业科学, 2017, 50(12): 2399-2410.doi:10.3864/j.issn.0578-1752.2017.12.020.
Yang D, Li Q, Wang G X, Ma Q H, Zhu L Q.Reference genes selection and system establishment for real-time qPCR analysis in Ping′ou hybrid hazelnut(C.heterophylla Fisch.× C.avellana L.)[J].Scientia Agricultura Sinica, 2017, 50(12): 2399-2410.
[32] 蒲小秋, 田嘉, 李疆, 张艳, 李鹏, 覃伟铭, 井春芝.梨果实细胞分裂期内参基因表达稳定性分析[J].经济林研究, 2020, 38(1): 66-74.doi:10.14067/j.cnki.1003-8981.2020.01.009.
Pu X Q, Tian J, Li J, Zhang Y, Li P, Qin W M, Jing C Z.Analysis on expression stability of internal reference genes at cell division stage of pear fruits[J].Non-wood Forest Research, 2020, 38(1): 66-74.
[33] Gimeno J, Eattock N, Van Deynze A, Blumwald E.Selection and validation of reference genes for gene expression analysis in switchgrass(Panicum virgatum)using quantitative real-time RT-PCR[J].PLoS One, 2014, 9(3): e91474.doi:10.1371/journal.pone.0091474.
[34] 陈笛, 陈雪津, 郭永春, 王鹏杰, 岳川, 陈桂信, 叶乃兴.茉莉花芳樟醇生物合成关键基因的克隆与表达分析[J].西北植物学报, 2019, 39(8): 1344-1352.doi:10.7606/j.issn.1000-4025.2019.08.1344.
Chen D, Chen X J, Guo Y C, Wang P J, Yue C, Chen G X, Ye N X.Clong and expression analysis of JsNEL/LINS from Jasminum sambac[J].Acta Botanica Boreali-Occidentalia Sinica, 2019, 39(8): 1344-1352.