牦牛(Bos grunniens)作为一种生活在高海拔、强紫外且低氧的高原地区的优势种群,是高原地区人民生活的必需种家畜,其具有“全能家畜”、“高原之舟”的美称,但与黄牛的繁殖情况不同,通常一年一胎或三年两胎,因此,其产仔率低下[1],严重阻碍了牧民的经济收益。KDM7A(Histone demethylase 7A)是一种染色质组蛋白修饰因子,能够参与哺乳动物生殖细胞分化发育过程。但该基因在牦牛中的表达模式及作用机制尚未可知,因此,开展KDM7A与牦牛生殖相关研究具有重要的科学意义。
表观遗传是一种在不改变碱基遗传序列情况下进行修饰的非孟德尔遗传,主要包括组蛋白修饰、DNA甲基化修饰、染色质结构修饰及复杂三维基因组排列[2-3]。组蛋白修饰是一种在组蛋白特定残基位点由特定相关酶作用的重要遗传修饰。甲基化、乙酰化、磷酸化等是其主要的修饰方式[4]。研究表明,组蛋白去甲基化在遗传过程中发挥重要的作用[5],甲基化水平通过组蛋白甲基化酶(HMTs)和去甲基化酶(HDMs)相互协调作用于氨基酸残基特定位点。根据是否含有JmjC结构域,可将组蛋白去甲基化酶(HDMs)分为FAD依赖性胺氧化酶KDM1家族与JHDM家族两大类,其主要作用于组蛋白H3/H4的赖氨酸和精氨酸N-端残基上。KDM1家族包含2个成员,KDM1A/LSD1和KDM1B/LSD2,只能作用于组蛋白单甲基化和二甲基化(mel/me2)位点;而JHDM家族利用2-酮戊二酸依赖性加氧酶(2-OGDO)机制修饰单甲基、二甲基和三甲基化(mel/me2/me3)位点,所以导致细胞分裂过程中染色体较大变异性,同时说明表观遗传标记具有多功能性[5-7]。
KDM7A又称为KIAA1718,在N-末端具有PhD结构域,在C-末端具有JmjiC结构域,其活性位点由一个Fe辅助因子、带电共底物酮戊二酸(2-OG)3个保守的氨基酸形成的His-Asp-His三联体组成的保守双链β-螺旋(DSBH)[8-9]。已有报道证实,KDM7A是一个组蛋白双位点去甲基化酶,作用于H3K9和H3K27 二甲基化残基位点上[10]。KDM7A能够调控癌症细胞的形成及增殖,Mallm等[11]发现KDM7A能够诱导人胶质母细胞瘤初级细胞(Human glioblastoma initiating cells, GICs)分化并促使其形成DNA损伤,Meng 等[12]利用基因编辑技术,敲除裸鼠KDM7A基因后,Krüppel样因子4(Krüppel-like factor 4, KLF4)和c-Myc癌基因表达量增强,能够维持乳腺癌干细胞(Breast cancer stem cells, BCSCs)的干性。KDM7A能影响生物繁殖过程,其主要参与调节哺乳动物胚胎神经元的分化发育,在成纤维细胞生长因子(Fibroblast growth factor,FGF4)的直接介导下促使小鼠胚胎干细胞和早期鸡胚的神经分化,该基因缺失致使出现神经发育缺陷和胚胎发育停滞的现象[10,13]。
目前,与KDM7A相关研究主要涉及该基因对人类癌症细胞的调控机制[11-12]及家禽和小鼠胚胎分化发育的影响[10-13],但其在牦牛各组织及卵母细胞成熟过程中的作用机制及表达模式尚未见报道。本研究以4周岁的未妊娠成年母牦牛作为试验对象,利用RT-PCR技术克隆获得牦牛KDM7A的CDS区序列,RT-qPCR检测KDM7A在牦牛各个组织中及卵母细胞成熟过程中的表达水平,旨在分析KDM7A理化性质及在牲牛生殖过程中的表达规律,为牦牛育种繁殖提供一定的理论基础。
1 材料和方法
1.1 试验动物与组织样品采集
试验动物源于四川成都周边屠宰场,选择4周岁未妊娠的母牦牛3头,屠宰后立即采集心、胃、肾、肝、肺、小肠、脾、肌肉、子宫及卵巢组织。用加入双抗(青霉素和链霉素混合物)的生理盐水反复冲洗。用无菌眼科剪处理为1.0 cm×0.5 cm×0.5 cm大小的块状后立即置于含有RNA保护剂冻存管中,随后带回实验室置于超低温冰箱保存备用。
1.2 主要试剂
TRIzol RNA提取试剂购自Invitrogen公司;核酸助沉剂购自百泰克公司;PrimeScrptTMRT Reagent Kit 反转录试剂盒、pMD19-T载体购自TaKaRa;ChamQ Universal SYBR qPCR Master Mix试剂盒、2×Phanta® Max Master Mix(Dye Plus)购自南京诺唯赞;DNA胶回收试剂盒购自康宁生命科学有限公司;DEPC、DNAMarker、感受态细胞DH5α皆购自天根生化科技有限公司;胚胎专用石蜡油(M-8410)(Sigma);0.5%透明质酸酶(Hyaluronidase,Hyase):0.5 g透明质酸酶粉末(Sigma)和100 mL Medium199;卵母细胞成熟液(OM培养液):10%胎牛血清(FBS)、Medium 199培养基购自赛默飞公司提供;1 μg/mL促卵泡素(Follicle-stimulating hormone,FSH)、1 μg/mL促黄体素(Luteinizing hormone, LH)购自Vetoquinol公司;1 μg/mL 17β-雌二醇(17β-estradiol, E2),0.02 mg/mL丙酮酸钠(C3H3NaO3)和0.03 mg/mL谷氨酰胺(Glutamine, Gln)购自Sigma公司。
1.3 卵巢获取及卵母细胞培养收集
牦牛屠宰后立即收集3~5周岁母牦牛(6头)的卵巢,用含有双抗的生理盐水冲洗3~5次,立即置于30 ℃无菌生理盐水暂作保存,于3 h内带至实验室。利用5 mL一次性无菌注射器抽取表面直径为2~10 mm大小卵泡的卵泡液。并放置在处于37 ℃恒温操作板的90 mm培养皿中。在体视显微镜下选取卵丘颗粒细胞致密、细胞质均匀无皱缩且结构紧密的卵丘-卵母细胞复合体(Cumulus-oocyte complexes, COCs),并用提前在培养箱中平衡10 h的OM培养液清洗2次。收集50颗颗粒细胞紧密包裹的COCs,用37 ℃预平衡3~6 h的5%透明质酸酶处理2 min,致使颗粒细胞全部从卵母细胞上脱离,用PBS清洗2~3次得到干净的GV期卵母细胞,收集于1.5 mL去酶EP管中备用。以30颗COCs为宜转入平衡10 h的1 mL OM成熟液中。放置于湿度100%、恒温37.5 ℃、二氧化碳通入量5%的条件下进行培养。待12 h后在体式显微镜下挑取卵丘颗粒细胞松散的COCs,并用上述方法收集50颗干净的卵母细胞置于1.5 mL 去酶EP管中备用,此时卵母细胞发育至MⅠ期。连续培养24 h后,用上述方法获得50颗干净卵母细胞收集备用,此时卵母细胞处于MⅡ期已排出第一极体。
1.4 牦牛各个组织及卵母细胞cDNA获取及检测
在液氮环境下将各个组织样进行充分研磨并加入1 mL TRIzol试剂振荡,将含有不同时期卵母细胞的EP管中加入200 μL TRIzol试剂,随后将以上样本在80 ℃环境进行过夜处理。待室温解冻后分别加入10 μL核酸助沉剂,轻微晃动再进行静止,最后依照TRIzol法进行后续试验获得各样本总RNA,并在核酸浓度测定仪中进行检测,选取OD260/280值位于1.8~2.0的RNA样本,根据PrimeScrptTM RT Reagent Kit 反转录试剂盒说明书步骤反转录获得每个组织和不同时期卵母细胞cDNA。最终收取OD260/280值处于1.8~2.0的cDNA样本,-20 ℃保存备用。
1.5 KDM7A基因克隆引物设计及合成
根据GenBank中登录的预测黄牛(Bos taurus)KDM7A序列(GenBank登录号:XM_027538848.1)结合引物设计软件Primer Premier 5.0设计扩增引物。利用NCBI中的Primer-Blast设计KDM7A基因定量引物及内参定量引物,引物由金斯瑞生物科技有限公司合成(表1)。
表1 PCR引物及产物长度
Tab.1 PCR primer sequences and predicted product length

引物Primers引物序列(5′-3′)Sequences(5′-3′)退火温度/℃Tm产物长度/bpProductlengthKDM7A-1F:CTGTATCACTGTCCCAACTG521137R:GTCTGGAATTTCAAAGGCKDM7A-2F:CTTATTTGTTCCTACAGGGTGG57978R:ACAGTGCAATCAGGTCCAGAGKDM7A-3F:GGGAGTGGAGAACGAAGAGTC591121R:TATTGCTGAGTGGGTGTGGCKDM7A-qPCRF:CTTGATATCAGGGGTGTGCCA60103R:AGGTCTCTTGGTGGGGTGAAβ-actin-qPCRF:AGGTGACAGCAGTCGGTTGGA60170R:CCTTAGAGAGAAGCGGGGTGG
1.6 牦牛KDM7A基因克隆测序
以牦牛子宫cDNA作为模板,3对引物PCR扩增体系均为50 μL,其中2×Phanta®Max Master Mix(Dye Plus)25.0 μL;上、下游引物(10 μmol/L)各2.0 μL;cDNA 2.0 μL;ddH2O 19.0 μL。PCR扩增程序:95 ℃预变性(3 min);95 ℃变性(15 s),52,57,59 ℃退火(15 s),72 ℃延伸(45 s),35个循环;72 ℃彻底延伸(5 min),置于4 ℃保存。配制2%琼脂糖凝胶并进行电泳检测,将目的条带在紫外光下切割回收,按照试剂盒说明进行DNA回收。在16 ℃温度下将4 μL DNA片段与pMD19-T载体连接16 h,转入大肠杆菌 DH5α感受态细胞中,并将其接种于基础培养基中增菌1.5 h后进行离心。涂布于含有氨苄青霉素(Amp)固体选择培养基中置于37 ℃培养箱过夜培养。挑取目的菌落,接种含有Amp液体培养基中摇床过夜增菌,凝胶电泳筛选阳性菌种送由生工生物工程(成都)有限公司进行测序。
1.7 牦牛KDM7A基因生物信息学分析
利用DNAMAN 7软件将KDM7A碱基拼接完整。通过在线比对工具Blast检索各物种KDM7A氨基酸序列,进行同源性比对。结合NCBI在线系统中开放阅读框ORF Finder(https://www.ncbi.nlm.nih.gov/orffinder/)预测分析牦牛KDM7A氨基酸序列。并通过ExPAsy在线软件中的ProtParam(http://web.expasy.org/protparam)、ProtScale(http://web.expasy.org/protscale/)和PredictPortein(http://www.predictprotein.org),以及MHMM 2.0 Server(http://www.cbs.dtu.dk/services/TMHMM/)、SWISS_MODEL(http://swissmodel.expasy)分析预测牦牛KDM7A蛋白质重要理化性质、疏水性、跨膜区域、二级结构及三级结构。利用MEGA 7软件构建系统进化树。
1.8 牦牛组织KDM7A基因表达谱分析
将各个组织样本cDNA作为模板。以β-actin(GenBank登录号:XM_027538848.1)作为内参基因(表1)分析KDM7A在牦牛各组织中相对表达量。反应体系为15 μL,其中ChamQ Universal SYBR qPCR Master Mix 7.5 μL,上、下游引物各0.5 μL;cDNA 1 μL;ddH2O 5.5 μL。定量反应条件为94 ℃预变性(3 min);94 ℃ 变性(15 s),60 ℃ 退火(30 s),72 ℃延伸(35 s),39个循环;溶解曲线:95 ℃ 15 s,60 ℃ 1 min。以上每个样本重复检测3次。
1.9 牦牛KDM7A基因在不同分裂时期卵母细胞中表达检测
利用RT-qPCR技术,β-actin作为内参基因,将1.4中得到的GⅤ期、MⅠ期、MⅡ期卵母细胞cDNA作为模板检测KDM7A在卵母细胞减数分裂过程中的表达量。反应体系及扩增条件参照1.8,每个样本设置3个重复。
1.10 数据处理与分析
将以上各样本获得的3个重复试验结果利用2-ΔΔCt法进行分析,结合IBM SPSS Statistic 22统计软件的单因素方差分析法分析各个组织及不同时期卵母细胞中KDM7A表达量差异显著性。其中,P<0.05表示差异显著,P>0.05表示差异不显著。
2 结果与分析
2.1 牦牛KDM7A基因克隆及序列分析
以子宫cDNA作为模板,利用分段扩增技术扩增到约1 137,1 121,978 bp的3个片段(图1)。分段扩增产物测序后,通过DNAMAN拼接获得了2 704 bp的KDM7A基因,其中CDS全长为2 409 bp,共编码802个氨基酸。同源性比对结果显示,与黄牛、野牛、绵羊和山羊同源性相对较高,分别为99.75%,99.57%,98.62%,98.46%。但与小鼠同源性相对较低为89.27%。利用MEGA 7软件构建系统进化树,结果(图2)表明,牦牛与黄牛和野牛同源关系最近,其次是绵羊。由此可见,在物种进化中KDM7A相对保守,即使发生碱基突变,也多为同义突变。
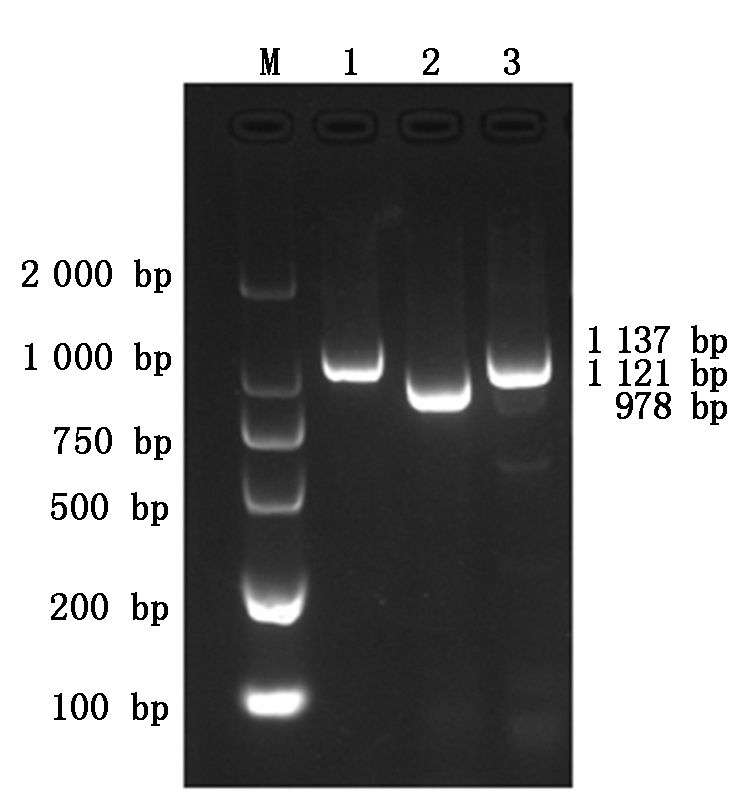
M.DL2000相对分子质量标记;1-3.KDM7A基因PCR扩增产物。
M.DL2000 DNA Marker; 1-3.KDM7A gene PCR amplification product.
图1 牦牛KDM7A基因扩增结果
Fig.1 The PCR amplification for KDM7A gene of yak
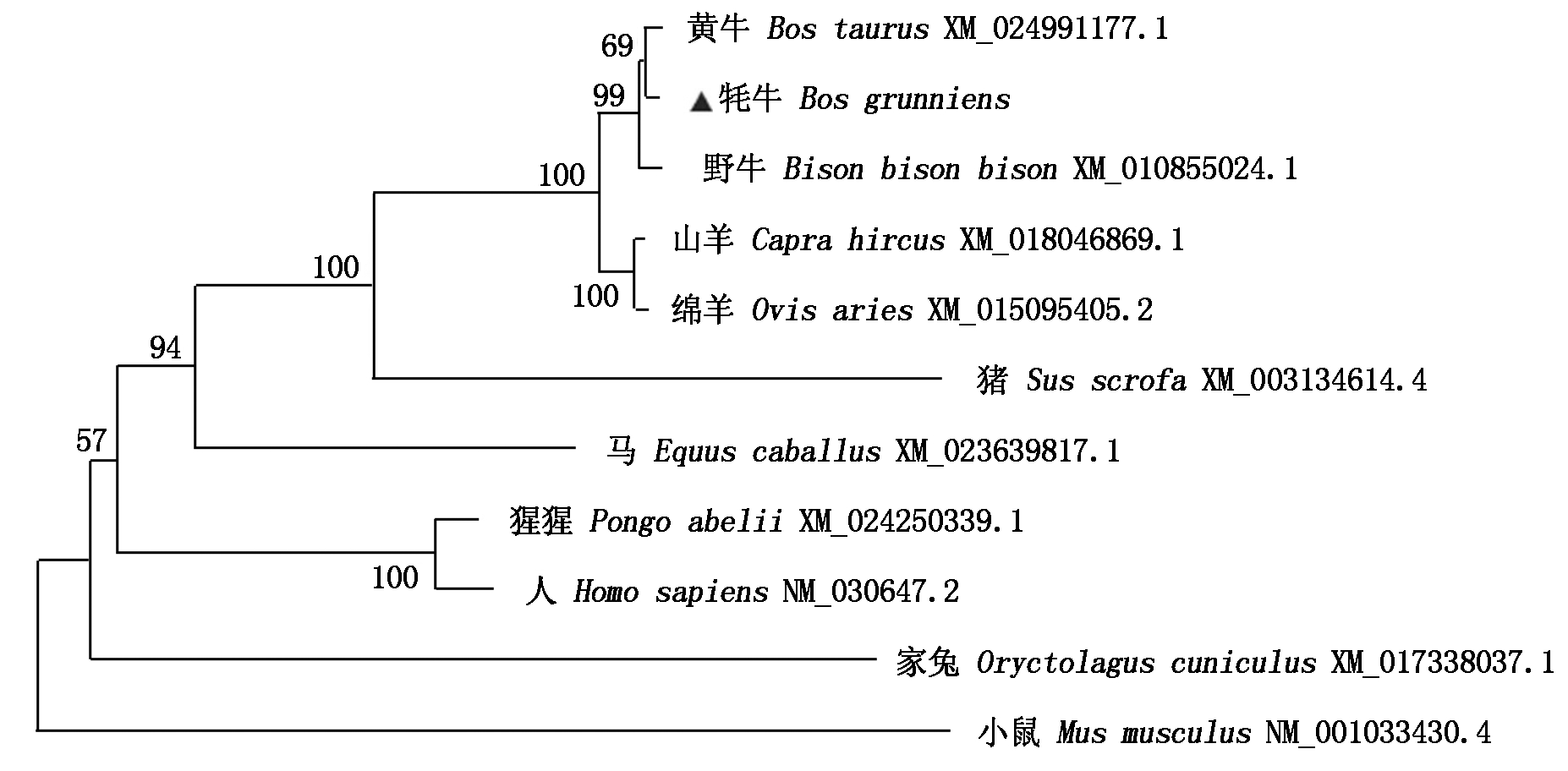
图2 KDM7A基因系统进化树
Fig.2 Phylogenetic tree analysis of KDM7A gene sequence
2.2 牦牛KDM7A蛋白质结构预测及功能分析
通过ExPAsy在线分析工具分析预测KDM7A蛋白质分子量91.551 ku,等电点为7.81,脂肪指数为69.93,不稳定指数为60.10,说明该蛋白质为不稳定蛋白,且预估半衰期为30 h。KDM7A蛋白含量较高的氨基酸有丝氨酸(Ser)、亮氨酸(Leu)和谷氨酸(Glu),含量分别为11.0%,8.2%和8.0%;含量较低的氨基酸有半胱氨酸(Cys)、甲硫氨酸(Met)和色氨酸(Trp),含量分别为2.0%,1.9%和1.5%;不含吡咯赖氨酸(Pyl)和硒半胱氨酸(Sec);正电残基精氨酸和赖氨酸(Arg+Lys)110个;带负电残基天冬氨酸和谷氨酸(Asp+Glu)108个。蛋白质亲疏水性分析,异亮氨酸(ILe)亲水性4.500,赖氨酸(Lys)亲水性为3.900,且该蛋白质中大多数氨基酸残基亲水性数值小于零,总亲疏水性平均值为0.715,因此KDM7A为亲水性蛋白。MHMM 2.0 Server和SignalP-4.1预测分析KDM7A没有跨膜结构和信号肽分泌蛋白。分析发现,KDM7A有98个磷酸化位点和1个N-端糖基化位点。结合SMART在线分析KDM7A含有JmjiC结构域。蛋白质二级结构预测分析α螺旋占25.44%,延伸链占20.32%,该蛋白质不含β-转角,无规则卷曲占54.24%(图3)。同时三级结构预测结果进一步证实二级结构的预测,KDM7A蛋白三级结构含有α螺旋,无规则卷曲和延伸链,且结果显示延伸链主要位于α螺旋和无规则卷曲结构之间(图4)。
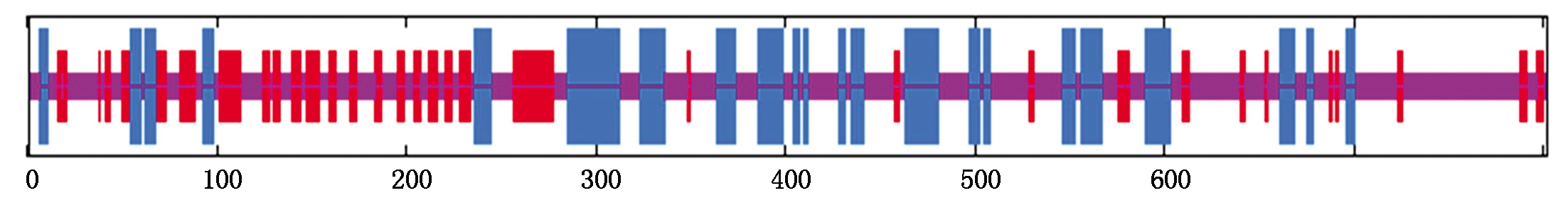
紫色.无规则卷曲(Cc);蓝色.α螺旋(Hh);红色.延伸链(Ee)。
Purple part.Irregular curl; Blue part.Alpha helix; Red part.Beta sheet.
图3 牦牛KDM7A氨基酸序列二级结构预测
Fig.3 Predicted secondary structure of amino acid sequence of yak KDM7A
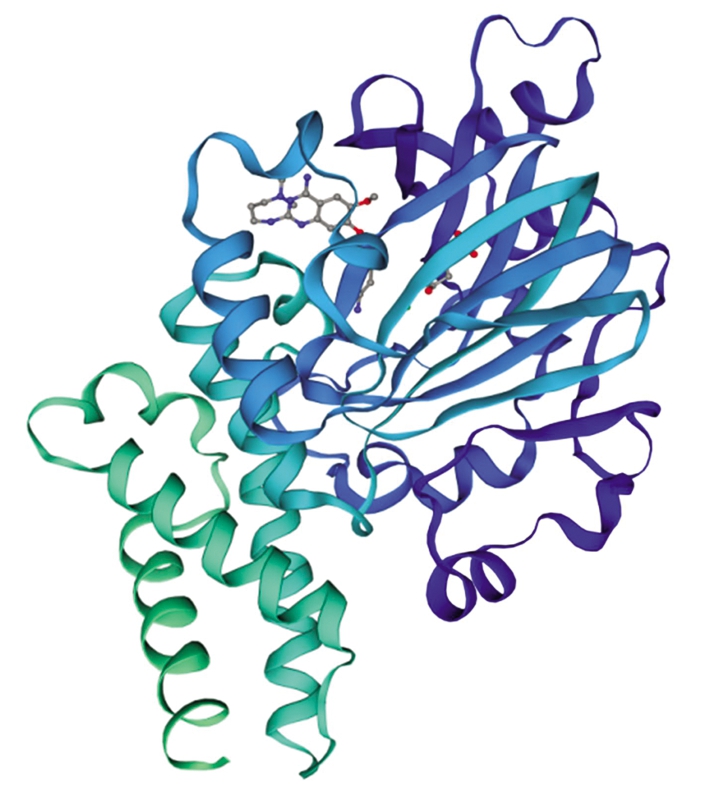
图4 牦牛KDM7A三级结构预测
Fig.4 Predicted tertiary structure of yak KDM7A
2.3 牦牛组织KDM7A基因表达谱
利用RT-qPCR技术,结合2-ΔΔCt法计算出KDM7A在各组织中的Ct值,并利用SPSS 22分析各组织中相对表达量差异性。以子宫Ct值作为参照,结果表明,KDM7A mRNA在子宫中表达量最高,在胃中表达量次之,子宫中KDM7A相对表达量显著高于除胃组织以外其他组织(P<0.05),肾脏和肌肉组织中KDM7A相对表达量显著低于其他组织(P<0.05)(图5)。
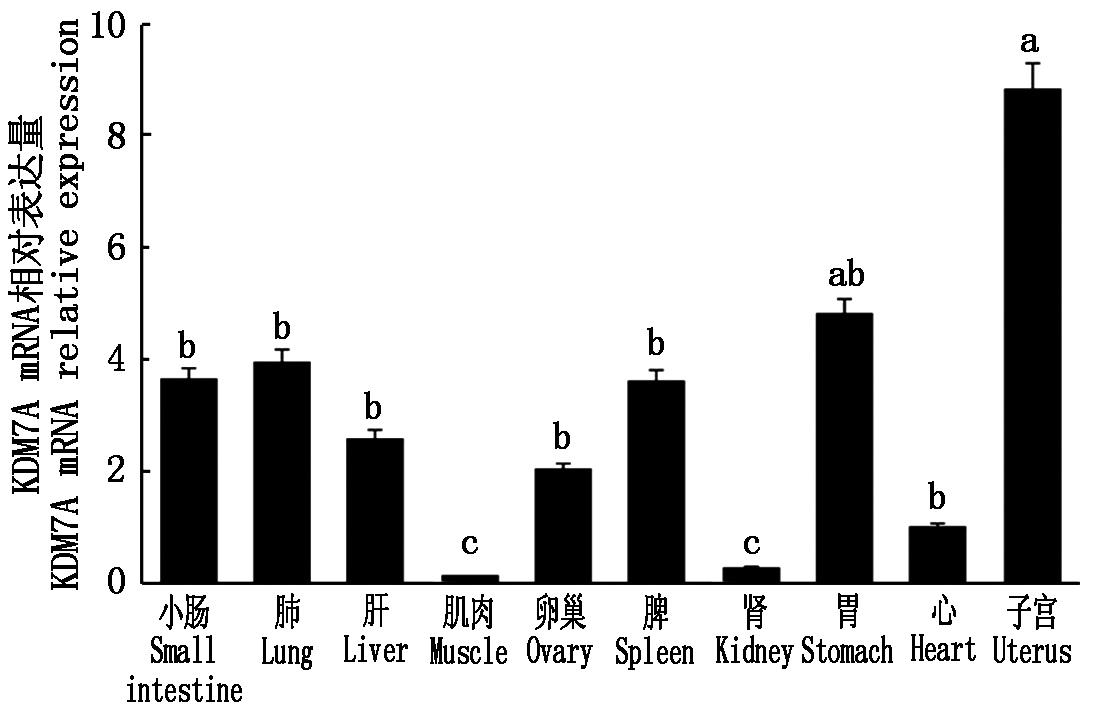
相同字母代表差异不显著(P>0.05);不同字母代表差异性显著(P<0.05);内参基因.β-actin。图6同。
The same letters suggest that the difference is not obvious(P>0.05);The difference letters suggest that the difference is obvious(P<0.05);The reference gene.β-actin.The same as Fig.6.
图5 牦牛KDM7A基因组织表达谱
Fig.5 The tissue expression profiles of yak KDM7A
2.4 牦牛KDM7A基因在不同分裂时期卵母细胞中表达
以MⅡ期卵母细胞中KDM7A mRNA相对表达量作为参照,利用RT-qPCR技术检测卵母细胞在成熟过程中表达规律(图6)。结果显示,MⅡ期卵母细胞KDM7A mRNA表达水平显著高于M Ⅰ期和GⅤ期(P<0.05),但MⅠ期与GⅤ期卵母细胞中KDM7A mRNA相对表达水平差异不显著(P>0.05)。
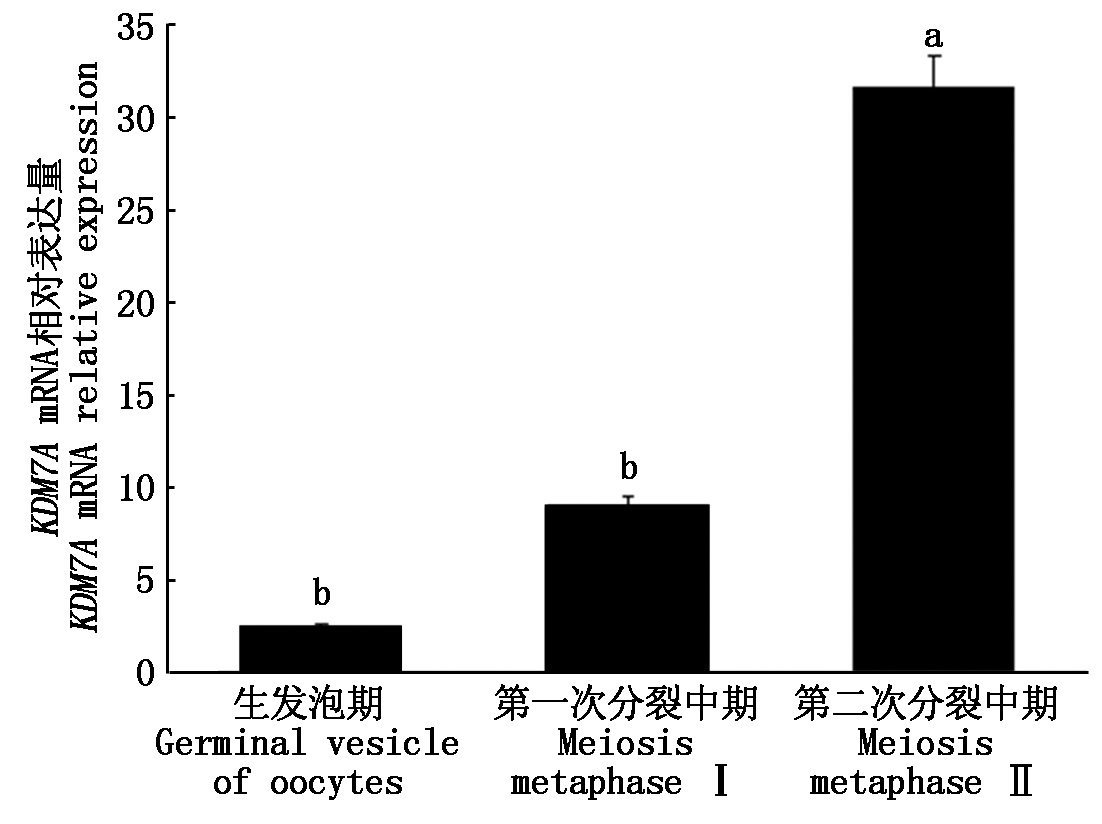
图6 牦牛不同分裂时期卵母细胞中KDM7A基因表达水平
Fig.6 The gene expression level of KDM7A in yak oocytes at different periods
3 讨论与结论
染色质、染色体是真核生物处于有丝分裂及减数分裂特定时期的特殊形式,其基本单位是由组蛋白H2A、H2B、H3和H4构成八聚体与DNA紧密缠绕形成的核小体[14]。目前大量研究报道表明,表观遗传修饰中DNA甲基化修饰在转录翻译调控过程中发挥重要作用[15-17],随着组蛋白修饰研究的开展,已证实组蛋白修饰与DNA甲基化在表观遗传中皆发挥举足轻重的作用[18]。最初对组蛋白去甲基化修饰的研究主要集中于植物昆虫[19-21]、基因互作[22]、化学干扰表达[23]、癌症调控[5]等方面,最新研究证实KDM7A影响动物早期胚胎神经分化及胚胎发育[13]。Kim等[24]发现赖氨酸脱甲基酶LSD1调节小鼠卵母细胞减数分裂进程,在卵母细胞成熟过程中LSD1条件性缺失导致纺锤体和染色体异常,致使卵母细胞无法从GⅤ期发育至MⅠ期,随后发生细胞凋亡。Kawakami等[25]通过敲除小鼠早期胚胎KDM2A,结合免疫组化、免疫荧光等技术发现KDM2A缺失导致10.5~12.5 d胎鼠致死并伴有严重的生长缺陷,细胞增殖减少并增加细胞凋亡;且KDM7A缺失在分子调控层面导致Polycomb组蛋白(PcG)介导的组蛋白H2A泛素化下调及细胞周期蛋白激酶抑制剂p21Cip1上调。但KDM7A在牦牛生殖过程中的研究仍较少。早期报道探究KDM7A在癌症细胞中的表达调控状况[5],近2 a出现少量研究报道关于KDM7A在哺乳动物生殖过程中的影响,但对牦牛生殖过程的影响仍未可知[10,26]。因此,探讨KDM7A在牦牛及其生殖过程中的表达模式及分子机制尤为重要,具有广泛深层研究价值。
本试验利用RT-PCR技术首次成功克隆获得牦牛KDM7A基因CDS区序列。通过进行同源性比对分析发现,KDM7A在不同物种之间同源性较高,同时发现牦牛、黄牛和野牛该核苷酸序列高度吻合,且KDM7A与KDM2/7亚家族中的KDM2A都具有JmjC和PHD结构域[27],且该结构域从酵母到哺乳动物细胞高度保守。综上表明,KDM7A基因不易突变具有高度保守性,该基因编码蛋白的功能也高度保守,这在物种进化过程中使处于自然界的动物之间具有生存协调稳定性。
KDM7A在动物不同组织中发挥作用不尽相同[5,28-29]。本研究选用β-actin作为稳定的内参基因,利用RT-qPCR技术检测牦牛各组织中KDM7A mRNA的相对表达量水平。结果表明,KDM7A在牦牛各组织中广谱表达,且在子宫和胃中表达量较高,在肌肉和肾中表达量较低。该结果与已有研究存在相同点,有报道已证实KDM2A在牦牛子宫组织中相对表达量最高[27],推测KDM2/7亚家族中各个去甲基化酶在发育繁殖过程中具有协同作用的可能。已知KDM7A作为维持胚胎神经分化过程中重要调节因子,该基因在早期胚胎神经分化的早期阶段表达增加且具有时序性[13]。子宫是胚胎着床及胎儿发育重要场所,因此,推断母体子宫KDM7A mRNA相对表达量较高或与胚胎神经分化及胎儿发育有关,但该基因在子宫中具体分子调节机制有待进行深一步研究。
为了进一步探究KDM7A mRNA在牦牛卵母细胞减数分裂进程的表达规律。本研究利用RT-qPCR检测出KDM7A mRNA在牦牛不同时期卵母细胞中相对表达量呈上升趋势。KDM2A mRNA在牦牛卵母细胞减数分裂进程与KDM7A mRNA在牛卵母细胞成熟过程中表达趋势相同[27,30]。进一步推测KDM2/7亚家族中各成员在繁殖发育过程中或存在关联,但与王帅[30]研究KDM7A mRNA在牛不同卵母细胞中相对表达量差异性却不一致,可能是由于物种特异性及牦牛特殊生存环境所致,具体原因有待进行深入研究。Marinho等[29]结合细胞免疫荧光方法检测猪卵母细胞中H3K27的一、二、三甲基化程度,证实JHDMs动态作用于哺乳动物卵母细胞减数分裂进程中,促使卵母细胞发生减数分裂,其中H3K27me1和H3K27me2甲基化荧光信号呈下降趋势。本研究结果显示,KDM7A mRNA在牦牛卵母细胞减数分裂进程中呈上升趋势,且已知KDM7A作用于H3K27me2位点,因此,本研究KDM7A mRNA表达趋势与H3K27me2位点甲基化荧光信号减弱结果相一致。但KDM7A在卵母细胞中具体调控机制有待深入研究。
本研究首次成功克隆获得牦牛KDM7A基因序列,生物进化树等分析发现KDM7A在物种之间具有高保守性,且其在子宫中的表达量较高,在肌肉和肾中表达量较低;此外,KDM7A参与整个卵母细胞减数分裂过程,在不同时期卵母细胞表达量具有显著差异,但其在牦牛卵母细胞减数分裂进程中的具体分子机制有待进一步研究。以上研究结果为牦牛繁殖研究提供可靠的基础理论依据。
[1] 秦文昌,殷实,李泽沛,王斌,杨柳青,周婧雯,李键.牦牛HDAC8基因特征及其在组织和卵母细胞成熟过程中的表达分析[J].华北农学报,2020,35(2):210-216.doi:10.7668/hbnxb.20190532.
Qin W C, Yin S, Li Z P,Wang B, Yang L Q, Zhou J W, Li J.The characteristic of yak HDAC8 gene and analysis of their expression in different tissues and during the oocyte maturation[J].Acta Agriculturae Boreali-Sinica,2020, 35(2):210-216.
[2] Marcho C,Oluwayiose O A,Pilsner J R.The preconception environment and sperm epigenetics[J].Andrology,2020,8(4):924-942.doi:10.1111/andr.12753.
[3] Li X G.Epigenetics and cell cycle regulation in cystogenesis[J].Cellular Signalling,2020,68:109509.doi:10.1016/j.cellsig.2019.109509.
[4] 肖婷,周菊,傅俊江.组蛋白修饰作为表观遗传肿瘤标志物的研究进展[J].西南医科大学学报,2019,42(3):284-288.doi:10.3969/j.issn.2096-3351.2019.03.018.
Xiao T,Zhou J,Fu J J.Research progress in histone modifications as epigenetic tumor markers[J].Journal of Southwest Medical University,2019,42(3):284-288.
[5] 魏猛.组蛋白去甲基化酶KDM7在肝细胞癌中的表达及其临床意义[D].济南:山东大学,2014.
Wei M.Expression of KDM7 enzymes in human hepatocellular carcinoma and its clinical significance[D].Jinan:Shandong University,2014.
[6] Reid M A,Dai Z W,Locasale J W.The impact of cellular metabolism on chromatin dynamics and epigenetics[J].Nature Cell Biology,2017,19(11):1298-1306.doi:10.1038/ncb3629.
[7] Fellous A,Earley R L,Silvestre F.The Kdm/Kmt gene families in the self-fertilizing mangrove rivulus fish,Kryptolebias marmoratus,suggest involvement of histone methylation machinery in development and reproduction[J].Gene,2019,687:173-187.doi:10.1016/j.gene.2018.11.046.
[8] Horton J R,Upadhyay A K,Qi H H,Zhang X,Shi Y,Cheng X D.Enzymatic and structural insights for substrate specificity of a family of jumonji histone lysine demethylases[J].Nature Structural & Molecular Biology,2010,17(1):38-43.doi:10.1038/nsmb.1753.
[9] Yue W W,Hozjan V,Ge W,Loenarz C,Cooper C D O,Schofield C J,Kavanagha K L,Oppermann U,McDonoughc M A.Crystal structure of the PHF8 jumonji domain, an Nε-methyl lysine demethylase[J].Febs Letters,2010,584(4):825-830.doi:10.1016/j.febslet.2009.12.055.
[10] Huang C Y,Xiang Y,Wang Y R,Li X,Xu L Y,Zhu Z Q,Zhang T,Zhu Q Q,Zhang K J,Jing N H,Chen C L.Dual-specificity histone demethylase KIAA1718(KDM7A)regulates neural differentiation through FGF4[J].Cell Research,2010,20(2):154-165.doi:10.1038/cr.2010.5.
[11] Mallm J P,Windisch P,Biran A,Gal Z,Schumacher S,Glass R,Herold-Mende C,Meshorer E,Barbus M,Rippe K.Glioblastoma initiating cells are sensitive to histone demethylase inhibition due to epigenetic deregulation[J].Internitional Journal of Cancer,2020,146(5):1281-1292.doi:10.1002/ijc.32649.
[12] Meng Z Z,Liu Y,Wang J,Fan H J,Fang H,Li S,Yuan L,Liu C C,Peng Y,Zhao W W,Wang L L,Li J,Feng J.Histone demethylase KDM7A is required for stem cell maintenance and apoptosis inhibition in breast cancer[J].Journal of Cellular Physiology,2020,235(2):932-943.doi:10.1002/jcp.29008.
[13] Huang C Y,Chen J,Zhang T,Zhu Q Q,Xiang Y,Chen C D,Jing N H.The dual histone demethylase KDM7A promotes neural induction in early chick embryos[J].Develomental Dynamics,2010,239(12):3350-3357.doi:10.1002/dvdy.22465.
[14] Kujirai T,Kurumizaka H.Transcription through the nucleosome[J].Current Opinion in Structural Biology, 2020,61:42-49.doi:10.1016/j.sbi.2019.10.007.
[15] 杨勤,柴志欣,王吉坤,王会,钟金城.牦牛ACTA1基因启动子区克隆、DNA甲基化与组织表达相关性分析[J].华北农学报,2020,35(1):222-229.doi:10.7668/hbnxb.20190124.
Yang Q,Chai Z X,Wang J K,Wang H,Zhong J C.Cloning of ACTA1 gene promoter region and correlation analysis between DNA methylation and tissue expression in yak[J].Acta Agriculturae Boreali-Sinica,2020,35(1):222-229.
[16] Thakali K M,Zhong Y,Cleves M,Andres A,Shankar K.Associations between maternal body mass index and diet composition with placental DNA methylation at term[J].Placenta,2020,93:74-82.doi:10.1016/j.placenta.2020.02.018.
[17] Bird A.DNA methylation: mega-year inheritance with the help of darwin[J].Current Biology,2020,30(7):319-321.doi:10.1016/j.cub.2020.02.034.
[18] Trojer P,Reinberg D.Histone lysine demethylases and their impact on epigenetics[J].Cell,2006,125(2):213-217.doi:10.1016/j.cell.2006.04.003.
[19] Yang W N,Jiang D H,Jiang J F, He Y H.A plant-specific histone H3 lysine 4 demethylase represses the floral transition in Arabidopsis[J].The Plant Journal,2010,62(4):663-673.doi:10.1111/j.1365-313X.2010.04182.x.
[20] Jeong J H,Song H R,Ko J H,Jeong Y M,Kwon Y E,Seol J H,Amasino R M,Noh B,Noh Y S.Repression of flowering locust chromatin by functionally redundant histone H3 lysine 4 demethylases in Arabidopsis[J].PLoS One,2009,4(11):e8033.doi:10.1371/journal.pone.0008033.
[21] Lin C H,Li B,Swanson S,Zhang Y,Florens L,Washburn M P,Abmayr S M,Workman J L.Heterochromatin protein 1a stimulates histone H3 lysine 36 demethylation by the Drosophila KDM4A demethylase[J].Molecular Cell,2008,32(5):696-706.doi:10.1016/j.molcel.2008.11.008.
[22] Sánchez-fernández E M,Tarhonskaya H,Al-Qahtani K,Hopkinson R J,Mccullagh J S O,Schofield C J,Flashman E.Investigations on the oxygen dependence of a 2-oxoglutarate histone demethylase[J].Biochemical Journal,2013,449(2):491-496.doi:10.1042/BJ20121155.
[23] Chen H B,Costa M.Iron-and 2-oxoglutarate-dependent dioxygenases: an emerging group of molecular targets for nickel toxicity and carcinogenicity[J].BioMetals,2009,22(1):191-196.doi:10.1007/s10534-008-9190-3.
[24] Kim J,Singh A K,Takata Y,Lin K,Shen J J,Lu Y,Kerenyi M A,Orkin S H,Chen T P.LSD1 is essential for oocyte meiotic progression by regulating CDC25B expression in mice[J].Nature Communications,2015,6(6):10116.doi:10.1038/ncomms10116.
[25] Kawakami E,Tokunaga A,Ozawa M,Sakamoto R,Yoshida N.The histone demethylase Fbxl11/Kdm2a plays an essential role in embryonic development by repressing cell-cycle regulators[J].Mechanisms of Development,2015,135(135):31-42.doi:10.1016/j.mod.2014.10.001.
[26] Rissi V B,Glanzner W G,De Macedo M P,Gutierrez K,Baldassarre H,Gonçalves P B D,Bordignon V.The histone lysine demethylase KDM7A is required for normal development and first cell lineage specification in porcine embryos[J].Epigenetics,2019,14(11):1088-1101.doi:10.1080/15592294.2019.1633864.
[27] 杨显英,熊显荣,蔡雯祎,韩杰,阿果约达,李键.牦牛KDM2A基因克隆及其在不同组织和减数分裂进程的表达[J].农业生物技术学报,2018,26(7):1186-1194.doi:10.3969/j.issn.1674-7968.2018.07.010.
Yang X Y,Xiong X R,Cai W Y,Han J,Aguo Y D,Li J.Cloning of yak(Bos grunniens)KDM2A gene and its expression in different tissues and mitotic progression[J].Journal of Agricultural Biotechnology,2018,26(7):1189-1194.
[28] Meng Z Z,Liu Y,Wang J,Fan H J,Fang H,Li S,Yuan L,Liu C C,Peng Y,Zhao W W,Wang L L,Li J,Fang J.Histone demethylase KDM7A is required for stem cell maintenance and apoptosis inhibition in breast cancer[J].Journal of Cellular Physiology,2020,235(2):932-943.doi:10.1002/jcp.29008.
[29] Marinho L S R,Rissi V B,Lindquist A G,Seneda M M,Bordignon V.Acetylation and methylation profiles of H3K27 in porcine embryos cultured in vitro[J].Zygote,2017,25(5):575-582.doi:10.1017/S0967199417000405.
[30] 王帅.牛HDM家族成员在卵母细胞中的表达规律及JMJD1C在早期胚胎发育中的功能[D].长春:吉林大学,2014.
Wang S.Expression pattern of HDM family members in bovine oocytes and function of JMJD1C in early embryo development[D].Changchun: Jilin University,2014.