中国是世界上第三大牛肉生产国和消费国,随着人们的生活水平不断提高,中国居民牛肉消费持续增加。牛肉品质的优劣直接影响到适口性,为了迎合大众口味,满足大众需求,提高牛肉品质是当前育种中亟待解决的问题,而研究脂肪沉积相关基因的功能和调控机制对改善肉质至关重要。安格斯牛和中国西门塔尔牛是具有不同脂肪沉积能力的2个品种,安格斯牛的背膘厚度显著高于中国西门塔尔牛[1],剪切力显著低于中国西门塔尔牛[2],肌内脂肪(Intramuscular fat, IMF)含量显著高于中国西门塔尔牛[3]。中国西门塔尔牛于20世纪80年代引入中国,是乳、肉、役兼用的全能牛,经育种改良的中国西门塔尔牛是科尔沁主要肉牛品种,在我国东北部、南部以及西部地区均有分布。中国西门塔尔牛肌肉丰满,生长速度较快,胴体肉多,脂肪少而分布均匀,屠宰率可达65%,相对于黄牛、荷斯坦牛和蒙古牛来说,西门塔尔牛在屠宰率、瘦肉率及肉用性能方面具有显著的优势,但其肉质要稍逊色于世界上其他著名的肉牛品种[4]。安格斯牛是20世纪70年代引入中国,经长期育种后,分布在新疆、内蒙古、东北、山东等地,被毛的颜色主要为黑色,其次为黑棕色或红色,身材矮小,耐寒能力、耐粗饲和抗病能力都很强。安格斯牛早熟、肉质质量好、净肉率高、大理石花纹明显,屠宰率达60%~65%,许多国家及地区常用安格斯牛作为母本对地方牛进行杂交来改良品种[5]。
脂肪沉积能力是牛肉品质的评价指标之一,影响脂肪沉积能力的因素有很多,如遗传因素、生长环境和营养水平等,其中遗传因素占据主导地位。目前发现参与脂肪代谢的分子主要有脂肪酸结合蛋白、脂肪酸合成酶、黑色素皮质素受体和脂联素等。另外,许多激素和细胞因子也参与脂质代谢,如胰岛素、糖皮质激素和成脂因子等,上述分子基因的多态性与脂肪沉积的相关性已经通过试验得到了证实,但目前关于安格斯牛和中国西门塔尔牛脂肪沉积的关键基因及调节机制的研究报道却很少。
为了研究与脂肪沉积相关的基因,本试验以安格斯牛和中国西门塔尔牛作为研究对象,对其背膘组织进行转录组测序,通过GO富集和KEGG代谢通路的分析,结合Cytoscape等分析与脂肪沉积有关的基因,并对与脂肪沉积相关的3个基因DKKL1、ACACB和PTGS2进行了qRT-PCR的验证,以期找出调控脂质代谢的候选基因,为中国西门塔尔牛和安格斯牛的后续选育提供科学依据。
1 材料和方法
1.1 试验材料
安格斯牛和中国西门塔尔牛都来自内蒙古通辽市余粮畜业育肥牛场,屠宰前牛场按照日粮标准进行集中统一饲喂,且都为阉牛,育肥期为60 d。肉牛样本是在统一条件下进行采集,及时取样,液氮保存。进行转录组测序的6头牛是通过聚类分析挑选的,能够很好地反映总体水平。
1.2 转录组测序及新转录本预测
TRIzol法提取RNA后用Nano-Drop2000测定RNA浓度。检测合格后按照说明建库后利用BGISEQ-500平台对安格斯牛和中国西门塔尔牛背膘样品的RNA测序并获得转录组数据。使用SOAPnuke软件对所得数据进行过滤,去除低质量reads以确保数据的准确性。
与参考基因组比对之后,先使用StringTie[6]对每个样品进行转录本重构,再使用Cuffmerge(Cufflinks的工具之一)将所有样品的重构信息整合在一起,最后使用Cuffcompare[7]将重构的转录本与参考注释信息比较得到新转录本。使用CPC[8]对新转录本进行蛋白编码潜力预测,最终将预测具有蛋白编码潜力的新转录本加入到参考基因序列中,得到一个完整的参考序列信息,后续将基于此参考序列进行分析。
1.3 SNP、差异剪接基因的检测
根据参考基因组比对结果,利用GATK[9]检测每个样品的SNP信息。通过比对基因组数据、标记重复序列、分割序列和重校准碱基对数据进行预处理。然后用HaplotypeCaller检测SNP。使用rMATS[10]检测不同样品之间的差异剪接基因(DSG)和样品自身的差异剪接基因,以FDR≤0.05为判断标准,用DEGseq[11]法进行差异基因的检测。并将P-values矫正为q values。为了提高DEGs的准确性,将差异倍数为2倍以上并且q value≤0.001的基因,筛选为显著差异表达基因。
1.4 GO功能注释和KEGG通路分析
对转录组数据中的差异表达基因进行GO(Gene Ontology)功能注释和KEGG通路富集分析。过滤后的Clean reads保存为FASTQ格式根据差异基因检测结果,对其GO功能进行分类以及富集分析。
根据GO注释结果以及官方分类,将差异基因进行功能分类,同时使用R软件中的phyper函数进行富集分析。然后对P-value进行FDR校正,通常FDR≤0.01的功能视为显著富集。
生物体内的生物学行为是由多种蛋白相互协调的,Pathway分析是基于公共数据库KEGG对生物学功能的进一步了解,可以初步了解蛋白质的功能,还有助于验证和阐明蛋白质的生物调控机制[12]。KEGG提供了通过Pathway作图过程将基因组与生命联系起来的参考知识库,可以将基因的基因组或转录组内容物映射到KEGG参考途径以推断细胞或生物体的系统行为[13]。KEGG资源提供了将基因组与生物系统连接起来的参考知识库,分类为基因组空间(KEGG GENES)和化学空间(KEGG LIGAND)中的构建块包括内源分子和外源分子,以及受体互作和反应网络(Pathway)的接线图以及计算机化的KeggBrite[12]。
1.5 荧光定量分析
荧光定量分析的主要目的是验证转录组数据的差异表达基因,首先提取样本总RNA并通过Nanodrop对RNA含量进行检测,然后将RNA逆转录成cDNA并对其进行凝胶电泳检测,配制25.0 μL的反应体系:SYBR 12.5 μL,上下游引物各0.5 μL,cDNA模板2.0 μL,去离子水9.5 μL;配好后将样品加到96孔板中,然后以4 000 r/min的转速离心30 s,最后放到已做好设定的荧光定量PCR仪中。qRT-PCR可以对差异表达基因的表达量进行分析。
2 结果与分析
2.1 测序数据分析
2.1.1 测序数据过滤 过滤后的reads质量统计结果见表1,通过BGISEQ-500平台对来自中国西门塔尔和安格斯牛的背膘的6个样品进行转录组测序,过滤前reads数为120.36 Mb。安格斯牛每个样品平均产出10.95 Gb,中国西门塔尔牛平均产出10.96 Gb。过滤后reads占比在安格斯牛为90.98%,中国西门塔尔牛为91.05%。数据过滤后,Q20(质量大于20的碱基数目占总碱基数目的比例)安格斯牛为97.49%,中国西门塔尔牛为97.56%,都大于97.00%,Q30安格斯牛为90.22%,中国西门塔尔牛为90.36%,都大于90.00%,表明测序结果良好,符合建库要求。
表1 过滤后的reads质量
Tab.1 Clean reads quality
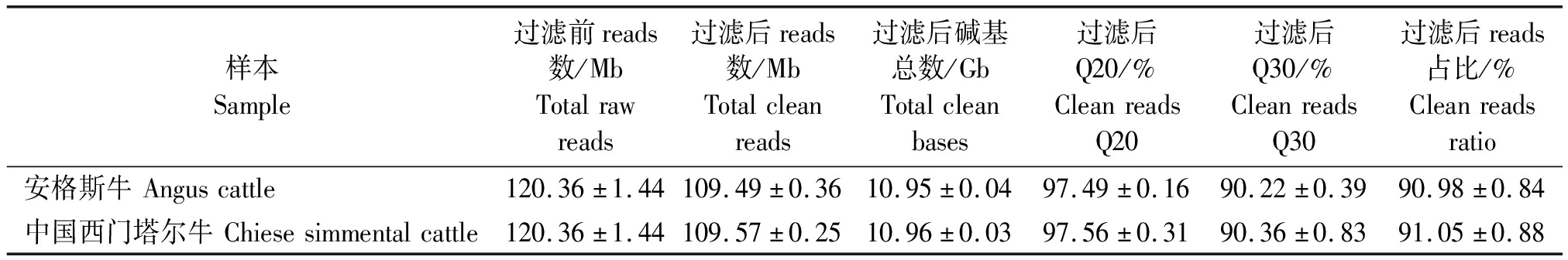
样本Sample过滤前reads数/MbTotalrawreads过滤后reads数/MbTotalcleanreads过滤后碱基总数/GbTotalcleanbases过滤后Q20/%CleanreadsQ20过滤后Q30/%CleanreadsQ30过滤后reads占比/%Cleanreadsratio安格斯牛Anguscattle120.36±1.44109.49±0.3610.95±0.0497.49±0.1690.22±0.3990.98±0.84中国西门塔尔牛Chiesesimmentalcattle120.36±1.44109.57±0.2510.96±0.0397.56±0.3190.36±0.8391.05±0.88
2.1.2 参考基因组比对 reads对基因的比对率统计见表2,使用HISAT[14]将Clean reads与参考基因组序列比对,结果显示,样品比对基因组的总比对率安格斯牛为92.19%,中国西门塔尔牛为92.47%,比对基因集的唯一比对率为安格斯牛为70.56%,中国西门塔尔牛为71.47%。同时样品间均匀的比对率表明,样品之间的数据具有可比性。
表2 参考基因组比对结果
Tab.2 Summary of genome mapping

样品Sample过滤后的reads总数Totalcleanreads总比对率/%Totalmappingratio唯一比对率/%Uniquelymappingratio安格斯牛Anguscattle109493996.00±363342.0492.19±0.5470.56±2.41中国西门塔尔牛Chinesesimmentalcattle109572867.00±246782.4592.47±0.6171.47±2.00
2.1.3 新转录本预测 与参考基因组比对之后,将转录本重构,与参考注释信息比较得到新转录本。共计得到新转录本19 747个,其中预测的16 576个为编码的转录本数,15 643个属于已知蛋白编码基因新的可变剪接亚型,933个属于新的蛋白编码基因的转录本,剩下的3 171个属于LncRNA。
2.2 SNP检测结果
与参考基因组比对之后使用GATK检测每个样品的SNP[9],SNP统计类型见表3。安格斯牛的A-G和C-T变异的SNP数目158 116个,A-C、A-T、C-G 和 G-T 变异的SNP数目为60 349个,所有变异类型的总数目为218 465个。中国西门塔尔牛的A-G和C-T变异的SNP数目179 321个,A-C、A-T、C-G 和 G-T 变异的SNP数目为68 589个,所有变异类型的总数目为247 910个。安格斯牛的变异的SNP数目普遍低于中国西门塔尔牛。
表3 SNP类型统计
Tab.3 Statistics of SNP variant type
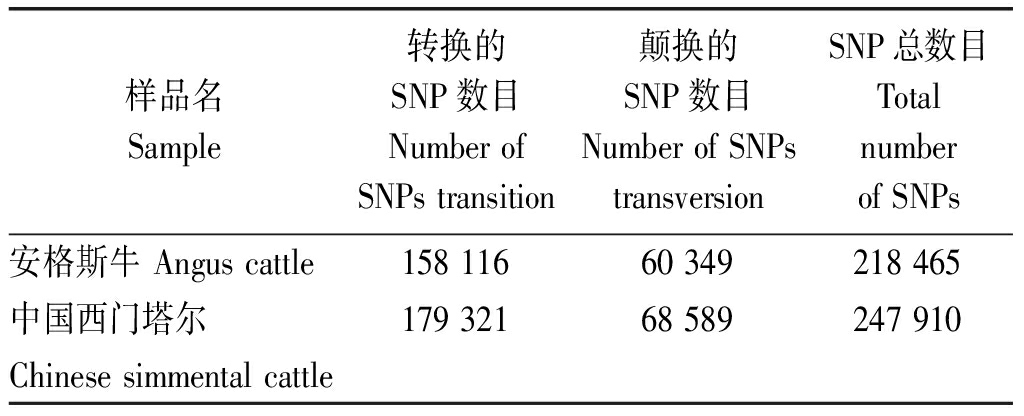
样品名Sample转换的SNP数目NumberofSNPstransition颠换的SNP数目NumberofSNPstransversionSNP总数目TotalnumberofSNPs安格斯牛Anguscattle15811660349218465中国西门塔尔17932168589247910Chinesesimmentalcattle
2.3 差异表达基因的筛选和功能注释
根据各个样品基因表达水平,检测样品之间的差异表达基因。中国西门塔尔牛对安格斯牛的差异表达基因的上下调关系见图1,中国西门塔尔牛对安格斯牛差异基因表达的分析中有1 296个差异表达基因,其中上调基因802个(Fold change≥2,q≤0.001),下调基因494个(Fold change≤-2,q≤0.001),即中国西门塔尔牛上调表达基因数高于安格斯牛的上调表达基因数。
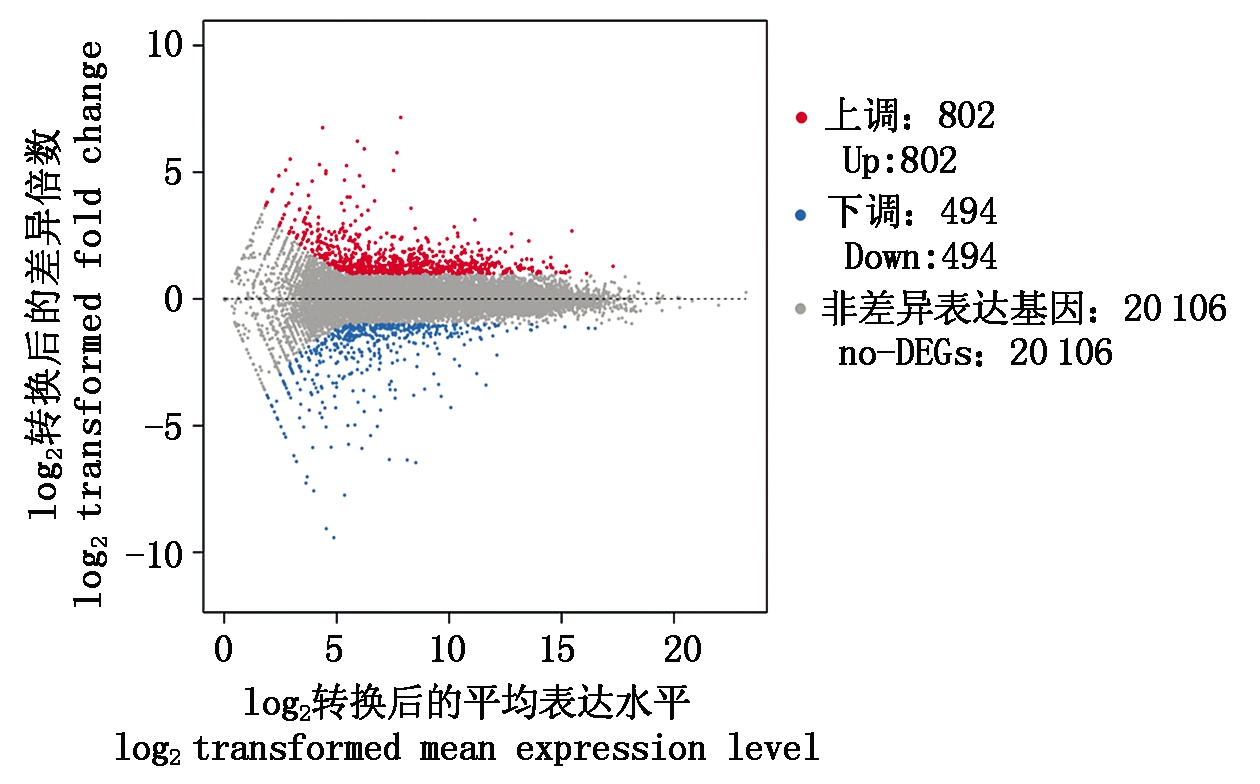
图1 DEGs的Scatter-plot分布
Fig.1 Scatter-plot of DEGs
根据差异基因检测结果,对所得数据进行进一步的分类及富集分析,由图2可看出,对筛选的6 244个基因进行GO功能分类注释和富集,其中2 205个DEGs注释到26个生物学进程(Biologicalprocess,BP)中,3 149个DEGs注释到15个细胞组分(Celluar component, CC)中,890个DEGs注释到12个分子功能(Molecular function, MF)中。进一步的分类和富集分析显示富集比较靠前且超过300个DEGs,BP中的细胞进程398个,占比6.37%;单细胞进程369个,占比5.91%;CC中的细胞组分396个,占比6.34%;膜组分394个,占比6.31%;MF中的结合功能380个,占比6.09%。
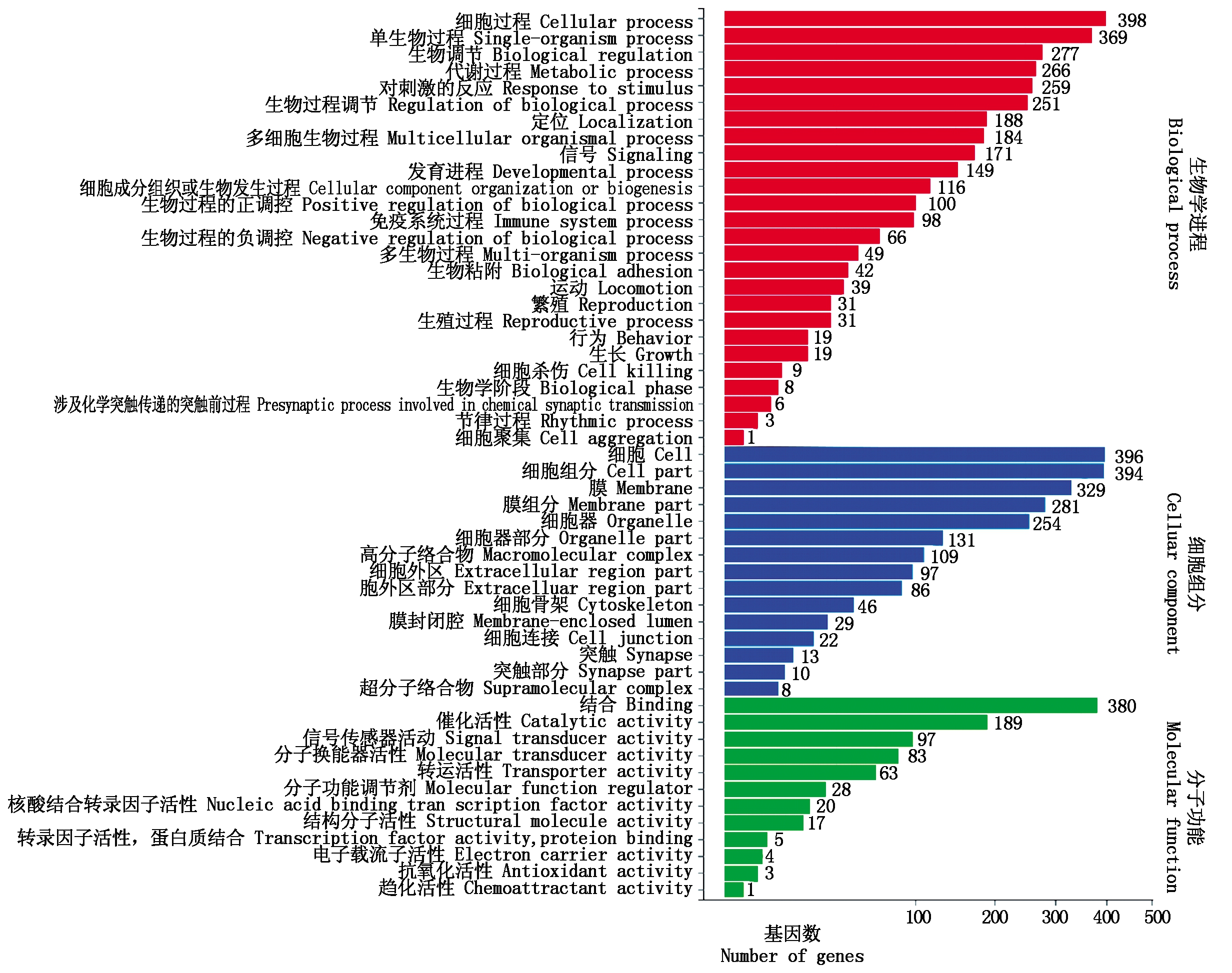
图2 差异基因GO功能分类
Fig.2 GO classification of DEGs
根据差异基因检测结果,对其进行KEGG生物通路分类以及富集分析,结果见图3,通过KEGG代谢通路分析牛背膘组织的Unigene参与了6大类共44小类的代谢通路,并且一共注释到了2 158个基因。其中基因数量排名靠前的代谢通路主要包括人类疾病通路,注释了616个基因,占比28.54%,有机系统注释了586个基因,占比27.15%,然后依次为环境信息进程、新陈代谢通路、细胞进程以及遗传信息进程,其依次占比为19.00%,12.00%,9.87%,3.43%。
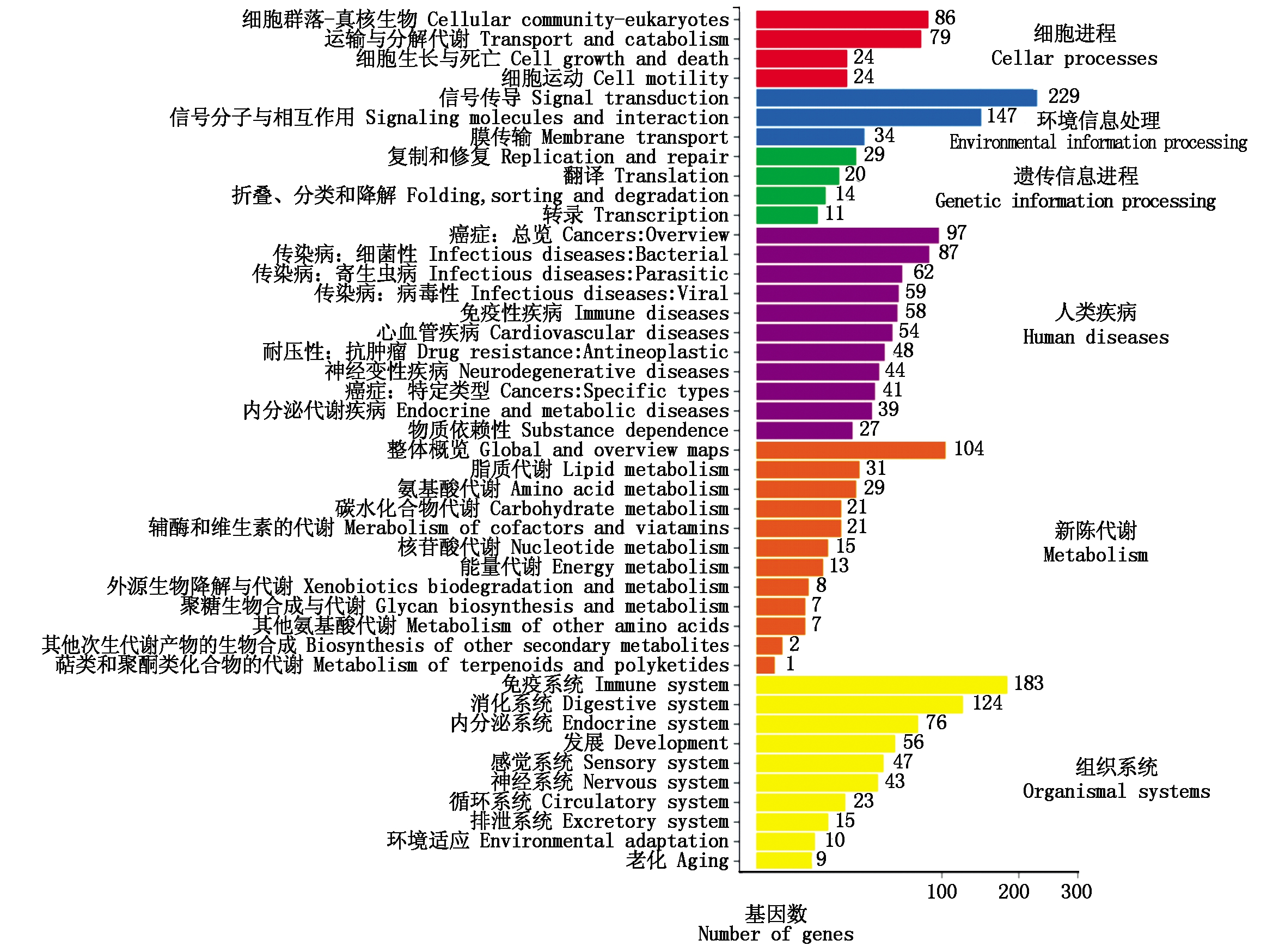
图3 差异基因Pathway分类
Fig.3 Pathway classification of DEGs
对差异表达基因的数据进行了Pathway富集,由图4可知在cAMP信号通路、神经活性配体-受体互作以及细胞因子受体互作3个通路上基因的富集程度最高,即数量最多且富集结果显著。

图4 差异基因Pathway富集分析
Fig.4 Pathway functional enrichment of DEGs
2.4 差异表达基因DKKL1、ACACB及PTGS2的荧光定量验证
图5为脂肪相关基因DKKL1、ACACB及PTGS2在转录组数据中表达量与qRT-PCR相对表达量的对比。荧光定量结果DKKL1、ACACB在中国西门塔尔牛的背膘中的相对表达量高于安格斯牛的背膘中的相对表达量(2-(ΔΔCt)>1),PTGS2在安格斯牛背膘中相对表达量高(2-(ΔΔCt)<1),差异表达模式均与RNA-seq表达模式一致,表明转录组数据可靠。可见DKKL1和ACACB在中国西门塔尔牛中表达量较高,PTGS2在安格斯牛体内表达较高。
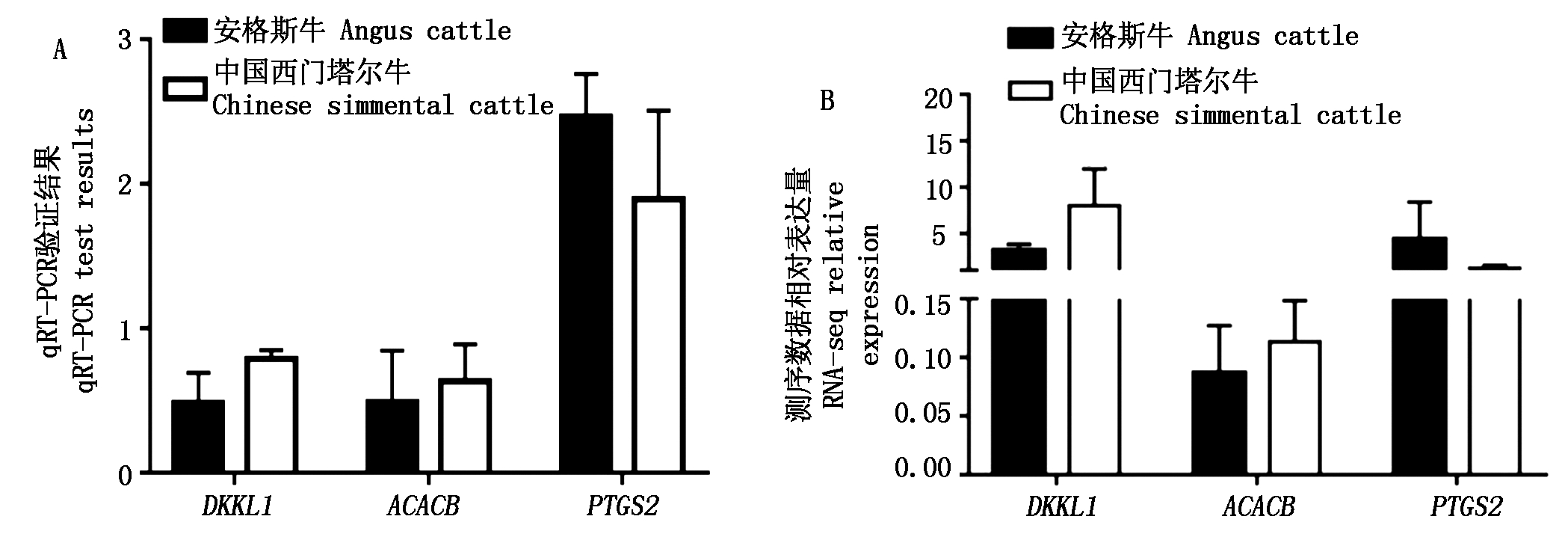
图5 DKKL1、ACACB及PTGS2的qRT-PCR中相对表达量(A)及RNA-seq表达量(B)
Fig.5 The expression levels of qRT-PCR(A)and RNA-seq(B)of DKKL1,ACACB and PTGS2
3 讨论与结论
在初期屠宰试验中得到中国西门塔尔牛剪切力显著高于安格斯牛,且安格斯牛背膘脂肪厚度显著高于中国西门塔尔牛(P<0.01)[1]。IMF含量与剪切力呈负相关,与嫩度呈正相关[15]。2个品种牛在脂肪沉积上有显著差异,故候选基因的筛选具有一定的意义。
本研究通过BGISE-500对被背膘组织进行转录组测序,并对测序数据进行富集和注释,从中找到与脂肪沉积相关功能和通路。从图 4中可以看出注释到cAMP信号通路中的差异基因数目最多且富集结果较为显著。同时cAMP能促进动物机体内脂肪的分解代谢[16],但可降低肉中硬脂酸含量,增加不饱和脂肪酸含量,增加犊牛肉中脂肪含量[17]。线粒体内由二氧化碳/碳酸氢盐调节的可溶性cAMP激活蛋白激酶A(PKA)使线粒体蛋白磷酸化从而调节ATP的产生[18]。cAMP增加会激活cAMP依赖蛋白激酶,使激素敏感脂酶(HSL)磷酸化进而催化甘油三酯水解为甘油和脂肪酸[19]。转变成酰基CoA,而后进入β-氧化和三羧酸循环(TCA)氧化产热,游离脂肪酸(FFA)可使线粒体进入解偶联状态,底物氧化耗能增强,最终导致脂肪分解加强[20]。
依据|log2(Fold chage)|≥1,q≤0.001对脂肪沉积表达差异基因进行筛选,初步筛选出3种基因DKKL1、ACACB及PTGS2。DKKL1(Dickkopf like acrosomal protein 1,Soggy)阻黑体,类似顶体蛋白1抑制物,位于牛的18号染色体,有7个外显子,长度为985 bp,编码233个氨基酸,属于分泌性蛋白,是Wnt抑制剂小蛋白家族Dickkopf的成员。DKKs编码的分泌蛋白通常通过阻止配体-受体相互作用[21]或抑制Wnt辅助受体LRP5/6来拮抗Wnt/β-catenin信号转导,可局部地抑制Wnt调节的过程而达到促进脂肪沉积的功能。Mao等[22]的研究认为DKK1通过Krm蛋白的降解,破坏Wnt蛋白配体与Fz-LRP6形成的受体复合物,从而抑制Wnt信号通路。研究显示DKK1、DKK2、DKK3及DKK4都可以抑制Wnt通路的传导,促进脂肪沉积。DKKL1虽是Wnt家族的一员,但与DKK1, DKK2和DKK4不同[23],尽管与DKK3拥有相同的Sgy结构域,但是却没有2个富含Cys的LRP相互作用结构域[24],同时也有研究表明,非特异性通路中,Wnt只与Fz作用,而不需要LRP5/6。Dakhova等[24]研究显示DKKL1可以通过负调节类固醇生成酶来调控睾酮的生成。睾酮可以降低胆固醇和体脂,但公阉牛体内的睾酮含量低于公牛,其作用机制和调节水平还有待进一步确定。因此,差异表达基因DKKL1对脂肪沉积的作用及在2个品种牛中的表达机制还需要后续试验的验证。
ACACB(Acetyl-CoA carboxylase beta,ACC2)乙酰辅酶A羧化酶β基因,位于牛的17号染色体,有56个外显子,全长8 924 bp,共编码2 431个氨基酸。ACC包含2种,ACC1和ACC2,其中ACC1位于胞质,主要参与脂肪的从头合成,ACC2主要存在于线粒体外膜,主要作用于脂肪酸的氧化。它们具有70%相同的酶活性和同源性。在AMPK信号通路当中,AMPK会抑制ACACA和ACACB的活性,而它们的作用是催化乙酰辅酶A(CoA)羧化为丙二酰辅酶A,丙二酰辅酶A通过抑制肉毒碱棕榈酰基转移酶1(CPT1)而抑制脂酰肉碱向线粒体内的转运从而导致游离脂肪酸增加[25],其中丙二酰辅酶A通常由线粒体外膜中的ACC2产生[26]。ACC2位于线粒体外膜,主要参与脂肪酸细胞氧化。在不同种属中脂肪酸调节也有差异,ACC2含量也不同。Oh等[26]认为ACC2将成为治疗肥胖和相关代谢疾病(如高脂血症、胰岛素抵抗和糖尿病)的靶基因。敲除ACACB会导致因为ACACB产生的Malonyl-COA水平的降低,进而导致脂肪酸降低,脂肪含量降低[27]。高丽南等[28]研究显示,不同饲料营养以及不同品种肉牛中AMPK的含量不同,其中,中国西门塔尔牛的AMPK含量较高。AMPK脂肪酸氧化中起着重要作用,即将脂肪分解为游离的脂肪酸。在肌肉中脂肪酸氧化的提高与明显降低的丙二酸单酰辅酶A水平有关,ACC2调控脂肪酸氧化,在肝脏中丙二酸单酰辅酶A水平就没有变化,这表明ACC2必须在一个没有ACC1产生的丙二酸单酰辅酶A的区域才能发挥作用。ACACB的作用机制和调节水平还有待进一步确定,差异表达基因ACACB对脂肪沉积的作用及在2个品种牛中的表达机制还需要后续试验的验证。
PTGS2(Prostaglandin-endoperoxide synthase 2,COX2)前列腺素内过氧化物合酶2,也称环氧化酶-2。位于牛的16号染色体,含有10个外显子,全长3 489 bp,共编码604个氨基酸。COX2是一种催化花生四烯酸转变成前列腺素前体(PGH2)的限速酶,是线粒体编码蛋白,也是生物活性脂类调节物[29]。在脂肪细胞分解调节通路(Regulation of lipolysis in adipocytes)中COX2会间接促进前列腺素E2(Prostaglandin E2,PGE2)的表达,PGE2在前列腺受体EP3的作用下会促进抑制性蛋白(Gi)的表达,从而抑制腺苷酸环化酶(Adenylatecyclase,AC)的表达,从而导致cAMP信号通路激活受阻,最终促进脂肪沉积。PTGS2的表达以体质量指数依赖的方式增加,并且与肥胖程度密切相关。COX2在成熟脂肪细胞中的表达受到被噻唑烷二酮特异性靶向的PPARγ2的影响而减少[30]。PPARγ可改善皮下脂肪细胞的形态,并降低了促炎蛋白COX2的表达[31]。Belda等[32]在研究10e12z-cla对3t3-l1细胞分化和完全分化的影响。10e12z-cla在整个分化期都能降低3t3-l1脂肪细胞分化的程度。这些变化仅在分化过程的中途才变得明显,并伴随着COX2表达的增加而发生,COX2表达是NF-κB活性的标志。10e12z-cla异构体抑制脂肪分化并诱导人脂肪细胞中的NF-κB转录激活。在体外完全分化的人脂肪细胞中,10e12z-cla也能诱导环氧合酶-2[32]。10e12z-cla在3t3-l1前脂肪细胞分化过程中显著抑制脂肪细胞表型并诱导炎症标志物COX2的表达[33]。但Yang等[34]在糖皮质激素相关的脂肪细胞分化的研究中发现E4bp4E4(启动子结合蛋白4)能够通过反式抑制PTGS2而促进脂肪沉积,而Fain等[35]的研究显示,COX2与抗脂肪性前列腺素的生成具有功能耦合。该原因可能是COX2与抗脂肪性前列腺素的生成具有功能耦合,另一方面,在分化期24 h后,AA的补充导致了COX2转录水平的显著降低,也可能是与脂肪细胞的功能调节有关。因此,PTGS2作为脂肪沉积的候选基因但还需更多试验的验证。
综上,可初步确定DKKL1、ACACB及PTGS2为脂肪沉积的候选基因,对安格斯牛和中国西门塔尔牛脂肪沉积的后续研究和肉牛育种工作具有一定的积累意义和参考价值。
[1] 王国富, 吴慧光, 赵新海, 季守财, 吴红君, 王东升, 刘胜敏, 高树新.安格斯牛、海福特牛和中国西门塔尔牛的部分胴体性状比较分析[J].内蒙古民族大学学报(自然科学版), 2010,25(5):535-537.doi:10.3969/j.issn.1671-0185.2010.05.016.
Wang G F, Wu H G, Zhao X H,Ji S C, Wu H J, Wang D S, Liu S M, Gao S X.Comparative study on some carcass traits in Angus,Hereford and Chinese Simmental cattles[J].Journal of Inner Mongolia University for Nationalities(Natural Sciences), 2010,25(5):535-537.
[2] 王小梅.内蒙古地区不同品种肉牛生产性能和肉品质及脂肪代谢相关基因mRNA表达量的比较研究[D].呼和浩特:内蒙古农业大学, 2012.
Wang X M.Comparative study on different breed beef cattle′s production,meat quality and mRNA expression of fatty metabolism related factors in Inner Mongolia[D].Hohhot:Inner Mongolia Agricultural University, 2012.
[3] 张国梁, 刘基伟, 胡成华, 吴健, 张嘉保.西门塔尔、夏洛来、安格斯杂交牛不同营养水平的育肥效果分析[J].中国畜牧兽医, 2007,34(7):10-12.doi:10.3969/j.issn.1671-7236.2007.07.002.
Zhang G L, Liu J W, Hu C H, Wu J, Zhang J B.Fattening effect analysis of Simmental, Charolais and Angus crossbred cattle at different nutritional levels[J].China Animal Husbandry & Veterinary Medicine, 2007,34(7): 10-12.
[4] 史新平, 陈燕, 徐玲, 金生云, 宝金山, 牛红, 王泽昭, 李俊雅, 孙少华.肉用西门塔尔牛与和牛杂交群体的肉品质分析[J].中国畜牧兽医, 2018,45(4):953-960.doi:10.16431/j.cnki.1671-7236.2018.04.05.
Shi X P,Chen Y,Xu L,Jin S Y,Bao J S,Niu H,Wang Z Z,Li J Y,Sun S H.Analysis of beef Simmental and Wagyu hybrid cattle on meat quality[J].Chinese Animal Husbandry and Veterinary Medicine,2018,45(4):953-960.
[5] Mazzucco J P, Goszczynski D E, Ripoli M V, Melucci L M, Pardo A M, Colatto E, ![]() A, Mezzadra C A, Depetris G J, Giovambattista G, Villarreal E L.Growth, carcass and meat quality traits in beef from Angus, Hereford and cross-breed grazing steers, and their association with SNPs in genes related to fat deposition metabolism[J].Meat Science, 2016,114:121-129.doi:10.1016/j.meatsci.2015.12.018.
A, Mezzadra C A, Depetris G J, Giovambattista G, Villarreal E L.Growth, carcass and meat quality traits in beef from Angus, Hereford and cross-breed grazing steers, and their association with SNPs in genes related to fat deposition metabolism[J].Meat Science, 2016,114:121-129.doi:10.1016/j.meatsci.2015.12.018.
[6] Pertea M, Pertea G M, Antonescu C M, Chang T C, Mendell J T, Salzberg S L.StringTie enables improved reconstruction of a transcriptome from RNA-seq reads[J].Nature Biotechnology,2015,33(3):290-295.doi:10.1038/nbt.3122.
[7] Trapnell C, Roberts A, Goff L, Pertea G, Kim D, Kelley D R, Pimentel H, Salzberg S L, Rinn J L, Pachter L.Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks[J].Nature Protocols,2012,7(3):562-578.doi:10.1038/nprot.2012.016.
[8] Kong L, Zhang Y, Ye Z Q, Liu X Q, Zhao S Q, Wei L P, Gao G.CPC: assess the protein-coding potential of transcripts using sequence features and support vector machine[J].Nucleic Acids Research,2007,35(2):345-349.doi:10.1093/nar/gkm391.
[9] McKenna A, Hanna M, Banks E, Sivachenko A, Cibulskis K, Kernytsky A, Garimella K, Altshuler D, Gabriel S, Daly M, DePristo M A.The Genome Analysis Toolkit: a MapReduce framework for analyzing next-generation DNA sequencing data[J].Genome Research,2010,20(9):1297-1303.doi:10.1101/gr.107524.110.
[10] Shen S H, Park J W, Lu Z X, Lin L, Henry M D, Wu Y N, Zhou Q, Xing Y.rMATS: robust and flexible detection of differential alternative splicing from replicate RNA-Seq data[J].Proceedings of the National Academy of Sciences of the United States of America, 2014,111(51):5593-5601.doi:10.1073/pnas.1419161111.
[11] Wang L K, Feng Z X, Wang X, Wang X W, Zhang X G.DEGseq: an R package for identifying differentially expressed genes from RNA-seq data[J].Bioinformatics, 2010,26(1):136-138.doi:10.1093/bioinformatics/btp612.
[12] Kanehisa M, Goto S, Hattori M, Aoki-Kinoshita K F, Itoh M, Kawashima S, Katayama T, Araki M, Hirakawa M.From genomics to chemical genomics: new developments in KEGG[J].Nucleic Acids Research,2006,34(S1):354-357.doi:10.1093/nar/gkj102.
[13] Kanehisa M, Araki M, Goto S, Hattori M, Hirakawa M, Itoh M, Katayama T, Kawashima S, Okuda S, Tokimatsu T, Yamanishi Y.KEGG for linking genomes to life and the environment[J].Nucleic Acids Research,2008,36(S1):480-484.doi:10.1093/nar/gkm882.
[14] Kim D, Langmead B, Salzberg S L.HISAT: a fast spliced aligner with low memory requirements[J].Nature Methods,2015,12(4):357-360.doi:10.1038/nmeth.3317.
[15] Okeudo N J, Moss B W.Interrelationships amongst carcass and meat quality characteristics of sheep[J].Meat Science, 2005,69(1):1-8.doi:10.1016/j.meatsci.2004.04.011.
[16] Ravnskjaer K, Madiraju A, Montminy M.Role of the cAMP pathway in glucose and lipid metabolism[J].Handbook of Experimental Pharmacology,2016,233:29-49.doi:10.1007/164_2015_32.
[17] 李常青.外源性cAMP对秦川犊牛生长发育及调控机理的研究[D].杨凌:西北农林科技大学, 2010.
Li C Q.The effect on the growth and controt mechanism in Qinchuan calves by exogenous camp[D].Yangling:Northwest A&F University, 2010.
[18] Acin-Perez R, Salazar E, Kamenetsky M, Buck J, Levin L R, Manfredi G.Cyclic AMP produced inside mitochondria regulates oxidative phosphorylation[J].Cell Metabolism,2009,9(3):265-276.doi:10.1016/j.cmet.2009.01.012.
[19] Okuda H, Morimoto C, Tsujita T.Effect of substrates on the cyclic AMP-dependent lipolytic reaction of hormone-sensitive lipase[J].Journal of Lipid Research,1994,35(7):1267-1273.
[20] McNeel R L, Mersmann H J.β-adrenergic receptor subtype transcripts in porcine adipose tissue[J].Journal of Animal Science,1995,73(7):1962-1971.doi:10.2527/1995.7371962x.
[21] Cruciat C M, Niehrs C.Secreted and transmembrane wnt inhibitors and activators[J].Cold Spring Harbor Perspectives in Biology,2013,5(3):a015081.doi:10.1101/cshperspect.a015081.
[22] Mao B Y, Wu W, Davidson G, Marhold J, Li M F, Mechler B M, Delius H, Hoppe D, Stannek P, Walter C, Glinka A, Niehrs C.Kremen proteins are Dickkopf receptors that regulate Wnt/beta-catenin signalling[J].Nature, 2002,417(6889):664-667.doi:10.1038/nature756.
[23] Krupnik V E, Sharp J D, Jiang C, Robison K, Chickering T W, Amaravadi L, Brown D E, Guyot D, Mays G, Leiby K, Chang B, Duong T, Goodearl A D J, Gearing D P, Sokol S Y, McCarthy S A.Functional and structural diversity of the human Dickkopf gene family[J].Gene,1999,238(2):301-313.doi:10.1016/s0378-1119(99)00365-0.
[24] Dakhova O, O′Day D, Kinet N, Yucer N, Wiese M, Shetty G, Ducy P.Dickkopf-like1 regulates postpubertalspermatocyte apoptosis and testosterone production[J].Endocrinology,2009,150(1):404-412.doi:10.1210/en.2008-0673.
[25] 李亮, 程彦伟.乙酰辅酶A羧化酶在治疗肥胖中的潜在作用[J].生命的化学, 2007,27(2):180-182.doi:10.3969/j.issn.1000-1336.2007.02.030.
Li L, Cheng Y W.The potential effects of acetyl-CoA carboxylase in curing obesity[J].Chemistry of Life, 2007,27(2):180-182.
[26] Oh W, Abu-Elheiga L, Kordari P, Gu Z W, Shaikenov T, Chirala S S, Wakil S J.Glucose and fat metabolism in adipose tissue of acetyl-CoA carboxylase 2 knockout mice[J].Proceedings of the National Academy of Sciences of the United States of America,2005,102(5):1384-1389.doi:10.1073/pnas.0409451102.
[27] Abu-Elheiga L, Oh W, Kordari P, Wakil S J.Acetyl-CoA carboxylase 2 mutant mice are protected against obesity and diabetes induced by high-fat/high-carbohydrate diets[J].Proceedings of the National Academy of Sciences of the United States of America, 2003,100(18):10207-10212.doi:10.1073/pnas.1733877100.
[28] 高丽南, 王思珍, 曹颖霞, 朱晓岩, 刘国文.营养与品种对肉牛血清中AMPK活性与血液生化指标关系的研究[J].中国畜牧杂志.2012,48(9):29-33.doi,10.3969/j.issn.0258-7033.2012.09.008.
Gao L N, Wang S Z, Cao Y X, Zhu X Y, Liu G W.The effects of nutrition and bread on AMPK activity in serum and relation ship between AMPK activity and blood indicators[J].Chinese Journal of Animal Science,2012,48(9):29-33.
[29] Warner T D, Mitchell J A.Cyclooxygenase-3(COX-3): filling in the gaps toward a COX continuum[J].Proceedings of the National Academy of Sciences of the United States of America,2002,99(21):13371-13373.doi:10.1073/pnas.222543099.
[30] Hu E, Tontonoz P, Spiegelman B M.Transdifferentiation of myoblasts by the adipogenic transcription factors PPAR gamma and C/EBP alpha[J].Proceedings of the National Academy of Sciences of the United States of America,1995,92(21):9856-9860.doi:10.1073/pnas.92.21.9856.
[31] Siebert A, Goren I, Pfeilschifter J, Frank S.Anti-Inflammatory effects of rosiglitazone in obesity-impaired wound healing depend on adipocyte differentiation[J].PLoS One,2016,11(12):e0168562.doi:10.1371/journal.pone.0168562.
[32] Belda B J, Thompson J T, Eser P O, Vanden Heuvel J P.10e12z CLA alters adipocyte differentiation and adipocyte cytokine expression and induces macrophage proliferation[J].The Journal of Nutritional Biochemistry,2012,23(5):510-518.doi:10.1016/j.jnutbio.2011.02.009.
[33] Kennedy A, Overman A, Lapoint K, Hopkins R, West T, Chuang C C, Martinez K, Bell D, McIntosh M.Conjugated linoleic acid-mediated inflammation and insulin resistance in human adipocytes are attenuated by resveratrol[J].Journal of Lipid Research,2009,50(2):225-232.doi:10.1194/jlr.M800258-JLR200.
[34] Yang Y, Wei H K, Song T X, Cai A L, Zhou Y F, Peng J, Jiang S W, Peng J.E4BP4 mediates glucocorticoid-regulated adipogenesis through COX2[J].Molecular and Cellular Endocrinology, 2017,450:43-53.doi:10.1016/j.mce.2017.04.015.
[35] Fain J N, Ballou L R, Bahouth S W.Obesity is induced in mice heterozygous for cyclooxygenase-2[J].Prostaglandins & Other Lipid Mediators,2001,65(4):199-209.doi:10.1016/S0090-6980(01)00136-8.