MADS-box家族基因编码的转录因子广泛存在于真菌、动物和植物中,其在植物中的功能主要是根、叶片、花和果实等的发育调控[1-3]。AP1和FUL是MADS-box家族基因中的两类基因,其序列具有很高的相似性,属于AP1/SQUA基因亚家族,它们在植物茎尖和花分生组织发育的过程中有着重要的作用[4-7]。
拟南芥中,AGL8/FUL基因在植株茎尖、花序和茎生叶中高调表达[8],该基因除了控制植株形态的转变、茎生叶的生长和复合叶的形态发育,还具有影响果荚的成熟和开裂等功能[9]。拟南芥FUL蛋白调控的靶标基因SAUR10参与茎和花序的分枝角度发育,影响植物的株型发育[10]。番茄FUL蛋白在乙烯调控合成途径中与1个MADS-box蛋白RIN作用来调控番茄果实的成熟[11-12]。桔梗中也有研究表明,FUL同源基因PlacFL1和PlacFL3可能在调节开花时间和花的发育中起保守作用,而PlacFL2可能参与桔梗休眠调节[13]。禾本科植物AP1/FUL基因重复在其小穗的进化过程中有重要的作用[14]。AP1基因的功能集中于花器官的识别,它在细胞分裂素途径中能抑制细胞分裂素的合成并激活细胞分裂素的降解,最终阻止萼片的形成[15-16],还可以调控树木光周期途径中树木季节性的生长[17]。然而,AP1/SQUA亚组中基因在棉花茎尖分生组织发育乃至花分生组织发育过程中的功能及调控通路研究仍未见详细报道。
棉花是我国重要的一种经济作物,目前,我国棉花种植在向新疆全程机械化推进,其中早熟性和株型是很重要的研究方向[18-20]。为探索MADS-box 家族基因中FUL基因在棉花茎尖分生组织和花发育调控过程中的功能,本研究以棉花栽培种中棉所50和早铃1号为材料,克隆FUL的同源基因GhMADS43,对其进行组织特异性表达模式及酵母自激活性分析,为后期筛选与该蛋白质相互作用的蛋白质奠定基础,从而为棉花早熟分子改良提供理论依据。
1 材料和方法
1.1 试验材料
所用棉花栽培种材料为中棉所50(CCRI50)和早铃1号(Zaoling No.1)。试验所用DNA聚合酶、反转录酶、pMD18-T载体、质粒提取试剂盒、胶回收试剂盒和荧光定量试剂盒均购自TaKaRa公司,植物多糖多酚总RNA抽提试剂盒购自天根公司,大肠杆菌感受态细胞DH5α购自康为世纪公司,诱饵空载体质粒pGBKT7、对照质粒pGBKT7-53及酵母菌株Y2HGold购自Clontech,引物合成及序列测定由英骏生物技术有限公司完成。
1.2 材料处理及取样
试验材料种植于河南科技学院D407人工气候培养室,培养条件为长日照,光照/黑暗为14 h/10 h,光照时间为8:00-22:00,温度为28 ℃,分别选取棉花材料中棉所50在二叶展平时期的根、茎、叶、顶芽、开花当天的花、10 d的纤维和胚珠等不同组织。并于四叶展平期到七叶展平期取2个棉花材料的茎尖样品,取样后快速放入液氮中冷冻,并于-80 ℃冰箱中保存备用。
1.3 GhMADS43基因克隆
根据陆地棉基因组序列,设计含有合适酶切位点的特异引物,以棉花茎尖的cDNA为模板,对目标基因进行扩增。PCR反应体系为50 μL,其中10×PCR Buffer 5 μL,dNTPs 4 μL,上下游引物(10 μmol/L)各2 μL,EX TaqDNA聚合酶(5 U/μL)0.25 μL,cDNA模板5 μL,灭菌ddH2O 31.75 μL。反应条件:94 ℃ 2 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 1 min,35个循环;72 ℃ 5 min。PCR反应产物用加了核酸染料Gelred的1% 琼脂糖凝胶电泳检测,并将阳性克隆送英骏生物技术有限公司测序验证。试验中所用PCR引物见表1。
1.4 基因的定量表达分析
使用天根多糖多酚RNA抽提试剂盒提取不同组织样品的总RNA,所使用RNA的质量需保证A260/280在1.8~2.1,A260/230大于1.8。使用TaKaRa 公司RR047A试剂盒进行反转录,使用gDNA Eraser消化总RNA中残留的基因组DNA,进行30 min反转录,合成cDNA。使用SYBR Green分析法并用TaKaRa公司荧光定量试剂盒RR420A对基因进行表达分析,所用仪器为Applied Biosystems Q6实时荧光定量PCR仪。试验中所用荧光定量PCR引物见表1。
表1 棉花GhMADS43基因克隆及表达所用引物
Tab.1 Primers used in GhMADS43 gene cloning and expression in upland cotton
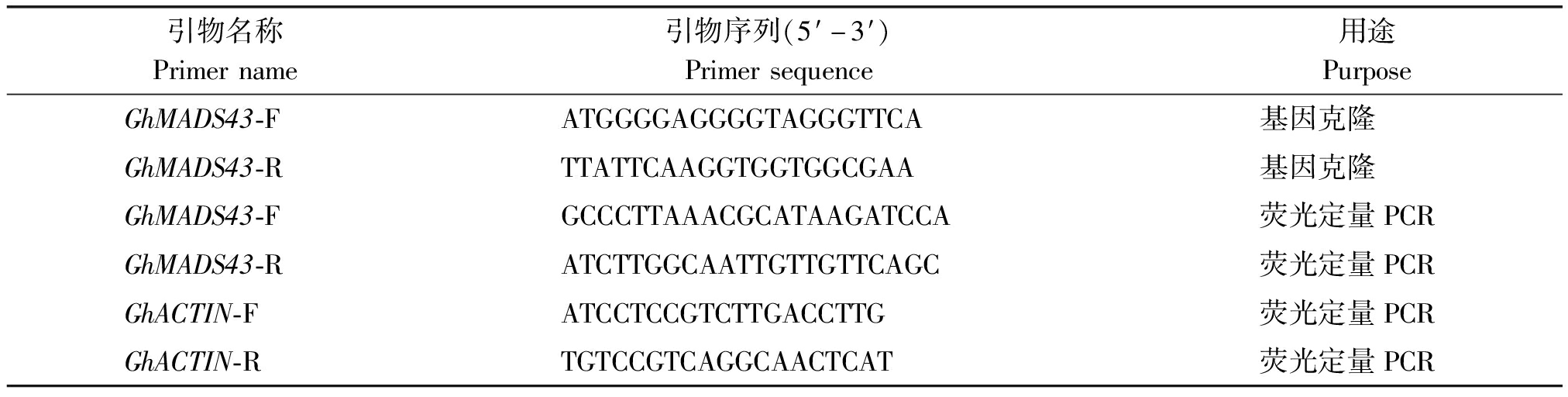
引物名称Primername引物序列(5′-3′) Primersequence 用途PurposeGhMADS43-FATGGGGAGGGGTAGGGTTCA基因克隆GhMADS43-RTTATTCAAGGTGGTGGCGAA基因克隆GhMADS43-FGCCCTTAAACGCATAAGATCCA荧光定量PCRGhMADS43-RATCTTGGCAATTGTTGTTCAGC荧光定量PCRGhACTIN-FATCCTCCGTCTTGACCTTG荧光定量PCRGhACTIN-RTGTCCGTCAGGCAACTCAT荧光定量PCR
1.5 诱饵重组载体构建
以测序正确的pMD18-GhMADS43菌液为PCR模板,通过特异性Infusion引物再次扩增GhMADS43的基因序列,并将载体 pGBKT7用限制性内切酶EcoRⅠ和PstⅠ双酶切形成黏性末端,利用Infusion连接试剂盒进行连接。将连接后的新鲜菌液送英骏生物技术有限公司测序,测序结果正确的阳性克隆即为重组诱饵质粒。
1.6 酵母转化及快速筛选
参照Clontech公司酵母双杂交手册进行酵母菌株Y2HGold 感受态细胞制备和质粒转化。分别将转化诱饵质粒后的Y2HGold 酵母菌涂布于SD/-Trp平板,倒置放30 ℃恒温箱培养3~5 d,挑取SD/-Trp平板上菌落生长良好、直径约2~3 mm的菌落于1.5 mL无菌离心管,加入含有终质量浓度为50 μg/mL卡那霉素的SD/-Trp液体培养基1 mL,以封口膜封口后在200 r/min的摇床上30 ℃培养10 h,进行PCR鉴定。具体方法: 吸取20 μL酵母菌液于PCR管中,6 000 r/min离心3 min,弃上清液,加入20 μL酵母菌裂解液,涡旋振荡1 min,沸水浴中处理10 min,-80 ℃冰箱中冷冻15 min,12 000 r/min离心30 s,重复沸水浴和超低温冰箱冷冻1次,取1 μL上清液作为PCR模板进行检测。取6 μL PCR 扩增产物用1%琼脂糖凝胶电泳检测。
1.7 诱饵蛋白毒性和自激活检测
参照Clontech公司Matchmaker TM Gold 酵母双杂交系统手册,将转化后的重组诱饵载体、pGBKT7-53载体和阴性对照的酵母菌Y2HGold分别加入6 μL 0.9%的NaCl溶液并点于SD/-Trp、SD/-Trp/X-α-Gal和SD/-Trp/-His/-Ade平板上。30 ℃恒温箱中倒置培养3~5 d,观察并记录结果。
2 结果与分析
2.1 GhMADS43基因克隆与分析
将从棉花栽培种材料中克隆到的GhMADS43基因的 CDS 序列,连接pMD-18载体,转化大肠杆菌感受态细胞DH5α并进行测序验证。结果表明,GhMADS43基因CDS全长696 bp,编码231氨基酸(图1-A)。进化树分析表明,GhMADS43蛋白与可可中的FUL的同源蛋白TcAGL8亲缘关系相近(图1-B)。
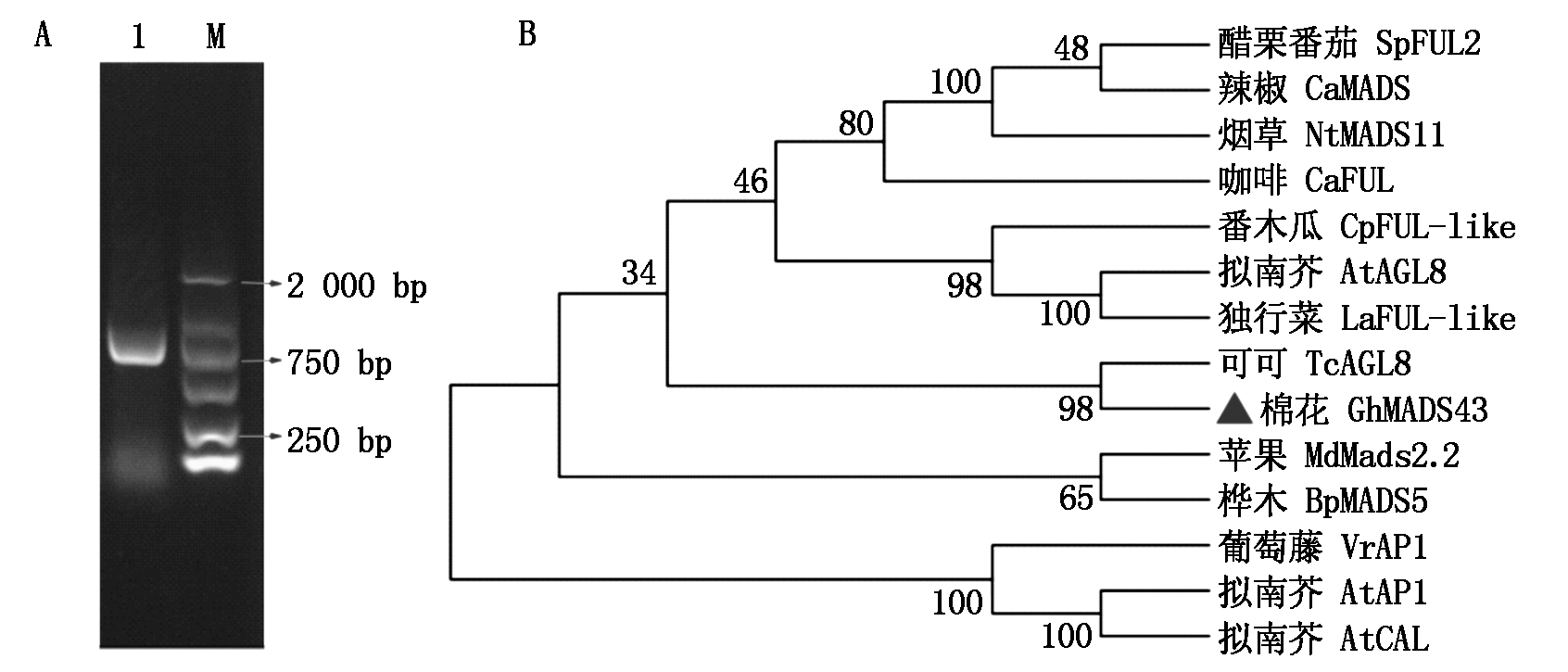
M.DL2000分子质量标记;1.GhMADS43基因的cDNA扩增产物。
M.DL2000 DNA Marker;1.Amplification product of GhMADS43 gene.
图1 GhMADS43基因克隆(A)与蛋白进化树分析(B)
Fig.1 Cloning of GhMADS43 gene(A)and phylogenetic tree of GhMADS43 and FUL proteins in different species(B)
2.2 GhMADS43基因的表达分析
组织特异性表达模式分析得出,GhMADS43基因在棉花的根和顶芽中优势表达,在成熟的花、纤维和胚珠中表达量显著降低(图2-A)。选取一式果枝早熟棉早铃1号和无限果枝早熟棉材料中棉所50从四叶展平时期至七叶展平时期的茎尖分生组织样品进行表达分析。结果表明,从五叶期开始GhMADS43基因在无限果枝中棉所50中的表达量高于一式果枝早铃1号,随着取样时间的推移,在中棉所50的六叶期和七叶期,GhMADS43基因的表达量显著高于早铃1号(图2-B)。
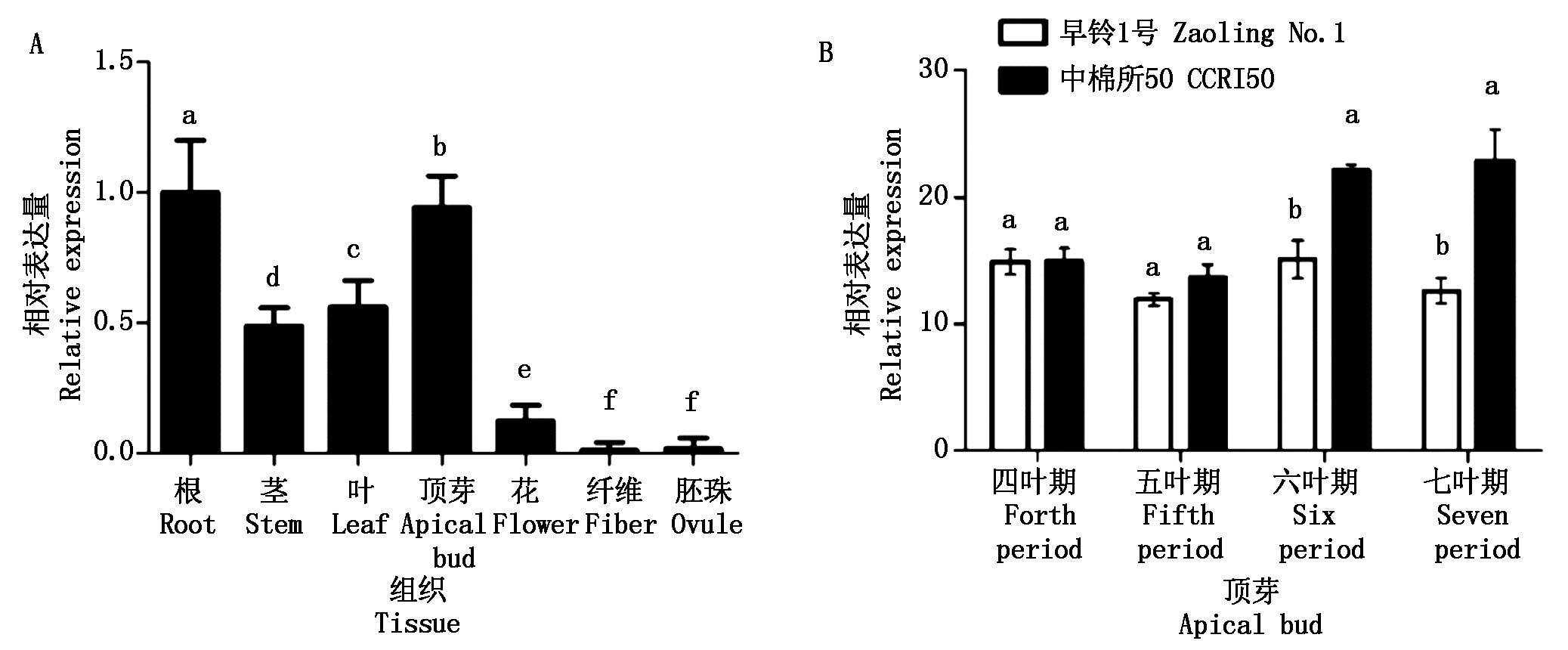
不同小写字母表示处理间差异显著(P<0.05)。
Different small letter indicates significant difference(P<0.05).
图2 棉花GhMADS43基因在不同组织(A)和不同茎尖发育时期表达特异性分析(B)
Fig.2 Relative expression analysis of GhMADS43 in different tissue(A)and different shoot apical meristem development period(B)
2.3 GhMADS43基因诱饵载体的构建和鉴定
根据测序正确的基因序列设计Infusion引物,以T载体为模板扩增基因序列,并与双酶切后的诱饵质粒pGBKT7进行重组,转化子以基因的特异性引物做菌液PCR鉴定。结果表明,成功扩增到GhMADS43全长片段,选取阳性克隆进行测序并对测序结果分析,获得与预期结果相符且读码框正确的序列,成功构建酵母诱饵表达载体pGBKT7-GhMADS43(图3)。
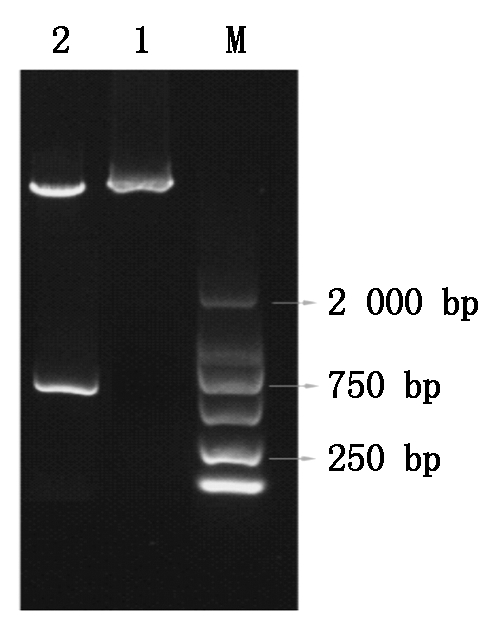
M.DL2000分子质量标记;1.pGBKT7-GhMADS43诱饵载体质粒提取检测;2.双酶切诱饵载体进行验证。
M.DL2000 DNA Marker; 1.Plasmid extraction of pGBKT7-GhMADS43 bait vector; 2.Double restriction enzyme digestion for bait vectors.
图3 重组诱饵载体双酶切验证
Fig.3 Double restriction enzyme digestion for bait vectors
2.4 诱饵质粒酵母菌的鉴定和毒性检测
将上述构建好的诱饵载体和阳性对照pGBKT7-53分别转化至酵母菌株Y2HGold 中,待生长3~5 d后挑取2 mm左右的酵母单克隆在SD/-Trp缺陷培养基中培养过夜,并进行菌液PCR,PCR产物用1%琼脂糖凝胶电泳检测。结果如图(图4-A)所示,诱饵重组质粒和阳性对照质粒转化后酵母菌落PCR产物条带大小与目标基因大小一致,说明重组诱饵质粒载体pGBKT7-GhMADS43和阳性对照质粒pGBKT7-53转化酵母成功。缺陷固体培养结果表明,转有重组诱饵载体在SD/-Trp平板上和转有对照的平板上菌落密集程度相似,生长正常,菌落大小均介于2~3 mm(图4-B)。重组诱饵质粒pGBKT7-GhMADS43和阳性对照质粒pGBKT7-53的菌液过夜培养后,OD600的检测值分别为1.002和1.121,均超过标准值0.8,说明重组诱饵蛋白对酵母菌Y2HGold 生长无毒性,可进行自激活活性检测。
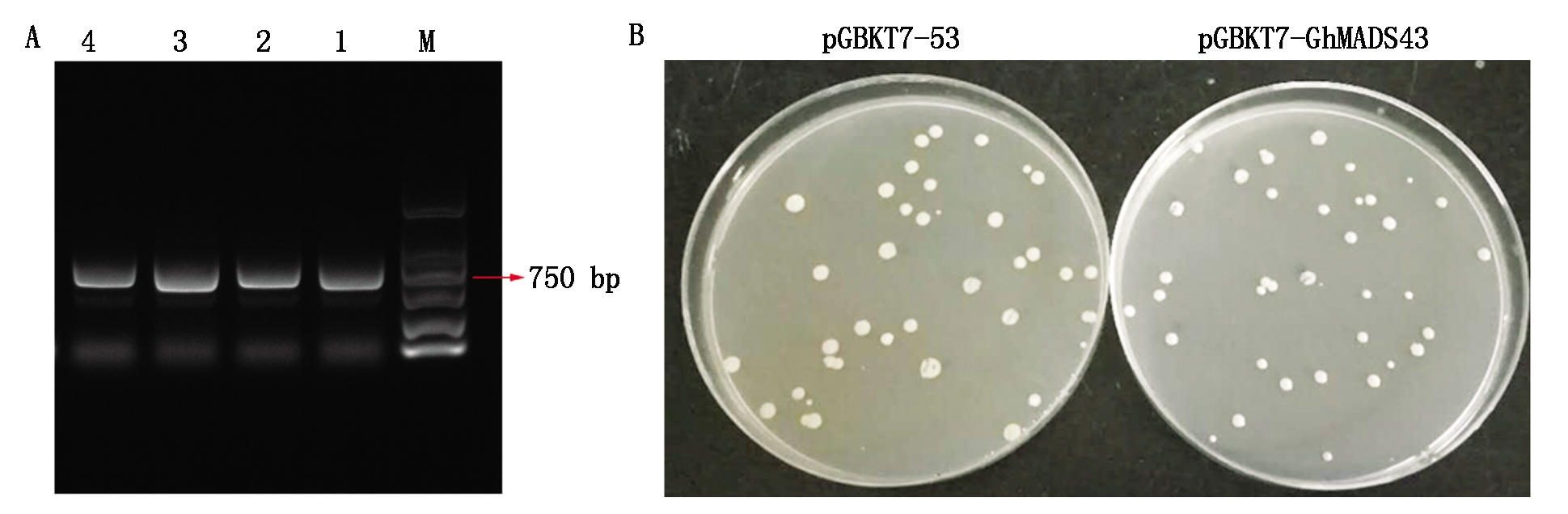
图4 诱饵载体转化酵母PCR检测(A)和毒性检测(B)
Fig.4 Verification of bait vectors in transformed yeast by PCR(A)and toxicity testing(B)
2.5 X-α-Gal验证诱饵载体自激活活性
通过LacZ报告基因的表达来检测诱饵载体的自激活活性。阳性酵母单克隆转移至SD/X-α-Gal/-Trp筛选培养基上,待酵母细胞生长3~5 d后,观察酵母细胞颜色变化情况,阳性对照的酵母细胞变为蓝色,阴性对照始终不变蓝,而诱饵质粒的酵母细胞的蓝色较弱(图5)。
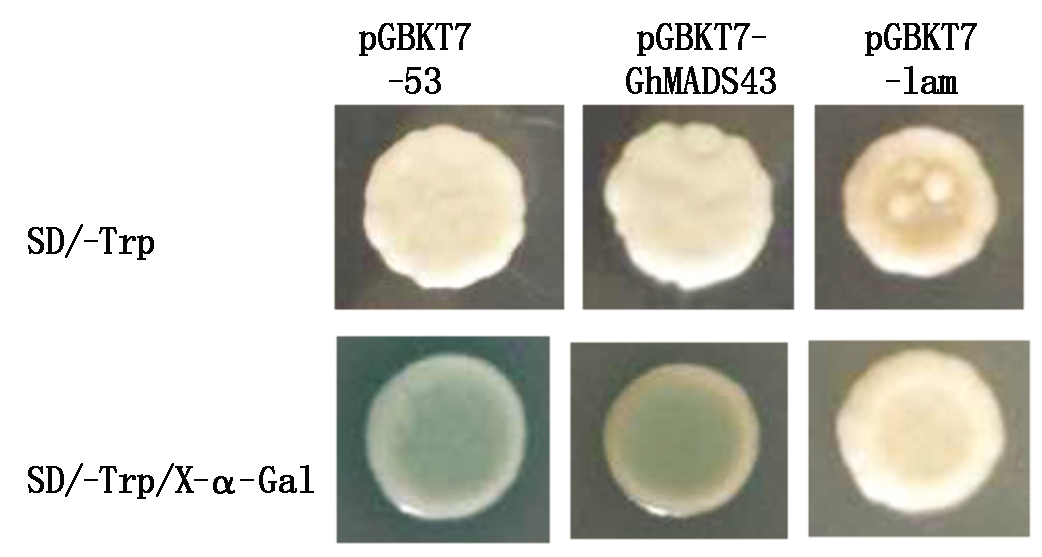
图5 X-α-Gal验证诱饵蛋白自激活结果
Fig.5 X-α-Gal assay for testing of bait protein self-activation
2.6 不同浓度3-AT检测诱饵载体自激活活性
进一步通过His3报告基因的表达与否检测诱饵载体的自激活活性。His3编码的酶参与组氨酸的生物合成,其功能可被特异性竞争抑制剂3-AT所抑制。若诱饵载体本身有激活作用,His3报告基因的转录被激活,则转化后的酵母菌能够在缺陷型平板SD/-Trp/-His/-Ade上生长。将阳性酵母菌株接种至分别添加0,5,10,25,50,75,100 mmol/L的3-AT三缺SD/-Trp/-His/-Ade培养基上。从图6可以看出,诱饵质粒转化后的酵母菌在不同浓度三缺培养基中,菌落均不生长,表明诱饵质粒在此浓度下对宿主酵母菌无自激活作用,后续可用于筛选酵母双杂交文库(图6)。
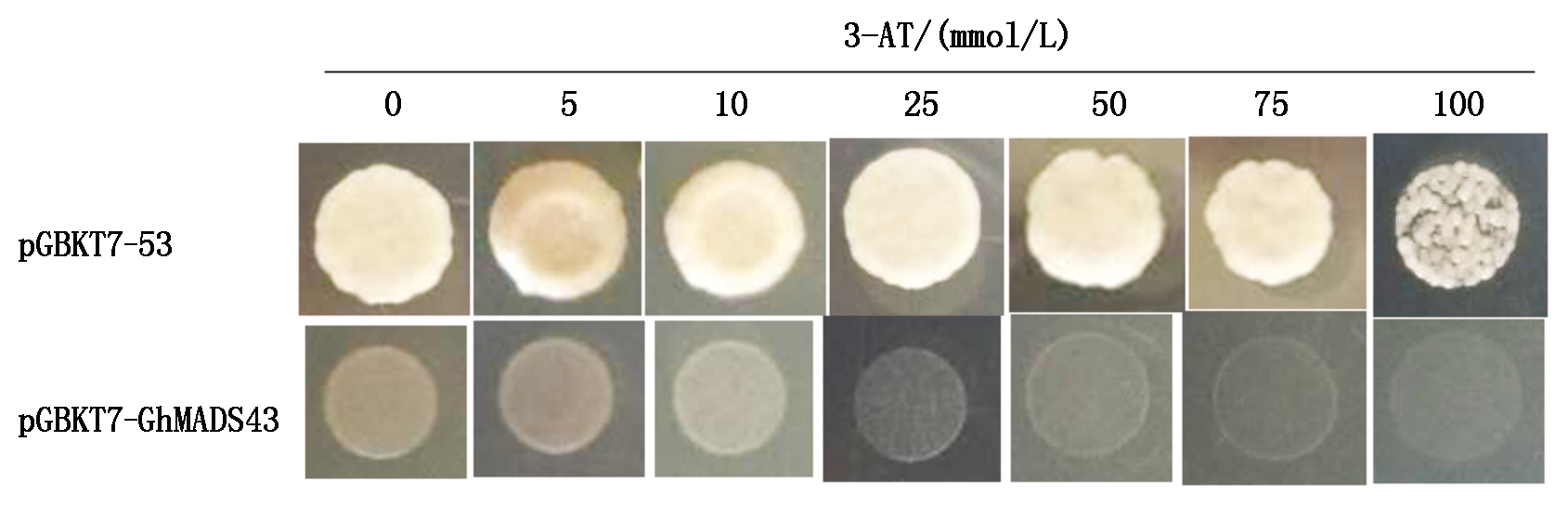
图6 不同 3-AT浓度检测诱饵蛋白自激活结果
Fig.6 Self-activation of bait protein in different concentration of 3-AT
3 讨论与结论
棉花是中国重要的经济作物,棉花机械化种植生产关乎棉农的切身利益。在综合性状发展的前提下,早熟也是棉花品种培育的一个重要目标[21-22]。MADS-box转录因子家族与植物叶片、茎尖和花分生组织发育相关[23]。其中FUL基因具有更广泛的功能,它既能影响叶片和茎尖的发育,又能在花发育的早期促进花序分生组织的形成,并在晚期心皮和角果等花器官发育中行使功能[24-26]。
本研究从棉花栽培种中克隆到FUL的同源基因GhMADS43,发现它在棉花的根和顶芽中表达量较高,而在成熟的花器官中表达量较低,推测该基因的功能可能与根尖和茎尖分生组织的发育和花蕾的前期形成有关。金鱼草中FUL的同源基因DEFH28,可以在花序顶端分生组织中表达,从而诱导花序分生组织转化成花分生组织[27]。AP1和FUL都在花分生组织的发育中起着重要的作用,两类基因的功能有重叠也有差异,其功能的分歧可能是由于序列的改变,FUL基因的功能主要参与茎叶和果实的发育[24]。FUL蛋白负调控茎尖分生组织中APETALA2基因的表达,导致分生组织和植物花序生长停滞,促进果实和种子出现[28-29]。在拟南芥果荚发育过程中,FUL能控制角果的生长,并促进果荚开裂[30]。棉花中GhMADS43在无限果枝中棉所50中的表达量高于一式果枝早铃1号,随着取样时间的延长表达量也呈增加趋势,那么该基因是否与棉铃的成熟和开裂有关,仍值得继续深入研究。
近年来,酵母双杂交技术在植物蛋白质互作研究中已成为一种重要手段,在探讨植物正常生理功能的分子机制等方面发挥很大的作用[31]。在利用酵母双杂交系统筛选文库的过程中,诱饵蛋白表达及自激活活性分析至关重要。一些转录因子具有转录自激活,严重影响后续的筛选工作。因此,将克隆到的GhMADS43基因构建到了酵母双杂交诱饵载体pGBKT7。分析发现,该蛋白质无自激活活性,可用于后续酵母双杂交文库的筛选,为研究该基因在茎尖分生组织发育过程中的功能及调控机理提供一定的基础。
[1] Sommer H,Beltrán J P,Huijser P,Pape H,Lönnig W E,Saedler H,Schwarz-Sommer Z.Deficiens,a homeotic gene involved in the control of flower morphogenesis in Antirrhinum majus:The protein shows homology to transcription factors[J].EMBO Journal,1990,9(3):605-613.doi:10.1002/j.1460-2075.1990.tb08152.x.
[2] Yanofsky M F,Ma H,Bowman J L,Drews G N,Feldmann K A,Meyerowitz E M.The protein encoded by the Arabidopsis homeotic gene agamous resembles transcription factors[J].Nature,1990,346(6279):35-39.doi:10.1038/346035a0.
[3] Magnaghi-Jaulin L,Masutani H,Lipinski M,Harel-Bellan A.Analysis of SRF,SAP-1 and ELK-1 transcripts and proteins in human cell lines[J].Febs Letters,1996,391(3):247-251.doi:10.1016/0014-5793(96)00745-4.
[4] Lee J,Lee I.Regulation and function of SOC1,a flowering pathway integrator[J].Journal of Experimental Botany,2010,61(9):2247-2254.doi:10.1093/jxb/erq098.
[5] Li H F,Liang W Q,Hu Y,Zhu L,Yin C S,Xu J,Dreni L,Kater M M,Zhang D B.Rice MADS6 interacts with the floral homeotic genes SUPERWOMAN1,MADS3,MADS58,MADS13,and DROOPING LEAF in specifying floral organ identities and meristem fate[J].The Plant Cell,2011,23(7):2536-2552.doi:10.1105/tpc.111.087262.
[6] Gu Q,Ferrándiz C,Yanofsky M F,Martienssen R.The FRUITFULL MADS-box gene mediates cell differentiation during Arabidopsis fruit development[J].Development,1998,125(8):1509-1517.
[7] Jaudal M,Monash J,Zhang L L,Wen J Q,Mysore K S,Macknight R,Putterill J.Overexpression of Medicago SVP genes causes floral defects and delayed flowering in Arabidopsis but only affects floral development in Medicago[J].Journal of Experimental Botany,2014,65(2):429-442.doi:10.1093/jxb/ert384.
[8] Mandel M A,Yanofsky M F.The Arabidopsis AGL8 MADSbox gene is expressed in inflorescence meristems and is negatively regulated by APETALA1[J].The Plant Cell,1995,7(11):1763-1771.doi:10.1105/tpc.7.11.1763.
[9] Pabón-Mora N,Wong G K S,Ambrose B A.Evolution of fruit development genes in flowering plants[J].Frontiers in Plant Science,2014,5:300.doi:10.3389/Fpls.2014.00300.
[10] Bemer M,van Mourik H,Mui o J M,Ferrándiz C,Kaufmann K,Angenent G C.FRUITFULL controls SAUR10 expression and regulates Arabidopsis growth and architecture[J].Journal of Experimental Botany,2017,68(13):3391-3403.doi:10.1093/jxb/erx184.
o J M,Ferrándiz C,Kaufmann K,Angenent G C.FRUITFULL controls SAUR10 expression and regulates Arabidopsis growth and architecture[J].Journal of Experimental Botany,2017,68(13):3391-3403.doi:10.1093/jxb/erx184.
[11] Fujisawa M,Shima Y,Nakagawa H,Kitagawa M,Kimbara J,Nakano T,Kasumi T,Ito Y.Transcriptional regulation of fruit ripening by tomato FRUITFULL homologs and associated MADS box proteins[J].The Plant Cell,2014,26(1):89-101.doi:10.1105/tpc.113.119453.
[12] Wang S F,Lu G,Hou Z,Luo Z D,Wang T T,Li H X,Zhang J H,Ye Z B.Members of the tomato FRUITFULL MADS-box family regulate style abscission and fruit ripening[J].Journal of Experimental Botany,2014,65(12):3005-3014.doi:10.1093/Jxb/Eru137.
[13] Zhang S S,Lu S J,Yi S S,Han H J,Zhou Q,Cai F F,Bao M Z,Liu G F.Identification and characterization of FRUITFULL-like genes from Platanus acerifolia,a basal eudicot tree[J].Plant Science,2019,280:206-218.doi:10.1016/j.plantsci.2018.11.016.
[14] Preston J C,Kellogg E A.Reconstructing the evolutionary history of paralogous APETALA1/FRUITFULL-like genes in grasses(Poaceae)[J].Genetics,2006,174(1):421-437.doi:10.1534/genetics.106.057125.
[15] Han Y Y,Zhang C,Yang H B,Jiao Y L.Cytokinin pathway mediates APETALA1 function in the establishment of determinate floral meristems in Arabidopsis[J].Proceedings of the National Academy of Sciences of the United States of America,2014,111(18):6840-6845.doi:10.1073/pnas.1318532111.
[16] Ge J T,Zhao D Q,Han C X,Wang J,Hao Z J,Tao J.Cloning and expression of floral organ development-related genes in herbaceous peony(Paeonia lactiflora Pall.)[J].Molecular Biology Reports,2014,41(10):6493-6503.doi:10.1007/s11033-014-3532-8.
[17] Azeez A,Miskolczi P,Tylewicz S,Bhalerao R P.A tree ortholog of APETALA1 mediates photoperiodic control of seasonal growth[J].Current Biology,2014,24(7):717-724.doi:10.1016/j.cub.2014.02.037.
[18] 狄佳春,陈旭升,赵亮.陆地棉短果枝的遗传与育种利用研究[J].中国棉花,2014,41(11):5-7.
Di J C,Chen X S,Zhao L.Genetic research and breeding evaluation on short branch trait of Gossypium hirsutum L.[J].China Cotton,2014,41(11):5-7.
[19] 王力,杨普,吴志旻.我国棉花生产布局的集聚变化及其驱动因素分析[J].石河子大学学报(哲学社会科学版),2019,33(6):30-36.doi:10.13880/j.cnki.cn65-1210/c.20191209.008.
Wang L,Yang P,Wu Z M.Analysis on agglomeration change and its driving factors of cotton production layout in China[J].Journal of Shihezi University(Philosophy and Social Science),2019,33(6):30-36.
[20] 李健伟,吴鹏昊,石洪亮,李春艳,崔建平,张巨松.不同机采棉种植模式对棉花主要植株形态影响效应分析[J].干旱地区农业研究,2018,36(5):82-87,93.doi:10.7606/j.issn.1000-7601.2018.05.13.
Li J W,Wu P H,Shi H L,Li C Y,Cui J P,Zhang J S.Characteristics of plant morphological parameters and correlation analysis for machine picking cotton in the different planting models[J].Agricultural Research in the Arid Areas,2018,36(5):82-87,93.
[21] 喻树迅,王寒涛,魏恒玲,宿俊吉.棉花早熟性研究进展及其应用[J].棉花学报,2017,29(S1):1-10.doi:10.11963/1002-7807.ysxysx.20170825.
Yu S X,Wang H T,Wei H L,Su J J.Research progress and application of early maturity in upland cotton[J].Cotton Science,2017,29(S1):1-10.
[22] 喻树迅,范术丽,王寒涛,魏恒玲,庞朝友.中国棉花高产育种研究进展[J].中国农业科学,2016,49(18):3465-3476.doi:10.3864/j.issn.0578-1752.2016.18.001.
Yu S X,Fan S L,Wang H T,Wei H L,Pang C Y.Progresses in research on cotton high yield breeding in China[J].Scientia Agricultura Sinica,2016,49(18):3465-3476.
[23] Ferrándiz C,Gu Q,Martienssen R,Yanofsky M F.Redundant regulation of meristem identity and plant architecture by FRUITFULL,APETALA1 and CAULIFLOWER[J].Development,2000,127:725-734.
[24] McCarthy E W,Mohamed A,Litt A.Functional divergence of APETALA1 and FRUITFULL is due to changes in both regulation and coding sequence[J].Frontiers in Plant Science,2015,6:1076.doi:10.3389/fpls.2015.01076.
[25] Pabon-Mora N,Sharma B,Holappa L D,Kramer E M,Litt A.The Aquilegia FRUITFULL-like genes play key roles in leaf morphogenesis and inflorescence development[J].Plant Journal,2013,74(2):197-212.doi:10.1111/tpj.12113.
[26] Yu X X, Duan X S, Zhang R, Fu X H, Ye L L, Kong H Z,Xu G X,Shan H Y.Prevalent exon-intron structural changes in the APETALA1/FRUITFULL,SEPALLATA,AGAMOUS-LIKE6,and FLOWERING LOCUS C MADS-box gene subfamilies provide new insights into their evolution[J].Frontiers in Plant Science,2016,7:598.doi:10.3389/fpls.2016.00598.
[27] Müller B M,Saedler H,Zachgo S.The MADS-box gene DEFH28 from Antirrhinum is involved in the regulation of floral meristem identity and fruit development[J].Plant Journal,2001,28(2):169-179.doi:10.1046/j.1365-313x.2001.01139.x.
[28] Balanzà V,Martínez-Fernández I,Sato S,Yanofsky M F,Kaufmann K,Angenent G C,Bemer M,Ferrándiz C.Genetic control of meristem arrest and life span in Arabidopsis by a FRUITFULL-APETALA2 pathway[J].Nature Communication,2018,9(1):565.doi:10.1038/s41467-018-03067-5.
[29] Balanzà V,Martínez-Fernández I,Sato S,Yanofsky M F,Ferrándiz C.Inflorescence meristem fate is dependent on seed development and FRUITFULL in Arabidopsis thaliana[J].Frontiers in Plant Science,2019,10:1622.doi:10.3389/fpls.2019.01622.
[30] Dinneny J R,Weigel D,Yanofsky M F.A genetic framework for fruit patterning in Arabidopsis thaliana[J].Development,2005,132(21):4687-4696.doi:10.1242/dev.02062.
[31] 翟玉山,彭磊,杨永庆,邓宇晴,程光远,郑艳茹,徐景升.甘蔗条纹花叶病毒HC-Pro、P3N-PIPO、CP和VPg基因酵母双杂交诱饵表达载体的构建及自激活检测[J].华北农学报,2016,31(1):83-89.doi:10.7668/hbnxb.2016.01.014.
Zhai Y S,Peng L,Yang Y Q,Deng Y Q,Cheng G Y,Zheng Y R,Xu J S.Construction and self-activated detection of the baits of HC-Pro,P3N-PIPO,CP and VPg from sugarcane streak mosaic virus for yeast two hybrid system[J].Acta Agriculturae Boreali-Sinica,2016,31(1):83-89.