GRAS蛋白家族是一类植物特有的转录因子家族,因最初包含GIBBERELLIN-INSENSITIVE(GAI)、REPRESSOR of ga1-3(RGA)和SCARECROW(SCR)3个成员而得名[1]。后来,Tian等[2]根据功能相似性和序列同源性,将当时在水稻和拟南芥中分别鉴定到的57,32个GRAS基因划归于8个亚家族,即:LISCL、PAT1、SCL3、DELLA、SCR、SHR、HAM和LS。迄今,已在拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa L.)、番茄(Solanum lycopersicum)、棉花(Gossypiumhirsutum)、油菜(Brassica napus L.)、红薯(Ipomoea trifida)等植物中对GRAS基因进行了全基因组水平的鉴定和分析[2-5],并发展出了新的基因亚家族[3-4, 6-8]。
研究表明,许多GRAS蛋白参与植物生长发育调控和激素信号转导。在拟南芥中,SCR在根内皮层特异表达,对皮层/内皮层干细胞的不对称分裂及根基本组织模式建成起重要调节作用[9]。3个GRAS转录因子SCR、SCARECROW-LIKE 23(SCL23)和SHORT-ROOT(SHR)在叶维管束鞘细胞的形成和外形的规整以及下胚轴内皮层(淀粉鞘)的形成中起着关键作用[10-11]。SCL3与DELLA相互抑制,维持正常的GA信号转导通路[12]。在根的伸长/分化区,SCL3-DELLA互作调控GA信号通路,主要促使根内皮层细胞的伸长;在分生区,SCL3-DELLA对GA信号通路的调控还需要SHR/SCR通路的参与,调控内皮层的生成[13]。
此外,一些GRAS在植物对逆境胁迫的应激响应中发挥重要作用。胡杨PeSCL7在叶片中的表达受干旱和高盐胁迫的诱导[14]。异源过表达PeSCL7能够提高转基因拟南芥对干旱和盐胁迫的耐受性[15]。玉米ZmSCL7的表达受NaCl、H2O2和低温处理诱导,过表达ZmSCL7有助于提高烟草的抗盐性[16]。独行菜LaSCL18的表达受低温胁迫的诱导,可能与其幼苗低温耐受性有关[17]。Fode等[18]研究发现,拟南芥SCL14可以和Ⅱ类TGA蛋白结合形成复合物,参与激活植物自身的解毒系统以减轻有害化学物质对细胞的伤害。小麦TaSCL14则在光氧化胁迫响应中起重要作用[19]。盐穗木HcSCL13的表达受盐、干旱的诱导,异源过表达HcSCL13能促进转基因拟南芥的生长并增强其耐盐性[20]。
水稻中至少存在60个GRAS基因[3],目前仅DHD1、DLT、MOC1、MOC3、NSP1、NSP2、OsGRAS19、OsGRAS23、OsSHR2、SCR、和SLR1等少数几个基因的功能被鉴定[21-30],其余大多数基因尚无功能研究的相关报道。OsGRAS39(LOC_Os10g22430.1)归属于PAT1亚家族[31-32]。为深入研究OsGRAS39的功能,本研究通过qRT-PCR技术分析了该基因在不同组织中、非生物逆境胁迫下及脱落酸(Abscisic acid, ABA)和赤霉素(Gibberellin A3, GA3)处理下的表达模式,并利用CRISPR/Cas9基因编辑技术创建了该基因敲除的突变体,旨在为后续的OsGRAS39功能研究奠定基础。
1 材料和方法
1.1 试验材料
本研究转基因受体材料为水稻粳稻品种日本晴(Oryza sativa L.spp.japonica cv.Nipponbare)。
1.2 菌株、载体和试剂
本研究所用菌株大肠杆菌(Escherichia coli)DH5α和根癌农杆菌(Agrobacterium tumefaciens)EHA105由江西省作物生长发育调控重点实验室保存。CRISPR/Cas9植物基因敲除试剂盒(pP1C.3 with DNA Recombinase)购于江苏吉锐生物技术有限公司(中国南京)。高保真酶PrimeSTAR®Max DNA Polymerase、微量DNA提取和PCR试剂盒MightyAmpTM Genotyping Kit、RNA提取试剂RNAiso Plus、逆转录试剂盒PrimeScriptTM RT reagent Kit with gDNA Eraser、qRT-PCR试剂盒TB Green®Premix DimerEraserTM均购于宝日医生物技术(北京)有限公司。限制性内切酶购自赛默飞世尔科技(中国)有限公司。DNA Marker、胶回收试剂盒、质粒提取试剂盒均购于天根生化科技(北京)有限公司。
1.3 水稻OsGRAS39表达模式分析
取三叶期水稻幼根和幼苗(地上部分)及生长至孕穗期的水稻的根、茎(穗下节间)、倒二叶的叶片、叶鞘和幼穗组织于液氮速冻后存于-80 ℃冰箱,用于部位表达分析。水稻种子萌发后于人工气候箱(25 ℃,80% RH,光周期为L∶D=12 h∶12 h)培养至三叶一心期,分别进行高温(42 ℃)、低温(4 ℃)、高盐(100 mmol/L NaCl)、模拟干旱(10% PEG6000)、ABA(100 μmol/L)和GA3(20 μmol/L)胁迫处理。在处理0,0.5,1,2,4,8,24 h取样(取地上部分,每5株混合成一个样品),液氮速冻后保存于-80 ℃冰箱备用。
水稻总RNA用RNAiso Plus提取后,利用PrimeScriptTM RT reagent Kit with gDNA Eraser试剂盒合成cDNA。以水稻UBQ5(Os01g0328400)基因作为内参进行荧光定量PCR分析。使用TaKaRa试剂盒TB Green® Premix DimerEraserTM(Perfect Real Time),在赛默飞StepOnePlusTM Real-Time PCR System中进行qRT-PCR。qRT-PCR体系(20.0 μL):TB Green Premix DimerEraser(2×)10.0 μL、上下游引物(10 μmol/L)各0.6 μL、ROX Reference Dye(50×)0.4 μL、cDNA 1.0 μL、补足灭菌ddH2O至20.0 μL。qRT-PCR反应程序:95 ℃ 30 s;95 ℃ 5 s,55 ℃ 30 s,72 ℃ 30 s,40个循环。试验设置3个生物学重复,采用2-ΔΔCT法[33]计算基因相对表达量,使用SPSS 25.0软件进行统计分析,采用Tukey法进行多重比较检验差异显著性(P<0.05),使用GraphPad Prism 8.0软件作图。
1.4 靶序列选择及引物设计
利用CRISPR-GE网站(http://skl.scau.edu.cn/home/)[34],分析筛选OsGRAS39序列上sgRNA靶位点。选择基因5′端处于外显子区且潜在脱靶效应评分低的靶位点。然后根据基因敲除试剂盒的要求设计扩增sgRNA表达盒的引物U3p.3-F/Oligo-R(表1)。
1.5 CRISPR/Cas9表达载体构建
本研究采用江苏吉锐生物技术有限公司的CRISPR/Cas9植物基因敲除试剂盒构建OsGRAS39的CRISPR/Cas9基因编辑表达载体。构建过程采用重组酶无缝克隆的方法进行。具体步骤:使用高保真DNA聚合酶PrimeSTAR® Max DNA Polymerase,以pP1C.3载体为模板,用U3p.3-F/Oligo-R引物扩增sgRNA表达盒;反应体系参照高保真DNA聚合酶说明书;PCR产物经过凝胶电泳后切胶回收;使用限制性内切酶EcoRⅠ和XbaⅠ将pP1C.3载体酶切线性化后通过凝胶电泳切胶回收;使用DNA重组酶将扩增获得的sgRNA表达盒和线性化的pP1C.3载体重组,构建重组载体;参照试剂盒说明书配置反应体系,于37 ℃孵育30 min后迅速置于冰上15 min;将重组产物转化大肠杆菌DH5α,37 ℃培养过夜;次日,随机挑取培养皿上2个单克隆菌落摇菌,送生工生物工程(上海)股份有限公司通用引物M13F(-47)进行测序验证;将测序正确的阳性克隆提取质粒,通过冻融法转入农杆菌菌株EHA105中。
1.6 水稻遗传转化
水稻遗传转化参考Toki等[35]的方法,经过水稻胚性愈伤组织诱导,农杆菌侵染及共培养,抗性愈伤筛选,愈伤组织的分化和幼苗的生根及移栽等过程,完成水稻遗传转化。
1.7 突变体鉴定及突变类型分析
剪取T0转基因植株幼苗大约2 mm2大小的叶片组织,使用试剂盒MightyAmpTM Genotyping Kit提取DNA。使用潮霉素抗性检测引物Hyg-JF/Hyg-JR(表1)筛选转基因阳性植株。在靶位点上下游设计1对突变体检测引物C-GRAS39-JF/C-GRAS39-JR(表1),对转基因阳性植株进行PCR扩增,PCR产物理论大小为499 bp。将PCR产物送生工生物工程(上海)股份有限公司测序,测序结果用在线软件工具DSDecodeM(http://skl.scau.edu.cn/dsdecode/)[34]进行峰图分析,以获得基因突变信息。
表1 本研究所用引物
Tab.1 Primers used in the study

引物Primer序列(5′-3′)Sequence用途ApplicationUBQ5-RTFGGAAGTAAGGAAGGAGGAGGAAqRT-PCRUBQ5-RTRCAGAGGTGATGCTAAGGTGTTCqRT-PCRGRAS39-RTFGGTGCTCATCCTGCCCATAqRT-PCRGRAS39-RTRTGTGGTCTGAGTGGCTATCATqRT-PCRU3p.3-FCAGGAAACAGCTATGACCATATTCAAGGGATCTTTAAAC表达载体构建Oligo-RGCTATTTCTAGCTCTAAAACATTAGCGGGAGCCCCCTGTCTTGCCACGGATCATCTGC表达载体构建Hyg-JFGATAGAGTTGGTCAAGAC潮霉素抗性检测Hyg-JRCAACATCTTCTTCTGGAG潮霉素抗性检测C-GRAS39-JFGGATGCTCACTACGCTTCTT突变体检测C-GRAS39-JRATCACCAGAGACAGACACCAT突变体检测M13F(-47)CGCCAGGGTTTTCCCAGTCACGAC表达载体测序
注:下划线部分为靶点序列。
Note:The target sequences are underlined.
2 结果与分析
2.1 OsGRAS39基因的组织表达模式
本研究通过qRT-PCR的方法检测了OsGRAS39在水稻不同组织中的表达情况。检测结果显示,OsGRAS39在所用组织中都有表达,其中在幼苗中的表达丰度最高,然后依次是幼根、叶片,在叶鞘、穗、根和茎中表达相对较弱(图1)。
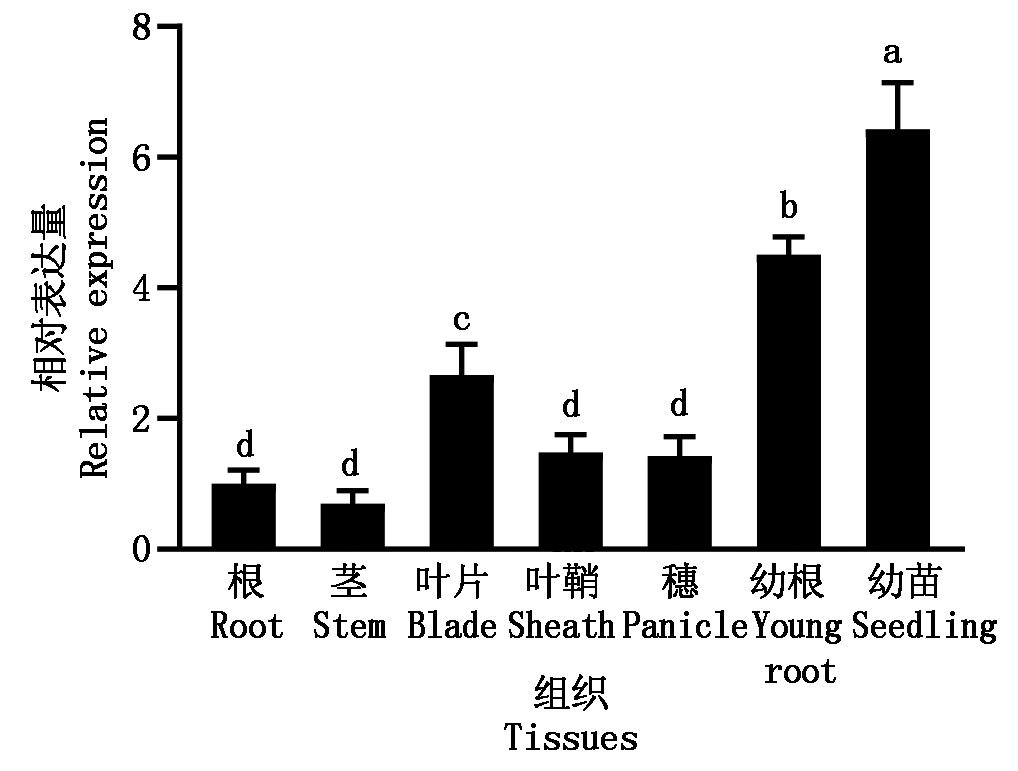
不同小写字母表示差异达到显著水平(P<0.05)。图2同。
Different lowercase letters indicate significant difference(P<0.05).The same as Fig.2.
图1 OsGRAS39的组织表达模式
Fig.1 Expression patterns of OsGRAS39 in different tissues
2.2 OsGRAS39逆境胁迫响应和激素应答
在不同的逆境胁迫和激素的处理下,OsGRAS39的表达受到不同程度的影响。在42 ℃高温处理下,OsGRAS39的表达在处理的最初2 h内无显著变化,在4 h后表达量显著下调至初始表达量的40%以下(图2-A)。OsGRAS39的表达受4℃低温处理的诱导,在1 h其表达丰度即显著上调至初始量的2.5倍左右,在2 h其表达强度约为初始水平的5.3倍,为最高值;随后其表达水平逐渐回落,在24 h其表达水平与初始值相当(图2-B)。OsGRAS39的表达受10% PEG6000处理的影响不大,其在所有处理时间点的表达水平无显著差异(图2-C)。在100 mmol/L NaCl处理下,OsGRAS39的表达从0.5 h开始即受到诱导显著上调,上调的幅度大都在2~3倍(图2-D)。在100 μmol/L ABA处理下,OsGRAS39的表达量在1 h显著上调至初始值的1.9倍,随后回复至初始水平,到8,24 h再次显著上调(图2-E)。OsGRAS39的表达在2 h开始受到20 μmol/L GA3的抑制,其表达量在8 h下调至最低点,大约为初始值的22%,但在24 h其表达水平显著上调,约为初始值的1.9倍(图2-F)。
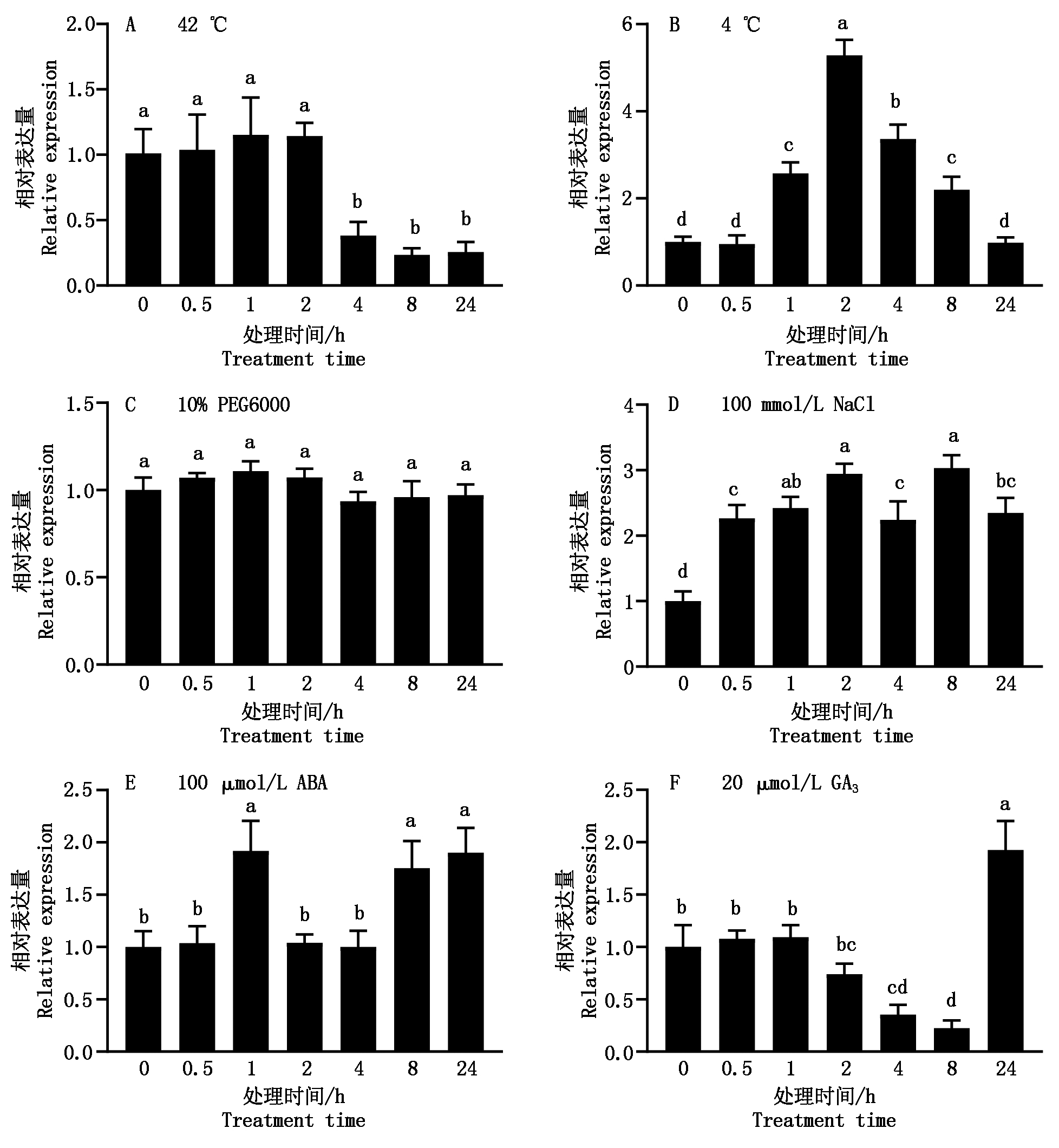
图2 OsGRAS39 在不同逆境胁迫下的表达模式
Fig.2 Expression patterns of OsGRAS39 under different stress treatments
2.3 sgRNA靶点选择和载体构建
本研究采用无缝克隆的方法将sgRNA表达盒克隆到CRISPR/Cas9载体pP1c.3上。pP1c.3载体的结构如图3-A所示。水稻OsGRAS39基因结构只有一个外显子序列,为了获得CRISPR/Cas9诱导的定点突变导致的功能缺失型突变体植株,本研究利用在线设计网站CRISPR-GE(http://skl.scau.edu.cn/)[34]分析筛选sgRNA靶位点。最终基因编辑的靶位点选择在反义链靠近起始密码子ATG的第290位碱基到271位碱基处(图3-B),靶点序列GC含量为65%。参照CRISPR/Cas9植物基因敲除试剂盒说明书上的方法,用引物U3p.3-F/Oligo-R扩增出sgRNA克隆框,通过重组酶作用与线性化的载体pP1C.3重组构建表达载体。重组载体经测序确认构建正确,命名为pP1C.3-OsGRAS39-gRNA。然后将质粒转入到农杆菌EHA105中。
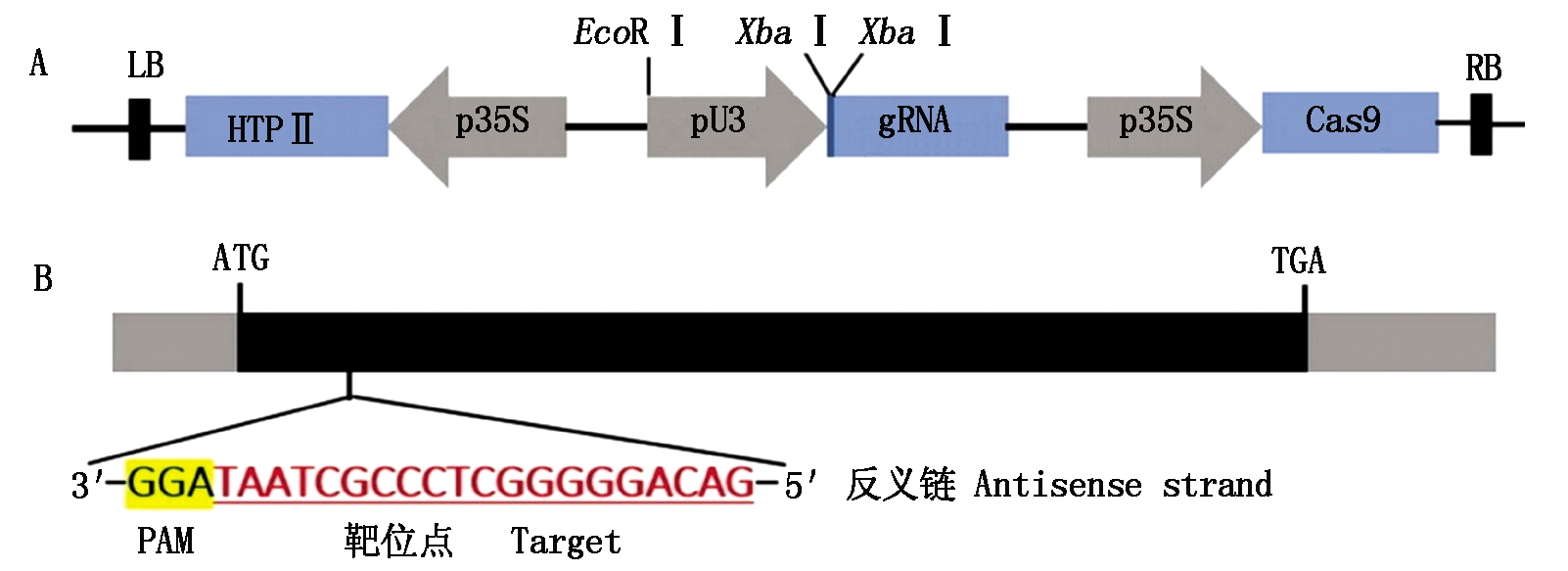
A.CRISPR/Cas9载体pP1C.3示意图;B.OsGRAS39基因结构及sgRNA靶位点位置。
A.Schematic diagram of the CRISPR/Cas9 vector pP1C.3; B.OsGRAS39 gene structure and the sgRNA target site.
图3 CRISPR/Cas9载体示意图及sgRNA靶位点信息
Fig.3 Diagram of CRISPR/Cas9 vector and information of the sgRNA target site
2.4 转基因植株的获得及突变类型分析
通过农杆菌介导的遗传转化,最终获得30株转基因苗(图4)。PCR检测结果显示全部为潮霉素抗性的阳性苗(图5)。通过测序和测序峰图分析显示,有8株在PAM附近发生了不同类型突变(图6),包括1个纯合突变体(#9),5个双等位突变体(#6、#7、#26、#29和#30),以及2个杂合突变体(#3和#8)。在正常生长条件下,突变体与野生型的表型无明显区别。
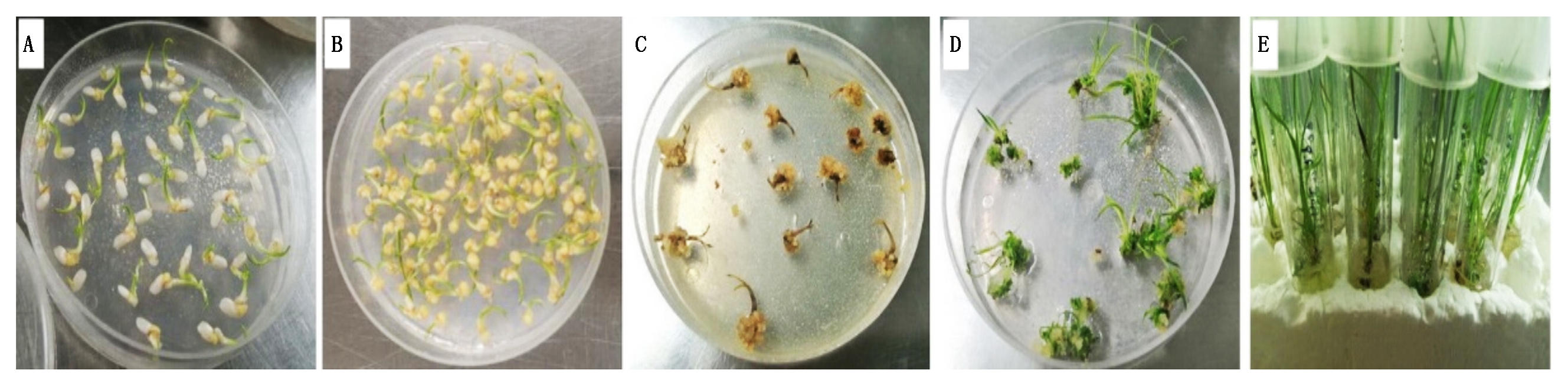
A.愈伤组织的诱导; B.愈伤组织与农杆菌共培养; C.愈伤组织的筛选; D.愈伤组织的分化培养; E.转基因苗的生根。
A.Callus induction; B.Callus co-culture with Agrobacterium; C.Screening of resistant callus; D.Callus regeneration; E.Rooting of transgenic plants.
图4 水稻遗传转化过程
Fig.4 Rice genetic transformation process
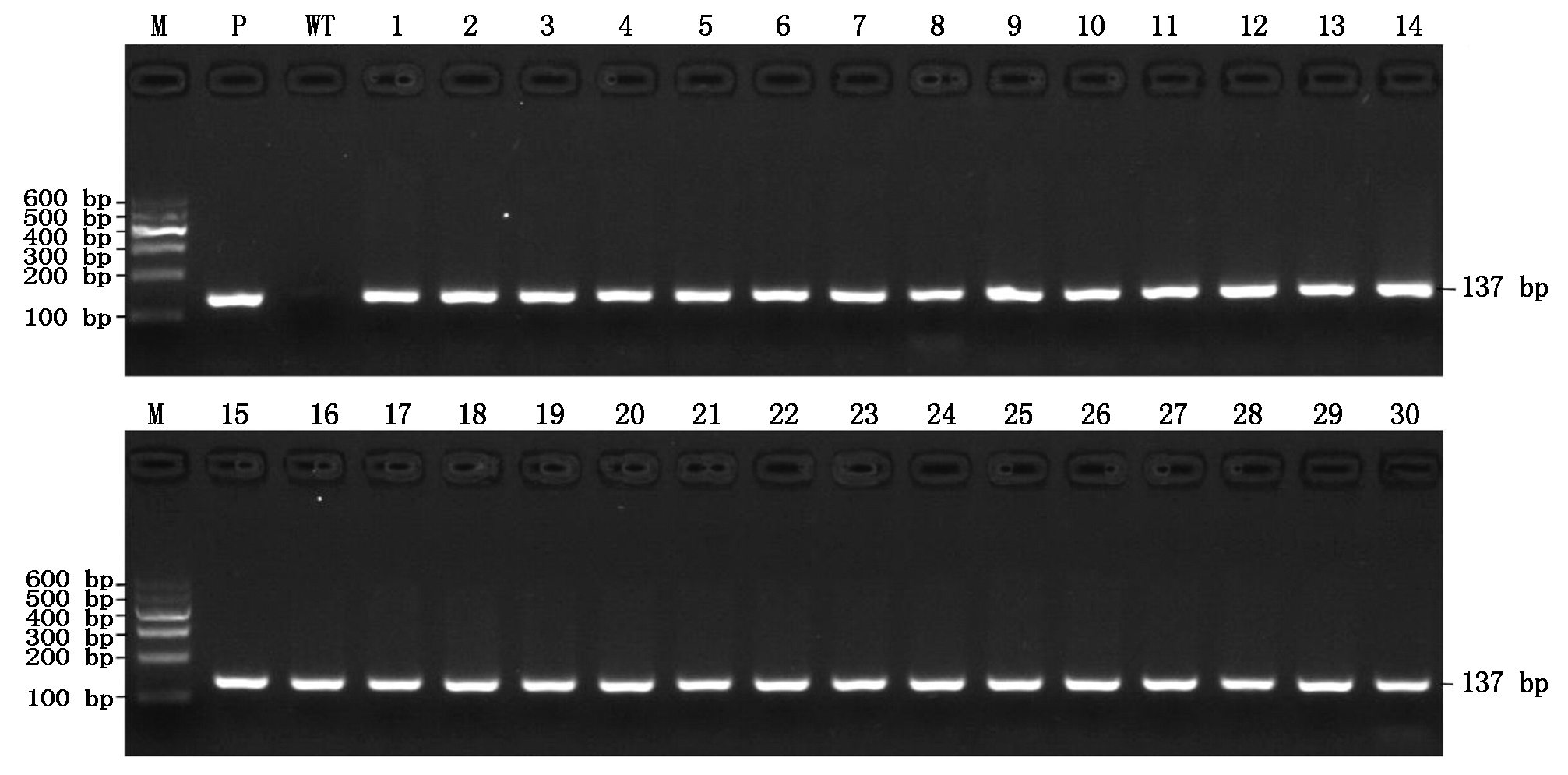
M.Marker Ⅰ; P.pP1C.3-OsGRAS39-gRNA质粒对照;WT.野生型;1-30.转基因植株。
M.Marker Ⅰ; P.pP1C.3-OsGRAS39-gRNA plasmid; WT.Wild type; 1-30.Transgenic plants.
图5 PCR鉴定转基因阳性植株
Fig.5 PCR identification of positive transgenic plants

WT.野生型; Ho.纯合突变; Bi.双等位突变;He.杂合突变;/.无突变;右边数字表示碱基插入(+)或缺失(-)数。
WT.Wild type; Ho.Homozygote; Bi.Bi-allele; He.Heterozygote;/.No mutation; The numbers on the right hand side indicate how many base pairs(bp)are involved(+.Insertion;-.Deletion).
图6 T0转基因株系中OsGRAS39基因突变类型的分析
Fig.6 Analysis of OsGRAS39 gene mutation types in T0 transgenic lines
3 结论与讨论
研究发现,一些GRAS基因的表达存在组织特异性。例如,拟南芥SCR在根的内皮层特异表达,对皮层/内皮层的分化起决定作用[9];葡萄VvSCL14-like在休眠芽中特异表达[36]。而一些GRAS基因在植物各个组织中呈组成型表达。胡杨PeSCL7在根、茎、叶中均有表达[14];水稻DHD1[22]和OsGRAS23[26]在所有检测的器官中表达,前者在叶片、叶鞘和穗中的表达较强,后者在根尖和穗中表达较强。Chen等[37]研究发现,OsGRAS19的表达在所有检测的器官,包括根、茎秆、茎尖、叶片、叶鞘和花序中都检测到,其在幼嫩器官中的表达丰度高于在成熟器官中的表达丰度。类似的,本研究中OsGRAS39在所有检测的器官中都有表达且在幼苗中表达丰度最高,然后是幼根,这意味着OsGRAS39参与了水稻生长发育的整个过程,且在苗期的生长发育过程中的作用更大。但是,经CRISPR/Cas9定向编辑产生的OsGRAS39敲除突变体表型并未表现出与野生型明显的差异,说明OsGRAS39在水稻生长发育方面的功能可能存在冗余。
本研究中,在各种逆境胁迫和激素处理下,OsGRAS39的表达受到不同程度的影响。其表达受低温、高盐和ABA的诱导,与山葡萄VaPAT1[38]的表达模式类似。与山葡萄VaPAT1[38]和番茄SlGRAS7[39]的表达被GA3持续抑制不同,本研究中OsGRAS39的表达在GA3处理2 h时开始下调,但在24 h表达上调。OsGRAS39的表达受低温的诱导,与山葡萄VaPAT1[38]、茶树CsCIGR[40]、番茄SlGLD1[41]和佛手CmsGRAS[42]相似。与本研究中PEG处理对OsGRAS39的表达无显著影响不同,山葡萄VaPAT1[38]和茶树CsCIGR[40]的表达受PEG的诱导,而盐穗木HcSCL13[20]和番茄SlGRAS7[39]的表达也能被水分胁迫(分别为暴露于空气中和甘露醇处理)诱导。与茶树CsCIGR[40]的表达被高温诱导相反,本研究中OsGRAS39的表达在高温处理4 h后下调。综上,OsGRAS39与同属PAT1亚家族的CmsGRAS、CsCIGR、HcSCL13、SlGLD1、SlGRAS7和VaPAT1在不同逆境胁迫和激素处理下的表达模式既存在一定的相似之处也存在不同之处。研究表明,在拟南芥中过表达HcSCL13能提高转基因植株的耐盐性[20];过表达VaPAT1的拟南芥增强了对低温、干旱和高盐的抗性[38];过量表达SlGRAS7能提高番茄干旱和高盐的耐受性[39]。鉴于OsGRAS39与HcSCL13、VaPAT1和SlGRAS7的表达模式存在不同程度的相似性,OsGRAS39可能在相应的逆境响应中发挥作用,这需要进一步的研究验证。
本研究中OsGRAS39的突变类型包含了单碱基插入、缺失及长片段缺失。这几种突变类型都存在移码现象而使蛋白翻译提前终止,说明成功获得了OsGRAS39功能缺失的突变株。这些突变体的获得,为进一步研究OsGRAS39在水稻生长发育过程和逆境胁迫响应中的功能提供了材料基础。
[1] Pysh L D, Wysocka-Diller J W, Camilleri C, Bouchez D, Benfey P N.The GRAS gene family in Arabidopsis: sequence characterization and basic expression analysis of the SCARECROW-LIKE genes[J].The Plant Journal, 1999, 18(1): 111-119.doi:10.1046/j.1365-313x.1999.00431.x.
[2] Tian C G, Wan P, Sun S H, Li J Y, Chen M S.Genome-wide analysis of the GRAS gene family in rice and Arabidopsis[J].Plant Molecular Biology, 2004, 54(4): 519-532.doi:10.1023/B:PLAN.0000038256.89809.57.
[3] Liu X Y, Widmer A.Genome-wide comparative analysis of the GRAS gene family in Populus, Arabidopsis and rice[J].Plant Molecular Biology Reporter, 2014, 32(6): 1129-1145.doi:10.1007/s11105-014-0721-5.
[4] Guo P C, Wen J, Yang J, Ke Y Z, Wang M M, Liu M M, Ran F, Wu Y W, Li P F, Li J N, Du H.Genome-wide survey and expression analyses of the GRAS gene family in Brassica napus reveals their roles in root development and stress response[J].Planta, 2019, 250(4): 1051-1072.doi:10.1007/s00425-019-03199-y.
[5] Chen Y, Zhu P P, Wu S Y, Lu Y, Sun J, Cao Q H, Li Z Y, Xu T.Identification and expression analysis of GRAS transcription factors in the wild relative of sweet potato Ipomoea trifida[J].BMC Genomics, 2019, 20(1): 911.doi:10.1186/s12864-019-6316-7.
[6] Zhang B, Liu J, Yang Z E, Chen E Y, Zhang C J, Zhang X Y, Li F G.Genome-wide analysis of GRAS transcription factor gene family in Gossypium hirsutum L.[J].BMC Genomics, 2018, 19(1): 348.doi:10.1186/s12864-018-4722-x.
[7] Song L L, Tao L, Cui H P, Ling L, Guo C H.Genome-wide identification and expression analysis of the GRAS family proteins in Medicago truncatula[J].Acta Physiologiae Plantarum, 2017, 39(4): 93.doi:10.1007/s11738-017-2393-x.
[8] Zhang H, Mi L M, Xu L, Yu C X, Li C, Chen C L.Genome-wide identification, characterization, interaction network and expression profile of GRAS gene family in sweet orange(Citrus sinensis)[J].Scientific Reports, 2019, 9(1): 2156.doi:10.1038/s41598-018-38185-z.
[9] Laurenzio L D, Wysocka-Diller J, Malamy J E, Pysh L, Helariutta Y, Freshour G, Hahn M G, Feldmann K A, Benfey P N.The SCARECROW gene regulates an asymmetric cell division that is essential for generating the radial organization of the Arabidopsis root[J].Cell, 1996, 86(3): 423-433.doi:10.1016/s0092-8674(00)80115-4.
[10] Long Y C, Goedhart J, Schneijderberg M, Terpstra I, Shimotohno A, Bouchet B P, Akhmanova A, Gadella T W J, Heidstra R, Scheres B, Blilou I.SCARECROW-LIKE23 and SCARECROW jointly specify endodermal cell fate but distinctly control SHORT-ROOT movement[J].The Plant Journal, 2015, 84(4): 773-784.doi:10.1111/tpj.13038.
[11] Cui H C, Kong D Y, Liu X W, Hao Y L.SCARECROW, SCR-LIKE 23 and SHORT-ROOT control bundle sheath cell fate and function in Arabidopsis thaliana[J].The Plant Journal, 2014, 78(2): 319-327.doi:10.1111/tpj.12470.
[12] Zhang Z L, Ogawa M, Fleet C M, Zentella R, Hu J H, Heo J O, Lim J, Kamiya Y, Yamaguchi S, Sun T P.SCARECROW-LIKE 3 promotes gibberellin signaling by antagonizing master growth repressor DELLA in Arabidopsis[J].Proceedings of the National Academy of Sciences, 2011, 108(5): 2160-2165.doi:10.1073/pnas.1012232108.
[13] Heo J O, Chang K S, Kim I A, Lee M H, Lee S A, Song S K, Lee M M, Lim J.Funneling of gibberellin signaling by the GRAS transcription regulator SCARECROW-LIKE 3 in the Arabidopsis root[J].Proceedings of the National Academy of Sciences, 2011, 108(5): 2166-2171.doi:10.1073/pnas.1012215108.
[14] 马洪双, 夏新莉, 尹伟伦.胡杨SCL7基因及其启动子片段的克隆与分析[J].北京林业大学学报, 2011, 33(1): 1-10.doi:10.13332/j.1000-1522.2011.01.014.
Ma H S, Xia X L, Yin W L.Cloning and analysis of SCL7 gene from Populus euphratica[J].Journal of Beijing Forestry University 2011, 33(1): 1-10.
[15] Ma H S, Liang D, Shuai P, Xia X L, Yin W L.The salt-and drought-inducible poplar GRAS protein SCL7 confers salt and drought tolerance in Arabidopsis thaliana[J].Journal of Experimental Botany, 2010, 61(14): 4011-4019.doi:10.1093/jxb/erq217.
[16] 郭鹏, 邢新, 金华, 董燕.玉米ZmSCL7的克隆及功能研究[J].中国农业科学, 2013, 46(12): 2584-2591.doi:10.3864/j.issn.0578-1752.2013.12.020.
Guo P, Xing X, Jin H, Dong Y.Cloning and functional study of ZmSCL7 in Zea mays[J].Scientia Agricultura Sinica, 2013, 46(12): 2584-2591.
[17] 李丽丽, 曾卫军, 李艳红, 葛风伟, 卢函, 雷维, 杜钰, 谢红桃, 赵和平, 赵惠新.独行菜GRAS转录因子家族分析及LaSCL18基因克隆与冷相关性研究[J].分子植物育种, 2017, 15(9): 3428-3437.doi:10.13271/j.mpb.015.003428.
Li L L, Zeng W J, Li Y H, Ge F W, Lu H, Lei W, Du Y, Xie H T, Zhao H P, Zhao H X.Analysis of GRAS family transcription factors, cloning and researching cold tolerance of LaSCL18 gene in Lepidium[J].Molecular Plant Breeding, 2017, 15(9): 3428-3437.
[18] Fode B, Siemsen T, Thurow C, Weigel R, Gatz C.The Arabidopsis GRAS protein SCL14 interacts with class Ⅱ TGA transcription factors and is essential for the activation of stress-inducible promoters[J].The Plant Cell, 2008, 20(11): 3122-3135.doi:10.1105/tpc.108.058974.
[19] Chen K M, Li H W, Chen Y F, Zheng Q, Li B, Li Z S.TaSCL14, a novel wheat(Triticum aestivum L.)GRAS gene, regulates plant growth, photosynthesis, tolerance to photooxidative stress, and senescence[J].Journal of Genetics and Genomics, 2015, 42(1): 21-32.doi:10.1016/j.jgg.2014.11.002.
[20] Zhang S, Li X W, Fan S D, Zhou L J, Wang Y.Overexpression of HcSCL13, a Halostachys caspica GRAS transcription factor, enhances plant growth and salt stress tolerance in transgenic Arabidopsis[J].Plant Physiology and Biochemistry, 2020, 151:243-254.doi:10.1016/j.plaphy.2020.03.020.
[21] 张占田, 孙雅菲, 艾昊, 罗闻真, 冯冰, 孙文献, 徐国华, 孙淑斌.水稻转录因子基因OsSHR2的表达特征及其在营养生长中的调控作用[J].中国水稻科学, 2018, 32(5): 427-436.doi:10.16819/j.1001-7216.2018.7037.
Zhang Z T, Sun Y F, Ai H, Luo W Z, Feng B, Sun W X, Xu G H, Sun S B.Expression patterns and regulation of transcription factor gene OsSHR2 in vegetative growth in rice[J].Chinese Journal of Rice Science, 2018, 32(5): 427-436.
[22] Zhang H, Zhu S S, Liu T Z, Wang C M, Cheng Z J, Zhang X, Chen L P, Sheng P K, Cai M H, Li C N, Wang J C, Zhang Z, Chai J T, Zhou L, Lei C L, Guo X P, Wang J L, Wang J, Jiang L, Wu C Y, Wan J M.DELAYED HEADING DATE1 interacts with OsHAP5C/D, delays flowering time and enhances yield in rice[J].Plant Biotechnology Journal, 2019, 17(2): 531-539.doi:10.1111/pbi.12996.
[23] Hirano K, Yoshida H, Aya K, Kawamura M, Hayashi M, Hobo T, Sato-Izawa K, Kitano H, Ueguchi-Tanaka M, Matsuoka M.SMALL ORGAN SIZE 1 and SMALL ORGAN SIZE 2/DWARF and LOW-TILLERING form a complex to integrate auxin and brassinosteroid signaling in rice[J].Molecular Plant, 2017, 10(4): 590-604.doi:10.1016/j.molp.2016.12.013.
[24] Liao Z G, Yu H, Duan J B, Yuan K, Yu C J, Meng X B, Kou L Q, Chen M J, Jing Y H, Liu G F, Smith S M, Li J Y.SLR1 inhibits MOC1 degradation to coordinate tiller number and plant height in rice[J].Nature Communications, 2019, 10(1): 2738.doi:10.1038/s41467-019-10667-2.
[25] Shao G N, Lu Z F, Xiong J S, Wang B, Jing Y H, Meng X B, Liu G F, Ma H Y, Liang Y, Chen F, Wang Y H, Li J Y, Yu H.Tiller bud formation regulators MOC1 and MOC3 cooperatively promote tiller bud outgrowth by activating FON1 expression in rice[J].Molecular Plant, 2019, 12(8): 1090-1102.doi:10.1016/j.molp.2019.04.008.
[26] Liu W, Kohlen W, Lillo A, den Camp R O, Ivanov S, Hartog M, Limpens E, Jamil M, Smaczniak C, Kaufmann K, Yang W C, Hooiveld G J E J, Charnikhova T, Bouwmeester H J, Bisseling T, Geurts R.Strigolactone biosynthesis in Medicago truncatula and rice requires the symbiotic GRAS-type transcription factors NSP1 and NSP2[J].The Plant Cell, 2011, 23(10): 3853-3865.doi:10.1105/tpc.111.089771.
[27] Lin Z M, Yan J W, Su J, Liu H Q, Hu C Q, Li G, Wang F, Lin Y.Novel OsGRAS19 mutant, D26, positively regulates grain shape in rice(Oryza sativa)[J].Functional Plant Biology, 2019, 46(9): 857-868.doi:10.1071/fp18266.
[28] Xu K, Chen S J, Li T F, Ma X S, Liang X H, Ding X F, Liu H Y, Luo L J.OsGRAS23, a rice GRAS transcription factor gene, is involved in drought stress response through regulating expression of stress-responsive genes[J].BMC Plant Biology, 2015, 15: 141.doi:10.1186/s12870-015-0532-3.
[29] Henry S, Dievart A, Divol F, Pauluzzi G, Meynard D, Swarup R, Wu S, Gallagher K L, Périn C.SHR overexpression induces the formation of supernumerary cell layers with cortex cell identity in rice[J].Developmental Biology, 2017, 425(1): 1-7.doi:10.1016/j.ydbio.2017.03.001.
[30] Kamiya N, Itoh J I, Morikami A, Nagato Y, Matsuoka M.The SCARECROW gene′s role in asymmetric cell divisions in rice plants[J].The Plant Journal, 2003, 36(1): 45-54.doi:10.1046/j.1365-313x.2003.01856.x.
[31] Niu Y L, Zhao T T, Xu X Y, Li J F.Genome-wide identification and characterization of GRAS transcription factors in tomato(Solanum lycopersicum)[J].Peer J, 2017, 5:e3955.doi:10.7717/peerj.3955.
[32] Zeng X, Ling H, Chen X M, Guo S X.Genome-wide identification, phylogeny and function analysis of GRAS gene family in Dendrobium catenatum(Orchidaceae)[J].Gene, 2019, 705:5-15.doi:10.1016/j.gene.2019.04.038.
[33] Livak K J, Schmittgen T D.Analysis of relative gene expression data using Real-time quantitative PCR and the 2-ΔΔCTmethod[J].Methods, 2001, 25(4): 402-408.doi:10.1006/meth.2001.1262.
[34] Xie X R, Ma X L, Zhu Q L, Zeng D C, Li G S, Liu Y G.CRISPR-GE: A convenient software toolkit for CRISPR-based genome editing[J].Molecular Plant, 2017, 10(9): 1246-1249.doi:10.1016/j.molp.2017.06.004.
[35] Toki S, Hara N, Ono K, Onodera H, Tagiri A, Oka S, Tanaka H.Early infection of scutellum tissue with Agrobacterium allows high-speed transformation of rice[J].The Plant Journal, 2006, 47(6): 969-976.doi:10.1111/j.1365-313X.2006.02836.x.
[36] 陈立勇, 柴丽娟, 陈尚武, 马会勤.葡萄SCARECROW Like 14-Like基因的表达特征及胁迫响应研究[J].中国农业大学学报, 2014, 19(3): 80-87.doi:10.11841/j.issn.1007-4333.2014.03.11.
Chen L Y, Chai L J, Chen S W, Ma H Q.Expression pattern and stress response of grape SCARECROW Like 14-Like gene[J].Journal of China Agricultural University, 2014, 19(3): 80-87.
[37] Chen L S, Xiong G S, Cui X, Yan M X, Xu T, Qian Q, Xue Y B, Li J Y, Wang Y H.OsGRAS19 may be a novel component involved in the brassinosteroid signaling pathway in rice[J].Molecular Plant, 2013, 6(3): 988-991.doi:10.1093/mp/sst027.
[38] Yuan Y Y, Fang L C, Karungo S K, Zhang L L, Gao Y Y, Li S H, Xin H P.Overexpression of VaPAT1, a GRAS transcription factor from Vitis amurensis, confers abiotic stress tolerance in Arabidopsis[J].Plant Cell Reports, 2016, 35(3): 655-666.doi:10.1007/s00299-015-1910-x.
[39] Habib S, Waseem M, Li N, Yang L, Li Z G.Overexpression of SlGRAS7 affects multiple behaviors leading to confer abiotic stresses tolerance and impacts gibberellin and auxin signaling in tomato[J].International Journal of Genomics, 2019, 2019: 4051981.doi:10.1155/2019/4051981.
[40] 王爽, 王永鑫, 王瑜, 李辉, 滕瑞敏, 庄静.茶树CsCIGR基因克隆及表达特性分析[J].西北植物学报, 2019, 39(5): 867-875.doi:10.7606/j.issn.1000-4025.2019.05.0867.
Wang S, Wang Y X, Wang Y, Li H, Teng R M, Zhuang J.Cloning and expression profile analysis of CsCIGR gene in Camellia sinensis[J].Acta Botanica Boreali-Occidentalia Sinica, 2019, 39(5): 867-875.
[41] 牛义岭, 姜秀明, 许向阳.番茄GRAS转录因子家族基因SIGLD1的克隆及VIGS载体构建[J].基因组学与应用生物学, 2016, 35(8): 2155-2160.doi:10.13417/j.gab.035.002155.
Niu Y L, Jiang X M, Xu X Y.Cloning and VIGS vector construction of GRAS transcription factors family gene SIGLD1 from tomato[J].Genomics and Applied Biology, 2016, 35(8): 2155-2160.
[42] 石瑞, 曹诣斌, 陈文荣, 郭卫东.佛手GRAS基因的克隆及表达分析[J].浙江师范大学学报(自然科学版), 2011, 34(4): 446-451.doi:10.3969/j.issn.1001-5051.2011.04.016.
Shi R, Cao Y B, Chen W R, Guo W D.On cDNA cloning and expression analysis of GRAS gene in fingered citron[J]. Journal of Zhejiang Normal University(Nat Sci),2011, 34(4): 446-451.