热激转录因子 (Heat shock transcription factor, HSF) 是生物体参与热胁迫信号转导与耐热性调控的关键转录因子[1]。研究表明,HSF在接收和传递信号、调控下游基因表达、激发植物抗逆反应以及提高植物耐热性的过程中发挥重要作用[2]。热激转录因子能够通过特异性识别并结合热激元件 (Heat shock element, HSE)激活热激蛋白 (Heat shock protein, HSP)。热激蛋白作为分子伴侣,协助受损蛋白重新折叠、组装、分配和降解,从而提高植物热抵抗能力[3-4]。研究表明,在真核生物中典型的HSF具有5个保守结构域,即DNA结合域 (DNA-binding domain, DBD)、寡聚化结构域 (Oligomerization domain, OD)、转录激活结构域 (C-terminal activation domain, CTAD)、核定位信号 (Nuclear localization signals, NLS)以及核输出信号 (Nuclear export signals, NES)。其中DBD与OD结构域最为保守,分别用于精准识别并结合HSE与HSP的启动子;NLS和NES结构域用于保障HSF的跨核运输,维持了基因在细胞核与细胞质中的自由分布;CTAD具有转录激活功能,可以直接调控热激过程中的相关基因表达[5-8]。
HSF基因最早在酵母中被克隆,随后多种哺乳动物HSF相继被挖掘。然而植物的第1个HSF基因是在番茄中克隆得到的[5],随着基因组测序技术的不断完善,HSF又相继在拟南芥、水稻、大豆、玉米等植物中被报道。不同植物的HSF基因家族成员数量差异较大,小麦中数量最多含有56个[8],大豆[9]、玉米[10]、拟南芥[11]中分别含有52,30,21个。目前有关植物HSF基因功能的研究主要集中在高温、干旱响应与生长发育等方面[2,7,12-16]。此外,其他研究表明HSF在冷胁迫应答中发挥着重要作用。朱华生[17]指出玉米ZmHsf04基因受低温胁迫诱导表达;余文才等[18]发现低温条件下巴西橡胶树HbHsfA4a基因的表达量显著上调。
蓖麻 (Ricinus communis L.) 是一种油料作物,被广泛应用于医药、化工、航空航天等领域,其含油量丰富,稳居世界十大油料作物之首,有“可再生绿色石油资源”的美称[19]。作为一种热带作物,蓖麻具有喜温不耐冷的特点,但目前关于蓖麻HSF基因的研究还未见报道。本研究对蓖麻HSF基因家族进行全基因组鉴定,利用生物信息学手段分析该基因家族成员的序列特征、染色体定位及基因组复制事件,结合冷胁迫下蓖麻叶片的转录组数据,分析蓖麻RcHSF基因在冷胁迫下的表达模式,以期为更深入研究蓖麻HSF基因家族成员的冷应答机制与生物学功能奠定基础。
1 材料和方法
1.1 试验材料
本研究选用通蓖5号为试验材料,由内蒙古通辽市农业科学研究院提供。将混合的草炭土与蛭石 (混合比例3∶1) 作为栽培土壤,设置室内培养温度24 ℃,相对湿度70%~75%,光照强度450 μmol/(m2·s)。当植株生长到四叶期时,取生长状态相同的植株分别进行0,2,6,8 h冷处理 (4 ℃)。每株幼苗取两片真叶作为样本,将3株幼苗混样作为1次生物学重复,共设置3次生物学重复。液氮速冻样本,用于转录组测序与基因表达试验。
1.2 试验方法
1.2.1 总RNA提取、cDNA合成及转录组测序 通过TRIzol法提取叶片的总RNA,利用DNase Ⅰ去除gDNA污染,Agilent2100 Bioanalyzer和NanoDrop分别检测RNA的完整性和纯度,质检合格后进行文库构建,随后使用Illumina HiSeq X-ten测序仪进行转录组测序。UEIris RT-PCR System (US Everbright,苏州,中国) 反转录合成cDNA第1条链,用于后续的qRT-PCR分析。
1.2.2 蓖麻HSF基因的鉴定与序列分析 蓖麻基因组数据与结构注释信息下载于Phytozome数据库 (https://phytozome.jgi.doe.gov/pz/portal.html),21个拟南芥HSF基因家族成员序列下载于Plant Tair数据库 (https://www.arabidopsis.org/),24个水稻HSF基因家族序列信息下载于NCBI数据库 (https://www.ncbi.nlm.nih.gov/)。利用基因组结构注释信息提取蓖麻全基因组CDS,成功构建蓖麻蛋白数据库。以拟南芥HSF基因家族成员为探针检索蓖麻蛋白数据库,去除冗余的蓖麻蛋白,预筛选HSF同源蛋白,E-value值小于0.01。提交预筛选蛋白于NCBI-swissprot数据库检索同源蛋白信息,去除蓖麻基因组中HSF家族成员的近源序列,最终将含有HSF保守结构域的蛋白序列保留。
1.2.3 蓖麻HSF家族成员的理化性质 利用Prot Param工具 (https://web.expasy.org/protparam/) 分析蓖麻HSF蛋白的氨基酸数量、等电点、分子量。利用CELLO工具(http://cello.life. nctu.edu. tw/)在线预测蓖麻HSF家族成员的亚细胞定位信息。利用TMHMM 2.0(http://www.cbs.dtu.dk/services/TMHMM/)工具统计蓖麻HSF家族成员的跨膜结构域。
1.2.4 蓖麻HSF保守基序与结构域分析 利用MEME在线工具 (http://meme.nbcr.net/meme/tools/meme) 预测蓖麻HSF蛋白的保守motif;参数设置:设置位点分布情况为任意重复次数,设置保守基序上限为10,设置基序大小为10~60 bp,其他参数均为默认。利用Batch CD-search 在线工具 (https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi) 分析蛋白的保守结构域。
1.2.5 蓖麻HSF家族成员进化分析和组分鉴定 利用MEGA 5.1工具对拟南芥、水稻与蓖麻HSF蛋白序列进行比对,最大迭代次数设置为100,其他参数默认;参考比对结果,采用邻接法 (Neighbor-Joining, NJ) 构建系统进化树,选择JTT模型,采用Partial deletion处理遗漏数据,设置Bootstrap检验次数为1 000。
1.2.6 蓖麻HSF染色体定位信息 从蓖麻基因组注释信息中获取RcHSF基因家族成员的染色体定位信息,使用TBtools绘制基因染色体分布图,通过微共线性分析,探讨基因复制事件。
1.2.7 蓖麻HSF基因家族成员顺式作用元件预测 提取RcHSF基因起始位点上游2 000 bp的启动子区序列,利用Plant CARE在线工具预测该家族成员与环境胁迫、激素调控等有关的顺式作用元件,以此进一步推测基因家族成员具有的潜在功能。
1.2.8 蓖麻HSF在冷胁迫下的转录组分析 结合蓖麻冷胁迫 (4 ℃) 下的转录组数据,对FoldChange标准化数值进行可视化分析,分析RcHSF基因在冷胁迫下的转录水平。
1.2.9 蓖麻HSF在冷胁迫下的表达验证 利用qRT-PCR验证差异表达基因在冷胁迫下的表达水平。参考王晓宇等[20]的研究方法,将RcADP (XP_002515227) 与RcEF1β (XP_002511200) 作为内参基因。利用Primer premier 5设计目的基因与内参基因的特异性引物 (表1)。qRT-PCR所用仪器为qTOWER 2.2 (Analytik Jena,德国),其反应体系与程序设置参考2×SYBR Green qPCR Master Mix (USEverbright,中国,苏州) 试剂盒说明书。数据采用平均数±标准差表示,并利用2-ΔΔCt法进行基因相对表达量的计算,两组间比较采用Students′t-test,当P<0.05时认为统计学上具有显著差异。
表1 qRT-PCR引物序列信息
Tab.1 Primers used for qRT-PCR
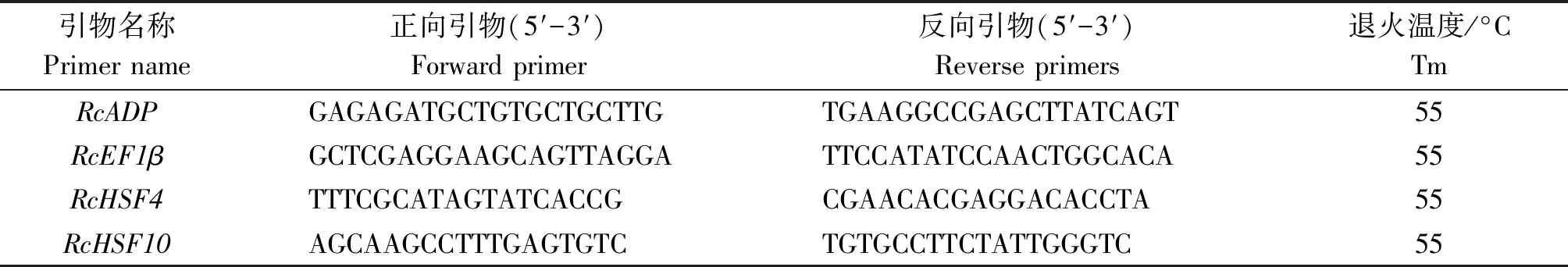
引物名称Primer name正向引物(5′-3′) Forward primer反向引物(5′-3′)Reverse primers退火温度/°CTmRcADPGAGAGATGCTGTGCTGCTTGTGAAGGCCGAGCTTATCAGT55RcEF1βGCTCGAGGAAGCAGTTAGGATTCCATATCCAACTGGCACA55RcHSF4TTTCGCATAGTATCACCGCGAACACGAGGACACCTA55RcHSF10AGCAAGCCTTTGAGTGTCTGTGCCTTCTATTGGGTC55
2 结果与分析
2.1 蓖麻HSF基因家族鉴定与结构分析
本研究从NCBI数据库获取蓖麻全基因组信息,通过拟南芥HSF基因家族比对和Swissprot数据库验证,筛选出20个蓖麻HSF基因家族成员。利用NCBI-Batch CD-search保守结构域预测去除1个错误比对 (30190.m011154.TIGRR0.1),最终成功鉴定到19个蓖麻HSF基因家族成员,并根据染色体定位顺序对所有成员依次命名 (表2)。CELLO工具预测显示所有蛋白均定位于细胞核;Prot Param工具分析表明,19个RcHSF蛋白的氨基酸个数在84 (RcHSF16)~521(RcHSF2),平均氨基酸个数为366;分子量 (ku) 大小在9.605 8(RcHSF16)~58.650 6(RcHSF2);等电点(pI)为4.68(RcHSF11)~9.76(RcHSF7),其中RcHSF11最小,显示出较弱的溶解度与更强的沉淀能力,而RcHSF7分子间相对作用力较大,推测沉降系数低。尽管RcHSF16的氨基酸数量明显少于HSF家族其他成员,但基于Domains保守结构域分析,该蛋白存在明显的HSF活性位点,推测能发挥完整的HSF蛋白功能。
RcHSFs基因结构分析显示,所有成员均含有长度与数量不等的UTR与CDS,基因RcHSF1、RcHSF2、RcHSF4、RcHSF5、RcHSF6、RcHSF7、RcHSF8、RcHSF9、RcHSF11、RcHSF14、RcHSF15、RcHSF16、RcHSF18均呈现以外显子开头和结尾的特殊结构,其中RcHSF15仅含有1个外显子,RcHSF1与RcHSF4含有3个外显子,其他成员均含有2个外显子 (图1)。
表2 蓖麻RcHSF基因家族成员信息
Tab. 2 The information of RcHSF gene family in Ricinus communis L.
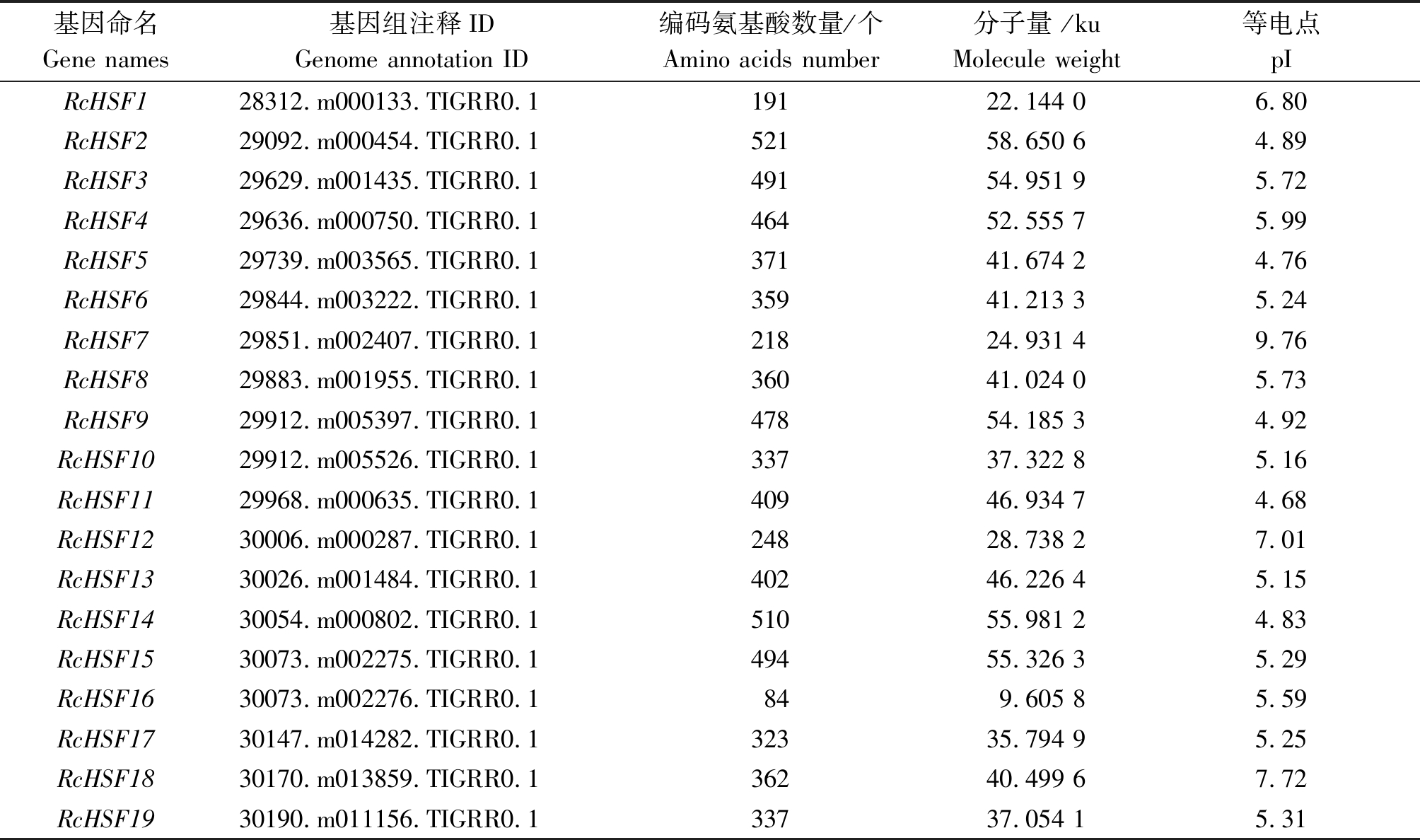
基因命名Gene names基因组注释IDGenome annotation ID编码氨基酸数量/个Amino acids number分子量 /kuMolecule weight等电点pIRcHSF128312.m000133.TIGRR0.119122.144 06.80RcHSF229092.m000454.TIGRR0.152158.650 64.89RcHSF329629.m001435.TIGRR0.149154.951 95.72RcHSF429636.m000750.TIGRR0.146452.555 75.99RcHSF529739.m003565.TIGRR0.137141.674 24.76RcHSF629844.m003222.TIGRR0.135941.213 35.24RcHSF729851.m002407.TIGRR0.121824.931 49.76RcHSF829883.m001955.TIGRR0.136041.024 05.73RcHSF929912.m005397.TIGRR0.147854.185 34.92RcHSF1029912.m005526.TIGRR0.133737.322 85.16RcHSF1129968.m000635.TIGRR0.140946.934 74.68RcHSF1230006.m000287.TIGRR0.124828.738 27.01RcHSF1330026.m001484.TIGRR0.140246.226 45.15RcHSF1430054.m000802.TIGRR0.151055.981 24.83RcHSF1530073.m002275.TIGRR0.149455.326 35.29RcHSF1630073.m002276.TIGRR0.1849.605 85.59RcHSF1730147.m014282.TIGRR0.132335.794 95.25RcHSF1830170.m013859.TIGRR0.136240.499 67.72RcHSF1930190.m011156.TIGRR0.133737.054 15.31
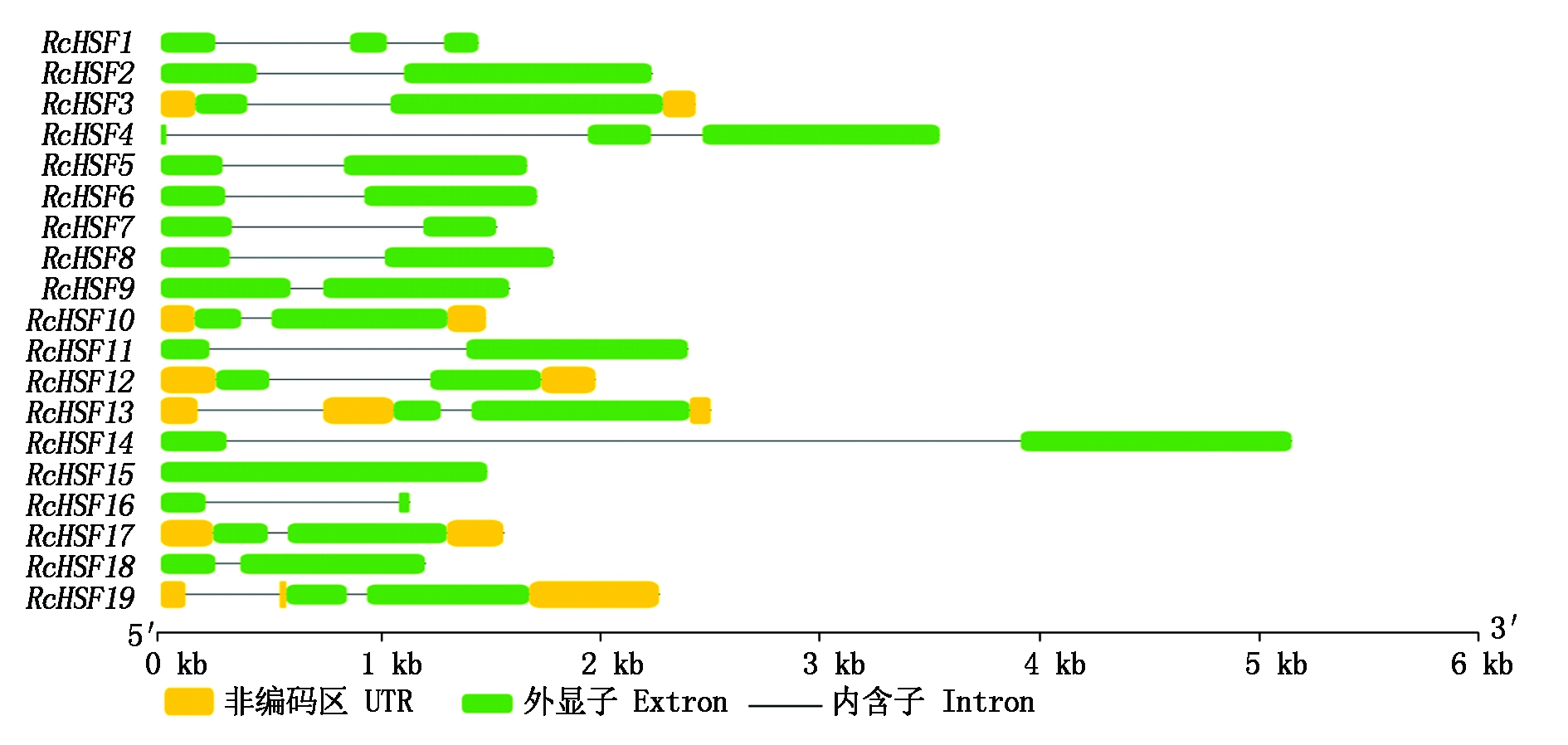
图1 蓖麻RcHSF基因结构分布
Fig.1 Structure of RcHSF gene in Ricinus communis L.
2.2 RcHSF蛋白保守基序及结构域鉴定
利用MEME进一步分析蓖麻HSF蛋白家族成员的保守基序,结果显示,所有RcHSFs的基序类型与排布方式基本相似 (图2-A),但无一例外都存在不同程度的motif缺失,其中RcHSF16仅含有2个保守基序,缺失最为严重。DNA结合域 (DBD) 与寡聚化结构域 (OD) 是保守度最高的结构域,确保了热激转录元件HES的精准定位,尽管本研究中RcHSF15没有DBD基序,但Domains分析显示该基因存在HSF活性,推测可以发挥完整的基因功能;核定位信号NLS是由Lys、Pro、Arg等组成的微小片段,参与并调控转录因子向核内运输的过程,但由于不同物种间NLS序列类型多样,导致转录因子的核运输方式同样存在差异[21],本研究中RcHSF2、RcHSF16、RcHSF17、RcHSF19蛋白没有检测到NLS信号区,故推测热激转录因子可能存在其他NLS非依赖性核运输方式。
Batch CD-search保守结构预测表明,所有RcHSF中均存在有关热激转录因子的活性区 (图2-B)。由于RcHSF15缺少DBD基序,导致其结构域比对率相对较低,该现象同样存在于龙眼HSF家族成员鉴定过程中[22],但在番茄与拟南芥中没有相似案例[11]。
通过氨基酸序列的同源比对,将所有RcHSFs聚为Ⅰ、Ⅱ、Ⅲ 3个组,其中RcHSF2、RcHSF15与其他氨基酸序列的同源性相对较低,不具有代表性,不作分组处理。又根据基序类型与数量又将Ⅰ组成员细化为Ⅰ-1、Ⅰ-2、Ⅰ-3 3个亚组。推测在同组的RcHSFs同源性较高,且存在功能冗余。
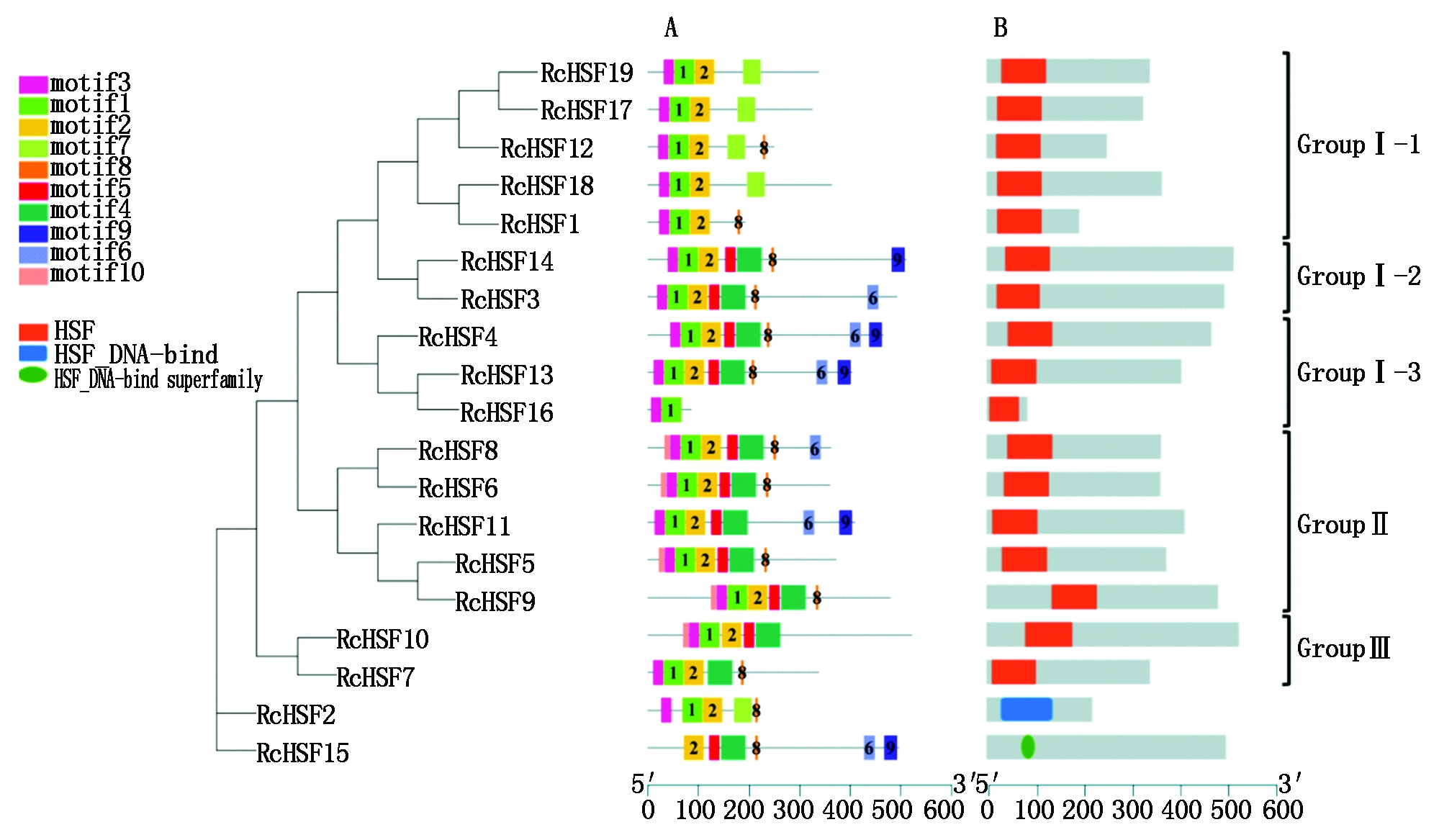
motif 1.DNA结合域 (DBD);motif 2.寡聚化结构域 (OD);motif 6.转录激活结构域 (AHA); motif 8.核定位信号 (NLS);motif 9.核输出信号 (NES)。 motif 1.A DNA binding domain (DBD); motif 2.An oligomerization domain (OD); motif 6.A transcription activation domain (AHA);motif 8.A nuclear localization signal (NLS);motif 9.A nuclear export signal (NES).
图2 蓖麻RcHSF蛋白的保守基序 (A)与结构域 (B)分布
Fig.2 Distribution of conserved motif (A) and conserved domain (B) of castor RcHSF proteins
2.3 RcHSF基因家族系统发育关系
通过比对不同植物的HSF基因,可以更准确分析基因家族成员间的进化关系,也为蛋白的精准分类提供依据。本研究对蓖麻、拟南芥与番茄的HSF氨基酸序列进行比对,并利用MEGA工具构建系统进化树 (图3)。根据序列的同源性将所有HSF蛋白聚为Ⅰ、Ⅱ、Ⅲ 3个主分支与多个亚分支,除了RcHSF4与RcHSF16为旁系同源基因,其他蓖麻HSF家族成员均与拟南芥AtHSF蛋白聚合为末端分支,故推测蓖麻与拟南芥HSF蛋白的同源性高于番茄HSF蛋白。
参考拟南芥HSF聚类方式,将所有HSF聚为A、B、C 3个组,其中只有A组蛋白含有C端启动基序 (AHA),B组蛋白的寡聚化基序 (HR-A/B) 比C组蛋白少7个碱基[23]。组内成员数按照A>B>C进行排序。根据基序的类型与数量,又将A组蛋白分为3个亚组 (A1、A2、A3)。推测在同组中,异源HSF可能具有相似功能。
2.4 RcHSF基因家族成员染色体定位信息
根据基因家族成员的染色体定位信息,使用TBtools工具绘制基因染色体分布图,由于蓖麻基因组拼接与染色体组装不完整,只能将RcHSFs基因定位在未完整拼接的染色体片段上。最终分析结果(图4)显示,所有RcHSF基因被定位在17个染色体片段上,呈不均匀分布。Gu等[24]将2个同源性均大于75%且比对长度大于较长序列80%的基因定义为串联重复基因。本研究中,由于RcHSF9与RcHSF10基因没有相连,RcHSF15与RcHSF16基因的同源性较低,且所有染色体片段之间没有表现出微共线性,充分证明在基因组复制过程中RcHSF基因家族成员并未发生串联重复与片段重复事件。

图3 蓖麻、拟南芥与番茄HSF的系统进化关系
Fig.3 Phylogenetic relationship between castor, Arabidopsis and tomato HSF
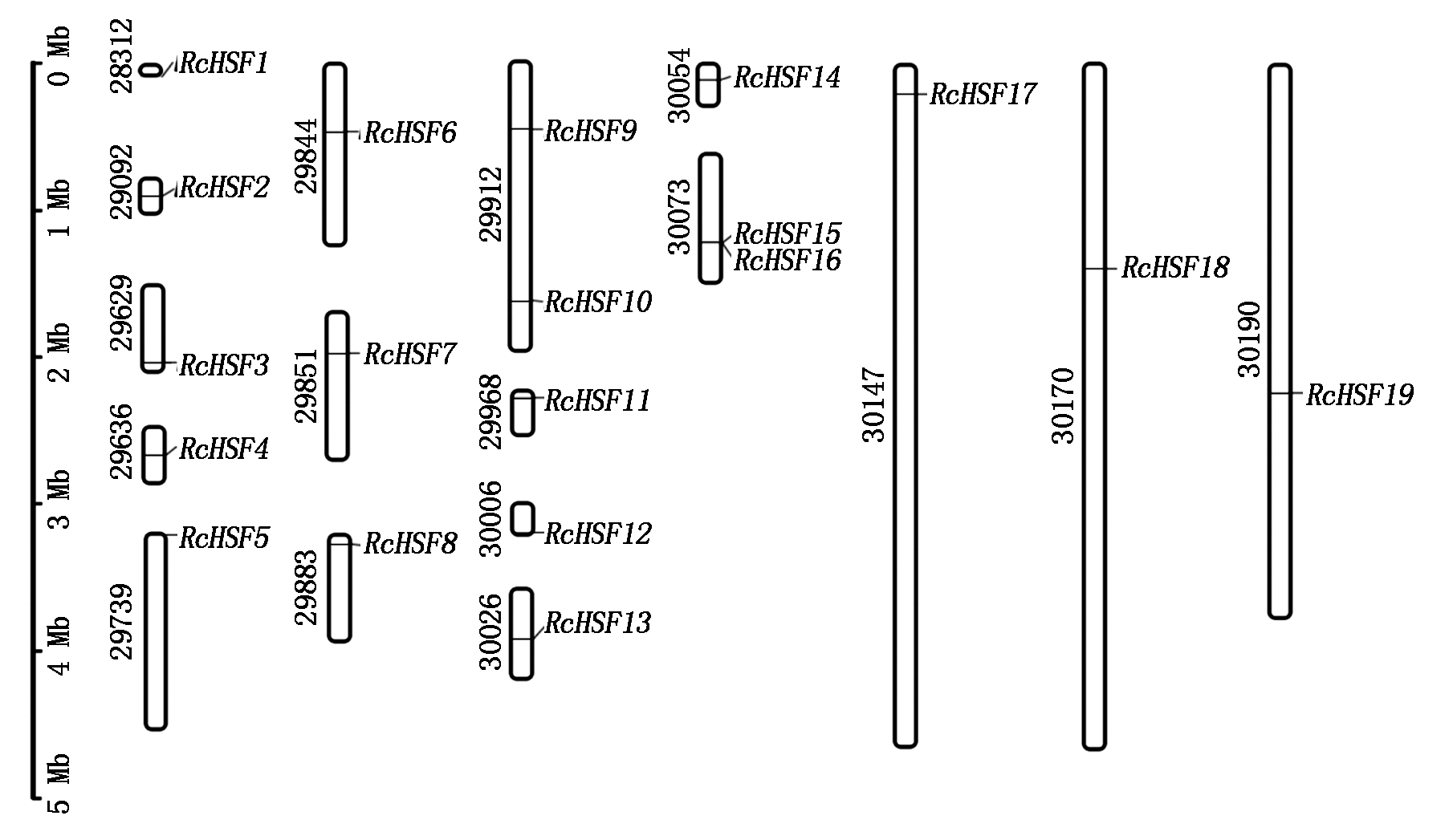
图4 蓖麻RcHSF基因的染色体分布
Fig.4 Chromosome distribution map of RcHSF gene in Ricinus communis L.
2.5 启动子顺式作用元件的预测分析
为挖掘蓖麻RcHSFs基因的潜在功能,利用Plant CARE工具在线预测处于基因起始位点上游2 000 bp的多种顺势作用元件,基于顺式作用元件的多样性,推测不同基因可能具有多种潜在功能。预测结果(表3)显示,该基因家族含有多种顺势作用元件,其中含有AAGAA-motif、MYB与MYC元件的基因数量最多,证明RcHSFs可能响应植物激素与逆境胁迫,如盐、干旱、低温胁迫与ABA诱导等。但基因表达受多种元件共同调控,复杂的调控类型与调控方式使基因功能多元化,基因功能的专一性仍需进一步验证。
2.6 RcHSF基因在冷胁迫下的表达模式分析
本研究采用DEseq2进行差异表达基因的筛选 (log2(FoldChange)>1;q value<0.001),发现蓖麻中仅有RcHSF4与RcHSF10能够响应低温诱导 (图5)。其中,RcHSF4在冷处理2,6 h时与对照组差异表达不显著,但冷处理8 h的表达量显著上调1.51倍;RcHSF10在冷处理2 h时与对照组差异表达不显著,但在冷处理6,8 h后差异表达水平均显著上调,上调倍数分别为1.05,1.61倍。该结果表明,RcHSF4与RcHSF10可能在蓖麻抗冷方面发挥着重要作用。
表3 蓖麻RcHSF基因家族成员启动子顺式作用元件分析
Tab.3 The cis acting element analysis of promoter region of castor RcHSF gene family members
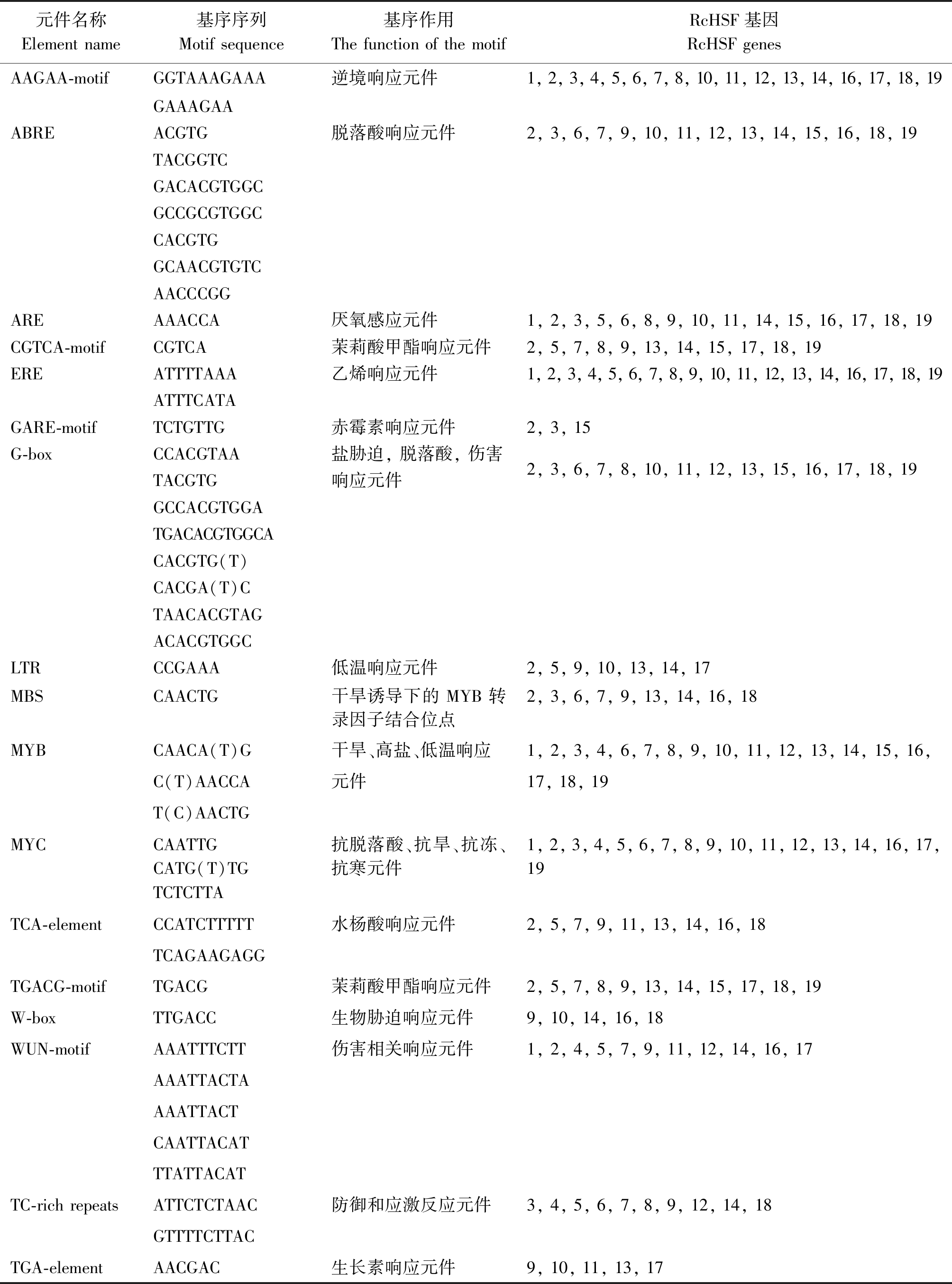
元件名称Element name基序序列Motif sequence基序作用The function of the motifRcHSF基因RcHSF genesAAGAA-motifGGTAAAGAAA逆境响应元件 1, 2, 3, 4, 5, 6, 7, 8, 10, 11, 12, 13, 14, 16, 17, 18, 19GAAAGAAABREACGTG脱落酸响应元件2, 3, 6, 7, 9, 10, 11, 12, 13, 14, 15, 16, 18, 19TACGGTCGACACGTGGCGCCGCGTGGCCACGTGGCAACGTGTCAACCCGGAREAAACCA厌氧感应元件1, 2, 3, 5, 6, 8, 9, 10, 11, 14, 15, 16, 17, 18, 19CGTCA-motifCGTCA茉莉酸甲酯响应元件2, 5, 7, 8, 9, 13, 14, 15, 17, 18, 19EREATTTTAAA乙烯响应元件1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 16, 17, 18, 19ATTTCATAGARE-motifTCTGTTG赤霉素响应元件2, 3, 15G-boxCCACGTAA盐胁迫, 脱落酸, 伤害TACGTG响应元件2, 3, 6, 7, 8, 10, 11, 12, 13, 15, 16, 17, 18, 19GCCACGTGGATGACACGTGGCACACGTG(T)CACGA(T)CTAACACGTAGACACGTGGCLTRCCGAAA低温响应元件2, 5, 9, 10, 13, 14, 17MBSCAACTG干旱诱导下的 MYB 转录因子结合位点2, 3, 6, 7, 9, 13, 14, 16, 18MYBCAACA(T)G干旱、高盐、低温响应1, 2, 3, 4, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, C(T)AACCA元件17, 18, 19T(C)AACTGMYCCAATTGCATG(T)TGTCTCTTA抗脱落酸、抗旱、抗冻、抗寒元件1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 16, 17, 19TCA-elementCCATCTTTTT水杨酸响应元件2, 5, 7, 9, 11, 13, 14, 16, 18TCAGAAGAGGTGACG-motifTGACG茉莉酸甲酯响应元件2, 5, 7, 8, 9, 13, 14, 15, 17, 18, 19W-boxTTGACC生物胁迫响应元件9, 10, 14, 16, 18WUN-motifAAATTTCTT伤害相关响应元件1, 2, 4, 5, 7, 9, 11, 12, 14, 16, 17AAATTACTAAAATTACTCAATTACATTTATTACATTC-rich repeatsATTCTCTAAC防御和应激反应元件3, 4, 5, 6, 7, 8, 9, 12, 14, 18GTTTTCTTACTGA-elementAACGAC生长素响应元件9, 10, 11, 13, 17
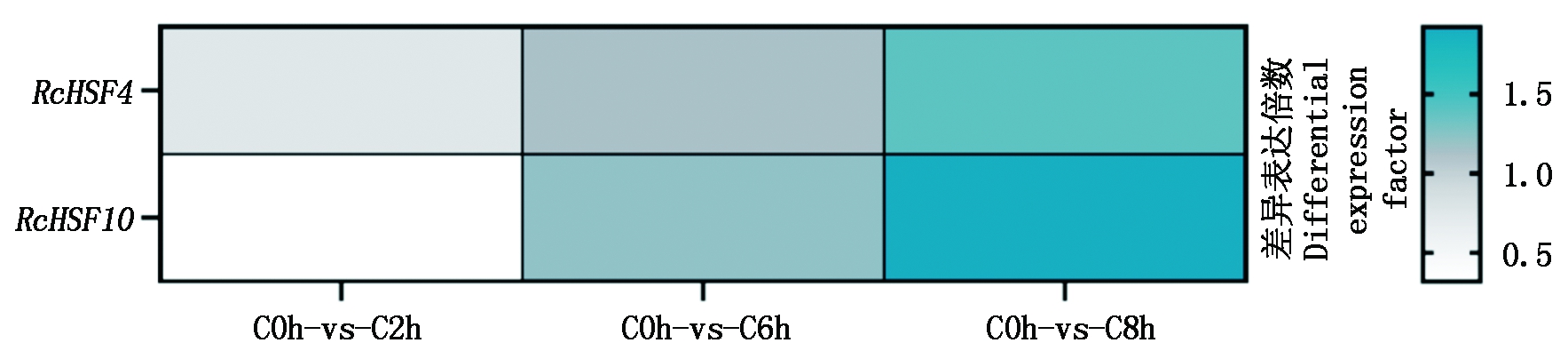
C0h-vs-C2h.冷处理0,2 h的差异表达倍数;C0h-vs-C6h.冷处理0,6 h的 差异表达倍数;C0h-vs-C8h.冷处理0,8 h的差异表达倍数。 C0h-vs-C2h.The differential expression multiple of cold treatment 0 h and 2 h;C0h-vs-C6h.The differential expression multiple of cold treatment 0 h and 6 h;C0h-vs-C8h.The differential expression multiple of cold treatment 0 h and 8 h.
图5 冷胁迫下 RcHSF差异表达基因的转录水平
Fig.5 Transcription level of differentially expressed genes of RcHSF under cold stress
2.7 RcHSF基因受冷胁迫的表达水平验证
为了验证转录组测序结果的可重复性,本研究对蓖麻RcHSF4与RcHSF10进行qRT-PCR分析(图6)。结果显示,这2个基因在不同持续时间冷处理下的表达水平与RNA-seq的数据基本一致,表明测序结果可靠。该结果进一步证明,RcHSF4与RcHSF10可能对于调节蓖麻冷耐受性具有重要意义,其作用途径有待进一步探究。
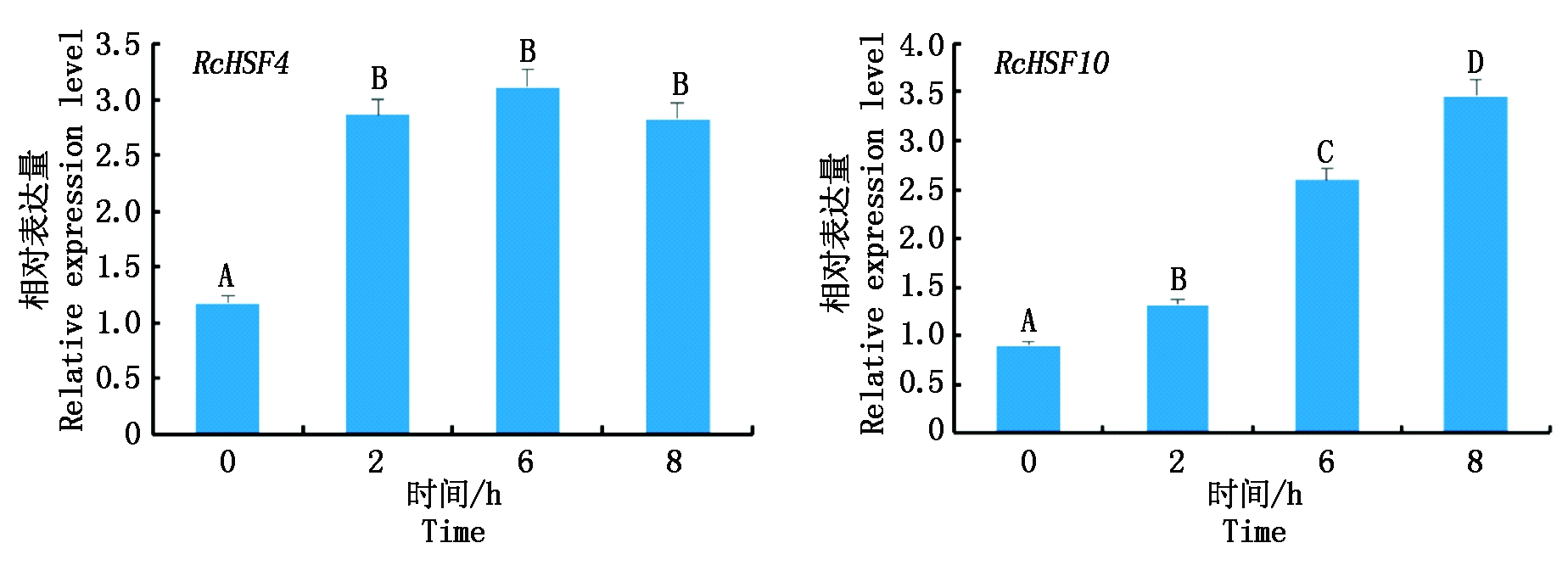
大写字母A、B、C、D代表基因受不同时间冷处理差异表达极显著 (P<0.01)。 The capital letters A, B, C and D represent that the differential expression of genes is extremely significant after cold treatment at different times(P<0.01).
图6 蓖麻RcHSF4与RcHSF10在冷胁迫下的表达水平
Fig.6 Expression levels of RcHSF4 and RcHSF10 of castor under cold stress
3 讨论与结论
蓖麻全基因组测序的实现为RcHSF基因家族的鉴定提供了便利,本研究在蓖麻全基因组范围内挖掘RcHSF基因家族成员,最终成功鉴定到19个热激转录因子,其数量约占所有编码基因(31 221)的0.000 6%。与拟南芥 (21)、番茄 (25)、玉米 (30)等植物相比,蓖麻HSF基因数量相对较少,这可能与蓖麻基因组注释不完全或染色体组装不完整有关。利用同源比对与多物种比对,将RcHSF基因进一步聚类为A、B、C 3个组,其中A组基因数量最多,C组数量最少,该聚类方式与拟南芥HSF基因家族成员的聚类方式相同[11],推测基序类型与排布顺序相似的同源基因,可能存在功能冗余,异源基因可能具有相同功能。雷娟利等[25]发现当拟南芥受到热激胁迫时,A类HSF的表达量上升最为明显,过表达AtHSFA2基因能够显著提高植物的基础耐热性;B类HSF也能够识别并结合热启动元件HSE,但受到羧基末端特征结构域 (-LFGV-) 的阻遏作用,导致既无法正常启动热响应基因的表达[21],又无法调节植物的热耐受性。因此,不同类型蓖麻RcHSF的蛋白功能与作用方式是否存在差异有待进一步考证。
进化分析表明,大部分蓖麻HSF基因与拟南芥HSF基因被聚为同一分支末端,属于直系同源基因,而在蓖麻基因组中仅存在RcHSF4与RcHSF16一对旁系同源基因,表明在植物漫长的进化过程中,大部分HSF基因均按照物种特异性的方式进行扩展,该现象在植物其他基因家族的研究中也得到了广泛验证[26-28]。
大多数RcHSFs启动子区均含有AAGAA-motif、MYB与MYC元件,暗示其响应植物激素与逆境胁迫。目前,植物HSF基因的功能研究主要集中在干旱与盐胁迫,而在调节植物冷适应性方面鲜有报道。郭丽红等[29]研究发现,低温胁迫下拟南芥AtHSFA1A可以通过负调控Caspase-3蛋白酶活性,进而抑制细胞程序性死亡。作物抗冷性是由多基因控制的复杂性状,因此,耐冷数量性状基因座的定位是抗冷基因克隆的前提。本研究结合蓖麻基因组与转录组数据对RcHSF进行分析,发现冷胁迫下仅RcHSF4与RcHSF10差异表达显著;其中 RcHSF4在冷处理早期差异表达倍数没有明显变化,而RcHSF10的差异表达倍数随冷处理时间的延长逐渐提高。该表达模式与茶树CsHSFA4、CsHSFA5的表达模式相似[30],推测RcHSF4与RcHSF10的上调表达对于蓖麻适应低温胁迫、触发冷响应机制具有重要意义。本研究为探索蓖麻HSF基因家族成员的冷应答机制提供重要数据,也为蓖麻抗冷新种质资源的建立提供候选基因。
[1] 翁锦周, 洪月云. 植物热激转录因子在非生物逆境中的作用[J]. 分子植物育种, 2006, 4(1): 88-94. doi:10.3969/j.issn.1672-416X.2006.01.016.
Weng J Z, Hong Y Y. The roles of plant heat shock transcription factors in abiotic stress[J]. Molecular Plant Breeding, 2006, 4(1): 88-94.
[2] Ohama N, Sato H, Shinozaki K, Yamaguchi-Shinozaki K. Transcriptional regulatory network of plant heat stress response[J]. Trends in Plant Science,2017, 22(1): 53-65. doi:10.1016/j.tplants.2016.08.015.
[3] Guo M, Liu J H, Ma X, Luo D X, Gong Z H, Lu M H. The plant heat stress transcription factors (HSFs): structure, regulation, and function in response to abiotic stresses[J]. Frontiers in Plant Science, 2016, 7: 114. doi:10.3389/fpls.2016.00114.
[4] Bharti K, Koskull-Döring P V, Bharti S, Kumar P, Tintschl-Körbitzer A, Treuter E, Nover L. Tomato heat stress transcription factor HsfB1 represents a novel type of general transcription coactivator with a histone-like motif interacting with the plant CREB binding protein ortholog HAC1[J]. The Plant Cell, 2004, 16(6): 1521-1535. doi:10.1105/tpc.019927.
[5] Scharf K D, Rose S, Zott W, Schöff F, Nover L,Schöff F. Three tomato genes code for heat stress transcription factors with a region of remarkable homology to the DNA-binding domain of the yeast HSF[J]. The EMBO Journal, 1990, 9(13): 4495-4501. doi:10.1002/j.1460-2075.1990.tb07900.x.
[6] Li P S, Yu T F, He G H, Chen M, Zhou Y B, Chai S C, Xu Z S, Ma Y Z. Genome-wide analysis of the Hsf family in soybean and functional identification of GmHsf-34 involvement in drought and heat stresses[J]. BMC Genomics, 2014, 15(1): 1009. doi:10.1186/1471-2164-15-1009.
[7] Zhu X Y, Huang C Q, Zhang L, Liu H F, Yu J H, Hu Z Y, Hua W. Systematic analysis of Hsf family genes in the Brassica napus genome reveals novel responses to heat, drought and high CO2 stresses[J]. Frontiers in Plant Science, 2017, 8: 1174. doi:10.3389/fpls.2017.01174.
[8] Scharf K D, Berberich T, Ebersberger I, Nover L. The plant heat stress transcription factor (Hsf) family: structure, function and evolution[J]. Biochimica et Biophysica Acta, 2012, 1819(2): 104-119. doi:10.1016/j.bbagrm.2011.10.002.
[9] Chung E, Kim K M, Lee J H. Genome-wide analysis and molecular characterization of heat shock transcription factor family in Glycine max[J]. Journal of Genetics and Genomics, 2013, 40(3): 127-135. doi:10.1016/j.jgg.2012.12.002.
[10] Lin Y X, Jiang H Y, Chu Z X, Tang X L, Zhu S W, Cheng B J. Genome-wide identification, classification and analysis of heat shock transcription factor family in maize[J]. BMC Genomics, 2011, 12(1): 76-89. doi:10.1186/1471-2164-12-76.
[11] Guo J K, Wu J, Ji Q, Wang C, Luo L, Yuan Y, Wang Y H, Wang J. Genome-wide analysis of heat shock transcription factor families in rice and Arabidopsis[J]. Journal of Genetics and Genomics, 2008, 35(2): 105-118. doi:10.1016/s1673-8527(08)60016-8.
[12] Prasinos C, Krampis K, Samakovli D, Hatzopoulos P. Tight regulation of expression of two Arabidopsis cytosolic Hsp90 genes during embryo development[J]. Journal of Experimental Botany, 2005, 56(412): 633-644. doi:10.1093/jxb/eri035.
[13] Swindell W R, Huebner M, Weber A P. Transcriptional profiling of Arabidopsis heat shock proteins and transcription factors reveals extensive overlap between heat and non-heat stress response pathways[J]. BMC Genomics, 2007, 8(1): 125. doi:10.1186/1471-2164-8-125.
[14] Hu W H, Hu G C, Han B. Genome-wide survey and expression profiling of heat shock proteins and heat shock factors revealed overlapped and stress specific response under abiotic stresses in rice[J]. Plant Science, 2009, 176(4): 583-590. doi:10.1016/j.plantsci.2009.01.016.
[15] Giorno F, Wolters-Arts M, Grillo S, Scharf K D, Vriezen W H, Mariani C. Developmental and heat stress regulated expression of HsfA2 and small heat shock proteins in tomato anthers[J]. Journal of Experimental Botany, 2010, 61(2): 453-462. doi:10.1093/jxb/erp316.
[16] Ishka M R, Brown E, Weigand C, Tillett R L, Schlauch K A, Miller G, Harper J F. A comparison of heat-stress transcriptome changes between wild-type Arabidopsis pollen and a heat-sensitive mutant harboring a knockout of cyclic nucleotide-gated cation channel 16 (cngc16)[J]. BMC Genomics, 2018, 19(1): 549. doi:10.1186/s12864-018-4930-4.
[17] 朱华生. 玉米热激转录因子ZmHsf04的克隆及抗逆功能研究[D]. 合肥:安徽农业大学, 2016.
Zhu H S. Cloning and stress-resistance study of heat shock transcription factor ZmHsf04 from Zea mays[D]. Hefei:Anhui Agricultural University, 2016.
[18] 余文才, 李言, 吴绍华, 晁金全, 张世鑫, 杨署光, 田维敏. 巴西橡胶树HbHsfA4a的克隆及其对低温胁迫的响应[J]. 热带作物学报, 2019, 40(5): 898-904. doi:10.3969/j.issn.1000-2561.2019.05.010.
Yu W C, Li Y, Wu S H, Chao J Q, Zhang S X, Yang S G, Tian W M. Cloning of HbHsfA4a and its transcriptional responses to cold stress in Hevea brasiliensis[J]. Chinese Journal of Tropical Crops, 2019, 40(5): 898-904.
[19] 李金琴, 朱国立, 吴国林, 何智彪, 李靖霞, 张春华, 田福东, 贾娟霞. 蓖麻种子含油量与主要数量性状的相关及通径分析[J]. 中国油料作物学报, 2004, 26(2): 43-46.
Li J Q, Zhu G L, Wu G L, He Z B, Li J X, Zhang C H, Tian F D, Jia J X. Correlation and path analysis beteween seed oil content and major quantitative characters in castor[J]. Chinese Journal of Oil Crop Sciences, 2004, 26(2): 43-46.
[20] 王晓宇, 李敏, 刘栩铭, 卜祥琪, 丁雪, 张继星. 蓖麻RcSEC14p基因的克隆及低温胁迫下的表达分析[J]. 分子植物育种, 2019, 17(13): 4204-4209. doi:10.13271/j.mpb.017.004204.
Wang X Y, Li M, Liu X M, Bu X Q, Ding X, Zhang J X. Cloning of RcSEC14p in Ricinus communis L. and expression analysis under cold stress[J]. Molecular Plant Breeding, 2019, 17(13): 4204-4209.
[21] 李家朋. 玉米热激转录因子ZmHsf-17的功能研究[D]. 合肥:安徽农业大学, 2015.
Li J P. Function analysis of heat shock transcription factor ZmHsf-17 of Zea mays[D]. Hefei:Anhui Agricultural University, 2015.
[22] 王云, 彭丽云, 孙雪丽, 高玉莹, 陈晓慧, 陈裕坤, 林玉玲, 赖钟雄. 龙眼Hsf基因家族全基因组鉴定及体胚发生过程中的表达分析[J]. 应用与环境生物学报, 2019, 25(2): 420-431. doi:10.19675/j.cnki.1006-687x.2018.06004.
Wang Y, Peng L Y, Sun X L, Gao Y Y, Chen X H, Chen Y K, Lin Y L, Lai Z X. Genome-wide identification of longan Hsf family members and their functional analysis during somatic embryogenesis in longan[J]. Chinese Journal of Applied and Environmental Biology, 2019, 25(2): 420-431.
[23] 陈先知, 王燕, 史建磊, 朱隆静, 王克磊, 徐坚. 黄瓜全基因组热激转录因子(HSFs)的鉴定与表达分析[J]. 遗传, 2014, 36(4): 376-386.doi:10.3724/SP.J.1005.2014.0376.
Chen X Z, Wang Y, Shi J L, Zhu L J, Wang K L, Xu J. Genome-wide identification, sequence characteristic and expression analysis of heat shock factors (HSFs) in cucumber[J]. Hereditas, 2014, 36(4): 376-386.
[24] Gu Z L, Cavalcanti A, Chen F C, Bouman P, Li W H. Extent of gene duplication in the genomes of Drosophila, nematode, and yeast[J]. Molecular Biology and Evolution, 2002, 19(3): 256-262. doi:10.1093/oxfordjournals.molbev.a004079.
[25] 雷娟利, 徐志豪. 转座子在植物功能基因组学中的应用[J]. 浙江农业学报, 2002, 14(5): 291-296. doi:10.3969/j.issn.1004-1524.2002.05.011.
Lei J L, Xu Z H. Application of transposons in the plant functional genomics[J]. Acta Agriculturae Zhejiangensis, 2002, 14(5): 291-296.
[26] Bai J F, Pennill L A, Ning J C, Lee S W, Ramalingam J, Webb C A, Zhao B Y, Sun Q, Nelson J C, Leach J E, Hulbert S H. Diversity in nucleotide binding site-leucine-rich repeat genes in cereals[J]. Genome Research, 2002, 12(12): 1871-1884. doi: 10.1101/gr.454902.
[27] Zhang S B, Chen C, Li L, Meng L, Singh J, Jiang N, Deng X W, He Z H, Lemaux P G. Evolutionary expansion, gene structure, and expression of the rice wall-associated kinase gene family[J]. Plant Physiology, 2005, 139(3): 1107-1124. doi:10.1104/pp.105.069005.
[28] Jain M, Tyagi A K, Khurana J P. Genome-ide analysis, evolutionary expansion, and expression of early auxin-responsive SAUR gene family in rice (Oryza sativa)[J]. Genomics, 2006, 88(3): 360-371. doi: 10.1016/j.ygeno.2006.04.008.
[29] 郭丽红, 徐娅, 郤秋霞, 李念, 檀文涛, 张学兰. 拟南芥热激因子AtHsfA1a在低温胁迫下对细胞程序性死亡中Caspase-3活性的影响[J]. 江苏农业科学, 2017, 45(21): 24-27. doi:10.15889/j.issn.1002-1302.2017.21.006.
Guo L H, Xu Y, Xi Q X, Li N, Tan W T, Zhang X L. Effect of Arabidopsis heat shock factor AtHsfA1a on Caspase-3 activity in programmed cell death under low temperature stress[J]. Jiangsu Agricultural Sciences, 2017, 45(21): 24-27.
[30] 熊洋洋, 顾雅琦, 刘志薇, 李彤, 吴致君, 庄静. 茶树中两个HSF转录因子基因分离与温度胁迫响应的比较分析[J]. 植物生理学报, 2015, 51(12): 2143-2151. doi:10.13592/j.cnki.ppj.2015.0532.
Xiong Y Y, Gu Y Q, Liu Z W, Li T, Wu Z J, Zhuang J. Isolation and expression profiles analysis of two HSF transcription factor genes under temperature stress in Camellia sinensis[J]. Plant Physiology Journal, 2015, 51(12): 2143-2151.
