全基因组重测序是对已完成基因组序列的同一物种或者近缘种的其他个体进行全基因组测序,并在此基础上对个体或群体进行差异性分析。新个体基因组序列通过映射参考基因组获得同源数据集,然后检测基因组范围内的单核苷酸多态性(Single nucleotide polymorphism,SNP)、插入缺失(Insertion/deletion,InDel)及结构变异(Structural variation,SV)等多态位点[1]。运用高通量的重测序技术进行遗传变异分析已经在谷子[2]、大豆[3]、水稻[4]、花生[5]、鼠尾草[6]、葡萄[7]、木兰[8]、烟草[9]、竹黄菌[10]和蘑菇[11]等植物上得到了广泛应用。
毛竹(Phyllostachys edulis)是我国特有的、利用历史悠久的重要经济竹种,也是栽培面积最广的竹种,其生长速度快,材性优良,繁殖能力强,一次造林可以永续利用。毛竹笋是一种全天然营养食品,含有丰富的蛋白质、脂肪、糖类、胡萝卜素、维生素、钙、磷、铁以及18种氨基酸等成分。因此,毛竹是优良的笋材两用竹种,有着巨大的开发和应用潜力[12]。毛竹广泛分布于我国南方16个省区,跨越北、中、南3种亚热带气候,在长期适应不同的生长环境和栽培经营措施下,加上自身发生遗传漂移等因素,发生了一系列的形态变异。目前,在天然毛竹林中已经发现21个毛竹变型[13],这些变型在形态上表现出丰富的多态性,尤其是秆形、秆色等形态性状差异显著。为研究这些形态差异究竟是因为基因变异而导致的可遗传变异还是由于生境不同而产生的表型可塑性,非常有必要在基因水平上揭示毛竹的遗传多样性和变异程度。
近年来,随着分子生物学技术的突飞猛进以及组学技术在竹类植物上的应用,特别是毛竹全基因组测序工作的完成[14],竹类植物基因组研究进入后基因组时代。目前,毛竹已得到一个完整的可变剪接图谱[15],对P450s[16]、AP2/ERF[17]、NAC[18]、SAUR[19]、AQP[20]、IQD[21]、SBP-like[22]、WRKY[23]、HD-Zip[24]和TCP[25]、Hsp[26]、CO-Like[27]等基因家族已采用生信方法进行了鉴定和功能验证,但尚未发现有关毛竹变型基因组序列变异的研究。本研究采用成熟且经济的二代测序技术,以毛竹全基因组为参考,对4个有代表性的毛竹变型进行重测序,对其SNP、Indel、结构变异SV等进行深度挖掘,从而揭示其全基因组的突变类型,分析其广适性状的变异基因,为从全基因组尺度分析毛竹的“种性”和变型种质“品性”获得海量基因组序列数据。
1 材料和方法
1.1 试验材料
试验材料为毛竹的4个变型(表1):圣音竹、黄槽毛竹、黄皮花毛竹和厚壁毛竹,编号分别为R01、R02、R03和R04,均采自国际竹藤中心安徽太平试验中心。选取刚长出且尚未展开的嫩叶,液氮速冻后超低温保存。
表1 4个毛竹变型形态特征
Tab.1 Morphological characteristics of 4 variations of Moso bamboo

编号ID变型Variation拉丁名Latin name形态特征Morphological characteristicsR01圣音竹P. edulis f.tubaeformis秆从上向基部逐渐膨大呈喇叭状,节间缩短 R02黄槽毛竹P. edulis f. luteosulcata秆绿色,但节间分枝一侧纵沟槽为黄色或淡黄色R03黄皮花毛竹P. edulis f.huamozhu秆黄色或黄绿色基本各半,有宽窄不等的绿色纵条纹;部分叶片还有少数淡黄色细纵条纹R04厚壁毛竹P. edulis f. pachyloen秆略呈四方形或椭圆形,壁厚,上部近实心
1.2 试验方法
1.2.1 DNA提取与基因组测序 基因组DNA采用改良CTAB法提取[28]。将检测合格的样品基因组DNA,用超声波方法将DNA进行片段化,然后进行DNA片段的纯化、末端修复、在3′端加A、连测序接头,进行琼脂糖凝胶电泳完成片段大小选择,通过PCR扩增建成测序文库,进行文库的质检,合格文库采用Illunima Hiseq 2500平台进行测序,得到原始测序序列(Raw reads),对其进行数据过滤,去掉带接头的reads,过滤N含量高(超过10%)和质量值低(低于10的碱基数超过50%)的reads,得到Clean reads,用作后续的信息分析。
1.2.2 与参考基因组进行比对统计 将重测序得到的测序reads重新定位到毛竹参考基因组上,进行后续的变异分析[14]。运用Bwa软件将测序获得的短序列与毛竹的参考基因组(毛竹基因组数据库 BambooGDB下载网址:http://www.bamboogdb.org/page/download.jsp)进行比对,通过Samtools flagstat命令对Clean reads比对定位到参考基因组上的位置,然后统计4个样品的测序深度和基因组覆盖度等方面的信息,进行序列变异的检测。
1.2.3 检测基因组变异 使用GATK软件工具包对样品的SNP和InDel进行检测[29]。根据参考基因组上Clean reads的定位结果,使用Picard软件去重复、GATK软件进行局部重比对和碱基质量值的校正等预处理,然后进行SNP和InDel的变异检测。对SNP进行严格的过滤,包括:SNP cluster过滤(5 bp内若有2个SNP过滤掉),InDel附近5 bp内的SNP过滤掉;相邻2个InDel距离小于10 bp过滤掉,从而获得SNP位点集。使用BreakDancer软件检测SV,主要是基于序列的比对结果,获得测序文库的插入片段大小、方差,再查找序列和参考基因组间比对的异常结果,寻找可能的结构变异。
1.2.4 注释SNP、InDel和SV 通过SnpEff软件进行SNP、InDel和SV的注释[30]。根据检测获得的变异位点比对到参考基因组的位置基因、CDS位置等,获得变异位点在基因组的发生区域(基因区、基因间区或CDS区等),以及产生的同义非同义突变等。
1.2.5 挖掘变异基因和功能注释 通过寻找样品间与参考基因组发生非同义突变SNP基因、CDS区发生的InDel基因与SV基因,挖掘可能存在的功能变异基因。通过Blast将变异基因与NR[31]、GO[32]、COG[33]、KEGG[34]等功能数据库进行比对,获得基因的注释,以便分析基因的功能。
2 结果与分析
2.1 与参考基因组比对统计
4个竹种通过高通量测序(Illunima Hiseq 2500等测序平台)得到原始图像数据文件,经碱基识别分析转化为原始测序序列(Raw reads),然后去除带接头的reads,过滤得到Clean reads,分别为180 506 436,184 142 480,213 567 146,188 998 942 bp,定位到毛竹参考基因组的占所有Clean reads数的百分比分别为96.13%,96.32%,96.33%和97.44%,双端均定位到毛竹参考基因组上并且距离符合测序片段的长度分布占所有Clean reads数的百分比分别为88.18%,88.42%,88.57%和89.65%。样品平均覆盖深度在10×以上,各深度对应的基因组覆盖比例如表2所示。
表2 4个毛竹变型的测序数据统计
Tab.2 Summary of sequencing data of 4 variants of Moso bamboo
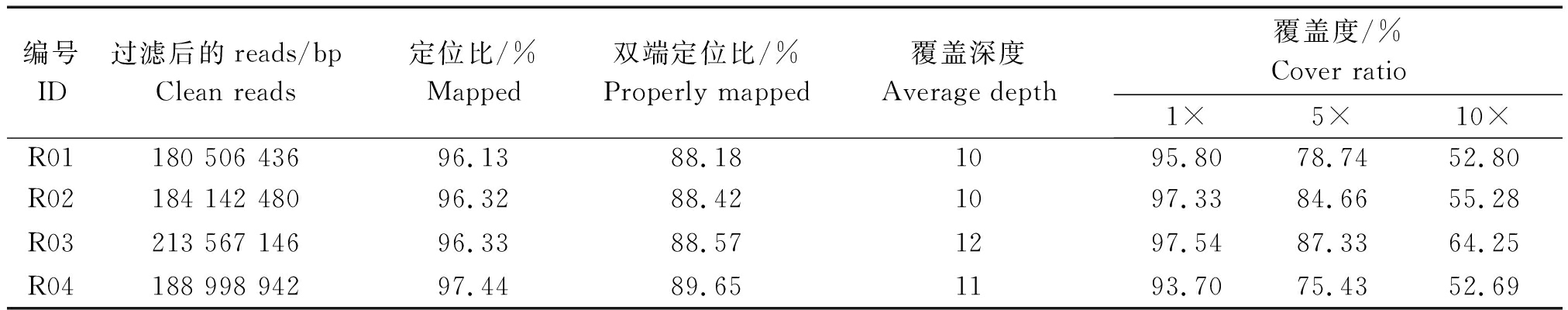
编号ID过滤后的reads/bpClean reads定位比/%Mapped双端定位比/%Properly mapped覆盖深度Average depth覆盖度/%Cover ratio1×5×10×R01180 506 43696.1388.181095.8078.7452.80R02184 142 48096.3288.421097.3384.6655.28R03213 567 14696.3388.571297.5487.3364.25R04188 998 94297.4489.651193.7075.4352.69
2.2 SNP的检测与注释
2.2.1 SNP检测 对4个毛竹变型的基因组DNA进行SNP检测,获得SNP位点集(表3),4个毛竹变型样品分别检测到的SNP数量为1 479 567~1 616 020,SNP总数为2 035 983,Ti/Tv值约为2.90。样品之间的SNP数统计结果见表4。
表3 4个毛竹变型SNP数据统计
Tab.3 Summary of SNPs detected in 4 variants of Moso bamboo
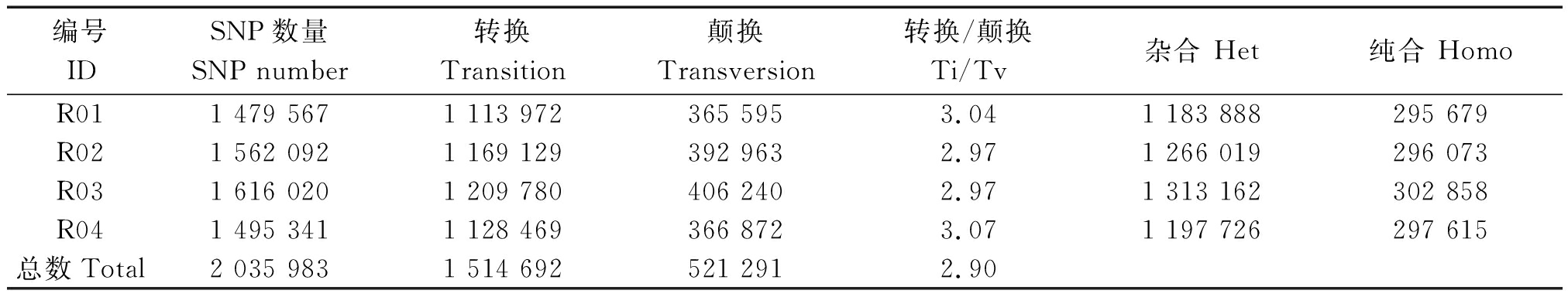
编号IDSNP数量SNP number转换Transition颠换Transversion转换/颠换Ti/Tv杂合 Het纯合 Homo R011 479 5671 113 972365 5953.041 183 888295 679 R021 562 0921 169 129392 9632.971 266 019296 073 R031 616 0201 209 780406 2402.971 313 162302 858 R041 495 3411 128 469366 8723.071 197 726297 615 总数Total2 035 9831 514 692521 2912.90
表4 毛竹变型间的SNP统计
Tab.4 Summary of SNPs detected between variants of Moso bamboo

编号IDR01R02R03R04R01 0480 449455 033460 206R02480 4490434 682469 846R03455 033434 6820438 693R04460 206469 846438 6930
2.2.2 SNP注释 运用SnpEff软件对4个毛竹变型全基因组SNP进行注释,得到其变异位点在基因组发生的区域或类型(表5)。圣音竹发生在CDS区域内的SNP共计24 426个,其中,同义突变占比为47.28%,非同义突变占比为51.56%。黄槽毛竹发生在CDS区域内的SNP共计25 319个,其中,同义突变占47.26%,非同义突变占51.50%。黄皮花毛竹发生在CDS区域内的SNP共计25 628个,其中,同义突变占47.26%,非同义突变占51.52%。厚壁毛竹发生在CDS区域内的SNP共计24 738个,其中,同义突变占47.49%,非同义突变占51.31%。
表5 4个毛竹变型SNP注释结果
Tab.5 SNP annotations in 4 variants of Moso bamboo
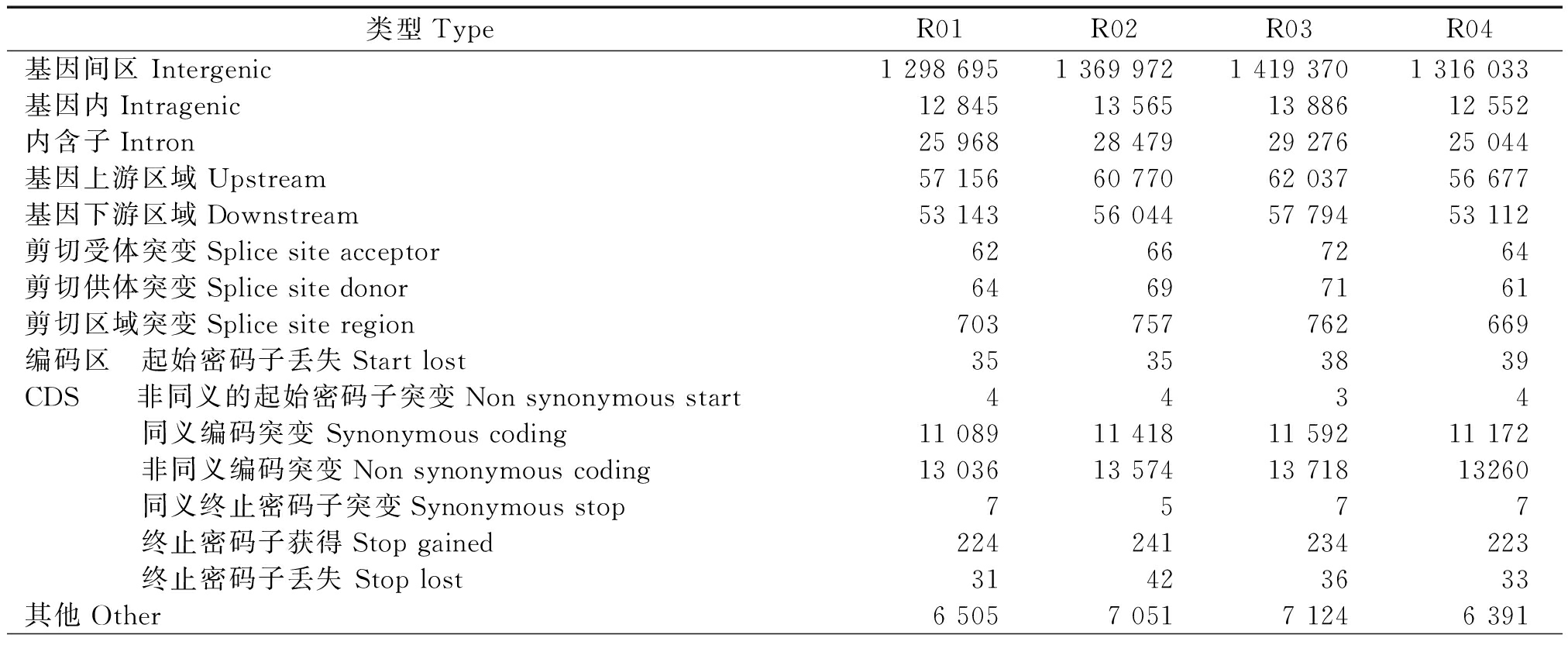
类型TypeR01 R02 R03 R04 基因间区 Intergenic1 298 6951 369 9721 419 3701 316 033基因内Intragenic12 84513 56513 88612 552内含子Intron25 96828 47929 27625 044基因上游区域Upstream57 15660 77062 03756 677基因下游区域Downstream53 14356 04457 79453 112剪切受体突变Splice site acceptor62667264剪切供体突变Splice site donor64697161剪切区域突变Splice site region703757762669编码区 起始密码子丢失Start lost35353839CDS 非同义的起始密码子突变Non synonymous start 4434 同义编码突变 Synonymous coding11 08911 41811 59211 172 非同义编码突变Non synonymous coding 13 03613 57413 71813260 同义终止密码子突变Synonymous stop7577 终止密码子获得Stop gained224241234223 终止密码子丢失 Stop lost31423633其他Other6 5057 0517 1246 391
2.3 InDel检测与注释
2.3.1 InDel检测 从4个样品InDel检测结果(表6)可以看出,圣音竹全基因组范围与编码区分别检测出174 657,3 173个InDel,CDS区有插入突变2 296个和缺失突变877个,其中纯合突变为578个。黄槽毛竹全基因组范围与CDS区分别检测出185 096,3 262个InDel,CDS区有插入突变2 323个和缺失突变939个,其中629个为纯合突变。黄皮花毛竹全基因组范围与CDS区分别检测出195 518,3 296个InDel,CDS区有插入突变2 371个和缺失突变925个,其中纯合突变为639个。厚壁毛竹全基因组范围与CDS区分别检测出176 393,3 121个InDel,CDS区有插入突变2 246个和缺失突变875个,其中纯合突变为611个。对样品在全基因组范围和CDS区的InDel长度进行统计(图1),基因组范围存在较多的+1、-1、+2、-2类型突变,在CDS区域存在较多的+1、-1、+3、-3类型突变。
表6 4个毛竹变型全基因组和编码区InDel统计
Tab.6 Summary of InDels detected in 4 variants of Moso bamboo
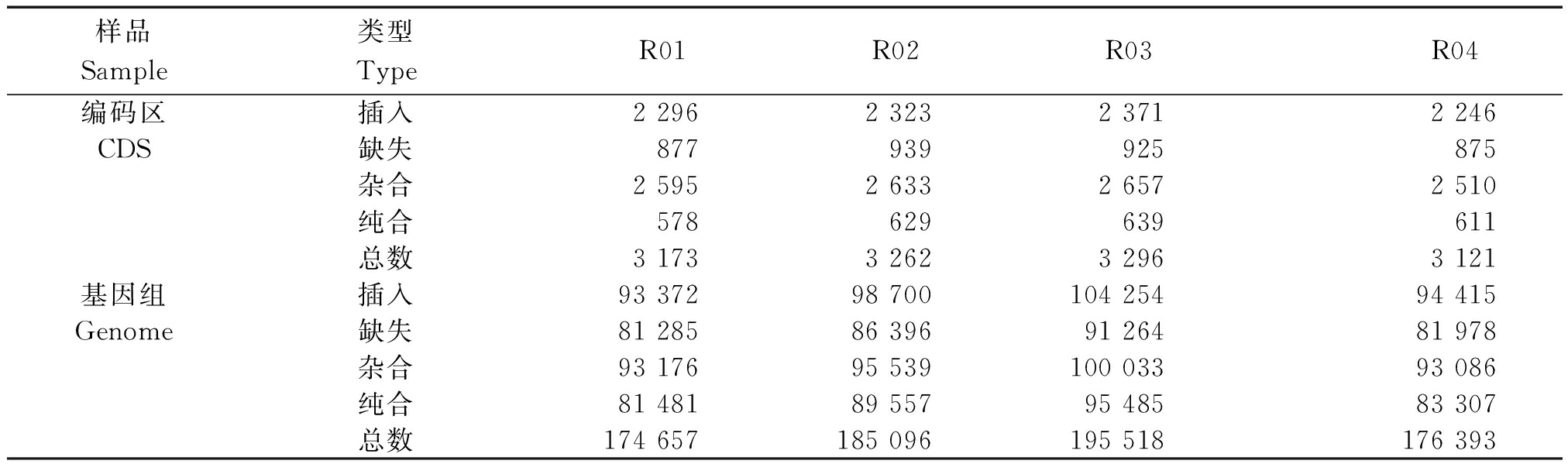
样品Sample类型Type R01 R02 R03 R04编码区插入2 2962 3232 3712 246CDS缺失877939925875杂合2 5952 6332 6572 510纯合578629639611总数3 1733 2623 2963 121基因组插入93 37298 700104 25494 415Genome缺失81 28586 39691 26481 978杂合93 17695 539100 03393 086纯合81 48189 55795 48583 307总数174 657185 096195 518176 393
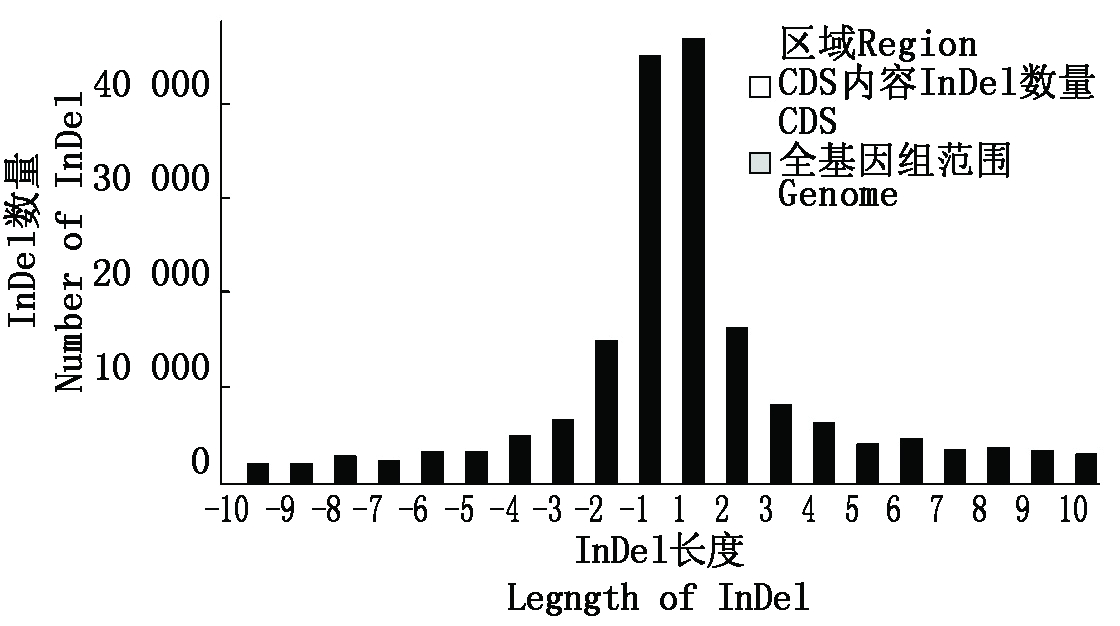
横坐标小于0为缺失,大于0为插入。
The abscissa less than 0 is missing,and greater than 0 is inserted.
图1 样品R01全基因组和编码区InDel长度分布
Fig.1 Length distribution of InDel in whole genome and CDS of sample R01
2.3.2 InDel注释 根据4个样品与参考基因组的InDel检测结果,进行两两比较,获得样品间的InDel变异位点(表7)。黄皮花毛竹与厚壁毛竹间的InDel数最少,为45 967;圣音竹与黄槽毛竹间InDel数最多,为48 551。
表7 毛竹变型间的InDel统计
Tab.7 Summary of InDels detected between variants of Moso bambooo
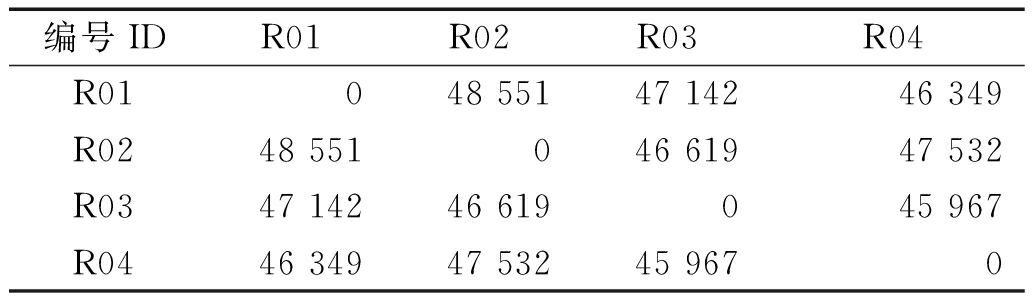
编号IDR01R02 R03 R04 R01 0 48 551 47 142 46 349 R02 48 551 0 46 619 47 532 R03 47 142 46 619 0 45 967 R04 46 349 47 532 45 967 0
2.4 SV检测与注释
2.4.1 SV检测 使用BreakDancer进行SV的检测(表8),圣音竹共检测到148 682个SV,其中插入3 701个,占总变异2.49%;缺失31 439个,占总变异21.15%;染色体间易位107 945个,占总变异72.60%;染色体内部易位4 991个,反转321个,其他285个。黄槽毛竹共检测到176 960个SV,其中插入17 377个,占总变异9.82%;缺失36 496个,占总变异20.62%;染色体间易位116 941个,占总变异66.08%。黄皮花毛竹共检测到181 687个SV,其中插入9 498个,占总变异5.23%;缺失37 880个,占总变异20.85%;染色体间易位127 646个,占总变异70.26%。厚壁毛竹共检测到148 968个SV,其中插入2 481个,占总变异1.67%;缺失30 971个,占总变异20.79%;染色体间易位110 054个,占总变异73.88%。
表8 4个毛竹变型结构变异数量统计
Tab.8 Summary of SVs detected in 4 variants of Moso bamboo
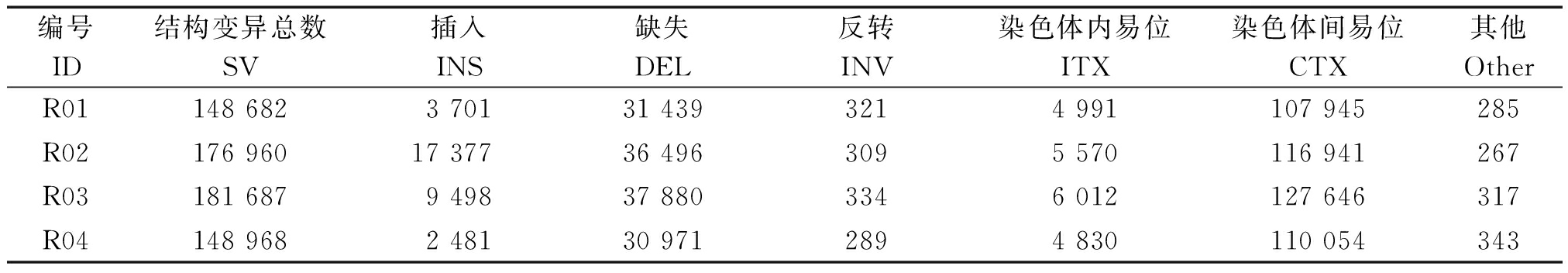
编号ID结构变异总数SV插入INS缺失DEL反转INV染色体内易位ITX染色体间易位CTX其他OtherR01148 6823 70131 4393214 991107 945285 R02176 96017 37736 4963095 570116 941267 R03181 6879 49837 8803346 012127 646317 R04148 9682 48130 9712894 830110 054343
2.4.2 SV注释 对结构变异深入分析表明(表9),圣音竹外显子区变异总数为1 894个,内含子区变异总数为789个,基因间区变异总数为32 778。黄槽毛竹外显子区变异总数为2 732个,内含子区变异总数为1 209个,基因间区变异总数为50 241个。黄皮花毛竹外显子区存在2 498个变异,内含子区存在1 064个变异,基因间区存在44 150个变异。厚壁毛竹外显子区分别存在1 731,内含子区存在773个,基因间区存在31 237个。
表9 4个毛竹变型结构变异注释结果
Tab.9 SV annotations in 4 variants of Moso bamboo
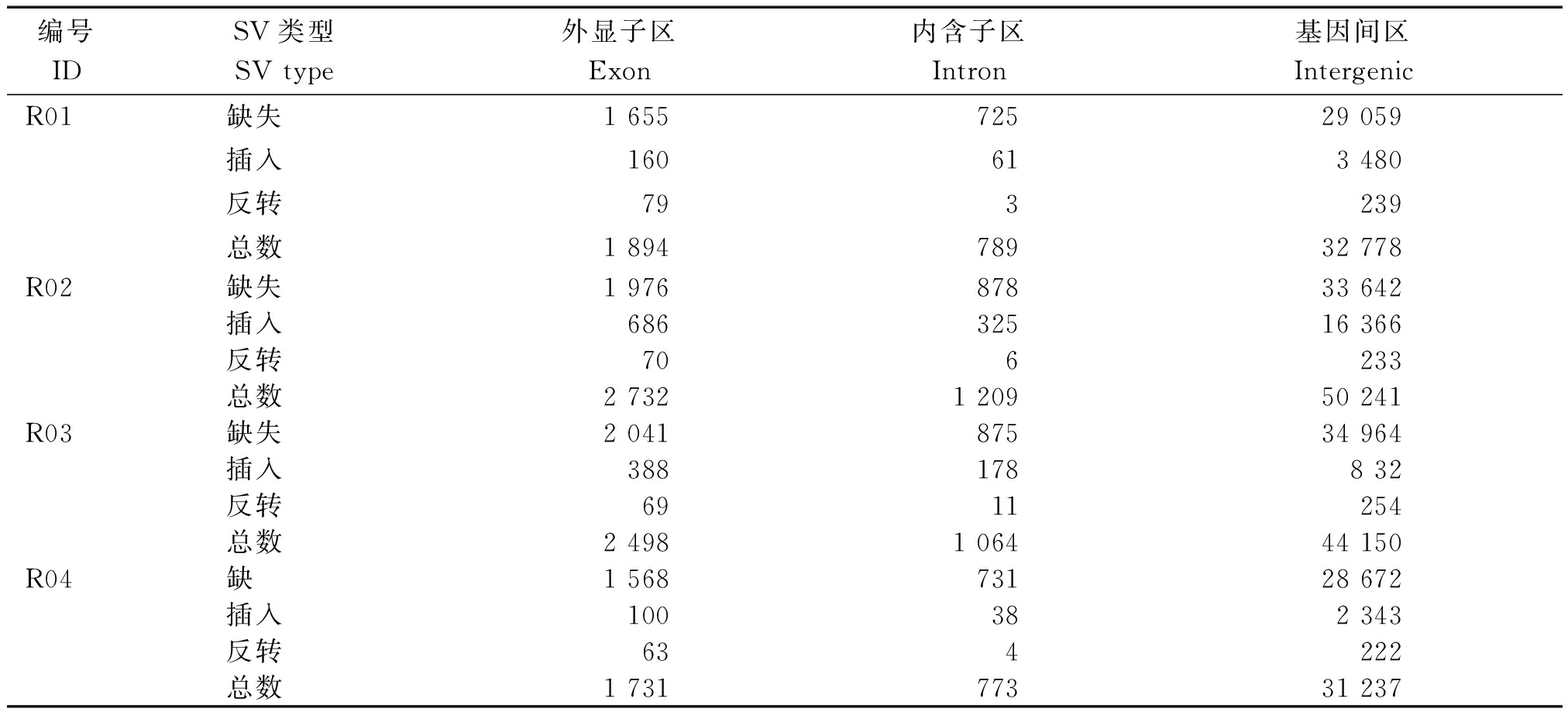
编号IDSV类型 SV type外显子区Exon内含子区Intron基因间区Intergenic R01缺失1 65572529 059插入160613 480反转793239总数1 89478932 778 R02缺失1 97687833 642插入68632516 366反转706233总数2 7321 20950 241 R03缺失2 04187534 964插入3881788 32反转6911254总数2 4981 06444 150 R04缺1 56873128 672插入100382 343反转634222总数1 73177331 237
2.5 DNA水平的变异基因挖掘与功能注释
2.5.1 变异基因挖掘 分别提取毛竹4个变型与参考基因组间发生非同义突变的SNP以及CDS区发生InDel与SV的基因,各样品的变异如表10所示。秆形变异的2个竹种圣音竹和厚壁毛竹基因组分别存在9 307,9 148个基因变异,其中SNP突变基因分别为4 973,4 981个,InDel基因分别为2 735,2 699个,SV突变的基因分别为1 599,1 468个,多个基因同时存在多种类型突变。秆色变异的2个竹种黄槽毛竹和黄皮花毛竹基因组分别存在10 194,9 977个基因变异,其中,SNP突变基因分别为5 094,5 095个,InDel基因分别为2 799,2 816个,SV突变基因2 301,2 066个,且多个基因同时存在多种类型突变。
表10 4个毛竹变型的变异基因统计
Tab.10 Summary of gene variations in 4 variants

编号ID非同义突变SNP基因Genes with non-synonymous SNP插入缺失基因Genes with InDel结构变异基因Genes with SV总数TotalR014 9732 7351 5999 307R025 0942 7992 30110 194R035 0952 8162 0669 977R044 9812 6991 4689 148
2.5.2 变异基因的功能注释 运用Blast软件,将挖掘的变异基因分别与GO、KEGG和COG等功能数据库进行比对,获得这些变异基因的注释,进而分析基因功能。4个毛竹变型注释到数据库的变异基因数分别为8 351,9 195,8 966,8 197个。
通过GO数据库,获得变异基因GO分类统计结果(图2)。从图中可以看出,细胞组件、分子功能和生物过程3个基因功能分类体系中,GO各分类内容对应的基因数以及基因数目所占百分比。对GO数据进一步分析发现,在细胞组分方面,叶绿素合成相关基因有1 848个,细胞壁相关基因750个,细胞骨架相关基因194个;在生物过程方面,参与类胡萝卜素合成过程的基因有60个,参与花青素合成过程中的调控以及紫外光下组织中花青素积累的相关基因有55个;在功能基因方面,参与信号转导的基因283个,转录因子475个,激素相关基因75个。
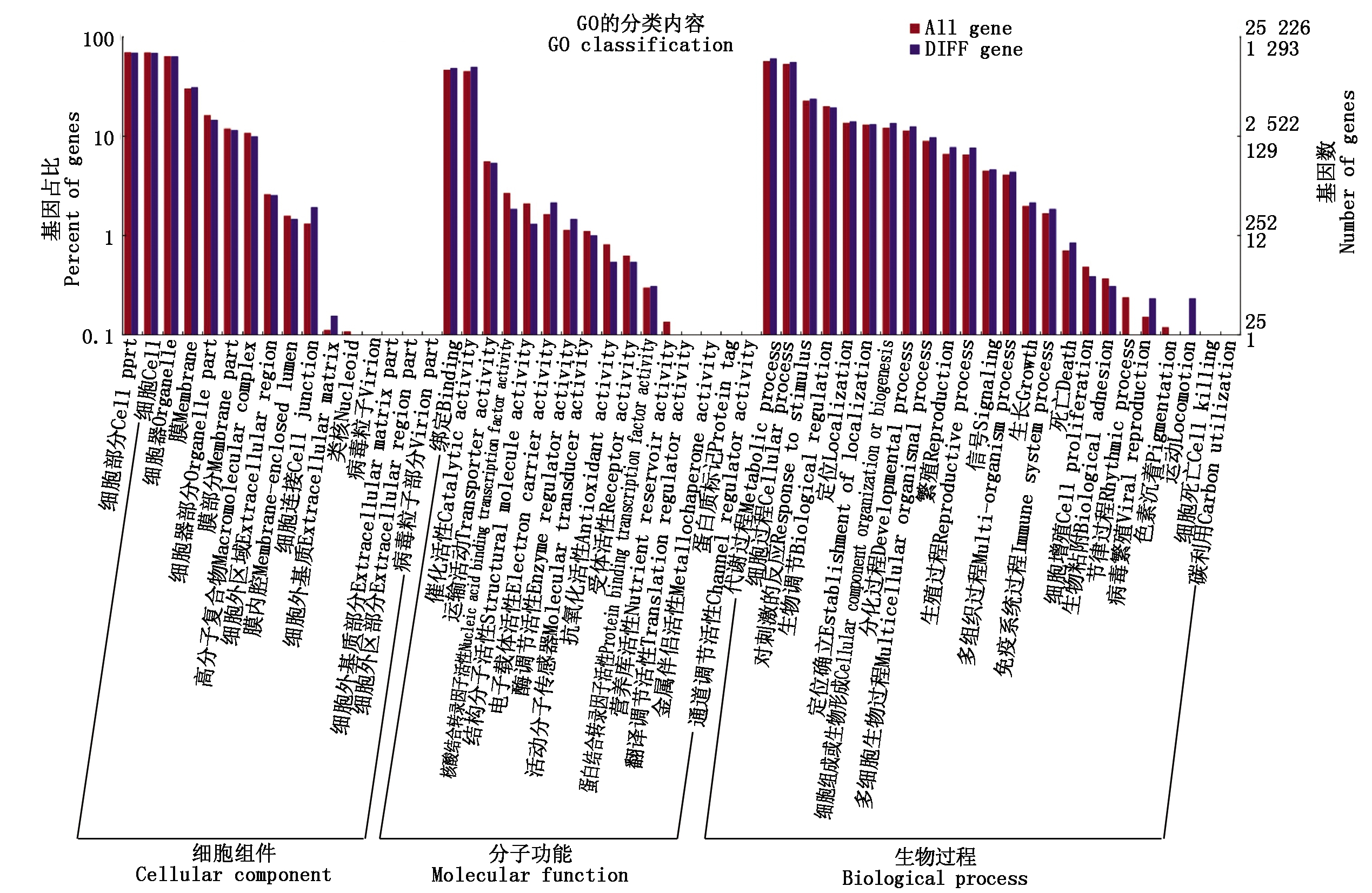
图2 变异基因的GO注释分类图
Fig.2 Classification of gene variations compared with GO database by Blast
COG数据库是对基因产物进行直系同源分类的数据库。从变异基因COG分类统计结果(图3)中可以看出,在不同的功能分类中,基因数目相差很大,其中参与功能预测、转录、复制重组修复和信号转导机制的基因数目较多。基因所占比例多少,反映对应时期和环境下代谢或者生理偏向等内容。COG注释获得的参与功能预测的基因1 131个,参与糖代谢途径的基因355个,细胞壁、细胞膜合成相关的基因有276个,信号转导机制的基因数为228个,细胞周期调控、细胞分裂和染色的相关基因145个。
通过KEGG数据库,变异基因注释到114个KEGG代谢通路中,分析共获得了1 310个转录因子及813个信号转导相关单基因;在功能基因方面,共获得618个与细胞壁构建相关的代谢基因。KEGG数据库能够系统地分析基因产物在细胞中的代谢途径和功能,有助于把基因及表达信息作为一个整体的网络进行研究。从变异基因的糖代谢通路(图4)可以看出,整个通路有46种不同的酶通过复杂的生化反应形成,框内的数字分别代表酶的号码。其中的4种酶:1.2.4.1(丙酮酸脱氢酶)、1.8.1.4(二氢硫辛酸脱氢酶)、5.3.1.1(磷酸丙糖异构酶)和5.4.2.2(葡萄糖磷酸变位酶)对应的框代表变异基因中与此通路相关。

图3 变异基因的COG注释分类图
Fig.3 Classification of gene variations compared with COG database
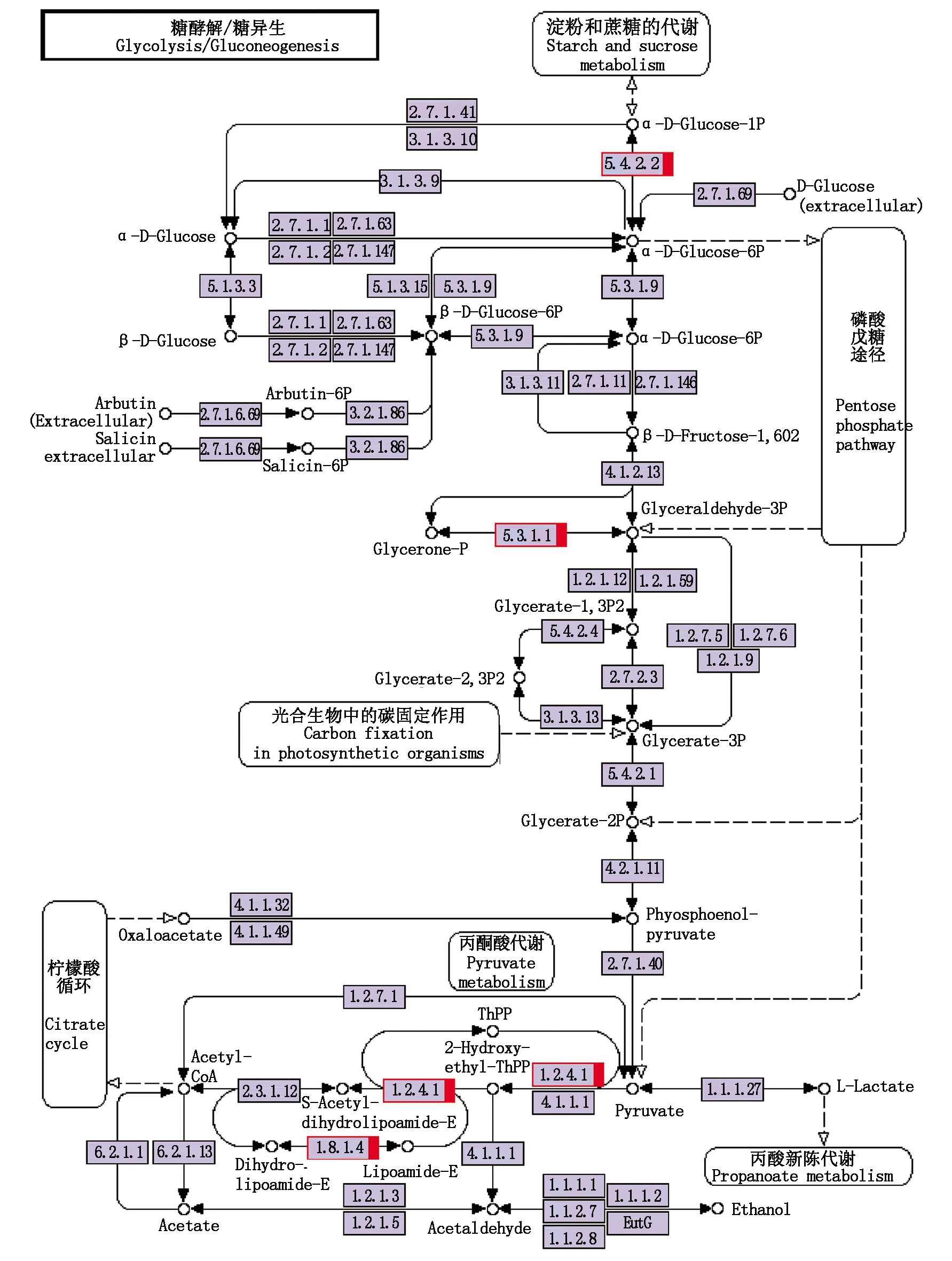
图4 变异基因的KEGG通路代谢图
Fig.4 Pathway of gene variations compared with KEGG database by Blast
将变异基因与GO、COG、KEGG等3个功能数据库进行比对、注释,共获得1 739个参与细胞壁和细胞膜合成的基因、501个细胞骨架构建相关基因、1 785个转录因子、1 324个信号转导、104个赤霉素及76个激素代谢相关基因、1 938个叶绿素合成相关的基因、73个类胡萝卜素合成代谢相关基因和68个花青素合成调控基因。
3 结论与讨论
3.1 4个毛竹变型的秆形、秆色变异表征
经过长期的自然演化和人工栽培后,毛竹种内产生丰富的遗传变异,从而形成不同的结构形态。圣音竹、黄皮花毛竹、黄槽毛竹和厚竹是毛竹种的4个变型,具有明显的秆形或秆色特征。圣音竹竹秆从上面向基部逐渐膨大呈喇叭状,同时节间缩短,且上粗下细或下部紧靠节处有不规则凹陷,节部呈波浪状歪斜。厚竹秆略呈四方形或椭圆形,壁厚,上部近实心,分枝以下节隆起较高。黄皮花毛竹秆黄色或黄绿色基本各半,有宽窄不等的绿色纵条纹。黄槽毛竹秆主要为绿色,但节间分枝一侧纵沟槽为黄色或淡黄色。这些在竹秆形态和颜色等方面的不同变异使其具有很高的观赏价值。
3.2 4个毛竹变型的基因组变异分析
通过对4个毛竹变型进行重测序,初步统计分析了这些形态变异发生的分子数据。样品检测到的SNP总数为2 035 983,Ti/Tv值约为2.90,说明同种类型的碱基之间突变比不同类型间更容易发生。分别提取毛竹4个变型与参考基因组间发生非同义突变的SNP、CDS区发生InDel与SV的基因,可以发现其基因组基因的变异数目和变异类型呈现一定的规律,其中,2个秆形变异竹种圣音竹和厚壁毛竹基因组基因的变异数目和变异类型相近,而2个秆色变异竹种黄槽毛竹和黄皮花毛竹基因组的变异数目和变异类型相近,但秆形和秆色变异的竹种两两之间差异较大。
3.3 变异基因的功能注释与变异性状的关联分析
DNA水平的变异基因挖掘和功能注释,可以分析基因产物在细胞中的代谢途径以及这些基因产物的功能。将变异基因通过多个数据库注释,共获得1 739个基因参与细胞壁、细胞膜合成,501个细胞骨架构建相关基因,以及1 785个转录因子、1 324个信号转导、104个赤霉素和76个激素代谢相关基因。参考矢竹的研究结果,其地下茎生长发育受到从各类激素、转录因子到其下游功能基因等组成的复杂分子网络的协同调控,细胞壁、细胞骨架下游功能相关基因在矢竹地下茎节间快速生长阶段表达最高,而赤霉素合成及信号传导相关基因则在节间伸长起始阶段最高[35]。因此,进一步对毛竹变型细胞组分、生物过程和分子功能相关变异基因的研究,有利于推测毛竹秆形变异的分子调控机制。
竹类植物中的色素主要有叶绿素、类胡萝卜素和花青素3大类,色素的变化会影响植株的呈色变异。通过数据库注释,发现的叶绿素合成相关基因、类胡萝卜素合成代谢相关基因和花青素合成调控基因,深入研究这些差异基因的调控途径利于从DNA水平上解释秆色的变异。研究表明[36],毛竹不同变异类型在净光合速率、叶绿素含量、β胡萝卜素含量、硝酸还原酶活性、游离氨基酸含量这5个生理指标中存在着显著性差异,其中,黄皮花毛竹最高,其次为厚壁毛竹,然后是圣音毛竹、黄槽毛竹。对毛竹的栽培变种进行ISSR和AFLP分子标记分析,分子遗传的分析结果与阮晓赛[37]进行的形态学研究结果相似,变型之间的遗传变异程度较小。进一步分析这4个毛竹变型基因产物的功能及其代谢途径,也有利于从基因水平上对其生物学和生理特性加以解释和分析。数据的挖掘与深入分析工作正在进行,后续分析将解析毛竹众多种质的分子遗传基础,阐析不同变异类型的分子品性、基因家族、功能基因等遗传根基。
[1] 王嘉,曾召琼,梁建秋,于晓波,吴海英,张明荣. 基于全基因组重测序的大豆分子标记开发及籽粒蛋白质含量QTL定位[J]. 中国农业科学,2019,52(16):2743-2757. doi:10.3864/j.issn.0578-1752.2019.16. 001.
Wang J,Zeng Z Q,Liang J Q,Yu X B,Wu H Y,Zhang M R. Development new molecular markers for quantitative trait locus (QTL) analysis of the seed protein content based on whole genome re-sequencing in Soybean[J]. Scientia Agricultura Sinica,2019,52(16):2743-2757.
[2] 贾小平, 张博, 全建章, 李剑峰, 王永芳, 袁玺垒. 不同光周期条件下谷子株高的全基因组关联分析[J]. 华北农学报,2019,34(4) :16-23. doi: 10.7668/hbnxb.201750856.
Jia X P,Zhang B,Quan J Z,Li J F,Wang Y F,Yuan X L. Genome-wide association analysis of plant height in foxtail millet under different photoperiod conditions[J]. Acta Agriculturae Boreali-Sinica,2019,34(4):16-23.
[3] 张羽,François Belzile. 大豆抗菌核病的全基因组关联研究[J]. 华北农学报,2020,35(1):205-213.doi:10.7668/hbnxb.20190364.
Zhang Y,François B. Genome-wide association study for Sclerotinia sclerotiorum resistance of soybean[J]. Acta Agriculturae Boreali-Sinica,2020,35(1):205-213.
[4] Wu Y B,Li G,Zhu Y J,Cheng Y C,Yang J Y,Chen H Z,Song X J,Ying J Z. Genome-wide identification of QTLs for grain protein content based on genotyping-by-resequencing and verification of qGPC1-1 in rice[J]. International Journal of Molecular Sciences,2020,21(2):408. doi:10.3390/ijms21020408.
[5] Zhao Y H,Ma J J,Li M,Deng L,Li G H,Xia H,Zhao S Z,Hou L,Li P C,Ma C,Yuan M,Ren L,Gu J Z,Guo B Z,Zhao C Z,Wang X J. Whole-genome resequencing-based QTL-seq identified AhTc1 gene encoding a R2R3-MYB transcription factor controlling peanut purple testa colour[J]. Plant Biotechnology Journal, 2020,18(1):96-105. doi:10.1111/pbi.13175.
[6] Liu T P,He J H,Dong K J,Wang X W,Wang W W,Yang P,Ren R Y,Zhang L,Zhang Z S,Yang T Y. QTL mapping of yield component traits on bin map generated from resequencing a RIL population of foxtail millet (Setaria italica)[J]. BMC Genomics,2020,21:141. doi:10.1186/s12864-020-6553-9.
[7] Jiang J F,Fan X C,Zhang Y,Tang X P,Li X M,Liu C H,Zhang Z W. Construction of a high-density genetic map and mapping of firmness in grapes (Vitis vinifera L.) based on whole-Genome resequencing[J]. International Journal of Molecular Sciences,2020,21(3):797. doi:10.3390/ijms21030797.
[8] Wang J Q,Li P,Mao D D,Zhang X M,Wang H,Li Y Y. The complete chloroplast genome sequence of Manglietia yuyuanensis (Magnoliaceae)[J]. Mitochondrial DNA Part B,2020,5(1):417-419. doi:10.1080/23802359.2019.1703568.
[9] Yang H J,Geng X Q,Zhao S M,Shi H Z. Genomic diversity analysis and identification of novel SSR markers in four tobacco varieties by high-throughput resequencing[J]. Plant Physiology and Biochemistry,2020,150:80-89. doi:10.1016/j.plaphy.2020.02.023.
[10] Ren X Y,Liu Y X,Tan Y M,Huan Y H,Liu Z Y,Jiang X L. Sequencing and functional annotation of the whole genome of Shiraia bambusicola[J]. Genome Report of Shiraia bambusicola,2020,10(1):23-35. doi:10.1534/g3.119.400694.
[11] Yu F,Song J,Liang J F,Wang S K,Lu J K. Whole genome sequencing and genome annotation of the wild edible mushroom,Russula griseocarnosa[J]. Genomics, 2020,112(1):603-614. doi:10.1016/j.ygeno.2019.04.012.
[12] 江泽慧. 世界竹藤[M]. 沈阳:辽宁科学技术出版社, 2002:3-10.
Jiang Z H. Bamboo and rattan in the world[M]. Shenyang:Liaoning Science and Technology Press, 2002: 3-10.
[13] 马乃训,赖广辉,张培新,张宏亮. 中国刚竹属[M]. 杭州:浙江科学技术出版社,2014:91-107.
Ma N X,Lai G H,Zhang P X,Zhang H L. The genus Phyllostachys in China[M]. Hangzhou: Zhejiang Science and Technology Press,2014:91-107.
[14] Peng Z H,Lu Y,Li L B,Zhao Q,Feng Q,Gao Z M,Lu H Y,Hu T,Yao N,Liu K Y,Li Y,Fan D L,Guo Y L,Li W J,Lu Y Q,Weng Q J,Zhou C C,Zhang L,Huang T,Zhao Y,Zhu C R,Liu X G,Yang X W,Wang T,Miao K,Zhuang C Y,Cao X L,Tang W L,Liu G S,Liu Y L,Chen J,Liu Z J,Yuan L C,Liu Z H,Huang X H,Lu T T,Fei B H,Ning Z M,Han B,Jiang Z H. The draft genome of the fast-growing non-timber forest species moso bamboo (Phyllostachys heterocycla)[J]. Nature Genetics,2013,45(4):456-461. doi:10.1038/ng.2569.
[15] Zhao H S, Gao Z M, Wang L, Wang J L, Wang S H, Fei B H, Chen C H, Shi C C, Liu X C, Zhang H L, Lou Y F, Chen L F, Sun H Y, Zhou X Q, Wang S N, Zhang C, Xu H, Li L C, Yang Y H, Wei Y L, Yang W, Gao Q, Yang H M, Zhao S C, Jiang Z H. Chromosome-level reference genome and alternative splicing atlas of moso bamboo (Phylostachys edulis)[J]. Giga Science,2018,7(10):1-12. doi:10.1093/gigascience/giy115.
[16] 林翩翩,白有煌,周明兵. 毛竹细胞色素P450的基因组学分析[J]. 植物生理学报,2014,50(9):1387-1400. doi:10.13592/j.cnki.ppj.2014.0205.
Lin P P,Bai Y H,Zhou M B. Genomics analysis of cytochrome P450 monooxygenase genes in Phyllostachys heterocycle [J]. Plant Physiology Journal, 2014,50(9):1387-1400.
[17] Wu H L,Lü H,Li L,Liu J,Mu S H,Li X P,Gao J. Genome-wide analysis of the AP2/ERF transcription factors family and the expression patterns of DREB genes in moso bamboo (Phyllostachys edulis)[J]. PLoS One,2015,10(5):e0126657. doi:10.1371/journal.pone.0126657.
[18] 黎帮勇, 胡尚连, 曹颖, 徐刚. 毛竹NAC转录因子家族生物信息学分析[J]. 基因组学与应用生物学,2015,34(8):1769-1777. doi:10.13417/j.gab.034.001769.
Li B Y,Hu S L,Cao Y,Xu G. Bioinformatics analysis of NAC gene family in moso bamboo[J]. Genomics and Applied Biology,2015,34(8):1769-1777.
[19] Bai Q S,Hou D,Li L,Cheng Z C,Ge W,Liu J,Li X P,Mu S H,Gao J. Genome-wide analysis and expression characteristics of small auxin-up RNA (SAUR) genes in moso bamboo (Phyllostachys edulis)[J]. Genome,2017,60(4):325-336. doi:10.1139/gen-2016-0097.
[20] Sun H Y,Li L C,Lou Y F,Zhao H S,Gao Z M. Genome-wide identification and characterization of aquaporin gene family in moso bamboo (Phyllostachys edulis)[J]. Molecular Biology Reports,2016,43(5):437-450. doi:10.1007/s11033-016-3973-3.
[21] Wu M,Li Y,Chen D M,Liu H L,Zhu D Y,Xiang Y. Genome-wide identification and expression analysis of the IQD gene family in moso bamboo (Phyllostachys edulis)[J]. Scientific Reports,2016,6:24520. doi:10.1038/srep24520.
[22] Pan F,Wang Y,Liu H L,Wu M,Chu W Y, Chen D M,Xiang Y. Genome-wide identification and expression analysis of SBP-like transcription factor genes in moso bamboo (Phyllostachys edulis)[J]. BMC Genomics,2017,18(1):486. doi:10.1186/s12864-017-3882-4.
[23] Li H,Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform[J]. Bioinformatics,2009,25(14):1754-1760. doi: 10.1093/bioinformatics/btp324.
[24] Chen D M, Chen Z,Wu M,Wang Y,Wang Y J,Yan H W,Xiang Y. Genome-wide identification and expression analysis of the HD-Zip gene family in moso bamboo (Phyllostachys edulis)[J]. Journal of Plant Growth Regulation,2017,2(36):323-337. doi:10.1007/s00344-016-9642-x.
[25] 刘俊, 黄容, 程占超,李雪平. 毛竹TCP基因家族全基因组鉴定与分析[J]. 基因组学与应用生物学,2018,37(12):5388-5397. doi:10.13417/j.gab.037.005388.
Liu J,Huang R,Cheng Z C,Li X P. Genome-wide identification and the whole analysis of TCP gene family in moso bamboo (Phyllostachys edulis)[J]. Genomics and Applied Biology,2018,37(12):5388-5397.
[26] Xie L H,Li X Y,Hou D,Cheng Z C,Liu J,Li J,Mu S H,Gao J. Genome-wide analysis and expression profiling of the heat shock factor gene family in Phyllostachys edulis during development and in response to abiotic stresses[J]. Forests, 2019,10(2):100. doi:10.3390/f10020100.
[27] Liu J,Cheng Z C,Li X Y,Xie L H,Bai Y C,Peng L X,Li J,Gao J.Expression analysis and regulation network identification of the CONSTANS-Like gene family in moso bamboo (Phyllostachys edulis) under photoperiod treatments[J]. DNA and Cell Biology,2019,38(7):607-626.doi:10.1089/dna.2018.4611.
[28] Zidani S,Ferchichi A,Chaieb M. Genomic DNA extraction method from pearl millet (Pennisetum glaucum) leaves[J]. African Journal of Biotechnology,2005,4(8):862-866.doi:10.5897/AJB2005.000-3218
[29] McKenna A,Hanna M,Banks E,Sivachenko A,Cibulskis K,Kernytsky A,Garimella K,Altshuler D,Gabriel S,Daly M,DePristo M A. The genome analysis toolkit:A mapreduce framework for analyzing next-generation DNA sequencing data[J]. Genome Research,2010,20(9):1297-1303.doi:10.1101/gr.107524.110.
[30] Cingolani P,Platts A,Wang L L,Coon M,Nguyen T,Wang L,Land S J,Lu X Y,Ruden D M. A program for annotating and predicting the effects of single nucleotide polymorphisms,SnpEff:SNPs in the genome of Drosophila melanogaster strain w1118;iso-2;iso-3[J]. Fly,2012,6(2) :80-92. doi:10.4161/fly.19695.
[31] Altschul S F,Madden T L,Schäffer A A, Zhang J H,Zhang Z,Miller W,Lipman D J. Gapped BLAST and PSI-BLAST:A new generation of protein database search programs[J]. Nucleic Acids Research,1997,25(17) :3389-3402.doi:10.1093/nar/25.17.3389.
[32] Deng Y Y, Li J Q, Wu S F, Zhu Y P, Chen Y W, He F C, Deng Y, Deng Y, Li J, Wu S, Zhu Y, Chen Y, He F, Li J, Wu S, Zhu Y, He F, Deng Y Y, Li J Q, Wu S F, Zhu Y P, Chen Y W, He F C, Chen Y W, Wu S, Deng Li, Li J, Wu S, Zhu Y, Chen Y, He F, Wu S F, Deng M, Li J T, Zhu Y, He F Y. Integrated nr database in protein annotation system and its localization[J]. Comput Eng,2006,32(5) :71-74. doi:10.1109/INFOCOM.2006.241.
[33] Tatusov R L,Galperin M Y,Natale D A,Koonin E V. The COG database:A tool for genome-scale analysis of protein functions and evolution[J]. Nucleic Acids Research,2000,28(1) :33-36. doi:10.1093/nar/28.1.33.
[34] Kanehisa M, Goto S, Kawashima S, Okuno Y, Hattori M. The KEGG resource for deciphering the genome[J]. Nucleic Acids Research,2004,32(S1):277-280. doi:10.1093/nar/gkh063.
[35] 魏强, 丁雨龙. 矢竹地下茎转录组测序及节间生长相关基因表达分析[J]. 南京林业大学学报(自然科学版),2017,41(5):42-48. doi:10.3969/j.issn.1000-2006.201601006.
Wei Q,Ding Y L. Transcriptome sequencing,de novo assembly and expression analysis of several genes related to internode development in Pseudosasa japonica[J]. Journal of Nanjing Forestry University (Natural Science Edition),2017,41(5):42-48.
[36] 陈建华,晏存育,王艳梅,张新明,曾翔,曹学优,贺峭. 6种不同变异类型毛竹叶片的生理特性研究[J].中南林业科技大学学报, 2011,31(4):70-73. doi:10.14067/j.cnki.1673-923x.2011.04.037.
Chen J H,Yan C Y,Wang Y M,Zhang X M,Zeng X,Cao X Y,He Q. Physiological characteristics of 6 different variation type bamboo′s leaves[J]. Journal of Central South University of Forestry & Technology,2011,31(4):70-73.
[37] 阮晓赛. 毛竹种源及栽培变种遗传变异的AFLP和ISSR分析[D].杭州: 浙江林学院,2008:30-31.
Ruan X S. Genetic variations of Phyllostachys edulis provenance and cultivars by ISSR and AFLP[D]. Hangzhou:Zhejiang Forestry College,2008:30-31.
