木质素是一种酚类聚合物,是植物体内含量仅次于纤维素的有机高分子物质,占地球生物圈有机碳的30%左右[1]。在形成细胞壁的过程中,木质素起很重要的作用,它主要存在于木质部中,通过形成交织网使细胞壁更加坚固[2],从而保护植物免受真菌侵害,给植物提供机械支撑,维持良好的硬度,承受整株植物的重量[3-4]。在陆生植物的进化过程中,木质素与植物的直立生长习性息息相关,其合成过程也是逐渐适应陆地生存的特征之一[5]。木质素不容易被食草动物消化,含量的高低可影响饲草的吸收效率和营养价值[6]。研究表明,木质素主要是由3种单体组成,分别是芥子醇、香豆醇和松柏醇,三者都是由苯丙烷衍生物通过不同的羟基化和甲基化形成的,再经化学键连接[7]。在不同的植物种类中,3种木质素单体所占比例皆不相同,G-S型木质素主要由松柏醇与芥子醇聚合而成,存在于双子叶植物;G型木质素主要由松柏醇聚合而成,存在于裸子植物与蕨类植物;H-G-S型木质素不同程度地包含着这3种木质醇单体,主要是单子叶植物[8]。研究这3种木质素单体形成过程中的关键酶基因的作用是当前研究的热点,很多研究者分析木质素与植物的抗性关系都是从木质素合成关键酶出发,但是由于过程中涉及的酶类众多,对于木质素的研究还有很长的路要走[9]。
象草(Pennisetum purpureum Schum)属禾本科,是一种原生于非洲草原的多年生草本植物[10]。秆直立,高可达3 m以上,是生物量最高的草本植物之一,最高可达90 t/hm2,同时被认为是最优秀的能源物质之一[10-11]。早在20世纪30年代,象草便从缅甸引入中国[12]。象草适应力强,适口性良好,已成为中国华南地区重要的牧草之一。除此之外,因其抗逆性强、热值高、生物量大、产量高等特点,所以也常常被用作造纸原料和新型生物能源作物[13-16]。近年来对象草木质素合成调控的研究相继展开。2014年,彭小群等[17]通过构建了PpCCR正义和反义载体,并利用农杆菌转化法转化到烟草中,发现PpCCR过表达烟草转基因烟草木质化细胞增多,而反义表达的烟草转基因烟草木质化细胞排列松散,笔者得出PpCCR在象草木质素合成中发挥很重要的作用,也为今后研究象草遗传改良奠定了基础。2015年,唐然等[18]对华南象草CAD基因进行克隆,构建表达载体在烟草中异源表达,证明了PpCAD基因参与植物木质素的合成过程,对继续研究象草木质素合成调控打下基础。钟天秀等[11]同样对华南象草中的Pp4CL基因进行克隆,并在烟草中异源表达,发现转基因烟草的木质素在叶柄和茎秆中有不同程度的提高,表明过表达Pp4CL基因能够显著提高植株木质素的含量。这些研究结果都为华南象草木质素改良工作打下了基础。
本研究以N51象草为试验材料,测定茎秆(以下简称为:倒一节间、倒三节间、倒五节间)和叶片(以下简称为:倒一叶、倒三叶、倒五叶、倒七叶、倒九叶)中的木质素含量以及木质素合成相关基因(PAL、C3H、4CL、C4H、CAD、HCT4、COMT、F5H、CCoAOMT和CCR)的表达量,进行象草木质素含量与木质素合成相关酶基因的相关性分析,并了解木质素合成基因在叶组织与茎组织中的功能和机制上可能存在的差异,为其他木质素相关研究奠定基础,也为今后深入地开展象草分子育种提供依据。
1 材料和方法
1.1 试验材料
供试材料取自华南农业大学草业科学系引种园中的N51象草,取样时象草已抽穗,选取长势相同的3株象草作为3次重复,各取茎秆倒数第一、第三、第五节间以及倒数第一、第三、第五、第七和第九叶片置于液氮中,带回实验室一同研磨后于-80 ℃超低温冰箱中保存。
1.2 试验试剂
象草RNA提取采用天根植物总RNA提取试剂盒,cDNA的合成采用全式金反转录试剂盒,Realtime-PCR采用MonAmpTM ChemoHS qPCR Mix(None ROX)试剂盒。
1.3 试验方法
1.3.1 木质素含量的测定 将新鲜的象草样品在60 ℃下烘干至恒质量。采用乙酰溴法测定木质素总含量,于分光光度计中测定OD280的吸光值,求得木质素的总含量,具体步骤参照王建庆等[19]的试验方法。
1.3.2 象草木质素合成关键酶编码基因的获取 由于目前仍没有完整可用的象草基因组和转录组序列,从NCBISRA(NationalCenter for Biotechnology Information Sequence Read Archive)数据库中下载了象草转录组的IlluminaHiSeq 2500测序数据(SRR3952080和SRR3952081),采用Trinity-v2.5.1(https://github.com/trinityrnaseq/trinityrnaseq/wiki)进行转录组拼接。从GenBank中获得了水稻木质素合成关键酶的基因序列(表1),通过tBlastN与拼接获得的象草转录组本地Blast库进行比对,获得相应的象草转录本序列。
表1 水稻木质素合成关键酶的基因序列
Tab.1 The gene sequence of key enzymes associated with rice lignin synthesis
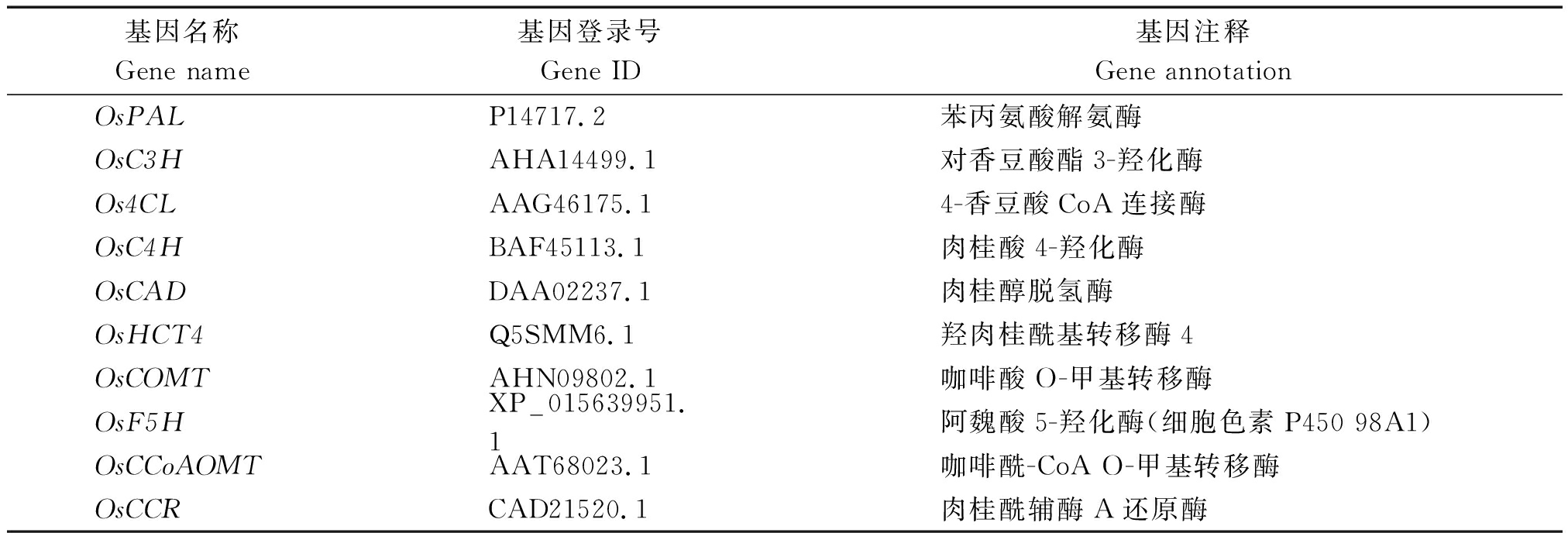
基因名称Gene name基因登录号Gene ID基因注释Gene annotationOsPALP14717.2苯丙氨酸解氨酶OsC3HAHA14499.1对香豆酸酯3-羟化酶Os4CLAAG46175.14-香豆酸CoA连接酶OsC4HBAF45113.1肉桂酸4-羟化酶OsCADDAA02237.1肉桂醇脱氢酶OsHCT4Q5SMM6.1羟肉桂酰基转移酶4 OsCOMTAHN09802.1咖啡酸O-甲基转移酶OsF5HXP_015639951.1阿魏酸5-羟化酶(细胞色素P450 98A1)OsCCoAOMTAAT68023.1咖啡酰-CoA O-甲基转移酶OsCCRCAD21520.1肉桂酰辅酶A还原酶
1.3.3 木质素合成关键酶编码基因表达量分析 采用天根植物总RNA提取试剂盒提取象草样品RNA,反转录成cDNA。利用MonAmp ChemoHS qPCR Mix(None ROX)试剂盒进行实时定量PCR试验,以获得的象草木质素合成关键酶编码基因序列为模板,利用 Primer 5.0 设计特异性实时定量PCR的引物,同时选择持家基因ACTIN作为内参(表 2)。实时荧光定量PCR的反应程序为:95 ℃ 3 min;95 ℃ 10 s,57 ℃ 10 s,72 ℃ 30 s,40个循环。
表2 象草木质素合成关键酶基因实时定量PCR引物序列
Tab.2 Real-time fluorescence quantitative PCR primer sequences for key enzyme genes of elephant grass lignin synthesis

基因名称Gene name引物序列(5'-3')Primer sequence(5'-3')PALF:GTCGATGAGTGGGTTGTCGTR:ACCCACTGATGAAGCCGAAGC3HF:ATTCCAGCAGTGATCATGTCR:TCATTGAAGAGCATGCTAAGGC4CLF:ATCGTCGTCCTCTCCAAGTAR:TCGATGAGCTCCTTGCTCAC4HF:AAGTTGTCCTCGAAGACCTTCR:GACATGTTCCGCATCATGTTCCADF:TTTAGCGGGCTGTACACTGTR:AGTACTGCAACAAGAGGATCTGHCT4F:TCCAGGTAGTCCACCAGCGAR:GGCTCTACTCCATGATCGA-CATGCOMTF:GTTCAAGGCCACCTACATCTAR:CAAGCAAAAGTACATGCACCGF5HF:GAACATGACGATGATGGACCAR:GTACGCGATAGCGATGGTCCoAOMTF:TTGTACAGGGCTTGGCTCTTR:ACACCACGGCTTTCGATTAGCCRF:AGTGACATCTGCTTCTCATCACGR:AAGTTCACGAACCAGAAGCTCCACTINF:GTTCCCTGGTATTGCTGACCR:CACCCTTCGAGATCCACATC
试验结果采用 2-ΔΔCt 算法进行分析,计算出每个关键酶基因的相对表达量。试验设置3次重复。
1.4 数据分析
通过Microsoft Excel 2010进行试验结果的统计和整理并进行绘图,运用SPSS进行方差分析、显著性分析以及相关性分析。
2 结果与分析
2.1 象草木质素的含量变化
在象草不同节间、不同叶片的木质素含量存在显著的差异,随着象草空间垂直结构逐渐向下,木质素的含量呈逐渐递增的趋势,随着取样叶片逐渐向下,木质素的含量也同样呈现逐级递增的趋势(图1-A),这与象草本身茎秆与叶片的硬度变化表现出较为一致的规律,说明随着象草的茎秆和叶片逐渐向下,木质素的合成与积累是逐渐增加的。但是将所有茎秆的木质素含量与叶片的木质素含量进行对比发现两者间不存在显著性差异(图1-B)。
2.2 象草转录组拼接和木质素合成基因的获取
通过Trinity对SRR3952080和SRR3952081测序数据进行de novo拼接,获得474 330条Contigs,共计265 490 867 bp,N50为1 048 bp,GC含量为53.66%。通过tBlastN将水稻木质素合成关键酶的氨基酸序列与象草转录组Contigs进行比对检索,选取每一种酶的最佳匹配序列,每条检索结果的E值都极低,表明检索匹配度非常高;对这些象草转录本序列进行ORF预测和翻译,并通过InterProScan将蛋白序列与Pfam、PANTHER和SUPERFAMILY数据库进行比对检索,结果表明,这些序列均包含相应关键酶的结构域和功能域(表3)。通过以上分析证明,tBlastN检索得到的转录本序列即象草木质素合成关键酶的编码基因序列,可用于实时荧光定量PCR的引物设计,检测相应基因的表达量。
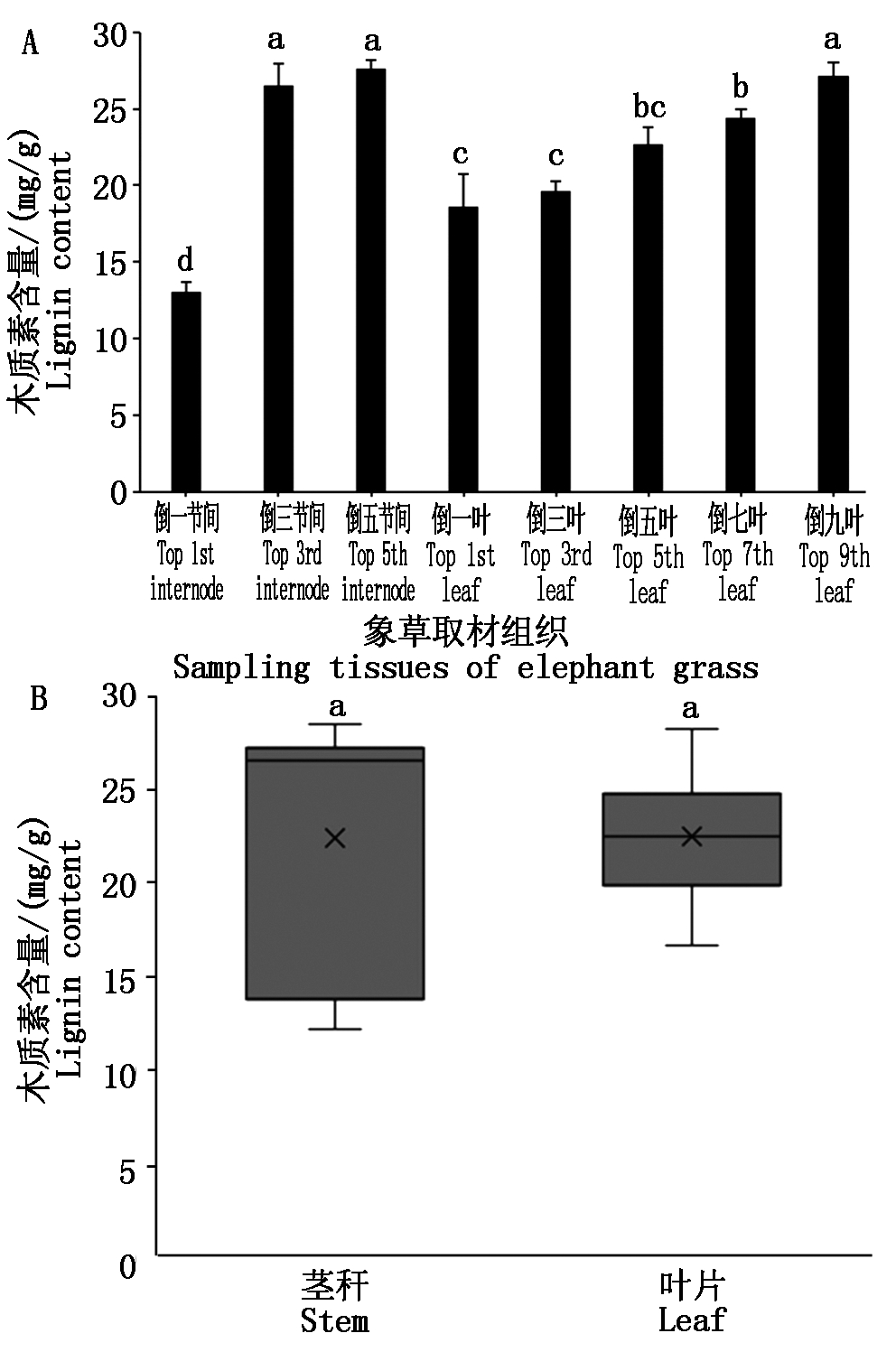
A.N51象草茎秆不同节间和不同叶片中木质素含量;B.N51象草茎秆和叶片木质素含量箱型图;不同小写字母表示差异显著P<0.05。图2-3同。
A. The lignin content in different internodes and different leaves of N51 elephant grass;B. Box plot of lignin content in N51 elephant grass stems and leaves;Different lowercase letters indicate significant differences P<0.05.The same as Fig.2-3.
图1 N51象草茎节间和叶片中木质素含量测定
Fig.1 Determination of lignin content in N51 elephant grass stems and leaves
2.3 象草木质素合成相关酶基因表达量分析
运用实时荧光定量PCR技术对象草的不同节间和不同叶片的木质素合成基因的相对表达量进行分析,结果显示(图2),各基因在象草不同组织中均有表达,但是每个基因的表达模式各不相同。CAD在倒五节间中表达量最高,约是倒一节间的5倍,且在叶中的表达量较低;同样的PAL在倒五节间中表达量最高,约是倒一节间的4倍,其在倒三叶片的表达量次之;CCoAOMT在倒一叶中的表达量是倒一节间的15倍;HCT4整体表达量不高;4CL在象草茎和叶中的表达量呈现递增趋势,其在倒九叶的表达量,均为倒一节间表达量的14倍;F5H表达量在茎中表达量较低并呈递增趋势,在叶片中的表达量先升高再下降,在倒七叶的表达量最高,约为倒九叶片的2倍;CCR和C4H在茎秆节间中的表达量都不高,其中倒三节间的表达量最低,随着叶片逐渐向下,CCR在叶片中的表达量逐级递减,C4H在倒三叶表达量最高,随后降低,COMT在茎中的表达量显著高于叶片,在倒五节间的表达量最高,约是叶片中表达量的6倍。
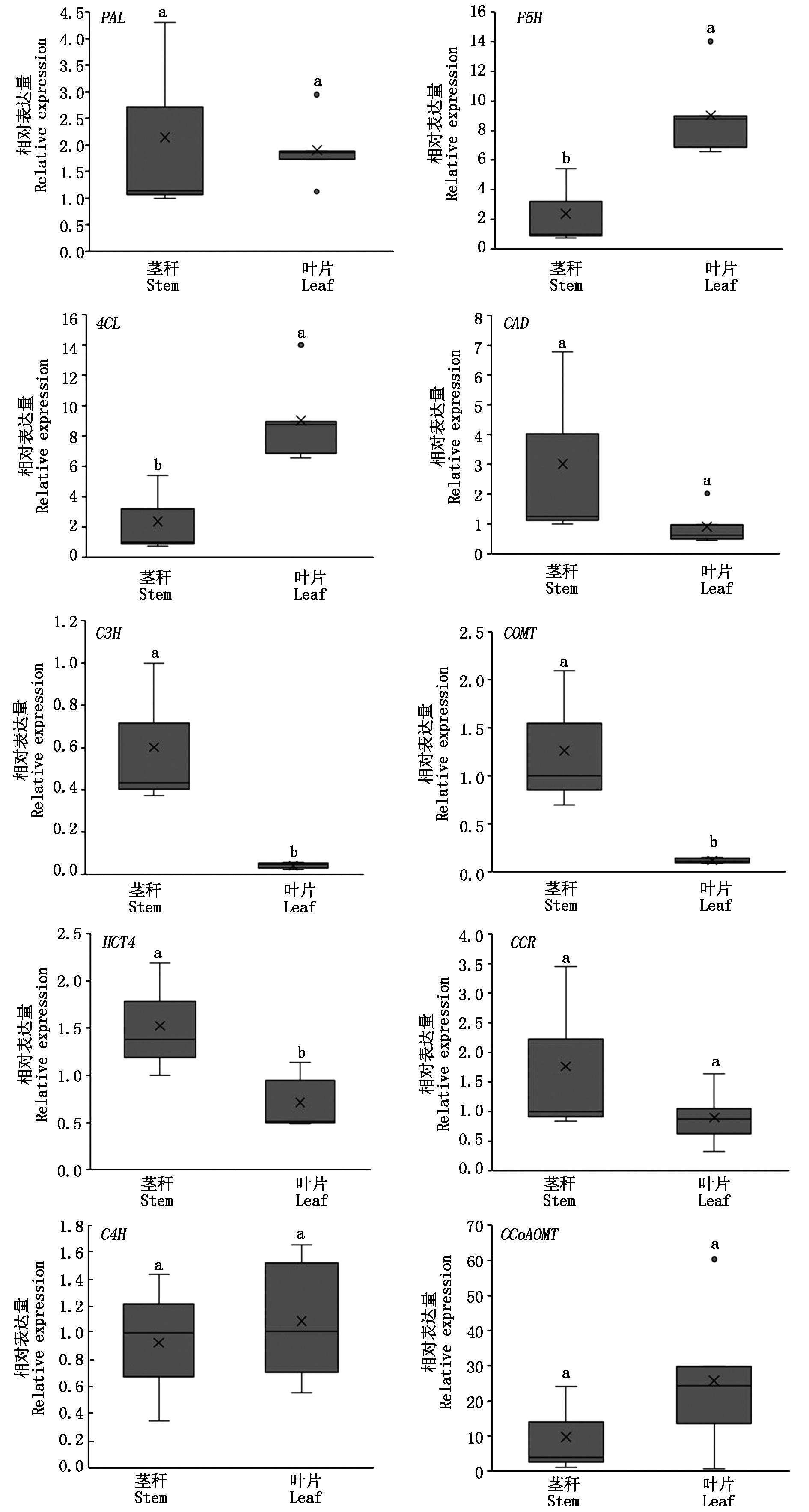
2.4 象草木质素合成相关酶基因在茎秆和叶片中的表达量比较
PAL、CAD、CCR、CCoAOMT和C4H基因在茎秆和叶片中的相对表达量无显著性差异,4CL和F5H在叶片中的表达量显著高于茎秆中的表达量,COMT、C3H和HCT4在叶片中的表达量显著低于茎秆中的表达量(图3)。这些结果说明,不同的木质素合成基因在象草不同组织中的表达丰度是不一样的,从而导致不同组织中木质素含量和构成的差异。
表3 象草木质素合成关键基因的tBlastN比对和结构域及功能域检索
Tab.3 tBlastN alignment and domain search for key genes in elephant grass lignin synthesis
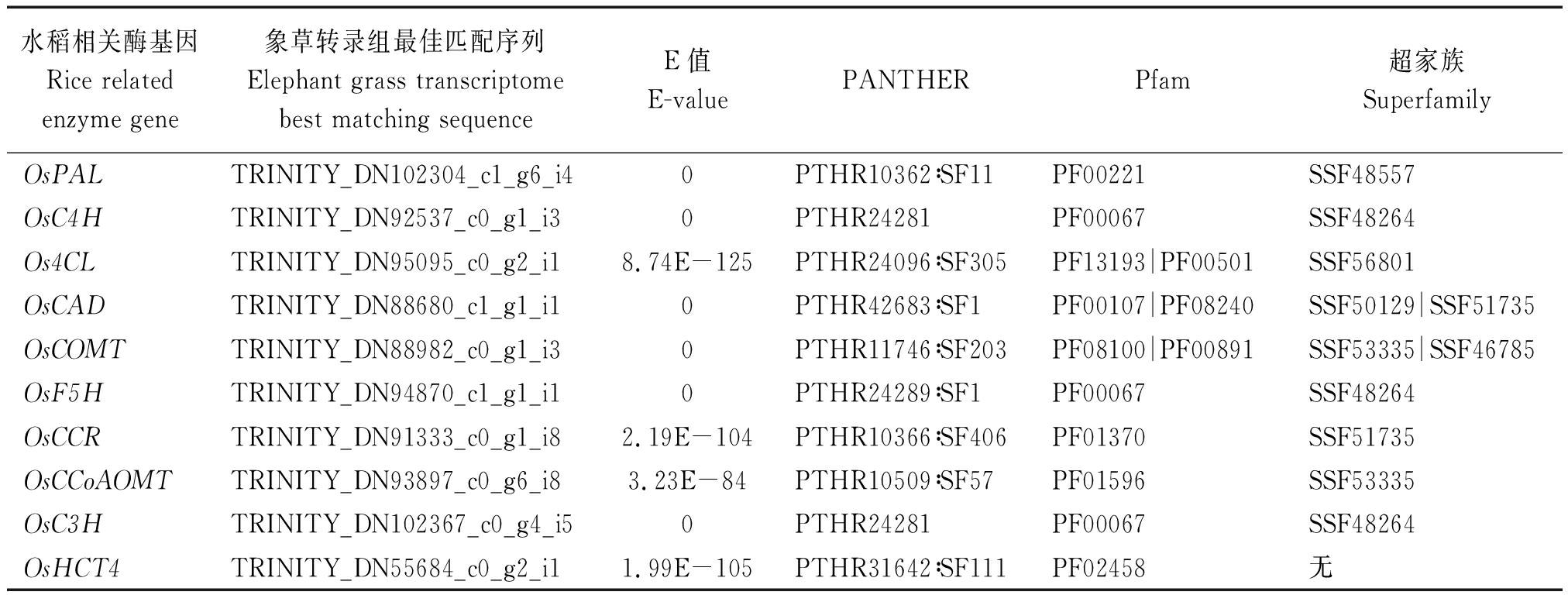
水稻相关酶基因Rice relatedenzyme gene象草转录组最佳匹配序列Elephant grass transcriptomebest matching sequenceE值E-valuePANTHERPfam超家族SuperfamilyOsPALTRINITY_DN102304_c1_g6_i40PTHR10362∶SF11PF00221SSF48557OsC4HTRINITY_DN92537_c0_g1_i30PTHR24281PF00067SSF48264Os4CLTRINITY_DN95095_c0_g2_i18.74E-125PTHR24096∶SF305PF13193|PF00501SSF56801OsCADTRINITY_DN88680_c1_g1_i10PTHR42683∶SF1PF00107|PF08240SSF50129|SSF51735OsCOMTTRINITY_DN88982_c0_g1_i30PTHR11746∶SF203PF08100|PF00891SSF53335|SSF46785OsF5HTRINITY_DN94870_c1_g1_i10PTHR24289∶SF1PF00067SSF48264OsCCRTRINITY_DN91333_c0_g1_i82.19E-104PTHR10366∶SF406PF01370SSF51735OsCCoAOMTTRINITY_DN93897_c0_g6_i83.23E-84PTHR10509∶SF57PF01596SSF53335OsC3HTRINITY_DN102367_c0_g4_i50PTHR24281PF00067SSF48264OsHCT4TRINITY_DN55684_c0_g2_i11.99E-105PTHR31642∶SF111PF02458无
2.5 象草木质素含量与木质素合成相关酶基因的相关性分析
运用SPSS软件对象草的茎和叶中的木质素含量和在其对应部位的木质素相关酶基因的表达量进行分析,结果如表4所示,在象草的茎秆中,HCT4和CCoAOMT的相对表达量与木质素含量呈现显著正相关,F5H的相对表达量与木质素含量呈现极显著正相关,C3H的相对表达量与木质素含量呈现极显著负相关。在象草的叶片中,C4H、COMT和CCR的相对表达量与木质素含量呈显著负相关,HCT4的相对表达量与木质素含量呈现极显著负相关,4CL的相对表达量与木质素含量呈现极显著正相关。
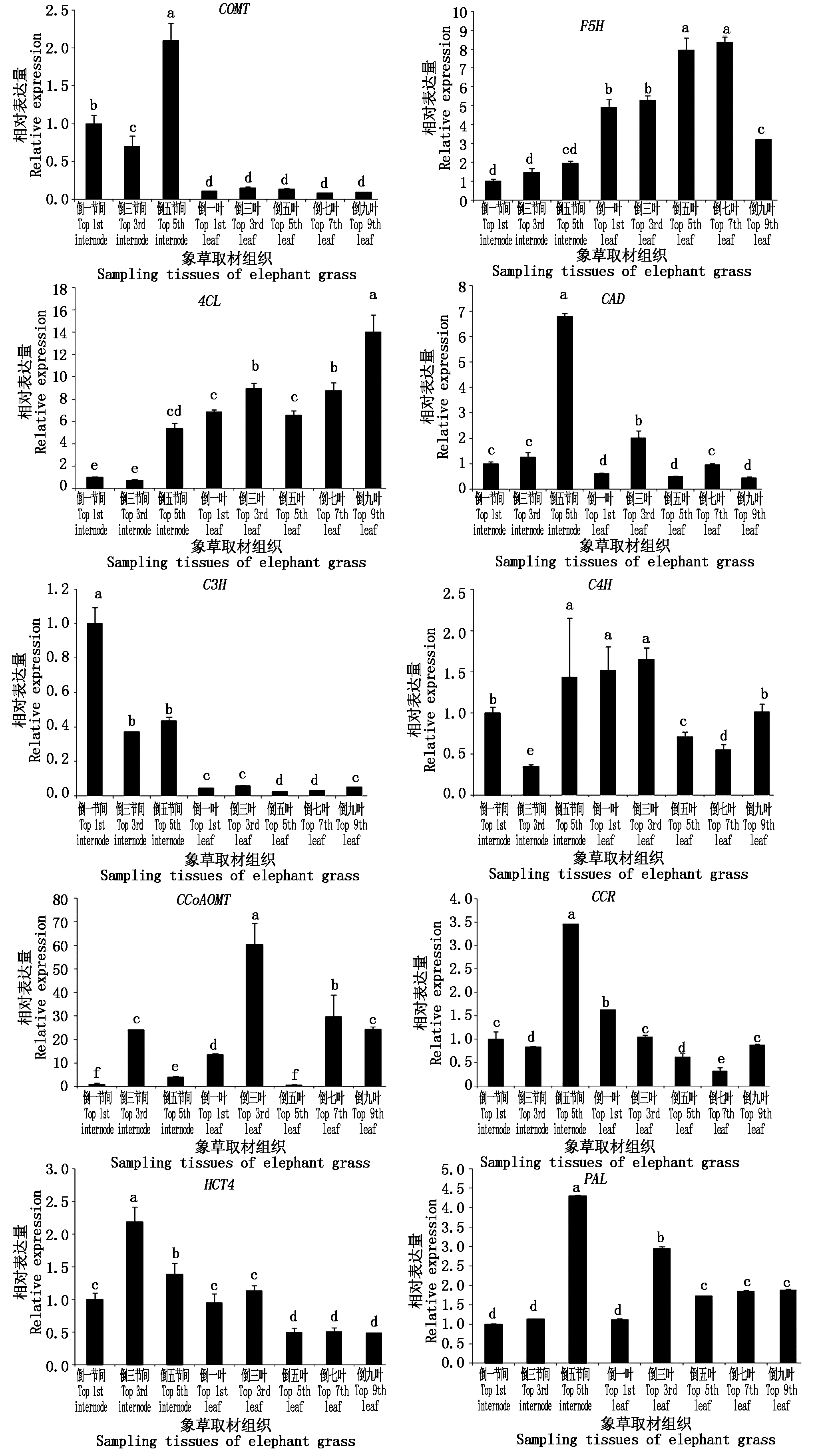
图2 N51象草木质素合成相关酶基因表达量
Fig.2 Expression level of N51 elephant grass lignin synthesis related enzyme gene
茎秆的表达量为所有节间表达量的平均值;叶片的表达量为所有叶片表达量的平均值。
The expression level of stems is the average of all internode expression levels;The expression level of leaves is the average of all leaf expression levels.
图3 N51象草木质素合成相关酶基因在茎秆和叶片中的表达量
Fig.3 Expression of N51 elephant grass lignin synthesis related enzyme genes in stems and leaves
表4 象草的茎和叶中的木质素含量与其合成相关酶的相对表达量之间的相关性
Tab.4 Correlation between lignin content in elephant grass stems and leaves and relative expression of their synthetic enzymes

木质素含量Lignin content木质素合成相关酶基因的相对表达量Relative expression of lignin synthesis-related enzyme genesPAL4CLC3HC4HCOMTF5HCADHCT4CCRCCoAOMT茎秆Stem(N=9)0.5600.509-0.971**-0.0580.3570.881**0.5830.668*0.5040.671*叶片Leaf(N=15)-0.0890.668**-0.204-0.556*-0.561*-0.044-0.431-0.807**-0.611*-0.174
注:*.P<0.05;**.P<0.01。
3 结论与讨论
3.1 象草木质素合成基因表达模式的组织特异性
尽管N51象草叶组织和茎组织的木质素总含量并没有显著性差异,但是不同木质素合成基因的表达却表现出了显著的组织特异性。叶组织的COMT、C3H和HCT4基因表达量显著低于茎组织,而4CL和F5H基因却显著高于茎组织。关联分析结果表明,茎秆中的木质素含量与F5H、HCT4以及CCoAOMT基因的表达呈正相关,与C3H基因的表达呈负相关,而叶片中的木质素含量与4CL基因的表达呈正相关,与C4H、COMT、HCT4以及CCR基因的表达呈负相关。在陈博雯等[20]研究中发现抑制CCoAOMT的表达可以显著降低G型木质素的合成效果;张婷[21]在研究酶基因4CL和CCoAOMT反义转化紫穗槐的试验中发现获得的转基因植株不仅可以正常生长,而且木质素的含量降低;师竹娟[22]在研究甘蓝型油菜木质素单体合成的基因调控中,为了提高甘蓝型油菜中G型木质素含量从而增强植物的抗倒伏能力,克隆了F5H基因,获得F5H反义表达转基因植株,发现茎秆木质素的沉淀分布发生了变化。其实目前大多数的研究都关注于木质素合成关键基因表达量对植株茎秆木质素含量之间的影响,鲜有关于影响叶片中木质素含量和构成的研究报道。借鉴以上的研究,注意到木质素的合成中不同的酶基因的表达量可能与合成的哪种木质素单体有关,于是本研究测定了象草不同部位的木质素含量及相关合成基因的表达量变化,结果表明,不同木质素合成基因在象草茎秆和叶片中的表达模式和作用机制都不甚相同。象草是非常重要的禾本科牧草,其叶片是牛羊食用的主要部位,叶片的木质素含量和构成能影响其适口性和营养价值,而本研究结果表明,木质素合成基因在叶组织中的功能和机制可能与茎组织存在差异,具有进一步研究的空间和价值。
3.2 G型和S型木质素单体合成关键基因的表达特性
植物细胞壁木质素含有H、G和S等3种类型的单体,其中G和S型单体的占比较高,因此木质素含量和成分的调控主要针对G型和S型单体的合成来进行操作。F5H(Ferulate 5-hydroxylase)将阿魏酸羟基化为5-羟基阿魏酸,5-羟基阿魏酸是合成S型木质素单体的前体。F5H编码基因的表达变化能显著影响被子植物中S∶G型木质素单体的比例。当敲除F5H基因时,植物只产生G型单体[23];当F5H基因过表达时,植物体内会大量积累S型单体,其含量可高达98%[24-25]。在N51象草中,叶片F5H基因表达量显著高于茎秆。F5H是合成S型单体的关键酶,叶片中F5H基因的高表达很可能意味着在叶片中S型单体的合成比茎组织更加旺盛。虽然茎秆中F5H基因的表达远低于叶片,但是其木质素含量与F5H基因的表达量呈极显著正相关,表明随着茎秆的成熟和木质素的积累,F5H基因的表达会不断上调,促进S型单体的合成。
COMT和4CL是S型和G型木质素单体合成通路中的关键酶,当COMT和4CL基因表达下调时,植物中的木质素总量和S/G型单体比例均会下降。当水稻中4CL基因被沉默时,植株的木质素总含量显著降低。通过RNA干涉技术下调杂交白杨(Populus tremula×Populus alba)中4CL基因的表达,能够使杂交杨枝条的木质素单体含量显著降低,减幅最高可达50%,S/G型单体比例下降,H型单体含量升高[26]。通过RNA干涉技术下调多年生黑麦草中(Lolium perenne)COMT1基因的表达,能够显著影响木质素的含量和构成,但不会对多年生黑麦草的植株健康和生物量产生负面影响,当COMT1基因的表达被显著下调时,多年生黑麦草茎中S型和G型单体含量都显著降低,S型单体下降幅度更大,从而导致S/G单体比例显著下降,而H型单体的含量则没有发生显著性变化[27]。在象草叶片中4CL基因的表达显著高于茎秆,且与木质素含量呈正相关,其表达量随着叶片的成熟和木质素的积累而逐渐上调,而COMT基因的表达却显著低于茎秆,且与木质素含量呈显著负相关,其表达量随着叶片的成熟和木质素的积累而逐渐下调,这表明4CL和COMT虽然同时参与S型和G型单体的合成,但是在叶片和茎秆木质素合成中的作用机制却不尽相同,叶片的成熟和木质素积累更依赖于4CL的作用,而茎秆则更依赖于COMT的作用。
3.3 H型木质素单体合成关键基因的表达特性
H型单体在木质素单体总量中占比不高,且主要在次生壁开始加厚的初期积累。H型木质素单体的合成与C3H和HCT基因密切相关[28-29]。在紫花苜蓿(Medicago sativa)中表达HCT反义载体,导致木质素含量显著降低,并显著改变了木质素构成,H型单体的比例会大幅度的提高。拟南芥c3h突变体表现出明显矮化特征,推测可能是因为H型木质素含量过低,不足以满足植物细胞壁合成所需[30]。在N51象草中,茎秆中的C3H和HCT4基因表达量均显著高于叶片,推测高表达的C3H和HCT4基因能促进茎秆中H型单体的合成,帮助茎秆形成厚实坚硬的次生壁,从而为整棵植株提供支撑作用。
3.4 结论
木质素合成是一个非常复杂的过程,参与调控的酶有多种,分别控制3个木质素单体的合成。本研究中,测定成熟象草的不同茎秆和叶片的木质素含量,在茎秆中,随着象草空间垂直结构逐渐向下,木质素含量逐渐增加,同样,在叶片中,随着象草空间垂直结构逐渐向下,木质素的含量也逐渐增加,这表明,在象草的空间垂直结构上,木质素含量在各组织中呈现逐级递增趋势,这与象草本身木质素的变化情况一致。测定了10种合成酶基因的表达量,并分析10种酶基因分别在茎秆和叶片中表达量对比结果,发现不同的木质素合成酶在不同的组织中呈现表达差异。这说明在不同组织中,合成的主要木质素不同。综上所述,象草木质素的合成受到多种酶基因的调控,在不同的组织中,木质素单体含量不同。
[1] 冯鹤翔, 涂轶. 木质素生物合成的研究[J]. 青岛大学学报(自然科学版),2018,31(1):46-54.doi:10.3969/j.issn.1006-1037.2018.02.10.
Feng H X, Tu Y.Research on lignin biosynthesis[J]. Journal of Qingdao University(Natural Science Edition),2018,31(1):46-54.
[2] 丁霄, 曹彩荣, 李朋波, 吴翠翠, 曹美莲, 杨六六. 植物木质素的合成与调控研究进展[J]. 山西农业科学,2016,44(9):1406-1411.doi:10.3969/j.issn.1002-2481.2016.09.43.
Ding X, Cao C R, Li P B, Wu C C, Cao M L, Yang L L. Research progress on synthesis and regulation of plant lignin[J]. Journal of Shanxi Agricultural Sciences, 2016, 44(9):1406-1411.
[3] 陈磊. 石榴籽粒木质素合成途径关键基因的表达及功能分析[D]. 合肥:安徽农业大学, 2018.
Chen L. Expession and functional analysis of pomegranate seed lignin biosynthesis key gene[D]. Hefei: Anhui Agricultural University, 2018.
[4] 李果. 油用牡丹‘凤丹’DFR基因克隆与功能验证及木质素合成基因发掘[D]. 杨凌:西北农林科技大学, 2017.
Li G. Molecular cloning and functional analysis of dihydro flavonol 4-reductas (DFR) gene and screening of lignin genes in Paeonia ostii[D]. Yangling: Northwest A&F University, 2017.
[5] 张旭, 王小佳, 黎思辰, 董甜甜, 汪志辉. 柑橘果实粒化过程中木质素生物合成与调控研究进展[J]. 浙江农业学报, 2019, 31(12): 2131-2140. doi:10.3969/j.issn.1004-1524.2019.12.22.
Zhang X, Wang X J, Li S C, Dong T T, Wang Z H. Research progress of lignin biosynthesis and regulation during granulation of citrus[J]. Acta Agriculturae Zhejiangensis, 2019, 31(12): 2131-2140.
[6] 丁忆然.甘蓝型油菜种皮木质素合成相关基因的差异表达分析[D]. 重庆:西南大学, 2019.
Ding Y R. Differential expression analysis of lignin synthesis related genes in the seed coat of Brassica napus L.[D]. Chongqing: Southwest University, 2019.
[7] Ralph J, MacKay J J, Hatfield R D, O'Malley D M, Whetten R W, Sederoff R R. Abnormal lignin in a loblolly pine mutant[J]. Science, 1997, 277(5323):235-239. doi:10.1126/science.277.5323.235.
[8] Whetten R W, MacKay J J, Sederoff R R. Recent advances in understanding lignin biosynthesis[J]. Annual Review of Plant Physiology and Plant Molecular Biology, 1998, 49: 585-609. doi: 10.1146/annurev.arplant.49.1.585.
[9] 郭璇. 木质素模型化合物的合成及解聚研究[D].北京:北京化工大学, 2018.
Guo X. Synthesis and depolymerization of lignin model compounds[D]. Beijing: Beijing University of Chemical Technology, 2018.
[10] Somerville C, Youngs H, Taylor C, Davis S C, Long S P. Feedstocks for lignocellulosic biofuels[J]. Science, 2010, 329(5993): 790-792. doi:10.1126/science.1189268.
[11] 钟天秀, 李有涵, 李菲, 彭小群, 柯善文, 陈曙, 解新明. 华南象草Pp4CL基因的克隆及其转基因烟草木质素含量分析[J]. 西北植物学报, 2015, 35(12): 2355-2364. doi:10.7606/j.issn.1000-4025.2015.12.2355.
Zhong T X, Li Y H, Li F, Peng X Q, Ke S W, Chen S, Xie X M.Isolation of 4-coumarate COA ligase gene from Pennisetum purpureum cv. Huanan and lignin content analysis of transgenic tobacco plants[J].Acta Botanica Boreali-Occidentalia Sinica, 2015, 35(12): 2355-2364.
[12] Liu X H, Shen Y X, Lou L Q, Ding C L, Cai Q S. Copper tolerance of the biomass crops elephant grass ( Pennisetum purpureum Schumach), vetiver grass (Vetiveria zizanioides) and the upland reed(Phragmites australis)in soil culture[J]. Biotechnology Advances, 2009, 27(5): 633-640. doi:10.1016/j.biotechadv.2009.04.017.
[13] 彭小群. 象草MYB4转录因子及CCR启动子的克隆与功能分析[D]. 广州:华南农业大学, 2016.
Peng X Q. Cloning and functional analysis of PpMYB4 transcription factor and PpCCR gene promoter in Pennisetum purpureum[D]. Guangzhou: South China Agricultural University, 2016.
[14] 赵新坤, 李赫龙, 佘雕, 孙润仓. 木质素模型化合物合成研究的现状与展望[J]. 生物质化学工程, 2018,52(1): 41-52. doi:10.3969/j.issn.1673-5854.2018.01.007.
Zhao X K, Li H L, She D, Sun R C. Status and prospect of lignin model compounds synthesis[J]. Biomass Chemical Engineering, 2018, 52(1): 41-52.
[15] 郭永翠, 秦江南, 张锐. 核桃内果皮木质素生物合成途径关键基因研究进展[J].现代园艺, 2019(7): 5-8. doi:10.14051/j.cnki.xdyy.2019.07.002.
Guo Y C, Qin J N, Zhang R. Research progress on key genes of lignin biosynthesis pathway in walnut endocarp[J]. Xiandai Horticulture, 2019(7): 5-8.
[16] 陈永忠, 谭晓风, David Clapham. 木质素生物合成及其基因调控研究综述[J]. 江西农业大学学报(自然科学), 2003, 25(4): 613-617. doi:10.13836/j.jjau.2003140.
Chen Y Z, Tan X F, Clapham D.Lignin biosynthesis and genetic regulation[J].Acta Agriculturae Universitatis Jiangxiensis(Natural Sciences Edition), 2003,25(4): 613-617.
[17] 彭小群, 张博雅, 刘洁雯, 王亚, 解新明. 象草PpCCR基因正、反义表达载体的构建及对烟草的转化[J]. 草业科学, 2015,9(10): 1586-1593. doi:10.11829/j.issn.1001-0629.2014-0572.
Peng X Q, Zhang B Y, Liu J W, Wang Y, Xie X M.Construction of sense and antisense expression vectors of PpCCR gene in Pennisetum purpureum and transformation to tobacco plants[J]. Pratacultural Science, 2015,9(10): 1586-1593.
[18] 唐然, 张博雅, 彭小群, 张向前, 江院, 解新明. 华南象草PpCAD基因的亚细胞定位及其在转基因烟草中的异源表达[J]. 西北植物学报, 2015, 35(6): 1069-1077. doi:10.7606/j.issn.1000-4025.2015.06.1069.
Tang R, Zhang B Y, Peng X Q, Zhang X Q, Jiang Y, Xie X M. Subcellular localization of PpCAD from elephant grass (Pennisetum purpureum cv. Huanan) and its heterologous expression in transgenic tobacco[J]. Acta Botanica Boreali-Occidentalia Sinica, 2015, 35(6): 1069-1077.
[19] 王建庆, 曹佃元, 张玉. 乙酰溴法测定棉籽壳中木质素的含量[J]. 纺织学报, 2013, 34(9): 12-16. doi: 10.13475/j.fzxb.2013.09.011.
Wang J Q, Cao D Y, Zhang Y.Acetyl bromide test method for determining amount of lignin in cottonseed coat[J].Journal of Textile Research, 2013, 34(9): 12-16.
[20] 陈博雯, 刘海龙, 肖玉菲, 覃子海, 张烨, 张晓宁. 尾叶桉COMT和CCoAOMT基因定向调控木质素单体合成的烟草转化研究[J]. 中国生物工程杂志, 2018, 38(3): 24-32. doi:10.13523/j.cb.20180304.
Chen B W, Liu H L, Xiao Y F, Qin Z H, Zhang Y, Zhang X N.Directional regulation of lignin monomer synthesis in tobacco by using COMT gene and CCo AOMT gene of Eucalyptus urophylla[J]. Chinese Biotechnology, 2018, 38(3): 24-32.
[21] 张婷. 木质素合成酶基因4CL和CCoAOMT反义转化紫穗槐的实验研究[D]. 长春:吉林大学, 2008.
Zhang T. Study on antisense transformation with lignin biosynthesis 4CL and CCoAOMT gene in Amorpha fruitcosa[D]. Changchun: Jilin University, 2008.
[22] 师竹娟. 甘蓝型油菜木质素单体合成基因COMT和F5H的克隆及表达调控[D]. 北京:中国农业科学院, 2006.doi:10.7666/d.Y880672.
Shi Z J. Monolignol biosynthesis gene COMT and F5H cloning and expression regulation in Brassica napus L.[D]. Beijing: Chinese Academy of Agricultural Sciences, 2006.
[23] Meyer K, Shirley A M, Cusumano J C, Bell-Lelong D A, Chapple C. Lignin monomer composition is determined by the expression of a cytochrome P450-dependent monooxygenase in Arabidopsis[J]. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(12): 6619-6623. doi:10.1073/pnas.95.12.6619.
[24] Shuai L, Amiri M T, Questell-Santiago Y M, Héroguel F, Li Y D, Kim H, Meilan R, Chapple C, Ralph J, Luterbacher J S. Formaldehyde stabilization facilitates lignin monomer production during biomass depolymerization[J]. Science, 2016, 354(6310): 329-333. doi:10.1126/science.aaf7810.
[25] Stewart J J, Akiyama T, Chapple C, Ralph J, Mansfield S D. The effects on lignin structure of overexpression of ferulate 5-Hydroxylase in hybrid poplar[J]. Plant Physiology, 2009, 150(2): 621-635. doi: 10.1104/pp.109.137059.
[26] Voelker S L, Lachenbruch B, Meinzer F C, Jourdes M, Ki C, Patten A M, Davin L B, Lewis N G, Tuskan G A, Gunter L, Decker S R, Selig M J, Sykes R, Himmel M E, Kitin P, Shevchenko O, Strauss S H. Antisense down-regulation of 4CL expression alters lignification, tree growth, and saccharification potential of field-grown poplar[J]. Plant Physiology, 2010, 154(2): 874-886. doi: 10.1104/pp.110.159269.
[27] Tu Y, Rochfort S, Liu Z Q, Ran Y D, Griffith M, Badenhorst P, Louie G V, Bowman M E, Smith K F, Noel J P, Mouradov A, Spangenberg G. Functional analyses of caffeic acid O-methyltransferase and Cinnamoyl-CoA-reductase genes from perennial ryegrass (Lolium perenne)[J]. The Plant Cell, 2010, 22(10): 3357-3373. doi: 10.1105/tpc.109.072827.
[28] Ong R G, Higbee A, Bottoms S, Dickinson Q, Xie D, Smith S A, Serate J, Pohlmann E, Jones A D, Coon J J, Sato T K, Sanford G R, Eilert D, Oates L G, Piotrowski J S, Bates D M, Cavalier D, Zhang Y P. Inhibition of microbial biofuel production in drought-stressed switchgrass hydrolysate[J]. Biotechnology for Biofuels, 2016, 9:237. doi: 10.1186/s13068-016-0657-0.
[29] Vanholme R, Cesarino I, Rataj K, Xiao Y G ,Sundin L,Goeminne G, Kim H,Cross J, Morreel K, Araujo P, Welsh L, Haustraete J,McClellan C, Vanholme B,Ralph J,Simpson G G,Halpin C,Boerjan W. Caffeoyl shikimate esterase (CSE) is an enzyme in the lignin biosynthetic pathway in Arabidopsis[J]. Science, 2013, 341(6150): 1103-1106. doi: 10.1126/science.1241602.
[30] Bonawitz N D, Kim J I, Tobimatsu Y, Ciesielski P N, Anderson N A, Ximenes E, Maeda J, Ralph J, Donohoe B S, Ladisch M, Chapple C. Disruption of mediator rescues the stunted growth of a lignin-deficient Arabidopsis mutant[J]. Nature, 2014, 509(7500): 376-380. doi:10.1038/nature13084.
