蛋白酶抑制剂是一种广泛存在于植物体内,可以使蛋白酶活力下降的一类蛋白质,很多蛋白酶抑制剂,特别是植物中的蛋白酶抑制剂,还具有抑制微生物病原体以及一些昆虫蛋白酶的作用,是一种天然的防御系统[1]。蛋白酶抑制剂分布较广,主要分为四大类:金属蛋白酶抑制剂、丝氨酸蛋白酶抑制剂、酸性蛋白酶抑制剂和半胱氨酸蛋白酶抑制剂[2]。半胱氨酸蛋白酶抑制剂于20世纪50年代首次在哺乳动物组织中发现,绝大多数的半胱氨酸蛋白酶抑制剂都含有特定的Q-V-G保守区域及色氨酸残基[3-4],在植物半胱氨酸蛋白酶抑制剂的氨基酸序列中,还包含有α螺旋结构序列[5]。近些年来,植物半胱氨酸蛋白酶抑制剂已逐步分离出来,形成一个独特的亚家族[6]。Kondo等[7]从水稻中发现并克隆了第一个植物半胱氨酸蛋白酶抑制剂基因,紧接着在其他植物中,许多其他的植物半胱氨酸蛋白酶抑制剂陆续被发现[8-9]。研究表明,植物半胱氨酸蛋白酶抑制剂在非生物逆境胁迫、病虫害防御以及植物种子萌发、果实发育、程序性细胞死亡等生理过程中发挥着十分重要的作用[10-14]。
到目前为止,拟南芥中已有多个植物半胱氨酸蛋白酶抑制剂基因被研究和报道[15]。在烟草中过量表达AtCYS1可以抑制和减少氧化胁迫或者氮氧胁迫造成的细胞损伤死亡[16];AtCYS3过量表达的拟南芥植株可以提高对盐碱和干旱等非生物逆境胁迫的耐受性[17];对AtCYS1和AtCYS2启动子的研究表明,它们在植物应对非生物逆境胁迫响应方面起着重要的作用机制[18];在拟南芥中过量表达AtCYS4和AtCYS5不仅可以促进种子的萌发以及幼苗的生长,还可以提高转基因植株对高温胁迫的耐受性[19-20]。这些研究表明,拟南芥半胱氨酸蛋白酶抑制剂基因在植物应对非生物逆境胁迫及生长发育中起着非常重要的作用。
本研究以拟南芥为研究对象,通过PCR扩增和测序方法克隆拟南芥半胱氨酸蛋白酶抑制剂AtCYS6基因全序列,分析其序列特征,构建其蛋白表达载体并进行蛋白诱导纯化及酶活性分析,以期更全面了解AtCYS6基因,为更详尽探索其参与调控的机理机制提供一定的研究基础。
1 材料和方法
1.1 试验材料
野生型拟南芥(哥伦比亚型)、大肠杆菌 JM109和BL21菌株及原核表达载体pGEX-6p-3由湖北省农业科学院中药材研究所资源与育种实验室保存,T4 DNA连接酶、限制性内切酶等购自TaKaRa公司,RNA提取试剂盒、琼脂糖凝胶回收试剂盒、反转录试剂盒和质粒提取试剂盒等购自天根生化科技有限公司,木瓜蛋白酶、半胱氨酸、GST标签蛋白纯化柱填料和尼龙膜等购自生工生物工程股份有限公司,检测GST标签蛋白的一抗和二抗购自碧云天生物技术研究所,其余试剂为国产分析纯。
1.2 试验方法
1.2.1 基因序列克隆 取培养14 d大小的拟南芥材料,在液氮下研磨均匀后,按照RNA提取试剂盒说明书的方法进行RNA提取,然后以提取的RNA为模板,按照反转录试剂盒说明书进行反转录试验。根据GenBank数据库中AtCYS6基因序列(序列号:NC_003074.8),以拟南芥cDNA为模板,利用上游引物F:5′-GTGAGAATGATGAGAAGCCG-3′,下游引物R:5′-GGAGAGGGACTAGTCATGGT-3′进行PCR扩增,退火温度为54 ℃,同时以水为模板作为空白对照。
1.2.2 序列生物信息学分析 将获得的AtCYS6氨基酸序列在GenBank数据库中进行Blast比对分析,利用DNAMAN等软件对其与荠菜(Capsella rubella;XP_006298448.1)、荠蓝(Camelina sativa;XP_010465015.1)、水稻(Oryza sativa;NP_001042702.1)、玉米(Zea mays;XP_008655485.1)、大豆(Glycine max;NP_001237734.1)、山嵛菜 (Eutrema salsugineum;XP_006407331.1)、烟草 (Nicotiana tomentosiformis;XP_009601821.1)、高粱 (Sorghum bicolor;XP_002457629.1)、谷子(Setaria italica;XP_004967724.1)等进行同源性分析,并利用MEGA 5软件构建系统进化树。在线网站分析,预测相对分子质量及等电点(http://web.expasy.org/protparam/),进行疏水性分析(http://web.expasy.org/protscale/),利用TMHMM预测跨膜结构域分析(http://www.cbs.dtu.dk/services/TMHMM/),预测信号肽分析(http://www.cbs.dtu.dk/services/SignalP),预测亚细胞定位分析(http://psort.hgc.jp/form.html),利用SWISS-MODEL(http://swissmodel.expasy.org/)进行结构域的三维同源建模。
1.2.3 AtCYS6-GFP蛋白的亚细胞定位 将AtCYS6-GFP瞬时表达载体利用聚乙二醇诱导法[21],转化拟南芥叶片细胞原生质体,原生质体先在黑暗条件过夜培养,然后在激光共聚焦显微镜下观察GFP荧光信号位置。
1.2.4 pGEX-6p-3-AtCYS6融合表达载体的构建 根据目的序列利用引物进行扩增,上游引物F:5′-GGATCCATGATGAGAAGCCG-3′,下游引物R:5′-GTCGACCTAGTCATGGTGTT-3′,退火温度为54 ℃。将PCR产物用BamH Ⅰ和Sal Ⅰ与载体pGEX-6p-3连接,构建pGEX-6-3-AtCYS6表达载体,然后将构建好的融合表达载体转化BL21菌株。
1.2.5 融合蛋白的诱导、纯化及Western Blot分析 小量诱导:分别挑取含有pGEX-6-3和pGEX-6-3-AtCYS6质粒的单克隆于LB液体培养基中振荡培养,待菌液的OD600达到对数中期时开始进行融合蛋白的诱导,诱导时间分别为0,15,30,60,120,210 min,诱导结束后收集每个时间的大肠杆菌进行SDS-PAGE电泳;大量诱导及纯化:根据小量诱导结果,利用三角瓶进行大量诱导,然后利用溶菌酶裂解菌液,菌液上清过GST 凝胶树脂进行GST和GST-AtCYS6融合蛋白的纯化,最后进行SDS-PAGE电泳及 Western Blot分析。
1.2.6 融合蛋白酶活性分析 将2.5~5.0 μg纯化的GST蛋白和 GST-AtCYS6蛋白与15 mL 1.0 mg/mL的木瓜蛋白酶溶液(100 mmol/L磷酸钠,10 mmol/L EDTA,5 mmol/L半胱氨酸)混合,37 ℃孵育20 min,孵育结束后,往混合液里面加入500 μL 1.0 mg/mL的偶氮酪蛋白溶液(100 mmol/L磷酸钠,15 mmol/L EDTA,15 mmol/L半胱氨酸),37 ℃孵育60~90 min,孵育结束后,往反应液里加入200 μL 10%(V/V)的三氯乙酸(TCA),于冰上放置30 min以终止反应,最后离心取上清于420 nm下测量OD值,所有试验测定至少重复3次。
2 结果与分析
2.1 基因序列克隆
利用设计的引物对AtCYS6目的条带进行扩增,扩增结果如图1所示,AtCYS6片段的大小约为730 bp,证明扩增结果正确。
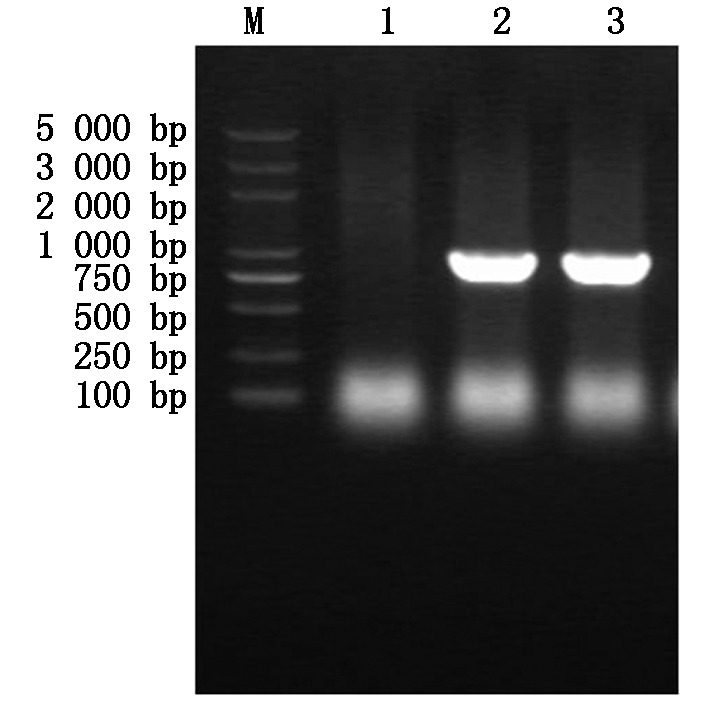
M.DNA Marker;1.空白对照;2,3.目的基因PCR扩增。
M.DNA Marker;1.Blank control;2,3.Target gene PCR amplification.
图1 AtCYS6基因序列克隆
Fig.1 Cloning of AtCYS6 gene sequence
2.2 序列生物信息学分析
利用在线网站(http://web.expasy.org/protparam/)预测AtCYS6蛋白相对分子质量为26.293 95 ku,等电点为5.85,化学式为C1179H1848N322O348S6,共含有3 703个原子数。蛋白亲/疏水性预测结果显示,约在第10个氨基酸位置,疏水性较大,峰值为3.025,在第65个氨基酸位置,亲水性较大,峰值为-2.995,整个分布区域亲水性氨基酸区域多于疏水性氨基酸区域,可认为AtCYS6为亲水性蛋白(图2-A)。SignalP 4.1 Server分析结果显示C值为0.476,Y值为0.674,S值为0.994,得分较高,表明AtCYS6蛋白有信号肽(图2-B)。TMHMM跨膜结构分析表明,AtCYS6蛋白含有1个跨膜区域(图2-C)。亚细胞定位分析表明,AtCYS6蛋白定位于细胞质的概率为0.82,内质网和溶酶体中的概率为0.1,其他细胞器的概率较低。在其整个氨基酸序列中,谷氨酸(Glu)含量最高,达到了12%,亮氨酸(Leu)、丙氨酸(Ala)、赖氨酸(Lys)、缬氨酸(Val)的含量也相对比较高,超过8.5%,而半胱氨酸(Cys)的含量最少,不到1%。蛋白质三级结构预测显示它包含2个部分,第一部分是由3个反向平行的β片层包裹着一个α-螺旋中心组成,第二部分是由5个反向平行的β片层包裹着一个α-螺旋中心组成(图2-D)。

A.疏水/亲水性预测;B.信号肽预测;C.跨膜结构域预测;D.蛋白质三级结构预测。
A.Hydrophobic/hydrophilic prediction;B.Signal peptide prediction;C.Transmembrane domain prediction;D.Protein tertiary structure prediction.
图2 AtCYS6序列生物信息学分析
Fig.2 Bioinformatics analysis of AtCYS6 sequence
2.3 氨基酸序列比对及系统进化树分析
本研究将AtCYS6与其他物种中的半胱氨酸蛋白酶抑制剂进行了比对,结果如图3所示,AtCYS6与十字花科的荠菜、荠蓝的同源性较高,同源性分别为81.1%,80.3%,同水稻、玉米和大豆的同源性较低,同源性分别为55.5%,54.3%,56.6%,且在氨基酸序列中间具有高度保守的Q-V-G(Gln-Val-Gly)序列。

图3 AtCYS6氨基酸同源性比对
Fig.3 Amino acid homology comparison of AtCYS6
本研究通过BioEdit、ClustalW和MEGA 5等生物软件对AtCYS6和其他物种的半胱氨酸蛋白酶抑制剂编码的氨基酸序列进行系统进化树分析,如图4所示,可以发现AtCYS6与荠菜、荠蓝的亲缘性最近,并且都同属于十字花科,同水稻、玉米、高粱、谷子等禾本科植物的亲缘性较远。
2.4 AtCYS6-GFP蛋白的亚细胞定位
将AtCYS6-GFP融合蛋白表达载体转化拟南芥叶片原生质体,首先在大视野下观察GFP荧光信号情况,如图5-A所示,在大视野下,观察到许多荧光信号,表明AtCYS6-GFP荧光信号可以正常表达;然后在小视野下选取原生质体进行观察,如图5-B所示,AtCYS6-GFP在整个细胞质均有表达,这与预测的结果相符合。
2.5 融合蛋白的诱导、纯化及Western Blot分析
为了检测融合蛋白的表达情况,本研究首先进行了小量诱导表达试验,在不同时间诱导之后,进行凝胶电泳检测,其中融合蛋白分子量约为52 ku,GST蛋白分子量约为26 ku。检测结果如图6所示:在IPTG诱导下,对照组中诱导0 min时未检测到GST蛋白(泳道1),诱导210 min后,检测到大量GST蛋白(泳道2);试验组诱导0 min时未检测到融合蛋白(泳道3),随着诱导时间的延长,开始检测到融合蛋白的表达,时间逐渐延长,融合蛋白的表达量也逐渐增加,同时结果还发现诱导120 min和诱导210 min融合蛋白的表达量差别不大。
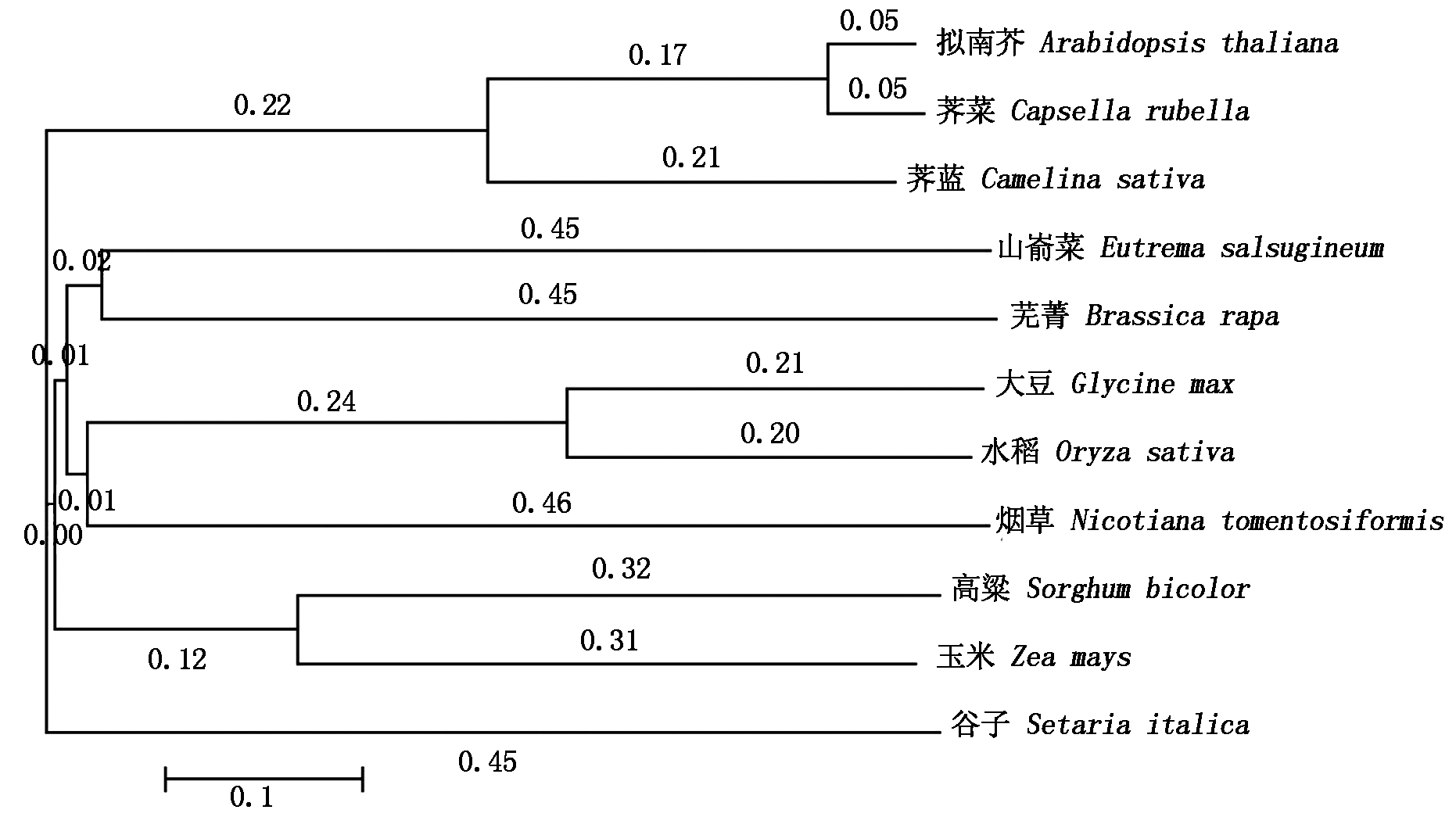
图4 系统进化树分析
Fig.4 Phylogenetic tree analysis
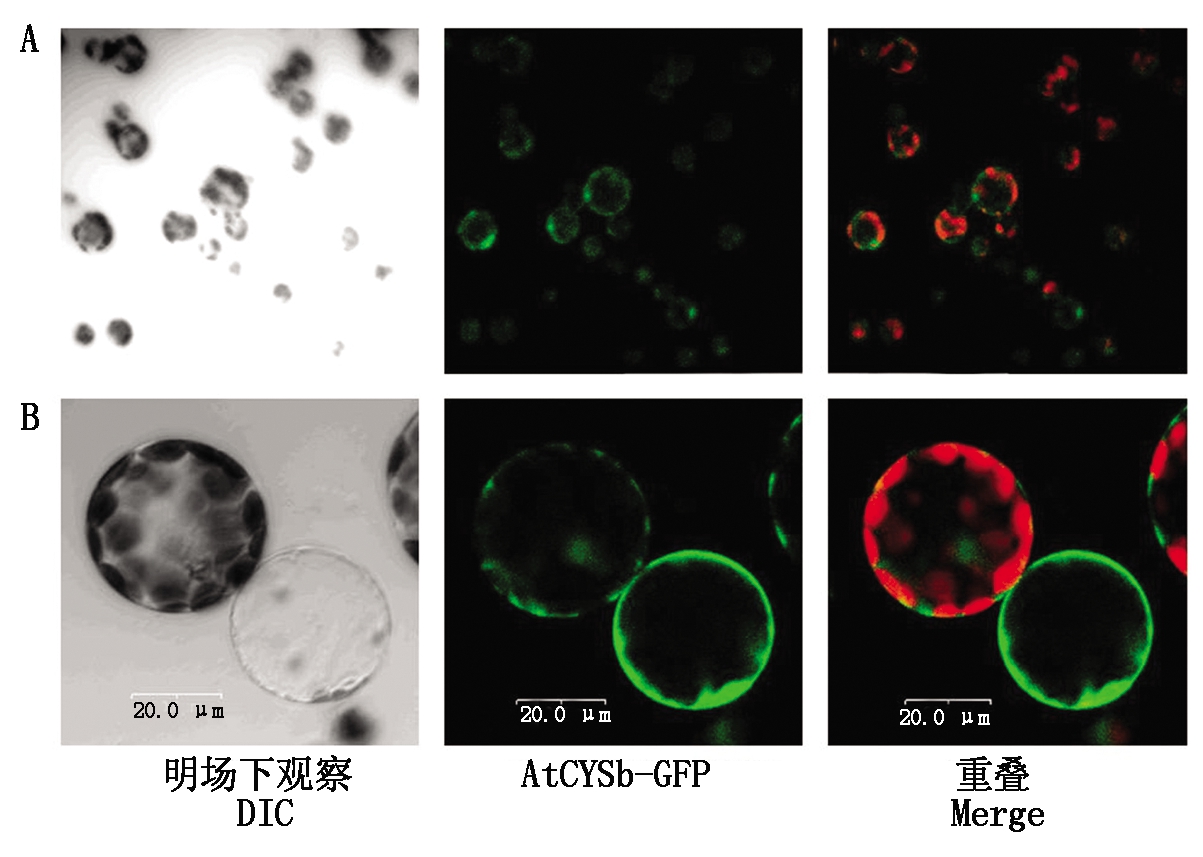
A.大视野下观察;B.小视野下观察。
A.Observation in the big view;B.Observation in the small view.
图5 AtCYS6-GFP亚细胞定位
Fig.5 Subcellular localization of AtCYS6-GFP
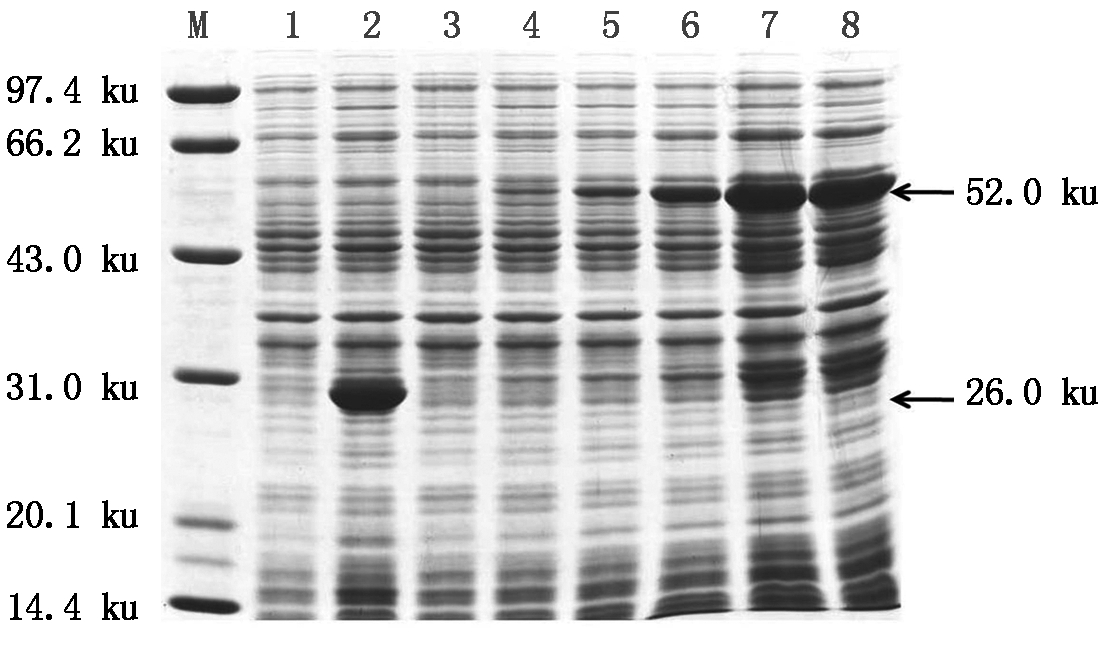
M.蛋白质Marker;1-2.对照组诱导0,210 min;
3-8.试验组诱导0,15,30,60,120,210 min。
M.Protein Marker;1-2.Control group induction for 0,210 minutes;
3-8.Experience group induction for 0,15,30,60,120,210 minutes.
图6 融合蛋白的小量诱导表达
Fig.6 Small amount of induced expression of fusion protein
根据小量诱导表达结果,本研究对融合蛋白进行大量诱导表达及纯化,结果如图7-A所示,得到了纯化的GST蛋白(泳道1)和GST-AtCYS6融合蛋白(泳道3);本研究进一步对GST-AtCYS6融合蛋白进行了Western Blot分析,如图7-B所示,检测到信号(泳道5)。这些结果说明,GST-AtCYS6融合蛋白正确翻译表达,另外纯化出来的蛋白也没有出现结构上的断裂或者降解的情况。
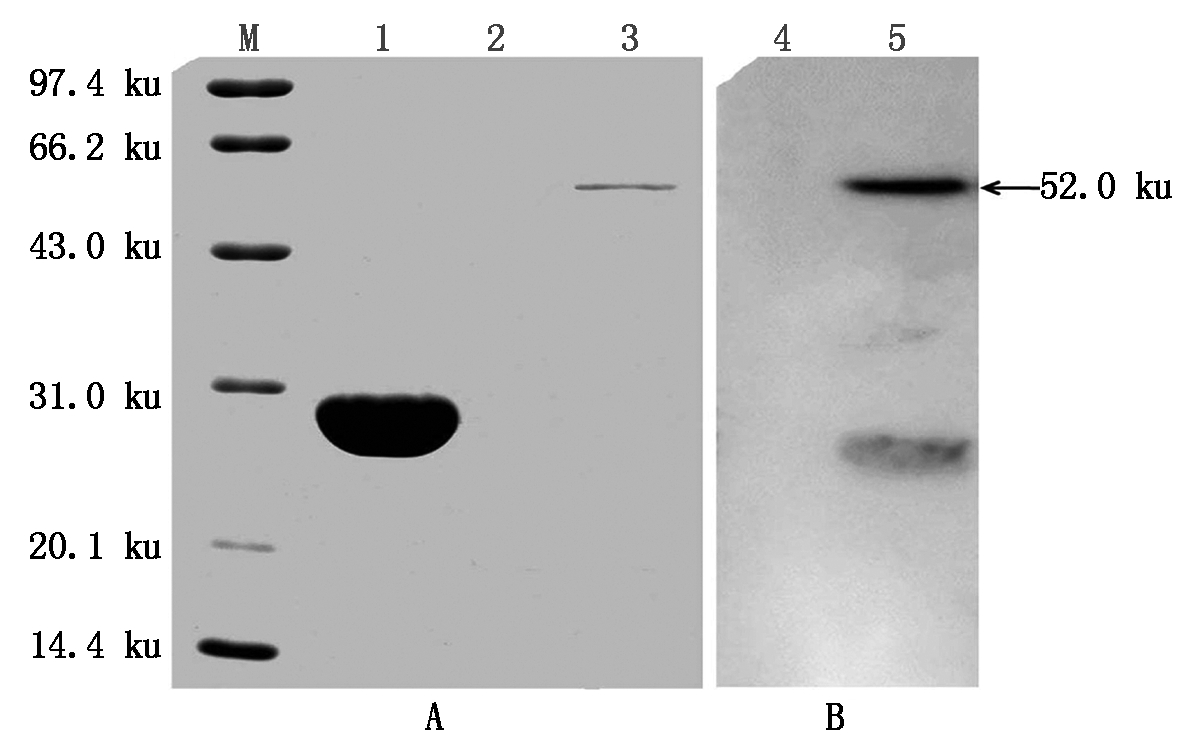
M.蛋白质Marker;1.纯化的GST蛋白;
2.空泳道;3.纯化的GST-AtCYSb蛋白;4-5.Western Blot分析。
M.Protein Marker;1.Purified GST protein;2.Empty lane;
3.Purified GST-AtCYSb protein;4-5.Western Blot analysis.
图7 GST、GST-AtCYSb蛋白纯化及Western Blot分析
Fig.7 GST,GST-AtCYSb protein purification and Western Blot analysis
2.6 融合蛋白酶活性分析
本研究对GST-AtCYS6融合蛋白的酶活性进行了检测分析,结果如图8所示:在没有添加融合蛋白的时候,酶活性抑制率为0,当添加融合蛋白后,木瓜蛋白酶的活性开始受到抑制,且随着蛋白浓度的升高,酶活性抑制率也逐渐升高,在2.5 μg/μL时,酶活性抑制率约为15.5%,当增加到5.0 μg/μL时,酶活性抑制率提高到27.9%。同时,在试验中本研究用纯化的GST蛋白作为对照,结果发现添加的GST蛋白对木瓜蛋白酶的活性没有抑制作用。
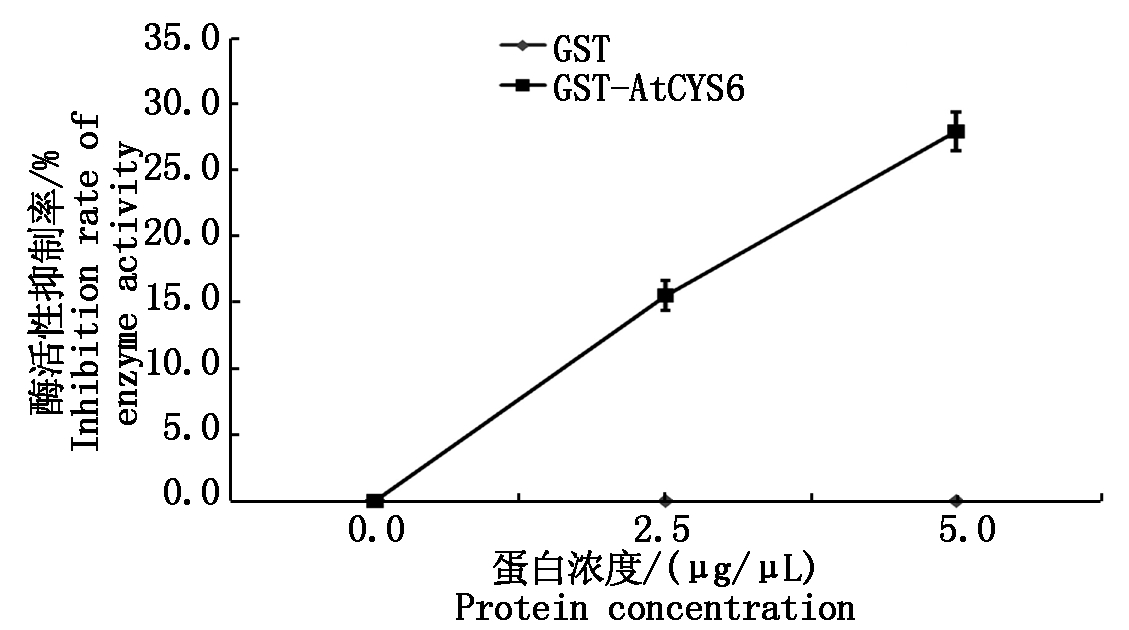
图8 融合蛋白酶活性分析
Fig.8 Enzyme activity analysis of fusion protein
3 结论与讨论
半胱氨酸蛋白酶抑制剂普遍存在于动植物和微生物体内,它在蛋白质水解、蛋白酶活性调节等许多生理过程中起着重要的作用[21-22],随着研究的进一步深入,人们发现半胱氨酸蛋白酶抑制剂在抗逆性[15-17]、植物程序性细胞死亡[23-24]和细胞增殖[25]等生命过程中也发挥着积极的作用。本研究从拟南芥中克隆了一种半胱氨酸蛋白酶抑制剂基因AtCYS6,序列比对结果表明它与十字花科的荠菜、荠蓝的同源性较高,同源性分别为81.1%,80.3%,同水稻、玉米和大豆的同源性较低,且同其他半胱氨酸蛋白酶抑制剂一样,在氨基酸序列中间具有高度保守的Q-V-G序列[3-4];预测AtCYS6氨基酸序列相对分子质量为26.293 95 ku,电泳结果表明,预测值较为准确;预测AtCYS6的等电点为5.85,具有一个跨膜结构域,含有信号肽的亲水性蛋白;蛋白质三维结构显示它包含2个部分,第一部分是由3个反向平行的β片层包裹着一个α-螺旋中心组成,第二部分是由5个反向平行的β片层包裹着一个α-螺旋中心组成;进化树分析表明,AtCYS6与同属于十字花科的荠菜、荠蓝的亲缘性较近,同水稻、玉米、高粱,谷子等禾本科植物的亲缘性较远;亚细胞定位结果表明其定位于细胞质,与预测结果相符合;蛋白诱导、纯化及Western Blot分析表明融合蛋白正确翻译表达,且没有出现结构上的断裂或者降解的情况;酶活性分析表明AtCYS6同一些报道一样,对木瓜蛋白酶具有抑制作用[26]。综上,AtCYS6在植物中具有重要的功能和调控作用,本研究结果为下一步在植物体内研究该基因的相关机理作用提供了基础。
[1] 王琛柱, 钦俊德. 植物蛋白酶抑制素抗虫作用研究进展[J]. 昆虫学报,1997,40(2):212-218. doi:10.16380/j.kcxb.1997.02.020.
Wang C Z,Qin J D. Protease inhibitors in plants contributing to resistance to insects: An overview[J].Acta Entomologica Sinica,1997,40(2):212-218.
[2] Ryan C A. Protease inhibitors in plants:Genes for improving defenses against insects and pathogens[J]. Annual Review of Phytopathology,1990,28:425-449. doi:10.1146/annurev.py.28.090190.002233.
[3] Barret A J. The cystatins:a new class of peptidase inhibitors[J]. Trends in Biochemical Sciences,1987,12:193-196. doi:10.1016/0968-0004(87)90092-2.
[4] Stubbs M T,Laber B,Bode W,Huber R,Jerala R,Lenarcic B,Turk V. The refined 2.4 A X-ray crystal structure of recombinant human stefin B in complex with the cysteine proteinase papain:a novel type of proteinase inhibitor interaction[J]. The EMBO Journal,1990,9(6):1939-1947. doi:10.1002/j.1460-2075.1990.tb08321.x.
[5] Margis R,Reis E M,Villeret V. Structural and phylogenetic relationships among plant and animal cystatins[J]. Archives of Biochemistry and Biophysics,1998,359(1):24-30. doi:10.1006/abbi.1998.0875.
[6] Kato H,Sutoh K,Minamikawa T. Identification,cDNA cloning and possible roles of seed-specific rice asparaginyl endopepti-dase,REP-2[J]. Planta,2003,217(4):676-685. doi:10.1007/s00425-003-1024-5.
[7] Kondo H,Abe K,Nishimura I,Watanabe H,Emori Y,Arai S. Two distinct cystatin species in rice seeds with different specificities against cysteine proteinases. Molecular cloning,expression,and biochemical studies on oryzacystatin-Ⅱ[J]. Journal of Biology Chemistry,1990,265(26):15832-15837. doi:10.5357/koubyou.57.12.
[8] Yamada T,Ohta H,Shinohara A,Iwamatsu A,Shimada H,Tsuchiya T,Masuda T,Takamiya K. A cysteine protease from maize isolated in a complex with cystatin[J]. Plant and Cell Physiology,2000,41(2):185-191. doi:10.1093/pcp/41.2.185.
[9] Hines M E,Osuala C I,Nielsen S S. Isolation and partial characterization of a soybean cystatin cysteine proteinase inhibitor of coleopteran digestive proteolytic activity[J]. Journal of Agricultural and Food Chemistry,1991,39(8):1515-1520. doi:10.1021/jf00008a030.
[10] Neuteboom L W,Matsumoto K O,Christopher D A. An extended AE-rich N-terminal trunk in secreted pineapple cystatin enhances inhibition of fruit bromelain and is posttranslationally removed during ripening[J].Plant Physiology,2009,151(2):515-527. doi:10.1104/pp.109.142232.
[11] Hong J K,Hwang J E,Lim C J,Yang K A,Jin Z L,Kim C Y,Koo J C,Chung W S,Lee K O,Lee S Y,Cho M J,Lim C O. Over-expression of Chinese cabbage phytocystatin 1 retards seed germination in Arabidopsis[J]. Plant Science,2007,172(3):556-563. doi:10.1016/j.plantsci.2006.11.005.
[12] Hwang J E,Hong J K,Je J H,Lee K O,Kim D Y,Lee S Y,Lim C O. Regulation of seed germination and seedling growth by an Arabidopsis phytocystatin isoform,AtCYS6[J]. Plant Cell Reports,2009,28(11):1623-1632. doi:10.1007/s00299-009-0762-7.
[13] Benchabane M,Schlüter U,Vorster J,Goulet M C,Michaud D. Plant cystatins[J]. Biochimie,2010,92(11):1657-1666. doi:10.1016/j.biochi.2010.06.006.
[14] Martinez M,Santamaria M E,Diaz-Mendoza M,Arnaiz A,Carrillo L,Ortego F,Diaz I. Phytocystatins:defense proteins against phytophagous insects and acari[J]. International Journal of Molecular Sciences,2016,17 (10):1747. doi:10.3390/ijms17101747.
[15] Martínez M,Abraham Z,Carbonero P,Díaz I. Comparative phylogenetic analysis of cystatin gene families from Arabidopsis,rice and barley[J]. Molecular Genetics and Genomics,2005,273 (5):423-432. doi:10.1007/s00438-005-1147-4.
[16] Belenghi B, Acconcia F, Trovato M, Perazzolli M, Bocedi A, Polticelli F, Ascenzi P, Delledonne M. AtCYS1,a cystatin from Arabidopsis thaliana,suppresses hypersensitive cell death[J]. European Journal of Biochemistry,2003,270(12):2593-2604. doi:10.1046/j.1432-1033.2003.03630.x.
[17] Zhang X X,Liu S K,Takano T. Two cysteine proteinase inhibitors from Arabidopsis thaliana,AtCYSa and AtCYSb,increasing the salt,drought,oxidation and cold tolerance[J]. Plant Molecular Biology,2008,68(1):131-143. doi:10.1007/s11103-008-9357-x.
[18] Hwang J E,Hong J K,Chan J L,Chen H,Je J Y,Yang K A,Kim D Y,Choi Y J,Lee S Y,Lim C O. Distinct expression patterns of two Arabidopsis phytocystatin genes,AtCYS1 and AtCYS2,during development and abiotic stresses[J]. Plant Cell Reports,2010,29(8):905-915. doi:10.1007/s00299-010-0876-y.
[19] Labudda M,Ró![]() ńska E,Szewińska J,Sobcak M,Dzik J M. Protease activity and phytocystatin expression in Arabidopsis thaliana upon Heterodera schachtii infection[J]. Plant Physiology and Biochemistry,2016,109:416-429. doi:10.1016/j.plaphy.2016.10.021.
ńska E,Szewińska J,Sobcak M,Dzik J M. Protease activity and phytocystatin expression in Arabidopsis thaliana upon Heterodera schachtii infection[J]. Plant Physiology and Biochemistry,2016,109:416-429. doi:10.1016/j.plaphy.2016.10.021.
[20] Song C,Kim T,Chung W S,Lim C O. The Arabidopsis phytocystatin AtCYS5 enhances seed germination and seedling growth under heat stress conditions[J]. Molecules and Cells,2017,40(8):577-586. doi:10.14348/molcells.2017.0075.
[21] Kiggundu A,Muchwezi J,Vyver C V,Viljoen A,Vorster J,Schlüter U,Kunert K,Michaud D. Deleterious effects of plant cystatins against the banana weevil Cosmopolites sordidus[J]. Archives of Insect Biochemistry and Physiology,2010,73(2):87-105. doi:10.1002/arch.20342.
[22] Yang A H,Yeh K W. Molecular cloning,recombinant gene expression,and antifungal activity of cystatin from taro (Colocasia esculenta cv. Kaosiung No. 1)[J]. Planta,2005,221(4):493-501. doi:10.1007/s00425-004-1462-8.
[23] Solomon M,Belenghi B,Delledonne M,Menachem E,Levine A. The involvement of cysteine proteases and protease inhibitor genes in the regulation of programmed cell death in plants[J]. The Plant Cell,1999,11(3):431-443. doi:10.1105/tpc.11.3.431.
[24] Gilroy E M,Hein I,Hoorn R,Boevink P C,Venter E,McLellan H,Kaffarnik F,Hrubikova K,Shaw J,Holeva M,López E C,Borras-Hidalgo O,Pritchard L,Loake G J,Lacomme C,Birch P R J. Involvement of cathepsin B in the plant disease resistance hypersensitive response[J]. The Plant Journal,2007,52(1):1-13. doi:10.1111/j.1365-313x.2007.03226.x.
[25] Tian L H,Zhang L,Zhang J,Song Y,Guo Y. Differential proteomic analysis of soluble extracellular proteins reveals the cysteine protease and cystatin involved in suspension-cultured cell proliferation in rice[J]. Biochimica et Biophysica Acta,2009,1794(3):459-467. doi:10.1016/j.bbapap.2008.11.023.
[26] Christova P K,Christov N K,Imai R. A cold inducible multidomain cystatin from winter wheat inhibits growth of the snow mold fungus Microdochium nivale[J]. Planta,2006,223(6):1207-1218. doi:10.1007/s00425-005-0169-9.
