抗坏血酸 (Ascorbic acid,AsA) 又称维生素C (Vitamin C,Vc) ,1932年首次从植物和哺乳动物来源中分离出来[1-2]。植物体内广泛存在的高丰度小分子物质,在高等植物的不同细胞中,都含有一定浓度的AsA,例如线粒体和叶绿体为10~300 mmol/L。AsA是植物体内不可缺少的辅酶和抗氧化剂[3],与植物生长发育代谢[4]、植物对逆境胁迫的抗性[5-7]以及参与乙烯和赤霉素等一些植物激素的生物合成密切相关[8]。由于人体内无法合成AsA[9],植物中天然的AsA与其他植化成分的互作效应,是人类最好的AsA来源[10-11]。
目前研究中,L-半乳糖途径 (Smirnoff-Wheeler途径) 是高等植物中最早被发现和公认的AsA生物合成主要途径[12-13]。在该途径中,GDP-甘露糖-3′,5′-差向异构酶 (GDP-mannose-3′,5′-epimerase,GME)分别在2个方向进行着反应,分别形成GDP-L-半乳糖和GDP-L-古洛糖[14],这是在糖核苷水平上植物抗坏血酸生物合成的第一步。GME已从拟南芥中纯化和克隆[15]。GME酶含有NAD结合域,因此,被氧化的烟酰胺核苷酸(NAD+和NADP+)催化,被其还原形式(NADH和NADPH)抑制,将酶活性与细胞的氧化还原状态联系起来。该酶活性还受GDP1,GDP-D-海藻糖、AsA抑制[16]。此外,Wolucka等[17]在纯化GME试验中,重组和天然酶都与热激蛋白Hsp70(分别为拟南芥Hsc70.3和大肠杆菌His和GST)发生共纯化现象,推测拟南芥Hsp70蛋白可能参与GME的折叠与调节。Valpuesta等[18]认为热激蛋白Hsp70与GME的共纯化在一定程度上说明这种相互作用是植物在逆境条件下的一种调控机制。但如何调节GME活性未见报道。GME属于短链脱氢酶/还原酶家族中的一个成员。Running等[19]在拟南芥细胞悬浮培养试验证明了抗坏血酸合成的“瓶颈”刚好发生在D-甘露糖和L-半乳糖之间,由于具有较低的Vmax值,该酶又被认为是抗坏血酸生物合成的限速酶。Wolucka等[20]通过茉莉酸甲酯调控GME转录水平,进而使拟南芥和烟草BY-2悬浮细胞中AsA含量增加。Watanabe等[21]对水稻GME基因OsGME进行研究,发现水稻GME基因与拟南芥GME基因氨基酸序列同源性达到91%,同样含有NAD结合域。Du等[22]对水稻进行低温诱导发现,GME受低温诱导。OsGME1、OsGME2在根茎叶中表达量都很高,但是在发育的种子中表达量很低,而且GA可能通过调控GME转录来影响AsA含量。同样在Cui等[23]的试验中也有同样发现,在寒冷胁迫的水稻幼苗中GME含量积累增加。Zhang等[24]在番茄中过表达SlGME1和SlGME2基因后发现,转基因植株体内抗坏血酸含量显著提高,且对环境胁迫的抵御能力也有着一定程度增加。Ma等[25]在研究胁迫对拟南芥影响时发现,植株体内过表达GME基因有助于其对抗氧化能力的提升。Liu等[26]发现蓝莓中GME基因的高表达使得果实中AsA含量上升。邹礼平[27]利用RFLP方法将GME2基因定位在9-J区域。Stevens等[28]对3个不同番茄种群利用QTL对番茄果实AsA进行定位,发现3个群体有共同的QTL位点9-J,说明GME2与抗坏血酸含量有密切关系。作为AsA生物合成中的限速酶,GME基因的表达将是调节AsA水平的良好候选者。
目前,AsA生物合成机制主要集中在拟南芥[29]和烟草[30]等模式植物中,在辣椒中还尚未有关于GME基因的研究报道。本研究报道了2种辣椒GME基因,分别命名为CaGME1和CaGME2。通过对辣椒GME基因家族生物信息学分析以及不同胁迫处理下GME基因表达特性研究,为辣椒逆境胁迫机制和GME基因的特征研究提供一定的参考依据。
1 材料和方法
1.1 试验材料
以甘肃省农业科学院蔬菜研究所选育地方日光温室主栽品种陇椒5号为供试材料。各差异胁迫处理在辣椒幼苗期真叶生长有6片以上展开叶时进行。
胁迫处理:采用100 μmol/L赤霉素 (Gibberellin,GA3) 、200 μmol/L茉莉酸甲酯 (Methyl jasmona,MeJA) 和300 mmol/L NaCl分别喷洒辣椒叶片的正反面,以喷洒超纯水为对照;对辣椒叶片进行低温 (Cold) 处理,光照培养箱的温度降至4 ℃ (昼) /0 ℃ (夜) ;对照组白天温度设置为28 ℃,晚上设置为26 ℃[31]。各种胁迫处理下的辣椒植株在光照强度为200 μmol/(m2·s) 的气候室中进行培养,光照周期分别为12 h的光照和12 h的暗处理。
1.2 试验方法
1.2.1 总RNA的提取与cDNA的合成 总RNA的提取:辣椒幼苗期真叶中总RNA的提取按照E.Z.N.A Plant RNA Kit试剂盒的说明书进行。
RNA逆转录为cDNA的步骤:以mRNA为模板,使用试剂盒为TaKaRa的PrimeScriptTM RT reagent Kit with gDNA Eraser (Perfect Real Time) ,使用过程中严格按照试剂盒说明进行。
1.2.2 辣椒GME家族基因成员的检索 根据拟南芥基因组数据库( https://www.arabidopsis.org)中获得的GME基因的编码序列,以获得的CDS为目标序列,利用茄科植物基因组数据库(https://solgenomics.net)中的BlastN程序对该序列进行搜索,获取该片段序列在茄科植物基因组数据库中相应序列的基因号、CDS及氨基酸序列,以确定其为GME基因家族的成员。
1.2.3 辣椒GME家族基因生物信息学分析 辣椒GME基因的各种理化特性通过ProtParam[32](http://expasy.org/tools/protparam.html)在线计算。SignalP 4.1 Server 分析信号肽;ProtScale分析亲水性/疏水性;TMHMM对蛋白的跨膜结构进行分析预测。WoLF PSORT(http://psort.ims.u-tokyo.ac.jp/form.html)对亚细胞定位分析[33]。MEME进行全长蛋白保守结构域的预测。利用FGENESH-C[34]对得到的目的序列进行内含子/外显子结构框架分析。采用Prabi在线对GME基因的二级结构特性进行预测研究。通过PlantCARE软件对辣椒GME基因的启动子进行分析整理。MEGA 5.0[35]和ClustalX[36]软件对辣椒GME蛋白进行分析比对绘制系统发育树。
1.2.4 辣椒GME家族基因的qRT-PCR表达分析 提取辣椒幼苗期叶片RNA;使用反转录试剂盒除DNA纯化以及反转录为cDNA;利用Primer premier 5.0设计引物,以辣椒Actin基因为内参,应用Bio-Rad iCycler iQ实时定量PCR仪,以不同胁迫处理下辣椒幼苗期叶片cDNA为材料,对不同胁迫处理辣椒GME基因家族进行特异性表达分析。
qRT-PCR 反应体系:含12.5 μL的SYBR Green Supermix,1.5 μL的 cDNA,0.5 μL的 10 μmol/L目的基因上下游引物,10.5 μL 的双蒸水,体系总共 25 μL。反应程序:95 ℃ 30 s;95 ℃ 5 s,60 ℃ 34 s,95 ℃ 15 s,60 ℃ 60 s,95 ℃ 15 s,共40个循环。相对表达量采用2-ΔΔCT法计算[36]。
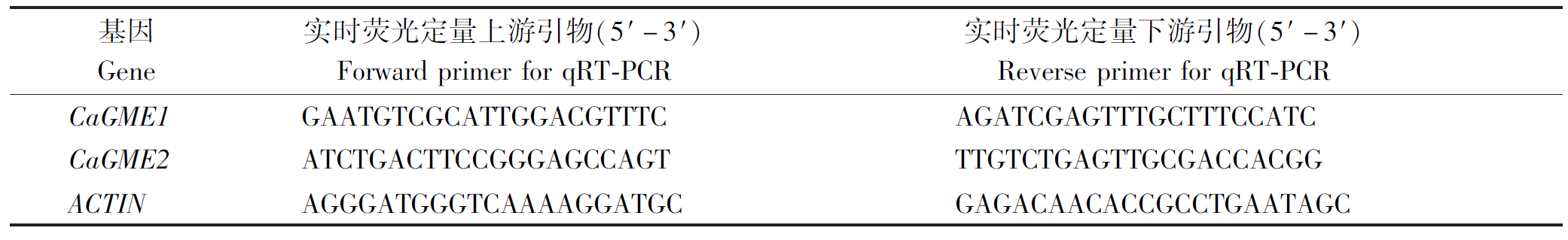
荧光定量PCR所用引物序列见表1。
表1 辣椒GME基因家族表达分析的实时荧光定量引物
Tab.1 qRT-PCR primers for expression on analysis of GME
1.2.5 统计分析与作图 所有试验均设3次生物重复,数据测定结果采用SPSS软件Duncan多重比较法进行方差分析,采用Excel作图。
2 结果与分析
2.1 辣椒GME基因家族信息及蛋白理化性质分析
利 用 ProtParam 对2个CaGMEs基因家族成员进行分析,结果表明:CaGME1和CaGME2基因分别定位于第3和第8染色体上,基因大小分别为4 170,2 730 bp,分别编码376,375 aa,对应的蛋白相对分子质量分别为42 544.35,42 378.23 ku,等电点分别为6.05和6.04,均分别有6个外显子(表2)。
表2 辣椒GME基因家族信息及理化性质
Tab.2 Physical and chemical property of CaGME
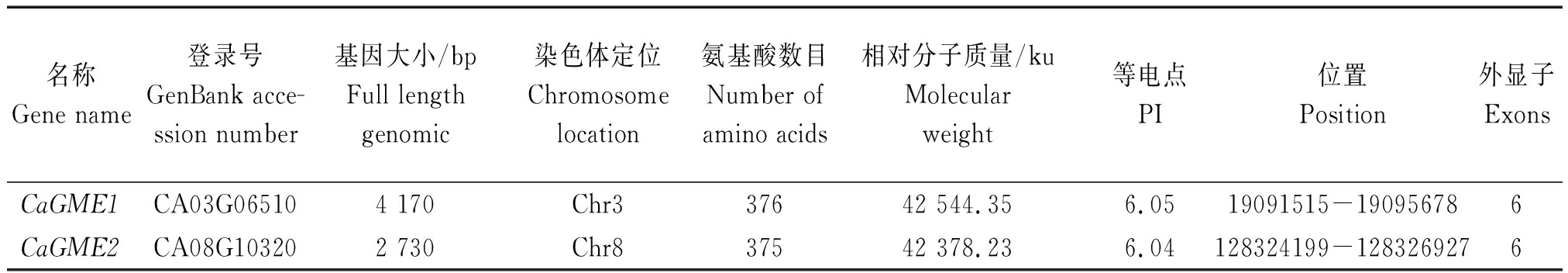
名称Gene name 登录号GenBank acce-ssion number 基因大小/bpFull lengthgenomic染色体定位Chromosomelocation氨基酸数目Number ofamino acids相对分子质量/kuMolecularweight等电点PI位置Position外显子ExonsCaGME1CA03G065104 170Chr337642 544.356.0519091515-190956786CaGME2CA08G103202 730Chr837542 378.236.04128324199-1283269276
2.2 辣椒GME蛋白的亲水性/疏水性、跨膜结构与亚细胞定位
通过SignalP 4.1 Server软件对CaGME蛋白所含有的信号肽预测分析发现,2个GME蛋白均不具有信号肽,因此,这2个蛋白都不属于分泌蛋白类型。
利用ProtScal软件对2个CaGMEs蛋白成员进行亲水性/疏水性的预测,结果(图1)表明,CaGME1和CaGME2中多肽链不同位置(CaGME1为64和36位,CaGME2为63和35位)出现亲水性和疏水性最强的氨基酸都相同分别是精氨酸(Arg)和异亮氨酸(Ile)且分值大小相近,亲水性氨基酸在整条肽链中分布较整体来说相对均匀,且数目远大于疏水性氨基酸,故推测CaGME蛋白为亲水性蛋白。这与根据平均疏水值预测的结果一致(CaGME1为-0.464, CaGME2为-0.430)。说明GME整条多肽链表现为亲水性,但是没有明显的亲水区域。
使用TMHMM对CaGMEs基因家族成员进行跨膜结构预测得到这2个GME蛋白没有明显的跨膜结构,都不属于跨膜蛋白。
对CaGMEs蛋白亚细胞定位预测表明(表3),CaGME1和CaGME2在细胞核和细胞质粒中均有表达,CaGME2在细胞质中也有表达。这2个蛋白在细胞质粒中的得分最高,推测定位在细胞质粒的可能性最高。

图1 辣椒GME蛋白亲水性/疏水性分析
Fig.1 Hydrophilic/hydrophobic analysis of pepper GME protein
表3 辣椒GME蛋白的亚细胞定位
Tab.3 Subcellular location prediction of GME proteins in pepper

名称Name细胞膜Cellmembrane内质网Endoplasmicreticulum线粒体Mitochondria液泡Vacuole细胞质Cytoplasm过氧化体Peroxide细胞核Nucleus细胞基质Cell matrix细胞质粒Cell plasmid叶绿体ChloroplastCaGME1------(1)-(13)-CaGME2----(1)-(1)-(12)-
注:括号内的数值代表预测的最后得分。
Note:The numbers in parenthesis mean the integral prediction scores of protein location.
2.3 辣椒GME基因家族保守结构域分析
利用MEME软件对CaGMEs保守结构域进行预测,该蛋白属于NADB Rossmann 超级家族,GME基因家族的2个基因所包含的氨基酸保守序列都一致,都有着8个结构域;N端主要是motif5,该氨基酸保守域结构含有NAD binding,在序列中高度保守。C端主要是motif7,含有扩展SDR具有典型的Rossmann折叠,是底物结合位点(图2)。对每个结构域进行分析(图3)发现,motif1、motif3、motif5均含有50个保守氨基酸,motif7和motif8含保守氨基酸较少,分别有29,19个。
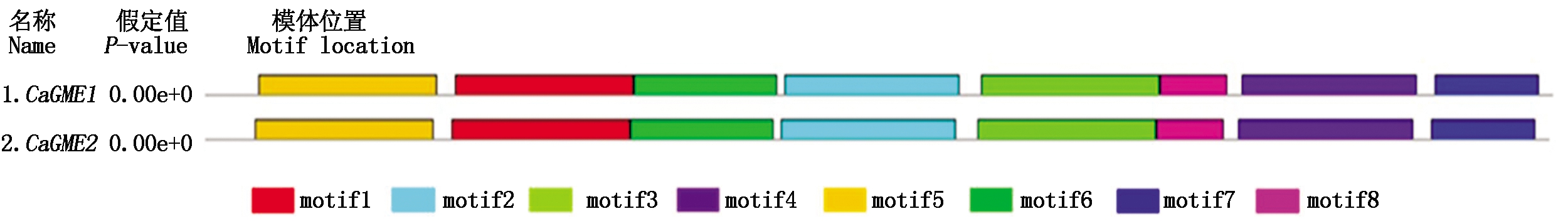
图2 辣椒GME基因家族motif分析
Fig.2 GME gene family motif analysis in pepper

图3 MEME预测的8个保守位点LOCO
Fig.3 LOCO of 8 conserved motif of domain
2.4 辣椒GME基因结构分析
利用FGENESH-C 对获得的辣椒GME基因的目的序列进行内含子/外显子结构框架分析。结果表明(图4),CaGMEs基因家族成员都含有上游和下游基因序列,基因结构完整。2个基因在长度上存在明显差别外,其内含子和外显子在数目上保持一致,在结构框架中都包含有6个外显子和5个内含子,基因结构相似。
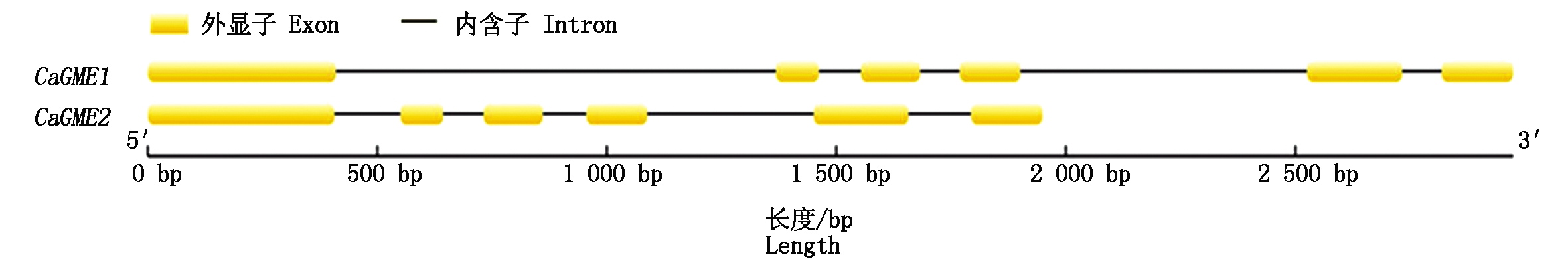
图4 辣椒GME基因结构
Fig.4 Gene structures of GME in pepper
2.5 辣椒GME蛋白二级结构对比分析
通过Prabi在线对CaGMEs基因所编码的蛋白二级结构预测分析,结果表明(表4),辣椒的2个CaGMEs蛋白质二级结构内容包含较完整均有α-螺旋、β-转角、延伸链、不规则卷曲,且不规则卷曲 (Random coil) >α-螺旋 (Alpha helix) >延伸链(Extended strand)>β-转角 (Beta turn)。CaGMEs的二级结构主要以不规则卷曲 (Random coil)和α-螺旋 (Alpha helix)为主。
表4 辣椒GME蛋白二级结构对比分析
Tab.4 The secondary structure of GME protein sequence in pepper %

名称Nameα-螺旋Alpha helixβ-转角Beta turn延伸链Extended strand不规则卷曲Random coilCaGME136.706.3817.0239.89CaGME238.136.4014.4041.07
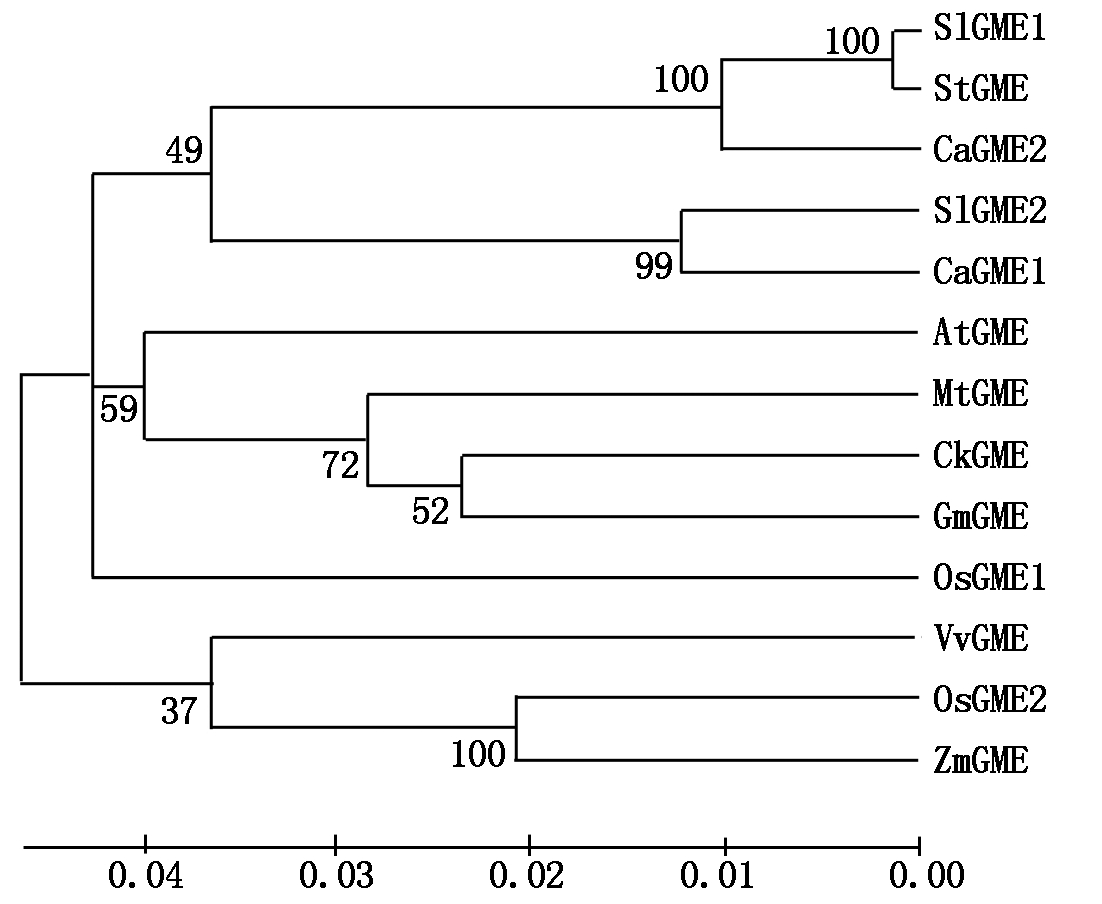
Ca.辣椒;At.拟南芥;Os.水稻;Sl.番茄;Zm.玉米;Vv.葡萄;Gm.大豆;Ck.柠条;Mt.截型苜蓿;St.马铃薯。
Ca.Capsicum annuum L.;At.Arabidopsis thaliana;Os.Oryza sativa L.;Sl.Solanum lycopersicum;Zm.Zea mays L.;Vv.Vitis vinifera L.;Gm.Glycine max(Linn.)Merr.;Ck.Caragana korshinskii Kom;Mt.Medicago sativa;St.Solanum tuberosum.
图5 多物种GME家族蛋白序列的系统发育树
Fig.5 Neighbor-joining phylogenetic tree of the GME family genes in multi-species
2.6 辣椒GME基因顺式作用元件分析
为深度解析CaGME家族成员的表达调控模式,将每条CaGME起始密码子ATG上游3 000 bp作为启动子区,通过采用PlantCare软件对上游区域启动子序列的相关顺式调控元件进行研究分析,进而鉴别到多种对环境因子和植株生长激素类信号物质能够形成应激响应的顺势元件,结果表明,CaGME表达可能受到多种因素制约影响进而使得其表达调控机制变得复杂化,极有可能受到植株体内和外界环境因素的双重应激调节。结果(表5)显示,2条CaGME具有的顺式作用元件主要分为植物激素响应元件,如脱落酸响应元件 (ABRE)、生长素响应元件 (TGA-element)、茉莉酸甲酯响应元件 (CGTCA-motif)、乙烯响应元件(ERE)、赤霉素响应元件 (P-box)等;植物生长发育相关顺式作用元件,如O2-site、CAT-box等;生物和非生物胁迫响应顺式作用元件,如干旱诱导元件 (MBS)、机械损伤信号响应元件(WUN-motif)、低温响应元件(LTR)、光响应元件(ACE)等。预测结果表明,辣椒CaGME在调节生长发育、激素信号物质和应对环境胁迫方面有重要作用。
2.7 辣椒GME蛋白系统发育树分析
为了揭示辣椒GME基因家族的进化关系,以拟南芥、水稻、番茄、玉米、葡萄、大豆、柠条、截型苜蓿、马铃薯GME家族基因作为参考。利用MEGA 5.0和ClustalX构建系统发育树。结果表明(图5),根据亲缘关系将该蛋白分为2个分支,其中SlGME1、SlGME2、StGME、CaGME1、CaGME2、AtGME、MtGME、CkGME、GmGME、OsGME1为一支,其余的为另一支。CaGME2与SlGME1和StGME的亲缘关系最近,其次是CaGME1与SlGME2。CaGME1和CaGME2在同一个分支当中,说明这2种蛋白质可能在辣椒中起到相似作用。
表5 辣椒GME家族成员启动子序列顺式作用元件分析
Tab.5 Analysis of cis-acting elements of promoter sequence of pepper GME family members

2.8 辣椒GME基因的表达分析
利用qRT-PCR分析了辣椒GME家族基因在逆境胁迫条件下的表达情况(图6),包括盐胁迫 (NaCl)、茉莉酸甲酯 (MeJA)、赤霉素(GA3)和冷胁迫 (Cold)。CaGME1在冷、盐胁迫和MeJA处理下,基因表达量上调且呈先上升后下降的趋势,分别于12,12,6 h时达到最大值,其中冷、盐胁迫上调表达为0 h的1.8,1.6倍左右;但在GA3胁迫下,表达量下调且呈先下降后上升的趋势,12 h时达到最小值,表达量下降到0 h的30%左右。CaGME2在胁迫环境下表达情况与CaGME1相似。
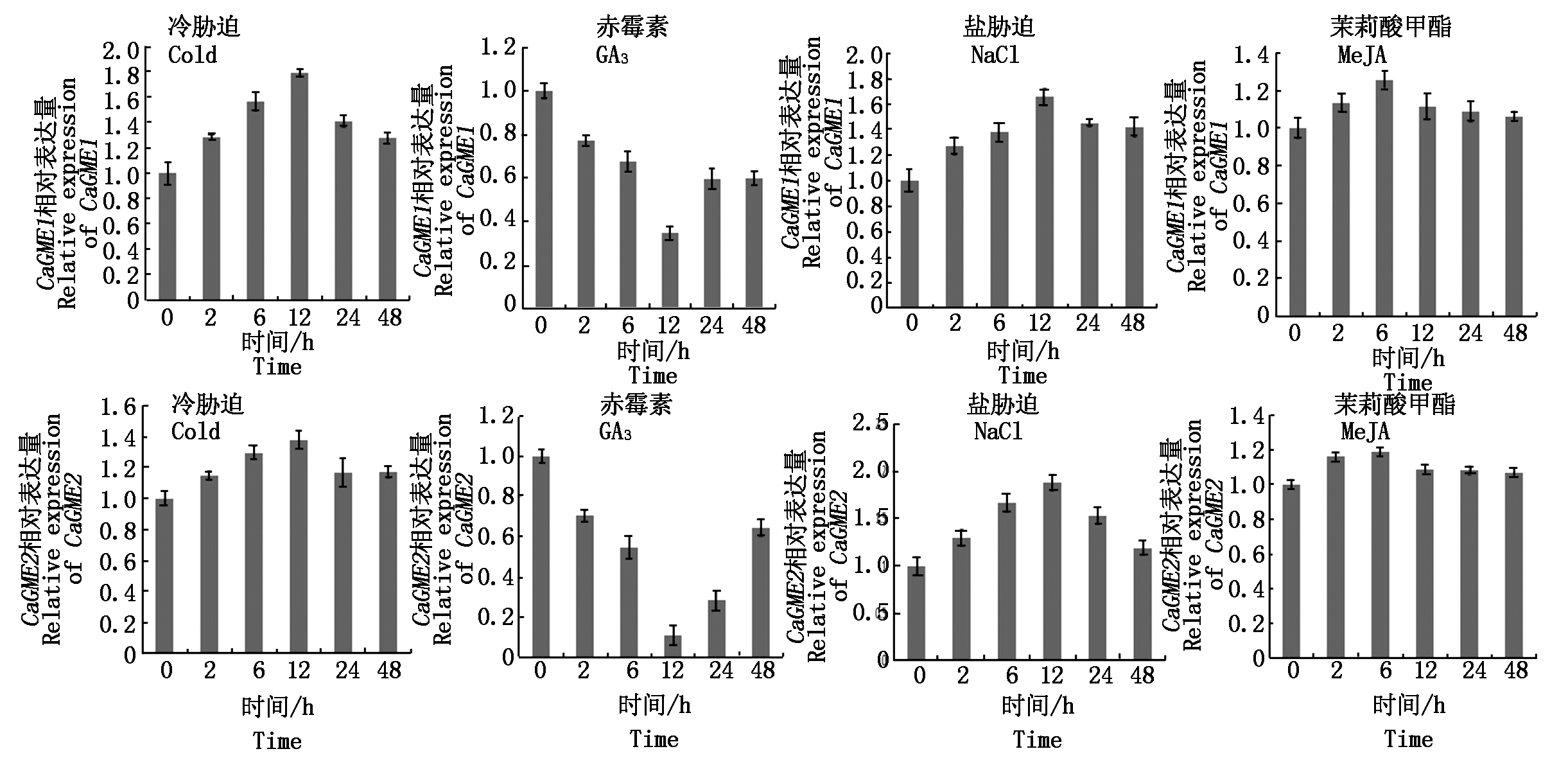
图6 辣椒GME基因对生物非生物胁迫响应
Fig.6 Response of pepper GME gene to abiotic stress
3 结论与讨论
目前,对GME基因家族有许多研究,在水稻[22]、棉花[37]、茶树[38]、番茄[39]等植物中有均有报道;Voxeur等[40]研究表明,GME不仅是L-半乳糖途径中Vc合成的关键限速酶,而且植物还会因GME失活导致细胞的分裂减慢和发育迟缓。Major等[41]研究表明,GME可以差向异构化,生成GDP-L-半乳糖和GDP-L-古洛糖。AsA生物合成是在GME水平上进行的。关于植物体内如何去分配和调节GME而控制AsA的合成,目前没有详细报道。
本研究通过生物信息学方法,对辣椒中得到的2个GME基因进行鉴定与表达分析,2个GME基因序列有较高的同源性,属于NADB Rossmann 超级家族,同时含有NADB结构域、NAD-dependent epimerase/dehydratase结构域和底物特异性结合位点,且在功能域上都是高度保守的。Watanabe等[21]对水稻GME基因OsGME进行研究,同样含有NAD结合域。2个GME基因的功能特征在理化性质、亲疏水性和二级结构等多个方面均高度相似,在发育树中处于同一分支,可能这2个基因在调控辣椒AsA合成中起到相似作用;启动子顺势元件分析中表明,辣椒CaGME在调节生长发育、激素信号和应对环境胁迫方面有重要作用。
本试验研究表明,在盐胁迫、冷胁迫和MeJA处理后,CaGME1和CaGME2在辣椒叶片中的相对表达量均呈上调趋势,这与Du等[22]和王美珍等[42]的研究结果基本相似;在GA3处理后,CaGME1和CaGME2在辣椒叶片中的相对表达量均呈下调趋势,这与王江英等[37]在对棉花进行GA3处理后发现,GME基因的表达量显著下降,及Du等[22]对水稻的GME基因GA3处理3 h后,发现OsGME1和OsGME2基因的表达量都下降的结果基本一致。
通过对辣椒GME基因家族的生物信息学分析和胁迫下基因表达特性的研究,可以为辣椒CaGME基因特征的探索以及在非生物胁迫下调节机制的下一步研究提供基础。
[1] Smirnoff N. Ascorbic acid:Metabolism and functions of amulti-facetted molecule[J]. Curr Opin Plant Biol,2000,3 (3):229-235. doi:10.1016/S1369-5266(00)80070-9.
[2] Zilva S S, Sc D. The isolation and identification of vitamin C[J]. Archives of Disease in Childhood,1935,10(58):253-264. doi: 10.1136/adc.10.58.253.
[3] Hancock R D,Viola R.Biosynthesis and catabolism of L-ascorbic acid in plants[J].Critical Review in Plant Sciences,2005,24(3):167-188. doi:10.1080/07352680591002165.
[4] Potters G,Gara L D,Asard H,Horemans N. Ascorbate and glutathione:Guardians of the cell cycle,partners in crime?[J]. Plant Physiology and Biochemistry,2002,40(6-8):537-548. doi:10.1016/S0981-9428(02)01414-6.
[5] Pavet V,Olmos E,Kiddle G,Mowla S,Kumar S,Antoniw J,Alvarez M E,Foyer C H. Ascorbic acid deficiency activates cell death and disease resistance responses in Arabidopsis[J].Plant Physiol,2005,139:1291-1303.doi:10.1104/pp.105.067686.
[6] Garnczarska M. Response of the ascorbate-glutathione cycle to re-aeration following hypoxia in lupine roots[J].Plant Physiol Biochem,2005,43(6):583-590. doi:10.1016/j.plaphy.2005.05.003.
[7] Yabuta Y,Mieda T,Rapolu M,Nakamura A,Motoki T,Maruta T,Yoshimura K,Ishikawa T,Shigeoka S. Light regulation of ascorbate biosynthesis is dependent on the photosynthetic electron transport chain but independent of sugars in Arabidopsis[J]. Journal of Experimental Botany,2007,58(10):2661-2671. doi:10.1093/jxb/erm124.
[8] Smirnoff N,Wheeler G L. Ascorbic acid in plants:Biosynthesis and function[J]. Crit Rev Biochem Mol Biol,2000,35 (4):291-314. doi:10.1080/10409230008984166.
[9] Smirnoff N. L-ascorbic acid biosynthesis[J].Vitam Horm,2001,61:241-266.doi:10.1016/S0083-6729(01)61008-2.
[10] Li Y,Schellhorn H E. New developments and novel therapeutic perspectives for vitamin C[J].The Journal of Nutrition,2007,137(10):2171-2184. doi:10.1093/jn/137.10.2171.
[11] Davey M W,Montagu M V,Inzé D,Sanmartin M,Kanellis A,Smiroff N,Benzie I J J,Strain J J,Favell D,Fletcher J. Plant L-ascorbic acid:Chemistry,function,metabolism,bioavailability and effects of processing[J]. Journal of the Science of Food and Agriculture,2000,80(7):825-860.doi:10.1002/(sici)1097-0010(20000515)80:7<825:aid-jsfa598>3.0.co;2-6.
[12] Wheeler G L,Jones M A,Smirnoff N. The biosynthetic pathway of vitamin C in higher plants[J].Nature,1998,393:365-369. doi:10.1038/30728.
[13] Linster C L,Clarke S G.L-ascorbate biosynthesis in higher plants:The role of VTC2[J].Trends in Plant Science,2008,13(11):567-573. doi:10.1016/j.tplants.2008.08.005.
[14] Wolucka B A,Montage M V. GDP-mannose 3′,5′-epimerase forms GDP-L-gulose,aputative intemnediate for the de novo biosynthesis of vitamin C in plants[J]. J Biol Chem 3,2003,278:47483-47490. doi:10.1074/jbc.M309135200.
[15] Wolucka B A,Persiau G, Doorsselaere J V ,Davey M W Demol H,Vandekerckhove J,Montagu M V,Zabeau M,Boerjan W. Partial purification and identify cation of GDP-mannose 3′,5′-epimerase of Arabidopsis thaliana,a key enzyme of the plant vitamin C pathway[J]. Proc Natl Acad Sci USA,2001,98(26):14843-14848. doi:10.1073/pnas.011578198.
[16] Ishikawal T,Dowdle J,Smirnoff N. Progress in manipulating ascorbic acid biosynthesis and accumulation in plants[J]. Physiologia Plant Arum,2006,126(3):343-355.doi: 10.1111/j.1399-3054.2006.00640.x.
[17] Wolucka B A,Van Montagu M. GDP-mannose 3′,5′-epimerase forms GDP-L-gulose,a putative intermediate for the de novo biosynthesis of vitamin C in plants[J]. J Biol Chem,2003,278(48):47483-47490. doi:10.1074/jbc.M309135200.
[18] Valpuesta V,Botella M A. Biosynthesis of L-ascorbic acid in plants:New pathways for an old antioxidant[J]. Trends Plant Sci,2004,9 (12):573-577. doi:10.1016/j.tplants.2004.10.002.
[19] Running J A,Burlingame R P,Berry A. The pathway of L-ascorbic acid biosynthesis in the colourless microalga Prototheca moriformis[J]. Journal of Experimental Botany,2003,54(389):1841-1849. doi:10.1093/jxb/erg207.
[20] Wolucka B A,Goossens A,Inzé D.Methyl jasmonate stimulates the de novo biosynthesis of vitamin C in plant cell suspensions[J]. Journal of Experimental Botany,2005,56(419):2527-2538. doi:10.1093/jxb/eri246.
[21] Watanabe K,Suzuki K,Kitamura S. Characterization of a GDP-D-mannose 3′,5′-epimerase from rice[J]. Phytochemistry,2006,67(4):338-346.doi:10.1016/j.phytochem.2005.12.003.
[22] Du Y,Yao Y,Liu J Y. Molecular characterization of two rice cDNAs encoding GDP-Mannose-3′,5′-Epimerase and their expression patterns[J]. Progress in Biochemistry and Biophysics,2006,33(4):368-376.
[23] Cui S X,Huang F,Wang J,Ma X,Cheng Y S,Liu J Y. A proteomic analysis of cold stress responses in rice seedlings[J].Proteomics,2005,5(12):3162-3172. doi:10.1002/pmic.200401148.
[24] Zhang Y Y,Li H X,Shu W B,Zhang C J,Ye Z B. RNA interference of a mitochondrial APX gene improves vitamin C accumulation in tomato fruit[J]. Scientia Horticulturae,2011,129(2):220-226. doi:10.1016/j.scienta.2011.03.025.
[25] Ma L C,Wang Y R,Liu W X,Liu Z P. Overexpression of an alfalfa GDP-mannose 3,5-epimerase gene enhances acid,drought and salt tolerance in transgenic Arabidopsis by increasing ascorbate accumulation[J].Biotechnology Letters,2014,36(11):2331-2341. doi: 10.1007/s10529-014-1598-y.
[26] Liu F H,Wang L,Gu L,Zhao W,Su H Y,Cheng X H. Higher transcription levels in ascorbic acid biosynthetic and recycling genes were associated with higher ascorbic acid accumulation in blueberry[J]. Food Chemistry,2015,188:399-405. doi:10.1016/j.foodchem.2015.05.036.
[27] 邹礼平.番茄抗坏血酸生物合成与代谢途径中相关酶基因的克隆与调控[D].武汉:华中农业大学,2005. doi:10.7666/d.y1428113.
Zou L P. Cloning and regulation of genes involved in tomato ascorbic acid biosynthesis and metabolism[D]. Wuhan:Huazhong Agricultural University,2005.
[28] Stevens R, Buret M, Duffé P, Garchery C, Baldet P, Rothan C, Causse M.Candidate genes and QTLs affecting fruit ascorbic acid content in three tomato populations[J]. Plant Physiology,2007,143(4):1943-1953. doi:10.1104/pp.106.091413.
[29] Laing W A,Bulley S,Wright M,Cooney J,Jensen D,Barraclough D,MacRae E. A highly specific L-galactose-1-phosphate phosphatase on the path to ascorbate biosynthesis.[J]. Proceedings of the National Academy of Sciences of the United States of America,2004,101(48):16976-16981. doi:10.1073/pnas.0407453101.
[30] Tabata K,Takaoka T,Esaka M.Gene expression of ascorbic acid-related enzymes in tobacco[J].Phytochemistry, 2002,61(6):631-635. doi:10.1016/S0031-9422(02)00367-9.
[31] 闫秋艳,董飞,段增强,李汛,王嫒华,汤英.土壤温度对不同施肥方式下辣椒生长及土壤理化性质的影响[J].华北农学报,2018,33(2):195-201. doi:10.7668/hbnxb.2018.02.027.
Yan Q Y,Dong F,Duan Z Q,Li X,Wang Y H,Tang Y. Effect of soil temperature on soil physical-chemical properties and pepper growth in different fertilizer treatments[J]. Acta Agriculturae Boreali-Sinica,2018,33(2):195-201.
[32] Bickmore W A,Sutherland H G E.Addressing protein localization within the nucleus[J].The European Molecular Biology Organization Journal,2002,21 (6) :1248-1254. doi: 10.1093/emboj/21.6.1248.
[33] 丛郁,杨顺瑛,宋志忠,郝东利,苏彦华.葡萄AMT基因家族生物信息学分析[J].中国农学通报,2011,27(25):193-199.
Cong Y,Yang S Y,Song Z Z,Hao D L,Su Y H. Bioinformatics analysis of AMT protein family in grape[J]. Chinese Agricultural Science Bulletin,2011,27(25):193-199.
[34] 刘炳臣,王茜,王跃进,张朝红.葡萄活性赤霉素合成关键基因VvGA3ox家族的克隆和表达分析[J].园艺学报,2016,43(12):2293-2303. doi: 10.16420/j.issn.0513-353x.2016-0294.
Liu B C,Wang Q,Wang Y J,Zhang C H.Cloning and expression analysis of the key genes family VvGA3ox in active gibberellin synthesis of grapevine[J]. Acta Horticulturae Sinica,2016,43(12):2293-2303.
[35] Tamura K,Dudley J,Nei M,Kumar S.MEGA4:Molecular evolutionary genetics analysis (MEGA) software version 4.0[J].Molecular Biology and Evolution,2007,24 (8) :1596-1599. doi: 10.1093/molbev/msm092.
[36] Chenna R,Sugawara H,Koike T,Lopez R,Gibsont T J,Higgins D G,Thompson J D.Multiple sequence alignment with the clustal series of programs[J].Nucleic Acids Research,2003,31 (13) :3497-3500. doi:10.1093/nar/gkg500.
[37] 王江英,张宁,司怀军,吴家和.棉花GDP-甘露糖-3′,5′-异构酶基因的克隆及其表达分析[J].华北农学报,2012,27(1):12-17.doi:10.3969/j.issn.1000-7091.2012.01.003.
Wang J Y,Zhang N,Si H J,Wu J H. Cloning and expression analysis of GhGME gene from Gossypium hirsuturm L.[J]. Acta Agriculturae Boreali-Sinica,2012,27(1):12-17.
[38] 李佼,余有本,周天山,肖瑶,柳洁,肖斌.茶树GDP-甘露糖-3′,5′-异构酶基因cDNA全长的克隆与表达[J].西北农林科技大学学报(自然科学版),2013,41(12):100-106.
Li J,Yu Y B,Zhou T S,Xiao Y,Liu J,Xiao B. Cloning and expression of GDP-Mannose-3′,5′-epimerase cDNA from Camellia sinensis[J]. Journal of Northwest A&F University(Natural Science Edition),2013,41(12):100-106.
[39] 刘军霞. 番茄抗坏血酸生物合成酶GME和MIOX的功能分析[D]. 武汉:华中农业大学,2009. doi:10.7666/d.y1812434.
Lui J X. Function analysis of GME and MIOX in ascorbic acid biosynthesis of tomato[D]. Wuhan:Huazhong Agricultural University,2009.
[40] Voxeur A,Gilbert L,Rihouey C,Driouich A,Rothan C,Baldet P,Lerouge P. Silencing of the GDP-D-mannose 3,5-epimerase affects the structure and cross-linking of the pectic polysaccharide rhamnogalacturonan Ⅱ and plant growth in tomato[J].The Journal of Biological Chemistry,2011,286(10):8014-8020.doi:10.1074/jbc.M110.198614.
[41] Major L L,Wolucka B A,Naismith J H.Structure and function of GDP-Mannose-3′,5′-epimerase:An enzyme which performs three chemical reactions at the same active site[J].J Am Chem Soc,2005,127 (51) :18309-18320. doi:10.1021/ja056490i.
[42] 王美珍,王学敏,王赞,高洪文,刘磊.柠条GDP-甘露糖-3′,5′-异构酶基因的克隆、序列分析及其植物表达载体的构建[J].中国草地学报,2009,31(5):75-83.
Wang M Z,Wang X M,Wang Z,Gao H W,Liu L. Cloning and sequence analysis of GDP-mannose-3′,5′-epimerase gene from Caragana korshinskii and construction of its plant expression vector[J]. Chinese Journal of Grassland,2009,31(5):75-83.
